Aplicação do pseudovírus Ha-CoV-2 para quantificação rápida de variantes do SARS-CoV-2 e anticorpos neutralizantes
In This Article
Summary
Este protocolo descreve a aplicação de um pseudovirus híbrido novo alphavirus-SARS-CoV-2 (Ha-CoV-2) como uma plataforma para a quantificação rápida da infecciosidade das variantes SARS-CoV-2 e sua sensibilidade aos anticorpos neutralizantes.
Abstract
A pandemia da doença 2019 do coronavirus (COVID-19) destacou a necessidade de ensaios rápidos para medir com precisão a infectividade das variantes emergentes do SARS-CoV-2 e a eficácia dos anticorpos neutralizantes induzidos pela vacina contra variantes virais. Esses ensaios são essenciais para a vigilância da pandemia e validação de vacinas e reforços específicos de variantes. Este manuscrito demonstra a aplicação de um pseudovirus alfavirus-SARS-CoV-2 híbrido novo (Ha-CoV-2) para a quantificação rápida da infectividade da variante SARS-CoV-2 e anticorpos neutralizantes vacina-induzidos às variantes virais. Ha-CoV-2 é uma partícula semelhante ao vírus SARS-CoV-2 que consiste em proteínas estruturais virais (S, M, N e E) e um genoma de RNA de expressão rápida derivado de um alfavírus, Semliki Forest Virus (SFV). O Ha-CoV-2 também contém proteína fluorescente verde (GFP) e genes repórteres de luciferase que permitem a quantificação rápida da infectividade viral. Como exemplo, a infectividade das variantes Delta do SARS-CoV-2 (B.1.617.2) e Omicron (B.1.1.529) são quantificadas, e suas sensibilidades a um anticorpo neutralizante (27VB) também são medidas. Esses exemplos demonstram o grande potencial do Ha-CoV-2 como uma plataforma robusta para a quantificação rápida de variantes do SARS-CoV-2 e sua suscetibilidade a anticorpos neutralizantes.
Introduction
Até maio de 2023, já foram mais de 766 milhões de casos de COVID-191. Apesar das campanhas mundiais de vacinação, o SARS-CoV-2 circula e infecta continuamente as pessoas, em grande parte devido ao surgimento de novas variantes como Delta (B.1.617.2) e Omicron (B.1.1.529), que impulsionam novas ondas de infecção 2,3,4. Dado que o SARS-CoV-2 está em constante evolução, é importante desenvolver ensaios rápidos que possam medir com precisão a infectividade de variantes emergentes e a eficácia dos anticorpos neutralizantes induzidos pela vacina contra essas variantes. Esses ensaios são essenciais para a vigilância da pandemia e para determinar a eficácia das vacinas e seus reforços específicos de variantes.
Devido à natureza altamente contagiosa do SARS-CoV-2, o Centro de Controle e Prevenção de Doenças (CDC) exige que o estudo do SARS-CoV-2 e suas variantes seja conduzido em instalações de nível de biossegurança (BSL) 3 5,6. Este requisito BSL-3 limita o uso de vírus vivos para quantificar a infectividade de variantes virais e seus anticorpos neutralizantes em pesquisas comuns e laboratórios clínicos. Além disso, os ensaios tradicionais de neutralização do SARS-CoV-2, tais como os ensaios baseados no efeito da placa ou do cytopathic usando vírus vivos competentes da replicação, são demorados e requerem longos períodos de incubação7. Vários pseudovírus SARS-CoV-2 pseudotipados com proteína spike (S) foram desenvolvidos para quantificar a eficácia de anticorpos neutralizantes 8,9,10,11,12. No SARS-CoV-2, a proteína S é a proteína principal que medeia a entrada viral13, e é o antígeno principal usado em vacinas SARS-CoV-2 9,10,14,15,16. Os virions pseudotipados da proteína S, como os do vírus da estomatite vesicular (VSV-G) ou lentivírus, têm sido utilizados para a quantificação de anticorpos neutralizantes 17,18,19. No entanto, o pseudovírus baseado em lentivírus normalmente requer 2 a 3 dias de infecção para quantificar os sinais do repórter. Sistemas pseudovírus baseados em VSV geralmente contêm vírus VSV residuais, que podem resultar em altas taxas de resultados falso-positivos e tipicamente requerem 24 h de infecção20.
Um novo sistema de pseudovírus SARS-CoV-2, o pseudovírus híbrido alphavirus-SARS-CoV-2 (Ha-CoV-2), foi recentemente desenvolvido por Hetrick et al12. O Ha-CoV-2 fornece uma nova ferramenta para a quantificação rápida da infectividade do vírus e da sensibilidade do vírus a anticorpos neutralizantes em laboratórios BSL-2 comuns. Estruturalmente, o Ha-CoV-2 se assemelha à partícula do virion do SARS-CoV-2, consistindo de proteínas estruturais do SARS-CoV-2, incluindo a proteína S (S), a membrana (M), o nucleocapsid (N) e o envelope (E), e não há nenhuma proteína estrutural de outros vírus. Além disso, a partícula Ha-CoV-2 contém um genoma de RNA de expressão rápida de um alfavírus para rápida expressão em células. Foi demonstrado que o Ha-CoV-2 mede rapidamente a atividade neutralizante de anticorpos no soro de indivíduos vacinados e convalescentes12. Como demonstrado por Hetrick et al., quando comparado com o pseudovírus SARS-CoV-2 baseado em lentivírus em um ensaio do curso do tempo, Ha-CoV-2 expressou o repórter de Luc tão cedo quanto 2-4 h pós-infecção, enquanto o pseudovirus do lentivirus expressou Luc após 24 h12. Além disso, a aplicação potencial de variantes do Ha-CoV-2 para quantificar anticorpos neutralizantes é demonstrada com o uso de um anticorpo neutralizante monoclonal padrão, o 27BV (ver Figura Suplementar 1)12. Este trabalho detalha o uso da plataforma Ha-CoV-2 para quantificação rápida da infectividade das variantes do SARS-CoV-2, usando as variantes Delta (B.1.617.2) e Omicron (B.1.1.529) como exemplos. Além disso, a aplicação potencial de variantes de Ha-CoV-2 para quantificar anticorpos neutralizantes é ainda demonstrada usando um anticorpo neutralizante monoclonal padrão, 27BV12.
Protocol
1. Montagem de vírus e partículas virais
- Vetores: Vetores de expressão de compra para SARS-CoV-2 M, E, ou N, bem como o SARS-CoV-2 S (Wild-type, Wt), Delta (B.1.617.2), e Omicron (B.1.1.529) comercialmente.
Observação : sequências de proteína dos vetores de expressão são fornecidas no arquivo suplementar 1. O fornecedor desses vetores de expressão também pode ser encontrado na Tabela de Materiais. - Células e cultura celular: Manter HEK293T células em meio de Eagle modificado (DMEM) de Dulbecco contendo 10% de soro fetal bovino (FBS) inativado pelo calor, 50 unidades/mL de penicilina e 50 μg/mL de estreptomicina.
NOTA: Todo o trabalho de cultura de células precisa ser feito em um gabinete de biossegurança de fluxo de ar laminar. A transfecção de polietilenoimina (PEI) normalmente requer 5-6 h de incubação. Os horários de início precisam ser considerados com cuidado antes. - Montagem de vírus e cotransfecção baseada em PEI: Montar partículas de Ha-CoV-2 por cotransfecção de células HEK293T. Células de sementes em uma placa de Petri de cultura celular de 10 cm (4-5 x 106 células por placa) em 10 mL de meio DMEM completo no dia anterior à cotransfecção. Para este estudo, três placas de Petri são usadas para semear células para a montagem de Ha-CoV-2 selvagem, Delta e Omicron, respectivamente.
- Incubar placas de Petri durante a noite em uma incubadora de CO2 a 37 °C. Verifique os pratos na manhã seguinte para garantir que as células sejam 80% confluentes. Remover o meio completo e substituí-lo por 9 mL de meio sem soro DMEM.
- Para cada prato, prepare uma mistura de cotransfecção com 2,5 μg de cada um dos vetores de expressão de proteína estrutural do SARS-CoV-2 (N, E, M), 10 μg de pAlphaPro-Luc-GFP-PreΨ (Genoma HaCoV2) e 2,5 μg do vetor de expressão da proteína S, variantes Delta ou Omicron S e 45 μL do reagente de transfecção baseado em PEI. Permitir que a mistura de cotransfecção forme complexos incubando por 13 min (não incubar por mais de 30 min).
- Após a incubação, adicione a mistura de cotransfecção a cada placa de Petri lentamente, gota a gota. Coloque os pratos dentro de uma incubadora de CO2 a 37 °C por 6 h. Após 6 h, remova o DMEM sem soro e substitua-o por meio DMEM completo. Vírus de colheita em 48-60 h pós-cotransfecção.
- Colheita e armazenamento do vírus: Colher partículas 48 h após a cotransfecção. Separar as células pipetando repetidamente sobre a superfície da monocamada e coletar células de cada placa, colocar em tubos de centrífuga de 15 mL e centrifugar a 400 x g por 5 min. Recolher o sobrenadante e passá-lo através de um filtro de 0,22 μM. Conservar o pseudovírus Ha-CoV-2 a -80 °C.
2. Ensaio de infectividade viral
- Células e cultura celular: Manter células HEK293T(ACE2/TMPRSS2) em meio de Eagle modificado (DMEM) de Dulbecco contendo 10% de SFB inativado pelo calor, 50 unidades/mL de penicilina e 50 μg/mL de estreptomicina.
- Semeadura de células HEK293T(ACE2/TMPRSS2): Um dia antes do ensaio de infectividade viral, sementes HEK293T(ACE2/TMPRSS2) em uma placa de 96 poços em 50 μL de meio DMEM completo. Para cada placa de 96 poços, semeie 2,5 x 104 células em cada poço, e um total de 2,5 x 106 células são necessárias para uma placa. Coloque a placa de 96 poços em uma incubadora de CO2 a 37 °C durante a noite.
- Infecção de HEK293T(ACE2/TMPRSS2): Use partículas variantes de Ha-CoV-2 para infectar células HEK293T(ACE2/TMPRSS2). Na manhã da infecção, remover 50 μL de DMEM da placa pré-semeada de 96 poços. Substitua o meio por 50 μL de Ha-CoV-2 Wild-type, Delta ou Omicron por 18 h a 37 °C.
NOTA: O protocolo pode ser interrompido aqui até que a infecção tenha atingido 18 h de incubação e a placa esteja pronta para ser analisada via Luciferase Assay. O sucesso da infecção é determinado pelo ensaio Luciferase resultante do gene Luciferase expresso nas células infectadas, portanto, quanto mais sinal é produzido, mais bem-sucedida é a infecção da variante Ha-CoV-2. - Ensaio de Luciferase: Após 18 h de incubação, adicionar 7,5 μL de tampão de lise celular diretamente a cada poço e misturar por agitação orbital por 2 min. Lise as células em tampão de lise por pelo menos 5 min à temperatura ambiente.
- Prepare a solução de ensaio de luciferase Firefly misturando a solução de D-luciferina com a solução de ensaio de luciferase Firefly numa proporção de 1:50. Para uma placa inteira de 96 poços, combinar 3 mL da solução de substrato de luciferase com 60 μL da solução de D-luciferina com 2940 μL da solução tampão de luciferase Firefly.
- Adicionar 25 μL da solução de ensaio de luciferase Firefly aos lisados celulares e misturar a placa por agitação orbital durante 1 min. Analise a atividade da luciferase usando um leitor comercial de microplacas de luciferase.
3. Extração de RNA de Ha-CoV-2 e PCR quantitativo de transcriptase reversa (RT-qPCR)
- Extração de RNA viral: Extraia o RNA viral do tipo Ha-CoV-2 Wild e das partículas variantes Delta e Omicron do Ha-CoV-2 usando um kit comercial de extração de RNA viral, seguindo as instruções do fabricante. Armazenar o RNA viral extraído a -80 °C ou usá-lo imediatamente para RT-qPCR.
- RT-qPCR: Realize RT-qPCR no RNA viral usando uma mistura mestre de uma etapa. Realizar a reação em uma máquina de PCR comercial. Observe que o alvo da amplificação é o RNA genômico do Ha-CoV-2. Use o DNA vetorial Ha-CoV-2 como padrão para criar uma curva padrão e calcular o número de cópias de RNA de cada variante.
4. Ensaio de anticorpos neutralizantes
- Semeadura de células HEK293T(ACE2/TMPRSS2): No dia anterior ao ensaio, semear células HEK293T(ACE2/TMPRSS2) em uma placa de 96 poços em 50 μL de meio DMEM completo. As células HEK293T(ACE2/TMPRSS2) são compradas comercialmente.
- Para a contagem de células, obter 20 μL de células de um frasco T75 contendo células HEK293T(ACE2/TMPRSS2) e misturar com 20 μL de solução de azul de tripano. Adicionar 20 μL desta mistura à câmara de contagem de células e contar o número de células por ml. Para semear uma placa de 96 poços para infecção, use 2,5 x 104 células por poço, e 2,5 x 106 células serão necessárias no total. Coloque a placa de 96 poços em uma incubadora de CO2 a 37 °C durante a noite.
- Ensaio de anticorpos neutralizantes: Em uma placa estéril de polipropileno de 96 poços, prepare o anticorpo neutralizante padrão 27BV e a mistura de Ha-CoV-2. Adicionar 8 μL de 27BV (45 mg/mL) à placa e realizar diluições seriadas do anticorpo com 6 μL de DMEM livre de soro.
NOTA: Certifique-se de trocar as pontas da pipeta entre as transferências de poço e certifique-se de que o meio livre de anticorpos e soro seja misturado completamente para produzir resultados precisos. - Ao anticorpo diluído em série, adicione 54 μL de partícula de Ha-CoV-2 e misture o vírus e o anticorpo. Pré-incubar partículas de Ha-CoV-2 com 27BV diluído em série por 1 h a 37 °C a 5% de CO2. Após 1 h de incubação, aplicar 50 μL da mistura de anticorpos e Ha-CoV-2 na placa de 96 poços contendo as células HEK293T(ACE2/TMPRSS2) (2,5 x 104 células por poço) semeadas no dia anterior.
- Para os controles, deixe pelo menos três poços contendo apenas as células HEK293T(ACE2/TMPRSS2). Adicionar 50 μL de meio completo a esses poços para servir como poços não infectados para o sinal de fundo das leituras do ensaio de luciferase.
NOTA: O protocolo pode ser interrompido aqui até que a infecção tenha atingido 18 h de incubação a 37 °C e, em seguida, a placa esteja pronta para ser analisada via ensaio de luciferase. - Ensaio de Luciferase: Após 18 h de incubação, adicionar 7,5 μL de tampão de lise diretamente a cada poço e misturar por agitação orbital por 2 min. Lise as células em tampão de lise por pelo menos 5 min à temperatura ambiente.
- Prepare a solução de ensaio de luciferase Firefly misturando a solução de D-luciferina com a solução de ensaio de luciferase Firefly numa proporção de 1:50. Para uma placa inteira de 96 poços, combinar 3 mL da solução de substrato de luciferase com 60 μL da solução de D-luciferina com 2940 μL da solução tampão de luciferase de vagalume.
- Adicionar 25 μL da solução de ensaio de luciferase Firefly aos lisados celulares e misturar a placa por agitação orbital durante 1 min. Analise a atividade da luciferase usando um leitor comercial de microplacas de luciferase.
5. Quantificação e análise estatística
- Coleta de dados: Realizar dosagens de infecção e luciferase em triplicata conforme indicado (Figura 1). Quantificar a expressão da luciferase com leituras de ensaio de luciferase. A média é o valor médio das três leituras do ensaio de luciferase. As leituras do sinal de fundo de poços não infectados são subtraídas desse valor médio. O desvio padrão (DP) é determinado a partir do valor médio das leituras do ensaio de luciferase.
- Análise dos dados: Plotar a atividade de neutralização de anticorpos e calcular os valores de ID50 (50% de dosagem de inibição) usando um software gráfico comercial. O valor de ID50 é definido como as diluições inibitórias de anticorpos nas quais se obtém uma redução de 50% na infecção de células HEK293T(ACE2/TMPRSS2) (com base nas leituras do ensaio de luciferase).
Representative Results
As partículas de Ha-CoV-2 foram montadas usando cinco vetores de DNA diferentes que expressam o genoma do RNA do Ha-CoV-2 e as proteínas estruturais (M, N, E e S) do SARS-CoV-2 em células HEK293T. O vetor da proteína S varia dependendo da variante S. A proteína S da cepa original Ha-CoV-2 de Wuhan (Wild-type, Wt) foi usada como controle positivo, e foi montada junto com a proteína S de cada uma das outras duas variantes: a Delta (B.1.617.2) ou a Omicron (B.1.1.529). Os mesmos M, N, E foram usados em todas as variantes. Ha-CoV-2(Wt) e partículas variantes foram coletadas 48 h após a cotransfecção e usadas para infectar células HEK293T(ACE2/TMPRSS2). A infectividade foi medida pela expressão de luciferase 18 h após a infecção. Nesse sistema, níveis mais elevados de expressão do sinal da luciferase refletem uma maior infecção de células por Ha-CoV-2. O sinal da luciferase foi normalizado com cópias de RNA genômico por RT-qPCR para cada variante. Como mostrado na Figura 1, a variante Ha-CoV-2 Omicron gerou sinal 4 a 10 vezes maior do que o Ha-CoV-2 original (Wt), sugerindo uma maior infectividade.
Além disso, a capacidade do 27BV de neutralizar as variantes HaCoV-2(Wt), Delta e Omicron foi quantificada. 27BV é um anticorpo monoclonal do coelho que foi desenvolvido contra o domínio RBD da proteína SARS-COV-2 S1. Para os ensaios de neutralização, diluições seriadas de 27BV foram realizadas em uma placa de 96 poços, pré-incubada com Ha-CoV-2 e, em seguida, adicionadas às células-alvo de HEK293T(ACE2/TMPRSS2). Os resultados demonstraram que o 27BV apresentou atividade neutralizante contra todas as variantes testadas (Figura 2). Curiosamente, o ID50 de 27VB para Omicron foi aproximadamente 10 vezes menos potente do que o ID50 para Ha-CoV-2(WT) e Ha-CoV-2(Delta; Gráfico 2). Esses resultados demonstram que a plataforma Ha-COV-2 pode ser usada como um método rápido para quantificar anticorpos neutralizantes induzidos por vacinas em variantes emergentes.
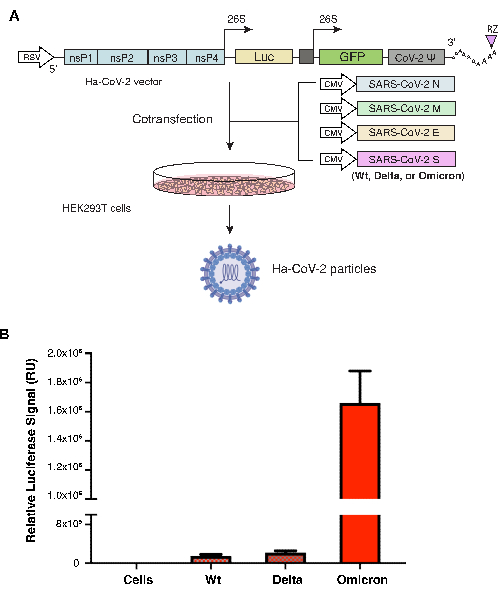
Gráfico 1. Montagem e quantificação de variantes do Ha-CoV-2. (A) Ilustração da montagem do Ha-CoV-2 e partículas variantes. Os vetores que expressam o genoma do repórter Ha-CoV-2 e as proteínas estruturais (M, S, N e E) são cotransfectados em células HEK293T. As partículas foram colhidas 48 h após a cotransfecção (a imagem da partícula do virion e as células HEK293T foram criadas com Biorender.com). (B) Quantificação da infectividade das variantes do Ha-CoV-2. A infectividade relativa das duas variantes (Delta e Omicron) é quantificada e normalizada usando cópias de RNA genômico de variantes individuais do Ha-CoV-2 (Luc). O tipo selvagem é o Ha-COV-2 (Wt), que é usado como um controle para comparação. Os ensaios de infecção e luciferase foram realizados 3x. UR, unidade relativa. A média e o desvio padrão (DP) são mostrados. Clique aqui para ver uma versão maior desta figura.
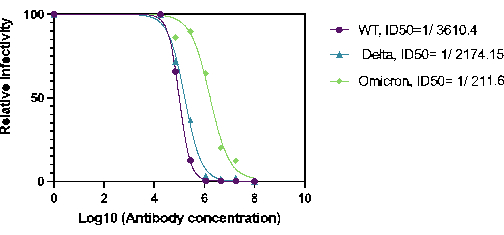
Gráfico 2. Quantificação da atividade de neutralização do 27BV contra variantes do Ha-CoV-2(Luc) A atividade de neutralização do 27BV foi analisada 18 h após a infecção de células HEK293T(ACE2/TMPRSS2). O ID50 foi calculado usando a taxa de infecção relativa (atividade da luciferase) versus a concentração de 27BV. Clique aqui para ver uma versão maior desta figura.
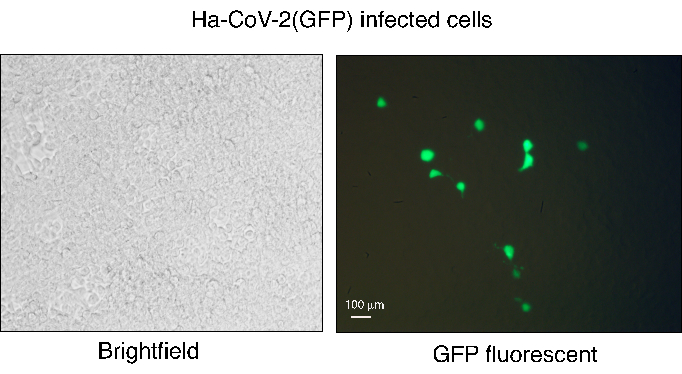
Gráfico 3. Infecção de HEK293T(ACE2/TMPRSS2) com Ha-CoV-2(GFP). Partículas de Ha-CoV-2 (GFP) foram montadas e usadas para infectar células HEK293T(ACE2/TMPRSS2). A expressão de GFP foi observada 48 h após a infecção usando microscopia fluorescente. O campo branco das células infectadas é mostrado à esquerda, e a imagem da GFP é mostrada à direita. A barra branca representa 100 μm. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1. Resumo Gráfico. A estrutura e aplicação do pseudovírus Ha-CoV-2. Imagem criada com Biorender.com. Clique aqui para baixar este arquivo.
Arquivo Suplementar 1. Sequências de Proteínas. Lista das sequências das proteínas S, M, N e E do SARS-CoV-2. As sequências da proteína S também incluem as variantes do SARS-CoV-2 Omicron (B.1.1.529) e Delta (B.1.617.2). Clique aqui para baixar este arquivo.
Discussion
A plataforma Ha-CoV-2 fornece um fluxo de trabalho rápido, robusto e simples para quantificar variantes virais e neutralizar anticorpos. No entanto, existem algumas etapas críticas que precisam de atenção. A produção do pseudovírus Ha-CoV-2 deve ser realizada utilizando células HEK293T com alta viabilidade. A eficiência da cotransfecção pode ser monitorada 24 h após a transfecção usando o gene repórter GFP do genoma do Ha-CoV-2. O genoma do Ha-CoV-2 pode conter dois repórteres (GFP e Luc), e a GFP pode ser expressa durante a cotransfecção e após a infecção por Ha-CoV-2 de células-alvo12. As células GFP+ da infecção estão normalmente em uma porcentagem baixa (1% a 5%), mas cada célula infectada expressa fortes sinais de GFP (Figura 3). Essa baixa porcentagem de GFP pode limitar o uso de GFP como uma leitura robusta para quantificar a neutralização de anticorpos, em comparação com o repórter Luc, que quantifica toda a população de células infectadas.
Ao realizar o ensaio de neutralização, é essencial trocar as pontas da pipeta entre as transferências de poço e garantir que o meio livre de anticorpos e soro sejam misturados completamente para produzir resultados precisos. Além disso, ao conduzir o protocolo de ensaio de luciferase, as células devem ser totalmente lisadas por pelo menos 3 min para garantir a lise completa das células e a liberação da enzima luciferase. Isso garantirá a precisão do ensaio. Além disso, uma vez que a solução de ensaio de luciferase Firefly é adicionada às placas ópticas de 96 poços de paredes brancas, a placa deve ser analisada dentro de 10 min, pois a emissão inicial de luz é alta, mas diminui com o tempo à medida que o ATP é esgotado21.
À medida que mais variantes do SARS-CoV-2 continuam a evoluir, há uma necessidade maior de plataformas como o Ha-CoV-2 para rastrear rapidamente a infectividade da variante e a sensibilidade da variante aos anticorpos neutralizantes induzidos pela vacina. A plataforma Ha-CoV-2 oferece velocidade mais rápida, maior relação sinal-ruído e um protocolo simples em comparação com os ensaios de neutralização baseados em pseudovírus existentes 8,9,10,11. A plataforma Ha-CoV-2 também oferece a vantagem de poder ser usada em laboratórios BSL-2 e não requer o uso de instalações BSL-3. Isso permite que a pesquisa do SARS-CoV-2 seja realizada em laboratórios clínicos e de pesquisa comuns. Além disso, a plataforma Ha-CoV-2 produz resultados rápidos em comparação com outros sistemas. Por exemplo, o estudo de anticorpos neutralizantes contra o vírus SARS-CoV-2 infeccioso frequentemente faz uso do teste de neutralização de redução de placa (PRINT)22. Embora o PRINT produza resultados confiáveis, a contagem manual de unidades formadoras de placas (UFP) é lenta e requer de 3 a 5 dias para obter os resultados23,24. Outros sistemas de pseudotipos, como o lentivírus-pseudovírus, necessitam de 24-72 h para produzir um sinal de repórter detectável12. Em comparação, o ensaio de neutralização do Ha-CoV-2 pode gerar resultados em 18 h. O Ha-CoV-2 fornece uma ferramenta conveniente para triagem rápida e quantificação de variantes virais e anticorpos neutralizantes para monitoramento de pandemias.
O monitoramento da infectividade do SARS-CoV-2 é essencial à medida que mais variantes de preocupação (VOCs) continuam a surgir. O Ha-CoV-2 oferece a vantagem de determinar rapidamente a infectividade dos VOCs. Estudos anteriores usaram modelagem baseada em inteligência artificial (IA) para analisar quantitativamente a infectividade da subvariante Omicron e de outras variantes do SARS-CoV-2, como a variante Delta25. Esses estudos mostraram que a variante Omicron é mais contagiosa do que o vírus original e mais propensa a escapar de anticorpos neutralizantes25. Nesses estudos, utilizando Ha-CoV-2, fenótipos semelhantes foram observados. Além disso, nos ensaios de neutralização de anticorpos, a variante Omicron tem dez vezes menos probabilidade de ser neutralizada por 27BV do que as cepas de Wuhan e Delta. Esses resultados também são consistentes com a maior transmissibilidade relatada da variante Omicron, que tem pelo menos 15 mutações em seu domínio de ligação ao receptor (RBD), provavelmente aumentando a afinidade de ligação viral ao receptor ACE2 para maior transmissibilidade e maior escape imunológico26.
Disclosures
Uma patente foi registrada pela Universidade George Mason e licenciada para a Virongy Biosciences Inc. Y.W. é fundador e membro do conselho consultivo da Virongy Biosciences. B. H é atualmente o CEO e Diretor Científico da Virongy Biosciences. Os autores não têm outros conflitos de interesse.
Acknowledgements
Este trabalho foi apoiado pelo fundo de pesquisa interno da George Mason University.
Materials
| Name | Company | Catalog Number | Comments |
| 27VB1 20 µg SARS-CoV-2 Standard Neutralizing Antibody | Virongy Biosciences | 27VBI-01 | |
| 500 mL - US Origin FBS | Neuromics | FBS001 | |
| AB Mixing Plate: Olympus 96-Well PCR Plate, Non-Skirted UltraThin Wall, Natural, 25 Plates/Unit | Genesee Scientific | Cat# 24-300 | |
| Allegra 6R Centrifuge | Beckman Coulter | 2043-30-1158 | |
| DMEM (1x) | ThermoFisher | 11995-073 | |
| GenClone 25-209, TC Treated Flasks, 250ml, Vent Growth Area: 75.0cm2, 5 per Sleeve, 100 Flasks/Unit | Genesee Scientfic | 25-209 | |
| GlowMax Discover Microplate reader | Promega | GM3000 | |
| Ha-CoV-2 E Vector | Virongy Biosciences | pCoV2_E | |
| Ha-CoV-2 M Vector | Virongy Biosciences | pCoV2_M | |
| Ha-CoV-2 N Vector | Virongy Biosciences | pCoV2_N | |
| Ha-CoV-2 WT S Vector | Virongy Biosciences | pCoV2_WT S | |
| Hek293T cells | ATCC | CRL-3214 | |
| Illumination Firefly Luciferase Enhanced Assay Kit 1000 assays | Gold Bio | I-930-1000 | |
| Infection Plate: 96-Well Tissue Culture Plate, Greiner Bio-One (With Lid, μClear White Flat Round, Chimney) | VWR | Cat# 82050-758 | |
| pAlphaPro-Luc-GFP-PreΨ (Ha-CoV-2 Genome) Vector | In house | ||
| PEI-based Transfection Reagent | Virongy Biosciences | Transfectin | |
| Penicillin-Streptomycin-Glutamine (100X) | Invitrogen | 10378016 | |
| Polyethylenimine, branched | Millipore Sigma | 408727-100ML | |
| QuantStudio 7 Pro Real-Time PCR System | ThermoFisher | A43163 | |
| Ready to use (HEK293T)(ACE2/TMPRSS2) Cells | Virongy Biosciences | Ready-To-Use-Cells | |
| SARS-CoV-2 S Omicron (B.1.1.529) Vector | Virongy Biosciences | pCoV2-B.1.1.529 | |
| SARS-CoV-2 S Delta (B.1.617.2) Vector | Virongy Biosciences | pCoV2- B.1.617.2 | |
| Syringe Filters, PES, 0.22µm | Genesee Scientfic | 25-244 | |
| TaqMan Fast Virus 1-Step Master Mix | ThermoFisher | 4444432 | |
| Trypan Blue Solution, 0.4% | ThermoFisher | 15250061 |
References
- World Health Organization. . Weekly epidemiological update on COVID-19. 143, 1 (2023).
- Dhama, K., et al. Global emerging Omicron variant of SARS-CoV-2: Impacts, challenges and strategies. Journal of Infection and Public Health. 16 (1), 4-14 (2023).
- Dhama, K., et al. Coronavirus Disease 2019-COVID-19. Clinical Microbiology Reviews. 33 (4), e00028-e00020 (2020).
- El-Shabasy, R. M., et al. Three waves changes, new variant strains, and vaccination effect against COVID-19 pandemic. International Journal of Biological Macromolecules. 204, 161-168 (2022).
- CDC. . Interim laboratory biosafety guidelines for handling and processing specimens associated with coronavirus disease 2019 (COVID-19). , (2021).
- Kaufer, A., Theis, T., Lau, K., Gray, J., Rawlinson, W. Laboratory biosafety measures involving SARS-CoV-2 and the classification as a Risk Group 3 biological agent. Pathology. 52 (7), 790-795 (2020).
- Vanderheiden, A., et al. Development of a Rapid Focus Reduction Neutralization Test Assay for Measuring SARS-CoV-2 Neutralizing Antibodies. Current Protocols in Immunology. 131 (1), e116 (2020).
- Dieterle, M. E., et al. A Replication-Competent Vesicular Stomatitis Virus for Studies of SARS-CoV-2 Spike-Mediated Cell Entry and Its Inhibition. Cell Host & Microbe. 28 (3), 486-496 (2020).
- Nie, J., et al. Quantification of SARS-CoV-2 neutralizing antibody by a pseudotyped virus-based assay. Nature Protocols. 15 (11), 3699-3715 (2020).
- Nie, J., et al. Establishment and validation of a pseudovirus neutralization assay for SARS-CoV-2. Emerging Microbes & Infections. 9 (1), 680-686 (2020).
- Yang, R., et al. Development and effectiveness of pseudotyped SARS-CoV-2 system as determined by neutralizing efficiency and entry inhibition test in vitro. Biosafety and Health. 2 (4), 226-231 (2020).
- Hetrick, B., et al. Development of a hybrid alphavirus-SARS-CoV-2 pseudovirion for rapid quantification of neutralization antibodies and antiviral drugs. Cell reports methods. 2 (3), 100181 (2022).
- Jackson, C., Farzan, M., Chen, B., Choe, H. Mechanisms of SARS-CoV-2 entry into cells. Nature Reviews. Molecular Cell Biology. 23 (1), 3-20 (2022).
- Jackson, L. A., et al. An mRNA Vaccine against SARS-CoV-2 - Preliminary Report. The New England Journal of Medicine. 383 (20), 1920-1931 (2020).
- Polack, F. P., et al. Safety and Efficacy of the BNT162b2 mRNA Covid-19 Vaccine. The New England Journal of Medicine. 383 (27), 2603-2615 (2020).
- Li, F. Structure, function, and evolution of coronavirus spike proteins. Annual Review of Virology. 3 (1), 237-261 (2016).
- Donofrio, G. A Simplified SARS-CoV-2 Pseudovirus Neutralization Assay. Vaccines. 9 (4), 389 (2021).
- Crawford, K. H. D., et al. Protocol and Reagents for Pseudotyping Lentiviral Particles with SARS-CoV-2 Spike Protein for Neutralization Assays. Viruses. 12 (5), 513 (2020).
- Zettl, F., et al. Rapid Quantification of SARS-CoV-2-Neutralizing Antibodies Using Propagation-Defective Vesicular Stomatitis Virus Pseudotypes. Vaccines. 8 (3), 386 (2020).
- Li, Q., Liu, Q., Huang, W., Li, X., Wang, Y. Current status on the development of pseudoviruses for enveloped viruses. Reviews in Medical Virology. 28 (1), e1963 (2018).
- Lundin, A. Optimization of the firefly luciferase reaction for analytical purposes. Advances in Biochemical Engineering/Biotechnology. 145, 31-62 (2014).
- Deshpande, G. R., et al. Neutralizing antibody responses to SARS-CoV-2 in COVID-19 patients. The Indian Journal of Medical Research. 152 (1 & 2), 82-87 (2020).
- Chen, C., et al. Research progress in methods for detecting neutralizing antibodies against SARS-CoV-2. Analytical Biochemistry. 673, 115199 (2023).
- Manischewitz, J., et al. Development of a novel vaccinia-neutralization assay based on reporter-gene expression. The Journal of Infectious Diseases. 188 (3), 440-448 (2003).
- Chen, J., Wang, R., Gilby, N., Wei, G. Omicron Variant (B.1.1.529): Infectivity, Vaccine Breakthrough, and Antibody Resistance. Journal of Chemical Information and Modeling. 62 (2), 412-422 (2022).
- Saxena, S. K., et al. Characterization of the novel SARS-CoV-2 Omicron (B.1.1.529) variant of concern and its global perspective. Journal of Medical Virology. 94 (4), 1738-1744 (2022).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved