Tomografia tridimensional de órgãos inteiros de alta resolução de infecções microbianas
In This Article
Summary
Aqui, descrevemos um procedimento que permite a detecção de bactérias patogênicas em todo o órgão durante a infecção e a quantificação das atividades do repórter fluorescente.
Abstract
A maioria das infecções ocorre dentro de tecidos tridimensionais do hospedeiro com anatomia intrincada e fisiologia do hospedeiro localmente variável. O posicionamento das células do patógeno dentro desse ambiente diversificado afeta significativamente seus níveis de estresse, respostas, destino e contribuição para a progressão geral da doença e falha do tratamento. No entanto, devido às dificuldades técnicas em localizar células patogênicas de tamanho μm dentro de órgãos hospedeiros de tamanho cm, essa área de pesquisa tem sido relativamente inexplorada. Aqui, apresentamos um método para enfrentar esse desafio. Empregamos tomografia serial de dois fótons e análise de imagem aprimorada por IA para localizar células individuais de Salmonella em todo o baço, lobos hepáticos e linfonodos inteiros de camundongos infectados. Usando repórteres fluorescentes e administração de anticorpos in vivo , a taxa de replicação de células únicas de Salmonella , sua interação local com células imunes específicas e as respostas bacterianas aos antibióticos podem ser determinadas. Essas metodologias abrem caminhos para um exame abrangente de infecções, sua prevenção e tratamento dentro do contexto do tecido tridimensional.
Introduction
As infecções ocorrem em tecidos com anatomia complexa e fisiologia compartimentada. Os diversos microambientes que coexistem no tecido infectado podem determinar o destino dos subconjuntos locais de patógenos e suas contribuições para o resultado geral da doença 1,2,3. No entanto, o mapeamento 3D abrangente de patógenos microbianos em tecidos de tamanho cm continua sendo um desafio4. A imagem do cérebro e de outros órgãos é um campo de pesquisa altamente ativo com estratégias experimentais em constante aprimoramento5, mas muitos métodos ainda carecem da resolução sub-μm que seria necessária para identificar patógenos bacterianos de tamanho μm com confiança. Em contraste, a tomografia serial de dois fótons (STP)6 permite imagens automatizadas multicoloridas e livres de deformação de tecidos inteiros com resolução no plano sub-μm, produzindo conjuntos de dados volumétricos completos. Este método combina seccionamento físico repetido do tecido usando um vibratomo com imagens intermitentes de dois fótons das faces do bloco emergente com luz infravermelha. A tomografia STP tem sido amplamente utilizada para mapear axônios finos no cérebro para estabelecer mapas de conectividade 7,8,9,10.
A tomografia STP também permite o mapeamento 3D de células microbianas individuais do patógeno (Salmonella, Toxoplasma) em todos os tecidos infectados11,12 usando um tomógrafo. A geração de segundo harmônico revela bainhas de colágeno ao redor das artérias e em bandas fibrosas, como as trabéculas do baço, fornecendo contexto anatômico. Anticorpos fluorescentes injetados in vivo podem ser usados para corar células hospedeiras para revelar interações entre células patogênicas individuais e células imunes infiltradas, como neutrófilos. Aqui, é descrito o pipeline envolvendo processamento do tecido, imagem, costura de ladrilhos de imagem com correção de iluminação, empilhamento de imagens em três dimensões e segmentação usando ferramentas de aprendizado de máquina. Este pipeline produz posições 3D de patógenos individuais, células e microcolônias dentro de seu contexto hospedeiro. Contar o número de células individuais dentro das microcolônias permanece difícil devido aos limites de resolução, mas esses números podem ser estimados com base no brilho integrado da microcolônia. O pipeline pode ser prontamente adaptado a outros modelos de infecção se patógenos recombinantes que expressam GFP ou YFP estiverem disponíveis.
Protocol
Todos os experimentos com animais descritos aqui foram aprovados pelas autoridades (licença 2239, Kantonales Veterinäramt Basel) e seguem as diretrizes locais (Tierschutz-Verordnung, Basel) e a lei suíça de proteção animal (Tierschutz-Gesetz).
1. Preparação e armazenamento de tecidos infectados
- Use um modelo de infecção de sua escolha. Aqui, camundongos de 10 a 16 semanas de idade de ambos os sexos são infectados com ~ 1.000 unidades formadoras de colônias (UFC) por injeção intravenosa ou 5 x 107 UFC por administração intragástrica com uma agulha de ponta redonda. As cepas de camundongos adequadas incluem BALB/c e C57BL/6.
NOTA: A mesma abordagem pode ser facilmente adaptada para outras espécies hospedeiras. - Certifique-se de que os patógenos expressem proteínas fluorescentes para permitir a identificação em tecidos não corados. Patógenos que expressam GFP.mut213, YPet14, TIMERbac15 ou mWasabi16 podem ser visualizados usando excitação de 940 nm17. Salmonella expressando mCherry18 em níveis prontamente detectáveis por citometriade fluxo 19 pode ser visualizada usando excitação de 800 nm, mas tem baixas relações sinal-fundo no baço e fígado infectados, que mostram autofluorescência substancial.
- Certifique-se de que todas as proteínas fluorescentes retenham >70% de sua intensidade de fluorescência após fixação com paraformaldeído em pH neutro com base na quantificação com citometria de fluxo. Aqui, são usados tecidos de camundongos infectados, ou seja, baço, fígado, gânglios linfáticos e adesivos de Peyer.
- Corar marcadores de superfície da célula hospedeira in vivo injetando anticorpos marcados com ficoeritrina (PE) 10 minutos antes da perfusão. Para corar neutrófilos, injete por via intravenosa 4 μg anti-Ly-6G-PE em 100 μL de PBS. Para corar macrófagos da zona marginal no baço, injete 4 μg de anti-CD169-PE em 100 μL de PBS (ver Tabela de Materiais).
- Fixar os tecidos infectados por meio de perfusão transcárdica padrão20 com 15 mL de tampão fosfato 50 mM gelado (PB; 10 mM NaH2PO4, 40 mMNa2HPO4, pH 7,4), seguido de 35 mL de paraformaldeído a 4% (PFA; fixador) em PB (Figura 1A).
- Em resumo, administrar anestesia profunda ao animal infectado por injeção intraperitoneal de 100 mg/kg de cetamina e 16 mg/kg de xilazina. Garanta a anestesia adequada, confirmando a perda do reflexo de pinça do dedo do pé.
- Desinfete o pelo com etanol a 70%, seque-o com uma toalha de papel estéril e abra cirurgicamente o peito com uma pinça e uma tesoura. Coloque uma agulha estéril no ventrículo esquerdo do coração, abra o átrio direito com uma tesoura estéril e passe o primeiro tampão e depois o fixador pela agulha, pelo ventrículo esquerdo e por todo o sistema vascular usando uma bomba. Isso corrige todos os tecidos de forma rápida e uniforme e causa eutanásia.
- Colete o tecido após a perfusão, incube-o em volume 20x de paraformaldeído frio a 4% em tampão PB e coloque-o em um agitador a 4 ° C durante a noite (Figura 1B). A imagem é possível para tecidos na faixa de tamanho de 0,05 a 2 cm3. Para tecidos maiores, corte em pedaços adequados.
- No dia seguinte, lave o tecido 3x por 10 min cada com tampão PB com agitação a 40 rpm em temperatura ambiente para remover o PFA.
NOTA: Devido à toxicidade do PFA, essas etapas devem ser realizadas dentro de uma capela de exaustão. Descarte os resíduos de PFA de acordo com as diretrizes institucionais. - Remova qualquer excesso de tampão PB e adicione 20x o volume de tampão de armazenamento crioprotetor21 (876 mM de sacarose, 0,25 mM de polivinilpirrolidona, 40% (v / v) de etilenoglicol, dissolvido em tampão PB 50 mM) à amostra com agitação a 40 rpm a 4 ° C por 6-8 h ou até que os tecidos afundem. Incubar amostras a -20 °C por pelo menos 3 dias para garantir a redução efetiva da autofluorescência do tecido11. As amostras podem ser usadas por até 5 anos quando armazenadas a -20 °C.
NOTA: O afundamento do tecido indica a penetração adequada do tampão de armazenamento no tecido.
2. Incorporação de amostras
- Incorpore o tecido em um bloco de agarose para um corte suave com um vibratome. Pré-ative a agarose quimicamente por oxidação com periodato e depois reduza com borohidreto para formar reticulação com o tecido para melhorar a estabilidade durante o processo de corte. Execute as etapas descritas abaixo.
- Prepare agarose oxidada a 4% pesando 2,25 g de agarose e adicione 0,21 g de NaIO4. Adicione 100 mL de PB à agarose e NaIO4. Agitar à temperatura ambiente num exaustor durante 2-3 h com proteção contra a luz.
NOTA: Etapa crítica: Não exceda a agitação por mais de 3 h ou a agarose polimerizará mal. - Recuperar a agarose por filtração por membrana num filtro de vácuo normalizado com membrana de 0,2 μm. Remova o NaIO4 restante lavando 3x com 50 mL de tampão PB. Ressuspenda a agarose oxidada em 50 mL de tampão PB.
NOTA: Não derreta a agarose durante esta fase. A agarose pode ser armazenada a 4 °C por até 2-3 semanas protegida da luz. - Prepare a solução de borohidreto adicionando 19 g de bórax e 3 g de ácido bórico a 1 L de água. Mexa até dissolver e ajuste o pH para 9-9,5 com 1 M NaOH. Numa placa de agitação numa hotte, aquecer 100 ml de tampão borato a 40 °C. Adicione 0,2 g de NaBH4 e mexa por 15-30 min enquanto protege da luz.
NOTA: A solução de borohidreto pode ser armazenada por várias semanas em temperatura ambiente no escuro. Etapa crítica: Ao adicionar NaBH4 ao tampão de borato, o gás será formado, portanto, conduza esta etapa dentro de uma capela de exaustão. Não execute esta etapa em um recipiente hermeticamente fechado, pois isso pode causar uma explosão. - Retire o tecido do freezer após incubação suficiente para redução da autofluorescência (≥ 3 dias), remova o excesso de tampão de armazenamento crioprotetor e lave o tecido 3x por 10 min cada com tampão PB com agitação a 40 rpm à temperatura ambiente (RT).
- Derreta aproximadamente 20 mL de suspensão de agarose oxidada (preparada na etapa 1) em um micro-ondas (700 W por ~ 30 s), deixe esfriar o suficiente para manusear (~ 45 ° C) e despeje em um molde de plástico.
NOTA: Etapa crítica: Não adicione a agarose diretamente após aquecê-la no micro-ondas na amostra, isso pode levar à destruição do tecido. - Use uma pinça para pegar suavemente o tecido e insira-o rapidamente no centro inferior da agarose. Incube o molde em RT por 15 min para permitir que a agarose polimerize.
- Após a polimerização da agarose, abra o molde de plástico com um bisturi dos quatro cantos e pegue cuidadosamente o cubo de agarose com os dedos enluvados.
- Mergulhar a amostra em solução de borohidreto (preparada no passo 2) durante 2-3 h a 4 °C para iniciar a reticulação da agarose oxidada e do tecido. Isso evita que o tecido se desprenda durante o seccionamento.
NOTA: Etapa crítica: A amostra imersa em borohidreto deve ser incubada protegida da luz a 4 °C. - Lave o cubo de agarose com tampão PB 2x por 10 min. Vire o cubo de agarose de modo que o tecido fique na parte superior e prenda o bloco a uma lâmina magnética (lâmina de microscópio com dois ímãs presos na parte de trás) usando algumas gotas de adesivo instantâneo. Leva de 10 a 20 minutos para a cola solidificar, então coloque um lenço de papel com gotas de tampão PB em cima da agarose, garantindo que a amostra permaneça adequadamente hidratada (Figura 1C).
3. Preparação do micrótomo e preparação do cenário
- Coloque a lâmina magnética no metal no centro da caixa de amostras do tomógrafo.
- Encha a caixa com tampão PB e posicione-a cuidadosamente no estágio e aperte o parafuso à esquerda para prender a caixa de amostra no estágio do tomógrafo.
NOTA: Não aperte demais os parafusos, pois isso danificará a caixa de plástico. - Coloque a lâmina no micrótomo e mude a posição da amostra cuidadosamente perto e embaixo do micrótomo. A superfície superior da agarose deve estar no mesmo nível da lâmina do micrótomo.
NOTA: Posicioná-lo muito baixo desperdiçará tempo de corte, enquanto posicioná-lo muito alto removerá um grande pedaço do bloco no primeiro corte. - Mova o palco para posicionar o cubo de agarose no centro da objetiva e clique repetidamente no botão Fatiar no software do tomógrafo até obter uma fatia intacta de todo o cubo de agarose.
4. Alocação da superfície e aquisição de imagens 2D
- Centralize a lente objetiva na amostra de tecido nas direções x e y.
- Abra o software do laser e ligue o laser no software. Ao ouvir um som de tique-taque, ligue o interruptor do laser e ajuste o comprimento de onda para 800 nm.
NOTA: A fonte de laser precisa ser aquecida por pelo menos 30 min, idealmente 1 h, antes da imagem. - Feche as portas do gabinete do microscópio, desligue todas as fontes de luz da sala, ligue os PMTs e ajuste a tensão para 750 V.
NOTA: Etapa crítica: Os PMTs são muito sensíveis à luz, mantenha-os desligados, a menos que as portas do armário estejam fechadas e todas as fontes de luz da sala estejam desligadas. - Ajuste as configurações do protocolo do software do tomógrafo conforme descrito e defina o obturador do microscópio para automático e a tensão para 20 e 3 para V1 e V2, respectivamente.
- Clique no botão 2D no software de tomógrafo para capturar uma foto da amostra.
- Assim que uma imagem com autofluorescência visível aparecer, ajuste o z-piezo para definir o plano focal na superfície do cubo de agarose. A primeira varredura será de 20 μm abaixo da superfície para adquirir uma imagem estável da amostra.
- Se nenhum fundo de autofluorescência estiver visível, aumente o contraste até que um sinal apareça. Pode ocorrer que não haja sinal visível (ou seja, por causa da agarose na parte superior do tecido). Neste caso, ajuste o eixo z usando o dispositivo z-piezo até atingir a superfície da agarose. O movimento manual da lente pode ser necessário se a posição desejada estiver fora do alcance do dispositivo z-piezo.
- Corte várias fatias de 50 μm depois de encontrar a superfície da amostra e adquira imagens 2D após cada corte para garantir que o plano focal esteja definido corretamente na superfície da face do bloco.
5. Encontrar as bordas das amostras e ajustar o laser para o ponto de partida
- Com base nas coordenadas xy da área de varredura a laser (4 coordenadas de canto), confine a área de imagem ao tecido com imagens mínimas do bloco de agarose circundante.
- Desligue os PMTs, defina o comprimento de onda do laser para 800 nm, mude o obturador do laser para aberto. A posição da luz laser que entra no bloco de agarose é visível como uma mancha vermelha de luz espalhada (Figura 2A, B).
Etapa crítica: Tenha especial cuidado com o laser de classe 4. Pode danificar rapidamente os tecidos e olhos humanos. O pessoal necessita de formação especial para operar este instrumento em segurança. - Use o ponto de laser enquanto desloca o palco para encontrar as coordenadas das bordas do tecido usando a sequência para trás, direita, frente e esquerda.
- Verifique novamente as coordenadas e posicione o ponto do laser no canto frontal direito, que é o ponto de partida padrão para a imagem do tecido.
- Converta as coordenadas da janela do console em tamanhos de bloco reconhecíveis por máquina, que são definidos pelas etapas xy. Insira essas medições de etapa no protocolo do software. Feche as portas do armário.
6. 3D digitalização/seccionamento
- Altere o comprimento de onda para 940 nm para excitar GFP, mWasabi ou YPet e PE.
NOTA: O laser é invisível ao olho humano neste comprimento de onda. Para melhorar o sinal-fundo para GFP, mWasabi e o componente emissor verde do TIMERbac, coloque um filtro passa-banda estreito de 510/20 nm na frente do PMT 2. Isso reduz a interferência da autofluorescência do tecido verde-amarelo. - Mude o obturador do microscópio para o modo automático e o volume do obturadortage configurações para 20 para V1 e 1.71 para V2.
- Ajuste os três parâmetros a seguir no software: Espessura da seção - para a maioria das finalidades, definida para 50 μm. Para o fígado, ajustado para 30 μm devido à profundidade de imagem limitada. O número de seções físicas - isso determina a profundidade total do tecido que será visualizada, definida de acordo. O número de planos a serem capturados para cada seção - isso determinará a resolução z. Use 5 planos (produzindo resolução vertical de 10 μm) para a maioria das finalidades. Para fígado, use 3 ou menos planos.
- Defina o ganho do laser. Isso precisa ser testado empiricamente para diferentes tecidos e profundidade de varredura.
- Defina o caminho da pasta e o nome para armazenar as imagens adquiridas e forneça as informações apropriadas para o metarquivo de imagem.
- Verifique novamente todos os valores inseridos.
- Clique na configuração do mosaico 3D para iniciar o processo de digitalização (Figura 1D).
- Após a conclusão da imagem, colete cuidadosamente as seções de tecido do tanque de água e armazene-as em tampão de armazenamento a -20 ° C até uso posterior (Figura 1E; Figura 2C, D).
7. Processamento de imagem e análise de dados
- Baixe os scripts MATLAB em um computador Linux a partir do https://github.com/BumannLab/Li_BumannLab_2020. Copie os scripts para uma pasta, por exemplo, /home/user/Program/.
NOTA: O complemento Computer Vision Toolbox para MATLAB e Fiji (ou ImageJ) deve ser instalado em computadores Linux para que o script seja totalmente funcional. - Costure imagens de ladrilhos conforme descrito abaixo.
- Transfira dados do servidor de tomógrafo para o computador Linux.
- Abra o script MATLAB StepOneStitchingAndArchive.m e localize a pasta de origem que contém os dados brutos. Defina as pastas de origem e destino no script.
- Alterne para a guia Editor e clique em Executar. As informações de progresso são exibidas na janela de comando. O processamento inclui a leitura de informações do arquivo Mosaic, geração de índice de ladrilhos antes da costura da imagem, correção de iluminação de fundo com Cidre22 (Figura 3), bem como correção de iluminação entre camadas ópticas adquiridas em diferentes profundidades. Encontre as imagens unidas em uma subpasta chamada stitchedImages_100 na pasta de origem. Compacte os dados brutos para armazenamento de longo prazo usando o comando tar e salve como um único arquivo com a extensão de nome de arquivo tar.bz2 (Figura 1F).
- Realize treinamento e segmentação de modelos com máquina de vetores de suporte. Para treinar a máquina de vetores de suporte e realizar a segmentação, siga as etapas abaixo.
NOTA: As imagens unidas para cada um dos três canais de fluorescência são armazenadas em 3 subpastas (denominadas 1, 2 e 3, correspondendo à fluorescência vermelha, laranja e verde) de stitchedImages_100. Cada subpasta contém todas as imagens unidas do mesmo canal.- Visualize as imagens unidas. Abra uma imagem com o mesmo nome de arquivo de cada subpasta com Fiji (ou ImageJ). Mescle os três canais em uma imagem colorida. Ajuste o brilho de cada canal até que sinais claros sejam vistos de bactérias e autofluorescência do tecido. Observe o nível de intensidade máxima ajustado de cada canal para a próxima etapa.
- Abra o script MATLAB StepTwoSegmentationAndAnalysis.m e navegue até a origem. Defina a pasta de origem e os nomes das imagens para treinamento. Por exemplo
sourceD = '/' ;
red_name = [sourceD '1/section_020_01.tif']; green_name = [sourceD '2/section_020_01.tif'];
blue_name = [sourceD '3/section_020_01.tif']; - Vá para a guia Editor e clique em Executar.
- Uma caixa de diálogo solicitando limites de cores aparecerá, preencha-a com base na verificação manual anterior com Fiji (etapa 7.3.1).
- Selecione regiões para fundo e regiões de interesse (ou seja, bactérias) de acordo com as caixas de diálogo. Gráficos aparecerão mostrando o quão bem o modelo foi treinado e perguntando se mais regiões precisam ser adicionadas. Adicione mais regiões de interesse e fundo até que as bactérias possam ser claramente segmentadas (Figura 4A, B).
- Nomeie e salve o modelo. O script carregará automaticamente as imagens e aplicará um filtro mediano (raio de 2 pixels).
- O processo de segmentação é executado automaticamente e as informações de progresso são mostradas na janela de comando. Observe o progresso da segmentação. Se a segmentação não funcionar bem com o modelo em outras imagens unidas, repita o treinamento do modelo para incluir mais regiões bacterianas e de plano de fundo.
- Para ver os resultados da segmentação, verifique o arquivo chamado Allpositions_filter3D.txt. Este arquivo contém as coordenadas do centro de gravidade para cada bactéria ou microcolônia bacteriana e a intensidade de fluorescência integrada correspondente (Figura 1G).
- Remova artefatos adicionais com rede neural convolucional, conforme descrito abaixo.
NOTA: Para algumas bactérias, a segmentação simples com a Máquina de Vetores de Suporte ainda inclui alguns artefatos de imagem. Isso é particularmente verdadeiro para bactérias que expressam YPet. A segmentação baseada em rede neural pode remover com eficiência esses artefatos (Figura 4C). O complemento Computer Vision Toolbox para MATLAB e Fiji (ou ImageJ) deve ser instalado em computadores Windows para que o script seja totalmente funcional. Para obter informações sobre a rede neural subjacente, consulte: https://ch.mathworks.com/help/deeplearning/ug/create-simple-deep-learning-network-for-classification.html- Prepare as imagens. Converta arquivos tif em arquivos jpg com diferentes ajustes de brilho usando Fiji. Identifique com curadoria manual pelo menos 600 bactérias e 600 artefatos e imagens recortadas com segurança desses objetos em pastas apropriadas.
- Abra o script MATLAB CNNtestV4.m e navegue até os arquivos de origem que contêm as imagens de objeto da etapa 7.4.1.
- Defina a pasta de imagens para treinamento. Por exemplo
digitDatasetPath = fullfile ('D:\20200602_CNN','fortrainingPixel12Folder2');
Defina o número de imagens para treinamento, por exemplo, numTrainFiles = 600. - Vá para a guia Editor e clique em Executar. Após o treinamento, o arquivo de modelo CNN é salvo como gregnet1.mat na pasta de origem.
- Defina uma pasta com imagens de teste. Por exemplo
digitDatasetPathTest = fullfile('D:\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
Os resultados são salvos em um YourFile.txt de arquivo que contém informações sobre o nome da imagem e a classificação como bactéria ou artefato. - Defina a pasta com imagens de teste. Por exemplo
digitDatasetPathTest = fullfile('D:\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
Os resultados serão salvos em um arquivo YourFile.txt, que contém informações sobre o nome da imagem e a classificação como bactéria ou artefato.
- Estime o tamanho das microcolônias com base na intensidade de fluorescência de uma única bactéria, conforme descrito abaixo.
NOTA: A resolução espacial do tomógrafo é insuficiente para resolver células bacterianas individuais dentro de microcolônias densamente compactadas. No entanto, o número de bactérias em cada microcolônia pode ser estimado com base na fluorescência total da microcolônia em relação à fluorescência de uma única bactéria. Essa precisão de estimativa depende da homogeneidade da intensidade de fluorescência em toda a população bacteriana.- Confirme a identidade de eventos positivos para GFP na tomografia STP corando componentes bacterianos como lipopolissacarídeo usando imuno-histoquímica de seções recuperadas do tomógrafo (Figura 1H; Figura 5).
- Identifique pelo menos 30 bactérias individuais e recupere sua intensidade de fluorescência a partir dos resultados de segmentação correspondentes. Utilize o valor de intensidade mediana como referência para uma única bactéria. Calcular o número bacteriano de cada microcolónia dividindo a intensidade total de fluorescência da microcolónia pelo valor de referência para uma única bactéria (figura 1I).
- Realize a reconstrução 3D com software de visualização, conforme descrito abaixo.
- Preparação de imagem
- Pegue as imagens do canal azul, que contêm sinais de segundo harmônico de colágeno, fornecendo uma referência anatômica útil, incluindo cápsulas de tecido, artérias e trabéculas. Use imagens segmentadas para bactérias como o2º canal.
- Coloque as imagens do1º canal 10 vezes nos eixos x e y e salve as imagens reduzidas em uma nova pasta. Faça 3 réplicas de imagens reduzidas dentro da mesma pasta. O nome das novas réplicas deve manter a mesma sequência. Por exemplo: section_001_01.tif, replique 1 com o nome section_001_01-copy.tif, section_001_01-copy-copy.tif. Aplique essas etapas também às imagens do2º canal. O downsizing produz tamanhos de arquivo mais gerenciáveis. As imagens triplas suavizarão o eixo z.
- Mescle as imagens de cada canal para formar pilhas de imagens com Fiji. Clique em Pilhas de > de imagem > Ferramentas > Classificador de pilhas > Classificar por rótulos.
- Execute a filtragem 3D. Clique em Processar > Filtros > Filtro 3D e defina o tamanho do filtro Z como 6. Salve as pilhas de imagens.
- Execute a visualização 3D conforme descrito abaixo.
- Abra o pacote de software de visualização na visualização Arena (configuração padrão). Use o ícone da pasta Assistir para adicionar pastas à visualização da Arena. Clique duas vezes para abrir o arquivo *.ims ou *.tif (preparado em 7.6.1).
- Usando Propriedades de Edição>Imagem, ajuste as representações de cores (LUTs) dos diferentes canais na janela Ajuste de Exibição.
- Clique em Avançado para definir manualmente os valores mínimo/máximo e um valor para correção de gama.
Clique nos nomes dos canais para alterar os nomes e LUTs. - Depois de ajustar a aparência da imagem, exporte a visualização atual usando a ferramenta Instantâneo.
- Use o ícone Animação para representar os dados 3D como um filme. Use o Ponteiro de Navegação para encontrar uma perspectiva/visualização e zoom e use + Adicionar para adicionar quadros-chave. Mova para outra posição e adicione o próximo quadro-chave. Pressione o botão vermelho de gravação para criar o filme. Salve-o na pasta de destino desejada e no tipo de arquivo (Figura 1J).
- Preparação de imagem
Representative Results
O procedimento descrito permite a detecção de células individuais de Salmonella em órgãos inteiros de camundongos, como baço, fígado, linfonodos mesentéricos e placas de Peyer11 (Figura 5 e Figura 6). Ele também detecta parasitas Toxoplasma gondii no cérebro de camundongos12. Alguns tecidos infectados, incluindo fígado, placas de Peyer e baço, emitem autofluorescência substancial na faixa verde-amarela. A autofluorescência é ainda mais reforçada pela fixação com paraformaldeído, que é necessária para preservar a estrutura do tecido. A detecção de fluorescência verde de GFP, mWasabi e o componente verde do TIMERbac contra esse fundo de autofluorescência é melhorada colocando um filtro passa-banda estreito de 510/20 nm (transmitindo a maioria das emissões de GFP, mas bloqueando uma grande parte do espectro de autofluorescência) na frente do fotomultiplicador 2 (que coleta emissões verdes) e reduzindo a autofluorescência do tecido armazenando tecidos fixos por 3 ou mais dias em crioprotetor11. No entanto, as bactérias ainda devem expressar pelo menos alguns milhares de cópias por célula de GFP ou outras proteínas fluorescentes. Por outro lado, níveis excessivos de proteína fluorescente devem ser evitados para minimizar os custos de aptidão que podem levar a uma virulência atenuada23.
A segmentação correta de bactérias fluorescentes nos tecidos pode ser confirmada por imuno-histoquímica de seções de tecido recuperadas após a imagem. Especificamente, objetos corados com um anticorpo para componentes da superfície bacteriana, como lipopolissacarídeo, podem ser alinhados com a imagem de fluorescência obtida por tomografia STP (Figura 5). É importante notar que algumas células de Salmonella coradas não possuem proteínas fluorescentes e, portanto, fluorescência detectável tanto na microscopia confocal quanto na tomografia STP. Essas células são Salmonella que foram mortas pelo sistema imunológico do hospedeiro, conforme demonstrado pela classificação por citometria de fluxo e culturas de crescimento de células classificadas simples, bem como a estreita correlação entre o número de unidades formadoras de colônias em placas de ágar e o número de células fluorescentes de Salmonella , conforme determinado por citometria de fluxo24. Além disso, a perda de plasmídeo pode resultar em células viáveis não fluorescentes e isso precisa ser testado por plaqueamento em meio com e sem antibióticos apropriados correspondentes ao marcador de seleção no plasmídeo. Para plasmídeos derivados de pSC101, a perda de plasmídeo in vivo é rara19. Para a maioria dos de expressão cromossomicamente integrados, como sifB :: gfp usado em11, a perda de expressão é indetectável in vivo. Se a segmentação for inconsistente com os dados de imuno-histoquímica, o pipeline de segmentação precisará ser modificado.
A resolução da tomografia STP é insuficiente para resolver células bacterianas individuais dentro de microcolônias densamente compactadas. No entanto, a intensidade total de fluorescência da microcolônia permite estimar o número de células de Salmonella . Isso requer uma cepa fluorescente com níveis de fluorescência altamente homogêneos, como Salmonella sifB::gfp11. A combinação do número estimado de células de Salmonella para todas as microcolônias e células únicas produz cargas totais de tecido bacteriano que são consistentes com métodos alternativos, como plaqueamento ou citometria de fluxo de homogeneizados de tecido11. O plaqueamento e a citometria de fluxo não podem ser feitos diretamente dos mesmos tecidos porque precisam ser fixados por perfusão para tomografia STP. Em vez disso, eles têm que ser feitos com animais adicionais que não são fixos. Se as cargas bacterianas medianas, conforme determinado pelas várias abordagens, diferirem em mais de 3 vezes, a viabilidade das bactérias fluorescentes pode ser comprometida (no caso de unidades formadoras de colônias inferiores) ou algumas bactérias podem ter perdido a construção repórter fluorescente (no caso de unidades formadoras de colônias mais altas). O experimento de controle será necessário para identificar a fonte de tais discrepâncias.
A tomografia STP fornece a localização de células bacterianas dentro da estrutura 3D dos tecidos infectados. Os sinais do segundo harmônico do colágeno fornecem marcos anatômicos, como artérias e trabéculas. Além disso, as células hospedeiras podem ser coradas in vivo injetando um anticorpo em marcadores de superfície antes da perfusão (Figura 5 e Figura 6). Essa coloração fornece pontos de referência adicionais para compartimentos de tecido e microambientes específicos, incluindo focos de inflamação (marcadores intracelulares e alguns compartimentos com barreiras difusionais, como a polpa branca esplênica ou o cérebro, não são facilmente acessíveis, adequados para essa coloração in vivo ). Essa abordagem revelou a polpa branca esplênica como um compartimento tecidual que permite a sobrevida a longo prazo de Salmonella durante a quimioterapia antimicrobiana11.
Finalmente, a Salmonella que se replica em taxas moderadas ou lentas pode ser identificada e localizada dentro da estrutura 3D dos tecidos usando cepas que expressam o temporizadorbac, um repórter de célula única para a taxa de replicação11,15.
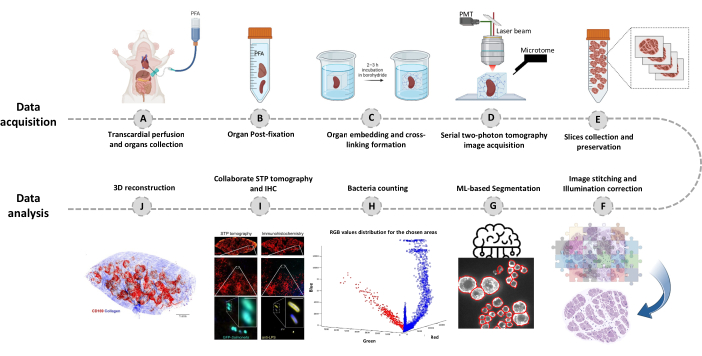
Figura 1: Procedimento para imagem de órgão inteiro usando tomografia seriada de dois fótons (STP). (A-B) O órgão é colhido após perfusão transcárdica e armazenado durante a noite em paraformaldeído (PFA) a 4% a 4 ° C. (C-E) O órgão é embebido em agarose oxidada e reticulado, então o tecido é escaneado e fatiado usando tomografia STP. As fatias são coletadas para imuno-histoquímica subsequente. (F-J) Pipelines de análise computacional para quantificar o número de bactérias, confirmação de bactérias fluorescentes e reconstrução 3D de posições bacterianas. Clique aqui para ver uma versão maior desta figura.

Figura 2: Configuração de tomografia serial de dois fótons (STP). (A) Tomógrafo que incorpora (B) imagem de 2 fótons com (C) seccionamento automatizado de tecido serial. (D) Fatias de tecido coletadas para investigações de acompanhamento. Clique aqui para ver uma versão maior desta figura.
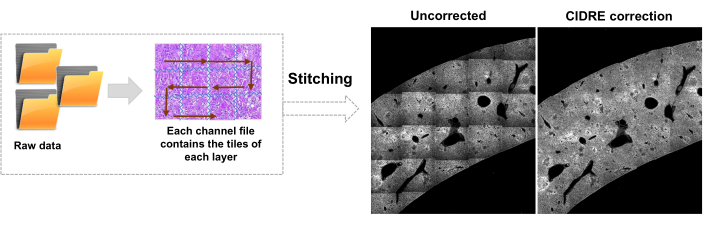
Figura 3: Costura de ladrilhos e correção de iluminação. Os ladrilhos são costurados e a iluminação irregular é corrigida para usar distribuições de intensidade corrigidas usando minimização de energia regularizada (CIDRE). Clique aqui para ver uma versão maior desta figura.
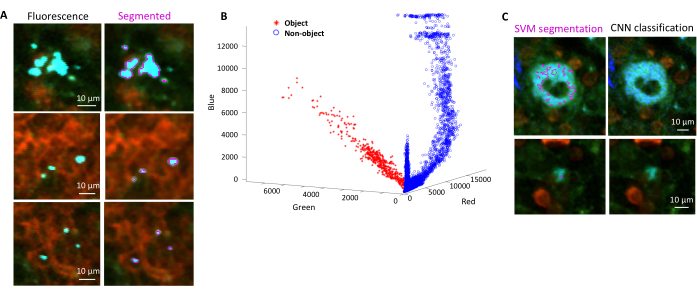
Figura 4: Segmentação de Salmonella expressando a proteína fluorescente verde (GFP) usando uma Máquina de Vetores de Suporte (SVM) e uma Rede Neural Convolucional (CNN). (A) Imagens representativas de objetos GFP segmentados por SVM (esquerda) e imagens correspondentes (direita) com regiões segmentadas por SVM (barra de escala: 10 μm). (B) Distribuição de valores clusterizados vermelho-verde-azul (RGB) para as regiões segmentadas. (C) Imagens representativas de objetos não GFP falsamente identificados pelo SVM como bactérias (à esquerda). A CNN os descarta corretamente como plano de fundo (à direita, barra de escala: 10 μm). Clique aqui para ver uma versão maior desta figura.
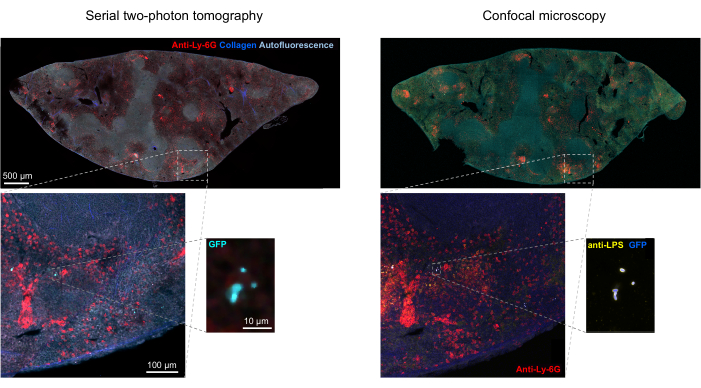
Figura 5: Detecção de Salmonella expressando a proteína fluorescente verde (GFP) por tomografia e confirmação por imuno-histoquímica. Imagens do mesmo corte adquiridas por tomografia (esquerda) ou microscopia confocal após coloração com um anticorpo para lipopolissacarídeo de Salmonella (direita). Os neutrófilos (vermelho) foram corados por injeção in vivo de um anticorpo anti-Ly-6G marcado com PE antes da perfusão. Clique aqui para ver uma versão maior desta figura.
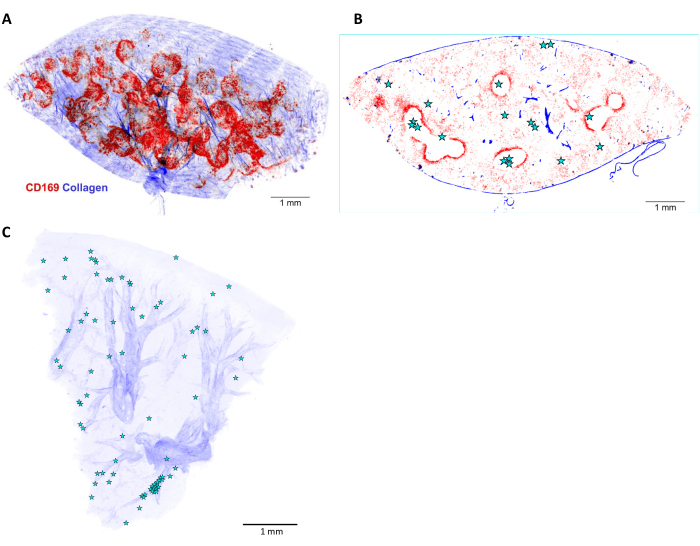
Figura 6: Reconstrução 3D e localização de Salmonella no baço de camundongo infectado. (A) Reconstrução tridimensional (3D) de um corte de baço de 5 mm de espessura e corado in vivo antes da perfusão com anticorpo anti-CD169 (vermelho). O sinal azul representa o colágeno detectado pelos segundos harmônicos. (B) Um plano óptico da pilha 3D mostrada em (A). As posições das células ou microcolônias de Salmonella são indicadas por estrelas. (C) Reconstrução 3D das posições de Salmonella (estrelas) no fígado infectado. As artérias são visíveis com base em suas bainhas de colágeno (azul). Barra de escala: 1 mm. Clique aqui para ver uma versão maior desta figura.
Discussion
O contexto tecidual local dos patógenos bacterianos é crucial para determinar os ataques locais do hospedeiro, as adaptações bacterianas, o resultado local das interações do patógeno hospedeiro e a quimioterapia antimicrobiana e as contribuições individuais para o resultado geral da doença. A imagem de bactérias do tamanho de micrômetros em órgãos de tamanho de centímetros tem sido um desafio. A tomografia seriada de dois fótons (STP) fornece resolução espacial suficiente para detectar células bacterianas individuais em órgãos inteiros, seccionamento e imagem automatizados e rendimento suficiente (~ 1 órgão por dia)11. Embora os antígenos do hospedeiro possam ser corados in vivo, as células do patógeno devem expressar proteínas fluorescentes adequadas para garantir a detecção abrangente de células patogênicas intracelulares. Os conjuntos de dados resultantes (0,5-1,5 TeraByte por órgão) representam desafios substanciais para as infraestruturas de TI para análise e armazenamento de dados.
Existem várias etapas críticas neste método. Primeiro, é necessária uma cepa de patógeno com expressão detectável e homogênea da proteína fluorescente GFP ou YFP. Idealmente, um de expressão cromossômica25 é usado para minimizar a heterogeneidade da fluorescência devido à variação do número de cópias do plasmídeo. É necessária intensidade de fluorescência suficiente, mas níveis excessivos de proteína fluorescente devem ser evitados para evitar prejuízos de aptidão do patógeno23. Níveis de expressão apropriados podem ser obtidos pela seleção de um promotor apropriado e ajuste fino do sítio de ligação ribossômica25 ou de toda a região 5 'não traduzida (UTR) 26 . Em segundo lugar, a fixação da perfusão deve envolver uma lavagem inicial com tampão para remover o maior número possível de eritrócitos da circulação sanguínea. Isso é particularmente crítico para o baço e o fígado (embora a remoção completa de eritrócitos desses órgãos seja difícil). Os eritrócitos remanescentes absorvem luz na parte visível do espectro, comprometendo a qualidade da imagem27. Em terceiro lugar, o armazenamento dos tecidos fixados no crioprotetor é fundamental para reduzir a autofluorescência do tecido, que é particularmente alta em tecidos inflamados e pode ofuscar a fluorescência comparativamente fraca das células do patógeno11. Quarto, a reticulação efetiva do tecido com o bloco de agarose circundante é crítica para o corte suave do vibratoma sem que o tecido salte do bloco de agarose. Em quinto lugar, os sinais fluorescentes e sua identificação como células patogênicas devem ser verificados de forma independente por meio de abordagens ortogonais, como coloração com anticorpos para componentes patogênicos (como lipopolissacarídeo para bactérias Gram-negativas) e microscopia confocal dos cortes recuperados do tomógrafo11. Alguns tecidos infectados contêm partículas autofluorescentes com formato semelhante e espectros de fluorescência sobrepostos que podem ser facilmente interpretados erroneamente como células patogênicas. Sexto, a quantidade de células patogênicas dentro das microcolônias deve ser comparada a abordagens ortogonais, como microscopia confocal, para avaliar a precisão. As cargas bacterianas globais baseadas nesses cálculos devem ser verificadas por comparação com abordagens ortogonais, como citometria de fluxo e plaqueamento.
Modificações importantes do protocolo STP amplamente utilizado incluem a colocação de um filtro passa-banda estreito de 510/20 nm na frente do fotomultiplicador 211, para reduzir a interferência da autofluorescência verde-amarela que é particularmente forte no fígado infectado e inflamado, baço e placas de Peyer. A forte autofluorescência e o aumento da dispersão de luz de tais órgãos em comparação com o cérebro (que domina outras aplicações do STP) também geram a necessidade de uma correção mais eficaz para iluminação irregular. Como outra modificação, este protocolo emprega a abordagem CIDRE22 para esse fim (Figura 3) e segmentação de bactérias baseada em IA. Finalmente, o pré-processamento do tecido foi alterado com a inclusão de uma etapa de incubação em crioprotetor a -20 ° C, o que reduz a autofluorescência do tecido e, portanto, facilita a detecção de pequenas células patogênicas com fluorescência relativamente fraca11.
A solução de problemas pode ser necessária se nenhum sinal de patógeno puder ser detectado ou a segmentação produzir sensibilidade insuficiente (muitas células do patógeno são perdidas) ou precisão insuficiente (muitas partículas de fundo são segmentadas como células do patógeno). Se a autofluorescência do tecido de fundo for detectável, mas houver poucos sinais de patógenos, os patógenos podem conter quantidades insuficientes de proteínas fluorescentes. Isso pode ser testado usando microscopia confocal de cortes de tecido do mesmo tecido infectado ou citometria de fluxo de homogeneizados de tecido19,28. As razões subjacentes podem ser níveis de expressão insuficientes ou instabilidade do de expressão. As estratégias de mitigação podem incluir promotores alternativos para impulsionar a expressão, adaptação de códons dos genes que codificam a proteína fluorescente para as espécies de patógenos, emprego de construções epissómicas com maior número de cópias ou estabilização de de expressão por integração cromossómica ou complementação letal equilibrada29. A escolha da proteína fluorescente também é importante, mas a detecção é possível com GFP.mut2, mWasabi, YPet e TIMERbac. Se a segmentação for imprecisa, isso pode ser causado por fluorescência de patógeno muito fraca, que pode ser tratada conforme descrito acima, ou fundo de autofluorescência de tecido muito alto. A perfusão extensa da solução de lavagem ou a incubação prolongada em tampão de armazenamento imediatamente antes da incorporação no bloco de agarose e na tomografia podem resolver esses problemas. Finalmente, é necessário treinamento suficiente da rede neural para uma classificação precisa, mas o treinamento excessivo pode levar ao sobreajuste que prejudica o desempenho de novas amostras.
Atualmente, nenhum outro método pode obter imagens de órgãos inteiros com resolução espacial suficiente em 3D para detectar bactérias individuais. Melhorias futuras na limpeza de tecidos e microscopia de folha de luz podem alcançar resolução semelhante. Isso pode permitir imagens em maior velocidade e com mais canais fluorescentes.
Uma limitação importante do STP é a resolução de pixels no plano de ~ 0,5 μm e a resolução vertical de 5 a 10 μm, o que é insuficiente para resolver bactérias localizadas próximas, por exemplo, dentro de uma microcolônia densamente compactada. No entanto, é possível recuperar cortes de tecido após a tomografia para microscopia confocal secundária de alta resolução de partes de tecido selecionadas. Outra limitação do STP é a disponibilidade de apenas três canais de fluorescência, o que restringe o número de fluoróforos que podem ser visualizados simultaneamente. Novamente, a análise secundária de seções de tecido recuperadas com métodos de multiplexação pode revelar a localização e a intensidade de muitos outros marcadores para partes de tecido selecionadas. Essas informações podem ser integradas à estrutura 3D geral do tecido circundante, conforme determinado com STP.
Em conclusão, este protocolo permite investigações detalhadas das interações patógeno-hospedeiro no nível local e de órgão inteiro. O protocolo deve ser facilmente adaptável a outros patógenos (desde que possam ser obtidos como cepas fluorescentes), outros órgãos e diferentes espécies hospedeiras.
Disclosures
Os autores não têm nada a divulgar.
Acknowledgements
O trabalho foi apoiado pela Swiss National Science Foundation 310030_156818, 310030_182315 e NCCR_ 180541 AntiResist (para DB).
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agarose Low Melt | Roth | Art. 6351.5 25g | |
| Boric acid | Sigma-Aldrich | 6768-500G | |
| Instant adhesive Loctite 435 | Henkel | ||
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Poly(ethylene glycol) | Sigma-Aldrich | P5413-1kg | |
| Polyvinylpyrrolidone | Sigma-Aldrich | PVP-100G | |
| Sodium borohydride | Sigma-Aldrich | 71321-25g | |
| Sodium hydroxide | Merck | 106453 | |
| Sodium periodate | Sigma-Aldrich | 311448-100G | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640-250G | |
| Sodium phosphate monobasic dihydrate | Sigma-Aldrich | 71500-1KG | |
| Sodium tetraborate | Sigma-Aldrich | 221732-100g | |
| Sucrose | AppliChem | A4734,1000 | |
| Tris-buffered saline (TBS) | Merck | T5912-1L | |
| Triton X-100 | Sigma-Aldrich | 9002-93-1 | |
| Vacuum filtration 500 | TPP | TPP99250 | |
| Equipment | |||
| Blade | Campden Instruments Limited | 01-01-4692 | |
| MAITAI Laser | Spectra-Physics | ||
| Peel away plastic mold | Sigma-Aldrich | E6032-1CS | |
| TissueCyte 1000 tomograph | TissueVision | ||
| Antibody/dyes | |||
| DAPI | Merck | D9542-5MG | |
| Primary antibodies | |||
| anti-LPS Salmonella, rabbit | Sifin | REF TS 1624 | |
| anti-CD169-PE, clone 3D6.112 | Biolegend | 142403 | |
| anti-Ly-6G-PE, clone 1A8 | Biolegend | 127608 | |
| Secondary antibodies | Invitrogen | ||
| chicken anti-rabbit Alexa 647 | Invitrogen | A-21443 | |
| Software | Company | Version | |
| Fiji | Image J | 1.54g or later | |
| MATLAB | MathWorks | 2017b/2018b or later | |
| Orchestrator (tomograph) | TissueVision | ||
| Visualization software Imaris | Oxford Instruments | 9.9.0 or later |
References
- Bjarnsholt, T., et al. The importance of understanding the infectious microenvironment. Lancet Infect Dis. 22 (3), e88-e92 (2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Bumann, D., Cunrath, O. Heterogeneity of Salmonella-host interactions in infected host tissues. Curr Opin Microbiol. 39, 57-63 (2017).
- Hofmann, J., Keppler, S. J. Tissue clearing and 3D imaging - putting immune cells into context. J Cell Sci. 134 (15), jcs258494 (2021).
- Blain, R., et al. A tridimensional atlas of the developing human head. Cell. 186 (26), 5910-5924.e17 (2023).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Meth. 9 (3), 255-258 (2012).
- Oh, S. W., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Kim, Y., et al. Brain-wide maps reveal stereotyped cell-type-based cortical architecture and subcortical sexual dimorphism. Cell. 171 (2), 456-469.e422 (2017).
- Matho, K. S., et al. Genetic dissection of the glutamatergic neuron system in cerebral cortex. Nature. 598 (7879), 182-187 (2021).
- Muñoz-Castañeda, R., et al. Cellular anatomy of the mouse primary motor cortex. Nature. 598 (7879), 159-166 (2021).
- Li, J., et al. Tissue compartmentalization enables Salmonella persistence during chemotherapy. Proc Natl Acad Sci U S A. 118 (51), e2113951118 (2021).
- Dogga, S. K., et al. Importance of aspartyl protease 5 in the establishment of the intracellular niche during acute and chronic infection of Toxoplasma gondii. Mol Microbiol. 118 (6), 601-622 (2022).
- Cormack, B. P., Valdivia, R. H., Falkow, S. FACS-optimized mutants of the green fluorescent protein (GFP). Gene. 173 (1 Spec No), 33-38 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Claudi, B., et al. Phenotypic variation of Salmonella in host tissues delays eradication by antimicrobial chemotherapy. Cell. 158 (4), 722-733 (2014).
- Ai, H. W., Olenych, S. G., Wong, P., Davidson, M. W., Campbell, R. E. Hue-shifted monomeric variants of Clavularia cyan fluorescent protein: identification of the molecular determinants of color and applications in fluorescence imaging. BMC Biol. 6, 13 (2008).
- Drobizhev, M., Makarov, N. S., Tillo, S. E., Hughes, T. E., Rebane, A. Two-photon absorption properties of fluorescent proteins. Nat Methods. 8 (5), 393-399 (2011).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Burton, N. A., et al. Disparate impact of oxidative host defenses determines the fate of Salmonella during systemic infection in mice. Cell Host&Microbe. 15 (1), 72-83 (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio Protoc. 11 (5), e3988 (2021).
- de Olmos, J., Hardy, H., Heimer, L. The afferent connections of the main and the accessory olfactory bulb formations in the rat: an experimental HRP-study. J Comp Neurol. 181 (2), 213-244 (1978).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nat Methods. 12 (5), 404-406 (2015).
- Wendland, M., Bumann, D. Optimization of GFP levels for analyzing Salmonella gene expression during an infection. FEBS Lett. 521 (1-3), 105-108 (2002).
- Barat, S., et al. Immunity to intracellular Salmonella depends on surface-associated antigens. PLoS Pathog. 8 (10), e1002966 (2012).
- Rollenhagen, C., Sorensen, M., Rizos, K., Hurvitz, R., Bumann, D. Antigen selection based on expression levels during infection facilitates vaccine development for an intracellular pathogen. Proc Natl Acad Sci U S A. 101 (23), 8739-8744 (2004).
- Chen, F., Cocaign-Bousquet, M., Girbal, L., Nouaille, S. 5'UTR sequences influence protein levels in Escherichia coli by regulating translation initiation and mRNA stability. Front Microbiol. 13, 1088941 (2022).
- Weissleder, R. A clearer vision for in vivo imaging. Nat Biotechnol. 19 (4), 316-317 (2001).
- Bumann, D. Examination of Salmonella gene expression in an infected mammalian host using the green fluorescent protein and two-colour flow cytometry. Mol.Microbiol. 43 (5), 1269-1283 (2002).
- Nakayama, K., Kelly, S. M., Curtiss III, R. J. B. t. Construction of an Asd+ expression-cloning vector: stable maintenance and high level expression of cloned genes in a Salmonella. vaccine strain. 6 (6), 693-697 (1988).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved