Imagem ao vivo e caracterização da dinâmica da microglia no embrião de peixe-zebra
In This Article
Summary
Demonstramos um método que aproveita a microscopia confocal de varredura rápida para realizar imagens ao vivo de células da microglia no tectum óptico do peixe-zebra em desenvolvimento, permitindo a análise da dinâmica dessas células in vivo.
Abstract
A microglia é uma célula altamente dinâmica e sua migração e colonização do parênquima cerebral é uma etapa crucial para o desenvolvimento e função adequados do cérebro. Os embriões de peixe-zebra em desenvolvimento externo possuem transparência óptica, que, juntamente com linhas repórter transgênicas bem caracterizadas que rotulam fluorescentemente a microglia, tornam o peixe-zebra um modelo de vertebrado ideal para tais estudos. Neste artigo, aproveitamos as características únicas do modelo de peixe-zebra para visualizar a dinâmica das células da microglia in vivo e sob condições fisiológicas. Usamos microscopia confocal para registrar um lapso de tempo de células da microglia no tectum óptico do embrião de peixe-zebra e, em seguida, extraímos dados de rastreamento usando o software IMARIS 10.0 para obter o caminho de migração das células, velocidade média e distribuição no tectum óptico em diferentes estágios de desenvolvimento. Este protocolo pode ser uma ferramenta útil para elucidar o significado fisiológico do comportamento da microglia em vários contextos, contribuindo para uma caracterização mais profunda dessas células altamente móveis.
Introduction
Como macrófagos residentes no sistema nervoso central (SNC), a microglia representa uma população não neuronal distinta que representa até 15% de todas as células gliais no cérebro adulto. O estudo da biologia da microglia tem ganhado cada vez mais atenção nos últimos anos devido à sua importância estabelecida no desenvolvimento, fisiologia e doença1. Em condições fisiológicas, as células microgliais são altamente dinâmicas, examinando continuamente o parênquima cerebral 2,3. Esse comportamento permite que a micróglia colonize o cérebro e desempenhe papéis fundamentais em seu desenvolvimento, como moldar os circuitos neuronais4, a poda sináptica5 e a vasculogênese6. Além disso, essa natureza dinâmica inerente permite que a microglia monitore constantemente o SNC em busca de sinais de infecção, lesão ou quaisquer desvios da homeostase7. Para dissecar essas intrincadas dinâmicas celulares, imagens ao vivo da microglia no espaço e no tempo são indispensáveis. Felizmente, a transparência óptica de embriões de peixe-zebra em desenvolvimento externo, juntamente com a disponibilidade de linhas repórter transgênicas bem caracterizadas que marcam fluorescentemente a microglia, posiciona o peixe-zebra como um modelo de vertebrado ideal para tais investigações. A imagem ao vivo em embriões de peixe-zebra oferece uma abordagem não invasiva que não requer cirurgia ou manipulação extensa de tecidos, minimizando possíveis perturbações no estado do SNC. Esta é uma consideração crítica ao estudar células microgliais, pois elas são altamente sensíveis até mesmo a mudanças sutis no ambiente extracelular8.
Aqui, fornecemos uma diretriz para rastrear com sucesso os movimentos das células microgliais 3D no embrião de peixe-zebra, permitindo uma visão sem precedentes do comportamento da microglia dentro da arquitetura intacta do parênquima cerebral em desenvolvimento (consulte a Figura 1 para obter uma visão geral gráfica do protocolo). Este protocolo passo a passo detalha como configurar e obter imagens da micróglia do peixe-zebra em diferentes estágios de desenvolvimento e como extrair dados de alta resolução sobre a motilidade das células da microglia para fornecer informações valiosas sobre seus padrões migratórios e respostas a pistas ambientais. Também demonstramos que este protocolo pode ser adaptado para realizar imagens multicoloridas ao vivo, estendendo assim sua aplicabilidade ao estudo da microglia em combinação com linhas transgênicas que marcam células vizinhas, incluindo neurônios3, oligodendrócitos9 e células endoteliais10 (como mostrado na Figura 2). Ao adicionar à caixa de ferramentas que permite observar e caracterizar diretamente a dinâmica do comportamento da microglia em tempo real e em seu ambiente natural, este protocolo provavelmente contribuirá para elucidar melhor a funcionalidade da microglia durante o desenvolvimento inicial, tanto na fisiologia quanto na doença.
Protocol
Os peixes-zebra foram mantidos em condições padrão, de acordo com FELASA42 e institucionais (Université Libre de Bruxelles, Bruxelas, Bélgica; ULB). Todos os procedimentos experimentais foram aprovados pelo Comitê de Ética para o Bem-Estar Animal da ULB (CEBEA) da ULB.
1. Criação de peixe-zebra e preparação de embriões
NOTA: A linhagem transgênica de peixe-zebra Tg(mpeg1:eGFP)gl22 que expressa a proteína fluorescente eGFP em macrófagos, incluindo microglia, foi usada para gerar os dados de rastreamento mostrados neste protocolo. Outras linhagens repórteres de macrófagos estão disponíveis no ZIRC e também podem ser usadas. Na escolha da linhagem transgênica, é importante considerar que uma alta intensidade de sinal facilitará a aquisição de imagens e a segmentação celular.
- Manter o peixe-zebra (Danio rerio) a uma temperatura de 28 °C e geri-lo de acordo com os protocolos estabelecidos11.
- Estimule o acasalamento colocando peixes fêmeas e machos juntos em um tanque de reprodução, com fundo de malha e separador, no final da tarde antes da data de nascimento dos embriões desejados. Remova o separador na manhã seguinte para cronometrar o acasalamento dos peixes.
- Colete os óvulos fertilizados usando uma tela e transfira-os para uma placa de Petri de 100 mm contendo meio E3 (5 mM NaCl, 0,17 mM KCl, 0,33 mM CaCl2, 0,33 mM MgSO4) com 0,01% de azul de metileno. Dependendo do histórico genético da linhagem transgênica de peixe-zebra usada, se a formação de pigmento for inibida para facilitar a imagem, adicione 0,003% de N-feniltioureia (PTU) ao tampão E3 no qual os embriões estão se desenvolvendo, começando em 6 h após a fertilização (hpf) e atualizando a solução a cada 2 dias até que o estágio de desenvolvimento desejado seja alcançado.
NOTA: Para evitar a exposição prolongada à PTU que pode afetar o desenvolvimento embrionário12, sugere-se o uso, se disponível, de uma linha transgênica em um fundo deficiente em pigmento, como Casperw2, que permite imagens nítidas sem a necessidade de expor embriões à PTU por um longo período de tempo13. - Se os embriões a serem fotografados tiverem menos de 3 dias após a fertilização (dpf), descorione-os a 24 hpf. Use um microscópio estéreo para confirmar que todos os embriões foram descorionados e não foram danificados pelo tratamento enzimático (consulte a etapa 1.4.2).
- No caso de um número limitado de óvulos, faça isso manualmente usando uma pinça de dissecação fina para beliscar o córion e puxá-lo para fora, liberando cuidadosamente cada embrião de seu córion.
- No caso de um número maior de ovos, realize a descorionação enzimaticamente incubando os ovos a granel à temperatura ambiente (TR) em uma solução de Pronase de 1 mg / mL em meio E3 por um breve período (normalmente 5-10 min, até que os córions se abram sob movimentos suaves). Em seguida, a solução de Pronase é descartada e os embriões são enxaguados abundantemente 3x com E3 fresco, para remover qualquer córion restante e enzima residual.
2. Montagem de peixe-zebra
- Prepare uma solução de agarose de baixo ponto de fusão a 1% em tampão E3. Resfrie a agarose a 37 ° C em um bloco de aquecimento de bancada e adicione uma solução de metanossulfonato de tricaína recém-feita (160 mg / L), que ajudará a manter os embriões anestesiados durante a imagem. Vortex a solução para homogeneizá-la completamente.
- Anestesiar os embriões adicionando 160 mg/L de metanossulfonato de tricaína à placa de Petri que contém os embriões em tampão E3. Verifique se o movimento parou e selecione os embriões de peixe-zebra com a fluorescência desejada. Verifique a saúde do embrião visualizando seus batimentos cardíacos usando um microscópio estéreo de campo claro. Com uma pipeta de transferência de ponta larga, retirar um ou até três dos embriões seleccionados e transferi-los para uma placa de imagem de vidro inferior.
- Sob um microscópio estéreo, use uma pipeta para remover o máximo de meio possível, tomando cuidado para não tocar ou danificar os embriões; Em seguida, substitua o meio pela solução de agarose de baixo ponto de fusão a 1% previamente preparada. Em seguida, use uma ponta plástica cônica fina fixada na extremidade de uma agulha provocadora para empurrar suavemente cada embrião para o fundo da placa de imagem, orientando-o de forma que seu lado dorsal fique voltado para o vidro para garantir uma boa visão do tectum óptico e uma quantidade mínima de agarose de baixo ponto de fusão entre o embrião e o vidro.
- Visualize o embrião montado imediatamente usando um microscópio confocal invertido com uma objetiva Plan Apo de alta qualidade (10x/0,45 Plan Apo ou 20x/0,75 Plan Apo usado aqui). Se o timelapse for executado por um longo período (mais de uma hora), adicione uma ou duas gotas de tampão E3 com metanossulfonato de tricaína (160 mg / L) à placa de imagem de vidro inferior para evitar que a agarose de baixo ponto de fusão seque excessivamente.
NOTA: É crucial esperar que a agarose solidifique completamente antes de adicionar tampão E3 e meio de metanossulfonato de tricaína ao disco de imagem; caso contrário, o peixe pode se mover lentamente lateral e axialmente, causando assim um desvio de amostra no timelapse. - Prossiga para configurar a aquisição de lapso de tempo para o tempo desejado: defina a resolução da imagem para 1.024 x 1.024, com um tamanho de pixel de 0,49 μm. Colete fatias ópticas em incrementos de 2-3 μm para garantir uma resolução aceitável no eixo z. No embrião de peixe-zebra, coloque o tectum óptico em uma pilha z de 100-150 μm, dependendo do estágio de desenvolvimento usado. Para garantir o rastreamento de células bem-sucedido, mantenha o intervalo de tempo entre os quadros entre 30 s e 60 s.
3. Análise de rastreamento e exportação de dados
- Converta o arquivo de lapso de tempo para o formato IMS usando o software conversor de arquivos Imaris (consulte Tabela de materiais). Ao iniciar o software, arraste e solte os arquivos na área de entrada ou utilize o botão Adicionar arquivos... para selecioná-los manualmente. No menu Saída , designe o local para os arquivos convertidos, optando pela mesma pasta dos arquivos de entrada ou especificando uma pasta específica. Certifique-se de que as dimensões do tamanho do voxel exibido estejam corretas clicando no botão Definir tamanho do voxel e, uma vez verificado o tamanho do voxel, pressione o botão Iniciar tudo para iniciar a conversão do arquivo (consulte o Arquivo Suplementar 1 para obter um guia passo a passo de toda a análise de dados).
NOTA: Se o timelapse foi realizado em dois ou mais embriões em paralelo, o arquivo será automaticamente dividido em posições únicas, separando cada embrião em arquivos diferentes. É possível usar o software em modo de lote e fazer upload e converter vários arquivos ao mesmo tempo. Os arquivos IMS serão criados e salvos na pasta de destino selecionada ou na pasta onde os arquivos de entrada estão localizados. - Uma vez iniciado, deixe o software de análise iniciar na Arena; clique em Observar pasta para abrir o arquivo convertido anteriormente. Depois que o arquivo for carregado no software, use a visualização Slice para rolar pela pilha z, usando o controle deslizante na barra de ferramentas Slice situada à esquerda da área de exibição, e meça alguns diâmetros de célula desenhando o diâmetro das células selecionadas usando o ponteiro. Clique e arraste o mouse para desenhar um segmento abrangendo o núcleo da célula; observe o comprimento do segmento desenhado na barra de ferramentas Medida, situada à direita da Área de Exibição.
NOTA: Usando as configurações de imagem na etapa 2.5, pode-se esperar que o diâmetro do corpo celular esteja entre 6 μm e 8 μm. - Para detectar automaticamente as células, volte para a vista 3D e clique no ícone de ponto na barra de ferramentas Objeto para adicionar um novo objeto Pontos na lista de objetos e abrir o assistente automático de criação de pontos. Na primeira etapa do assistente, marque a opção de segmentar apenas uma região de interesse. Em seguida, ative a opção Rastrear pontos (ao longo do tempo) para calcular os dados de rastreamento, bem como as Estatísticas de objeto-objeto para permitir a comparação entre pontos e expandir o intervalo de dados que estarão disponíveis posteriormente na análise. Por fim, prossiga no assistente clicando no botão Avançar no canto inferior direito da janela do assistente.
- Na segunda etapa, prossiga para especificar uma área de região de interesse (ROI) que envolve o tectum óptico do embrião. Modifique o tamanho do ROI clicando e arrastando as pequenas setas brancas presentes em cada face dele. Depois que o tamanho do ROI for definido, estenda-o a todos os quadros gravados ajustando o intervalo de tempo.
- Em seguida, selecione como Canal de Origem o canal usado para detectar as células e defina a medição do diâmetro da célula, obtida anteriormente na etapa 3.2, como o Diâmetro XY Estimado. Ative a opção Subtração em segundo plano e clique em Avançar.
- Neste ponto do assistente de criação de pontos, o software incorpora uma etapa de filtragem de qualidade em que os pontos são filtrados pela intensidade do sinal medida no centro de cada ponto; no canal onde as manchas foram detectadas. Ajuste o limite de intensidade inserindo o valor diretamente no campo de dados para as caixas de limite inferior e superior ou arrastando as linhas coloridas correspondentes no histograma mostrado na parte inferior do assistente de criação, para que todas as células sejam detectadas, incluindo aquelas localizadas mais profundamente no tecido, que geralmente mostram uma intensidade de sinal mais baixa. Todas essas alterações são visíveis simultaneamente na Área de Exibição.
NOTA: É possível observar vários pontos rotulando uma única célula neste ponto, mas isso pode ser corrigido posteriormente na análise.
Em timelapses mais longos, se algum fotobranqueamento for visto, verifique a área de visualização de vários quadros antes de definir o limite para garantir que todas as células também sejam detectadas nos quadros mais recentes e, portanto, mais fracos. - Quando estiver satisfeito, clique em Avançar para validar o limite de qualidade selecionado. Todos os pontos que satisfaçam o parâmetro de limite de qualidade e tenham um diâmetro correspondente ao diâmetro XY estimado serão rotulados como um ponto. Visualize isso como uma visualização na Área de Exibição, onde os pontos que satisfazem esses dois parâmetros serão sobrepostos no Canal de Origem.
- Na próxima etapa, selecione o Movimento Autorregressivo como o algoritmo de rastreamento para rastrear células que têm um movimento mais ou menos contínuo. Julgue a distância mais longa que uma célula se move entre dois pontos de tempo observando cuidadosamente o movimento dos pontos entre dois quadros e insira um número estimado no campo Distância máxima . Esta é a distância que um ponto pode se desviar da posição futura prevista.
- Defina o tamanho máximo da lacuna para o menor número de pontos de tempo em que o ponto pode desaparecer sem interromper a faixa. Se o intervalo entre os quadros for inferior a 60 s, use um tamanho de 3. Se o intervalo de tempo entre os quadros for maior, aumente o tamanho máximo da lacuna permitido para reduzir a fragmentação da trilha.
- Ative o Preencher lacunas com todos os objetos detectados para diminuir o limite de detecção próximo à posição esperada prevista pelo algoritmo de rastreamento e conectar trilhas interrompidas por uma falha na detecção de ponto. Por fim, clique em Avançar e deixe o software gerar automaticamente faixas para todos os pontos gerados anteriormente.
- Se os rastros obtidos parecerem muito numerosos ou fragmentados, volte ao assistente de criação, clicando no botão Anterior , e ajuste a distância máxima definida anteriormente, aumentando ou diminuindo o número até que a maioria dos rastros obtidos seja representativa dos movimentos da célula.
NOTA: O número de rastros deve corresponder aproximadamente ao número de células da microglia, portanto, variando entre 30 e 50, dependendo da idade do embrião. No entanto, antes da conclusão da análise, espera-se um número maior devido a trilhas errôneas. - Uma vez que as faixas tenham sido calculadas, o software incorpora uma etapa de filtragem para filtrar todas as faixas muito breves, pois geralmente são imprecisas ou não informativas. Exclua faixas com menos de 3 a 8 minutos (min) inserindo o valor diretamente no campo de dados para o limite inferior do filtro de duração da faixa ou arrastando as linhas coloridas correspondentes no histograma mostrado na parte inferior do assistente de criação. Clique em Avançar para validar o filtro e prosseguir no assistente de criação.
NOTA: Como mencionado anteriormente, modificando o limite máximo (máx) e mínimo (min) de duração da faixa, é possível visualizar as faixas que serão filtradas e excluídas da análise. As trilhas filtradas podem então ser duplicadas em um novo objeto para análise posterior. O limite de tempo exato para filtragem pode variar com base nos resultados da etapa anterior do assistente de criação . Recomendamos ajustar essas configurações de filtro revisando a visualização na Área de Exibição para obter os melhores resultados. - Como a linha transgênica Tg(mpeg1:eGFP) marca todos os fagócitos mononucleares, os macrófagos da pele, além da microglia, também serão detectados em filmes de lapso de tempo. Remova manualmente os rastros gerados pelos macrófagos da pele da análise para focar na micróglia parenquimatosa cerebral (Figura 3).
- Corrija manualmente outros possíveis erros nas trilhas no editor de trilhas, onde cada ponto de dados é exibido como um objeto na trilha. Ative o botão de modo de seleção de círculo à direita da Área de visualização e destaque as faixas que requerem modificações ou ajustes. Uma vez selecionada a faixa problemática, usando os botões de conexão e desconexão , edite-a para representar corretamente os movimentos da célula. Use o editor de trilha para também selecionar e excluir pontos únicos duplicados ou pequenos fragmentos de trilha que representam erroneamente a mesma célula, selecionando-os com o modo de seleção de círculo e pressionando Canc ou Delete para eliminá-los.
NOTA: Se as configurações do rastreamento estiverem corretas, esta etapa deve ser necessária apenas para uma pequena porcentagem das células rastreadas. - Se a linha repórter da microglia for usada em combinação com transgênicos fluorescentes que marcam outros tipos de células no parênquima, execute a segmentação como um novo objeto de superfície ou objeto pontual para visualizá-los. Para isso, repita os passos acima (dos passos 3.2 a 3.14), modificando a entrada do Canal de Origem no passo 3.5, para que seja a utilizada para a imagem da segunda população de células, e cuidando de adaptar os parâmetros descritivos específicos das células de interesse, como Diâmetro XY Estimado (passo 3.5), Distância Máxima (passo 3.8) para a nova população de células. Outras etapas podem ser repetidas sem alterações.
NOTA: No caso de uma nova criação de superfície, o Diâmetro XY estimado é substituído por uma opção de limite com base na Subtração de fundo, onde a entrada a ser fornecida é o diâmetro da maior esfera que se encaixa no objeto. Esse valor pode ser estimado na exibição Fatia usando a barra de ferramentas Medir , conforme explicado na etapa 3.2. - Quando estiver satisfeito com as faixas obtidas, extraia as estatísticas de rastreamento desejadas da guia Estatística . Clicando no botão de configurações , selecione quais estatísticas serão calculadas para cada objeto de ponto ou objeto de superfície criado anteriormente. Caso mais objetos tenham sido criados na mesma análise, dois objetos pontuais ou um objeto pontual e um objeto de superfície, as estatísticas que descrevem a posição relativa de um objeto em relação ao outro, como a distância mais curta entre os dois, também estarão disponíveis para exportação.
NOTA: Todos os valores desejados podem ser valores específicos ou médios, e todos os dados brutos podem ser baixados como um arquivo CSV (Valores Separados por Vírgula) usando os botões na parte inferior da guia Estatísticas . Desde que a análise também inclua outros tipos de células, estatísticas adicionais sobre a distância relativa de um objeto ao outro também estarão disponíveis.
Representative Results
Células microgliais que expressam proteína fluorescente verde (eGFP) e células endoteliais que expressam DsRed em Tg(mpeg1:eGFPgl22; KDRL: CRES898; actb2:loxP-STOP-loxP-DsRedexpress,sd5) embriões transgênicos triplos14 foram fotografados a 3 dpf, de acordo com o protocolo descrito. Um único embrião de peixe-zebra foi montado em agarose de baixo ponto de fusão a 1% em uma placa de vidro inferior e o processo de imagem não impediu o crescimento do embrião durante o tempo de aquisição. O timelapse foi registrado usando um sistema comercial de microscopia confocal de varredura pontual equipado com uma lente objetiva seca de 10x 0,45 NA, e lasers de excitação de 488 nm e 561 nm foram usados para imagens de células microgliais e endoteliais, respectivamente. Além disso, o intervalo de tempo, a resolução da imagem, o tamanho do pixel e o passo z foram de 30 segundos (s), 1024x1024, 0,49 μm e 2,5 μm, respectivamente. O registro em timelapse teve duração de 3,5 horas (h).
O campo de visão adquirido cobria toda a cabeça do embrião, mas a análise se concentrou especificamente no tecto óptico, pois durante a primeira semana de desenvolvimento, as microglias ficam restritas principalmente à camada de soma neuronal dessa região do mesencéfalo dorsal, permitindo que os pesquisadores visualizem toda a população simultaneamente15. A análise de rastreamento 3D foi realizada usando o Imaris 10.0, conforme explicado acima. Conforme mostrado na Figura 4, o rastreamento foi bem-sucedido, resultando em 25 trilhas, correspondendo ao número esperado de células da microglia presentes no tectum óptico em 3 dpf16. Uma correção manual mínima dos rastros foi necessária. A Figura 5 mostra um exemplo dos dados que é possível extrair de um experimento de rastreamento bem-sucedido. A marcação simultânea de macrófagos e células endoteliais permite a quantificação da posição da microglia em relação a estas últimas, permitindo que os pesquisadores visualizem a distância relativa de cada célula até a célula endotelial mais próxima no tempo e examinem a frequência e o número de interações potenciais (Figura 5).
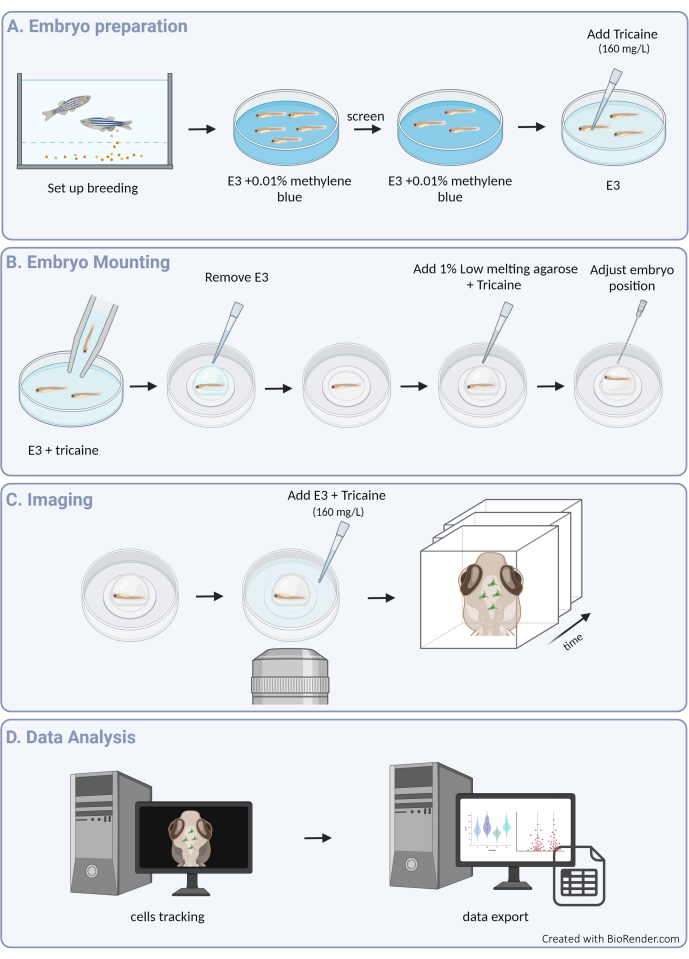
Figura 1: Visão geral do procedimento experimental. (A) Preparação e anestesia de embriões de peixe-zebra. (B) Montagem e posicionamento da amostra. (C) Aquisição de imagens. (D) Processamento de imagem e extração de dados de motilidade. Clique aqui para ver uma versão maior desta figura.
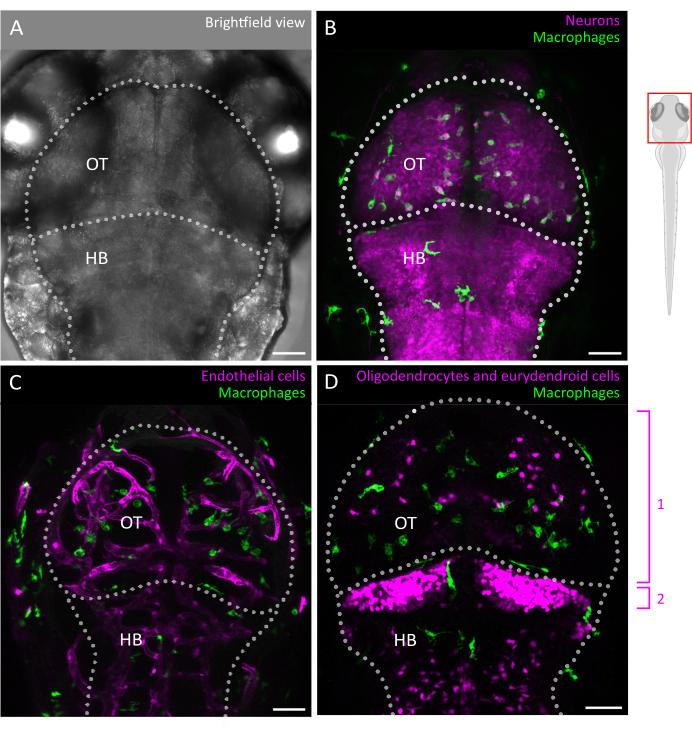
Figura 2: Imagem da microglia e interações com o ambiente celular do cérebro no embrião de peixe-zebra. (A) Imagem de campo claro de uma cabeça e cérebro embrionários de peixe-zebra de 3 dpf (vista dorsal), rotulando o tectum óptico e o rombencéfalo. O cérebro do peixe-zebra embrionário pode ser fotografado em sua totalidade nesta fase devido ao seu pequeno tamanho e transparência óptica. (BD) A localização e o comportamento da microglia podem ser visualizados pelo uso de linhagens transgênicas como (B,C) Tg(mpeg1:eGFP)gl22 e (D) Tg(mpeg1:mcherry)gl23, e com foco no OT. (B) Os neurônios e seus corpos celulares podem ser identificados usando a linha repórter Tg (XlTubb :D sRed) zf148 e as interações microglia-neurônio podem ser visualizadas pela fusão dos dois canais em linhas que co-expressam ambos os transgenes. Visto é uma fusão dos sinais verde (microglia) e vermelho (neurônios, em magenta). (C, D) As linhas repórter também podem lançar luz sobre as interações da microglia (em verde) com células endoteliais, aqui marcadas em vermelho usando o (C) duplo Tg (kdrl: cres898; actb2:LOXP-STOP-LOXP-Dsredexpress, sd5) transgênicos, ou com células gliais como oligodendrócitos e seus precursores, usando o (D) duplo Tg(olig2:EGFP; MPEG1:mCherry ) linha transgênica. 1: oligodendrócitos e células progenitoras no OT, 2: neurônios eurydendroides no cerebelo. Barras de escala = 50 μm. Abreviaturas: dpf = dias pós-fertilização; OT = tectum óptico; HB = rombencéfalo. Clique aqui para ver uma versão maior desta figura.
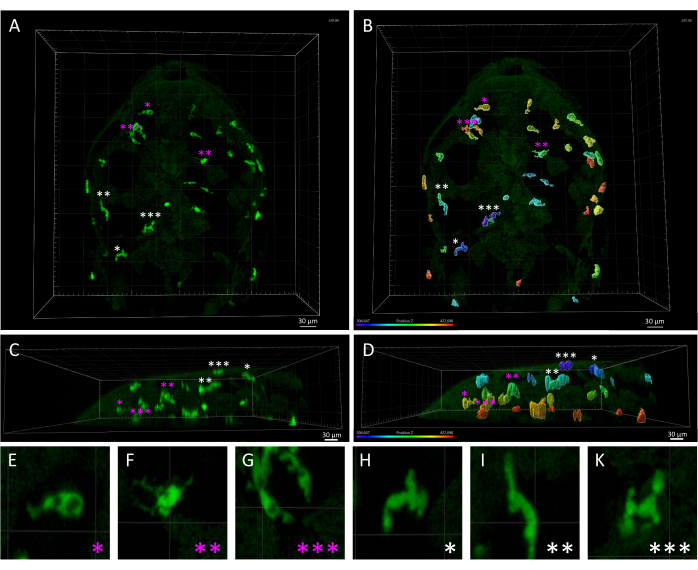
Figura 3: Disparidade morfológica e espacial entre macrófagos da pele e micróglia em um embrião de peixe-zebra de 3 dpf. (A, B) Vista dorsal de um embrião de peixe-zebra Tg (mpeg1: eGFP) gl22 a 3 dpf, representando (A) células da microglia parenquimatosa (marcadas por asteriscos magenta) versus macrófagos da pele (asteriscos brancos), identificados com base em sua posição relativa ao longo do eixo z, como visto em (B), que mostra a imagem 3D monocromática renderizada com codificação de cores do eixo z. (C, D) Vista lateral de (C) A e (D) B, mostrando as profundidades z das células mpeg1: eGFP + dentro da cabeça do embrião e destacando a localização superficial dos macrófagos da pele em comparação com as células microgliais. (E-K) Alta ampliação de cada célula indicada por um asterisco em A e B, fornecendo uma visualização detalhada das morfologias distintas entre a micróglia amebóide (EG) e os macrófagos cutâneos alongados (HK). Barras de escala = 30 μm. Abreviatura: dpf = dias após a fertilização. Clique aqui para ver uma versão maior desta figura.
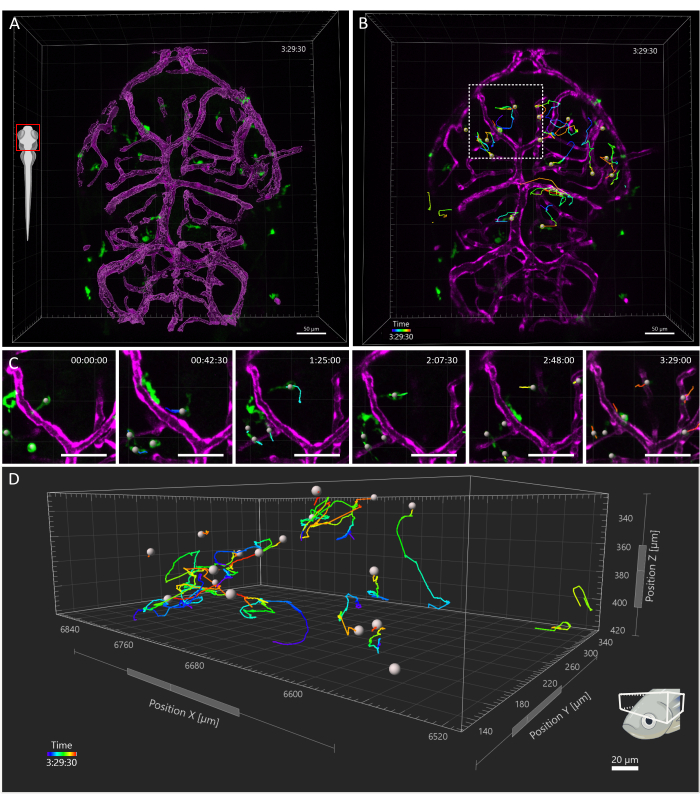
Figura 4: Rastreamento de células microgliais em um embrião de peixe-zebra de 3 dpf. (A) Vista dorsal da cabeça de um Tg de 3 dpf (kdrl: cres898; actb2: LOXP-STOP-LOXP-Dsredexpresso, sd5; mpeg1:eGFPgl22) embrião triplo transgênico, mostrando microglia (verde) e a renderização da superfície dos vasos (magenta). (BD) Rastreamento representativo dos movimentos da microglia em uma janela de tempo (B) de 3,5 h. As células individuais são vistas seguindo trajetórias complexas. (C) Detalhe do timelapse, mostrando uma visão ampliada da região em B delimitada pelo quadrado tracejado. Seis quadros do filme (45 minutos de intervalo) são apresentados, documentando a micróglia estabelecendo contatos transitórios com células endoteliais em seu microambiente. (D) Trajetórias de migração de células microgliais individuais dentro do tectum óptico, com os eixos X, Y e Z representando dimensões espaciais. Barra de escala = 50 μm. Abreviatura: dpf = dias após a fertilização. Clique aqui para ver uma versão maior desta figura.
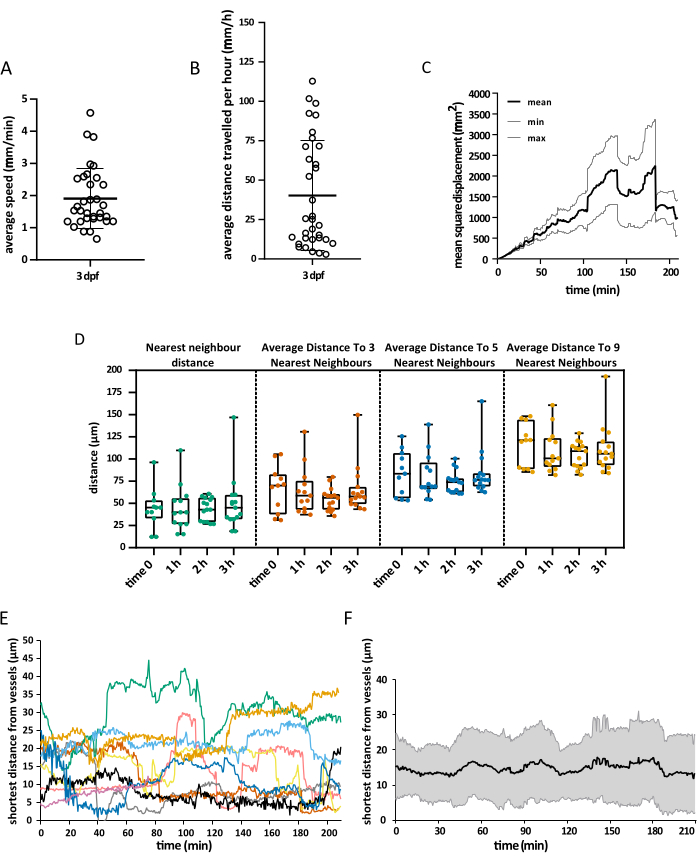
Figura 5: Visualização dos dados obtidos. Exemplo dos dados espaciais que podem ser obtidos usando o protocolo descrito. Os gráficos mostram (A) a velocidade média das células microgliais, (B) a distância média que percorrem em 1 h, (C) seu deslocamento quadrático médio e (D) sua distribuição no espaço em diferentes momentos. A renderização da superfície do vaso também permitiu a medição da distância mais curta entre a microglia e as células endoteliais em um determinado momento, tanto no nível (E) de célula única quanto na média global (F). Para A, B e D, cada ponto representa uma célula individual. N = um embrião. Clique aqui para ver uma versão maior desta figura.
Arquivo Suplementar 1: Clique aqui para baixar este Arquivo.
Discussion
O protocolo atual permite imagens in vivo da dinâmica da microglia em um embrião de vertebrado e visualização dos dados de motilidade adquiridos. A colonização da micróglia do cérebro em desenvolvimento ocorre muito cedo durante a embriogênese e precede eventos críticos, como picos de neurogênese, astrogliogênese, oligodendrogênese e muitos outros processos celulares17. Portanto, não é surpreendente que a microglia desempenhe funções importantes na formação de aspectos específicos do desenvolvimento do cérebro18, por exemplo, por meio da regulação da diferenciação, migração e sobrevivência neuronal19 , 20 , 21 , bem como poda sináptica5 e mielinização22 , 23 , 24 .
A contribuição da microglia disfuncional para a patogênese e/ou progressão dos distúrbios do neurodesenvolvimento também está sendo cada vez mais reconhecida25. De fato, a presença precoce de microglia no cérebro em formação expõe essas células a estados fisiológicos distintos26 e mudanças ambientais. Isso pode ter um impacto significativo, uma vez que a microglia são células de vida longa em roedores e humanos, sendo mantidas durante a vida útil por meio da auto-renovação de progenitores locais 27,28,29. Acreditamos que este protocolo pode servir como uma ferramenta poderosa para caracterizar melhor o comportamento da microglia nesses estados fisiológicos distintos, à medida que se desenvolvem, amadurecem e estabelecem sua rede durante as etapas sucessivas da morfogênese cerebral.
Usando a configuração descrita aqui, temos imagens e adquirimos com sucesso dados sobre larvas de peixe-zebra com até 6 dpf. Estender a análise para estágios posteriores de desenvolvimento provavelmente será bem-sucedido, mas exigirá o ajuste da configuração da imagem para levar em consideração o aumento do tamanho da amostra, especialmente ao longo do eixo z. Ao tentar isso, sugerimos focar na manutenção de uma baixa relação sinal-ruído e tempo de varredura rápido, pois são parâmetros-chave para uma análise bem-sucedida.
Sugerimos um tempo mínimo de imagem de 1 h para permitir o rastreamento da microglia; A janela de imagem mais longa que foi testada com este protocolo é de 8 h. Além disso, é importante que a análise de rastreamento mantenha o intervalo de tempo entre os quadros o mais curto possível, idealmente entre 30 s e 60 s. Isso permitirá dados de rastreamento mais precisos e detalhados em análises posteriores. Portanto, especialmente se detectar mais de um fluoróforo, é fundamental evitar a sobreposição espectral e garantir separação suficiente entre os dois espectros de emissão de fluoróforos para permitir uma aquisição simultânea, sem sangramento de sinal.
Outros protocolos para gravação de lapso de tempo de alta qualidade do cérebro do peixe-zebra estão disponíveis30, mas este é o primeiro que mostra como rastrear com sucesso todo o movimento da microglia durante o desenvolvimento embrionário por um período prolongado. Embora o fluxo de trabalho apresentado aqui tenha se concentrado no rastreamento da microglia em um contexto fisiológico, ele pode ser facilmente aplicado à análise da microglia em patologia. De fato, vários modelos de distúrbios do neurodesenvolvimento, como autismo31, epilepsia32 e esquizofrenia33, mas também neurodegeneração34 e câncer35, foram estabelecidos em peixes-zebra que fornecem oportunidades únicas para determinar a resposta e o comportamento microglial em condições de doença.
Notavelmente, este protocolo de rastreamento é altamente versátil e também pode ser fundamental para lançar luz sobre os padrões de migração de vários tipos de células em diversas regiões anatômicas do embrião de peixe-zebra, abrindo assim potencialmente caminhos para aplicações adicionais, além do escopo de investigação microglial descrito neste artigo. Além disso, ao aproveitar a capacidade de combinar várias linhas transgênicas fluorescentes, ganhamos a capacidade de discernir a relação espacial entre a microglia e outros tipos de células do microambiente cerebral, com o potencial de visualizar interações celulares e conversas cruzadas ao longo de gravações de lapso de tempo, de forma não invasiva. Isso pode ser fundamental para desvendar o significado fisiológico do comportamento da microglia e contribuir para uma caracterização mais profunda dessas células altamente móveis.
Disclosures
Os autores declaram não haver conflitos de interesse.
Acknowledgements
Os autores gostariam de expressar sua sincera gratidão ao professor Nicolas Bayens por generosamente fornecer acesso ao microscópio confocal essencial para este estudo. Este trabalho foi financiado em parte pelos Fundos para Pesquisa Científica (FNRS) sob os números de concessão F451218F e UG03019F, a Alzheimer Research Foundation (SAO-FRA) (para VW), AM é apoiado por uma bolsa de pesquisa do FNRS. A Figura 1 foi criada em biorender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 1 L Breeding tanks | Tecniplast | ZB10BTE | |
| 1-phenyl-2-thiourea (PTU) | Sigma-Aldrich | P7629 | Diluted to 0.2 mM in E3 to prevent embryo pigmentation |
| Bottom glass imaging dish | FluoroDish | FD3510-100 | |
| Disposable Graduated transfer pipette | avantor | 16001-188 | |
| Dry block heater | Novolab | Grant QBD4 | To keep low melting agarose at 37 °C |
| Ethyl 3-aminobenzoate methanesulfonate (Tricaine) | Sigma-Aldrich | E10521-50G | |
| Imaris 10.0 | Oxford Instruments | analysis software | |
| Imaris File Converter | Oxford Instruments | https://imaris.oxinst.com/big-data | |
| Laser-scanning confocal microscope | Nikon | Eclipse Ti2-E | |
| Methylene blue | Sigma-Aldrich | M9140-25G | |
| microloader tips | Eppendorf | 5242956003 | |
| NuSieve GTG Agarose | Lonza | 50081 | |
| Petri dishes (90 mm) | avantor | 391-0559 | |
| Pronase | Sigma-Aldrich | 11459643001 | |
| Stainless Steel Forceps Dumont No. 5 | FineScienceTools | 11254-20 | |
| Stereo microscope | Leica | Leica M80 | To mount the embryos |
| teasing needle | avantor | 76549-024 |
References
- Wolf, S. A., Boddeke, H. W., Kettenmann, H. Microglia in physiology and disease. Annu Rev Physiol. 79, 619-643 (2017).
- Nimmerjahn, A., Kirchhoff, F., Helmchen, F. Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Sci Rep. 308 (5726), 1314-1318 (2005).
- Peri, F., Nusslein-Volhard, C. Live imaging of neuronal degradation by microglia reveals a role for v0-ATPase a1 in phagosomal fusion in vivo. Cell. 133 (5), 916-927 (2008).
- Marın-Teva, J. L., et al. Microglia promote the death of developing Purkinje cells. Neuron. 41 (4), 535-547 (2004).
- Paolicelli, R. C., et al. Synaptic pruning by microglia is necessary for normal brain development. Science. 333 (6048), 1456-1458 (2011).
- Hattori, Y. The microglia-blood vessel interactions in the developing brain. Neurosci Res. 187, 58-66 (2023).
- Colonna, M., Butovsky, O. Microglia function in the central nervous system during health and neurodegeneration. Annu Rev Immunol. 35, 441-468 (2017).
- Xu, H. -. T., Pan, F., Yang, G., Gan, W. -. B. Choice of cranial window type for in vivo imaging affects dendritic spine turnover in the cortex. Nat Neurosci. 10 (5), 549-551 (2007).
- Shin, J., Park, H. -. C., Topczewska, J. M., Mawdsley, D. J., Appel, B. Neural cell fate analysis in zebrafish using olig2 BAC transgenics. Methods Cell Sci. 25 (1), 7-14 (2003).
- Chi, N. C., et al. Foxn4 directly regulates tbx2b expression and atrioventricular canal formation. Genes Dev. 22 (6), 734-739 (2008).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. J Vis Exp. (69), e4196 (2012).
- Bohnsack, B. L., Gallina, D., Kahana, A. Phenothiourea sensitizes zebrafish cranial neural crest and extraocular muscle development to changes in retinoic acid and IGF signaling. PLoS ONE. 6 (8), e22991 (2011).
- Ariga, J., Walker, S. L., Mumm, J. S. Multicolor time-lapse imaging of transgenic zebrafish: visualizing retinal stem cells activated by targeted neuronal cell ablation. J Vis Exp. (43), e2093 (2010).
- Ferrero, G., et al. Embryonic microglia derive from primitive macrophages and are replaced by cmyb-dependent definitive microglia in zebrafish. Cell Rep. 24 (1), 130-141 (2018).
- Xu, J., Wang, T., Wu, Y., Jin, W., Wen, Z. Microglia colonization of developing zebrafish midbrain is promoted by apoptotic neuron and lysophosphatidylcholine. Dev Cell. 38 (2), 214-222 (2016).
- Svahn, A. J., et al. Development of ramified microglia from early macrophages in the zebrafish optic tectum. Dev Neurobiol. 73 (1), 60-71 (2013).
- Thion, M. S., Garel, S. Microglial ontogeny, diversity and neurodevelopmental functions. Curr Opin Genet Dev. 65, 186-194 (2020).
- Bohlen, C. J., Friedman, B. A., Dejanovic, B., Sheng, M. Microglia in brain development, homeostasis, and neurodegeneration. Annu Rev Genet. 53, 263-288 (2019).
- Squarzoni, P., et al. Microglia modulate wiring of the embryonic forebrain. Cell Rep. 8 (5), 1271-1279 (2014).
- Schafer, D. P., et al. Microglia sculpt postnatal neural circuits in an activity and complement-dependent. Neuron. 74 (4), 691-705 (2012).
- Parkhurst, C. N., et al. Microglia promote learning-dependent synapse formation through brain-derived neurotrophic factor. Cell. 155 (7), 1596-1609 (2013).
- Green, L. A., O'Dea, M. R., Hoover, C. A., DeSantis, D. F., Smith, C. J. The embryonic zebrafish brain is seeded by a lymphatic-dependent population of mrc1+ microglia precursors. Nat. Neurosci. 25 (7), 849-864 (2022).
- Hughes, A. N., Appel, B. Microglia phagocytose myelin sheaths to modify developmental myelination. Nat Neurosci. 23 (9), 1055-1066 (2020).
- Yu, Q., et al. C1q is essential for myelination in the central nervous system (CNS). iScience. 26 (12), 108518 (2023).
- Zengeler, K. E., Lukens, J. R. Innate immunity at the crossroads of healthy brain maturation and neurodevelopmental disorders. Nat Rev Immunol. 21 (7), 454-468 (2021).
- Paolicelli, R. C., et al. Microglia states and nomenclature: A field at its crossroads. Neuron. 110 (21), 3458-3483 (2022).
- Réu, P., et al. The lifespan and turnover of microglia in the human brain. Cell Rep. 20 (4), 779-784 (2017).
- Ajami, B., Bennett, J. L., Krieger, C., Tetzlaff, W., Rossi, F. M. V. Local self-renewal can sustain CNS microglia maintenance and function throughout adult life. Nat Neurosci. 10 (12), 1538-1543 (2007).
- Mildner, A., et al. Microglia in the adult brain arise from Ly-6ChiCCR2+ monocytes only under defined host conditions. Nat Neurosci. 10 (12), 1544-1553 (2007).
- Cook, Z. T., Brockway, N. L., Weissman, T. A. Visualizing the developing brain in living zebrafish using Brainbow and time-lapse confocal imaging. J Vis Exp. (157), e60593 (2020).
- Tayanloo-Beik, A., et al. Zebrafish modeling of autism spectrum disorders, current status and future prospective. Front Psychiatry. 13, 911770 (2022).
- D'Amora, M., et al. Zebrafish as an innovative tool for epilepsy modeling: State of the art and potential future directions. Int J Mol Sci. 24 (9), 7702 (2023).
- Langova, V., Vales, K., Horacek, J. The role of zebrafish and laboratory rodents in schizophrenia research. Front Psychiatry. 11, 549232 (2020).
- Chia, K., Klingseisen, A., Sieger, D., Priller, J. Zebrafish as a model organism for neurodegenerative disease. Front Mol Neurosci. 15, 940484 (2022).
- Astell, K. R., Sieger, D. Zebrafish in vivo models of cancer and metastasis. Cold Spring Harb Perspect Med. 10 (8), 037077 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved