Medição quantitativa multiplex monocromática do comprimento do telômero do PCR
* These authors contributed equally
In This Article
Summary
Aqui apresentamos um protocolo para a medição do comprimento relativo dos telômeros (TL) usando o ensaio de reação em cadeia da polimerase quantitativa multiplex monocromática (MMqPCR). O ensaio MMqPCR é um método repetível, eficiente e econômico para medir a LT do DNA humano em estudos populacionais.
Abstract
Os telômeros são estruturas ribonucleoproteicas no final de todos os cromossomos eucarióticos que protegem o DNA de danos e preservam a estabilidade cromossômica. O comprimento dos telômeros (TL) tem sido associado a várias exposições, processos biológicos e resultados de saúde. Este artigo descreve o protocolo de ensaio de reação em cadeia da polimerase quantitativa multiplex monocromática (MMqPCR) conduzido rotineiramente em nosso laboratório para medir a TL média relativa do DNA humano. Existem vários métodos diferentes de medição de LT baseados em PCR, mas o protocolo específico para o método MMqPCR apresentado nesta publicação é repetível, eficiente, econômico e adequado para estudos de base populacional. Este protocolo detalhado descreve todas as informações necessárias para que os investigadores estabeleçam este ensaio em seu laboratório. Além disso, este protocolo fornece etapas específicas para aumentar a reprodutibilidade da medição de TL por este ensaio, definido pelo coeficiente de correlação intraclasse (ICC) em medições repetidas da mesma amostra. O CCI é um fator crítico na avaliação do poder esperado para uma população de estudo específica; como tal, relatar ICCs específicos de coorte para qualquer ensaio de LT é uma etapa necessária para aumentar o rigor geral dos estudos populacionais de LT. Exemplos de resultados utilizando amostras de DNA extraídas de células mononucleares do sangue periférico demonstram a viabilidade de gerar dados TL altamente repetíveis usando este protocolo MMqPCR.
Introduction
Os telômeros são complexos protetores encontrados no final de todos os cromossomos eucarióticos, compostos por sequências de DNA repetitivas e altamente conservadas e proteínas associadas. O telômero protege a integridade do DNA, preservando a estabilidade cromossômica. O encurtamento progressivo dos telômeros ocorre na divisão das células como resultado da síntese incompleta de DNA de fita retardada, danos ao DNA e outros fatores 1,2. O aumento das evidências que apóiam o comprimento dos telômeros (LT) como um biomarcador de envelhecimento e doenças relacionadas à idade ao longo da vida humana foi acompanhado por um aumento nos tipos de ensaios de medição de TL utilizados para avaliar o papel da LT em estudos de exposição humana, doença e saúde 3,4,5. Metanálises relataram associações de LT com mortalidade geral, exposições ambientais e desfechos de saúde, incluindo câncer, doenças cardiovasculares e diabetes 6,7,8,9,10,11,12,13 . Essas meta-associações derivam de estudos que utilizam uma das mais de duas dúzias de diferentes metodologias de medição de TL, onde os pontos fortes das associações tendem a variar entre diferentes metodologias 14,15,16. Selecionar o método ideal de medição de LT para um estudo de pesquisa é uma etapa crucial para garantir resultados precisos, pois cada método possui suas próprias vantagens e desvantagens 5,17.
Devido ao custo relativamente baixo dos reagentes, rápido retorno do ensaio, escalabilidade e menor necessidade inicial de DNA, as técnicas de medição de TL baseadas em PCR são frequentemente utilizadas preferencialmente na realização de estudos com grandes populações de amostras, estudos com acesso limitado a amostras com altas concentrações de DNA ou estudos que priorizam alto rendimento. O primeiro método de medição de TL baseado em PCR, a reação em cadeia da polimerase quantitativa singleplex (qPCR) originalmente desenvolvida por Richard Cawthon, utiliza a razão dos sinais de fluorescência do telômero (T) e amplificação do gene de cópia única (S), executados em placas de PCR separadas18. Nesta abordagem, primers complementares às sequências repetidas de DNA dos telômeros (T) são usados para amplificar o conteúdo total de DNA telomérico na amostra e quantificados pela detecção do repórter fluorescente SYBR green. Da mesma forma, primers complementares a uma região intergênica de um gene conservado de cópia única (S) são usados para quantificar o número de cópias do genoma. Essas duas estimativas são quantificadas em relação a uma curva padrão de DNA genômico utilizada em todos os ensaios em um projeto para controlar a variação placa a placa. A divisão do DNA telomérico total (T) pelo número de cópias do genoma único (S) produz a razão T / S, uma medida relativa sem unidade que representa o conteúdo telomérico médio por célula para uma amostra de DNA individual18 , 19 . Assim, a relação T/S não é uma medida específica do comprimento funcional; no entanto, de acordo com as normas da literatura, utilizamos o termo TL médio por amostra em todo este protocolo.
Este método foi desenvolvido em 2009, quando Richard Cawthon descreveu o ensaio de qPCR multiplex monocromático (MMqPCR) como uma abordagem para reduzir potencialmente a variabilidade na relação T/S em relação ao método original de qPCR singleplex19. O ensaio MMqPCR possui os benefícios do ensaio qPCR com a vantagem adicional de medir os sinais T e S dentro do mesmo poço de reação usando um fluoróforo de relatório, diminuindo assim o erro em relação ao qPCR singleplex e resultando em maior precisão e reprodutibilidade19. Além disso, esse método multiplex reduz potencialmente os custos e aumenta o rendimento, uma vez que metade das reações são necessárias em comparação com o ensaio singleplex19.
Dadas as vantagens da medição de LT MMqPCR, este método é adequado para estudos populacionais de associações de LT com exposições, resultados de saúde e processos biológicos. No entanto, iniciar o método pode ser um desafio. Para enfrentar esses desafios, descrevemos, em detalhes, o protocolo de medição MMqPCR TL utilizado em nosso laboratório, destacando as principais etapas implementadas para aumentar a precisão do ensaio, diminuir o risco de contaminação e melhorar a repetibilidade.
Além disso, esse protocolo descreve etapas para limpeza de dados e cálculo do coeficiente de correlação intraclasse (CCI), uma importante medida estatística da reprodutibilidade das medidas de LT2. Com nossos resultados representativos, demonstramos a capacidade de gerar altos ICCs usando este protocolo. Além disso, identificamos o controle de qualidade (CQ) e as etapas de solução de problemas que devem diminuir a variação nas medições de TL e aumentar o ICC resultante. Devido à alta repetibilidade, eficiência e custo-benefício deste método, a medição de TL MMqPCR é ideal para pesquisas epidemiológicas de LT. A Figura 1 fornece uma visão geral do método MMqPCR conforme descrito neste protocolo.
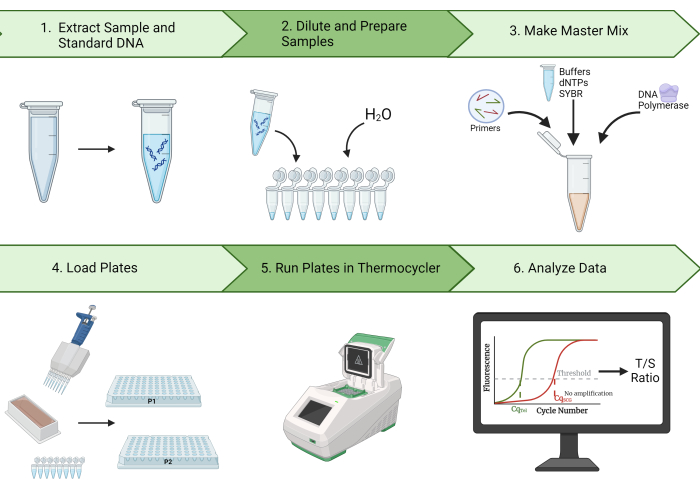
Figura 1: Visão geral do método. Uma ampla visão geral do método de reação em cadeia da polimerase quantitativa multiplex monocromática para medir o comprimento dos telômeros. Clique aqui para ver uma versão maior desta figura.
Protocol
Esta pesquisa foi realizada em conformidade com as diretrizes institucionais. Este protocolo descreve o ensaio MMqPCR conduzido a uma profundidade de medição de triplicatas duplicadas, ou seja, medições triplicadas de cada amostra repetidas em placas duplicadas, implementadas usando dois termocicladores simultaneamente para aumentar o rendimento. O uso de placas duplicadas é uma consideração primordial feita na implementação deste protocolo para alcançar alta reprodutibilidade, conforme indicado por altos ICCs. Embora seja possível usar menos repetições, o impacto no ICC e, subsequentemente, o poder e o tamanho da amostra necessários, precisam ser cuidadosamente considerados e medidos com cada coorte por cada laboratório 20,21. Se apenas um termociclador estiver disponível, sugerimos manter a profundidade de medição (ou seja, triplicados duplicados) e executar 2 placas sequencialmente.
1. Condições de preparação e armazenamento de estoque
- Para todos os materiais e equipamentos usados para este protocolo, consulte a Tabela de Materiais. Além disso, para os reagentes específicos da mistura principal e as quantidades utilizadas para o ensaio MMqPCR, ver Quadro 1. Se estiver executando duas placas sequencialmente, metade do volume da mistura principal, mantendo a concentração, garantindo que as mesmas alíquotas de reagente sejam usadas em placas sequenciais do mesmo ensaio.
NOTA: Todos os reagentes de estoque devem ser preparados com antecedência e os reagentes alíquotas devem ser completamente descongelados antes do uso. - Alicote e armazene 1x Tris-EDTA, pH 7,6 (TE) em tubos de 5 mL em volumes de 3 mL em temperatura ambiente por até 2 anos.
- Alicotar e armazenar 5 M de betaína em tubos de 5 mL em volumes de 1.280 μL a -20 °C por até 1 ano.
- Alicotar e armazenar SYBR Green em volumes de 3 μL em tubos de tiras PCR individuais a -20 °C por até 2 anos, cobertos com papel alumínio e minimamente expostos à luz.
- Armazene a DNA polimerase em tubos originais a -20 °C por até 1 ano.
- Aliquotar e armazenar tampão polimerase 10x em tubos de 1,5 mL em volumes de 660 μL a -20 °C por até 1 ano.
- Para diluições de tampão 1x de tampão polimerase 10x, adicione 9,9 μL de tampão 10x a 89,1 μL de PCR grau H2O em tubos de 0,5 mL; Conservar a -20 °C até 1 ano.
- Alicotar e armazenar 1 M MgCl2 em tubos de 0,5 mL em volumes de 70 μL a 4 °C longe da luz por até 1 ano.
- Armazene os primers liofilizados de oligonucleotídeos de telômero direto e reverso (T) e de gene de cópia única (S) em temperatura ambiente, longe da luz. As quatro sequências específicas de oligonucleotídeos são apresentadas na Tabela 2.
NOTA: O gene de cópia única neste protocolo é a albumina; se escolher um gene de cópia única diferente, as concentrações do primer podem precisar ser ajustadas para garantir uma eficiência de PCR aceitável.- Reconstituição do primer: Vórtice cada tubo de primer liofilizado por 10 s e, em seguida, coloque os tubos em uma mini centrífuga por 5 s para garantir que o pellet liofilizado esteja no fundo do tubo antes de adicionar 1x tampão TE.
- Verifique cada tubo de primer individual quanto à concentração de nM ([nM]= alfa) e reidrate cada tubo de primer adicionando 1x TE igual a 10x do valor alfa em μL (por exemplo, se a concentração de nM no tubo for de 24,6 nM, adicione 246 μL de 1x TE ao tubo de primer) para criar uma solução de 100 μM de cada primer.
- Alíquota de iniciadores de telômeros diretos e reversos separadamente em volumes de 16 μL e primers de albumina direta e reversa separadamente em volumes de 11 μL. Conservar os quatro tipos de alíquotas de primário a -20 °C durante um período máximo de 6 meses.
- Mistura dNTP: Vórtice 4-8 tubos de cada um dos quatro tipos dNTPS de soluções estoque de 25 mM (ou seja, 16-32 tubos no total) por 10 s, seguido de uma centrifugação rápida à temperatura ambiente usando uma mini centrífuga. Coloque os tubos em uma distribuição uniforme nas posições dos tubos na mini centrífuga e, em seguida, feche a tampa para permitir que a máquina atinja a velocidade máxima, girando por cerca de 5 s.
- Em um tubo de 5 mL, adicione 200-250 μL de cada tubo, garantindo que partes iguais de cada um dos quatro tipos de dNTP sejam adicionadas e faça um vórtice da mistura de dNTP.
- Alíquota de 210 μL de mistura em tubos de 0,5 mL e armazenar a -20 °C por até 1 ano.
- Armazene o estoque de DTT a -20 °C e o estoque de acetato de sódio 3 M em temperatura ambiente.
- Solução DTT: Meça 0,1545 g DTT em um barco de pesagem em uma balança usando uma espátula de aço inoxidável.
CUIDADO: O ditiotreitol (DTT) é um reagente perigoso. A TDT é prejudicial se ingerida, causa irritação na pele e pode causar sérios danos aos olhos. Os indivíduos devem usar luvas de proteção, proteção para os olhos e proteção facial ao manusear a TDT. Além disso, os indivíduos devem evitar respirar vapores de TDT, trabalhar com capuz de PCR ao manusear DTT e evitar exposição prolongada ou repetida. - Faça 10 mL de acetato de sódio 0,01 M adicionando 33,33 μL de acetato de sódio 3 M e 9.967 μL de PCR grau H2O em um tubo de 15 mL e poço de vórtice. Adicione a TDT medida no tubo de 15 ml de acetato de sódio 0,01 M. Transferir cerca de 100 μL da solução de acetato de sódio 0,01 M para o recipiente de pesagem para recolher a TDT residual. Pipete os 100 μL de volta para o tubo de 15 mL. Quando toda a TDT estiver na solução, bata o tubo até que ele se dissolva completamente.
- Despeje o conteúdo do tubo de 15 mL em uma calha de carregamento, aspire toda a solução em uma seringa de plástico de 10 mL de uma extremidade da calha de carregamento. Encaixe um filtro de seringa de acetato de celulose estéril de 0,2 μm a 25 mm na extremidade da seringa cheia, deixe saturar completamente por 1 min e pingue lentamente a solução em um novo tubo de 15 mL empurrando levemente o êmbolo da seringa para baixo, garantindo que toda a solução escorra pelo filtro para o novo tubo de 15 mL.
- Solução de alíquota DTT em volumes de 200 μL em tubos de 0,5 mL e armazenar a -20 °C por até 6 semanas.
- Solução DTT: Meça 0,1545 g DTT em um barco de pesagem em uma balança usando uma espátula de aço inoxidável.
Tabela 1: Volumes finais e concentrações de reagentes. Volumes e concentrações de reagentes em alíquotas individuais, master mix e em alvéolos de PCR. Clique aqui para baixar esta tabela.
Tabela 2: Sequências de primers de oligonucleotídeos de telômeros e genes de cópia única. Lista das sequências de primers do gene de cópia única dos telômeros e da albumina usadas na metodologia. Clique aqui para baixar esta tabela.
2. Extração de DNA genômico e preparação de amostras
- Extração de amostras analíticas: Realize uma extração de DNA genômico para amostras de acordo com as diretrizes do fabricante usando kits ou métodos estabelecidos no laboratório.
- Verificar a qualidade da amostra de ADN com um espectrofotómetro e ADN de cadeia dupla (dsDNA) utilizando um fluorómetro. Amostras com proporções médias inaceitáveis de 260/280 e 260/230 ou uma concentração de dsDNA abaixo da detecção (<0,20 ng/μL) não devem ser analisadas para TL22,23. Além disso, se a concentração de dsDNA for muito alta para gerar uma leitura usando o fluorômetro (>1000 ng/μL), dilua a amostra e repita o ensaio.
NOTA: Para os resultados aqui apresentados, os valores aceitáveis para a pureza dos ácidos nucléicos foram uma proporção de 260/280 entre 1,6 e 2,0 e uma proporção de 260/230 entre 2,0 e 2,2.
- Verificar a qualidade da amostra de ADN com um espectrofotómetro e ADN de cadeia dupla (dsDNA) utilizando um fluorómetro. Amostras com proporções médias inaceitáveis de 260/280 e 260/230 ou uma concentração de dsDNA abaixo da detecção (<0,20 ng/μL) não devem ser analisadas para TL22,23. Além disso, se a concentração de dsDNA for muito alta para gerar uma leitura usando o fluorômetro (>1000 ng/μL), dilua a amostra e repita o ensaio.
- Extração de amostras de controle: Realize uma extração de DNA genômico em uma amostra de controle derivada do mesmo material biológico que as amostras em questão que estão sendo analisadas para um determinado projeto. Certifique-se de extrair DNA de controle suficiente para todas as placas que devem ser executadas para toda a coorte, de modo que um único padrão de DNA seja utilizado para todos os ensaios.
- Verifique a qualidade da amostra de DNA de controle por espectrofotômetro e fluorômetro. A amostra de controle deve ter proporções médias aceitáveis de 260/280 e 260/230 e concentrações detectáveis de dsDNA22,23.
- Alíquota DNA de controle em uma concentração de 2 ng/μL em quantidades de 150 μL em tubos de 0,5 mL marcados com o tipo e data da amostra. Armazene alíquotas de ADN de controlo a -20 °C durante um período máximo de 5 anos. Use uma amostra de DNA de controle para todas as placas de todas as amostras da mesma coorte ou projeto de pesquisa. Prepare essas alíquotas de estoque com antecedência.
- Preparação de amostras analíticas: Para amostras com uma concentração inicial de >5 ng/μL, criar alíquotas de diluição usando um fator de diluição de número inteiro, adicionando a quantidade apropriada de amostra de DNA extraída (Arquivo Suplementar 1 MMqPCR Folha de Configuração, Coluna F) e PCR grau H2O (Arquivo Suplementar 1 MMqPCR Folha de Configuração, Coluna G) para diluir a concentração entre 2,5 - 5,0 ng/μL (Arquivo Suplementar 1 Folha de Configuração do MMqPCR, Coluna K). Não dilua amostras com uma concentração <5 ng/μL, no entanto, alíquota de volume suficiente para analisar a amostra 3x usando este protocolo.
NOTA: As alíquotas de diluição devem ser feitas durante a preparação da amostra para que 15 ng de DNA possam ser adicionados ao tubo de tira de PCR correspondente de cada amostra. As alíquotas da amostra permitem que os técnicos evitem que ciclos desnecessários de congelamento e descongelamento afetem a integridade do DNA da amostra de estoque se a amostra não passar nos critérios de qualidade e precisar ser executada novamente. O modelo MMqPCR de amostra encontrado no Arquivo Suplementar 1 pode ajudar a identificar o fator de diluição e os volumes corretos para preparar alíquotas de diluição (Arquivo Suplementar 1, Folha de Configuração do MMqPCR, Colunas EI).- Encha as tiras de PCR com amostras A1-8 na tira de PCR A, amostras B1-8 na tira de PCR B e amostras C1-8 na tira C da RPC, conforme detalhado na Folha de Configuração de Arquivo Suplementar 1 MMqPCR e listado na Tabela 3 com quantidades de amostra adequadamente diluídas (Arquivo Suplementar 1 Folha de Configuração de MMqPCR, Coluna L) e quantidades de PCR grau H2O (Arquivo Suplementar 1 Folha de configuração de MMqPCR, coluna M) para uma quantidade total de 15 ng de DNA por tubo de PCR e um volume total de 75 μL. Reserve.
NOTA: As amostras em tiras de PCR podem ser armazenadas durante a noite a 4 °C para serem plaqueadas no dia seguinte.
- Encha as tiras de PCR com amostras A1-8 na tira de PCR A, amostras B1-8 na tira de PCR B e amostras C1-8 na tira C da RPC, conforme detalhado na Folha de Configuração de Arquivo Suplementar 1 MMqPCR e listado na Tabela 3 com quantidades de amostra adequadamente diluídas (Arquivo Suplementar 1 Folha de Configuração de MMqPCR, Coluna L) e quantidades de PCR grau H2O (Arquivo Suplementar 1 Folha de configuração de MMqPCR, coluna M) para uma quantidade total de 15 ng de DNA por tubo de PCR e um volume total de 75 μL. Reserve.
- Preparação da curva padrão: Descongelar uma alíquota de DNA de controle, vórtice por 30 s, centrifugar por 5 s e aspirar o volume total (150 μL) para o primeiro tubo na tira de tubo de PCR da curva padrão (SC).
- Pipetar 70 μL de PCR grau H2O no segundo ao oitavo tubos da tira de PCR SC. Ressuspenda completamente o DNA de controle no primeiro tubo antes de aspirar 70 μL e dispensá-lo no segundo tubo de PCR. Aguarde 30 s e, em seguida, ressuspenda completamente a solução no segundo tubo de PCR antes de aspirar 70 μL e dispensá-la no terceiro tubo de PCR. Repita isso para os tubos de três a sete para criar uma diluição serial de 2 vezes de sete padrões. O tubo final deve conter apenas PCR grau H2O para funcionar como um controle não molde (NTC). Coloque a tira de PCR SC de lado.
Tabela 3: Organização das amostras e padrão de controle em placa. Localização de todas as amostras e padrões em uma placa de PCR de 96 poços. Clique aqui para baixar esta tabela.
3. Preparação da mistura mestra de MMqPCR
- Recolher as alíquotas de reagentes da mistura principal enumeradas no quadro 1, excepto a ADN polimerase, e deixá-las atingir a temperatura ambiente no interior da cabina de PCR. Adicione 1 μL da alíquota SYBR Green à alíquota 1x tampão.
NOTA: Essa quantidade exata é necessária para gerar a concentração correta de SYBR devido às limitações de precisão da pipeta durante o ensaio, portanto, mesmo que apenas uma placa seja executada por vez, essa quantidade deve permanecer inalterada. - Vortex todas as alíquotas por 10 s, centrifugue por 5 s e, em seguida, adicione as seguintes quantidades de reagentes ao tubo de 5 mL que contém a betaína 5 M (agora o tubo master mix): 1.235,2 μL de PCR grau H2O, 640 μL do tampão 10x, 204,8 μL dos dNTPs, 192 μL do DTT, 64 μL de MgCl2, 48 μL de SYBR Green / tampão 1x (da etapa 3.1), 14,4 μL de ambos os primers de telômeros e 9,6 μL de ambos os primers de albumina, conforme listado na Tabela 1.
- Retire a DNA polimerase do armazenamento de -20 °C, vórtice por 10 s, centrifugue por 5 s e, em seguida, adicione lentamente 128 μL à mistura principal; mova imediatamente a DNA polimerase de volta para armazenamento a -20 °C.
- Vortex o tubo master mix de 5 mL por 30 s.
4. Preparação de placa de 96 poços
- Coloque duas placas de 96 poços, calha de carregamento e ponteiras de pipeta multicanal dentro da coifa de PCR. Rotule cada placa com o número da placa (1 ou 2). Gire a placa um (a placa P1) 180 ° para que os números das colunas fiquem de cabeça para baixo. Deixe a placa dois (P2) voltada para o técnico.
- Despeje o conteúdo do tubo de mistura principal de 5 mL na calha de carregamento. Use uma ponta de pipeta para coletar e dispensar qualquer solução restante no tubo.
- Carregue as ponteiras na pipeta multicanal, empurrando a base de cada ponteira para garantir uma vedação hermética. Verifique se as pontas não possuem nenhum filtro dentro da região de saída onde a mixagem master será aspirada; Se houver um pedaço de filtro, substitua a ponta.
- Use pipetagem reversa para encher as placas com a mistura principal viscosa, gire a calha de carregamento e pressione o êmbolo da pipeta após a primeira parada, aspirando mais de 15 μL nas pontas da pipeta, certificando-se de que as pontas sejam preenchidas ao mesmo tempo com o mesmo volume. Ao expelir a mistura principal em uma coluna, pressione o êmbolo até a primeira parada, deixando a mistura principal extra nas pontas da pipeta. Deixando o êmbolo pressionado, mergulhe as pontas de volta na calha de carregamento e solte o êmbolo para encher as pontas com mais 15 μL. Use as mesmas pontas para encher as duas placas.
- Encha as placas expelindo a mistura principal em todas as colunas pares ou ímpares primeiro, alternando entre as placas (ou seja, se o técnico optar por começar com números de coluna ímpares, a coluna 1 em P2 será preenchida primeiro, seguida pela coluna 11 em P1, depois pela coluna 3 em P2, etc.). Depois que o técnico tiver preenchido todos os poços da série (ímpar ou par) trabalhando da esquerda para a direita, faça a outra série (par ou ímpar) de colunas da mesma maneira. Consulte a Figura 2 para obter um gráfico desse processo.
NOTA: Esta maneira de enchimento de placas funciona para controlar os efeitos de posição nas placas.
- Encha as placas expelindo a mistura principal em todas as colunas pares ou ímpares primeiro, alternando entre as placas (ou seja, se o técnico optar por começar com números de coluna ímpares, a coluna 1 em P2 será preenchida primeiro, seguida pela coluna 11 em P1, depois pela coluna 3 em P2, etc.). Depois que o técnico tiver preenchido todos os poços da série (ímpar ou par) trabalhando da esquerda para a direita, faça a outra série (par ou ímpar) de colunas da mesma maneira. Consulte a Figura 2 para obter um gráfico desse processo.
- Vortex as quatro tiras de PCR fechadas contendo as amostras e a curva padrão e, em seguida, gire cada uma em uma mini centrífuga por 5 s.
- Alinhe as tiras de PCR no suporte de tubos de PCR na ordem em que serão colocadas na placa. Para P1, as primeiras 3 colunas são amostras (tira de PCR A), as colunas 4-6 são o padrão (tira de PCR S), as colunas 7-9 são amostras (tira de PCR B) e as colunas 10-12 são amostras (tira de PCR C; ver Figura 3 e Tabela 3). Como P2 é preenchido enquanto girado 180° em relação ao técnico, as colunas serão o oposto.
- Gire ambas as placas 180° para que os números das colunas sejam invertidos em relação ao técnico para cada placa (ou seja, se o técnico estava voltado para a etiqueta P2 anteriormente, agora ele estará voltado para a etiqueta P1 na borda da placa).
- Defina a pipeta multicanal para 10 μL e carregue as pontas da pipeta na pipeta multicanal conforme descrito na etapa 4.3.
- Use a pipeta multicanal para ressuspender as soluções e, em seguida, execute a pipetagem reversa conforme descrito na etapa 4.4 para aspirar e dispensar 10 μL das amostras e soluções padrão, começando com a tira de PCR A.
- Preencha as 3 primeiras colunas de cada placa usando a tira PCR A, alternando as colunas de maneira semelhante à que a mistura principal foi colocada na placa (ou seja, par ou ímpar).
- Preencha as próximas três colunas usando a tira de PCR de curva padrão. Para esta tira, ressuspenda o7º tubo de diluição padrão (penúltimo), usando uma pipeta de 200 μL regulada para ~90 μL, completamente antes de ressuspender toda a tira usando a pipeta multicanal.
NOTA: O maior volume e a menor concentração de DNA neste tubo frequentemente resultam em medições variadas nos poços da placa de PCR devido à má mistura. Usar um volume maior para misturar este tubo homogeneíza melhor a solução de DNA. - Preencha as próximas três colunas com a tira de PCR B e as três últimas com a tira de PCR C. Recoloque as pontas da pipeta entre cada tira de PCR, preenchendo todos os 96 poços em ambas as placas com as amostras correspondentes e o padrão de controle, conforme descrito na Figura 3.
- Depois que as placas estiverem cheias, bata suavemente na placa na bancada para garantir que o líquido nas laterais dos poços escorra para o fundo e, em seguida, cubra os topos das placas com filmes de vedação. Use as pontas dos dedos para pressionar todas as bordas do filme para garantir uma vedação firme.
- Misture as placas girando na parte superior do capô por 30 s e, em seguida, coloque as placas seladas no girador de placas por 2 min com as aberturas do poço voltadas para o centro.
- Coloque as placas em um termociclador, com os números das colunas das placas em ordem legível. Use um lenço de papel limpo para limpar a parte superior de cada placa antes de fechar a parte superior do termociclador.
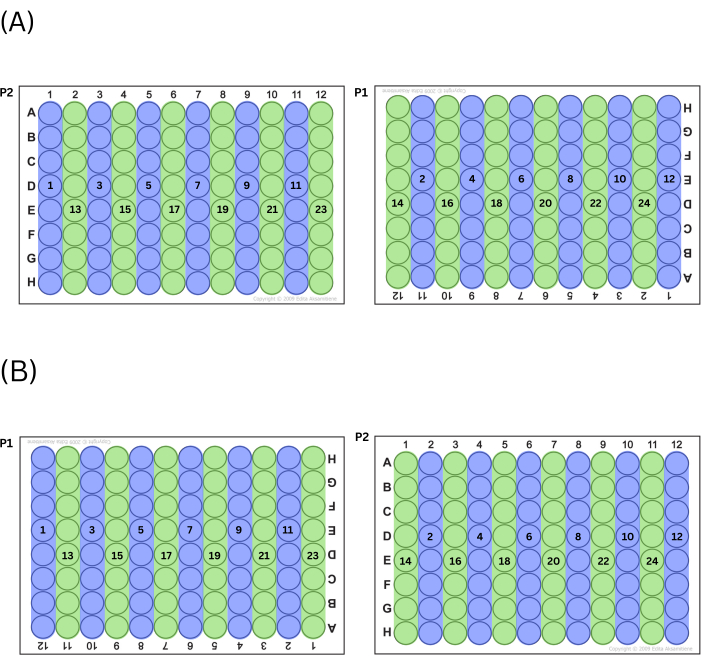
Figura 2: Processo de enchimento de placas. (A) Se optar por preencher colunas ímpares primeiro, esta é a ordem em que os poços são preenchidos. (B) Se optar por preencher colunas pares primeiro, esta é a ordem em que os poços são preenchidos. Clique aqui para ver uma versão maior desta figura.
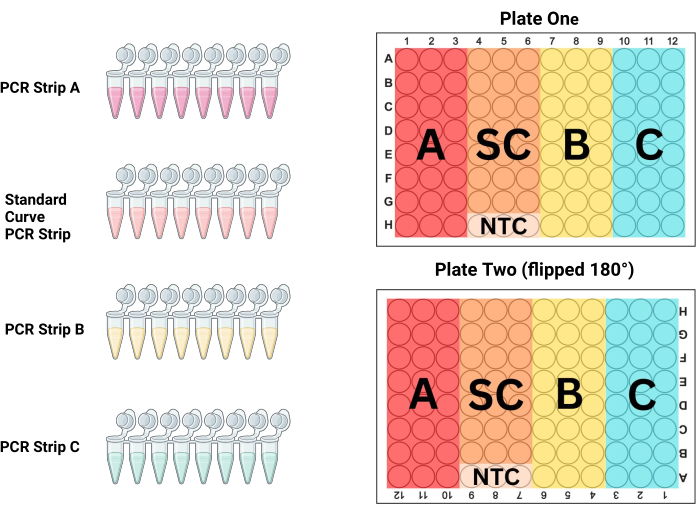
Figura 3: Layout da placa. A tira de PCR A, a tira B, a tira C e a tira de curva padrão (SC) devem ser usadas para preencher três colunas em cada placa para produzir triplicados duplicados de cada amostra e diluição padrão. Este diagrama mostra qual das colunas deve ser preenchida com cada faixa. A placa dois é virada 180° (observe que os cabeçalhos das colunas e linhas estão de cabeça para baixo) antes do carregamento, mas a placa é preenchida de forma idêntica à placa um, eliminando possíveis erros de pipetagem e ainda controlando os efeitos de posição nas placas. Clique aqui para ver uma versão maior desta figura.
5. Termociclagem MMqPCR
- Enquanto as placas estão girando na etapa 4.8.1, ligue o computador e os termocicladores. Abra o software termociclador. Este protocolo descreve o uso do software CFX Maestro.
- Crie o protocolo de termociclagem TL de acordo com o protocolo de termociclagem MMqPCR19 (Figura 4).
- Adicione uma etapa de incubação para ativar a DNA polimerase a 95 °C por 15 min.
- Para evitar a ligação primer-dímero, execute dois ciclos de 94 ° C por 15 s, 49 ° C por 1 min, depois 3 ciclos de 94 ° C por 15 s, 59 ° C por 15 s.
- Para amplificação dos telômeros, adicione 27 ciclos de 85 ° C por 15 s, 74 ° C por 30 s e, em seguida, aquisição de sinal.
- Para amplificação de albumina, adicione 31 ciclos de 94 ° C por 15 s, 84 ° C por 30 s e, em seguida, aquisição de sinal.
- Incluir uma curva de fusão de 59 °C a 95 °C em intervalos de 5 s para cada grau crescente no protocolo de ciclagem térmica.
- Clique em Iniciar execução para ambos os termocicladores. Quando solicitado, forneça um título para os arquivos de análise.
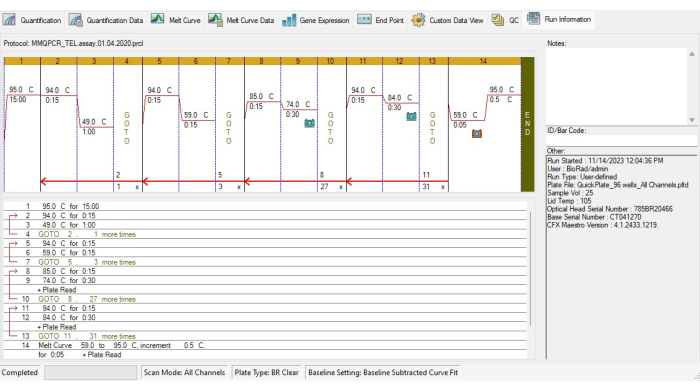
Figura 4: Perfil de termociclagem do ensaio MMqPCR. Protocolo MMqPCR criado no software de acordo com o protocolo original de termociclagem19. Clique aqui para ver uma versão maior desta figura.
6. Análise de dados de MMqPCR
- Quando o termociclo estiver concluído, analise os dados da seguinte maneira para produzir valores de TL para as amostras que foram processadas.
- No software, selecione o recurso Configuração de placa . No menu suspenso, clique em Exibir/Editar chapa.
- Realce todos os poços e clique em Selecionar fluoróforos, marque a caixa SYBR e desmarque todas as outras caixas. Clique em OK.
- Enquanto todos os poços ainda estão destacados, ao lado da palavra Load, marque a caixa ao lado de SYBR. Agora todos os poços devem ter SYBR escrito neles.
- Em seguida, destaque os três poços NTC na parte superior ou inferior da diluição serial de controle padrão e selecione NTC no menu de tipo de amostra à direita. Os três poços agora devem ser amarelos e chamados de NTC. A Figura 5A mostra quais poços devem ser selecionados para P1 e a Figura 5B mostra quais poços devem ser selecionados para P2.
NOTA: P2 será invertido de P1, então o NTC estará na parte inferior das colunas 4-6 para P1 e no topo das colunas 7-9 para P2. - Realce os 21 poços da diluição serial de controle padrão e selecione Padrão no menu do tipo de amostra. Esses poços devem ser verdes. Enquanto esses poços ainda estiverem destacados, clique em Replicação técnica. Selecione 3 no menu Tamanho da replicação e, em seguida, selecione Horizontal > Aplicar. Esses poços devem ser rotulados em conjuntos de três de Std-1 a Std-7.
- Enquanto o padrão ainda estiver realçado, role para baixo e selecione Série de diluição. No campo Fator de diluição, insira 2 e digite a concentração inicial de diluição e a direcionalidade de acordo com o número da placa, conforme descrito nas etapas a seguir.
- Para a placa P1, insira 2.00E-03 no campo de concentração inicial e marque a caixa Decrescente e selecione Aplicar. Os valores nos 21 poços devem ter os valores de concentração escritos em cada poço variando de 2,00E-03 a 3,13E-05 de cima para baixo.
- Para a placa P2, insira 3.13E-5 no campo de concentração inicial e marque a caixa Aumentando e selecione Aplicar. Os valores nos 21 poços devem ter os valores de concentração escritos em cada poço variando de 3,13E-05 a 2,00E-03 de cima para baixo.
- Para as amostras, realce as colunas da tira de PCR A (1-3 para P1 e P2), selecione Desconhecido no menu de tipo de amostra e, em seguida, selecione Replicação técnica. Selecione 3 no menu Tamanho da replicação e, em seguida, selecione Horizontal > Aplicar. Os poços para essas colunas devem ser azuis e rotulados por linha em conjuntos de 3 de Unk-1 a Unk-8.
- Repita a etapa 6.3 para colunas para a tira de PCR B (colunas 7-9 para P1 e 4-6 para P2). Eles devem ser rotulados como Unk-9 a Unk-16.
- Repita a etapa 6.3 para colunas para a tira de PCR C (colunas 10-12 para P1 e P2). Eles devem ser rotulados como Unk-17 a Unk-24. Quando as etapas 6.2-6.3.2 forem concluídas, as janelas de configuração da placa devem ser semelhantes à Figura 5A,B.
- Selecione OK no canto inferior direito da janela do editor de placas. Clique em Sim para aplicar as alterações quando solicitado.
- Para CQ em nível de ensaio, certifique-se de que as curvas na guia de quantificação sejam apropriadas (por exemplo, sem curvas de amplificação invertidas) para a Etapa 9 e a Etapa 12 do perfil de termociclagem, que correspondem aos amplicons do telômero e da albumina, respectivamente. As curvas são acessadas pelo menu suspenso no meio do lado direito da janela do software. A Figura 6A, B exibe as curvas apropriadas.
- Certifique-se de que as eficiências de PCR relatadas na Etapa 9 e na Etapa 12 estejam entre 90% e 110% e que essas duas eficiências não difiram uma da outra em mais de 10% (ou seja, 92,4% para a Etapa 9 e 98% para a Etapa 12 são apropriados, mas 92,4% para a Etapa 9 e 108% para a Etapa 12 não são apropriados). Além disso, certifique-se de que a curva R2 padrão seja >0,995. Se P1 ou P2 não atenderem a esses critérios, as placas precisam ser executadas novamente.
- Para P1, selecione a Etapa 9 e destaque os 21 poços correspondentes à curva padrão, conforme visto na Figura 6A,B. Copie os valores Cq no canto inferior direito da janela do software. Abra o modelo de folha de dados do telômero disponível como Arquivo Suplementar 1 e cole esses valores na planilha padrão 1, coluna B. Selecione a Etapa 12 e copie esses valores Cq. Cole esses valores na planilha padrão 1, coluna I.
- Identifique o valor da inclinação na Etapa 9 e digite-o na célula C4 na planilha padrão 1. Identifique o valor da inclinação na Etapa 12 e digite-o na célula J4 na planilha padrão 1.
NOTA: O sistema calcula a eficiência percentual dos primers usando as inclinações da curva padrão identificadas na etapa 6.6.1. A porcentagem de eficiência pode ser calculada manualmente usando a seguinte fórmula: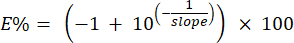
- Assegurar que o coeficiente de variação (CV) para cada triplicado de diluição-padrão é inferior a 0,1. Se for superior ou igual a 0,1, excluir até três alvéolos individuais de todas as concentrações de amostras de controlo incluídas na curva de diluição seriada padrão de 7 pontos na placa. Apenas um poço deve ser excluído de qualquer conjunto triplicado.
NOTA: A exclusão de pontos padrão da análise no software alterará a inclinação, R, interceptação y e eficiência relatadas para ambos os primers. Assim, depois que qualquer poço padrão é removido da análise, as etapas 6.5.1-6.6.1 devem ser repetidas. - Repita as etapas 6.6.1 a 6.6.2 para o arquivo de análise P2 e a folha padrão 2 correspondente.
- Somente quando as folhas padrão 1 e padrão 2 forem preenchidas com currículos aceitáveis, colete dados de amostra individuais dos arquivos de análise. Certifique-se de que os ajustes de CQ sejam relatados na coluna L da planilha P1 v P2, por exemplo, quantos poços foram excluídos no arquivo de análise P1.
- Identifique o valor da inclinação na Etapa 9 e digite-o na célula C4 na planilha padrão 1. Identifique o valor da inclinação na Etapa 12 e digite-o na célula J4 na planilha padrão 1.
- Certifique-se de que apenas os 72 poços de amostra no arquivo de análise P1 estejam destacados em azul na guia Quantificação, conforme visto em Figura 7A,B. Selecionar Passo 9. Copie os valores Cq e SQ no canto inferior direito da janela do software. Cole esses valores na planilha P1 de amostras, começando na célula D3. Selecionar Passo 12 em seguida, copie esses valores Cq e SQ e cole na folha P1 de amostras, começando na célula F3.
- Repita esta etapa para as amostras no arquivo de análise P2 e na folha P2 das amostras correspondentes. As relações telômero para cópia única (T/S) (coluna H) são calculadas automaticamente, mas podem ser calculadas manualmente usando a seguinte fórmula:
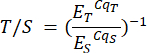
Em que ET/S é a eficiência da amplificação exponencial para reações direcionadas ao telômero ou ao gene de cópia única, respectivamente, e CqT/S é o ciclo no qual um determinado replicado direcionado ao conteúdo telomérico ou o gene de cópia única atinge o limiar crítico de quantificação de fluorescência. As relações T/S médias (coluna I) são calculadas em seis medições por amostra.
NOTA: Os números de identificação da amostra no CFX não correspondem à mesma amostra biológica nas duas placas. O modelo de folha de dados dos telômeros leva em conta essa diferença e alinhará os triplicados duplicados apropriados. - No CFX Maestro, verifique se os valores de Cq para todas as amostras analíticas estão entre o valor de Cq mais baixo e o mais alto da curva padrão, como visto na Figura 7B. Se uma amostra estiver fora do intervalo, conforme visto na Figura 7A, ela precisará ser executada novamente após o ajuste da concentração. Uma amostra com um valor de Cq inferior ao padrão de concentração mais alto deve ser diluída por um fator aproximado ao número de ciclos de amplificação que está fora da faixa, e uma amostra com um valor de Cq superior ao padrão de concentração mais baixo precisaria ser concentrada por um fator aproximado ao número de ciclos de amplificação além do limiar.
NOTA: A grande variação na concentração de amostras analíticas introduz um nível potencial adicional de variação e deve ser feita com cautela. - Certifique-se de que a concentração inicial no NTC seja inferior a 5% da quantidade média de DNA presente nos poços de amostra. Isso pode ser verificado visualizando a % de contaminação da água nas amostras P1 e amostras P2 folhas do MMqPCR. Se o NTC indicar contaminação, execute novamente as placas.
NOTA: Devido à sensibilidade do ensaio MMqPCR, uma pequena contaminação incontrolável pode ocasionalmente fazer com que os poços NTC se amplifiquem. No entanto, essa amplificação deve ser menor e ocorrer vários ciclos após a amplificação do gene de cópia única na Etapa 12 do protocolo de termociclagem. - Para CQ em nível de amostra, revise os desvios padrão (SD) (Coluna J) e VCs (Coluna K) nas amostras P1 e amostras de Folhas P2. Certifique-se de que os CVs intraplaca nas relações T/S triplicadas da amostra sejam inferiores a 0,10 (10%). Para corrigir quaisquer CVs intraplaca maiores que 0,1, uma relação T/S de um conjunto de triplicatas pode ser excluída.
NOTA: Apenas uma réplica total nas seis medições por amostra pode ser excluída, ou seja, uma réplica para a mesma amostra não pode ser excluída em P1 e P2. - Verifique se os CVs interplacas de amostra individuais na folha P1 v P2, coluna G, são inferiores a 0,05 (5%). Para corrigir quaisquer CVs maiores que 0,05, execute a exclusão de uma relação T/S das seis medições por amostra em ambas as placas. Exclua as amostras após inspeção visual em triplicados intraplaca e todas as seis medições, conforme necessário.
NOTA: Somente amostras que passam por esses critérios de CQ, CV intraplaca < 10% e CV interplaca < 5%, podem ser incluídas nos resultados finais da LT. Caso contrário, a amostra deve ser excluída das folhas de dados P1 v P2 e ICC para esta corrida, e a amostra deve ser reavaliada em um segundo ensaio MMqPCR. - Para CQ em nível de placa, verifique se a variação média intraplaca em todas as amostras em cada placa, localizada na parte inferior das amostras P1 e folhas de amostras P2, é inferior a 0,05 (5%). Para CQ adicional no nível da placa, verifique se a variação geral entre placas na folha P1 v P2, célula G29, é inferior a 0,06 (6%). Certifique-se de que as amostras que não passaram no CQ sejam removidas do cálculo do CV entre placas na célula G30.
NOTA: Ao trabalhar com grupos de amostra relacionados (por exemplo, membros da família, pontos de tempo diferentes, etc.), todas as amostras em um grupo precisam ser executadas na mesma placa. Portanto, se uma amostra do grupo não passar nos critérios de CQ, todas as amostras do grupo deverão ser executadas novamente. - Em um arquivo de dados de coorte ou experimento geral separado, registre os dados finais de TL para cada amostra que passou no CQ (Folha P1 v P2, Coluna I), os cálculos de ICC para cada amostra que passou no CQ (Folha de Dados ICC, Colunas EK) e os dados finais de CQ da Folha P1 v P2 (Colunas JL)
- Repita esta etapa para as amostras no arquivo de análise P2 e na folha P2 das amostras correspondentes. As relações telômero para cópia única (T/S) (coluna H) são calculadas automaticamente, mas podem ser calculadas manualmente usando a seguinte fórmula:
- Use o Arquivo Suplementar 2 criado pela Telomere Research Network (TRN) para calcular o ICC para o projeto20.
- Por fim, para melhorar a comparabilidade entre os estudos de LT, transforme os dados finais de TL para cada amostra na coorte geral separada ou no arquivo de dados do experimento em pontuações Z usando a seguinte equação, em que a razão T / S média para todas as amostras da coorte é subtraída da relação T / S da amostra individual e dividida pelo DP em todas as amostras da coorte.
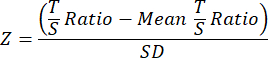
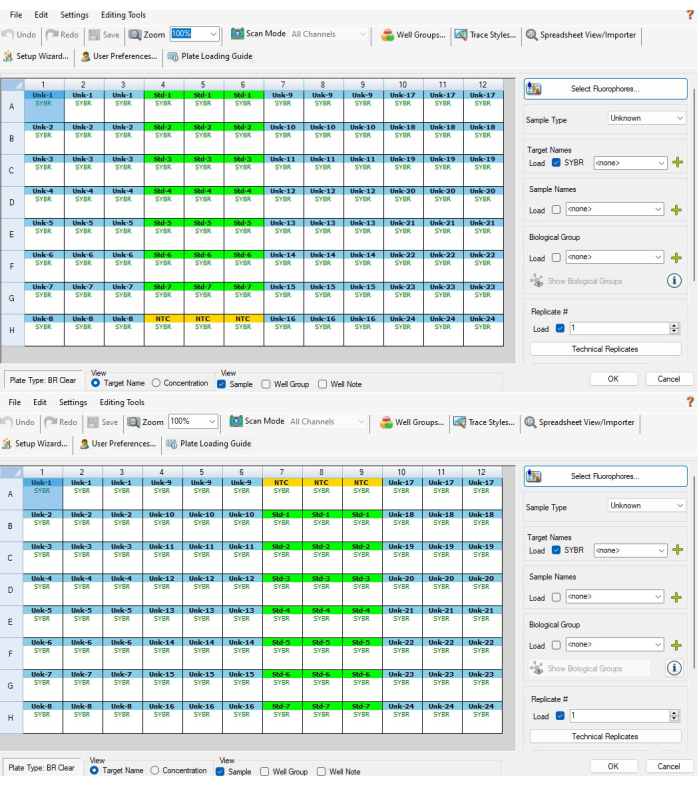
Figura 5: Configuração de placa baseada em software. (A) Configuração de placa baseada em software para uma placa P1 após concluir as etapas 6.2-6.4. (B) Configuração de chapa baseada em software para uma chapa P2 após concluir as etapas 6.2 a 6.4. Os IDs de amostra e padrão não estão alinhados entre as duas placas CFX devido à maneira como o software atribui IDs de amostra com base na posição do poço (ou seja, a amostra CFX 1 em P1 é a amostra CFX 24 em P2). O modelo do Excel fornecido no Arquivo Suplementar 1 leva isso em consideração, garantindo que as medições entre placas feitas na mesma amostra biológica estejam devidamente alinhadas umas com as outras Clique aqui para ver uma versão maior desta figura.
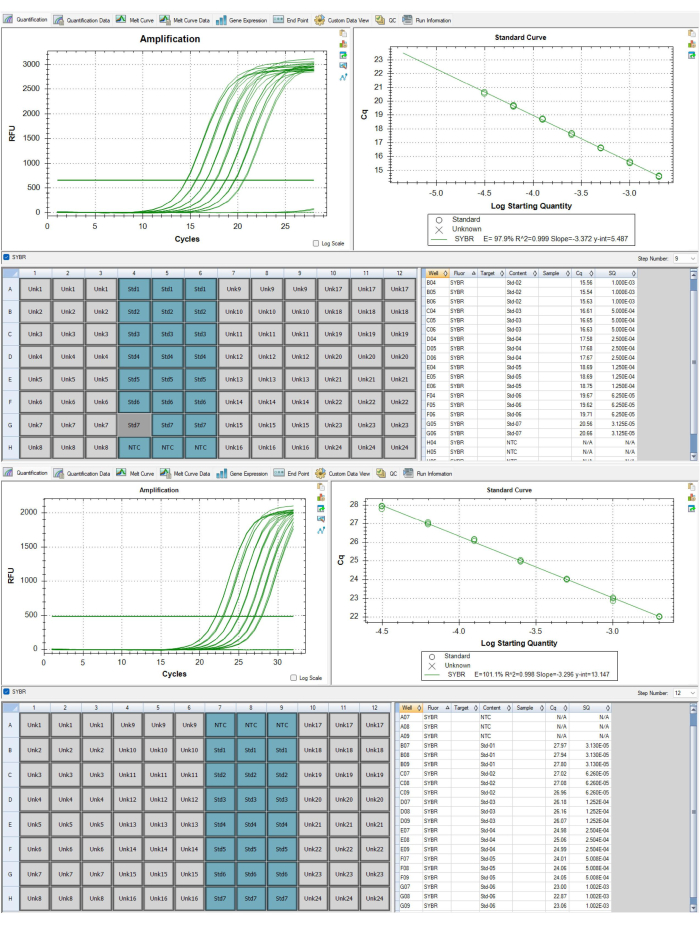
Figura 6: Curvas padrão para amplicons deelômeros t e albumina. (A) Esta curva padrão é do amplicon de telômeros P1 do conjunto de dados de resultados representativos. Um padrão foi removido porque não atendia aos critérios de CQ. (B) Esta curva padrão é do amplicon de albumina P2 do conjunto de dados de resultados representativos. Clique aqui para ver uma versão maior desta figura.
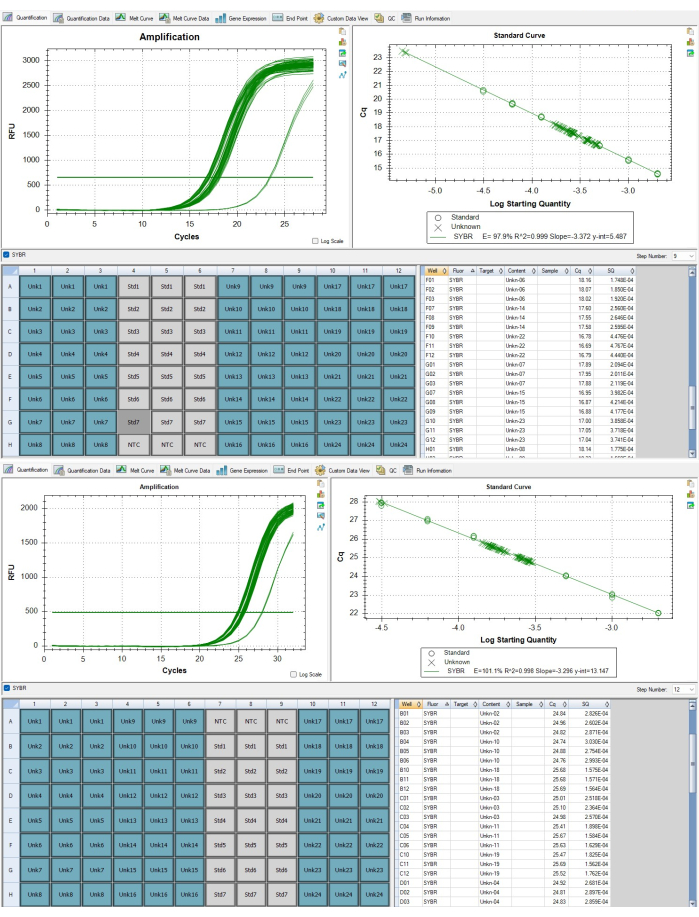
Figura 7: Telômeros e amplificadores de genes de cópia única. (A) Amplificação dos telômeros e (B) amplificação do gene da albumina de amostras relatadas em resultados representativos exibidos no software. Clique aqui para ver uma versão maior desta figura.
7. Relatório de dados MMqPCR
- Ao relatar os resultados da pesquisa usando os dados TL criados a partir do MMqPCR, certifique-se de relatar os itens descritos nas Diretrizes Mínimas de Relatórios do TRN. Essas diretrizes podem ser encontradas no site da TRN (https://trn.tulane.edu/resources/reporting-guidelines/) e um modelo para relatar essas informações é fornecido no Arquivo Suplementar 3. Cite este protocolo e outras diretrizes e recursos da TRN, quando apropriado.
Representative Results
Os resultados apresentados na Tabela 4 e na Tabela 5 oferecem um exemplo de medições de TL altamente repetíveis obtidas seguindo o protocolo. Para esses resultados, o DNA foi extraído de 24 amostras de células mononucleares do sangue periférico (PBMC) usando um kit comercial de acordo com as diretrizes dos fabricantes. Essas 24 amostras foram executadas em duas placas de 96 poços. Todas as amostras de DNA foram verificadas quanto à qualidade por meio de espectrofotômetro e fluorômetro, usando a proporção média de 260/280, proporção média de 260/230 e concentração de dsDNA para determinar a elegibilidade do ensaio e o fator de diluição da amostra (Tabela 6). A Tabela 6 também destaca a importância de quantificar a concentração de dsDNA, que pode diferir da concentração de DNA medida usando um espectrofotômetro. Essa variabilidade é resultado de diferentes abordagens de quantificação. Especificamente, o espectrofotômetro deriva a concentração de DNA com base na absorção a 280 nm e é suscetível a flutuações devido a contaminantes (por exemplo, proteína, sal, etc.) que afetam as leituras de absorbância. Por outro lado, as concentrações de dsDNA medidas usando um fluorômetro são determinadas pela fluorescência de um corante que se liga especificamente ao dsDNA e, como tal, presume-se que seja um reflexo mais preciso do conteúdo de DNA. As amostras de DNA experimentaram até três ciclos de congelamento e descongelamento antes da análise de TL MMqPCR. O DNA de controle foi criado a partir de extrações de DNA agrupadas de PBMCs de um indivíduo, que foi usado para criar uma diluição serial de sete pontos de 2 ng / μL a 0,0313 ng / μL de DNA. As curvas padrão independentes criadas para a Etapa 9 da PCR (amplicon dos telômeros) e Etapa 12 (amplicon do gene de cópia única, albumina neste protocolo) são apresentadas na Figura 6A,B. Os ensaios foram executados em um sistema comercial de detecção de PCR em tempo real, que gerou uma curva de fusão que mostra os produtos individuais do amplicon sendo produzidos em temperaturas distintas, conforme visto na Figura 8.
Tabela 4: Dados de saída da medição de MMqPCR para resultados representativos ideais. Razões T/S médias, DPs, CVs e TL com pontuação Z para 24 amostras. Clique aqui para baixar esta tabela.
Tabela 5: Saída de dados do comprimento dos telômeros. Os dados brutos do ensaio MMqPCR são analisados no software e, em seguida, adicionados ao modelo de planilha anexado como Arquivo Suplementar 1 para análise posterior e controle de qualidade. Esta planilha de saída do modelo TL apresenta um resumo dos dados, exibindo IDs de amostra, TLs médios, SDs e CVs para cada amostra em ambas as placas. Clique aqui para baixar esta tabela.
Tabela 6: Métricas de qualidade de DNA de espectrofotômetro e fluorômetro para resultados de amostra. Dados de controle de qualidade da análise do espectrofotômetro duplicado e medição do fluorômetro singular do dsDNA e de quaisquer contaminantes por amostra. Clique aqui para baixar esta tabela.
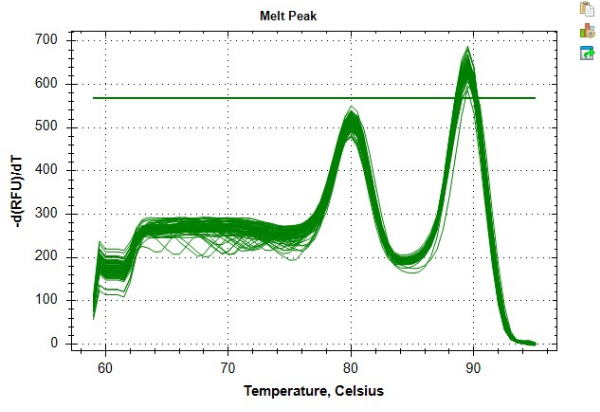
Figura 8: Exemplo de curva de fusão. Curva de fusão para dados de amostra conforme gerados no software. O pico anterior ~ 80 ° C representa o amplicon dos telômeros e o pico secundário a ~ 89 ° C representa o amplicon de albumina. Clique aqui para ver uma versão maior desta figura.
Para este projeto, a eficiência média dos telômeros foi de 98,6% com um intervalo de 90,7 a 102,1 e a eficiência média da albumina foi de 102,3% com um intervalo de 93,6 a 108,2 em todas as execuções. Houve uma média de 0,97 repetições removidas das curvas padrão, o que atende aos critérios de CQ deste protocolo. A média da VC interplacas foi de 1,83% com um DP de 0,00616 e a média da VC intraplaca foi de 3,78% com um DP de 0,00658. As TLs médias para essas amostras representativas são apresentadas na Tabela 4 e na Tabela 5. A média de TL foi de 1,37 com DP de 0,24 e variação de 0,84 a 2,32. A relação T/S, DP, CV e TL com escore Z por amostra estão listadas na Tabela 4. Como a saída da relação T/S do ensaio MMqPCR é uma medida relativa de TL, as proporções foram transformadas neste escore Z para permitir a comparação entre os estudos. O ICC para este projeto foi calculado usando o script R, conforme descrito pelas diretrizes TRN encontradas no Arquivo Suplementar 2, contabilizando os efeitos de lote e execução20. Para calcular o ICC intra-projeto, executamos novamente 10% das amostras aprovadas, certificando-nos de preencher as placas ICC com pelo menos uma amostra de cada placa da coorte. O ICC geral do projeto de 0,801 [IC: 0,703, 0,86] indica a alta reprodutibilidade dos resultados da LT.
Nem todos os resultados serão ótimos. Os resultados na Figura 9 e na Tabela 7 mostram resultados abaixo do ideal do ensaio MMqPCR. A Figura 9 exibe uma curva padrão com uma eficiência de telômero abaixo de 90%, que está abaixo dos padrões de CQ, exigindo que toda a placa seja repetida. Problemas com eficiências de primer geralmente são devidos a um problema com reagentes, por isso é importante rastrear as datas em que os reagentes são aliquotados e quando eles expiram como um primeiro passo para determinar qual reagente é responsável pela baixa eficiência. Os reagentes vencidos devem ser substituídos antes que a placa seja executada novamente. A Tabela 7 exibe uma placa que passou nos critérios iniciais de CQ em nível de amostra, mas tem um alto nível de variabilidade entre as placas, levando à falha de CQ em nível de placa. A variação entre as placas geralmente se deve a erros nas técnicas de pipetagem e enchimento de placas. Nesse caso, o técnico deve avaliar quaisquer problemas ocorridos durante a configuração da placa e garantir que as pipetas estejam calibradas.
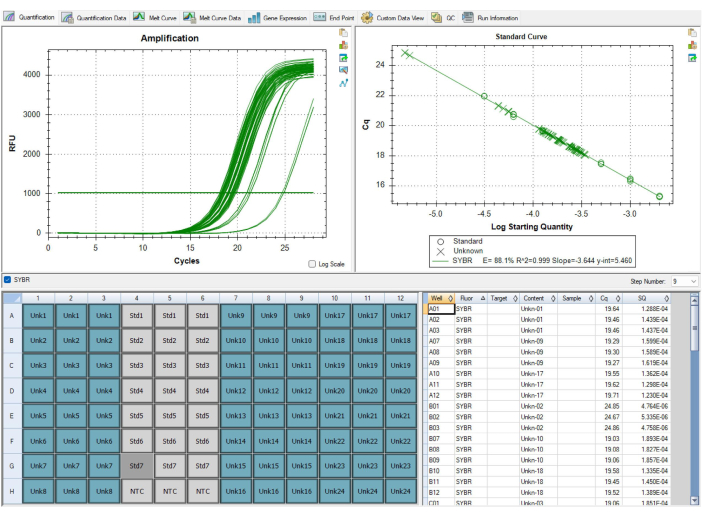
Figura 9: Resultados abaixo do ideal. Essa curva padrão exibida no software não passou no controle de qualidade, uma vez que a eficiência de amplificação para o primer telômero, foi inferior a 90%. A imagem é de P1, mas P2 teve eficiências igualmente baixas. Os dados dessa execução não puderam ser usados e todas as amostras precisaram ser executadas novamente após a substituição do reagente causal que estava vencido. Clique aqui para ver uma versão maior desta figura.
Tabela 7: Resultados abaixo do ideal. A tabela exibe o modelo de dados de telômero para uma execução em que muitas das amostras não atenderam aos padrões de CQ. As amostras que precisavam ser executadas novamente foram determinadas com base nos valores CV e, em seguida, o nome da amostra foi alterado para uma fonte vermelha para facilitar a identificação. Clique aqui para baixar esta tabela.
Arquivo suplementar 1: Modelo de planilha de dados de telômero. Clique aqui para baixar este arquivo.
Arquivo Suplementar 2: Cálculo do ICC. Este protocolo foi criado pela Telomere Research Network (TRN). Este arquivo foi modificado de20. Clique aqui para baixar este arquivo.
Arquivo Suplementar 3: Diretrizes de relatórios TRN. Clique aqui para baixar este arquivo.
Discussion
O método de medição de LT mais comumente usado em estudos populacionais maiores, antes de 2002, foi a análise de Southern blot de comprimentos de fragmentos de restrição terminal (TRF)24,25. O TRF, apesar de fornecer excelente precisão e reprodutibilidade em laboratórios especializados, é limitado em aplicabilidade devido à quantidade e qualidade do DNA necessário e ao rendimento limitado, fornecendo assim o pano de fundo para o aumento da utilização de ensaios TL baseados em qPCR e, posteriormente, o ensaio MMqPCR. O método MMqPCR TL fornece medição de TL repetível quando configurado, otimizado e mantido com atenção cuidadosa a cada critério de CQ. O cálculo e o relatório do ICC específico para cada coorte analisada são necessários para garantir a confiabilidade do ensaio. Embora sujeito à qualidade do DNA e ao conhecimento técnico, o método MMqPCR é adequado para grandes estudos populacionais que investigam a LT porque requer pequenas quantidades de DNA, é mais confiável do que a PCR singleplex e é mais eficiente nos custos de reagentes e no tempo do técnico do que outros métodos. A capacidade de gerar altos ICCs fornece dados adicionais em apoio ao uso de MMqPCR para grandes estudos populacionais de LT. A medição de TL por MMqPCR pode ser aplicada a uma ampla gama de estudos que buscam definir o papel da LT como um biomarcador de mortalidade por todas as causas, envelhecimento, estresse ao longo da vida, exposições ambientais e resultados de saúde física, como doenças cardiovasculares e câncer 4,7,8,10,11,12,13,26,27,28, 29,30,31.
Uma limitação do método MMqPCR é que ele relata TL como uma relação T/S, uma estimativa relativa de comprimento que varia dependendo da seleção do gene de cópia única, composição da mistura principal e parâmetros de ciclo de PCR32. A relação T/S não tem unidade. Assim, sem combinar com outras metodologias de medição de TL, este método é incapaz de relatar estimativas em valores de pares de bases 18,33,34. Como resultado, a relação T/S deve ser transformada em um escore Z para ter relevância entre os estudos35. Deve-se tomar cuidado substancial ao fazer isso em laboratórios, métodos e ensaios. Além disso, este método, como nos ensaios de hibridização baseados em gel, pode incluir a quantificação de telômeros intersticiais. No entanto, essas sequências compreendem uma proporção muito pequena do conteúdo total de DNA dos telômeros por genoma. Além disso, as sequências teloméricas intersticiais são mais propensas a conter sequências de pares de bases incompatíveis que se desviam das repetições canônicas dos telômeros, diminuindo a probabilidade de ligação e amplificação do primer. Além disso, embora a quantidade mínima de DNA necessária para o ensaio MMqPCR seja vantajosa, é importante observar que as medições baseadas em qPCR de TL são influenciadas por fatores pré-analíticos que afetam a qualidade e a integridade do DNA, incluindo condições de armazenamento de amostras, metodologia de extração de DNA e tecido biológico36,37. Foi demonstrado que o controle analítico desses fatores pode aumentar a validade externa das medições de LT geradas usando qPCR38. Mesmo assim, o impacto das diferenças na qualidade do DNA na LT gerada pelo ensaio MMqPCR precisa ser avaliado sistematicamente, pois não existe nenhuma orientação atual informada por dados para determinar se uma amostra é de qualidade suficiente para gerar uma estimativa precisa de TL usando essa abordagem. Apesar dessas limitações, as aplicações deste ensaio para estudos sobre resultados de saúde em nível populacional são consideráveis.
Ao utilizar o ensaio MMqPCR, é necessária uma avaliação contínua da precisão e do rendimento. Conforme projetado atualmente, os técnicos executam triplicatas de amostras simultaneamente em placas duplicadas usando dois termocicladores. Na ausência de vários termocicladores, recomendamos manter medições triplicadas duplicadas e placas de execução sequencialmente para maior precisão, mesmo ao custo de menor rendimento. Qualquer decisão de priorizar o rendimento em vez da precisão, por exemplo, empregando medições de triplicado único, deve ser acompanhada de testes e avaliações completos dos ICCs resultantes antes de prosseguir com a análise de amostras analíticas. Ao tomar decisões sobre rendimento e precisão, deve-se levar em consideração o tamanho da amostra e a qualidade do DNA da amostra. Um tamanho de amostra menor ou amostras de DNA de baixa qualidade exigem priorização de maior precisão23. Isso é ainda mais importante quando se trabalha com amostras de grupos relacionados (por exemplo, membros da família, sujeitos com vários pontos no tempo). Nesses casos, um planejamento cuidadoso, por exemplo, atribuir amostras relacionadas à mesma placa antes de iniciar o experimento, é uma maneira de evitar a perda de poder estatístico por meio de confusão inadvertida de grupo por placa.
Para este ensaio, um espectrofotômetro foi usado para avaliar a qualidade da amostra de DNA: amostras dentro da faixa de 1,6-2,0 para proporções 260/280 e 2,0-2,2 para proporções 260/230 foram consideradas aceitáveis. Essa avaliação de qualidade e a avaliação precisa do DNA de fita dupla via fluorômetro são etapas críticas neste protocolo para obter dados TL repetíveis. Outras medidas mais descritivas da integridade do DNA, como tamanho do fragmento e/ou medidas resumidas da qualidade do DNA determinada via gel de agarose (por exemplo, número de integridade do DNA) também podem ser utilizadas na determinação da qualidade da amostra38. Recomendamos também que a diluição das amostras ocorra apenas no momento do preparo da amostra para a execução do ensaio MMqPCR. Isso garante que as alíquotas de DNA sofram a menor quantidade possível de manipulação após a extração, diminuindo a variabilidade no manuseio pré-ensaio. Caso seja necessário transportar amostras de DNA, elas devem ser enviadas em gelo seco na concentração mais alta possível para mitigar a degradação que ocorre em amostras de DNA em concentrações mais baixas 39,40. Devido à degradação do DNA que ocorre com os ciclos de congelamento e descongelamento, o número de congelamentos e descongelamentos para estocar DNA deve ser minimizado41. Alíquotas do ADN de controlo agrupado devem ser criadas antes da execução da coorte e alíquotas de amostras individuais de ADN analítico devem ser criadas antes da execução do ensaio.
As principais métricas de desempenho do ensaio e CQ incluem sinal NTC, CVs interplaca e intraplaca e curva padrão R2. O não cumprimento dos critérios de CQ pode ser mitigado de várias maneiras. A troca regular dos estoques de PCR grau H2O e a alíquota dos subestoques de PCR grau H2O para cada par de placas executadas minimizarão as fontes de contaminação, bem como a amplificação do NTC. As etapas adicionais para reduzir a contaminação incluem o seguinte: designar um capuz de PCR específico dedicado apenas ao ensaio MMqPCR; limpar o capô e o equipamento de PCR com uma solução descontaminante de DNA; irradiando a sala com luz ultravioleta; e praticar técnica estéril quando no capuz de PCR. Para aumentar a repetibilidade do ensaio e diminuir os CVs, recomenda-se vortex amostras, diluições e tiras de PCR vigorosamente em suas respectivas etapas de vórtice e ressuspender completamente ao pipetar amostras de DNA. Uma curva R2 padrão abaixo do limite (<0,995) é provavelmente atribuível a erros de pipetagem durante o carregamento da placa. Para evitar isso, preste muita atenção à pipetagem precisa e calibre as pipetas anualmente, misture vigorosamente as tiras de PCR padrão antes de carregar e organize cuidadosamente os suprimentos para promover um fluxo de trabalho eficiente. Se for observado que o uso de duas máquinas e uma chapa produz CVs consistentemente mais altos, a máquina deve ser reparada como uma maneira potencial de melhorar o problema. As placas de controle de qualidade do fabricante devem ser executadas nas placas regularmente para avaliar o desempenho do termociclador.
Se os problemas persistirem mesmo após a aplicação das etapas recomendadas acima, as etapas a seguir poderão ser usadas para solucionar problemas do protocolo. Manter um registro de quando todos os reagentes foram aliquotados e quaisquer datas de validade pertinentes pode ajudar a agilizar o processo de solução de problemas quando inevitavelmente surgirem dificuldades. Uma parte importante de qualquer processo de solução de problemas é ajustar apenas um reagente por vez para determinar a causa específica das placas que não passam nos critérios de CQ, começando com o reagente mais barato em questão. Por exemplo, se o telômero e o gene de cópia única tiverem baixa eficiência, reagentes compartilhados, como DTT, dNTPs ou alíquota SYBR, são mais prováveis de causar do que primers específicos para amplicon. Ao preço listado, novas alíquotas devem ser testadas na ordem de TDT, depois dNTPs e, finalmente, se o problema persistir, novas alíquotas SYBR. Por outro lado, se apenas um dos amplicons (telômero ou gene de cópia única) tiver baixa eficiência, a causa da dificuldade é mais provável que seja um dos primers. A interpretação dos picos da curva de fusão apresentados na Figura 8 pode servir como uma fonte de informações importantes para a solução de problemas. A visualização dos dois picos da curva de fusão pode ser usada para identificar possíveis problemas com uma amostra específica, pois uma amostra de problema individual se destacará da tendência geral de picos exibidos por padrões ou amostras analíticas restantes. A curva de fusão também pode ser usada para diagnosticar problemas com um primer específico se os picos de um determinado amplicon forem sistematicamente menos nítidos do que o outro.
Este manuscrito detalha como configurar com sucesso o ensaio MMqPCR para medir TL com ampla aplicabilidade à pesquisa em saúde pública e apresenta as principais recomendações para CQ e solução de problemas com o objetivo de aumentar a acessibilidade e a confiabilidade desse método eficiente e econômico.
Disclosures
Os autores não têm nada a divulgar.
Acknowledgements
Os autores gostariam de agradecer ao Comitê Consultivo da Rede de Pesquisa de Telômeros e ao financiamento do Instituto Nacional de Envelhecimento / Instituto Nacional de Ciências da Saúde Ambiental (U24 AG066528 e U24 AG066528-S1) que tornaram este trabalho possível.
Materials
| Name | Company | Catalog Number | Comments |
| 0.5mL Tubes | USA Scientific | 1605-0099 | Seal-Rite 0.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 1.5mL Tubes | USA Scientific | 1615-5599 | Seal-Rite 1.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 100mM DTT | In House | Not Applicable | Made with stock DTT, diluted sodium acetate, and PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 15mL Tubes | Thermo Fisher | 14-959-53A | Corning 352196 Falcon 15mL Conical Centrifuge Tubes Storage Temperature and Conditions: Room temperature |
| 1M MgCl2 | Thermo Fisher | 50152107 | Biotang Inc 1M MgCl2 1M Magnesium Chloride Solution, Prepared in 18.2 Megohms Water and Filtered through 0.22 Micron Filter Storage Temperature and Conditions: 4 °C |
| 1x Gold Buffer | In House | Not Applicable | 10X Gold Buffer diluted with PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 25mM dNTPs | New England BioLabs | N0446S | Deoxynucleotide Solution Set Storage Temperature and Conditions: minus 20 °C |
| 5mL Tubes | Thermo Fisher | 3391276 | Argos Technologies Microcentrifuge Tubes – 5mL Storage Temperature and Conditions: Room temperature |
| 96 Well Plate | Bio-Rad | HSP9601 | Hard-Shell 96-Well PCR Plates, Low Profile, Thin Wall, Skirted, White / Clear Storage Temperature and Conditions: Room temperature |
| Aluminum Foil | Office Depot | 3489072 | Reynolds Wrap Stanard Aluminum Foil Roll, 12" x 75', Silver Storage Temperature and Conditions: Room temperature |
| AmpliTaq Gold Kit – Polymerase and Buffer | Thermo Fisher | 4311806 | AmpliTaq Gold DNA Polymerase with Gold Buffer and MgCl2 (MgCl2 in this kit is not used), 10X Gold Buffer, 2.5U AmpliTaq Gold Polymerase Storage Temperature and Conditions: minus 20 °C |
| Betaine | Thermo Fisher | AAJ77507AB | Betaine, 5M Solution, Molecular Biology Grade, Ultrapure, 10mL Storage Temperature and Conditions: minus 20 °C |
| Big Tube Rack | Thermo Fisher | 344817 | Fisherbrand 4-Way Tube Rack Storage Temperature and Conditions: Room temperature |
| CFX Maestro Software | Bio-Rad | 12004110 | Software for real-time PCR plate setup, data collection, statistics, and graphiing of results Storage Temperature and Conditions: Room temperature |
| CFX96 Optical Reaction Module for Real-Time PCR Systems with Starter Package | Bio-Rad | 1845096 | 96-well optical module for real-time PCR Storage Temperature and Conditions: Room temperature |
| DTT | Fisher Scientific | AAJ1539706 | Dithiothreitol, >99.5+ Molecular Biology Grade, 5 g Storage Temperature and Conditions: minus 20 °C |
| ELIMINase | Fisher Scientific | 04-355-32 | ELIMINase Laboratory Decontaminant Storage Temperature and Conditions: Room temperature |
| HEPA Filter | USA Scientific | Replacement Filters | High-Efficiency Particulate Air Filter for AirClean Workstations Storage Temperature and Conditions: Room temperature |
| Kimwipes | Thermo Fisher | 06666A | Kimberly-Clark Professional Kimtech Science Kimwipes Delicate Task Wipers, 1-Ply Storage Temperature and Conditions: Room temperature |
| Loading Trough | Thermo Fisher | 14387069 | Thermo Scientific Matrix Reagent Reservoirs Storage Temperature and Conditions: Room temperature |
| Microsoft Excel | Microsoft | Not Applicable | Microsoft 365 package, Excel software application Storage Temperature and Conditions: Room temperature |
| Mini Centrifuge | Genesee Scientific | 31-500B | Poseidon 31-500B Mini Centrifuge, Blue Lid Storage Temperature and Conditions: Room temperature |
| PCR Grade H2O | Thermo Fisher | AM9937 | Nuclease-Free Water (not DEPC-Treated) Storage Temperature and Conditions: Room temperature |
| PCR Hood | USA Scientific | 4263-2588 | Nucleic Acid Workstation with HEPA Filtration, AirClean Systems Combination PCR Workstation Storage Temperature and Conditions: Room temperature |
| PCR Strips | Thermo Fisher | AB0776 | Low Profile Tubes and Flat Caps, Strips of 8 Storage Temperature and Conditions: Room temperature |
| PCR Tube Rack | Thermo Fisher | 344820 | Fisherbrand 96-Well PCR Tube Rack Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Multichannel) | Ranin | 17005860 | Pipette Tips SR LTS 20µL F 960A/5, 20µL Maximum Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1181-3850 | 10µL Graduated TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1180-1850 | 20µL Beveled TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-0880 | 200µL Natural TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-2890 | 1000µL Natural Graduated TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013802 | Pipet-Lite Multi Pipette L8-10XLS, 0.5 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013803 | Pipet-Lite Multi Pipette L8-20LS+, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F144802G | Gilson Pipetman Classic Pipets, 1 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123600 | Gilson Pipetman Classic Pipets, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123601 | Gilson Pipetman Classic Pipets, 20 to 200µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123602 | Gilson Pipetman Classic Pipets, 200 to 1000µL Storage Temperature and Conditions: Room temperature |
| Plate Sealing Film | Bio-Rad | MSB1001 | Microseal “B” PCR Plate Sealing Film, Adhesive, Optical Storage Temperature and Conditions: Room temperature |
| Plate Spinner | Thermo Fisher | 14-100-141 | Fisherbrand Mini Plate Spinner Centrifuge, 230 V Storage Temperature and Conditions: Room temperature |
| Pre-Hood Filter | USA Scientific | 4235-3724 | Prefilter for AirClean Systems Workstations Storage Temperature and Conditions: Room temperature |
| R Software | The R Project for Statistical Computing | Not Applicable | R version 4.2.2 Storage Temperature and Conditions: Room temperature |
| Scale | Thermo Fisher | 01-922-329 | OHAUS 30430060 PR Series Analytical Balance, 62g Capacity Storage Temperature and Conditions: Room temperature |
| Scissors | Office Depot | 458612 | Office Depot Brand Scissors, 8”, Straight, Black, Pack of 2 Storage Temperature and Conditions: Room temperature |
| Sharpies | Sharpie | 2151734 | Brush Twin Permanent Markers, Black Storage Temperature and Conditions: Room temperature |
| Single Copy Gene Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Single Copy Gene Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Small Tube Rack | Thermo Fisher | 21-402-17 | Thermo Fisher 8601 Reversible Microtube Racks with Lid Storage Temperature and Conditions: Room temperature |
| Sodium Acetate | Thermo Fisher | J63560.EQE | 3M NaOAc pH 5.2 Storage Temperature and Conditions: Room temperature |
| Stainless Steel Spatula | Thermo Fisher | 3990240 | Bel-Art SP Scienceware Stainless-Steel Sampling Spoon and Spatula Storage Temperature and Conditions: Room temperature |
| SYBR Green | Thermo Fisher | S7563 | SYBR Green I Nucleic Acid Gel Stain – 10,000X Concentrate in DMSO Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Syringe Filters | Fisher Scientific | 09-927-55A | GD/X 25 mm Sterile Syringe Filter, cellulose acetate filtration medium, 0.2 μm Storage Temperature and Conditions: Room temperature |
| Syringes | Thermo Fisher | 148232A | BD Luer-Lok Disposable Syringes without Needles, 10mL Storage Temperature and Conditions: Room temperature |
| TE Buffer | Fisher Scientific | BP2474100 | TE Buffer, Tris-EDTA, 1X Solution, pH 7.6, Molecular Biology, Fisher BioReagents Storage Temperature and Conditions: Room temperature |
| Telomere Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Telomere Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| UV Light | USA Scientific | 4288-2540 | UV Light Bulb for Workstations Storage Temperature and Conditions: Room temperature |
| Vortex | Thermo Fisher | 14-955-151 | Fisherbrand Mini Vortex Mixer, 115 V, 50/60 Hz Storage Temperature and Conditions: Room temperature |
| Weigh Boat | Thermo Fisher | 01-549-752 | Fisherbrand Sterile Hexagonal Weighing Boat, 10mL Storage Temperature and Conditions: Room temperature |
References
- Hemann, M., Strong, M., Hao, L., Greider, C. The shortest telomere, not average telomere length, is critical for cell viability and chromosome stability. Cell. 107 (1), 67-77 (2001).
- Xu, L., Li, S., Stohr, B. A. The role of telomere biology in cancer. Ann Rev Pathol: Mech Dis. 8, 49-78 (2013).
- Lindrose, A., Drury, S. Minimum reporting recommendations for pcr-based telomere length measurement. Telomere Research Network. , (2020).
- Verhulst, S., et al. Commentary: The reliability of telomere length measurements. Int J Epidemiol. 44 (5), 1683-1686 (2015).
- Aubert, G., Hills, M., Lansdorp, P. M. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1-2), 59-67 (2012).
- Mundstock, E., et al. Effect of obesity on telomere length: Systematic review and meta-analysis. Obesity. 23 (11), 2165-2174 (2015).
- Haycock, P. C., et al. Leucocyte telomere length and risk of cardiovascular disease: Systematic review and meta-analysis. BMJ. 349, 4227 (2014).
- Astuti, Y., Wardhana, A., Watkins, J., Wulaningsih, W. Cigarette smoking and telomere length: A systematic review of 84 studies and meta-analysis. Environ Res. 158, 480-489 (2017).
- Ridout, K. K., Ridout, S. J., Price, L. H., Sen, S., Tyrka, A. R. Depression and telomere length: A meta-analysis. J Affect Disord. 191, 237-247 (2016).
- Wang, Q., Zhan, Y., Pedersen, N. L., Fang, F., Hägg, S. Telomere length and all-cause mortality: A meta-analysis. Ageing Res Rev. 48, 11-20 (2018).
- Hu, R., Hua, X. G., Jiang, Q. C. Associations of telomere length in risk and recurrence of prostate cancer: A meta-analysis. Andrologia. 51 (7), e13304 (2019).
- Schneider, C. V., et al. Association of telomere length with risk of disease and mortality. JAMA Inter Med. 182 (3), 291-300 (2022).
- Deng, Y., et al. Telomere length and the risk of cardiovascular diseases: A mendelian randomization study. Front Cardiovasc Med. 9, 1012615 (2022).
- D'mello, M. J., et al. Association between shortened leukocyte telomere length and cardiometabolic outcomes: Systematic review and meta-analysis. Circulation: Cardiovasc Gene. 8 (1), 82-90 (2015).
- Wilbourn, R. V., et al. The relationship between telomere length and mortality risk in non-model vertebrate systems: A meta-analysis. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160447 (2018).
- Gardner, M., et al. Gender and telomere length: Systematic review and meta-analysis. Exp Gerontol. 51, 15-27 (2014).
- Lai, T. -. P., Wright, W. E., Shay, J. W. Comparison of telomere length measurement methods. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160451 (2018).
- Cawthon, R. Telomere measurement by quatitative pcr. Nuc Acid Res. 30 (10), e47-e53 (2002).
- Cawthon, R. M. Telomere length measurement by a novel monochrome multiplex quantitative pcr method. Nuc Acid Res. 37 (3), e21-e21 (2009).
- Lindrose, A. R., et al. Method comparison studies of telomere length measurement using qpcr approaches: A critical appraisal of the literature. PLoS One. 16 (1), e0245582 (2021).
- Lin, J., et al. Effects of DNA extraction, DNA integrity, and laboratory on the precision of qpcr-based telomere length measurement - a multi-lab impartial study. bioRxiv. , (2022).
- Wilfinger, W. W., Mackey, K., Chomczynski, P. Effect of ph and ionic strength on the spectrophotometric assessment of nucleic acid purity. Biotechniques. 22 (3), 474-481 (1997).
- Boesenberg-Smith, K. A., Pessarakli, M. M., Wolk, D. M. Assessment of DNA yield and purity: An overlooked detail of pcr troubleshooting. Clin Microbiol Newsletter. 34 (1), 1-6 (2012).
- Harley, C. B., Futcher, A. B., Greider, C. W. Telomeres shorten during ageing of human fibroblasts. Nature. 345 (6274), 458-460 (1990).
- Kimura, M., et al. Measurement of telomere length by the southern blot analysis of terminal restriction fragment lengths. Nat Protoc. 5 (9), 1596-1607 (2010).
- Ye, Q., et al. Telomere length and chronological age across the human lifespan: A systematic review and meta-analysis of 414 study samples including 743,019 individuals. Ageing Res Rev. 90, 102031 (2023).
- Axelrad, M. D., Budagov, T., Atzmon, G. Telomere length and telomerase activity; a yin and yang of cell senescence. J Vis Exp. (75), e50246 (2013).
- Liu, M., et al. Immune-mediated inflammatory diseases and leukocyte telomere length: A mendelian randomization study. Front Genetics. 14, 1129247 (2023).
- Van Ockenburg, S., et al. Stressful life events and leukocyte telomere attrition in adulthood: A prospective population-based cohort study. Psychol Med. 45 (14), 2975-2984 (2015).
- Zong, Z. Q., et al. Ambient air pollution exposure and telomere length: A systematic review and meta-analysis. Public Health. 215, 42-55 (2023).
- Tang, L., Li, D., Wang, J., Su, B., Tian, Y. Ambient air pollution, genetic risk and telomere length in uk biobank. J Expo Sci Environ Epidemiol. , (2023).
- Aubert, G., Hills, M., Lansdorp, P. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1), 59-67 (2012).
- Lin, J., et al. Systematic and cell type-specific telomere length changes in subsets of lymphocytes. J Immunol Res. 2016, 5371050 (2016).
- Needham, B. L., et al. Socioeconomic status, health behavior, and leukocyte telomere length in the national health and nutrition examination survey, 1999-2002. Soc Sci Med. 85, 1-8 (2013).
- Verhulst, S. Improving comparability between qpcr-based telomere studies. Mol Ecol Res. 20 (1), 11-13 (2020).
- Dagnall, C. L., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PloS one. 12 (9), e0184098 (2017).
- Lin, J., Smith, D. L., Esteves, K., Drury, S. Telomere length measurement by qpcr-summary of critical factors and recommendations for assay design. Psychoneuroendocrinology. 99, 271-278 (2019).
- Wolf, S. E., et al. Cross-tissue comparison of telomere length and quality metrics of DNA among individuals aged 8 to 70 years. PLOS One. 19 (2), e0290918 (2024).
- RoDer, B., FruHwirth, K., Vogl, C., Wagner, M., Rossmanith, P. Impact of long-term storage on stability of standard DNA for nucleic acid-based methods. J Clin Microbiol. 48 (11), 4260-4262 (2010).
- Dagnall, C., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PLoS One. 12 (9), e0184098 (2017).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreserv Biobank. 10 (1), 4-11 (2012).
- Institute for Statistics and Mathematics of WU. The Comprehensive R Archive Network Available from: https://cran.r-project.org/ (2024)
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biol Rev. 85 (4), 935-956 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved