Remoção de bacteriófagos de culturas de Salmonella infectadas
In This Article
Summary
Os bacteriófagos, onipresentes e diversos na Terra, infectam e se replicam dentro de hospedeiros bacterianos, desempenhando um papel crucial nos ecossistemas microbianos. Apesar de sua importância, sua presença pode atrapalhar os processos industriais. Desenvolvemos um método usando lipopolissacarídeos bacterianos para eliminar bacteriófagos de culturas de Salmonella .
Abstract
Os bacteriófagos, ou simplesmente fagos, desempenham um papel vital em ambientes microbianos, impactando as populações bacterianas e moldando sua evolução e interações. Esses organismos são vírus que infectam e se replicam dentro de hospedeiros bacterianos. Os fagos são onipresentes na Terra, altamente diversos e muito abundantes. Embora os bacteriófagos tenham papéis valiosos em diferentes ambientes e sejam uma área-chave de pesquisa em microbiologia e ecologia, sua presença pode ser indesejável em certos processos ou produtos industriais. Considerando a abundância e onipresença de bacteriófagos na Terra, o desenho de procedimentos para a remoção de bacteriófagos de culturas bacterianas é crucial em diversas aplicações laboratoriais e industriais para preservar a integridade das culturas e garantir resultados experimentais precisos ou qualidade do produto. Aqui, ajustamos um protocolo para eliminar os bacteriófagos de culturas de Salmonella enterica infectadas, usando uma estratégia baseada no uso de lipopolissacarídeos (LPS) localizados na membrana externa de bactérias Gram-negativas. O LPS bacteriano desempenha um papel importante no reconhecimento do hospedeiro por fagos, e fazemos uso dessa propriedade para projetar um procedimento eficaz para a remoção de fagos, que usam LPS como receptor, em culturas bacterianas de Salmonella .
Introduction
As populações microbianas enfrentam múltiplos desafios em ambientes naturais, e uma ameaça especialmente grave é o potencial de infecção por bacteriófagos, os vírus que infectam as bactérias1. Esses vírus estão disseminados no planeta, exibindo grande diversidade e abundância 2,3,4,5. Embora os bacteriófagos sejam diversos em tamanho, morfologia e organização genômica, todos compartilham a mesma estrutura: um genoma de DNA ou RNA envolto por um capsídeo formado por proteínas codificadas por fagos6. As bactérias desenvolveram uma gama diversificada de mecanismos de defesa contra elas7. Um aspecto fundamental da infecção por bacteriófagos, que é relevante para a caracterização e também para a detecção, são os domínios de ligação ao receptor presentes nas fibras da cauda. Os bacteriófagos têm proteínas em sua superfície chamadas proteínas de ligação ao receptor ou fibras da cauda para reconhecer e se ligar a locais receptores específicos na superfície da célula bacteriana. No caso de bactérias Gram-negativas, o reconhecimento de estruturas superficiais, como lipopolissacarídeos (LPS), proteínas da membrana externa, pili e/ou flagelos, estão envolvidos na interação fago-bactéria8. Essa interação entre bacteriófagos e bactérias é altamente específica e depende principalmente de sua capacidade de se ligar às superfícies do hospedeiro. O antígeno O do lipopolissacarídeo é um receptor comumente usado9.
A investigação das interações bacteriófagos-bactérias não é apenas fascinante do ponto de vista biológico, mas também tem aplicações práticas em áreas como terapia fágica e biotecnologia. Embora os bacteriófagos tenham papéis valiosos em vários contextos, por exemplo, alterando populações microbianas10, sua presença pode ser indesejável em certos processos industriais. Em produtos farmacêuticos, biotecnologia e produção de alimentos, a presença de bacteriófagos pode impactar a qualidade e a segurança dos produtos finais, tornando sua remoção essencial para atender aos padrões de qualidade. No bioprocessamento e biofabricação, onde as culturas bacterianas são usadas para produzir vários compostos (por exemplo, proteínas, enzimas ou antibióticos), a presença de bacteriófagos pode levar à interrupção dos processos de produção devido à sua capacidade de equilibrar a população bacteriana em todos os ambientes compartilhados. Os fagos podem, ocasionalmente, transformar a vida profissional do microbiologista industrial em um pesadelo11. O design de procedimentos eficazes para remover fagos é fundamental para garantir uma produção consistente e confiável, aumentando a eficiência do processo. Além desses aspectos industriais, em um ambiente de laboratório de pesquisa, onde a precisão e a reprodutibilidade são cruciais, a eliminação de bacteriófagos é essencial para a obtenção de resultados precisos e confiáveis. Além disso, a remoção de fagos também pode ser usada para simular ambientes variados para testar diferentes hipóteses12. A remoção de fagos também pode ser muito útil no ambiente de pesquisa, uma vez que muitos estudos baseados em fagos, como a enumeração de bactérias após a aplicação de fagos, se beneficiariam de uma etapa para remover fagos a fim de produzir contagens viáveis muito mais confiáveis.
A remoção de fagos com base no isolamento de colônias levaria vários dias para garantir que as colônias estejam livres de fagos, enquanto o procedimento descrito aqui permite a geração de culturas livres de fagos em horas. Este protocolo nos permite acompanhar a evolução das culturas bacterianas sem impedi-las de isolar as colônias. Nesse sentido, é possível simular ambientes flutuantes (presença e/ou ausência de fagos) para testar diferentes hipóteses. Além disso, este protocolo permite a análise qualitativa e quantitativa da presença de fagos em uma cultura bacteriana.
Em resumo, projetar procedimentos econômicos para a remoção de bacteriófagos é crucial para manter a qualidade, segurança e eficiência do processo do produto em vários setores e para avanços na pesquisa básica e aplicada. Aqui, descrevemos um protocolo altamente eficaz baseado no uso de LPS para a remoção de bacteriófagos, que usam LPS como receptor, de culturas de Salmonella infectadas, que é eficiente em termos de tempo e requer equipamento mínimo.
Protocol
NOTA: Antes de realizar o procedimento de eliminação de fagos, descrevemos a preparação de uma cultura de Salmonella infectada com bacteriófagos 9NA. Na Figura 1, é ilustrada uma representação geral do procedimento completo para remoção de bacteriófagos em culturas bacterianas.
1. Preparação de culturas de Salmonella infectadas com bacteriófagos
- Preparação de cultura bacteriana
NOTA: Neste estudo, foi utilizada Salmonella enterica subsp. enterica sorovar Typhimurium cepa ATCC 14028 opvAB::lacZ (SV8011)13 .- Descongele lentamente o frasco para injetáveis recuperado a -80 °C que contém a estirpe de interesse de S. enterica , colocando-o sobre gelo.
- Retire a cepa de S. enterica do estoque de dimetilsulfóxido descongelado em um prato de caldo Lennox (LB) (10 g / L de triptona, 5 g / L de NaCl, 5 g / L de extrato de levedura e 15 g / L de ágar, consulte a Tabela de Materiais). Incubar a placa a 37 °C durante 24 h.
- Usando uma agulha de inoculação, transfira uma colônia da placa de S. enterica e inocule-a em 5 mL de LB líquido em um tubo de vidro. Incubar a 37 °C durante 24 h com arejamento por agitação a 200 rpm.
- Preparação de bacteriófagos
NOTA: O bacteriófago 9NA, pertencente à família Siphoviridae (ordem Caudovirales)14, foi utilizado neste protocolo. Notavelmente, outros bacteriófagos de Salmonella enterica que usam o antígeno O do lipopolissacarídeo como receptor podem ser usados.- Prepare o caldo de bacteriófago adicionando 0,1 mL de lisado de bacteriófago concentrado a uma mistura de 100 mL de caldo nutritivo (NB; 5 g/L de peptona e 3 g/L de extrato de levedura), 2 mL de 50x Sais E (38,78 mM MgSO47 H2O, 520,5 mM de ácido cítricoH2O, 2,87 M K2HPO4 anidro e 829 mM NaNH4HPO44 H2O) e 0,4 mL de 50% glicose. Misture bem por inversão.
- Um exemplo representativo de concentração de bacteriófagos pode ser 109 PFU/mL (Unidades Formadoras de Placas/mL), mas concentrações mais baixas podem ser usadas com resultados semelhantes (ver Tabela de Materiais). Estimar a concentração de bacteriófagos seguindo o passo 1.2.3.
- Prepare o lisado seguindo as etapas descritas abaixo.
- Prepare o inóculo de S. enterica em 5 mL de LB (10 g/L de triptona, 5 g/L de NaCl e 5 g/L de extrato de levedura). Incubar a 37 °C e 200 rpm durante 24 h.
- Misture 4 mL de caldo de bacteriófago com 1 mL da cultura noturna de S. enterica. Incubar a mistura a 37 °C e 200 rpm durante 8 h.
- Para discriminar entre bactérias e bacteriófagos, centrifugue a mistura a 2.377 x g por 20 min. Recupere o sobrenadante em um tubo de vidro, adicione 800 μL de clorofórmio (consulte a Tabela de Materiais) e vortexe-o. Certifique-se de transferir apenas a fase superior quando uma mistura com clorofórmio for usada.
NOTA: É importante evitar a transferência de clorofórmio residual, o que pode afetar a eficiência do procedimento. A etapa do clorofórmio é comum em protocolos de isolamento e propagação como forma de maximizar os fagos liberados. No entanto, o clorofórmio pode inativar alguns fagos ou destruir sua infectividade15. Portanto, esta etapa é aplicável apenas a fagos resistentes ao clorofórmio. - Manter os lisados à temperatura ambiente durante 2 h e conservá-los a 4 °C.
- Titular o lisado para quantificar o número de bacteriófagos/número de PFU por ml presentes no lisado preparado. Para fazer isso, execute a técnica de sobreposição descrita abaixo.
- Prepare 5 mL de LB de inóculo para S. enterica. Incubar a 37 °C durante 24 h com arejamento por agitação a 200 rpm.
- Realize diluições seriadas do lisado em LB para atingir um número contável de PFU/mL. Para determinar os factores de diluição necessários, identificar o lisado bacteriófago inicial concentrado. Para um exemplo representativo de concentração de bacteriófagos em torno de 109 PFU/mL, os fatores de diluição que resultam em um número contável de placas estão entre 106-10 10.
- Adicionar 100 μL da diluição apropriada do lisado e 60 μL da cultura noturna de S. enterica preparada no dia anterior a 5 mL de ágar LB mole, preparado misturando LB líquido e LB-ágar na proporção de 1:1 e mantendo a 56 °C para evitar a solidificação. Misture com cuidado, evitando bolhas.
- Despeje cada mistura no topo de um prato LB e mantenha na bancada até que o meio solidifique.
- Incubar as placas durante 24 h a 37 °C. Conte o número de placas em cada placa. Para determinar o número de PFU/mL, multiplique o número de placas de cada placa pelo fator de diluição (FD). Para garantir uma infecção eficiente por fago, são necessários 10 9-1012 PFU/mL.
UFP/mL= Não. placas na placa x FD
- Prepare o caldo de bacteriófago adicionando 0,1 mL de lisado de bacteriófago concentrado a uma mistura de 100 mL de caldo nutritivo (NB; 5 g/L de peptona e 3 g/L de extrato de levedura), 2 mL de 50x Sais E (38,78 mM MgSO47 H2O, 520,5 mM de ácido cítricoH2O, 2,87 M K2HPO4 anidro e 829 mM NaNH4HPO44 H2O) e 0,4 mL de 50% glicose. Misture bem por inversão.
- Infecção de culturas de Salmonella com bacteriófagos
- Dilua uma cultura noturna de S. enterica a 1:100 em um volume final de 5 mL de LB e adicione 0,1 mL de lisado (concentrado a 10 9-1012 PFU/mL). Incubar a 37 °C e 200 rpm durante 24 h.
- Determinação do título de fagos como PFU em ensaio em placa
- Para quantificar o número de bacteriófagos na cultura infectada, realize ensaios em placas para determinar o título de fagos como PFU/mL, conforme descrito abaixo.
- Transfira 1 mL da cultura para um tubo. Centrifugue a 17,115 x g por 2 min e despeje o sobrenadante em um novo frasco rotulado.
- Adicione 100 μL de clorofórmio ao frasco e ao vórtice para remover os detritos bacterianos da solução. Siga o passo 1.2.3.
- Para quantificar o número de bacteriófagos na cultura infectada, realize ensaios em placas para determinar o título de fagos como PFU/mL, conforme descrito abaixo.
2. Remoção de bacteriófagos de culturas de Salmonella enterica infectadas (ver Figura 2)
NOTA: Para monitorar o processo de remoção do bacteriófago, diferentes alíquotas são tomadas ao longo do protocolo de limpeza para titulação. No total, existem oito alíquotas para confirmar que o número de PFU/mL está diminuindo ao longo do processo de limpeza até a eliminação completa.
- Remoção do bacteriófago em suspensão
- Diluir a cultura bacteriana contendo fagos a 1:100 em 5 mL de LB filtrado a 0,22 μm em um tubo de centrífuga cônico de 50 mL. Ponto de titulação 1: Esta é a primeira alíquota para titulação (a cultura inicial de Salmonella infectada).
- Centrifugue a suspensão por 10 min a 2.377 x g. Remova cuidadosamente o sobrenadante. Não use uma pipeta; despeje o tubo. Tente deixar algum volume na parte inferior do tubo cônico da centrífuga para garantir a presença de células.
- Lave as células bacterianas com 10 mL de meio LB filtrado com 0,22 μm. Repita as etapas de centrifugação e lavagem 3x. Ressuspenda o pellet em 5 ml de LB filtrado a 0,22 μm. Ponto de titulação 2: Esta é a segunda alíquota para titulação.
- Transfira 50 μL da suspensão bacteriana acima através de um filtro estéril de 0,45 μm (consulte a Tabela de Materiais) usando um sistema de filtração a vácuo. Enxágue o filtro com 100 mL de LB filtrado com 0.22 μm.
NOTA: O tamanho da cabeça do bacteriófago 9NA é de 0,067 μm. Em contraste, a Salmonella tem 2-5 μm de comprimento e 0,5-1,5 μm de largura. Assim, se membranas de 0,45 μm forem usadas, a maioria das bactérias não passará porque são maiores que 0,45 μm, enquanto a maioria dos fagos passará pelos poros do filtro de 0,45 μm.
- Remoção do bacteriófago contido no interior das células de Salmonella
- Para facilitar a liberação de fagos das células bacterianas, incube o filtro de 0,45 μm contendo células por 2 h a 37 ° C dentro da incubadora sem desmontar o sistema.
NOTA: Esta etapa de incubação é necessária para promover a lise das bactérias infectadas e a liberação de fagos intracelulares no filtro. - Lave o filtro de 0,45 μm usando um sistema de filtragem a vácuo, enxaguando o filtro com 100 mL de LB filtrado de 0,22 μm.
- Recupere as células bacterianas do filtro em 2 mL de 2x LB (20 g / L de triptona, 10 g / L de NaCl, 10 g / L de extrato de levedura; consulte a Tabela de Materiais). Para fazer isso, transfira o filtro de 0,45 μm para um frasco usando uma pinça estéril. Em seguida, adicione 2 mL de 2x LB sobre o filtro e pipete várias vezes para liberar as células do filtro.
- Centrifugue 1 mL desta suspensão por 2 min a 10.354 x g. Descarte o sobrenadante e lave as células bacterianas 3x com 1 mL de meio LB filtrado. Ressuspenda as células em 1 mL de 2x LB. Ponto de titulação 3: Esta é a terceira alíquota para titulação.
NOTA: O uso de um LB (2x LB) mais concentrado é altamente recomendado, pois, conforme descrito abaixo, será adicionado lipopolissacarídeo comercial (LPS), e este LPS se dissolve em água, diluindo assim o meio.
- Para facilitar a liberação de fagos das células bacterianas, incube o filtro de 0,45 μm contendo células por 2 h a 37 ° C dentro da incubadora sem desmontar o sistema.
- Evite a reinfecção enganando os bacteriófagos liberados das células bacterianas com lipopolissacarídeo comercial, conforme descrito abaixo.
- Incubar 20 μL da suspensão bacteriana na presença de um lipopolissacarídeo comercial de S. enterica (LPS; ver Tabela de Materiais) até uma concentração final de 3,75 mg / mL em 1 mL de LB filtrado com 0,22 μm por 2 h a 37 ° C com agitação a 200 rpm.
NOTA: Os fagos liberados das células para o meio podem se ligar ao LPS comercial de S. enterica em vez do LPS bacteriano. - Após 2 h de incubação, transfira 1 mL de cultura para um tubo de microcentrífuga e centrifugue a suspensão por 2 min a 10.354 x g. Ponto de titulação 4: Utilizar o sobrenadante como quarta alíquota para a titulação.
- Lave o pellet 3x com 1 mL de meio LB filtrado.
- Após a lavagem, ressuspenda o pellet em 1 mL de 2x LB. Incube 20 μL desta mistura em 1 mL de LB filtrado com 0,22 μm com 0,8 mg / mL de LPS comercial por 2 h a 37 ° C com agitação a 200 rpm. Ponto de titulação 5: Utilizar o resto da suspensão bacteriana para a quinta titulação.
- Após 2 h de incubação, transfira 1 mL de cultura para um tubo de microcentrífuga e pellet por centrifugação por 2 min a 10.354 x g. Ponto de titulação 6: Utilizar o sobrenadante como sexta alíquota para a titulação, deitando o sobrenadante num novo frasco para injetáveis.
- Lave as células bacterianas 3x com LB filtrado. Após a lavagem, ressuspenda as células coletadas em 1 mL de LB filtrado com 0,22 μm.
- Passe 1 mL de suspensão bacteriana por um filtro estéril de 0,45 μm usando um sistema de filtração a vácuo. Enxágue o filtro usando 100 mL de LB filtrado com 0.22 μm.
- Recupere células do filtro de 0,45 μm em 2 mL de meio 2x LB. Para o efeito, transferir o filtro de 0,45 μm para um balão com uma pinça esterilizada. Em seguida, adicione 2 mL de 2x LB sobre o filtro e pipete várias vezes para tentar liberar as células do filtro.
- Pellet 1 mL de células por centrifugação por 2 min a 10.354 x g. Lave 3x com LB filtrado de 0,22 μm. Após a lavagem, ressuspenda as células coletadas em 1 mL de 2x LB. Ponto de titulação 7: Esta é a sétima alíquota para titulação. Esta alíquota informará sobre a eficiência da remoção de fagos em culturas bacterianas.
- Incubar 20 μL da suspensão bacteriana na presença de um lipopolissacarídeo comercial de S. enterica (LPS; ver Tabela de Materiais) até uma concentração final de 3,75 mg / mL em 1 mL de LB filtrado com 0,22 μm por 2 h a 37 ° C com agitação a 200 rpm.
3. Preparação da cultura de Salmonella isenta de bacteriófagos após remoção de bacteriófagos
- Preparar o inóculo isento de fagos. Adicione 100 μL de células da suspensão bacteriana a 900 μL de LB contendo 0,45 mg / mL de LPS. Incubar a 37 °C durante 24 h com agitação a 200 rpm.
- Ponto de titulação 8: Preparar a oitava alíquota para titulação (a cultura final potencial não infectada). Este ponto é para confirmar a ausência de reinfecção por fagos.
Representative Results
Salmonella enterica e outras bactérias Gram-negativas têm uma membrana externa contendo LPS. O antígeno O do LPS é um receptor comumente usado pelos bacteriófagos 9NA para infectar culturas de Salmonella 16,17.
Dada a afinidade específica dos bacteriófagos pelo antígeno O ou regiões polissacarídicas centrais do LPS, queríamos examinar se o LPS comercial de Salmonella enterica poderia ser usado como isca para excluir bacteriófagos 9NA. Para isso, misturamos concentrações conhecidas do LPS comercial e lisado de 9NA, seguido de titulação. Os bacteriófagos LPS e 9NA comerciais foram misturados em um volume total de 200 μL e incubados por 2 h a 37 °C sem agitação. Para a titulação, 100 μL da mistura LPS-9NA e 60 μL de uma cultura noturna foram adicionados a 5 mL de ágar LB mole e despejados no topo de uma placa LB. As placas foram incubadas por 24 h a 37 °C. Conforme mostrado na Figura 3, o título de lisado diminui proporcionalmente quando a concentração de LPS comercial de S. enterica aumenta. Esses resultados indicam que o LPS comercial funciona corretamente como um chamariz para bacteriófagos 9NA.
Curiosamente, o tempo necessário para o fago 9NA lisar as células de Salmonella e liberar fagos é facilitado pelo número de bactérias e fagos (Figura 4). Para estudar como esse aspecto poderia afetar o protocolo de remoção de fagos, testamos diferentes proporções de bactérias: fagos (1:1, 1:10, 1:100 e 1:1000). Como visto na Figura 4, há uma queda na DO600 nm da cultura em 1,5-2,5 h após a adição do fago 9NA. Valores de OD600 nm próximos de zero são indicativos de que a cultura bacteriana está sendo lisada18. Por esse motivo, os tempos de incubação neste protocolo foram definidos como 2 h, a fim de garantir tempo suficiente para que o fago contido dentro das células de Salmonella lisasse as bactérias e fosse liberado. Este tempo deve ser estimado para cada sistema hospedeiro-fago antes de realizar este protocolo.
Uma vez determinado que o LPS comercial funciona como um chamariz e o tempo de lise para bactérias infectadas por 9NA, realizamos o protocolo descrito para limpeza de culturas de Salmonella infectadas dos bacteriófagos (Figura 1 e Figura 2). Para monitorar a presença de bacteriófagos ao longo de cada etapa do protocolo, realizamos ensaios de placa para calcular a infectividade das culturas resultantes em 100 μL da mistura resultante em diferentes pontos (Figura 5). Podemos observar que a lavagem e filtragem repetidas não são suficientes para a eliminação de bacteriófagos das culturas (pontos de titulação 1-3); no entanto, o número de fagos diminui assim que empregamos uma etapa de incubação com LPS comercial (ponto de titulação 4). A etapa crucial para a remoção completa de bacteriófagos em uma cultura bacteriana é a segunda incubação com LPS comercial (ponto de titulação 6). Esta etapa é essencial para a remoção bem-sucedida do bacteriófago 9NA em culturas de Salmonella .
Um aspecto interessante deste protocolo seria conhecer o nível de resistência do fago após as etapas de remoção do fago. Um procedimento que separa fagos de bactérias resistentes a fagos não tem utilidade. Por esse motivo, é crucial revelar que as bactérias que permanecem na cultura são suscetíveis a fagos. Para demonstrar que as células suscetíveis permanecem nas culturas após o procedimento, usamos o ensaio em placa de uranina azul de Evans (EBU) para rastrear a contaminação por fagos19. As placas EBU foram confeccionadas em meio LB suplementado com 10 mL/L K2HPO4 25%, 5 mL/L de glicose 50%, 2,5 mL/L de fluoresceína 1%, 1,25 mL/L Evans Blue 1% e 15 g/L de ágar. O cross-streaking em placas de EBU com fago 9NA foi usado para discriminar isolados resistentes a fagos e sensíveis a fagos (Figura 6). As culturas bacterianas obtidas ao final do protocolo de limpeza foram utilizadas para obtenção de colônias isoladas, as quais foram verificadas quanto à contaminação por fagos (Figura 6B). Podemos observar a existência de células resistentes e sensíveis. Este protocolo não favorece a seleção de células resistentes a fagos; elimina apenas bacteriófagos.
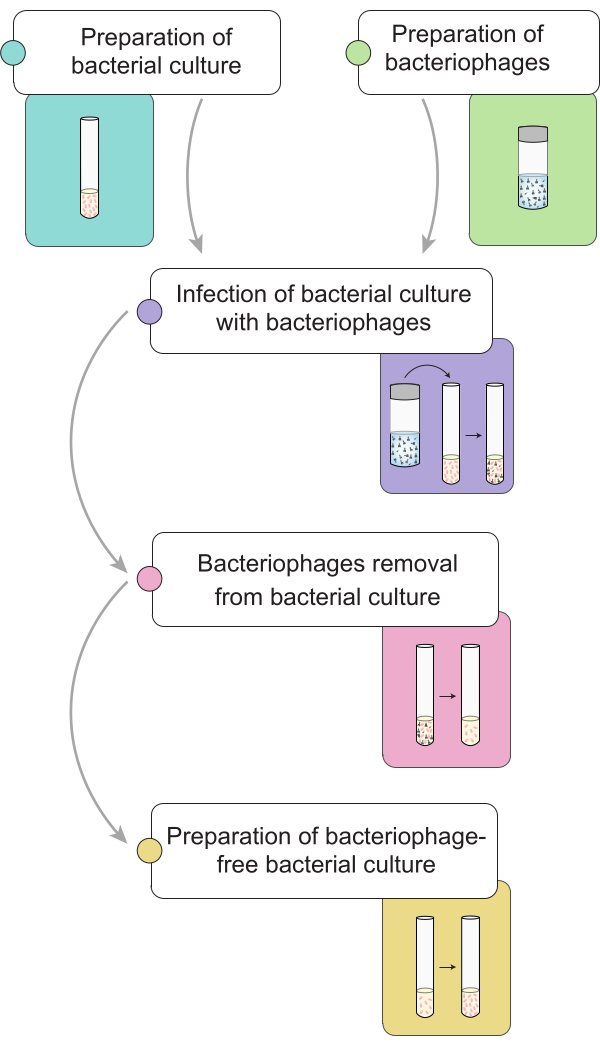
Figura 1: Breve esboço do procedimento para eliminação de bacteriófagos em culturas de Salmonella . O fluxo de trabalho é dividido em diferentes etapas: preparação da cultura bacteriana e do lisado, infecção da cultura bacteriana com bacteriófagos, remoção de bacteriófagos de culturas bacterianas infectadas e preparação de um inóculo bacteriano livre de fagos. Clique aqui para ver uma versão maior desta figura.
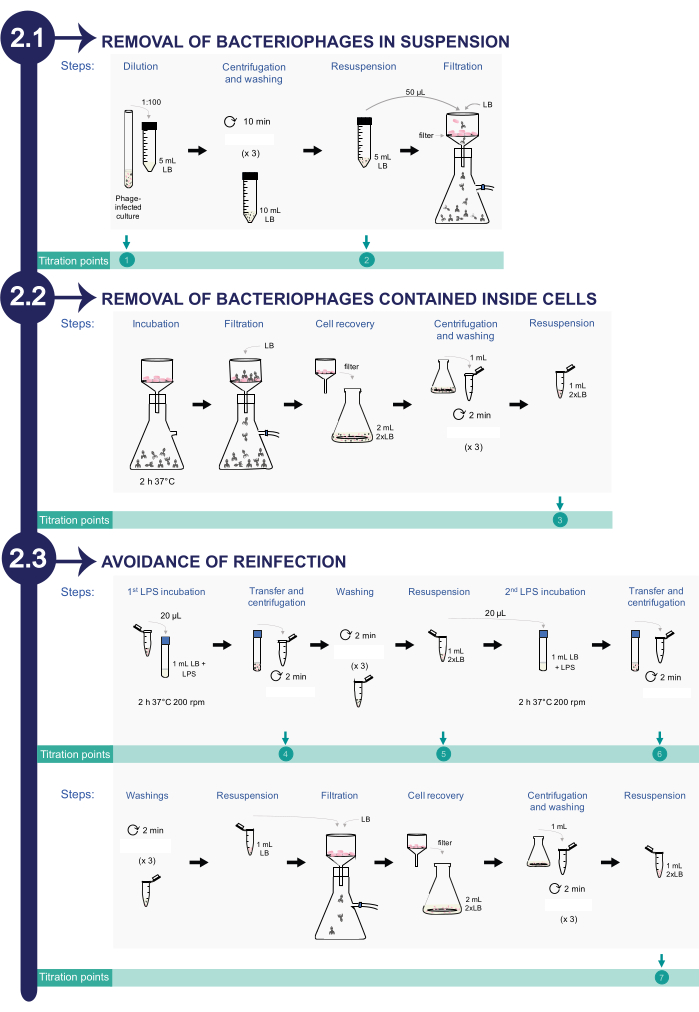
Figura 2: Procedimento para remoção de bacteriófagos de culturas infectadas de Salmonella enterica. O processo consiste em três fases: 1) Remoção de bacteriófagos em suspensão, 2) Remoção de fagos contidos dentro das células bacterianas e 3) Prevenção de reinfecção. Clique aqui para ver uma versão maior desta figura.
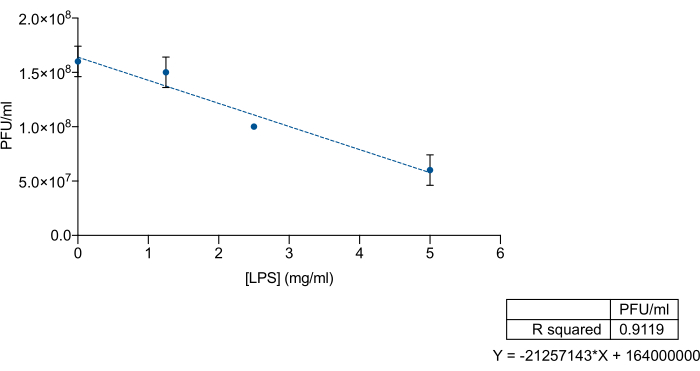
Figura 3: Ensaio de chamariz LPS para medir a eficiência do LPS comercial de Salmonella enterica para se ligar a bacteriófagos. Titulação de um lisado de 9NA (PFU/ML) em concentrações crescentes de LPS comercial de Salmonella enterica . O experimento foi conduzido em triplicata. Média e desvio padrão são apresentados. Clique aqui para ver uma versão maior desta figura.
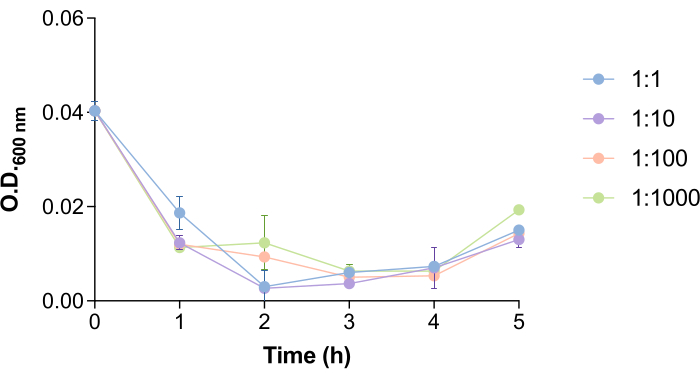
Figura 4: Tempo de lise do bacteriófago 9NA em culturas de Salmonella . Curvas de crescimento das culturas de Salmonella enterica na presença do bacteriófago 9NA nas proporções bactéria:fagos de 1:1, 1:10, 1:100 e 1:1000. O experimento foi conduzido em triplicata. Média e desvio padrão são apresentados. Clique aqui para ver uma versão maior desta figura.
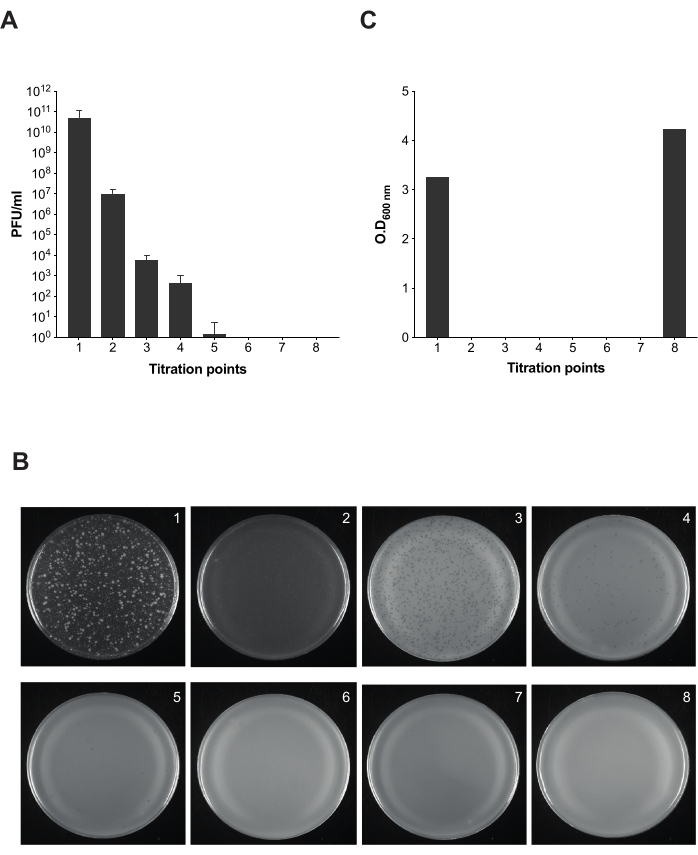
Figura 5: Ensaio de placa para teste de infectividade durante a remoção de bacteriófagos em culturas de Salmonella . (A) A titulação de oito alíquotas foi feita em diferentes pontos do protocolo (os pontos de titulação 1-7 estão marcados na Figura 2). Para este experimento, foram utilizados Salmonella enterica sorovar Typhimurium cepa ATCC 14028 opvAB::lacZ (SV8011) e bacteriófago 9NA. Os experimentos foram realizados em triplicata, e a média e os desvios-padrão são mostrados. (B) Placas de ágar mole com Salmonella enterica foram obtidas seguindo a técnica de sobreposição usando alíquotas de oito pontos de titulação. As placas correspondem da esquerda para a direita com os pontos de titulação 1-8. (C) Densidade óptica a 600 nm de cultura bacteriana em diferentes momentos do protocolo de remoção de fagos. Clique aqui para ver uma versão maior desta figura.
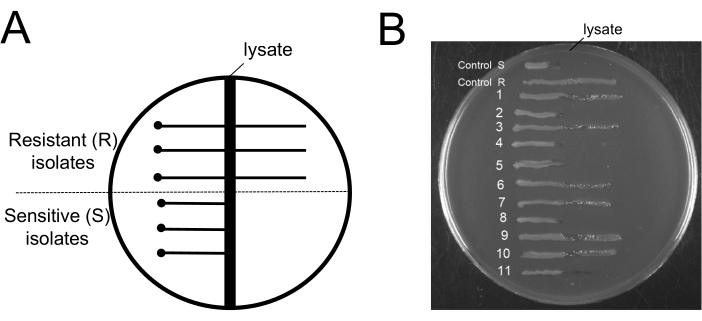
Figura 6: Teste de bactérias resistentes a fagos após procedimento de remoção de bacteriófagos. (A) Diagrama esquemático de uma placa de ágar EBU típica usada para o ensaio de ágar de faixa cruzada: a região escura vertical no centro representa a zona do lisado de 9NA. O ponto representa o local onde as células testadas são inoculadas a uma distância segura da zona de lisado, e as linhas sólidas horizontais representam as células resistentes a fagos que estão crescendo na zona lisada ou as células sensíveis a fagos que não estão crescendo além da zona de lisado. (B) Ensaios em placa EBU para testar 11 colônias foram obtidos no final do protocolo de remoção. Os controles R e S são exemplos de isolados resistentes a fagos e sensíveis a fagos, respectivamente. Clique aqui para ver uma versão maior desta figura.
Discussion
Diversas estratégias são empregadas pelos bacteriófagos para reconhecer e infectar hospedeiros bacterianos. Diferentes estruturas moleculares na superfície das bactérias podem atuar como receptores de fagos: proteínas, polissacarídeos, lipopolissacarídeos (LPS) e porções de carboidratos20. Em bactérias Gram-negativas, o LPS é um receptor comum para fagos. Além disso, outros receptores são proteínas da membrana externa, pili e flagelos21.
A interação específica entre bacteriófagos e bactérias baseada no reconhecimento de LPS8 foi explorada neste trabalho para o desenvolvimento de um protocolo altamente eficiente para a eliminação de bacteriófagos em culturas bacterianas infectadas (Figura 1 e Figura 2). Nosso protocolo não favorece a seleção de células resistentes a fagos; elimina apenas bacteriófagos (Figura 6). Tanto as células suscetíveis quanto as resistentes a fagos permanecem em cultura bacteriana após a realização deste protocolo de remoção de fagos.
A prática padrão tradicional quando ocorre uma infecção por fago é tentar eliminar todo o material contaminado, seguido de limpeza e esterilização22. O procedimento de descontaminação envolve a exposição da cultura bacteriana a condições estressantes, como altas temperaturas, a fim de eliminar parcial ou completamente as células bacterianas. Conforme descrito em resultados representativos, a etapa crucial neste protocolo é a incubação de culturas bacterianas infectadas por fagos com LPS comercial, uma substância não prejudicial para culturas bacterianas. Isso ajuda a preservar a viabilidade das culturas bacterianas e oferece vantagens significativas para aplicações industriais em fermentadores e biorreatores.
O tempo de incubação neste protocolo é de 2 h para garantir tempo suficiente para a lise do fago das células bacterianas. Se forem utilizadas diferentes estirpes bacterianas e bacteriófagos, este parâmetro deve ser considerado e definido pelo utilizador. Neste caso, deve ser efectuado um ensaio semelhante ao descrito na figura 4 antes da experiência.
Curiosamente, a eficácia desse protocolo de limpeza também pode ser analisada empregando um ensaio que monitora o conteúdo de fagos de uma determinada amostra. Nesse sentido, os biossensores epigenéticos são uma nova ferramenta para a detecção de bacteriófagos23. Um biossensor de fagos bem conhecido capaz de detectar colifagos, que usam LPS como receptor, é o sistema opvAB::gfp 13,18,23,24. Este biossensor de fagos detecta um aumento na subpopulação OpvABON na presença de fagos que usam o antígeno O como receptor. Nesse sentido, poderíamos usar uma fusão opvAB :: gfp para monitorar fagos de ligação a LPS em várias etapas deste protocolo e / ou diversos meios e condições. Essas abordagens podem ser valiosas para determinar o momento e os locais em que um protocolo eficaz pode ser necessário.
Embora o reconhecimento de LPS seja comum, os fagos também podem utilizar uma variedade de outros receptores de superfície nas células bacterianas para fixação e infecção. Aqui, usamos a Salmonella Gram-negativa como uma enterobactéria representativa e o bacteriófago 9NA que usa LPS como receptor e gatilho de ejeção do genoma. Outros fagos de enterobactérias (por exemplo, Escherichia coli T5) ligam-se frouxamente ao LPS e requerem uma proteína de membrana externa para injeção no genoma. O protocolo descrito é aplicável para bacteriófagos que reconhecem e precisam do antígeno O do LPS para infecção bem-sucedida, como 9NA, Det7 e P22 13,25,26,27. Assim, a implementação bem-sucedida deste protocolo para a descontaminação de fagos de culturas bacterianas envolve determinar se a fonte da infecção por fagos requer o reconhecimento de LPS no hospedeiro.
Em conclusão, e apesar das limitações potenciais do protocolo, nossos resultados representativos demonstram claramente que este método é uma ferramenta poderosa para limpar culturas bacterianas de Salmonella de bacteriófagos que usam LPS como receptor e gatilho de ejeção do genoma.
Disclosures
Os autores não têm nada a divulgar.
Acknowledgements
Agradecemos à Dra. Carmen R. Beuzón e Rocío Carvajal-Holguera pelas discussões e sugestões úteis. Este trabalho foi apoiado pela bolsa PID2020-116995RB-I00 financiada pelo MICIU/AEI/ 10.13039/5011100011033 e pelo VI Plano de Investigação e Transferência da Universidade de Sevilha.
Materials
| Name | Company | Catalog Number | Comments |
| 20 mL syringe | BD Discardit II | 300296 | No special requirements |
| 50 mL conical tubes | Avantor | 525-0610 | No special requirements |
| 90 x 14 mm Petri dishes | Deltalab | 200209 | No special requirements |
| Agar | Sigma-Aldrich | A1296 | No special requirements |
| Bacteriophage lysate | Minimal concentration: 109 PFU/mL | ||
| Centrifuge | Eppendorf | No special requirements | |
| Chloroform | Panreac | 131252 | No special requirements |
| Citric acid · H2O | Merck | 1.00247 | |
| Colony counter | No special requirements | ||
| Evans Blue | Sigma-Aldrich | E-2129 | |
| Flasks | No special requirements | ||
| Fluorescein sodium salt | Sigma-Aldrich | F-6377 | |
| Forceps | No special requirements | ||
| Glass tubes | No special requirements | ||
| Glass tubes for lysate | No special requirements | ||
| Glucose | Sigma-Aldrich | G7021 | |
| K2HPO4 | Merck | 1.05104.1000 | |
| K2HPO4 anhydrous | Merck | 1.05104 | |
| Lipopolysaccharide from Salmonella enterica serotype Typhimurium | Sigma-Aldrich | L6511-25 mg | Dissolved in sterile water |
| Membrane 0.45 µm | MF-Millipore | HAWP02500 | No special requirements |
| MgSO4 · 7 H2O | Merck | 1.05886 | |
| NaCl | Sigma-Aldrich | S9888 | No special requirements |
| NaNH4HPO4 · 4 H2O | Sigma-Aldrich | S9506 | |
| Peptone | iNtRON | Ba2001 | No special requirements |
| Syringe Filter 0.22 µm | Millex | SLGSR33SB | No special requirements |
| Toothpicks | No special requirements | ||
| Tryptone | Panreac | 403682.1210 | No special requirements |
| Vacuum pump | Thermo Scientific | No special requirements | |
| Yeast extract | iNtRON | 48045 | No special requirements |
References
- Kasman, L. M., Porter, L. D. . Bacteriophages. , (2024).
- Lawrence, D., Baldridge, M. T., Handley, S. A. Phages and human health: More than idle hitchhikers. Viruses. 11 (7), 587 (2019).
- Beller, L., Matthijnssens, J. What is (not) known about the dynamics of the human gut virome in health and disease. Curr Opin Virol. 37, 52-57 (2019).
- Roux, S., Hallam, S. J., Woyke, T., Sullivan, M. B. Viral dark matter and virus-host interactions resolved from publicly available microbial genomes. eLife. 4, 08490 (2015).
- Wommack, K. E., Colwell, R. R. Virioplankton: Viruses in aquatic ecosystems. Microbiol Mol Biol Rev. 64 (1), 69-114 (2000).
- Louten, J. Virus structure and classification. Essentl Human Virol. , 19-29 (2016).
- Egido, J. E., Costa, A. R., Aparicio-Maldonado, C., Haas, P. J., Brouns, S. J. J. Mechanisms and clinical importance of bacteriophage resistance. FEMS Microbiol Rev. 46 (1), 048 (2022).
- Rakhuba, D. V., Kolomiets, E. I., Dey, E. S., Novik, G. I. Bacteriophage receptors, mechanisms of phage adsorption and penetration into host cell. Polish J Microbiol. 59 (3), 145-155 (2010).
- Lindberg, A. A. Bacteriophage receptors. Ann Rev Microbiol. 27 (1), 205-241 (1973).
- Koskella, B., Meaden, S. Understanding bacteriophage specificity in natural microbial communities. Viruses. 5 (3), 806-823 (2013).
- Marcó, M. B., Moineau, S., Quiberoni, A. Bacteriophages and dairy fermentations. Bacteriophage. 2 (3), 149-158 (2012).
- Fernández-Fernández, R., et al. Evolution of a bistable genetic system in fluctuating and non-fluctuating environments. bioRxiv. , (2024).
- Cota, I., Blanc-Potard, A. B., Casadesús, J. STM2209-STM2208 (opvAB): a phase variation locus of Salmonella enterica involved in control of O-antigen chain length. PloS one. 7 (5), 36863 (2012).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods Mol Biol. 501, 69-76 (2009).
- Ackermann, H. W. Tailed bacteriophages: The order Caudovirales. Adv Virus Res. 51, 135-201 (1998).
- Wilkinson, R. G., Gemski, P., Stocker, B. A. D. Non-smooth mutants of Salmonella typhimurium: Differentiation by phage sensitivity and genetic mapping. J Gen Microbiol. 70 (3), 527-554 (1972).
- Casjens, S. R., Leavitt, J. C., Hatfull, G. F., Hendrix, R. W. Genome sequence of Salmonella Phage 9NA. Genome Announc. 2 (4), 00531-00614 (2014).
- Cota, I., et al. Epigenetic control of Salmonella enterica O-antigen chain length: A tradeoff between virulence and bacteriophage resistance. PLoS Genet. 11 (11), 1005667 (2015).
- Chan, K., Botstein, D., Watanabe, T., Ogata, Y. Specialized transduction of tetracycline resistance by phage P22 in Salmonella typhimurium. Virology. 50, 883-898 (1972).
- Bertozzi Silva, J., Storms, Z., Sauvageau, D. Host receptors for bacteriophage adsorption. FEMS Microbiol Lett. 363 (4), 002 (2016).
- Sørensen, M. C. H., et al. Bacteriophage F336 recognizes the capsular phosphoramidate modification of Campylobacter jejuni NCTC11168. J Bacteriol. 193 (23), 6742-6749 (2011).
- Ogata, S., Hongo, M. Bacteriophages of the Genus Clostridium. Adv Appl Microbiol. 25, 241-273 (1979).
- Olivenza, D. R., Casadesús, J., Ansaldi, M. Epigenetic biosensors for bacteriophage detection and phage receptor discrimination. Environ Microbiol. 22 (8), 3126-3142 (2020).
- Olivenza, D. R., et al. A portable epigenetic switch for bistable gene expression in bacteria. Sci Rep. 9 (1), 862 (2019).
- Walter, M., et al. Structure of the receptor-binding protein of bacteriophage Det7: a Podoviral tail spike in a Myovirus. J Virol. 82 (5), 2265-2273 (2008).
- Davies, M. R., Broadbent, S. E., Harris, S. R., Thomson, N. R., van der Woude, M. W. Horizontally acquired Glycosyltransferase operons drive Salmonellae lipopolysaccharide diversity. PLoS Genet. 9 (6), 1003568 (2013).
- Wahl, A., Battesti, A., Ansaldi, M. Prophages in Salmonella enterica: a driving force in reshaping the genome and physiology of their bacterial host. Mol Microbiol. 111 (2), 303-316 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved