基于琼脂糖的模型生态系统,用于在甲烷-氧反梯度中培养嗜甲烷菌
In This Article
Summary
描述了一种协议,用于准备一个简单的模型生态系统,该生态系统重建了在好氧甲烷氧化细菌的自然栖息地中发现的甲烷-氧反梯度,从而能够在空间分辨的背景下研究它们的生理学。还描述了对用于基于琼脂糖的模型生态系统的常见生化测定的修改。
Abstract
好氧甲烷氧化细菌,称为嗜甲烷菌,在生物地球化学循环中起着重要作用。在土壤和沉积物中发现的甲烷-氧反梯度中,嗜甲烷菌占据特定的环境生态位,这会影响它们在个体和社区层面上的行为。然而,研究这些温室气体减排微生物生理学的常规方法通常使用均相浮游生物培养物,这并不能准确代表环境中发现的空间和化学梯度。这阻碍了科学家对这些细菌 在原位行为的理解。在这里,描述了一种称为梯度注射器的简单、廉价的模型生态系统,它使用半固体琼脂糖来重现嗜甲烷菌自然栖息地特征的陡峭甲烷-氧反梯度。梯度注射器允许培养甲烷营养菌株并从环境样品中富集混合甲烷氧化化合物,揭示仅在这种空间分辨环境中可见的表型。该方案还报告了各种生化测定,这些测定已被修改为与半固体琼脂糖基质兼容,这对于在其他基于琼脂糖的系统中培养微生物的研究人员可能很有价值。
Introduction
生活在缺氧-含氧界面的微生物通常发挥着重要的生态作用1。一个例子是好氧甲烷氧化细菌(嗜甲烷菌),它存在于土壤和沉积物中甲烷和氧气的逆梯度中 2。这些微生物具有独特的代谢和生理特性,使它们能够利用环境中存在的气体梯度,几十年来一直是持续研究的主题 3,4,5。目前,大多数已发表的关于甲烷营养生物和甲烷氧化群落的研究都是基于对均质浮游生物培养物的研究,这些培养物往往无法捕捉其自然微生物栖息地固有的空间和化学梯度。这种限制阻碍了我们对微生物生理学的理解以及我们将基因组信息与表型性状联系起来的能力。
该协议报告了一个简单的、基于实验室的模型生态系统,它为研究特定的甲烷营养生物(如甲基单胞菌属菌株 LW13)和直接来自环境土壤样品的甲烷氧化群落创造了可重复的条件。重要的是,在梯度注射器中培养会产生同质浮游生物培养物中不存在的反梯度特异性表型6,突出了该系统揭示甲烷菌生理学新方面的能力。受先前发布的模型生态系统 7,8,9 的启发,梯度注射器是一种简化的方法,可用于从使用这种方法培养的微生物中收集化学和分子信息。
已对遗传、化学和分子分析的报道程序进行了修改,以可靠地处理在半固体琼脂糖基质中生长的微生物培养物。这些程序也可用于分析在其他基于半固体琼脂糖的系统(例如用于细菌软琼脂游泳测定的系统)中生长的细菌。将这些分析应用于空间解析的背景可能会为研究更具生态相关性的环境中的微生物生命开辟新途径。
Protocol
材料 表中列出了研究中使用的试剂和设备的详细信息。
1. 梯度注射器的制备和挤出
注:梯度注射器制备应使用无菌技术进行。
- 使用在平板上新鲜生长的 几个甲基单胞 菌属 LW13 菌落,在 18 mm x 150 mm 玻璃管中接种 6 mL 硝酸盐矿物盐 (NMS) 培养基。用血清塞和铝压接密封密封管,并使用注射器将甲烷添加到空气中甲烷的最终气氛中,使其达到 50% (v/v) 的气体。在室温下以 200 rpm 的速度摇动这种浮游液体培养物直至浑浊(约一天)。
- 将液体培养物以 1:10 的比例传代到新鲜培养基中。继续培养嗜甲烷菌的液体培养物至对数期生长(OD600 为 ~0.5)并调整至 OD600 =1.0。
- 通过取下随附的柱塞并将其保存在无菌容器中来准备注射器。将无菌 PTFE 过滤器尖端连接到注射器上,并将其放在标准试管架上,尖端朝下。
- 对于每个 10 mL 注射器,将步骤 1.2 中的 1 mL 细胞与 5 mL NMS 和 4 mL 熔融琼脂糖(0.5% m/v,冷却至 55 °C)在无菌锥形管中彻底混合。这些体积可以放大以平行填充多个注射器。
- 缓慢倒入或使用血清移液管将混合物添加到每个注射器中,直至达到 8 mL 标记。让注射器内的琼脂糖凝固(~15 分钟),然后用无菌的 20 mm 橡胶丁基胶塞盖住。使用实验室胶带将塞子固定在注射器上,并在注射器内容物上贴上标签。
- 要将甲烷添加到注射器顶部空间,请在大 (60 mL) 注射器中填充 100% CH4 ,并将 PTFE 过滤器尖端(0.2 μm,25 mm)连接到无菌针头 (23 G)。用大注射器刺穿橡胶塞,然后将第二个无菌针头穿过塞子,形成气体出口。
- 按下大注射器上的柱塞,让 20 mL 的 100% CH4 通过顶部空间冲洗,当大注射器中还剩下 1-2 mL 的 CH4 时,小心取下出口针头,以防止氧气通过出口针头回流。
- 将注射器在18°C下孵育,每天重复步骤1.6和1.7以补充甲烷。
- 要挤出琼脂糖,请用无菌 23 G 针头更换 PTFE 过滤头,并用随附的注射器柱塞更换橡胶塞。缓慢按下柱塞,以 1 mL 的增量分配到单独的无菌 1.5 mL 微量离心管中。
2. 测定反梯度气体浓度
- 测量溶解氧梯度
- 使用剃须刀片切开靠近 PTFE 过滤器的填充琼脂糖注射器(按照步骤 1.1-1.8 制备)的宽度。将打开的注射器固定在朝向 Clark 型微电极的注射泵上,开口端朝向电极尖端。
- 调整注射泵的设置,以 1 mL/min (0.6 cm/min) 的速率将注射器移向微电极;注射泵开始移动后,立即开始在 Unisense Logger 软件上记录溶解氧测量值。
- 测量甲烷梯度
- 在挤出之前,立即用连接到 23 G 针头的单向旋塞更换针头过滤器尖端,并快速将橡胶塞换成注射器柱塞。加入 8 个 1 mL 琼脂糖等分试样,以分离抽空的 12 mL 气密样品瓶,并让样品在室温下平衡 1 小时。
- 通过裂开并立即重新密封样品瓶或刺穿并快速取出针头,将样品瓶平衡至大气压。使用气密注射器将 500 μL 样品瓶顶空注入采用火焰离子化检测 (GC-FID) 的气相色谱仪中。创建源自 CH4 标准品的校准曲线,将峰面积 (pA*min) 转换为 μmol/L。
3. 对梯度进样针中的细胞进行计数
- 流式细胞术
- 如步骤 1.9 中所述,从接种有野生型或突变型 LW13 的梯度注射器中挤出 1 mL 琼脂糖片段。此外,从无细胞、无菌注射器中制备并挤出琼脂糖作为阴性对照。
- 向所有挤出的琼脂糖样品中加入 0.75 mL 0.85% (m/v) NaCl 的水溶液,并通过涡旋定质。此外,将 100 μL 转移到新的微量离心管中,并加入 900 μL 盐溶液,以 1:10 的比例稀释样品。
- 加入 3 μL SYTO9 和碘化丙啶染料的 1:1 混合物,然后在室温下避光孵育 15 分钟。为了测定每 mL 琼脂糖中的细胞数,请在水浴中对微球计数珠悬浮液进行超声处理 5 分钟。然后,在流式细胞术分析之前,向每个样品中加入 10 μL 悬浮液。
- 使用流式细胞仪10,11 分析具有以下参数的样品:触发绿色荧光,流速为 10 μL/s,颗粒分析速率低于 1,000 个颗粒/s。
- 比较 SSC 与FITC 点图在无细胞对照样品和接种的琼脂糖样品之间绘制,以绘制排除背景琼脂糖颗粒的“细菌事件”电压门。此外,为微球计数珠绘制电压门,样品之间应保持一致。
- 要确定梯度注射器内每个琼脂糖片段中的细胞浓度,请使用以下公式,注意上述方案的稀释因子为 17.7275,10-6 mL 是一个微球珠的体积。
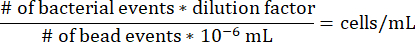
- 对梯度注射器内的菌落形成单位进行计数
- 将 1 mL 琼脂糖片段挤出到单独的无菌 2 mL 微量离心管中,加入 800 μL NMS,涡旋 10 秒以帮助移液。
- 通过向每个孔中加入 180 μL NMS 来制备无菌 96 孔板。向第一列的每个孔中加入 20 μL 稀释的琼脂糖样品,然后吸取移液混合。
- 使用多通道移液器,将 20 μL 从第一排孔转移到第二排孔中,然后移液 10 次以混合,将样品连续稀释 10 倍。继续此过程,直到板的最后一行。
- 标记包含 NMS 琼脂或所选培养基的方格板。使用多通道移液器,从 96 孔板的柱中点样 5 μL 到琼脂板上。根据琼脂板的大小,可以在同一板上点样多个柱子。
- 在空气中使用 40% 甲烷孵育板,并在 18 °C 下生长。 2-3 天后对细菌菌落进行计数,并确定每毫升的菌落形成单位 (CFU/mL)。
4. 生物分子检测检测
- 多糖测定
- 将 1 mL 琼脂糖片段挤出到单独的 2 mL 微量离心管中,并与 1 mL 1% (m/v) Na2CO3 水溶液混合。将样品加热至 80 °C 30 分钟,每 5-10 分钟涡旋一次,然后在 4 °C 下以 4,000 x g 离心 20 分钟。
- 收集上清液与三体积的 100% 乙醇混合,并在 20 °C 下孵育至少 2 小时(或过夜)。
- 通过在 4 °C 下以 16,100 x g 离心 30 分钟来收集乙醇沉淀的多糖。去除上清液并风干沉淀。将沉淀重悬于 100 μL 去离子水中。
- 使用苯酚 - 硫酸比色测定法测量每个琼脂糖片段的相对多糖含量12,13。将 50 μL 重悬提取物与 150 μL 浓硫酸和 30 μL 5% (v/v) 苯酚的水溶液混合在一个透明的 96 孔板中。
- 使用酶标仪测量 490 nm 处的吸光度,并计算每个琼脂糖片段的相对多糖含量,作为最靠近 PTFE 过滤器的琼脂糖片段吸光度的百分比。
- 蛋白质检测
- 将琼脂糖挤出到单独的 1.5 mL 微量离心管中,并将每个等分试样的 100 μL 转移到玻璃试管中。
- 使用 BCA 蛋白质测定试剂盒的试管方案测定总蛋白质浓度。用从无菌注射器挤出的琼脂糖作为稀释剂制备白蛋白 BSA 标准品。
- 细胞外 DNA 检测
- 将琼脂糖挤出到单独的 1.5 mL 微量离心管中,并将每种管 20 μL 转移到 0.2 mL 微量离心管中。
- 按照制造商的方案使用市售的 1x dsDNA 高灵敏度检测试剂盒测量 DNA 浓度。
5. RNA 提取
- 通过在 800 mL 不含 RNase 的水中混合以下内容来制备提取缓冲液:2.0 g CTAB、2.0 g 聚乙烯吡咯烷酮 (PVP 40)、81.8 g NaCl、100 mM Tris-HCl (pH 8.0) 和 20 mM EDTA。将体积提高到 1 L 并高压灭菌;储存在 4 °C。
- 分装制备的提取缓冲液,并在使用前加入 1% (v/v) 终浓度的 β-巯基乙醇。使用水浴或加热块将缓冲液加热至 65 °C。
- 按照步骤 1.9 中的程序,将琼脂糖挤出到单独的不含 RNase 的 2 mL 微量离心管中,将每个梯度注射器分成 1 mL 切片。
- 将样品在 21,000 x g , 4 °C 下离心 15 分钟,弃去上清液,将样品保持在冰上。
- 向每个沉淀的 1 mL 挤压琼脂糖中加入 600 μL 预热的提取缓冲液。加入大约 200 μL 的氧化锆/二氧化硅珠子,并使用珠子打浆器以 30 Hz/s 的速度匀浆样品 3 分钟,中途暂停将样品放在冰上 2 分钟。
- 将样品在 15,000 x g , 4 °C 下离心 2 分钟以减少起泡。向试管中加入 600 μL 氯仿:异戊醇 (24:1) 并涡旋 10 秒,提取样品。
- 将样品在 15,000 x g 、4 °C 下离心 8 分钟。小心地将上层水相转移到新的不含 RNase 的微量离心管中,并向转移的上层相中加入 600 μL 氯仿:异戊醇 (24:1) 并涡旋 10 秒。
- 将样品在 15,000 x g 、4 °C 下离心 8 分钟,然后将新的上层水相转移到新的无 RNase 微量离心管中。向转移的上层相中加入等体积的异丙醇,并在 -20 °C 下孵育样品数小时。 可选:样品可以在 -20 °C 下放置过夜。
- 通过在 16,100 x g 、4 °C 下离心样品 30 分钟来收集含 RNA 的沉淀物。
- 弃去上清液,用 300 μL 用不含 RNase 的水制成的冷 75% (v/v) 乙醇洗涤沉淀,并在 16,100 x g、4 °C 下离心 5 分钟。
- 按照步骤 5.10 再次洗涤沉淀。
- 去除乙醇上清液后,让沉淀风干 15 分钟。将沉淀溶解在 100 μL 不含 RNase 的水中。
- 按照制造商的方案,在 37 °C 下用 DNase I 处理样品 30 分钟。
- 通过添加 300 μL 酸性苯酚:氯仿:IAA(125:24:1,pH 4.5)灭活 DNase I。涡旋 10 秒并在室温下孵育 5 分钟。
- 以 16,100 x g , 4 °C 离心 5 分钟,并通过将其转移到新的无 RNase 微量离心管中来保持上层水相。
- 可选:通过将上层相混合到锥形管中,将来自所有复制注射器中同一片段的 RNA 混合。
- 加入 1 体积等于含 RNA 的上层相体积的异丙醇,并加入 7.5 M LiCl 至终浓度为 0.8 M,倒置数次以混合。
- 将样品在 -20 °C 下孵育数小时。 可选:样品可以在 -20 °C 下放置过夜。
- 将样品在 16,100 x g , 4 °C 下离心 15 分钟,然后小心弃去上清液。通过在不含 RNase 的水中加入冷的 70% (v/v) 乙醇,以 16,100 x g 、4 °C 离心 5 分钟,并去除上清液,洗涤含 RNA 的沉淀两次。
- 让沉淀在室温下风干 10 分钟,然后重悬于 50 μL 不含 RNase 的水中。将 RNA 样品储存在 -80 °C。
- 可选,具体取决于 RNA 质量:可以使用 RNA 纯化试剂盒重新纯化样品,以去除小 (<200 nt) RNA。
- 为了确认 RNA 样品不含残留 DNA,使用 1 μL 纯化的 RNA 作为模板,使用通用细菌 16S rRNA 基因引物 27F/1492R 进行 PCR 扩增。包括两个额外的 PCR 反应,分别含有 1 μL 基因组 DNA(1:100 稀释)或无核酸酶水,分别用作阳性和阴性对照。
- 使用凝胶电泳14 在 1% 琼脂糖 TBE 凝胶上运行产物。
注:没有 DNA 污染的 RNA 样品应导致样品泳道中没有条带。如果 RNA 样品显示 DNA 污染,请从步骤 5.13 开始重新处理样品。RNA 可直接用于下游分析,并且可以选择通过测量 RNA 完整性值 (RIN) 来分析以量化其完整性。
Representative Results
在这里,梯度注射器模型生态系统用于培养单个菌株(嗜甲烷菌 甲基单胞 菌属菌株 LW13)(图 1A)6,但它也可用于通过直接土壤接种来富集甲烷氧化微生物群落(图 1B)。通过测量无细胞和接种注射器中的甲烷和氧气浓度来验证甲烷-氧反梯度的存在(图 1C)。对于接种 LW13 的梯度注射器,在冲洗注射器后 1 天内形成反梯度,在孵育 3 天内变陡。在同一时间段内,在相同深度形成水平带,两种气体底物都达到最低浓度(图 1A)。陡峭的气体梯度以及甲烷和氧气的消耗超过水平带深度表明,LW13 有氧代谢甲烷并产生在均质浮游生物培养中未观察到的表型。这种表型也由从与 LW136 相同的环境样本中分离的其他嗜甲烷菌产生。不同嗜甲烷菌菌株之间水平带发展的时间和深度的应变依赖性变化表明,在空间分辨环境中培养时,水平带会受到每种微生物的特定行为的影响6。
使用流式细胞术和菌落计数 (CFU/mL) 测量整个琼脂糖塞中的细胞数(图 2A)。该方法用于比较野生型 LW13 与含有岩藻糖 4-O-乙酰转移酶 (OAT) 基因缺失的 LW13 突变菌株的细胞分布和存活率,该基因先前已被证明会影响水平带发育6。与野生型相比,LW13 的 ΔOAT 突变体在 6 天内在梯度注射器中的总体生长较低,这种效果在相同菌株的均质浮游培养物中未观察到6。在梯度注射器中培养时,突变菌株未形成与 LW13 野生型相同的明显水平带(图 2B)。在突变菌株中进行基因互补后,细胞数量和水平条带外观恢复到与野生型相似的水平。这些结果表明,梯度注射器可用于将基因与仅存在于甲烷-氧反梯度中的特定表型联系起来。
各种遗传、化学和分子技术适用于在半固体琼脂糖基质中生长的细菌。梯度注射器模型生态系统可以很容易地用于标准生物分子定量测定,包括未接种的琼脂糖作为阴性对照。测量了细胞外聚合物物质和生物膜中常见的三种不同生物分子的浓度:多糖、蛋白质和细胞外 DNA15 (图 3)。在 LW13 接种的注射器片段中,水平带的多糖含量明显高于其他片段,蛋白质或细胞外 DNA 没有显着增加。
RNA-seq 用于测量在注射器不同深度生长的 LW13 的转录差异。使用基于 CTAB 的提取缓冲液,然后进行常规苯酚:氯仿提取和沉淀步骤,实现稳健的 RNA 提取。RNA-seq 分析的结果后来用于鉴定与水平多糖条带的产生有关的基因。这些结果表明,对于创建空间分辨模型生态系统至关重要的半固体琼脂糖不会阻止通常保留用于浮游和基于板的培养物的进一步生化分析。
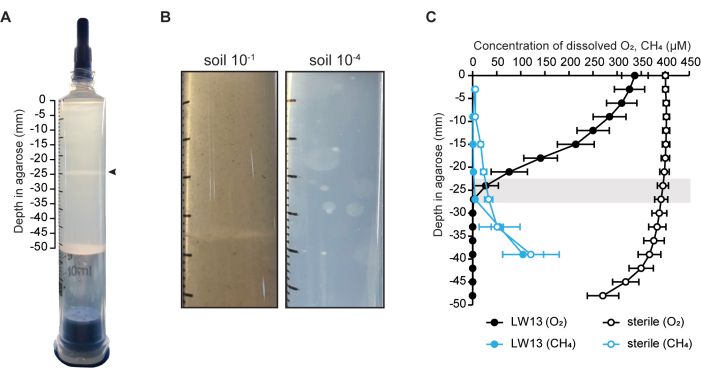
图 1:梯度注射器模型生态系统。(A) 接种了嗜甲烷菌甲基单胞菌属 LW13 的梯度注射器。用 100% 甲烷冲洗注射器后两天内形成明显的水平带(箭头)。(B) 接种了稀释 10-1 和 10-4 的土壤并孵育两周的梯度注射器的特写照片。含有更多稀释土壤的梯度注射器导致整个琼脂糖出现球形菌落,而更浓缩的土壤接种物导致明显的条带。(C) 孵育三天后 LW13 接种和无菌梯度注射器中甲烷-氧反梯度的表征。灰色条表示多糖条带所在的深度范围;数据显示 3 个独立实验的平均 SD ± 每个实验有 3 个技术重复。面板 (A) 和 (C) 是从 Beals 等人 6 修改而来的。请单击此处查看此图的较大版本。
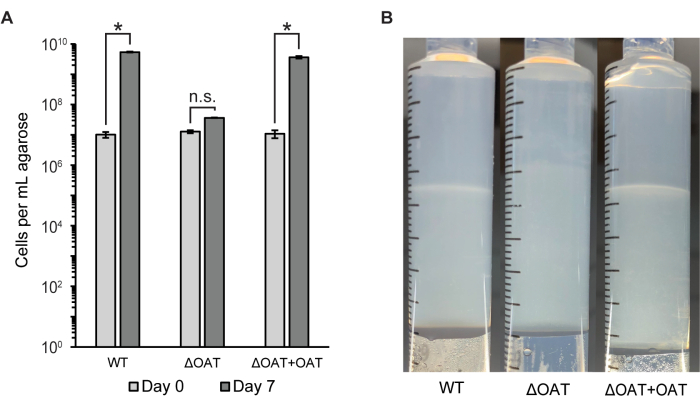
图 2:在梯度注射器中孵育后 LW13 野生型和突变体的定量。(A) 通过流式细胞术测量第 0 天和第 7 天从梯度注射器中回收的每 mL 挤出琼脂糖的 LW13 细胞总数。ΔOAT 包含岩藻糖 4-O-乙酰转移酶基因的缺失,该基因在位于多糖条带深度的细胞中高度表达。ΔOAT+OAT 包含插入 ΔOAT 基因组远端位置的 OAT 基因。*,显著不同(双尾异方差 t 检验,α = 0.05);n.s. 的 n.s. 没有显著差异。数据显示了三个独立实验的平均 ± SD 两个技术重复。(B) 孵育 7 天后,接种 LW13 野生型、ΔOAT 或 ΔOAT+OAT 的梯度注射器中的水平带发展。该图是从 Beals 等人 6 修改而来的。请单击此处查看此图的较大版本。
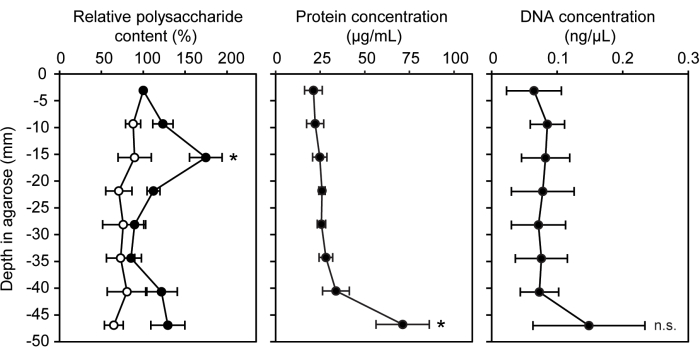
图 3:梯度注射器深度增加时生物分子的定量。孵育 7 天的 LW13 接种梯度注射器的 8 个切片中的相对多糖含量 (%)、蛋白质浓度 (μg/mL) 和 DNA 浓度 (ng/μL)(实心圆圈;空心圆圈表示无菌梯度注射器的值)。数据显示了 3 个独立实验的平均 ± SD 和 3 个技术重复。对于相对多糖含量,* 表示在等效深度下与无菌对照存在显著差异(双尾异方差 t 检验,α = 0.05)。对于蛋白质和 DNA 浓度,* 表示与包含水平带的部分存在显著差异(使用 Tukey-Kramer 事后分析的单因素方差分析);n.s.,不显著。该图是从 Beals 等人 6 修改而来的。请单击此处查看此图的较大版本。
Discussion
甲烷菌培养方法
几十年来,人们一直对营养甲烷菌进行研究,以了解它们的生理机能、它们在自然环境中的个体和群落行为,以及它们在工业应用中减少甲烷的潜力。在这些研究中,进行的大部分研究都是使用同质浮游生物培养物进行的,其中空间背景丢失。梯度注射器模型生态系统的开发是为了在实验室中复制天然嗜甲烷菌栖息地的甲烷-氧反梯度特征,使研究人员能够研究在更接近这些生物进化环境中生长的嗜甲烷菌。
在过去的 30 年里,研究人员使用各种方法在实验室中重建了甲烷-氧反梯度,其主要目标是从混合甲烷氧化联盟中分离和分类甲烷营养生物。这些方法可分为两种方法,都涉及使用相对的甲烷和氧气室:将相对未受干扰的土壤悬浮在膜上 16,17,18,或将少量土壤或纯细菌培养物接种到琼脂糖 7,8,19 中的最小培养基中。这里描述的梯度注射器方法结合了 Dedysh 及其同事9 的基于注射器的方法与 Amaral 和 Knowles8 以及 Schink 及其同事7 之前工作中甲烷菌的培养。这些方法中的后一种为在甲烷-氧反梯度中培养甲烷菌奠定了基础,并在琼脂糖栓的两侧使用甲烷和氧气的连续流动。虽然这提供了更稳定的环境,但这种方法增加了实验设置的复杂性,并且需要专用的气源。
相比之下,此处描述的梯度注射器依靠每天冲洗注射器来提供新鲜的甲烷,每个注射器只需不到一分钟,同时通过无菌 PTFE 过滤器尖端提供持续获取大气中的氧气。这种更简单的方法可能使该模型生态系统更广泛地用于在空间分辨背景下研究甲烷营养生物。所描述的方案还详细说明了可以直接对在半固体琼脂糖中孵育的细菌进行的化学和分子水平分析。因此,在分析前无需在琼脂糖基质外切除和培养细菌,从而在采样时保持气体梯度条件。
关于协议的评论
由于细菌是在聚丙烯容量法注射器内培养的,因此研究人员可以使用随附的注射器柱塞准确且可重现地分割琼脂糖塞,同时保持仍保留在注射器针筒中的琼脂糖基质的空间完整性。如果没有注射器固有的气密设计,则需要从注射器针筒中取出琼脂糖塞并切片,这给琼脂糖链段的体积带来了不确定性,并向大气中释放了无法量化的甲烷和溶解氧。通过无菌针头进行琼脂糖挤出简化了样品制备,并有助于在不剪切细菌细胞的情况下使挤出的片段均质化。该方法允许研究人员将每个接种的梯度注射器分成至少 8 个琼脂糖片段,并对在氧气和甲烷浓度范围内生长的嗜甲烷菌进行平行实验。
在优化高多糖含量琼脂糖的 RNA 提取时,发现硫氰酸胍和 TRIzol 等常见试剂会导致琼脂糖凝胶化,从而阻碍纯化柱并抵抗离心沉淀。RNA 产量和质量低也是一个问题,因为大多糖分子可以捕获核酸,而小多糖可以与 RNA20 共沉淀。相反,使用含有阳离子表面活性剂 CTAB 的提取缓冲液,该缓冲液可溶解脂质膜20;和 NaCl,它阻止 CTAB-核酸复合物形成并允许核酸沉淀,但将多糖保留在溶液中21。通过在 CTAB 缓冲液中加入 β-巯基乙醇来使 RNase 变性。对于 RNA-seq 实验,包括一个可选的基于柱的纯化步骤,以在文库制备之前排除小 (<200 个核苷酸) RNA。
限制和注意事项
虽然 NMS 和琼脂糖为培养甲烷营养细菌提供了最小的培养基基质,但此处描述的梯度注射器仅再现了甲烷营养栖息地的气体梯度,而不重现这些环境中存在的其他梯度,例如痕量金属22、盐度23 或其他营养物质24。将来可能会将这些梯度添加到类似的系统中。此外,注射器的体积(8 mL 琼脂糖)限制了每个注射器的总生物量,因此需要汇集多个注射器进行某些分析(如步骤 5.16 中所述)。虽然手持式注射器可以方便地将琼脂糖分割成 1 mL 等分试样,但其尺寸也将顶部空间限制在大约 4 mL,从而限制了可储存用于培养微生物的大量甲烷量。由于甲烷氧化速率与好氧嗜甲烷菌25 的生长速率成正比,因此建议每天补充顶空甲烷。虽然这仍可能导致甲烷限制期,但这些期可以在实验室中重现,并且可能模拟自然环境中发现的情况。
使用梯度注射器时,琼脂糖多糖的存在需要对用于分析在该系统中生长的嗜甲烷菌的分析进行一些调整。例如,需要转移小体积挤出琼脂糖的方案需要多个稀释步骤,每次稀释之间进行彻底均质化,以实现准确移液。此外,在多糖测定等情况下,琼脂糖基质固有的多糖将与硫酸-苯酚试剂反应,因此必须包含无菌、无细胞的琼脂糖阴性对照。通过加入琼脂糖水解酶 β-琼脂酶来缓解这些问题的早期尝试未成功,并为生物实验引入了一个未知变量。使用多个技术重复、彻底稀释、均质化和包含对照可用于缓解琼脂糖基质固有的大部分挑战。
应用
除了单菌株研究外,梯度注射器还可以支持多种菌株的共培养,并且可以使用土壤代替纯细菌培养作为接种物。梯度注射器模型生态系统的简单设计适用于通过使用不同的气体底物(如 H2 或 CO)代替甲烷,培养存在于缺氧和含氧环境界面的其他类型的微生物。总之,使用简单的空间分辨模型生态系统使研究人员能够研究缺氧-含氧微生物的独特生理学和代谢适应,并可用于将基因与生物体表型联系起来。
Disclosures
作者声明没有利益冲突。
Acknowledgements
这项工作得到了犹他大学化学系和 NSF CAREER Award #2339190 的启动资金的支持。我们感谢 Puri 实验室的成员进行有益的讨论。我们感谢 Rachel Hurrell(犹他大学)对流式细胞术实验的初步指导。
Materials
| Name | Company | Catalog Number | Comments |
| 1% Gas mix analytical standard | Supelco | 22561 | 1% each component in nitrogen: carbon monoxide, carbon dioxide, hydrogen, methane and oxygen |
| 100% Methane | Airgas | ME CP300 | chemically pure grade |
| 15 ppm Gas mix analytical standard | Supelco | 23470-U | 15 ppm each component in nitrogen: methane, ethane, ethylene, acetylene, propane, propylene, propyne, and n-butane |
| 1x Nitrate mineral salts | see CAS numbers below | Dissolve the following in Mili-Q water and autoclave: 0.2 g/L MgSO4·7H2O, 0.2 g/L CaCl2·6H2O, 1 g/L KNO3, and 30 μM LaCl3. Before use, add trace elements to a 1X final concentration and phosphate buffer (pH 6.8) to a final concentration of 5.8 mM. | |
| 23 G needle | BD Biosciences | 305194 | sterile, Luer-Lok |
| 500x Trace elements | see CAS numbers below | Dissolve the following in Milli-Q water: 1.0 g/L Na2-EDTA, 2.0 g/L FeSO4·7H2O, 0.8 g/L ZnSO4·7H2O, 0.03 g/L MnCl2·4H2O, 0.03 g/L H3BO3, 0.2 g/L CoCl2·6H2O, 0.6 g/L CuCl2·2H2O, 0.02 g/L NiCl2·6H2O, and 0.05 g/L Na2MoO·2H2O. | |
| 96 Well plate | CELLTREAT | 229596 | sterile |
| Acid phenol:chloroform:IAA (125:24:1) | Invitrogen | AM9720 | pH 4.5 |
| Agarose | Fisher Scientific | BP160 | molecular biology grade, CAS 9012-36-6 |
| Aluminum crimp seals | VWR | 30618-460 | 20 mm |
| Bead beater | Qiagen | 9003240 | TissueLyser III |
| Butyl rubber stopper | Chemglass Life Science | 50-143-854 | 20 mm, blue |
| Chloroform:isoamyl alcohol (24:1) | Millipore Sigma | 25666 | BioUltra, for molecular biology |
| Clark-type O2 microelectrode | Unisense | OX-500 | |
| DEPC-treated water | Thermo Scientific | R0601 | |
| DNase I (Ambion) | Invitrogen | AM2222 | |
| Flow cytometer | Beckman Coulter | CytoFLEX | |
| Gas chromatograph (flame ionization detection) | Agilent | 6890N | |
| Gastight analytical syringe | Hamilton | 81220 | 1750 TLL |
| Gastight analytical syringe needle | Hamilton | 7729-07 | 22 G, metal hub needle, 2 in, point style 5 |
| Gas-tight vials | Labco | 938W | Exetainer vial: 12 mL, round bottom |
| Glass culture tubes | Bellco Glass | 2048-00150 | 18 x 150 mm |
| LiCl precipitation solution (7.5 M) | Invitrogen | AM9480 | |
| One-way stopcock | VWR | MFLX30600-00 | inlet port: female luer, outlet port: male luer lock |
| Petri dish, square | Fisher Scientific | FB0875711A | 100 x 100 mm |
| Phosphate buffer, 0.2 M (pH 6.8) | see CAS numbers below | Dissolve the following in Milli-Q water and autoclave: 12.24 g/L KH2PO4, 26.29 g/L Na2HPO4 · 7H2O | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| PTFE syringe filter tip | Thermo Scientific | 03-050-469 | hydrophobic, pore size: 0.2 µm, diameter: 4 mm |
| Qubit 1x dsDNA High Sensitivity Assay Kit | Invitrogen | Q33230 | |
| Qubit 4 Fluorometer | Invitrogen | Q33238 | |
| RNA Clean & Concentrator-5 | Zymo Research | R1013 | |
| Serum stopper | Fisher Scientific | 03-340-302 | 20 mm |
| Syringe | BD Biosciences | 302995 | Luer-Lock, 10 mL, single use, sterile |
| Syringe pump | New Era Pump Systems Inc. | 1000-US | NE-1000 one channel programmable |
| SYTO9, propidium iodide, microspheres | Invitrogen | L34856 | LIVE/DEAD BacLight Bacterial Viability Kit |
| Zirconia/silica beads | BioSpec Products | 11079101z | 0.1 mm diameter |
| Chemical reagents | CAS number | ||
| CaCl2·6H2O | 7774-34-7 | ||
| CoCl2·6H2O | 7791-13-1 | ||
| Concentrated sulfuric acid | 7664-93-9 | ||
| CTAB, cetrimonium bromide | 57-09-0 | ||
| CuCl2·2H2O | 10125-13-0 | ||
| Ethanol | 64-17-5 | ||
| FeSO4·7H2O | 7782-63-0 | ||
| H3BO3 | 10043-35-3 | ||
| Isopropanol | 69-63-0 | ||
| KH2PO4 | 7778-77-0 | ||
| KNO3 | 7757-79-1 | ||
| LaCl3 | 10099-58-8 | ||
| MgSO4·7H2O | 10034-99-8 | ||
| MnCl2·4H2O | 13446-34-9 | ||
| Na2CO3, sodium carbonate | 497-19-8 | ||
| Na2-EDTA | 139-33-3 | ||
| Na2HPO4 · 7H2O | 7782-85-6 | ||
| Na2MoO·2H2O | 10102-40-6 | ||
| NaCl, sodium chloride | 7647-14-5 | ||
| NiCl2·6H2O | 7791-20-0 | ||
| Phenol (90% solution in water) | 108-95-2 | ||
| PVP40, polyvinylpyrrolidone | 9003-39-8 | ||
| Tris-HCl | 1185-53-1 | ||
| ZnSO4·7H2O | 7446-20-0 | ||
| β-Mercaptoethanol | 60-24-2 |
References
- Brune, A., Frenzel, P., Cypionka, H. Life at the oxic-anoxic interface: microbial activities and adaptations. FEMS Microbiol Rev. 24 (5), 691-710 (2000).
- Auman, A. J., Stolyar, S., Costello, A. M., Lidstrom, M. E. Molecular characterization of methanotrophic isolates from freshwater lake sediment. Appl Environ Microbiol. 66 (12), 5259-5266 (2000).
- Whittenbury, R., Phillips, K. C., Wilkinson, J. F. Enrichment, isolation and some properties of methane-utilizing bacteria. J Gen Microbiol. 61 (2), 205-218 (1970).
- Koo, C., Rosenzweig, A. Biochemistry of aerobic biological methane oxidation. Chem Soc Rev. 50 (5), 3424-3436 (2021).
- Strong, P. J., Xie, S., Clarke, W. P. Methane as a resource: Can the methanotrophs add value. Environ Sci Technol. 49 (7), 4001-4018 (2015).
- Beals, D. G., Puri, A. W. Linking methanotroph phenotypes to genotypes using a simple spatially resolved model ecosystem. ISME J. 18 (1), wrae060 (2024).
- Bussmann, I., Rahalkar, M., Schink, B. Cultivation of methanotrophic bacteria in opposing gradients of methane and oxygen. FEMS Microbiol. Ecol. 56 (3), 331-344 (2006).
- Amaral, J. A., Knowles, R. Growth of methanotrophs in methane and oxygen counter gradients. FEMS Microbiol Lett. 126 (3), 215-220 (1995).
- Danilova, O. V., et al. A new cell morphotype among methane oxidizers: a spiral-shaped obligately microaerophilic methanotroph from northern low-oxygen environments. ISME J. 10 (11), 2734-2743 (2016).
- Ou, F., McGoverin, C., Swift, S., Vanholsbeeck, F. Absolute bacterial cell enumeration using flow cytometry. J Appl Microbiol. 123 (2), 464-477 (2017).
- Krause, S. M. B., et al. Lanthanide-dependent cross-feeding of methane-derived carbon is linked by microbial community interactions. Proc Natl Acad Sci USA. 114 (2), 358-363 (2017).
- Masuko, T., et al. Carbohydrate analysis by a phenol-sulfuric acid method in microplate format. Anal Biochem. 339 (1), 69-72 (2005).
- Felz, S., Al-Zuhairy, S., Aarstad, O. A., van Loosdrecht, M. C. M., Lin, Y. M. Extraction of structural extracellular polymeric substances from aerobic granular sludge. J Vis Exp. (115), e54534 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), e3923 (2012).
- Costa, O. Y. A., Raaijmakers, J. M., Kuramae, E. E. Microbial extracellular polymeric substances: Ecological function and impact on soil aggregation. Front Microbiol. 9, 1636 (2018).
- Sinke, A. J. C., Cottaar, F. H. M., Buis, K., Keizer, P. Methane oxidation by methanotrophs and its effects on the phosphate flux over the sediment-water interface in a eutrophic lake. Microb Ecol. 24 (3), 259-269 (1992).
- Murase, J., Frenzel, P. A methane-driven microbial food web in a wetland rice soil. Environ Microbiol. 9 (12), 3025-3034 (2007).
- Reim, A., Lüke, C., Krause, S., Pratscher, J., Frenzel, P. One millimetre makes the difference: High-resolution analysis of methane-oxidizing bacteria and their specific activity at the oxic-anoxic interface in a flooded paddy soil. ISME J. 6 (11), 2128-2139 (2012).
- Rahalkar, M., Bussmann, I., Schink, B. Methylosoma difficile gen. nov., sp. nov., a novel methanotroph enriched by gradient cultivation from littoral sediment of Lake Constance. Int J Syst Evol Microbiol. 57 (5), 1073-1080 (2007).
- Wang, L., Stegemann, J. P. Extraction of high-quality RNA from polysaccharide matrices using cetlytrimethylammonium bromide. Biomaterials. 31 (7), 1612 (2010).
- Liyanage, N. M. N., Chandrasekara, B. C. H. W. M., Bandaranayake, P. C. G. A CTAB protocol for obtaining high-quality total RNA from cinnamon (Cinnamomum zeylanicum Blume). 3 Biotech. 11 (4), 201 (2021).
- Semrau, J. D., DiSpirito, A. A., Gu, W., Yoon, S. Metals and methanotrophy. Appl Environ Microbiol. 84 (6), e02289-e02317 (2018).
- Zhang, S., et al. Salinity significantly affects methane oxidation and methanotrophic community in Inner Mongolia lake sediments. Front Microbiol. 13, 1067017 (2023).
- Fox, A. L., Trefry, J. H. Nutrient fluxes from recent deposits of fine-grained, organic-rich sediments in a Florida estuary. Front Mar Sci. , (2023).
- He, L., et al. A methanotrophic bacterium to enable methane removal for climate mitigation. Proc Natl Acad Sci. 120 (35), e2310046120 (2023).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved