Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Rastgele Konik Tilt Yöntemiyle Exosome Kompleksi Tek Parçacık Elektron Mikroskobu İmar
Bu Makalede
Özet
Bu makale, negatif boyama elektron mikroskobu (EM) kullanarak biyolojik makromoleküllerin üç boyutlu (3B) rekonstrüksiyonu almak için standart bir yöntem açıklanır. Bu protokolde, rasgele konik eğim rekonstrüksiyon yöntemi (RCT) ile orta çözünürlükte Saccharomyces cerevisiae exosome kompleks 3D yapısı nasıl açıklayabilir.
Özet
Tek parçacık elektron mikroskobu (EM) yeniden yapılanma büyük makromoleküler kompleksleri, üç boyutlu (3D) bir yapı elde etmek için son zamanlarda popüler bir araç haline gelmiştir. X-ışını kristalografisi ile karşılaştırıldığında, bazı benzersiz avantajlar vardır. İlk olarak, tek bir parçacık EM yeniden yapılanma, özellikle büyük makromoleküler kompleksleri için X-ışını kristalografisi darboğaz protein örnek, kristalize değildir. İkincisi, büyük miktarda protein örneklerinin gerek yoktur. Kristalizasyon için gerekli proteinlerin miligram ile karşılaştırıldığında, tek parçacık EM yeniden yapılanma sadece negatif boyama EM yöntemi kullanarak nano-molar konsantrasyonlarda protein çözüm çeşitli mikro-litre ihtiyacı var. Ancak, yüksek simetri birkaç makromoleküler meclisleri olmasına rağmen, tek bir parçacık EM özellikle de simetri olmadan birçok örnekler için (1 nm den daha düşük çözünürlük) nispeten düşük çözünürlükte sınırlıdır. Bu teknik, aynı zamanda çalışmanın altında moleküllerin büyüklüğü ile sınırlıdır, yani genel olarak dondurulmuş sulu numuneler için olumsuz lekeli örnekleri ve 300 kDa 100 kDa.
Yapısı bilinmeyen yeni bir örnek için, genelde negatif boyama ile moleküller gömmek için bir heavy metal çözümü kullanın. Daha sonra örnek moleküllerin iki boyutlu (2D) mikrograflar almak için bir transmisyon elektron mikroskopta incelenir. İdeal olarak, protein molekülleri, homojen bir 3D yapıya sahip ancak farklı yönelimleri mikrograflar gösterirler. Bu mikrograflar "tek parçacıklar" olarak sayısallaştırılmış ve bilgisayarlara işlenir. Iki boyutlu bir uyum ve sınıflandırma teknikleri kullanarak, aynı görüşleri homojen moleküllerin sınıfa kümelenmiş. Onların ortalamalar molekülün 2D şekilleri sinyal artırır. Biz doğru bağıl yönelimi (Euler açıları) parçacıklar atadıktan sonra, biz, 2 boyutlu, 3 boyutlu bir sanal hacmin içine parçacık görüntüleri yeniden inşa etmek mümkün olacak.
Tek parçacık 3D rekonstrüksiyon, önemli bir adım, tek tek her parçacığın uygun yönlendirmeyi doğru atamak. Açısal sulandırıldıktan 1 ve rastgele konik tenteli (RCT) yöntem 2 de dahil olmak üzere, her parçacık görünümü atamak için çeşitli yöntemler vardır. Bu protokol, maya exosome kompleksi negatif boyama EM ve RCT kullanarak 3 boyutlu rekonstrüksiyon almak bizim uygulama açıklar. Elektron mikroskobu ve görüntü işleme protokol RCT temel prensibi izler ama yöntemi gerçekleştirmek için tek yol olduğunu belirtmek gerekir. Önce, sürekli ince bir karbon film tabakası ile kaplı bir holey karbon ızgarası kullanılarak, uranil-biçimleri protein boyutu ile karşılaştırılabilir bir kalınlığında bir tabaka haline protein örnek gömmek nasıl açıklar. Sonra örnek toplamak için bir transmisyon elektron mikroskobu eklenir untilted (0 derece) ve işleme ve maya exosome bir ilk 3D model elde etmek için daha sonra kullanılabilir olacak mikrograflar eğimli (55 derece) çifti. Bu amaçla, biz RCT gerçekleştirmek ve daha sonra arıtma yöntemini 3 uyan projeksiyon kullanarak ilk 3D model rafine .
Protokol
1. Rastgele Konik Tilt Yönteminin Prensibi
- Rasgele konik tilt yönteminin ilkesi, elektron mikroskobu içinde numunenin aynı bölgede mikrograflar, bir çift alarak gerektirir. Bir resim bir untilted konumda (Şekil 1A) numune alınır ve başka bir resim, 50 ila 70 derece (bizim durumumuzda, biz 55 derece) arasında bir açıyla eğik numune alınır. (Şekil 1B)
- Seçilen bilgisayar kullanarak, sayısallaştırılmış mikrografı çifti aynı parçacıklar ve görüntüleri yan yana konur. (Şekil 1C)
- Üç boyutlu koordinatları, untilted parçacıklar ve eğimli ortakları görüntüler eğim ekseni ve eğim açısı yönünde birbirine ilişkilidir. (Şekil 1D)
- Untilted parçacık görüntüleri uyum, bunlara karşılık gelen düzlem yerlere eğilmiş parçacıklar görüntüleri getiriyor. (Şekil 1E)
- Düzlem alanı doldurarak eğimli parçacıkların birden fazla görüntü kullanarak, molekülün üç boyutlu yapısı, arka projeksiyon algoritması kullanılarak elde edilebilir. (Şekil 1F)
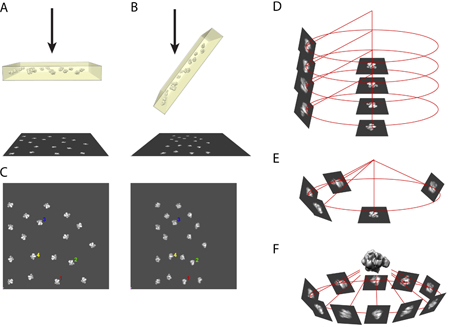
Şekil 1 ilke RCT yeniden inşası için bir örnek.
2. İnce Karbon kaplı Delikli Karbon Izgaralar hazırlayın
Gerekçe: Biz rasgele konik eğim yeniden inşası için protein örnek düzeltmek için negatif boyama yöntemi kullanır. Makromoleküller çok kuruma sırasında düzleşme olmaksızın korumak için, biz, proteinler 4 boyutu hakkında kalınlığı ile derin bir leke protein molekülleri gömmek için çalışıyoruz . Genel olarak, sürekli karbon olumsuz vitray örneklerinin yapımında kullanılır. Ancak, bu tür karbon protein parçacıkları etrafında leke kalınlığı kontrol etmek zor. Böylece ev yapımı holey olumsuz vitray örnekleri yapmak için karbon film (~ 5 mil kalınlığı) ince bir tabaka ile kaplı karbon ızgaraları. Optimal bir leke kalınlığında protein gömmek için çok daha kolay bu yüzden delikleri tarafından oluşturulan küçük bir kuyu protein çözüm ve ızgara üzerinde leke çözüm istinat izin. Ayrıca, karbon delik üzerinde ince bir tabaka büyük ölçüde arka plan gürültüsünü azaltır.
- % 0.5 'lik Formvar çözeltisi hazırlayın. Davlumbaz, 0.45 gr, 100 ml cam beher polivinil resmi reçine ve 90 mL kloroform ekleyin. Beher kapağı için alüminyum folyo kullanın ve manyetik karıştırıcı resmi reçine çözme yardımcı olmak için küçük bir heyecan çubuğunu kullanın. Reçine çözmek için yaklaşık 15 dakika sürer.
- Formvar dağılması sırasında, temiz mikroskopi cam, metanol slaytlar ve Kimwipes ile kurulayınız.
- Formvar reçine tamamen kloroform çözünmüş sonra, çözüm yüzeyine 1 ml% 50 gliserol ekleyin. Gliserol ses düzeyini ayarlama holey karbon deliklerin yoğunluğunu etkiler. Yaklaşık 1 inç derinlikte ultrasonicator bir çözüm ucu batırın ve Formvar çözüm gliserol damlacıklarının emülsiyon yapmak için 1 dakika sonikasyon maksimum güç kullanmak. Çözüm, bu adımdan sonra sütlü olur. Daha uzun sonication holey karbon delikler daha küçük boyut neden olur.
- Sonication hemen sonra, 1 saniye emülsiyon içine dikey olarak temizlenmiş cam slaytlar daldırma onları almak ve slaytlar yüzey üzerinde ince bir plastik film oluşturmak için filtre kağıdı kullanarak slaytlar 'dipleri kurulayın. Choloroform buharlaşır sonra film delik yoğunluğu ve boyutu, ışık mikroskobu altında kontrol edin. Hazırlık durumu ihtiyaçlarına göre ayarlayın. Burada anlatılan durum, genelde 3 ~ 4 mikro-metre ve 400 mesh ızgara her kare 10 ~ 20 delik çapı delik almak.
- Cam slaytlar kuru sonra, slayt yüzeyinde plastik film kenarında kesti. Distile su yüzeyinde film kaydırın. Su yüzeyinde ince bir film tabakası ışık yansıması karşı bakarak açıyla görülebilir. Film aşağı bakacak ızgaraları pürüzsüz bir yüzey ile tek tek Yeri 400 örgü bakır ızgaralar.
- Plastik film üzerine bir parça kağıt kullanarak ızgaraları ile toplayınız. Kağıdı ters çevirin ve bir petri kurumaya bırakın. Deliklere kalan gliserol çıkarın ve kağıt havada kurumaya bırakın metanol kağıt bekletin.
- Karbon evaporatör ~ 20 nanometre kalınlığında bir karbon tabakası ile Coat ızgaraları. Kalınlığı karbon gri renk ile tespit edilebilir.
- Izgaraları kloroform, karbon Formvar çıkarmak için yarım saat bekletin. Izgaraları kuruladıktan sonra, biz almış, ev yapımı holey karbon ızgaraları.
- Taze bölünmüş mika yüzeye yaklaşık 5 nanometre kalınlığında ince bir tabaka ile karbon buharlaşır.
- Holey karbon ızgaraları distile su altında dikkatlice koyun. Su yüzeyine mika yüzeyinden ince bir karbon Float ve holey karbon üzerine yatırmakyavaş yavaş ızgaraları. Davlumbaz ızgaraları Kuru.
3. Exosome Kompleksi Negatif Boyama
Gerekçe: uranil asetat, uranil formik fosfotungstik asit, amonyum molibdat, ve diğerleri de dahil olmak üzere oldukça negatif boyama EM için kullanılabilir bir kaç ağır metal leke çözümler, vardır. Farklı leke çözüm farklı benzersiz özelliklere sahiptir. Örneğin, uranil asetat parçacığın yüksek kontrast sağlar ama asitli bir ortama sevmiyorum protein kompleksleri çökebilir. Bu örnekleri için, nötr pH phosphotungestic asit, iyi bir leke çözüm olabilir. Biz ince taneciklilik molekülleri içine yüksek penetrasyon yeteneği nedeniyle doymuş uranil biçimleri (UF) çözümü seçin.
- 1 dakika için distile su kaynatın. Yavaş yavaş oda sıcaklığına kadar soğumasını. Bu adım, suyun çözünmüş oksijen kaldırmak için.
- Taze% 2 uranil biçimleri (UF) çözümü olun. 1.5 mL tüp içinde 1 ml su ve 20 mg UF karıştırın. 10 dakika karıştırın.
- 10 M potasyum hidroksit 2 mikrolitrelik ekleyerek 5.0 pH değerini ayarlayın. Hemen karıştırın. Çözelti rengi sarı olmalıdır. Çözeltinin pH çok yüksek olmamalıdır, aksi takdirde leke çökeltileri.
- Başka bir 10 dakika süreyle vortexer tüp koyun.
- 10 dakika süreyle maksimum hızda bir masaüstü santrifüj çözüm Spin.
- 0.2 mikrometre PVDF membran yoluyla çözüm Filtresi. Bu taze UF çözümdür. Işık kirliliğini önlemek için, bir parça alüminyum folyo çözüm tüp örtün. Çözümü, aynı gün içinde kullanılmalıdır.
- Glow deşarj biri ince karbon-over-holey akımı 25 mA 30 saniye boyunca parlayan bir deşarj cihazları kullanarak karbon ızgara.
- Bankta, temiz parafilm bir parça koyun. 3 damlacıklar 50 mikrolitrelik UF leke çözüm parafilm üstüne koyun.
- Seyreltme tamponu (25 mM Tris-HCl, pH 7.5, 100 mM NaCl, 2 mM DTT) 50 ~ 100 nM bir konsantrasyon exosome kompleks sulandırınız. Kızdırma taburcu ızgara üzerinde seyreltilmiş protein 4 mikrolitrelik koyun. Örnek bir dakika süreyle ızgara kalsınlar. (Not: Böyle bir moleküllerin nihai konsantrasyonu genellikle negatif boyanan bir ızgara üzerinde iyi dağılmış parçacıklar optimal yoğunluk verir uranil format veya uranil acecate leke çözüm için, fosfat ya da genel olarak yüksek tuz konsantrasyonu (0.5 M fazla) iyi boyama sonuçları elde etmek için iyi değil. Deneyimlerimiz HEPES veya Borular uranil leke çözümleri ile iyi çalışır olduğunu göstermektedir.)
- Kalan çözüm ızgara kenarından kurulayın ve leke damlacıklarının hemen ızgara çevirmek ve her damla yaklaşık 10 saniye süreyle ızgara durulama için bir parça filtre kağıdı kullanın.
- Son durulama sonra başka bir 1 dakika süreyle ızgara üzerinde leke kalmasına izin ve sonra filtre kağıdı bir parça uzakta leke kurulayın. Iyi bir derin leke sonuçlar elde etmek amacıyla, ızgara yüzeyinde leke çözüm ince bir tabaka tutun. Izgara, davlumbaz hızlı kurumasını bekleyin.
4. Exosome Kompleksi Elektron Mikroskobu
Gerekçe: devrilir bir aşaması ile herhangi bir transmisyon elektron mikroskobu tenteli RCT yeniden inşası için örnek çiftleri toplamak için kullanılabilir. Teorik olarak, yüksek açılı bir numune, daha iyi veri toplamak için yatırılabilir. Uygulamada, numune tutucu ve ızgara geometri tasarımı nedeniyle, maksimum uçlarıyla açısı 50 ila 70 derece sınırlıdır. Bu protokol, biz sadece bir FEI Tecnai-12 elektron mikroskobu kullanarak prosedürü tarif. Mikroskopların diğer modeller için, işlemleri, proje ve cihaz mülkiyet ihtiyacına göre ayarlanması gerekir.
- Numune tutucu örnek ızgara koyun ve sonra bir FEI Tecnai -12 elektron mikroskobu sahibinin koymak. Mikroskop 120 kV işletilmektedir. Biz Gatan Ultrascan4000 CCD kamera ile fotoğraf çekmek için kullanabilirsiniz. Dijital Mikrografik arayüzü, 'config kamera' iletişim 'dikey ekseni etrafında Flip' doğru ellilik belirlenmesini sağlamak için işaretlenmemiş olduğundan emin olun. (Not: okuyucu 3D model almak için SPIDER RCT rekonstrüksiyon prosedürleri dayanmaktadır özellikle bu önemli)
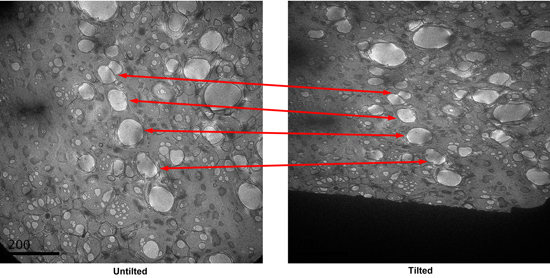
- Iyi lekeli kareler bulmak için düşük büyütme örnek ızgara kontrol edin. Bu tür kareler yaklaşık 1 boyut delik bir düzine ~ 2 mikrometre ve onları koyu leke alanlarda olmalıdır. (Şekil 2)

Şekil 2 iyi ve kötü lekeler gösteren bir kılavuz kareler düşük bir büyütme mikrografı. - FEI Kullanıcı Arayüzü düşük doz modunu açın ve arama hizalamak, odak ve düşük doz modunda pozisyon maruz. Biz, pozlama ve arama için kırınım 1,5 metre kamera süresini odak, 52.000 150.000 büyütülerek kullanın. Pozlama süresi 1 saniye olarak ayarlanır. Maruz poz odak pozisyonu 2 mikrometre ayarlayınition devrilir ekseni boyunca.
- Arama modunda iyi boyanması ile delik bulun ve kaydedebilirsiniz. Iyi delikler genellikle koyu leke degrade arama modu altında gözlenen var. Meydanın bir CCD resim çekin. 55 dereceye kadar numune Tilt Başka bir resim çekmek. Iki fotoğraf, iki mikrograflar eşleştirilmiş delik belirlemek. (Şekil 3)
Şekil 3 arama modunda bir kare mikrograflar bir eğim çifti. Sorumlu eşleştirilmiş delikleri gösterilir. - 0 derece geri sahne yatırın. Yüksek büyütmede arama modunda tanımlanan her delik fotoğraf çekmek için düşük doz kiti kullanarak. Kullanılan bulanıklaştırma yaklaşık -0.7 mikrometre.
- Tüm delikleri resimleri çekildikten sonra, 55 dereceye kadar sahne yatırın. -1.2 Hakkında mikrometre bir bulanıklaştırma untilted olanlar aynı büyütme eğimli örneğinin mikrograflar çekin. Düşük büyütme mikrograflar desenler dayalı mikrograflar ilgili eğimli çiftleri belirleyin. Ev yapımı holey karbon ızgaraları düzensiz desen korelasyon yardımcı olur. Mikrograflar eğim çift inceleyin ve sığ lekeli alanlar (parçacıklar eğimli koşul altında onların etrafında bir hale görünen) gibi kötü lekeler mikrograflar kaldırmak. (Şekil 4)
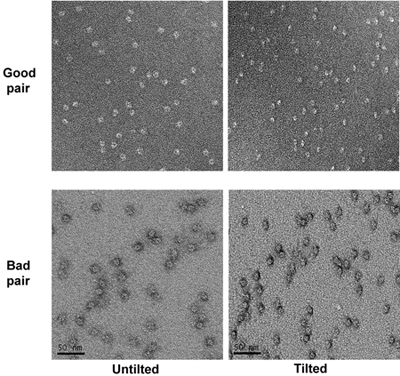
Şekil 4 yüksek büyütmede numune mikrograflar iki eğim çiftleri. Iyi ve kötü mikrograflar işaretlenmiştir.
5. Veri Görüntü İşleme
Gerekçe: bilgisayar RCT yeniden yapılanma gerçekleştirmek için farklı seçenekleri ve yazılım paketleri vardır. En genel olarak kullanılan ÖRÜMCEK 5. ÖRÜMCEK RCT gerçekleştirmek temel bir protokol http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html web sayfası bulunabilir . ÖRÜMCEK RCT gerçekleştirmek ayrıntılı bir protokol Şeyh ark makalede açıklanan 6 protokolde, IMAGIC-5 7 ve protokol video versiyonu ÖRÜMCEK bir kombinasyonunu kullanabilirsiniz. Biz de sadece protokol metni sürümü ÖRÜMCEK kullanmak alternatif bir prosedür sağlar.
- Programları Kur. ÖRÜMCEK Gatan Dijital görüntü görüntü formatı görüntü biçimini değiştirmek için EMAN 8 paket proc2d kullanın. IMAGIC-5 2D hizalama yapmak için kullanılır. ÖRÜMCEK 3D yeniden yapılanma ve arıtma yapmak için kullanılır.
Alt bölüm 1: Toplama tenteli parçacıkların çiftlerini.
- *. Dm3 Gatan Dijital görüntü EMAN proc2d komutunu kullanarak ÖRÜMCEK görüntü biçimi dönüştürme. Eğik ve untilted çifti sırasıyla *** t.spi ve *** u.spi desen olarak adlandırılır.
- ÖRÜMCEK program paketi içinde dağıtılan 'İNTERNET' programı kullanarak parçacık çiftleri seçin. Talimat izleyin Koordinatlar dcu ***. spi ve DCT ***. spi sırasıyla untilted ve eğimli parçacıklar için otomatik olarak kaydedilir. Teta sahip olmadığından, eğim çiftleri arasındaki WEB montaj sırasında üç açıları son rekonstrüksiyon doğru ellilik garanti etmez. Dcb ***. spi başka bir belge, eğimli ve untilted parçacıklar arasındaki eğim açısı (Not içerir işareti (pozitif değer) ve phi ve gama, onlara belirlenen başlangıç değerleri güveniyor. eğimli mikrografı bulanıklaştırma degrade yönünde incelenmesi doğru elde etmek için doğru başlangıç değerleri takmadan önce phi ve gama belirlemenize yardımcı yeniden yapılanma Şekil 5 ellilik doğru kongre göstermektedir.)
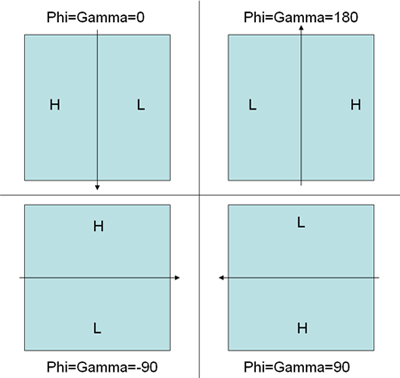
Şekil 5 bulanıklaştırma degrade tarafından belirlenen montaj modu seçmek WEB eğim çifti parçacık açısı belirlenmesi kuralı açıklamak için bir resim. L düşük Odak alanı temsil ederken H yüksek Odak alanı temsil eder. Ok eğilme ekseni temsil eder. PHI ve GAMMA ilgili doğru ilk açıları her düzeni için gösterilir. - ÖRÜMCEK script web sayfası gösterildiği gibi modifiye edilmiş bir sürümünü kullanarak tüm aldı partiküller dışarı Kutusu http://www.wadsworth.org/spider_doc/spider/docs/partpick.html . Komut untilted ve eğimli parçacık yığınları u.spi ve t.spi olarak kaydeder. Mikrografı parçacıkların sayıları particle_list.spi kaydedilmiş olmalıdır. (Not: Bu doğru Euler açısı dosyaları üretmek için çok önemlidir.RCT.)
Sub-Bölüm 2: untilted parçacık görüntülerin iki boyutlu bir uyum ve sınıflandırma.
- Untilted parçacıklar Imagic-5 paket em2em programını kullanarak IMAGIC-5 formatına dönüştürün. Hizalayın ve yinelemeli IMAGIC-5 programları (Ek A) kullanılarak homojen sınıflara parçacıklar sınıflandırmak. IMAGIC-5 msa-isimleri-in-class komutunu kullanarak, sınıfları imagic_classes.lis olarak kaydetmek parçacıklar, masa arama oluşturmak için. (Not: her sınıfın sınıflandırılması ve uyum aynı şekli ile parçacıkların sayısını artırmak gibi bir sınıf farklılıkları azaltmak Varyans harita sınıfın kalitesi hakkında bilgi verebilir.)
- IMAGIC-5 başlık komutunu kullanarak her parçacığın uyum çevirme ve döndürme değerler için bir komplo dosyası (video gösterimi ali_50.plt) oluşturun.
- ÖRÜMCEK belge dosyaları base_file ***. spi sınıfları look-up tablosu dağıtılan bir perl betiği lis2spi.pl kullanarak dönüştürme http://cryoem.berkeley.edu .
- ÖRÜMCEK belge dosya ali_50.spi dağıtılan bir komut dosyası plt2spi.pl kullanarak içine adım 5.6 çeviri ve dönme değerleri için oluşturulan çizim dosyadan her parçacığın uyum çeviri ve dönme değerleri dönüştürme http://cryoem.berkeley.edu .
Elimizde bu iş için en iyi performansı verir çünkü 2B uyum ve sınıflandırılması için IMAGIC-5. ÖRÜMCEK iki boyutlu bir uyum ve sınıflandırma için alternatif strateji http://www.wadsworth.org/spider_doc/spider/docs/align.html bulunabilir . Ayrıca exosome untilted partikülleri 2B analizi gerçekleştirmek için SPIDER kullandık. Aşağıda basit bir işlemdir.
Alternatif 5.5) açıklandığı gibi referans ücretsiz uyum kullanın görüntüleri aynı hizaya getirin. İki basit programlar bulunabilir Bir belge dosya angular_file.spi tüm parçacıkların dönme ve kayma kaydedin.
Alternatif 5.6) açıklandığı gibi aynı görüşü ile gruplar halinde hizalanmış parçacıklar sınıflar Biz sınıflandırılması için K-ortalamalar yöntemi kullandık. Generate base_file ***. spi sınıflandırma dayalı.
Alt-bölüm 3: Üç boyutlu rekonstrüksiyon eğimli parçacık görüntüleri kullanarak.
- Band-pass filtresi, maske ve merkez IMAGIC-5 untilted partiküller için eğimli parçacıklar. Başlıklı partiküller için yeni bir veri kümesi oluşturun. (Not: Bu adım isteğe bağlıdır ÖRÜMCEK yapılabilir.)
- Sınıflar look-up tablosu belgeleri ali_50.spi adım 5.3 ve Ek-B'de olarak ÖRÜMCEK komut dosyası kullanarak adım 5.4 parçacık liste dosya partile_list.spi web tarafından oluşturulan dcb ***. spi dosyaları anglular belge dosyaları oluşturun
- Açıklandığı gibi her sınıfın yeniden yapmak ÖRÜMCEK yeniden yapılanma scriptler kullanın http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Parçacıkların Her sınıf bir 3D rekonstrüksiyon hacmine katkıda bulunuyor. 3D modelleri UCSF-Chimera 9 incelenebilir. Euler açısı (0,0,0) 3 boyutlu model iki boyutlu projeksiyon rekonstrüksiyonun kalitesini doğrulamak için untilted parçacıklar ilgili 2B sınıf ortalaması ile karşılaştırıldığında olabilir. Benzer hacimleri bul hizalamak ve prosedürü izleyerek ilk ciltleri üretmek için onları birleştirmek http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html .
Alt-bölüm 4: untilted parçacık görüntüleri kullanarak 3 boyutlu rekonstrüksiyon iyileştirildi.
- Daha yüksek bir çözünürlük volum almak için untilted partiküllere karşı birleştirilmiş ilk hacmi projeksiyon eşleşen arıtma yapınaçıklandığı gibi ÖRÜMCEK komut dosyalarını kullanarak eksik koni ve düzleşme artifakı olmadan e http://www.wadsworth.org/spider_doc/spider/docs/techs/recon/refine.html .
6. Temsilcisi Sonuçlar:
RCT yöntemi kullanarak, toplam 5.000 eğim çifti (Şekil 6) exosome yaklaşık 50 rekonstrüksiyonlar elde edilmiştir. 50 3D modelleri, biz esas olarak iki ortogonal manzarasına sahip ızgara üzerinde karmaşık oturma farklı yönlerde görebilirsiniz. Düzleşme artifakı karbon yüzeyine dik yönde birçok birimlerin de saptanabilir. Biz hizalama yapılır ve dik kez ilk iki cildi oluşturmak için 3D hacimleri birleştirilmesi. 5.000 untilted parçacık görüntüleri kullanarak, hem ilk modelleri (Şekil 7) yaklaşık 18 Angstrom çözünürlükte exosome aynı nihai 3 boyutlu rekonstrüksiyon elde edilmiştir. Maya exosome mimari yapısı ortaya ve RNA substrat işe alım yolu 10 üzerine bilgi sağlamaktadır.

Şekil 6 50 RCT rekonstrüksiyon exosome kompleks 3D modeller.
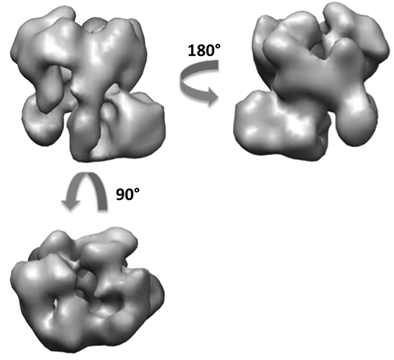
Şekil 7 arıtma sonra exosome kompleks 3D rekonstrüksiyon.
Ek:
Ek A. IMAGIC-5 2D uyum ve sınıflandırma için komut dosyası.
Dosya: auto_align_i.sh
Dosya için buraya tıklayın
Ek B. SPIDER 3D rekonstrüksiyon için açısal dosyasını oluşturmak için komut dosyası.
Dosya: generate_angular_file.spi
Dosya için buraya tıklayın
Tartışmalar
Bu yazıda, numune hazırlama, ayrıntılı protokol ve negatif boyama elektron mikroskobu kullanarak exosome kompleks üç boyutlu rekonstrüksiyon sunuyoruz. Bu yöntemi kullanarak, yapının herhangi bir ön bilgi olmadan rastgele konik eğim yöntemi kullanarak 3 boyutlu rekonstrüksiyon aldı. Rastgele konik eğim yöntemi mutlaka homojen bir örnek gerektirmez, ancak aşağıdaki projeksiyon eşleşen arıtma adım yüksek çözünürlük elde etmek için homojen bir numune gerekir.
Ol...
Açıklamalar
Teşekkürler
Yazarlar tam protokolleri kurmak için onların yardımına Yale Üniversitesi'nde ilk protokolleri ve Wang laboratuar kurmak yardımcı UC-Berkeley Nogales laboratuar üyelerine teşekkür etmek istiyorum. Ayrıca, verdikleri destek için Yale Tıp Okulu'nda cryo-EM tesis ve Yüksek Başarımlı Hesaplama Merkezi değnek kabul etmiş oluyorsunuz. HW Smith Aile Awardee.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Polyvinyl Formal Resin | Electron Microscopy Sciences | 63450-15-7 | |
| Uranyl Formate | Electron Microscopy Sciences | 22451 | |
| Superfrost Microscope Slides | Thermo Fisher Scientific, Inc. | 4951F-001 | |
| 400 mesh grid regular | SPI Supplies | 3040C | |
| Carbon coater Auto 306 | Edwards Lifesciences | ||
| Tecnai-12 Electron Microscope | FEI | ||
| Glow Discharger | BAL-TEC | Sputter Coater SCD 005 |
Referanslar
- van Heel, M. Angular reconstitution: a posteriori assignment of projection directions for 3D reconstruction. Ultramicroscopy. 21, 111-123 (1987).
- Radermacher, M. Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J Electron Microsc Tech. 9, 359-394 (1988).
- Penczek, P. A., Grassucci, R. A., Frank, J. The ribosome at improved resolution: new techniques for merging and orientation refinement in 3D cryo-electron microscopy of biological particles. Ultramicroscopy. 53, 251-270 (1994).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative staining and image classification - powerful tools in modern electron microscopy. Biol Proced Online. 6, 23-34 (2004).
- Frank, J., Radermacher, M., Penczek, P., Zhu, J., Li, Y., Ladjadj, M., Leith, A. SPIDER and WEB: processing and visualization of images in 3D electron microscopy and related fields. J Struct Biol. 116, 190-199 (1996).
- Shaikh, T. R., Gao, H., Baxter, W. T., Asturias, F. J., Boisset, N., Leith, A., Frank, J. SPIDER image processing for single particle reconstruction of biological macromolecules from electron micrographs. Nat Protoc. 3, 1941-1974 (2008).
- Heel, M. v. a. n., Harauz, G., Orlova, E. V., Schmidt, R., Schatz, M. A new generation of the IMAGIC image processing system. J Struct Biol. 116, 17-24 (1996).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: semiautomated software for high-resolution single-particle reconstructions. J Struct Biol. 128, 82-97 (1999).
- Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C., Ferrin, T. E. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25, 1605-1612 (2004).
- Wang, H. W., Wang, J., Ding, F., Callahan, K., Bratkowski, M. A., Buttler, J. S., Nogales, E., Ke, A. Architecture of the yeast Rrp44 exosome complex suggests routes of RNA recruitment for 3' end processing. Proc Natl Acad Sci USA. 104, 16844-16849 (2007).
- Scheres, S. H., Nunez-Ramirez, R., Sorzano, C. O., Carazo, J. M., Marabini, R. Image processing for electron microscopy single-particle analysis using Xmipp. Nat Protoc. 3, 977-990 (2008).
- Yoshioka, C., Pulokas, J., Fellmann, D., Potter, C. S., Milligan, R. A., Carragher, B. Automation of random conical tilt and orthogonal tilt data collection using feature-based correlation. J Struct Biol. 159, 335-346 (2007).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır