Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Genomik DNA 5-hydroxymethylcytosine seçici Yakalama
Bu Makalede
Özet
Açıklanan kolay ve yoğunluk bağımsız zenginleştirme için bir biyotin bağlayıcının aktarmak tıkırtı kimya ve ardından, 5-HMC için bir azit-glikoz aktarmak için β-glukoziltransferaz (β-GT) ile iki aşamalı bir etiketleme bir işlemdir. Bu etkili ve spesifik bir etiketleme yöntemi nesil dizileme ile son derece düşük arka plan ve yüksek verimlilik epigenomic haritalama ile 5-HMC zenginleşmesine olanak sağlar.
Özet
5-metilsitozin (5-mC) insan genomik DNA'daki toplam sitozinler arasında ~% 2-8 oluşturan ve gen ekspresyonu, genom bütünlüğünün korunması, ebeveyn baskılama, X kromozomu inaktivasyonu, düzenlenmesi de dahil olmak üzere biyolojik fonksiyonları, geniş bir yelpazede etkiler gelişme, yaşlanma ve kanser 1. Son zamanlarda, bir okside 5-mC, 5-hydroxymethylcytosine (5-HMC), varlığında embriyonik sap (ES) hücreleri ve nöronal hücreler 2-4, özellikle de memeli hücrelerinde tespit edilmiştir. 5-HMC TET aile demir (II) / α-ketoglutarat-bağımlı dioxygenases 2, 3 ile 5-mC katalizli oksitlenmesi ile elde edilir. 5-HMC embriyonik kök (MES) hücre normal hemopoeziste ve maligniteler ve zigot gelişimi 2, 5-10 bakımı dahil olmak üzere önerilmiştir. Iyi 5-HMC fonksiyonunu anlamak için, güvenilir ve kolay sıralama sistemi esastır. Geleneksel bisülfit dizileme 5-mC 11 5-HMC ayırt edemez 12 glikoz parçası ekler bir bakteriyofaj enzim yararlanarak, etiketlemek ve 5-HMC yakalamak için oldukça etkili ve seçici kimyasal bir yaklaşım geliştirdik.
Burada 5-HMC seçici kimyasal etiketleme için basit bir iki aşamalı bir prosedür açıklanmaktadır. Birinci adımda etiketleme, genomik DNA 5-HMC 5-HMC için 6-azit-glikoz aktaran bir şekilde, β-GT, T4 bakteriyofaj bir glukoziltransferaz ile bir 6-azit-glikoz katalize ile etiketlenir Modifiye kofaktör, UDP-6-N3-GLC (6-N3UDPG). İkinci adımda, biyotinilasyon, bir disülfid biyotin linker klik kimyası ile azid grubu eklenir. Iki adım genomik bölgelerde 5-HMC bolluğu bakılmaksızın etiketlenmesi ve son derece düşük arka plan vererek tamamlamak için lider, son derece spesifik ve etkili. 5-HMC takibi biyotinilasyon, 5-HMC içeren DNA parçaları daha sonra seçici yakalanırbir yoğunluk-bağımsız bir şekilde streptavidin boncuklar kullanarak. Sonuçta ortaya çıkan 5-HMC-zenginleştirilmiş DNA fragmanları, yeni nesil dizi analizi de dahil olmak üzere aşağı akım analizleri için kullanılabilir.
Bizim seçici etiketleme ve yakalama protokol çeşitli / değişken 5-HMC bolluğu ile genomik DNA herhangi bir kaynak için geçerli yüksek hassasiyet, bahşeder. Bu protokolün amacı ve mansabındaki uygulaması (yani., Yeni nesil dizileme genomunda 5-HMC dağıtım haritaya) olsa da, tek-molekül, bir gerçek zamanlı SMRT (DNA) dizi ile uyumlu 5-HMC tek-baz çözünürlük sıralama sunma yeteneğine.
Protokol
1. Genomik DNA Parçalanma
İstediğiniz bir boyut aralığına sonication kullanarak Fragment genomik DNA genom dizileme platformu için uygundur. (Genellikle ~ 300 bp ile sonikasyon.)% 1 agaroz jel (Şekil 1) üzerinde dağınık genomik DNA'nın boyutu dağılımı kontrol edin.
2. DNA Preparation
Genomik DNA 5-HMC bolluğu dayalı başlayarak DNA miktarları belirleyin. 5-HMC düzeyleri farklı doku tiplerinin anlamlı farklılık olduğu, başlangıç DNA miktarları numunelerin 5-HMC seviyeleri bağlıdır. Örnekler için Tablo 1'e bakınız.
3. β-GT katalizörlü reaksiyonu (Glikoz Transferi Reaksiyon)
- 1 saat için bir 37 ° C su banyosu içinde Tablo 2 ve inkübe de gösterildiği gibi, karışım pipetleme ile karıştırın.
- Inkübasyondan sonra, DNA 10 mg başına kullanarak, QIAquick Nükleotid Kaldırma Kiti ile reaksiyonu temizlemeksütun. Kolon başına 30 ul su Zehir ve birleştirir.
4. Biyotinilasyon Reaksiyon (Tıklayınız Kimya)
- 150 uM'lik bir son konsantrasyon (yani, 30 ul DNA solüsyonu başına çözelti çalışma 5 ul) ile yıkandı, DNA çözeltisi (adım 3) içinde DBCO-SS-PEG3-Biotin konjugat çalışma çözeltisi (1 mM) ilave edilir.
- 2 saat boyunca 37 ° C su banyosunda pipetleme ve inkübe Mix by.
- QIAquick Nükleotid Kaldırma Kiti ile reaksiyonu temizleyin. İdeal toplam elüsyon hacmi 100 ul olduğunu.
- Mikrolitre ölçekli spektrofotometre (örn., Nanodrop) kullanılarak kurtarıldı DNA miktarını ölçmek.
5. 5-HMC-içeren DNA Yakalama
- Üreticinin talimatlarına göre 1X B & W tampon 1 ml Dynabeads MyOne Streptavidin C1 50 ul 3 kere yıkayın. Manyetik bir stand ile boncuk ayırın.
- Kurtarılan biotinlenmiş D 2X & B tamponu eşit hacimde ekleYıkanmış boncuklar NA (100 ul).
- Bir rotator üzerine nazik rotasyonu ile oda sıcaklığında 15 dakika inkübe edin.
- Manyetik bir stand ile boncuk ayırın ve 1X B & W tampon 1 ml boncuk 3 kere yıkayın.
- Bir döndürücü üzerine nazik dönme ile, oda sıcaklığında 2 saat süreyle taze hazırlanmış, 50 mM DTT 100 ul olarak boncuklar ile inkübe ederek DNA Zehir.
- Manyetik bir stand ile boncuk ayırın. DTT kaldırmak için üretim talimat doğrultusunda bir Mikro Bio-Spin 6 Sütun üzerine solvent ve yük aspire. Hedef DNA artık çözelti içindedir.
- EB tamponu 10 ul Qiagen MinElute PCR Arıtma Kit ve elute DNA tarafından önceki adımdaki Elüsyonlanan DNA arındırın. Konsantrasyonunun 20 ng / ul daha yüksek ise Qubit, florimetre veya Nanodrop kullanılarak DNA ölçülmesine imkan verirler. DNA aşağı genom sekanslama kütüphane hazırlanması için hazır hale gelir.
6. Temsilcisi Sonuçlar
Kalite o takdirdef genomik DNA yüksek, β-GT ve biyotinilasyon tepkilerden sonra tipik kurtarma verimleri ~% 60-70 vardır. Ancak, yakalama verim numunelerin 5-HMC seviyelerine bağlı olarak farklı doku tipleri ile önemli ölçüde değişmektedir. Tipik olarak, beyin genomik DNA için yakalama verimliliği ~ 4-9% ve bazı aşırı durumlarda, verimliliği% 12'ye kadar çıkabilir. ES hücreleri için, ortalama yakalama verim sinir hücreleri için ~% 0.5 'e aksine, ~% 2-4 olup. Görülen düşük verim şimdiye kadar kanser hücrelerinden genomik DNA için idi. Tüm zenginleştirilmiş DNA standardının yeni nesil kütüphane hazırlık protokolleri için hazırdır. Buna ek olarak, yakalanan DNA ayrıca, ilgili primerlerin mevcut olduğunda giriş DNA ile karşılaştırıldığında bazı parçalar zenginleştirme tespit etmek için gerçek-zamanlı PCR için şablon olarak kullanılır.
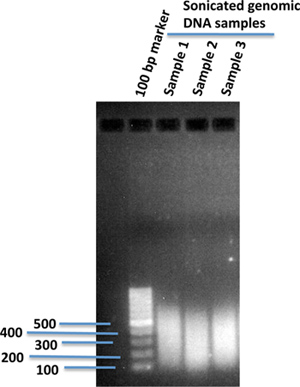
Şekil 1. Içinde sonike insan genomik DNA için BamHI% 1 agaroz jel. 1X TE tamponu, 120 ul, insan iPS hücreleri, izole edilen genomik DNA için 10 ug bir sonikasyon cihazı (Covaris) kullanılarak sonike edildi. Sonikasyon sonrasında sonicated DNA'nın 2 ul sonicated DNA parçalarının boyutları karşılaştırmak için DNA işaretleyici 100 bp ile% 1 agaroz jel üzerine yüklendi.
| Bileşen | Hacim | Nihai Konsantrasyon |
| Su | _ Ul | |
| 10 X β-GT Reaksiyon Tamponu | 2 ul | 1 X |
| Dakikada 10 mikrogram genomik DNA | _ Ul | Up 500 ul ng / |
| UDP-6-N GLC 3-(3 mM) | 0.67 ul | 100 uM |
| β-GT (40 uM) | 1 ul | 2 mcM |
| Toplam hacmi | 20 ul |
doku genomik DNA (yüksek 5-HMC içeriği>% 0,1) için: i)
| Bileşen | Hacim | Nihai Konsantrasyon |
| Su | _ Ul | |
| 10 X β-GT Reaksiyon Tamponu | 10 ul | 1 X |
| En fazla 20 mikrogram genomik DNA | _ Ul | Up 500 ul ng / |
| UDP-6-N3-GLC (3 mM) | 1.33 ul | 100 uM |
| β-GT (40 uM) | 2 ul | 2 mcM |
| Toplam hacmi | 40 ul |
ii) kök hücre genomik DNA (ortanca 5-HMC içeriği ~% 0.05) için
| Bileşen | Hacim | Nihai Konsantrasyon |
| Su | _ Ul | |
| 10 X β-GT Reaksiyon Tamponu | 10 ul | 1 X |
| Up 50 mikrogram genomik DNA | _ Ul | Up 500 ul ng / |
| UDP-6-N3-GLC (3 mM) | 3.33 ul | 100 uM |
| β-GT (40 uM) | 5 ul | 2 mcM |
| Toplam hacmi | 100 ul |
iii) kanser hücresinin genomik DNA (düşük 5-HMC içeriği ~ 0.01% için)
Tablo 1. Giriş DNA ve seçici kimyasal etiketleme yöntemi ile çeşitli 5-HMC seviyeleri ile numuneleri kullanılarak etiketleme reaksiyon miktarı örnekleri.
| Örnek | 5-HMC düzeyi | Başlangıç DNA (mikrogram) | Işaretlendikten sonra Kurtarma (boncuk giriş) (mikrogram) | Kurtarma verim | Pull-down DNA (ng) | Pull-down verim |
| Yetişkin fare beyincik | 0.4% | 10 | 7.5 | % 75 | 236 | 3.1% |
| Postnatal gün 7 fare beyincik | % 0.1 ' | 11 | 9 | % 82 | 140 | 1.6% |
| Fare ES hücre E14 | 0.05% | 60 | 42 | 70% | 350 | 0.8% |
Tablo 2. Fare beyin dokuları ve ES hücreleri Temsilcisi sonuçları.
Tartışmalar
Hydroxymethylcytosine 5-(5-HMC) belirli memeli hücre türleri içinde önemli miktarda yeni tanımlanmış bir epigenetik modifikasyon mevcuttur. Burada sunulan yöntem, 5-HMC arasında genom dağılımı belirlemek içindir. Biz 5-HMC bir hidroksil grubu üzerine bir azid grubu ihtiva eden bir mühendislik glikoz yarımı aktarmak için β-glukoziltransferaz T4 bakteriyofaj kullanır. Azid grubu kimyasal memeli genomları 5-HMC içeren DNA parçalarının tespiti, afinite zenginleştirme ve sıralama için biotin ile ...
Açıklamalar
Çıkar çatışması ilan etti.
Teşekkürler
Bu çalışma, Ulusal Sağlık Enstitüleri (CH ve NS051630/MH076090/MH078972 için PJ için GM071440) tarafından kısmen desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Isim | Şirket | # Katalog | Yorumlamak |
| Reaktifler | |||
| 5M Sodyum klorür (NaCl) | Promega | V4221 | |
| 0.5 M pH 8.0 Ethylenediaminetetraacetic asit (EDTA) | Promega | V4231 | |
| 1M Trizma tabanı (Tris) pH7.5 | Invitrogen | 15567-027) | |
| HEPES 1M pH7.4 | Invitrogen | 15630 | |
| Magnezyum klorür (MgCl2) 1M | Ambion | AM9530g | |
| Dimetil sülfoksit (DMSO) | Sigma | D8418 | |
| Tween 20 | Fisher BioReagents | BP337-100 | |
| DBCO-SS-PEG3-Biotin konjugat | Kimya Araçlar'ı tıklayın | A112P3 | |
| 1,4-ditiyotretol, son derece saf (DTT) Superpure | Invitrogen | 15508-013 | |
| QIAquick Nükleotid Kazıma Seti | Qiagen | 28304 | |
| Mikro Bio-Spin 6 Sütun | Bio-Rad | 732-6222 | |
| Dynabeads MyOne | Invitrogen | 650-01 | |
| Streptavidin C1 | |||
| Qiagen MinElute PCR Arıtma Kiti | Qiagen | 28004 | |
| Ultrapure Agaroz | Invitrogen | 16500500 | |
| UDP-6-N 3-glikoz | Aktif Motif | 55013 | |
| Enzim | |||
| β-glukoziltransferaz (β-GT) | New England Biolab | M0357 | |
| Ekipman | |||
| Sonikasyon cihazı | Covaris | ||
| Masaüstü santrifüj | |||
| Su banyosu | Fisher Scientific | ||
| Jel çalışan cihazlar | Bio-Rad | ||
| NanoDrop1000 | Thermo Scientific | ||
| Labquake Tüp Çalkalayıcı | Barnstead | ||
| Labquake Tüp Çalkalayıcı | Thermolyne | ||
| Manyetik Ayırma Standı | Promega | Z5342 | |
| Qubit 2.0 Fluorometre | Invitrogen | ||
| Reaktif kurulum 10 X β-GT Reaksiyon Tamponu (500 mM HEPES pH 7.9, 250 mM MgCl2) 2 X bağlayarak ve (10 mM Tris, pH 7.5, 1 mM EDTA, 2 M NaCl,% 0.02 Tween 20) (B) tampon yıkama . |
Referanslar
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. , 245-254 (2003).
- Ito, S. Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specification. Nature. 466, 1129-1133 (2010).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Kriaucionis, S., Heintz, N. The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain. Science. 324, 929-930 (2009).
- Ko, M. Impaired hydroxylation of 5-methylcytosine in myeloid cancers with mutant TET2. Nature. 468, 839-843 (2010).
- Koh, K. P. Tet1 and tet2 regulate 5-hydroxymethylcytosine production and cell lineage specification in mouse embryonic stem cells. Cell Stem Cell. 8, 200-213 (2011).
- Iqbal, K., Jin, S. G., Pfeifer, G. P., Szabo, P. E. Reprogramming of the paternal genome upon fertilization involves genome-wide oxidation of 5-methylcytosine. Proceedings of the National Academy of Sciences of the United States of America. 108, 3642-3647 (2011).
- Wossidlo, M. 5-Hydroxymethylcytosine in the mammalian zygote is linked with epigenetic reprogramming. Nat. Commun. 2, 241 (2011).
- Gu, T. P. The role of Tet3 DNA dioxygenase in epigenetic reprogramming by oocytes. Nature. 477, 606-610 (2011).
- Dawlaty, M. M. Tet1 is dispensable for maintaining pluripotency and its loss is compatible with embryonic and postnatal development. Cell Stem Cell. 9, 166-175 (2011).
- Huang, Y. The behaviour of 5-hydroxymethylcytosine in bisulfite sequencing. PLoS One. 5, e8888 (2010).
- Song, C. X. Selective chemical labeling reveals the genome-wide distribution of 5-hydroxymethylcytosine. Nat. Biotechnol. 29, 68-72 (2011).
- Pastor, W. A. Genome-wide mapping of 5-hydroxymethylcytosine in embryonic stem cells. Nature. 473, 394-397 (2011).
- Matarese, F., Pau, C. a. r. r. i. l. l. o. -. d. e. S. a. n. t. a., E, ., Stunnenberg, H. G. 5-Hydroxymethylcytosine: a new kid on the epigenetic block. Mol. Syst. Biol. 7, 562 (2011).
- Szwagierczak, A., Bultmann, S., Schmidt, C. S., Spada, F., Leonhardt, H. Sensitive enzymatic quantification of 5-hydroxymethylcytosine in genomic DNA. Nucleic Acids Res. 38, 181 (2010).
- Terragni, J., Bitinaite, J., Zheng, Y., Pradhan, S. Biochemical characterization of recombinant β-glucosyltransferase and analysis of global 5-hydroxymethylcytosine in unique genomes. Biochemistry. , (2012).
- Rusmintratip, V., Sowers, L. C. An unexpectedly high excision capacity for mispaired 5-hydroxymethyluracil in human cell extracts. Proc. Natl. Acad. Sci. U.S.A. 97, 14183-14187 (2000).
- Globisch, D. Tissue distribution of 5-hydroxymethylcytosine and search for active demethylation intermediates. PLoS One. 5, e15367 (2010).
- Yildirim, O. Mbd3/NURD Complex Regulates Expression of 5-Hydroxymethylcytosine Marked Genes in Embryonic Stem Cells. Cell. 147, 1498-1510 (2011).
- Szulwach, K. E. Integrating 5-hydroxymethylcytosine into the epigenomic landscape of human embryonic stem cells. PLoS Genet. 7, e1002154 (2011).
- Szulwach, K. E. 5-hmC-mediated epigenetic dynamics during postnatal neurodevelopment and aging. Nat. Neurosci. 14, 1607-1616 (2011).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır