Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Yüksek verim SHAPE kullanarak RNA İkincil Yapısı Tahmin
Bu Makalede
Özet
Astar uzantısı (SHAPE) tarafından analiz yüksek verimli seçici 2 'hidroksil asilleme tek nükleotid çözünürlükte binlerce nükleotid birkaç yüz ile RNA'lar yapılarını belirlemek için bir teknoloji sondalama yeni kimyasal, ters transkripsiyon, kapiler elektroforez ve ikincil yapı tahmini yazılımı kullanır.
Özet
Biyolojik süreçlerinde RNA işlevini anlama RNA yapısı ayrıntılı bir bilgi gerektirir. Bu sonuna doğru, dublaj metodoloji, veya SHAPE "astar uzantısı ile analiz yüksek verimli seçici 2 'hidroksil asilleme", tek nükleotid çözünürlükte RNA ikincil yapı tahmini sağlar. Bu yaklaşım, tercihen sulu çözelti içinde RNA tek sarmallı ya da esnek bölgeler asil ile kimyasal tarama maddeler kullanır. Bir kimyasal modifikasyon Yer modifiye RNA ters transkripsiyon ile tespit edilir ve bu reaksiyonun ürünleri, otomatik kapiler elektroforez (CE) ile fraksiyone edilmiştir. Ters transkriptaz ŞEKİL reaktifleri ile değiştirilmiş olanlar RNA nükleotid duraklar yana Sonuçta elde edilen cDNA kütüphanesi dolaylı olarak tek kapandı RNA bağlamında mahsur bu ribonükleotidler eşler. ShapeFinder yazılımı kullanarak, otomatik CE tarafından üretilen electropherograms işlenmiş ve nu dönüştürülürcleotide tepkime tablolar kendilerini RNAStructure (v5.3) tahmini algoritması kullanılan sözde enerji kısıtlamaları dönüştürülür. Siliko RNA ikincil yapı tahmininde ile sondalama SHAPE birleştirerek elde edilen iki boyutlu RNA yapıları tek başına her iki yöntemi kullanarak elde edilen yapılara göre çok daha doğru olduğu tespit edilmiştir.
Giriş
Yapıştırma, çeviri, virüs çoğaltma ve kanser düzenlenmesinde yer alan katalitik olmayan kodlama RNA'lar işlevlerini anlamak için, RNA yapısının detaylı bilgi 1,2 gereklidir. Ne yazık ki, RNA katlama doğru tahmin müthiş bir meydan okuma sunuyor. Klasik tarama maddeler muzdarip toksisite, eksik nükleotid kapsamı ve / veya deney başına 100-150 nükleotidleri ile sınırlı verim gibi birçok dezavantajları. Çıplak ikincil yapı tahmini algoritmaları benzer, dezavantajlı etkili enerjik benzer yapıları arasında ayırt etmek için kendi yetersizlik kaynaklanan yanlışlıklar nedeniyle vardır. Özellikle büyük RNA'lar, aynı zamanda X-ışını kristalografisi ve şekilsel esnekliği ve bu teknikler için gerekli son derece saf bir numune büyük miktarlarda nedeniyle spektroskopisi (NMR) nükleer manyetik rezonans olarak 3D yapı belirleme yöntemleri genellikle alınamamaktadır.
High verimli SHAPE tek nükleotid çözünürlükte büyük RNA'lar yapıları sondalama için etkili, basit bir yaklaşım sağlayarak bu sorunların çoğu çözer. Ayrıca, şekil için kullanılan reaktiflere işlemek için güvenli, kolay ve reaktifler tarama çoğu diğer kimyasal aksine, dört ribonükleotidler ile reaksiyona girerler. Bu reaktifler de mümkün bağlamda in vivo (lar) 3 kendi RNA'lar araştırmak için yapım, hücre membranları içine nüfuz edebilir. Başlangıçta hafta laboratuar 4 geliştirilen ŞEKLİ RNA'lar, geniş bir yelpazede ~ 9 kb HIV-1 RNA genomunun 5 tam ikincil yapısının belirlenmesi; en önemli örneği analiz etmek için kullanılmıştır. Şeklini kullanarak Diğer başarıları bulaşıcı viroidler 6, insan uzun kodlayıcı olmayan RNA'lar 7, maya ribozomlar 8 ve 9 riboswitches hem de tespit etmek virionu ile ilişkili HIV-1 RNA 3 protein bağlanma yerleri yapılarının aydınlatılması içerir. WhSHAPE protokol Ile orijinal ve yüksek verimli varyasyonlar başka bir yerde 10-12 yayınlanmıştır, Bu çalışmada floresan oligonükleotidler, Beckman Coulter CEQ 8000 Genetic Analyzer kullanarak yüksek verimli SHAPE RNA ikincil yapı tayini ayrıntılı bir açıklamasını sağlar ve SHAPEfinder ve RNAStructure (v5.3) yazılımı. Daha önce yayınlanmamış teknik ayrıntı ve sorun giderme tavsiye de dahildir.
SHAPE varyasyonları
ŞEKİL ve varyasyonları özü seçici modifikasyonu sitelerinin hantal katılma ürünleri üreten, 2'-hidroksi (2'-OH) riboz grupları açilleme elektrofilik anhidritler, sulu çözelti içinde RNA maruz kalmasıdır. Baz eşleştirilmiş veya mimari Yapım ise bu kimyasal reaksiyon, tek iplikli nükleotidler bu reaktif ile elektrofilik saldırı için elverişli konformasyonlar benimsemeye daha yatkındır gibi, yerel RNA yapısal dinamikleri sorgulayan bir araç olarak hizmet vermektedirained nükleotid daha az ya da 10 reaktif değillerdir. Adukt formasyonu Yer modifiye RNA ("(+)" primer uzatma reaksiyonu) ile ilgili belirli bir site hibridize floresan veya radyoaktif primerlerle başlatarak, ters transkripsiyon ile tespit edilir. Ters transkriptaz (RT) asile ribonucleotides hareket başarısız olduğunda, cDNA ürünleri bir havuz olan uzunlukları değişiklik siteleri denk üretilir. Bir kontrol, "(-)" astar uzantısı reaktif maruz olmamıştır RNA kullanarak reaksiyon da çok yapılır bu yapı, non-spesifik RNA zincir kırıkları, vb olabilir RNA nedeniyle DNA sentezi (yani "durur") erken sona ermesi. kimyasal modifikasyonu ile üretilen duraklatma ayırt edilebilir. Son olarak, aynı primerlerle başlatılması iki-dideoksi sekanslama reaksiyonları RNA primer dizisi elektroforezi ile aşağıdaki reaktif nükleotid ilişkilendirmek için marker olarak kullanılmaktadır.
SHAP orijinal uygulamasında(-) Ve iki dizi reaksiyonlar E aynı 32 P-uç-etiketli astar, (+) için kullanılmaktadır. Bu reaksiyonların ürünleri bir% 5-8 poliakrilamid jel levha bitişik kuyu içine yüklenir ve poliakrilamid jel elektroforezi (PAGE, Şekil 1), denatüre edici ile fraksiyone edilmiştir. Geleneksel şekli ile üretilen jel görüntüleri kantitatif analiz SAFA, yarı-otomatik bir ayak izi analiz yazılımı 13 kullanılarak da gerçekleştirilebilir.
Buna karşılık, yüksek verimli SHAPE floresan etiketli astar ve otomatik kapiller elektroforez kullanır. Spesifik olarak, inceleme altındaki RNA ortak bir dizi ancak farklı 5 'floresan etiketler olarak sentezlenebilir ya da satın alınması gerekir olan dört DNA primerleri kümesi her bir bölgesi için. Bu farklı-etiketli oligonükleotidler asal iki SHAPE reaksiyonlar ve iki sıralama reaksiyonları, toplanmış ve fraksiyone / otomatik kapiller elektroforez (CE) tarafından tespiti yapılan ürünler için hizmet vermektedir. NereyeEAS RNA 100-150 nt reaktivite profili temel yaklaşımı kullanarak, dört reaksiyonlar bir dizi elde edilebilir, yüksek verimli bir şekli, bir tek numune toplanmış 3 300-600 nt çözünürlüğü sağlar. Gibi pek çok 96 gibi, örnekler 12 ardışık CE çalışır (Şekil 2) boyunca fraksiyonasyon için hazırlanmış olabilir iken, aynı zamanda, fraksiyonlara ayrılmış olabilir üste reaksiyonlar 8 setleri için. Ayrıca, CEQ ve diğer genetik analiz çıkan verilerin işlenmesi ve analiz etmek için geliştirilen SHAPEfinder yazılım, daha otomatik ve SAFA 13 veya diğer jel-analiz paketleri çok daha az kullanıcı müdahalesi gerektirir.
Daha gelişmiş yüksek verimli yöntemleri son zamanlarda elde etmek için teknikler sıralama yeni nesil ile birlikte yapı-belirli enzimler yerine alkilleme reaktifleri kullanmak PARS (RNA yapısı paralel analiz) 14 ve Frag-Seq (fragman-dizi) 15, olarak ortaya çıkmıştır BİLİŞİMRNA yapısı hakkında n. Bu tekniklerin çekiciliğini rağmen, sondalama nükleaz doğasında birçok sınırlamalar hala 16 kalır. Bu sorunlar, yeni nesil sıralama geleneksel SHAPE'e gerçekleştirilen edilene benzer bir şekilde kimyasal modifikasyonu ve RNA'ların ters transkripsiyon öncesinde ŞEKLİ dizi analizleri (Şekil-Seq) 17 protokol bölgesi atlatılabilir. Bu yöntemler RNA yapı tayini geleceği temsil olsa da, bu yeni nesil sıralama çok pahalı ve birçok laboratuvarlara kullanılamaz durumda olduğunu hatırlamak önemlidir.
SHAPE Veri Analizi
Genetik analiz üretilen veri taşıma zaman indeksi karşı çizilen kılcal detektörü akan numunenin floresan yoğunluğu (ler) in bir electropherogram şeklinde sunulmuştur. Bu grafik, dört flüoresan kanala tekabül eden üst üste izleri formunda alırin farklı bir florofor tespit etmek için kullanılır ve tek tek her bir iz cDNA'sı ya da sekanslama ürünlerine karşılık gelen tepe oluşur burada. Elektroferogram veri bir sekme ile sınırlandırılmış metin dosyası olarak genetik analiz ihraç ve ShapeFinder dönüşüm ve analiz yazılımı 18 alınır.
ShapeFinder ilk geçiş süreleri ve yüksek miktarlar doğru sırasıyla reaksiyon ürünleri, kimlikleri ve miktarları yansıtmasını sağlamak için veri matematiksel dönüşümleri bir dizi gerçekleştirmek için kullanılır. Peaks sonra uyumlu ve entegre ve sonuçlar birincil RNA dizisi ile birlikte tablo vardır. RNA ilgili bölümü için bir "reaktivite profili" (+) nükleotid Her RNA ile ilişkili değerleri kontrol değerleri çıkarılarak, ve aşağıda tarif edildiği gibi verileri normalize edilmesiyle elde edilir. Bu profil normalize tepkime val dönüştürür (v5.3) yazılım 19,20, RNAstructure alınırRNA ikincil yapı katlama algoritması dahil edilmiştir sözde enerji kısıtlamaları içine ues. Bu şekilde algoritma sondalama ve katlama kimyasal birleştiren önemli ölçüde tek başına 12,21 ya yöntemine göre yapı tahmini doğruluğunu artırır. RNAstructure çıktısı (v5.3) en düşük enerji görüntüleri içeren RNA ikincil yapılar metin nokta-dirsek gösterimde SHAPE reaktivite profil (ler), hem de aynı yapılarla renk kodlu. İkincisi daha sonra bu Varna 22 ve PseudoViewer 23 olarak RNA'nın ikincil yapının grafik ekran için özel yazılım ihraç edilebilir.

Şekil 1. SHAPE 4,10 ile RNA yapısı belirlenmesi Akış Şeması. (A) RNA mAy biyolojik numunelerin veya in vitro transkripsiyonu sonucunda elde edilebilir. (B) kaynağına bağlı olarak, RNA, katlanmış ya da başka şekilde işlem ve ŞEKİL reaktifi ile modifiye edilir. (C) ters transkripsiyon floresan veya radyoaktif olarak etiketlenmiş primerler kullanılarak. (D) cDNA ürünlerdir ya kılcal veya levha jel tabanlı elektroforezi ile fraksiyonlara ayrılmış. (E) Parça analizi. (F) RNA yapısı tahmini. büyük rakam görmek için buraya tıklayın.
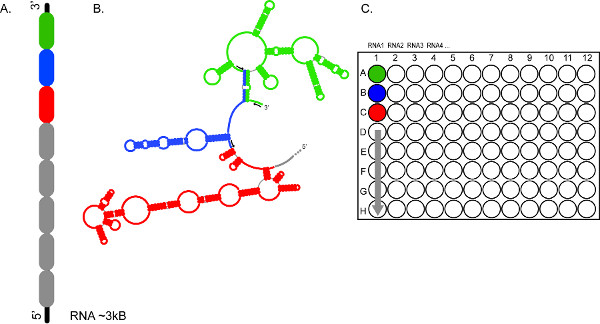
Şekil 2. CE tabanlı SHAPE yüksek verimli karakter hızlı çoklu RNA'lar analizi ve / veya aynı RNA oluşan parçalara ayır sağlar. (A) bir RNA 300-600 nt bölümler (renk yeşil, mavi ve kırmızı kodlu) (B) RNA bölümleri floresan astar farklı ayarlar (siyah ok) ile bağımsız tanınacak olan (C) takımları ayrılabilir nasıl temsil reaksiyonları ~ 3 kb RNA1 için tam koruma sağlar, sırasıyla kuyu A1, B1, C1, vb, içine toplanmış ve yüklenir. RNA'lar 2, 3, 4, vb reaksiyon ürünleri benzer ardışık elektroforetik çalıştırır fraksiyon için hazırlanabilir. büyük rakam görmek için buraya tıklayın.
Protokol
RNA 3 'ucu Primer tasarımı ve uzatma
Yüksek verimli şekli ile uzun RNA'lar analiz etmek için, primer hibritleşme sitelerinin bir dizi de (i) ve (ii) (iii) / RNA 20-30 nt uzunluğunda, ve, ~ 300 nt ile ayrılmış olduğu şekilde seçilmelidir Bu siteler DNA tavlanmasıyla üretilmiştir DNA hibridi> 50 ° C arasında bir erime sıcaklığına sahiptir beklenen Böyle bir tespit çoğu zaman kullanılamaz RNA yapının bazı önceden bilme gerektirir bulunmasına rağmen Buna ek olarak, son derece yapılandırılmış olması için tahmin edilmektedir RNA segmentleri kaçınılmalıdır. Bu siteler hibridize DNA primerleri sonra stabil dimer ya da intrastrand ikincil yapılar oluşturmak için beklenmeyebilecektir emin olmak için özen tasarlanmış olmalıdır.
Bir kez tasarlanmış, primer setleri ya satın (Integrated DNA Technologies, Ames, Iowa gibi) veya 24,25 sentez edilmelidir. Astarlar, Cy5, Cy5.5 ile 5'-etiketliWellRedD2 (Beckman Coulter) ve IRDye800 (Lycor) / WellRedD1 (Beckman Coulter) ile en iyi karışmasını en aza indirirken, iyi bir sinyal yoğunluğu veren, Beckman Coulter CEQ 8000 için uygundur. Etiketli oligonükleotidler -20 ° C'de küçük, 10 mcM alikotları süresiz saklanabilir, tekrar donma / çözülme döngüsü kaçının.
Bu şekilde tasarlanan primerler kullanılarak, herhangi bir uzunlukta hemen hemen bütün bir RNA şekil verileri elde etmek mümkündür. (Örneğin bir "yapı kaset") hangi bir astar 4 melezleşmiştir olabilir terminali uzatma Ancak, 3 ya da yakın dizisi 'RNA 3 içeren tasarlanmıştır sürece bir RNA ucuna, SHAPE her zaman ulaşılmaz'.
Kapiler Elektroforez ile Hazırlık RNA
Biyolojik örneklerden RNA'lar, yüksek verimli bir şekil için kullanılır, bununla birlikte, burada verilen protokol in vitro transkripsiyon tarafından üretilen RNA için optimize edilmiştir. Ticari traBu tür POLYCLEAR RNA saflaştırma sütunlar (Ambion) ile bağlantılı olarak kullanılan MegaShortScript (Ambion) ve oyma yazıyı kitler alanda iyi saf RNA büyük miktarlarda üretmek için uygundur. RNA -20 ° C ile -80 ° C arasında bir TE tampon maddesinde muhafaza edilmelidir En iyi sonuç için, RNA'lar hem denatüre olmayan denatüre poliakrilamid jel elektroforezi ile homojen görünmelidir.
1. RNA Katlanır
- 0.5 ml mikrosantrifüj tüp, su ile 18 ul RNA 12 pmol sulandırmak ve 10X renatürasyon tampon 2 ul ekleyin. İyice karıştırın.
- Isı 85 ° C 4 ila sonra soğuk 1 dakika boyunca 0.1 ° C arasında bir oranda ° C / saniye.
- Su ve 100 ul 5X katlama tamponu 30 ul ekle.
- Katlandıktan RNA bağlı olarak 30-60 dakika boyunca 37 ° C'de inkübe edin. Genel olarak, Mg 2 daha uzun ve daha yapısal RNA'lar + bağımlı katlama uzun inkübasyon sürelerinde gerektirir.
- Iki adet 0.5 ml mikrosantrifüj tüpleri her birine bir 72 ul kısım aktarın: Değişikled (+) ve Kontrol (-).
2. RNA kimyasal modifikasyonu
Evet özelliği, elektrofilik reaktifler ŞEKLİ izatoik anhidrit (IA) içinde, N-methylisatoic anhidrit (NMIA), 1-metil-7-nitro-izatoik anhidrid (1M7) 26 ve benzoil siyanür (BzCN) 27 içerir. Bunlardan, en yaygın olarak yüksek verimli bir şekil için kullanılan 1M7 ve NMIA ve sadece ikinci (Life Technologies), ticari olarak mevcuttur. Reaktif modifiye nihai konsantrasyonu, "tek-hit" değişiklik kinetiğinin çözümü en RNA'lar 11 analiz edilen RNA bölgede bir kez değiştirilmiş edildiği durumda, yani her bir RNA için optimize edilmelidir. Bu en iyi konsantrasyon reaktif maddenin konsantrasyonu, aşağıda Bölüm 2.1 'de verilen tabloda belirtilen aralık (lar) boyunca farklılaştığı birden tepkimeler gerçekleştirilerek belirlenebilir. Kolayca saptanabilir sinyal üretir reaktif maddenin konsantrasyonu kullanım sırasında dakikauzun ve kısa DNA sentezi ürünleri (örneğin Şekil 3) arasında sinyal yoğunluğu arasındaki fark imizing.
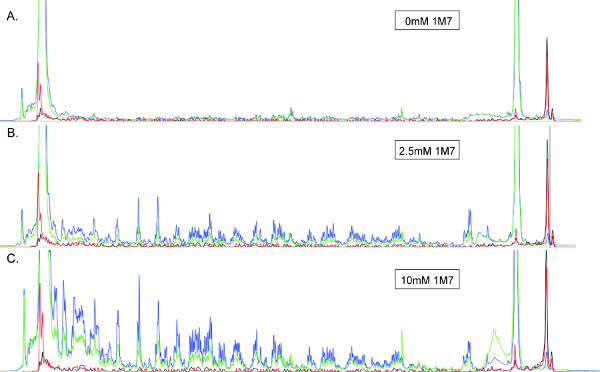
Şekil 3,. (A) 0 (B) 2.5 mm ve (C), 10 mM 1M7 ile tedavi edilen bir ~ 360 nt RNA 'dan üretilen ŞEKİL electropherograms. Tüm electropherograms aynı ölçekte gösterilir. Mavi, yeşil, kırmızı ve siyah izleri (+) reaksiyon ürünleri (Cy5), karşılık (-) reaksiyon ürünleri (Cy5.5), ve iki sıralama merdivenler (WellRed D2 ve IRDye800), sırasıyla. Görüntü (B) üretmek için kullanılan RNA iz (sol) boyunca en az sinyal bozulması olmadan, iyi pik çözünürlük ve şiddeti gösteren, 1M7 en uygun miktarı ile tedavi edilmiştir. Uzunluğu, bu koşullar altında maksimumdur okuyun. Buna karşılık, orta int yokluğundadensite, (A), içinde iyi çözüldü tepe 1M7 bir alt-optimal konsantrasyonunu göstermektedir. Bunun tersine, (C) ile belirgin sinyal çürüme, tek isabet kinetik gösterir görülmez ve RNA aşırı değiştirilir. Bu gibi durumlarda, oda sıcaklığında RNA şablonunun 5 'ucunu karşılaşmaya beklenmeyebilecektir, özellikle de uzunluğu optimal okuma olacaktır.
- SHAPE reaktif (NMIA veya 1M7) 10X stok hazırlayın. Bu en iyi sonra istenen konsantrasyonu elde etmek için DMSO ilavesiyle, 1.5 ml mikrofüj tüpe reaktif küçük bir miktarının ilave edilmesi ile elde edilir Dikkat:. RNA ile karıştırılır kadar ŞEKİL reaktif çözeltiler susuz kalmalıdır. Mağaza oda sıcaklığında desikatörde DMSO ve stok çözümler hazırlamak hemen önce ortam su buharı maruziyeti en aza indirmek amacıyla kullanılacak.
Reaktif Optimum 10X konsantrasyonu (DMSO içinde) ZamanReaktif 27 tam bozulması NMIA 10-100 mM ~ 20 dakika 1M7 10-50 mM 70 sn
Tablo 1. Elektrofilik reaktifler RNA modifikasyonu için kullanılabilir. - . (-) Modifiye edilmiştir (+) ve kontrol etmek için 8 ul 10X NMIA/1M7 veya susuz DMSO ekleme sırasıyla karışımları Not: 2.5 mM bağımsız olarak RNA analiz edilen, NMIA 1M7 ve hem de etkili bir başlangıç konsantrasyonunda olduğu kanıtlanmıştır.
- 50 dakika (NMIA) ya da uygun olarak 5 dakika (1M7) boyunca 37 ° C'de inkübe edin.
- 3 M NaOAc (pH 5.2), 8 ul 100 mM EDTA, soğuk etanol ve 1 ul 10 mg / ml glikojen 240 ul (3 hacim) içinde 8 ml (0.1 hacim) ilave edilerek çökelti RNA. 4 ° C'de 30 dakika boyunca 14,000 x g'de santrifüj daha sonra 2 saat boyunca buzdolabında ve Soğuk% 70 etanol ile iki kez pelet yıkayın Dikkat:. Bu önemlidirBu olumsuz elektroforez sırasında pik çözünürlüğü etkileyebilir olarak, tuz eşçökeltmesi en aza indirmek amacıyla soğutma süresi, santrifüj zaman ve hız en aza indirir.
- Bir mikropipet ile süpernatant kaldırmak ve hava kuru pelet oda sıcaklığında 5 dakika için.
- 10 ul TE tamponu içinde çökelen RNA çözülür ve oda sıcaklığında 5 dakika inkübe edin. Bu yeterli iki, ters transkripsiyon reaksiyonları için RNA çözülür. -20 ° C Dikkat de kullanılmayan kısmı Mağaza: pelet mekanik tabanda genellikle gerekli değildir, ve RNA zarar verebilir.
3. Transkripsiyon Ters
Bu adım, dolaylı olarak RNA nükleotid bir SHAPE reaktif tarafından modifiye edilmiş derecesini tanımlamak için kullanılan floresan etiketli cDNA ürünleri üretir. SHAPE için, Üstsimge III (Invitrogen) RT performansını diğer RTS test daha üstün olduğunu ve bu kullanım için seçilen enzimdirprotokolü. Sırasıyla reaksiyonları (-), Cy5 ve Cy5.5 ile etiketlenmiş oligonükleotidler asal (+) ve kullanılmaktadır. Kısa RNA'lar için, primerler terminalinde 4 ', 3 ile ilgili bilgiler elde etmek için doğal RNA ucu uzantısı (örneğin bir "yapı kaset")', bir 3 hibridize edilir Dikkat:. Bu noktadan itibaren CE üzerinden, örnekler korunmalıdır ışık.
- Hazırlama (+) ve (-) 0,5 ml mikrofüj tüplerine ters transkripsiyon için örnekler. (+) Çözeltisine oda sıcaklığında tepkime için, modifiye edilmiş RNA (+) 5 ul, 6 ul su ve 1 ul Cy5 etiketli primer (10 uM) karışımı; için (-) (-), bir RT reaksiyonu, 5 ul kontrol RNA karıştırın . 6 ul su ve 1 ul Cy5-etiketli astar (10 mcM) Dikkat: Sarstedt PCR tüpleri (REF 72.735.002) bu uygulama için tavsiye edilir.
- Yeri bir termal döngü tüpler ve aşağıdaki programı uygulayarak RNA ve ters transkripsiyon hazırlanmak için astar tavlama: 85 ° C, 1 dakika, 60 ° C, 5 dakika;35 ° C, 5 dk, 50 ° C, basılı tutun.
- Tavlama adımı sırasında, yapılması gereken reaksiyon sayısı, ayrıca en az% 50 (-) reaksiyonları, ölçek 4,5-iki kat (+) ve iki (örneğin) için yeterli 2.5X RT karışımı hazırlamak. 4 ul 5 z RT tamponu ul, 1 ul 100 mM DTT, 1.5 ul su, 1 ul 10 mM dNTP, 0.5 ul Üst Simge III RT aşağıdaki gibidir: Bir reaksiyon, 8 ul gerektirir. Buz üzerinde tutmak Dikkat:. 5X RT tamponu ve 100 mM DTT Üst Simge III RT ile sağlanır.
- (-) Tavlama karışımları sıcaklık, 50 ° C'ye ulaştığında, (+) ve 2.5X RT karışımı 8 ul reaksiyon şekli:. Sıcak karışım oda sıcaklığında ° C 37-5 dakika boyunca reaksiyon eklemeden önce .
- . 50 dakika daha uzun süre RT reaksiyon Kuluçka anormal cDNA ürünleri neden olabilir: 50 50 dk ° C, 4 ° C ve / veya yer buz üzerinde Not sonra serin inkübe edin.
- 95-1 ul 4 M NaOH ekleme ve ısıtma ile RNA Hidroliz ° C 3 dak. Buz ve ardından serin reaksiyonlar 2 M HCl 2 ul ekleyerek onları nötralize Dikkat:. CDNA ürün kötü kalite ayrılması bu adımı sonuçları ihmali.
- Kombine (+) ve (-) reaksiyonları ve 3 M NaOAc, 0.1 hacim, 100 mM EDTA, 0.1 hacim, soğuk etanol ve 1.5 hacim 10 mg / ml glikojen 1 ul ekleyerek cDNA hızlandırabilir. 2 saat boyunca buzdolabında, daha sonra 4 ° C'de 30 dakika boyunca 14,000 x g'de santrifüj Soğuk% 70 etanol ile iki kez pelet yıkayın Dikkat:. Santrifüj yüksek oranlarda veya pelet (ler) resuspending zorluklar daha uzun bir süre sonuç için.
- Daha 30 dakika boyunca kuvvetli bir vorteks ardından 10 dakika boyunca 65 ° C'ye kadar ısıtılarak deiyonize formamid 40 ul cDNA, pelet yeniden süspanse Dikkat:. Granüller görünmez olabilir. Sinyal ya da zayıf sinyal aşağıdaki elektroforez olmaması yeterince bu aşamada pelet çözmek için başarısızlık sonucu olabilir.
Sıralama merdivenler veri işleme sırasında nükleotid pozisyon bulmak için işaretleri olarak hizmet vermektedir. Bu WellRed D2 veya D1/Lycor 800 ile işaretlenmiş bir USB Döngüsü Sıralama RNA çalışılan aynı sekansı olan kiti (# 78500), DNA ve primerler kullanılarak oluşturulur. Tipik olarak, bu reaksiyonda kullanılan DNA'nın söz konusu RNA için bir transkripsiyon şablon olarak kullanılan bu olacaktır. Reaksiyon protokolü burada sunulan rağmen yakından kiti üretici tarafından önerilen, reaksiyon birkaç kat büyütülüyor olduğunu benzer. DdA ve DDT, aşağıda tarif edilen reaksiyonlar, zincir sonlandırıcı olarak kullanılır ise, sonlandırıcı herhangi bir çift dizi merdivenler üretmek için kullanılabilir.
- DDA sonlandırma karışımı 40 ul, DNA örneğinin 5 pmol 10X tampon Sequenase, WellRed D2 işaretli primer, 82 ul toplam hacmi getirmek için Sequenase su ve 4.6 ul 10 ul 4.6 ul karıştırın. Sequenase son ekleyin. PrepaBunun yerine DDT ve Licor IR800 işaretli primer kullanılarak, aynı şekilde ikinci bir dizi reaksiyon, tekrar.
- USB önerilen koşullar kullanarak PCR devam Dikkat:. Madeni yağ ilavesi gerekli ne de protokolleri / ısıtmalı kapak kullanmak thermal cycler'ları için tavsiye edilmez.
- Bir 1.5 ml mikrofuge'de tüp (~ 164 ul toplam) içine reaksiyonlar sıralama ddA ve ddT birleştirin.
- Çökelme DNA aşağıdaki gibi: 16 ul 3 M NaOAc (pH 5.2), 16 ul 100 mM EDTA, 1 ul 10 mg / ml glikojen ve 480 ul% 95 etanol ekleme. İyice karıştırın, 4 ° C'de inkübe edin ve 30 dakika santrifüj 30 dakika için 14,000 x g'de 4 ° C'de bir
- En az 30 dakika boyunca kuvvetli bir vorteks ardından 10 dakika boyunca 65 ° C'ye kadar ısıtılarak deiyonize formamid 100 ul cDNA pelet yeniden süspanse edin.
5. Kapiler Elektroforez ile Reaksiyon Ürünleri Fraksiyonu
Kapiler elektroforez aynı anda sağlartek bir örnek olarak toplanmış dört reaksiyonlardan cDNA sentezi ürünlerin ayrılması. Gibi birçok gibi, örnekler 96, tek bir çalışma (Şekil 2) ve fraksiyonlara ayrılmış olabilir, sekiz numune, aynı zamanda, fraksiyonlara ayrılmış olabilir.
- 10 toplanmış sıralama merdiven ul, ve 96-örnek plakaları transfer ile toplanmış SHAPE örneklerin 40 ul karıştırın Dikkat:. Zorunludur olduğunu Beckman Coulter reaktifler ve plakaları (, LPA-I jel dahil olmak üzere tampon, mineral yağ, örnek çalışan yükleme çözümü ve örnek ve tampon plakaları) Beckman Coulter CEQ-8000 Genetik Analiz Cihazı ile kullanılabilir.
- Program ve kapiller elektroforez enstrüman hazırlamak ve üretici firma bilgileri göre çalıştırmak Not başlatmak:. Örneklerin iyi çözünürlük için, daha önce yayınlanmış CAFA yöntem parametreleri 28 kullanın.
İdeal olarak, astar ve güçlü-stop zirveleri, sinyallerin dışında dört elektroferogram t her zirve içinyarış lineer aralığında olmalıdır; sinyali bırakma kademeli bir kabul edilebilir. Ancak bazen, büyük tepe (durak) bile kontrol reaksiyon belirgindir ve bu daha sonraki veri işleme engelleyebilir. Bu zirveleri neden Kesik cDNAs ters transkripsiyon (örneğin RNA ikincil yapı) veya RNA bozulması sırasında doğal bir engel sonucu olabilir. Eski durumda, bu tür betain gibi katkı RT processivity geliştirmek ve / erken sonlandırma duraklatma RT azaltabilir.
Veri işleme
ShapeFinder yazılım kullanıcı görselleştirmek ve dönüşümü CE izleri ve SHAPE reaktivite profilleri 18 dönüştürmek sağlar. Tepkime değerleri tablo sonra, onlar normalize ve ikincil yapısal modeller oluşturmak ve geliştirmek için RNAStructure (v5.3) alınır.
6. ShapeFinder Yazılım
BaseFinder iz işleme plat bir uzantısıformu 29, ShapeFinder en yayınlanan sürümü ticari olmayan kullanım için 18 serbestçe kullanılabilir. ShapeFinder içinde veri işleme için ayrıntılı talimatlar yazılım belgelerine ile sağlanır.
- Electropherograms onlar (i) floresan arka plan için düzeltmek için ayarlanır ShapeFinder, içine CEQ ithal edilmektedir (ii) bir floresans yoğunluğu floresan kanallar arasında spektral örtüşme, (iii) hareketlilik farklı tagged astar tarafından kazandırdığı vardiya, (iv) farkları ortak ürünler farklı fluorophores etiketli, ve (v) sinyal çürüme ters transkripsiyon erken sonlandırma kaynaklanan.
- ShapeFinder olarak "aynı hizaya getirin ve entegre" aracı "Setup" fonksiyonu otomatik olarak bireysel zirveleri için kimlik atar ve kullanıcı girişi ve iki sıralama merdivenler tarafından tanımlanan RNA dizisi için bu ilişkilidir. Ilk atamaları genellikle kusurlu olmasına rağmen, hataların "Değiştir" işlevini kullanarak elle düzeltilebilirAynı aracı. Tepki zirveleri ve bir sekme ile sınırlandırılmış metin dosyasına karşılık gelen nükleotid numarası ile birlikte bu tepkime değerleri tabulates - () Son olarak, "Fit" fonksiyonu hizalanmış (+) ve altındaki alanları hesaplar.
Not: Verilerin analizi SHAPE doğruluğundan önemlidir ve bazı hususlar da dahil olmak üzere, bu analizi sırasında çok önemlidir:
- Sinyal-gürültü: sinyal-gürültü oranı bireysel zirveleri düşük tepkime ile pozisyonlar için bile kolayca tanımlanabilir olması gerektiğini bu olmalıdır. ShapeFinder bir veri düzeltme seçeneği sağlasa da, bu alternatif bu çarpık sonraki analiz olabildiğince, son derece dikkatli kullanılmalıdır.
- Analiz Bölge: Tipik olarak, güvenilir veri arka plan gürültü ayırt etmek zor seviyeleri için sinyal bozunur olarak astar 3 'ucu kaldırılır bir bölgede 40-80 nt başlayan ve biten, cDNAs 300-600 nt uzun elde edilebilir. Çoklu kullanımıple primer setleri RNA uzamaktadır analiz etmek için gerekli olacaktır. Bu durumda, bu primer seti arasında güvenilir bir sinyal örtüşme 30-50 nt aralığında olması tavsiye edilir. Kısa RNA üzerinde, ters transkriptaz sık RNA şablonu sonuna ulaştığında burada, bakım olan sinyal gürültü oranı, DNA sentezi güçlü durdurma etkilenir, bu tepe bertaraf edilmesi gerekir.
- Sinyal bozunma: Sinyal çürüme deney hem de RT kusurlu Processivity sırasında RNA modifikasyon derecesinin ile ilgilidir. İdeal olarak, analiz edilen RNA bölgeye göre tek-hit kinetiği boyu okumak maksimize etmek için elde edilmelidir. Shapefinder sinyal çürüme için düzeltmede etkili bir araç içerir, bu analize hata tanıtmak eğilimindedir çünkü ancak, - tekil hit kinetik gözlenmez, özellikle sinyal çürüme az olduğunda, en iyi (yani, kullanıldığında dağılımı zirveleri tek vuruş kine ile tutarlıdırtikler). Son zamanlarda, sinyal sinyal çürüme dönüştürmek için geliştirilmiş algoritmalar 30 yayınlanmıştır ve sinyal çürüme belirli bir deneyde özel endişe ise araştırılmalıdır.
- Ölçekleme Sinyal. (-) Izleri eşittir Muhtemelen, kontrol profilini minimal reaktif (+) ve arasında pik yoğunlukları, böylece Şekil Veri işleme bölgesindeki en rasgele adım ölçeklendirilmelidir. Çok büyük bir ölçüde kontrol iz ölçekleme (aşağıya Veri Normalizasyon bakınız) ilk çeyrekte negatif reaktivite değerleri bolluğu neden olacaktır. Bu durumda, ölçek faktörü, buna göre azalır ve veri kazandırılması gerekmektedir.
- Peaks atama. Genel olarak, en yüksek atama otomatik versiyon iyi çalışır. Işlemi başarısız olduğunda, ancak kullanıcı sinyal-gürültü oranı düşük, özellikle tüm zirveleri yazılım tarafından kabul edilmiştir sağlamak zorunludur. Omuz zirveler, örneğin, her zaman G-zengin SE tespit edilir ve edilmezüstesinden gelmek amacıyla çoğu kez sıkıştırılmıştır.
7. Veri Normalleştirme
RNAStructure (v5.3) yazılım tarafından kullanılan ikincil yapı algoritma içine nükleotid reaktivite profilleri dahil, ve / veya yakından ilgili RNA'lar profillerinin karşılaştırılması için, SHAPE veri standart bir şekilde 12 normalize edilmelidir. Bu içerir: (i) her reaktivite değerlerine bölünmesi ile "etkili olacak azami" reaktif (örn., reaktivite değeri en yüksek% 8 arasında ortalama aykırı hariç) belirlenmesi sonraki hesaplamalardan aykırı, (ii) ve (iii) normalleştirme hariç "etkili maksimum", aşağıdaki gibi:
- Uyum ve entegrasyon sonrası oluşturulan sekme ile sınırlandırılmış metin dosyasını açın ve bir Excel elektronik tabloya içeriği kopyalayın. Bu dosyanın en sağdaki sütunda (RX.area-BG.area) RNA her nükleotid için hesaplanan mutlak SHAPE tepkime değerleri içerir. En soldaki sütun RNA SEQU bu tepkime ilgilience.
- Ilk ve üçte çeyrek (yani, 25. ve 75. yüzdelik) değerleri (RX.area-BG.area) "= QUARTILE (dizi, litre)" Excel işlevini kullanarak hesaplamak ve kaydetmek
- Hesaplayın ve çeyrekler arası fark saklamak "= QUARTILE (dizi, 3)-QUARTILE (dizi, 1)"
- Formül "= (QUARTILE, dizi, 3) +1.5 * ((QUARTILE (dizi, 3)-QUARTILE (dizi, 1))" kullanarak "aykırı kesme değeri" hesaplamak ve saklayın. Tüm tepkime bu değerden büyük olan değerleri sonraki hesaplamalar dışında edilecek.
- Gelen reaktivite değerleri (RX.area-BG.area) kopyalayın ve bitişik, boş bir sütuna yapıştırın, daha sonra en büyük sütunun üstünde olduğu gibi bu değerleri sıralayabilirsiniz.
- Yeni oluşturulan "sıralama kriteri değerleri sütun" olarak, aykırı kesim değerinden daha büyük değerler silin.
- "Sıralama kriteri değerleri colu kalan reaktivite değerleri en büyük% 8 ortalama hesaplayın ve saklamakmn ". Bu değer bir" etkili maksimum "reaktivite.
- "Normalize reaktivite değerleri" elde etmek için "etkili maksimum" reaktivite değeri (aykırı dahil) her nükleotid (RX.area-BG.area) sınıflandırılmamış bölün. Sola boş bir sütun bırakarak, boş bir sütununda bu saklayın. Daha sonra, tablonun sol tarafındaki nükleotid numara kopyalamak ve doğrudan "normalize reaktivite değerleri" solundaki boş sütuna yapıştırın.
- Kopyalayıp, bir metin editörü içine nükleotid pozisyonu-normalize tepkime değer çiftleri yapıştırın.
- Bu muhtemelen RT şablonun kimyasal değişiklik dışındaki nedenlerle cDNA sentezi sırasında duraklatma sonucu olarak, -0.09 (yani boşluk boş bırakın) aşağıda değerleri ortadan kaldırmak. Ayrıca, duraklatma güçlü değiştirilmemiş şablonu (olarak "aynı hizaya getirin ve entegre" ShapeFinder profili görsel inceleme ile belirlenir) üzerine görülmektedir hangi nükleotid için herhangi bir tepkime değerleri, ekarte edilmelidir.
- SaRNAstructure (v5.3) yazılımı ile yapısal analiz kullanılmak üzere bir. şekli uzantılı dosyayı ettik.
8. Veri Modelleme
RNAstructure (v5.3) yazılım şekil analizi 19 türetilen sözde serbest enerji kısıtlamaları kullanarak deneysel olarak desteklenen RNA ikincil yapı (lar) tahmin etmek için kullanılır. Yazılım en düşük enerji 2D RNA yapıların grafik gösterimleri yanı sıra nokta-köşeli parantez açıklaması bu yapıların metin temsilini sağlar. İkinci yayın kalitesinde görüntüler üretmek için kullanıcının tercihine, örneğin Pseudoviewer 23 veya Varna 22, bir RNA yapısı görüntüleyici olarak alınabilir.
Not: RNAstructure (v5.3) yazılım tarafından üretilen yapı göz önüne alındığında dikkat edilmelidir. Örneğin, yazılım gibi pseudoknots ve öpüşme döngüler gibi üçüncül etkileşimleri çözemez, ne de bunun olmadığını eksikliği ayırt edebilirsinizBelirli bir bölgede tepkime bağlı proteinler tarafından basepairing veya sterik koruma kaynaklanmaktadır. Kesin bir yapısal model sunulması sırasında bir sonucu olarak, bu faktörler, her yapı için rapor edilen enerji ile birlikte, dikkat edilmesi gerekir.
Sonuçlar
RNA mevcudiyetinde, HIV-1 devir tepki elemanı (RRE) ve bir 3 'terminal yapısı kaseti 4 bunun 37' de ısıtma, soğutma ve inkübasyon tarafından katlanmış ve bundan sonra in vitro transkripsiyon ve doğrusallaştırılmış bir plasmidden elde edilmiştir ° C ihtiva eden MgCl2. RNA NMIA maruz bırakıldı ve daha sonra 3 'terminal yapısı kaseti hibridize bir 5'-uç-etiketli DNA primeri transkripsiyonu ters. Sonuçta ortaya çıkan şekil cDNA kütüphanesi...
Tartışmalar
Burada yüksek verimli SHAPE için ayrıntılı bir protokol, her boyutta RNA'lar için tek nükleotid çözünürlük sekonder yapı tayini sağlayan bir teknik size sunmak. Ayrıca, ikincil yapı tahmini algoritmaları ile deneysel SHAPE veri bağlantısı tek başına ya yöntemiyle mümkün olandan daha yüksek doğruluk derecesi ile RNA 2D modellerin üretimi kolaylaştırır. Floresan-etiketli primerlerin ve otomatik CE kombinasyonu tek bir deneyde uzun RNA sekanslarının, çözünürlük kolaylaştırmak gel...
Açıklamalar
Çıkar çatışması ilan etti.
Teşekkürler
S. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Rausch ve SFJ Le Grice Ulusal Kanser Enstitüsü, Ulusal Sağlık Enstitüleri, ABD İntramural Araştırma Programı tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5' labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied - including any 3'terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies | ||
Referanslar
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 '-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 '-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2'-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues - A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır