Method Article
Kromatin Regülatörleri ve Devletleri'nde genom Anlık
Bu Makalede
Özet
nasıl kromatin düzenleyiciler ve kromatin devletlerin soru in vivo genom hücre kaderi kararları gelişen embriyonun yapılır ne kadar erken anlayışımıza önemli olduğunu etkileyebilir. ChIP-Seq-küresel düzey bir Xenopus embriyolar için burada özetlenen kromotin özelliklerini araştırmak için en popüler yaklaşım.
Özet
The recruitment of chromatin regulators and the assignment of chromatin states to specific genomic loci are pivotal to cell fate decisions and tissue and organ formation during development. Determining the locations and levels of such chromatin features in vivo will provide valuable information about the spatio-temporal regulation of genomic elements, and will support aspirations to mimic embryonic tissue development in vitro. The most commonly used method for genome-wide and high-resolution profiling is chromatin immunoprecipitation followed by next-generation sequencing (ChIP-Seq). This protocol outlines how yolk-rich embryos such as those of the frog Xenopus can be processed for ChIP-Seq experiments, and it offers simple command lines for post-sequencing analysis. Because of the high efficiency with which the protocol extracts nuclei from formaldehyde-fixed tissue, the method allows easy upscaling to obtain enough ChIP material for genome-wide profiling. Our protocol has been used successfully to map various DNA-binding proteins such as transcription factors, signaling mediators, components of the transcription machinery, chromatin modifiers and post-translational histone modifications, and for this to be done at various stages of embryogenesis. Lastly, this protocol should be widely applicable to other model and non-model organisms as more and more genome assemblies become available.
Giriş
The first attempts to characterize protein-DNA interactions in vivo were reported about 30 years ago in an effort to understand RNA polymerase-mediated gene transcription in bacteria and in the fruit fly1,2. Since then, the use of immunoprecipitation to enrich distinct chromatin features (ChIP) has been widely adopted to capture binding events and chromatin states with high efficiency3. Subsequently, with the emergence of powerful microarray technologies, this method led to the characterization of genome-wide chromatin landscapes4. More recently, chromatin profiling has become even more comprehensive and high-resolution, because millions of co-immunoprecipitated DNA templates can now be sequenced in parallel and mapped to the genome (ChIP-Seq)5. As increasing numbers of genome assemblies are available, ChIP-Seq is an attractive approach to learn more about the genome regulation that underlies biological processes.
Here we provide a protocol to perform ChIP-Seq on yolk-rich embryos such as those of the frog Xenopus. Drafts of the genomes of both widely used Xenopus species—X. tropicalis and X. laevis—have now been released by the International Xenopus Genome Consortium6. The embryos of Xenopus species share many desirable features that facilitate and allow the interpretation of genome-wide chromatin studies, including the production of large numbers of high-quality embryos, the large size of the embryos themselves, and their external development. In addition, the embryos are amenable to classic and novel manipulations like cell lineage tracing, whole-mount in situ hybridisation, RNA overexpression, and TALEN/CRISPR-mediated knockout technology.
The following protocol builds on the work of Lee et al., Blythe et al. and Gentsch et al.7-9. Briefly, Xenopus embryos are formaldehyde-fixed at the developmental stage of interest to covalently bind (cross-link) proteins to their associated genomic DNA. After nuclear extraction, cross-linked chromatin is fragmented to focus subsequent sequencing on specific genomic binding or modification sites, and to minimize the contributions of flanking DNA sequences. Subsequently, the chromatin fragments are immunoprecipitated with a ChIP-grade antibody to enrich those containing the protein of interest. The co-immunoprecipitated DNA is stripped from the protein and purified before creating an indexed (paired-end) library for next-generation sequencing (NGS). At the end, simple command lines are offered for the post-sequencing analysis of ChIP-Seq data.
Protokol
NOT: Tüm Xenopus çalışmalar İngiltere Hayvanlar (Bilimsel Prosedürler) Tıbbi Araştırma MAM Ulusal Enstitüsü tarafından yürütülen Yasası 1986 ile tamamen uyumludur.
1. Hazırlıklar
- ChIP deney (tartışma) için gerekli olan embriyoların sayısını tahmin edin.
- Aşağıdaki oda sıcaklığında saklanır çözelti hazırlayın: 7.5'e ayarlandı ve otoklav ile sterilize EDTA, pH olmadan 10x Marc Modifiye Ringers (KKK), 500 ml (1 M NaCI, 20 mM KCI, 20 mM CaCI2, 10 mM MgSO 4, 50 mM HEPES pH 7.5), 10, 1% 1 SDS), 1 5x DNA yükleme tamponu (% 0.2 Portakal G,% 30 gliserol mi, 60 SDS elüsyon tamponu (50 mM Tris-HCI, pH 8.0, 1 mM EDTA, ml mM EDTA, pH 8.0).
- 4 ° C'de saklanır aşağıdaki çözümleri hazırlayın: 50 HEG tamponu (50 mM HEPES-KOH, pH 7.5, 1 mM EDTA, pH 8.0,% 20 gliserol), ekstraksiyon tampon E1 (50 mM HEPES-KOH, pH değeri, 500 ml mi 7.5, 150 mM NaCI, 1 mM EDTA. % 10 gliserol,% 0.5 Igepal CA-630,% 0.25 Triton X-100) ve E2 (10 mM, pH 8.0 Tris-HCI, 150 mM NaCI, 1 mM EDTA, 0.5 mM EGTA) ve E3 (10 mM Tris-HCI pH 8.0, 150 mM NaCI, 1 mM EDTA,% 1 Igepal CA-630,% 0.25 Na-Deoksikolat,% 0.1 SDS), RIPA tamponu (50 mM HEPES-KOH, pH 7.5, 500 mM LiCİ, 1 mM EDTA, 500 ml % 1 Igepal CA-630, 0.7% Na-deoksikolat) ve TEN tamponu (10 mM Tris-HCI, pH 8.0, 1 mM EDTA, 150 mM NaCl), 50 ml.
- Skor ve 7 ml işaretine 15 ml konik polistiren tüp klibi. Sonikasyon geçiren nükleer özler içeren bu tüp kullanın.
- Post-dizi analizi için, en az 8 GB RAM ve 500 GB boş disk alanı ile bir çok çekirdekli Unix tarzı işletim bilgisayarı kullanın. Yerel bunların çoğu komut satırında kullanılan aşağıdaki yazılımı yükleyin: FastQC, Illumina casava-1.8 kaliteli filtre, Papyon 11, SAMtools 12, HOMER 13, MACS2 14, IGV 15,16, Cluster3 17, Java TreeView, ŞOK + 18, ve b2g4pipe 19. Derleyiciler ve üçüncü parti yazılımlar için kurulum talimatları ve gereksinimleri kontrol edin.
- Kısa NGS Xenopus genomunun okur hizalanması için bir Papyon endeksi oluşturun. Bir örnek X için burada gösterilir Xenbase ftp sunucusu (/ pub / Genomik / JGI) bir FASTA dosyası (genome.fa) olarak indirebilirsiniz tropicalis genom v7.1 (Kasım 2011). Bowtie dizin alt dizinine FASTA dosya taşımak.
- XenTro7 indeks dosyaları oluşturmak için (> istemi karakterden sonra burada) aşağıdaki komut satırını kullanın:
> Papyon-inşa /path/to/bowtie/index/genome.fa xenTro7
> Ihracat BOWTIE_INDEXES = / / yol / papyon / index /
- XenTro7 indeks dosyaları oluşturmak için (> istemi karakterden sonra burada) aşağıdaki komut satırını kullanın:
- UCSC Genom Tarayıcı veya Araçlar / Masa Tarayıcı üzerinden en son genom sürümleri (genomes.nimr.mrc.ac.uk) için Nimr sunucusunda ayna sitesinden gen açıklama dosyası (GTF) indirin. Genom FAŞTA dosyası ve H özelleştirmek için GTF dosyasını kullanınXenopus için OMER (örneğin, X tropicalis genom v7.1, - xenTro7 adı).
- Alternatif olarak, Xenopus genomunun bazı eski sürümleri için önceden oluşturulmuş HOMER paketleri kullanın.
> LoadGenome.pl name xenTro7 -ORG boş -fasta /path/to/genome.fa -gtf / yol / genes.gtf
- Alternatif olarak, Xenopus genomunun bazı eski sürümleri için önceden oluşturulmuş HOMER paketleri kullanın.
- Ve açıklama dosyası (genes.gtf) (aynı klasörde genome.fa.fai dosyası ile genome.fa) bir dizinlenmiş FASTA dosyasını yükleyerek genom tarayıcısı IGV için bir genom parça (.genome dosyası) oluşturun. Aşağıdaki gibi Ksenopus genomunun bir genom iskele indeksi (genome.fa.fai) oluştur:
> Samtools faidx /path/to/genome.fa - Aşağıdaki gibi Xenopus genler üzerine birkaç modeli türlerinin (insan, fare, zebra balığı, meyve sineği ve maya) den Gen Ontoloji (GO) terimleri geçmek patlama + kullanın:
- Araçlar aracılığıyla UCSC Genom Tarayıcı tek bir FASTA dosyası (cds.fa) gibi tüm kodlama dizileri (CDS) indirin/ Tablo Tarayıcı ve olmayan gereksiz proteinlerin (nr) önceden biçimlendirilmiş ŞOK veritabanı ile güncelleme ŞOK +:
> Update_blastdb.pl nr - Insan (txid9606) ara, fare (txid10090), Zebra balığı (txid7955), meyve sineği (txid7227) ve maya (txid4932) gelişmiş arama fonksiyonu ile NCBI sitesinden proteinler (http: //www.ncbi.nlm.nih. gov / protein / gelişmiş) ve bilgisayara sonuçtaki GI (dizi tanımlayıcı) liste (sequence.gi.txt) göndermek.
- Belirli bir bekliyoruz (E) değeri kesme (burada 10 -20) ile BLASTX yürüterek GI listeden en benzer proteinlere Ksenopus genleri atayın. Emin çıkış biçimi xml olduğundan emin olun (-outfmt 5 -out blastx_results.xml). Mevcut bilgisayar çekirdek sayısı ile ilişkili zaman tasarrufu iş parçacığı (-num_threads) yararlanın.
> BLASTX -DB / / yol / nr -gilist /path/to/sequence.gi.txt -query / yol / tonO / cds.fa -evalue 1e-20
-outfmt 5 -out /path/to/blastx_results.xml -num_threads [# konuları] - Bir metin editörü ile b2g4pipe klasörünün b2gPipe.properties dosyasını açın ve Dbacces.dbname = b2go_sep13 ve Dbacces.dbhost = publicdb.blast2go.com veritabanı özelliklerini güncelleyin. Yükleme klasöründen çalıştırın b2g4pipe.
> Java Xmx1000m -cp *: ext / *: es.blast2go.prog.B2GAnnotPipe -in /path/to/blastx_results.xml
-out sonuçlar / xenTro7 -prop b2gPipe.properties v -annot
Not: Bu program ekstreleri vurmak her BLAST koşullarını GO ve ilgili Xenopus genlerinin (xenTro7.annot) bunları atar. En güncel veritabanı ayarları Araçları altında bulunabilir / Genel Ayarlar / Blast2GO Java Web Start uygulaması DataAccess Ayarları (9.11.1 bakınız).
- Araçlar aracılığıyla UCSC Genom Tarayıcı tek bir FASTA dosyası (cds.fa) gibi tüm kodlama dizileri (CDS) indirin/ Tablo Tarayıcı ve olmayan gereksiz proteinlerin (nr) önceden biçimlendirilmiş ŞOK veritabanı ile güncelleme ŞOK +:
2. Kromatin Çapraz bağlama
- Xenopus yumurta, de-jöle ve kültür em gübreleyinbryos standart protokollere 20 göre yöntem.
- Kapaklı bir 8 ml cam numune şişesine ilgi gelişim aşamasında dejellied embriyolar (maksimum 2.500 X. laevis veya 10.000 X. tropicalis) aktarın ve 0.01x MMR ile kısaca bir kez yıkayın.
- Oda sıcaklığında 40 dakika için 15 (örneğin, 8 ml 0.01x MMR ile 36,5-38% formaldehit 225 ul ekleyin) 0.01x MMR% 1 formaldehit ile embriyolar Fix (sabitleme zaman ve gerekli embriyoların sayısı için bkz Tartışma ) ChIP deney başına.
NOT: Formaldehit korozif ve son derece toksiktir. Bu göz ve cilt teması, hazımsızlık, ve solunması durumunda zararlı olduğunu. Flakon formaldehit eklerken davlumbaz kullanın. - Kısaca soğuk 0.01x MMR ile embriyolar üç kez yıkanarak tespiti durdurun. Yüzey gerilimi onları yırtmak için neden çünkü Embry os sıvı yüzeyi ile temas etmesine izin vermeyin.
- Buz üzerinde 2 ml mikrosantrifüj tüplerine embriyolar alikosuYaklaşık 250 ul (X. tropicalis) veya kuluçkalık önce 600 ul (X. laevis) bir hacim işgal tüp başına 250 embriyoların, maksimum.
- Mümkün olduğunca uzak Pipet kadar 0.01x MMR. Eğer bölüm 3 ile hemen devam ederse, aşağıdaki adımı atlayın.
- Soğuk HEG tamponu 250 ul embriyolar dengelenmesi. Embriyolar tüpün dibine yerleşmiş sonra mümkün olduğunca fazla sıvı çıkarın ve ek-freeze sıvı azot içinde. -80 ° C'de saklayın.
3. Kromatin Ekstraksiyon
NOT: Xenopus embriyolar çapraz bağlı kromatin aşağıdaki çıkarma aşamasında 2.3 ve 50-80 X belirtilen sabitleme süreleri ile en verimli çalışır 40 X'e tropicalis veya 25 ekstre alma tampon maddesi E1, E2 ve E3 ml'si başına embriyolar laevis. Tampon iki hesaplanan miktarı, gerekli şekilde Her bir ekstraksiyon aşaması tekrarlanır. Yükseltme için çok sayıda 2 mi microcentri kullanımıfuge tüpler veya 50 ml santrifüj tüpleri. Kromatin ekstraksiyon sırasında buz üzerinde örnekleri ve tamponlar tutun.
- Tamponlar, E1, E2, ve 1 mM DTT ile E3 ve proteaz inhibitörü, tabletler yeterli miktarlar Ek. Fosfo-spesifik antikor, 5 mM NaF, 2 mM Na 3 VO 4 ile daha fazla ek tampon fiş gerçekleştirmek gerekebilir.
- Yukarı ve aşağı pipetleme E1 ile sabit embriyolar homojenize. 2 dakika boyunca 1000 x g'de soğutmalı santrifüj (4 ° C) santrifüje homojenatları (ya da 5 dakika 50 ml tüpler kullanılması halinde). Süpernatant ve duvara bağlı herhangi lipidler aspire.
- E1 pelet yeniden süspanse. 10 dakika boyunca buz üzerinde örneklerin tutun. Adım 3.2 gibi Santrifüj ve ıskarta süpernatanları.
- E2 pelet yeniden süspanse. Adım 3.2 gibi Santrifüj ve ıskarta süpernatanları.
- Adımı tekrar 3.4 ama santrifüj işleminden önce 10 dakika boyunca buz üzerinde örnekleri tutmak.
- E3 pelet yeniden süspanse. En az 10 dakika boyunca buz üzerinde örneklerin tutun. Santrifüj ve atın suAdım 3.2 gibi pernatants.
NOT: Bu aşamada, resuspensions oldukça şeffaf hale gelmelidir. E3 anyonik deterjanlar çözünür kalan yumurta sarısı trombositlerin en hale getirilmesi ile çapraz bağlanmış çekirdek ekstrakte edin. - Çapraz-bağlanmış çekirdeklerin süspanse ve havuz topaklar E3 1 ml'lik toplam hacim içinde (normal olarak çözünmemiş pigment granüllerinin kahverengi renkte). Çok viskoz görünür ve pipet zor ise 2 ya da 3 ml E3 ile örnek seyreltilir. Buz üzerinde tutun veya 4 ° C'de aynı veya ertesi gün, adım 4 ile devam etmek. Daha sonra kullanılmak üzere -80 ° C'de, sıvı azotun ve mağaza ek bileşen dondurma.
4. Kromatin Parçalanma
Not: Sonikasyon çözündürmek için ve çapraz bağlantılı kromatin kesme hem de kullanılır. İşte 1/16 inç konik mikrotip ve ses muhafaza ile donatılmış Misonix Sonikatör 3000 çalıştırmak için parametrelerdir. Diğer selenleyici kullanıyorsanız, üreticilerin önerileri kesme takipçapraz-bağlanmış kromatin veya toplam olarak 4-8 dakika için 12 W 6 kullanın.
- Sonikasyonun (adım 1.4) için özel yapılmış bir tüp içine adım 3.7 nükleer örnek aktarın. Kısa bir termometre kelepçe ile buzlu su ile doldurulmuş bir 800 ml'lik plastik çanağa bağlı tüp sağlayarak sonikasyon sırasında soğutulmuş örnek tutun.
- Bir laboratuar jakı Beher yerleştirin. Sonikatör mikrotip tüp duvar dokunmadan ses derinliği yaklaşık üçte ikisi numunede submersed ve ortalanmış şekilde kriko ayarlayın.
- Toplam 7 dakika örnek sonikasyon, 1 dakika duraklar her 30 saniyede kesildi. 1.0 güç ayarlayın. Sonikasyon başlatın ve hemen örnek köpüğe başlarsa W. hemen Pause 9 ila 12 arasında bir okuma ulaşmak için (normalde 2-4) güç ayarını artırın. Boruyu ve köpük tamamen kayboldu zaman yeniden başlatın.
- 15,0> (tam hızda önceden soğutulmuş 1.5 ml mikrosantrifüj tüpler ve döndürme makaslanmış kromatin aktarın4 ° C'de 5 dakika boyunca 00 x g).
- 1.5 ml mikrosantrifüj tüpleri öncesi soğutulmuş süpernatant aktarın. Kromatin parçalanması (bölüm 5) derecesini görselleştirmek için (ideal etrafında 400,000 veya daha fazla çekirdeğin kromatin içeren) süpernatant 50 ul toplayın. ChIP (bölüm 6) için süpernatant kalanını kullanın.
- Kadar bir gün için 4 ° C'de saklayın örnekleri. Parçalar halinde Ek dondurma örnekleri -80 ° C 'de, uzun süreli depolama için sıvı azot içinde (çip deney başına bir tane).
5. Görüntüleme Kromatin Parçalanma
- SDS yürütme tamponunun 50 ul 5 M NaCI 4 ul ve aşama 4,6 süpernatan 50 ul proteinaz K (20 ug / ml), 1 ul ekle.
- Fırın 65 ° C'ye ayarlanmış bir melezleme 6-15 saat (O / N) inkübe edilir.
- Ticari bir PCR saflaştırma kiti kullanılarak DNA arındırın. Gerekirse, üretici tarafından tavsiye edildiği gibi pH değerini ayarlamak için, sodyum asetat (pH 5.2), 3 M kullanın. ZehirDNA, iki kez yıkama tamponu 11 ul (10 mM Tris-HCI, pH 8.5) ile.
- 100 bp ile birlikte tüm numune ve elektroforez ile% 1.4 agaroz jel üzerinde 1 kb DNA merdiveni, çalıştırmadan önce RNaz 0.4 ul (20 ug / ml) ile 5x DNA yükleme tamponu içinde 5 ul ekle. Elektroforez sonra güvenli bir nükleik asit boyama solüsyonu ile optimum sonuçlar, leke jel için.
6. Kromatin İmmunopresipitasyon
NOT: Bu bölümde, 4 ° C'de 5 dakika boyunca manyetik boncuklar yıkamak için düşük tutma 1.5 ml mikrosantrifüj tüpleri ve tüp başına belirtilen tampon en azından 1 ml kullanılır. Boncuk tampon çıkarmadan önce, 20 her zaman sn 30 veya çözelti berrak olana kadar manyetik rafa tüpleri bırakın.
- Yeni bir tüp adım 4.6 den süpernatan (makaslanmış kromatin) 30 ul transfer 10 ChIP için kullanılan toplam kromatin yaklaşık% 1'ine tekabül giriş numune olarak daha sonra kullanılmak üzere. 4 MağazaÇip sample çapraz bağlar tersine çevirmek için hazır olana kadar ° C.
- Yeni bir tüpe kalan kromatin aktarın. Bir antikor kontrolü gerektiren ChIP-qPCR deneyleri için, iki tüplere kromatin eşit hacimlerde dağıtmak.
- Kromatine ChIP dereceli antikoru (veya ilgili antikor kontrolü) ekleyin. Bir kaba bir kılavuz olarak, yaklaşık 1 ug ilgi epitopunu ifade bir milyon hücre başına antikor kullanımı.
- Daha doğru antikorun çeşitli miktarlarda aynı ChIP çalıştırın, (örneğin, 0.25 ug, 1 ug ve 2.5 ug) ChIP deney başına gereken antikor miktarını tahmin ve ChIP-qPCR tarafından negatif ve pozitif kontrol loci de verim karşılaştırmak için (bkz bölüm 10). Bir antikor bir kontrol olarak, bir antikor ile aynı izotip ve ana hayvan türleri normal serum kullanılır.
- 4 ° C'de / bir rotator (10 rpm) O üzerinde N inkübe edin.
- 5 dakika için a E3 ile bir kez antikor uyumlu manyetik boncuk yeterli miktarda yıkayınt, 4 ° C. Kontrol boncuk antikor bağlama kapasitesi üreticinin özellikleri (boncuk genellikle 5 ila 20 ul IgG antikor 1 ug bağlamak).
- Antikor ön kuluçkaya kromatine yıkanmış boncuk ekleyin. Daha fazla 4 saat süre ile, bir döndürücü (10 rpm) ile inkübe edilir.
- Yıkama önceden soğutulmuş TEN tamponu ile bir kez daha sonra dört kez (ChIP-qPCR) ya da ön-soğutulmuş RIPA tamponu ile on defa (ChIP-Seq), ve boncuklar.
- Bir ChIP-Sıra deneyi gerçekleştirmek Yalnızca bu adımı yürütmek.
- Süspanse tüp başına TEN tamponu 50 ul boncuk yıkanmıştır. Yeni bir tüp aktararak bir tek ChIP deney Havuz tüm boncuklar. Tüpün alt kısmında boncuk toplamak için 1.000 xg'de manyetik raf ve buzdolabında (4 ° C) santrifüj kullanın. Boncuk pelet bozmadan kadar sıvı mümkün olduğunca atın.
- SDS elüsyon tamponu ve Vort ve 50-100 ul boncuk resuspending boncuk kapalı Şerit cips malzemesinin65 ° C sıcaklıkta 15 dakika boyunca bir termomikser (1000 rpm) ile sürekli olarak bunları exing. 30 saniye için tam hızda (> 15.000 xg) bu santrifüj sonra. Yeni bir tüp süpernatant (ChIP eluatı) aktarın.
- Son adımı tekrarlayın ve ChIP eluatları birleştirir.
7. Kromatin Arka çapraz bağlanması ve DNA saflaştırma
- 100 ila 200 ul (adım 6.10) 'dir ChIP numune, hacmini ulaşmak için giriş numune (adım 6.1) için yeterli SDS elüsyon tamponu ekleyin. 5 M NaCl 1/20 hacmi ile hem ChIP ve giriş örnekleri Ek. , Bir hibridizasyon etüvü içinde 65 ° C sıcaklıkta 6-15 saat (O / N) örnekleri inkübe edin.
- 200 ug / ml TE tamponu ve RNaz A 1 hacim. 37 ° C'de 1 saat süreyle inkübe edin.
- 200 ug / ml proteinaz K ilave. 55 ° C'de 2 saat ila 4 inkübe edin.
- Fenol DNA arındırın: kloroform: etanol çökeltme izlenmiş ve izoamil alkol çıkarma, daha önce 9 gösterildiği gibi üretilebilir. ChIP-Sıra için, 32 ekleyinelüsyon tamponu (10 mM Tris-HCI, pH 8.5), ul DNA pelet eritin. 30 dakika DNA, tamamen çözülene sağlamak için buz üzerinde örneklerin bırakın.
Not: Ticari PCR saflaştırma kiti düşük bir DNA kurtarma ama daha uygundur, ve talaş qPCR örnekler için kullanılabilir. - ChIP-Sek için fluorometri dayalı yöntemler kullanılarak çip ve giriş DNA 1 ul konsantrasyonunu belirler. Üreticinin talimatlarını izleyin ve DNA konsantrasyonu florometre güvenilir algılama aralığında düşüyor emin olun.
8. ChIP-Seq Kütüphanesi İnşaatı ve Doğrulama
NOT: DNA kütüphanesi hazırlanması için Güncel yöntemler 1'den 2'ye ng NGS için yüksek karmaşıklık kütüphaneleri inşaatına izin. Bazı karmaşıklık pahasına, kütüphaneler DNA az 50 pg (Belirli Malzemeleri / Ekipman Tabloya bakınız) yapılabilir. ChIP ve giriş kütüphanede hem de DNA aynı miktarda kullanın. Kısaca, mak içine endeksli ChIP-Seq kütüphaneler, ChIP ve giriş DNA sonu-tamir olması gerekir (paired-end), (Belirli Malzeme / Ekipman bkz: İçindekiler) özel adaptörler lige, boyut seçilen ve PCR ile çoğaltılan.
- ChIP-Seq kütüphaneler yapmak için üreticinin yönergeleri izleyin. Daha fazla öneriler için tartışma bakın.
- Elüsyon tamponu 12 ul her kütüphane Zehir ve bir flüorometre kullanarak her ChIP ve giriş kütüphane 1 ul konsantrasyonunu belirlemek. 5-25 ng / ul konsantrasyonlarında bekliyoruz. Konsantrasyonları daha yüksek 25 ng / ml ise PCR döngüleri (az 18 siklus) sayısının azaltılması düşünün.
NOT: Doğru ölçümü en iyi NGS sonuçları elde anahtarıdır. 18 PCR döngüsünden sonra 1 ng / ul kadar düşük konsantrasyonlarda Kütüphane dizilenmiştir, ancak sık sık daha düşük karmaşıklık girmektedir. - Fragman boyutu dağılımını belirlemek için kütüphane 1 ul kullanın ve c herhangi bir adaptör dimer kontaminasyon (bant yaklaşık 120 bp) kontrol etmek içinKalça-tabanlı kapiller elektroforez. 1: 1 bir boncuk-örnek bir oranı olan katı faz döner hareketsizleştirme arıtma tekrar (yerine 1.6: 1) kütüphane adaptör dimerleri içeriyorsa.
- Benzer DNA zenginleştirme eğilimleri öncesi ve kütüphane hazırlık sonra gözlenen olup olmadığını kontrol etmek için onaylanmış pozitif ve negatif kontrol loci (bakınız bölüm 10) üzerine qPCR gerçekleştirin. Sıralama için kalite kontrol onaylı kütüphaneleri gönderin.
9. Post-sıralama Analiz ve Veri Görselleştirme
NOT: Günümüzde, NGS genellikle içi veya ticari sıralama tesisleri (bazı NGS kılavuzlar için tartışmaya bakınız) tarafından yürütülmektedir. Standart çıkış sıralaması okur milyonlarca depolanması tek veya çoklu gzip-sıkıştırılmış fastq dosyaları (* .fastq.gz) vardır. Normalde, çoğaltılmış kendi endeksine göre zaten ayrılmış okur ve her okuma, her bas için bir dizi tanımlayıcı ve kalite kontrol puanı (Illumina için Phred + 33 1.8+) içerirE çağrısı. Bu yaklaşım, buradan NGS verilerini analiz etmek için kaç yollarından sadece biridir. okuyucu aşağıdaki komut satırlarından herhangi bu alan hızla ilerlemektedir ve güncellemeler düzenli yaşanıyor değişiklikleri gerektiren olmadığını kontrol etmek teşvik edilmektedir.
- Gzip-sıkıştırılmış fastq dosyaları birleştirmek ve FastQC script kullanarak sıralama verilerinin kalitesini kontrol edin. Bu Yürütme ve hem ChIP ve giriş sıralama verileri için aşağıdaki komutları en Terminalden (örnekler çipi için gösterilmiştir):
> Kedi /path/to/*.fastq.gz> ChIP.fastq.gz
> Fastqc ChIP.fastq.gz
NOT: Bir yüksek karmaşıklık ChIP-Sıra kütüphane başarılı sıralanması Ham veriler çoğu testleri geçmelidir. Başarısızlıklar kötü sıralama çalışır ve böyle önyargılı PCR amplifikasyonu veya adaptör kirlenme gibi deneysel eserler ağırlıklı kaynaklanır. Gereksiz iyi niyetli DNA zenginleştirme temsil edebilir okur olarak çoğaltılması (fazlalık) belli bir derecesi bekleniyor 21. 5 'ucunu veya okur - - Ancak, bir sonraki okuma etiketleri kısıtlayabilirsiniz birine baz çifti başına herhangi bir gereksiz (adım 9.4) 21 zirveleri algılama hassasiyetini etkilemeden okur ortadan kaldırmak için. - Ön işlem sıralama verileri (homerTools Döşeme -3 ) sağlayan bir uyumsuzluk (-mis 1) adaptör kontaminasyonu temizlemek için. Ligasyon üzerine ilgi DNA fragmanı proksimal (3 '5) (endeksli) adaptörün ilk 20 üsleri kullanın (Özel Malzeme / Ekipman Tablo listelenen adaptörü için gösterilen).
> Gzip -cd ChIP.fastq.gz | fastq_illumina_filter -VN> ChIP.fastq
> HomerTools Döşeme -3 GATCGGAAGAGCACACGTCT 1 -min 36 ChIP.fastq -mis
NOT: süzüldü kaldırılması (N) Sadece Illumina 1.8 tarafından oluşturulan fastq dosyaları varsayılan olarak gereklidir okur. Fastq_illumina_filter komutu ihmal (yani. "| Fastq_illumina_filter -VN ')1.8 daha eski bir sürüm dizisi tanımlayıcı oluşturulan eğer. - Hizalama önceden işlenmiş Bowtie kullanarak referans genom (xenTro7) okur. Sadece benzersiz eşlenen tutmak okur (-m 1) (-S) ilk 28 üsleri ve SAM formatında maksimum 70. Rapor uyum okuma başına tüm uyumsuzlukları toplam Phred + 33 kalite puanı varsayılan, yani ayarları, maksimal iki uyumsuzlukları kullanarak . Öbek bellek bitkin ise parçacığı başına megabayt (--chunkmbs) sayısını artırın:
> Papyon -m 1 -S -p [# konuları] --chunkmbs [örneğin 200] xenTro7 ChIP.fastq.trimmed> ChIP.sam
NOT: Papyon varsayılan Phred + 33 kalite puanları bekliyor. Seçeneği ekle - phred64-bıldırcınlar fastq dosyası 1.8 daha eski Illumina tarafından Phred + 64 kalite puanları ile oluşturulan eğer. - Bir kodaman dosyası (.bw) içine hizalama (SAM) dosyasını dönüştürmek için iki HOMER komutları kullanın:
> MakeTagDirectory ChIP / -tek -tbp 1 ChIP.sam
> MakeUCSCfile ChIP /ChIP.bw -o -bigWig / / yol / genome.fa.fai -fsize 1e20 -norm 1E7
NOT: dönüşüm referans genom (adım 1.8) olarak iskele indeksi (genome.fa.fai) gerektirir. İşte profili (-norm 1E7), 10 milyona baz çifti (-tbp 1) ve normalize başına bir etiketi okur sınırlıdır. kodaman dinamik böyle IGV (adım 9.12) gibi bir genom tarayıcı ile kromatin profilleri görselleştirmek için tercih edilen biçimi biridir. - (Örneğin, +/- 25 bp bidonları, 10 kb -si ze 20000 -hist 25) gibi transkripsiyon başlangıç (TSS, örnek burada gösterilmektedir) ve sonlandırma (genomik yerlerinden de etiketleri dağılımı (-d ChIP /) belirleyin tts) siteleri. Xenopus açıklamaları xenTro7 (adım 1.7) ile HOMER Perl komut annotatePeaks.pl çalıştırın:
> AnnotatePeaks.pl TSS xenTro7 -size 20000 -hist 25 d ChIP /> ChIP_tagDensity.tss - ChIP arasındaki DNA zenginleştirme önemli zirveleri bul(ChIP.sam -t) ve X Girdi (c Input.sam) tropicalis genom model yapımı için 200 bp (--bw = 200) içindeki bir (0.01 q)% 1 FDR kesme ve (sonikasyon) sonra, DNA fragmanları ile MACS2 kullanılmıştır. Böyle histon işaretleri veya RNA polimeraz gibi ilgi kromatin özelliği geniş bir dağıtım bekliyor bu komut satırına bayrak --broad ekleyin.
> Macs2 callpeak -t ChIP.sam -c Input.sam -f SAM n ChIP -g 1.4376e9 q 0.01 --bw = 200
Not: X etkili boyutu tropicalis genom montaj v7.1 yaklaşık 1437600000 bp (-g 1.4376e9) 'dir. MACS2 kendi genomik yerleri ile doruklarına askere bir YATAK dosyası (ChIP_peaks.bed) oluşturur. - Bir kümelenmiş İlgi haritası şeklinde birkaç kromatin profilleri karşılaştırın:
- Ilgi etiket yoğunluğu dizinleri bir etiket dağıtım matrisi oluşturma (-d ChIP / other_ChIP /) MACS2 doruklarına (en örn, 25 bp kutuları, -size 2000 -hist 2 +/- 1 kb5 -ghist):
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 ölçülü 2000 -hist 25 -ghist -d çip / other_ChIP /> ChIP.matrix - ChIP.matrix dosya upload ve hiyerarşik en yakın sentroidinden minimal Öklid mesafeye göre bu etiket yoğunlukları küme Cluster3 grafik kullanıcı arabirimini kullanın. Kümelenmeyi görselleştirmek Java TreeView oluşturulan CTD dosyasını açın.
- Ilgi etiket yoğunluğu dizinleri bir etiket dağıtım matrisi oluşturma (-d ChIP / other_ChIP /) MACS2 doruklarına (en örn, 25 bp kutuları, -size 2000 -hist 2 +/- 1 kb5 -ghist):
- Pik zirvelerinde zenginleştirilmiş yeni ve önceden bilinen bağlama motifleri, +/- 100 bp (200 -size) bulabilirsiniz. Motifi oluşumları harita ve motif yoğunlukları arsa annotatePeaks.pl kullanın:
> FindMotifsGenome.pl ChIP_peaks.bed xenTro7 ChIP_motifs / -size 200 -p [# konuları]
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -m motif1.motif> ChIP_peaks.motif1
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 -m motif1.motif -size 800 -hist 25> motif1.density
NOT: findMotifsGenome.pl komut INFErs karşılaştırmadan zenginleştirme rastgele gen-merkezli arka plan dizileri seçme. En zenginleştirilmiş yeni motifi bir pozisyon ağırlığı matris biçiminde motif1.motif altında kaydedilir. okuyucu gibi cisFinder 22 ve MEME 23 gibi diğer de novo motifi keşif yöntemleri ile bu sonuçları kanıtlamak için teşvik edilmektedir. - En yakın gene kendi mesafeyi hesaplayarak ve 400 bp pencereler (400 -size) içindeki normalize okuma sayısını belirleyerek doruklarına ek açıklama:
> AnnotatePeaks.pl ChIP_peaks.bed xenTro7 400 d ChIP / Giriş /> ChIP_peaks.genes -size - Numarası (N), konum ve bireysel (R) normalize okuma sayısı ve yakın (hedef) genin başına tüm (LR) zirveleri listelemek için aşağıdaki awk komutunu kullanarak çıktıyı özetler.
> Awk '{ t' 7 $> = -5000 && $ 7 <= 1000 FS =} BEGIN '
{N- [$ 8] + = 1 'dir; R [$ 8] + = $ 9; LR [$ 8], 'LR [$ 8] = &# 39; 7 $ '(' 9 $ ')'} END {(i N)
{Baskı i ' t' N [i] '' t R [i] '' t substr (LR [i], 2)}} 'ChIP_peaks.genes> ChIP_peaks.summary
Not: $ izleyen sayı, önceki adımda oluşturulan ChIP_peaks.genes dosyasını uyacak şekilde değiştirilmesi gerekir olabilir sütun numarası, ifade eder. Bu komut exemplar 5 kb memba ve mansap TSS 1 kb ötesinde doruklarına filtreler. 7 $, 8 $ ve $ 9 TSS, sırasıyla gen tanımlayıcı ve tepe başına normalize okuma sayısı, uzaklığa bakın. - Aşağıdaki gibi hedef genlerin arasında zenginleştirilmiş GO açısından analiz gerçekleştirin:
- Java Web Start (javaws) 19 üzerinden komut satırından Blast2GO grafik kullanıcı arayüzü başlatın.
> Javaws http://blast2go.com/webstart/blast2go1000.jnlp - Açıklamayı yüklemek için geliştiricilerin talimatları izleyinXenopus 1.9.4 oluşturulan gibi genlerin (xenTro7.annot) ve belirlenen hedef genlerin düz bir dosya için dosya. Aynı gen tanımlayıcıları, hem dosyaları kullanıldığından emin olun.
- Java Web Start (javaws) 19 üzerinden komut satırından Blast2GO grafik kullanıcı arayüzü başlatın.
- Parça olarak IGV içine kodaman (ChIP.bw, Input.bw) ve YATAK dosyaları (ChIP_peaks.bed) ekleyerek kromatin profillerini gözünüzde canlandırın. RNA Sıra ile Kompleman veri gelişme aynı aşaması için varsa izler. Bir oturumda gibi sonuçları kaydedin.
- Programlama platformları R kullanın (www.r-project.org) yukarıda oluşturulan gibi daha fazla veri işlemek ve görselleştirmek veya MATLAB. Alternatif olarak, Excel ile küçük veri setleri arsa.
10. ChIP-qPCR Testi ChIP ve Onaylanması ChIP-Seq
- Pozitif (pik spesifik) ve negatif kontrol lokuslarının hem de 60 ° C (Tm) yaklaşık olarak 100 bp'lik bir DNA çevreleyen primerler tasarlamak üzere on-line bir platform Primer3 kullanın. Astar özgüllüğü kullanarak onaylayın silikotüberküloz PCR arama UCSC Genom Tarayıcı içine uyguladı.
- Yaklaşık% 1 girişinden itibaren üç kat dilüsyonlarının 8-noktalı bir standart eğri oluşturma veya 2 kullanın - DNA zenginleştirme ölçümü için ΔΔC (T) yöntemi 8,24.
- Gerekirse, standart eğri örnekleri Tüm örneklerde, yani, ChIP, kontrol için teknik üçlü gerçek zamanlı PCR Yürütme ve.
- Giriş DNA yüzdesi olarak pozitif ve negatif kontrol loci hem de kontrol numunesi karşısında çip oranı olarak Konu DNA zenginleştirme.
Sonuçlar
Protokol de idam eğer burada sunulan eşdeğer sonuçlar beklenmektedir ve kullanım antikor ChIP dereceli kalitesi (tartışma) biridir. Bu protokol formaldehit sabit Xenopus embriyolar çekirdeklerin çıkarma ve sonikasyon ile kromatin verimli kesme (Şekil 1A-C) izin verir. Kesilmiş kromatin esas olarak 100 ila 1000 bp arasında değişen ve bp (Şekil 1C) 300 ila 500 zirve DNA fragmanlarının asimetrik dağılımı gösterilmektedir. Çökeltilir DNA minimal 50 pg başarıyla benzer büyüklükteki DNA ekler (Şekil 2A) ile bir dizine eşleştirilmiş-uç ChIP-Sıra kütüphane yapmakla yükümlüdür. Kütüphane, yaklaşık 120 bp Elektroforegramda görülebilir adaptör dimerlerinin, büyük ölçüde yoksun olmalıdır.
Sekanslama-sentez sırasında, okuma ön işlemden genom (Şekil 2B, C) eşleştirilir. Başarılı bir deneyde In X. tropicalis embriyolar, tek sonu normalde 50 ila 70% maksimum iki uyumsuzlukları ile V7.1 genom düzeneğine benzersiz eşlenebilir 40 bp okur. Giriş genomu boyunca oldukça eşit hizaya okur iken, ChIP hizalama ilgi kromatin özelliğini kanadını iplikçik özgü zenginleşme sonuçları okur. Bütün parçalar 5 'ucuna (Şekil 2C) 25 dizilenmiştir olmasıdır. Ortalama fragman büyüklüğü yön okuma uyum uzatılması gibi transkripsiyon faktörü bağlayıcı olaylar gibi tek kromatin özellikleri doğru profiller üretmektedir. IGV veya başka bir uyumlu genom tarayıcıda görüntülendi zaman bu DNA doluluk zirveleri olarak görünür. MACS gibi Tepe arayanlar bu zirveleri (Şekil 3A) konumunu belirlemek için kullanılır. Onlarca bağlayıcı sitelerin binlerce Bu şekilde X tespit edilmiştir tropicalis genom gibi vegt 26 gibi T-box transkripsiyon faktörleri. ChIP-qPCR deneyrından ChIP-Sek (Şekil 3B) tarafından bulundu yerel zenginleştirme teyit etmelidir.
ChIP-Seq deneyler kromatin özellikleri genom özelliklerini keşfetmek izin. Örneğin, transkripsiyon başlangıç ve sonlandırma siteleri gibi genomik elemanları üzerinde okuma dağılımı hesaplanırken genlerin etrafında herhangi bir mekansal bağlama tercihleri (Şekil 3C) vurgulamak olabilir. Benzer şekilde, tepe yerlerde okuma dağıtımları bir heatmap bir genom ölçeğinde (Şekil 3D) farklı kromatin özelliklerini karşılaştırmak için kullanılır. Bazı transkripsiyon faktörleri potansiyel eş-faktörler (Şekil 3E) eş-zenginleştirilmiş motifler içeren bu tür bilgileri alabilirsiniz genomik DNA yatan zirveleri DNA dizisi-özellikle. De novo motifi analizini bağlamak. hedef genlerin büyük bir çoğunluğu DNA düşük doluluk ziyade yüksek düzeyde (Şekil 3F) gösterir. Bu ölçek serbest özelliği tr arasında oldukça yaygın gibi görünüyoranscription faktörleri ve hedef genlerin yalnızca küçük bir bölümünün doğrudan biyolojik uygunluğu 27,28 düzenlenmiştir olduğunu göstermektedir. Böyle daha Xenopus embriyo (Şekil 3G) kromatin özelliğinin biyolojik fonksiyonu anlayışlar ortaya çıkarabilir hedef genlerin diferansiyel ifadesi olarak zenginleştirilmiş GO açısından veya diğer niteliklerin analizi.
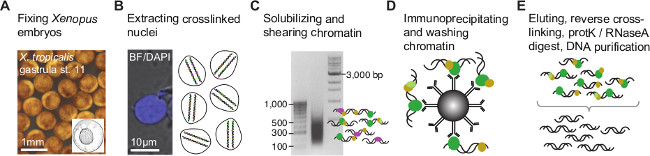
Xenopus embriyolar 1. Kromatin immüno-çökeltmesi prosedürü Şekil. (A) Embriyolar formaldehit-sabit kovalent bağlama (cross-link) ilgi genomik DNA ile ilişkili herhangi bir proteinlerin gelişimsel aşamadadır. Nükleer ekstraksiyon (B) üzerine, çapraz bağlı kromatin bağlayıcı genomik DNA veya yan DN minimize ederek kromatin modifikasyon siteleri daraltmak için parçalanmışBir dizi (° C). Daha sonra, kromatin fragmanları ilgi (D) epitop ihtiva eden ettirecek bir çip dereceli antikoru ile imüno-çökeltilir. ko-immunoprecipitated DNA protein sıyrıldı ve NGS (Şekil 2) için ChIP fragmanı kütüphane oluşturmadan önce (E) arıtılır. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.

Şekil 2. ChIP-Sıra kütüphane hazırlık, sıralama-sentez, haritalama ve pik arama. (A) 'elektroferogramıdır 250 bp 450 DNA şablonları ile iyi talaş-Dizi kütüphane gösterir. Bu şablonlar evrensel (58 bp) tarafından çevrili ilgi DNA insert ve dövize endeksli (63 bp) adaptörü gerektirir. (B) Milyonlarca, geri dönüşümlü, farklı floroforu ve aynı sonlandırma özelliklere sahip dört nükleotidin varlığında tabanı tarafından sıralı taban vardır. Floresan görüntüleri okur içine sonuçta monte karşılık gelen bazlar, aramak için gerçek zamanlı olarak işlenir. (C) Sadece benzersiz Ksenopus genomuna haritası tutulmasını okur. Bütün parçalar 5 'ucundan dizilenmiştir gibi, yonga eşleme ilgi kromatin özelliği kuşatan şerit spesifik pikler okur. Böylece, pik arayanlar doğru kromatin özelliklerini lokalize bir ortalama fragman uzunluğu okur immunoprecipitation kaynaklanan ve genişletmek zenginleştirme tespit. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.

Şekil 3. Zigotik T-box transkripsiyon faktörü vegt (zVegT). Hepsi burada gösterilen sayıları 10 milyona normalize olan okuma benzersiz haritalanmış ve olmayan gereksiz okur. (A) alıntı vasıtasıyla sonrası dizi analizi ve veri görselleştirme örneği zVegT genom profil X. bağlanma (Nieuwkoop ve Faber 29 sonra 12.5 sahne 11) Tropicalis gastrula embriyolar. Her tepe, genişletilmiş okur yığılma, tek bir bağlanma yeri temsil eder. Bu doruklarına% 1'den daha az bir yanlış keşif oranı (FDR) ile MACS2 tarafından denir. Her MESP gen bağlanma oldukça yakın ve üst baş zVegT gösterir, ancak MESPA ve mespb Bu aşamada (RNA'ya Dizi verileri 30) ile ifade edilir. (B) 'zVegT DNA doluluk seviyeleri birkaç loci talaş-qPCR tarafından (bir non içeren kb yukarısında β aktin arasında TT-bağlı bölgesi 0.5) konfiChIP-Sıra tarafından bulunan özel zenginleştirme rm. (A) (kırmızı çubuk) olarak adlandırılan tepe MESPA sonuçları karşılaştırın. DNA doluluk seviyesi hem de giriş yüzdesi, vegt antikor (IgG izotipinin tavşan poliklonal) ve antikor kontrolü (normal tavşan IgG) ile ChIP ile ChIP olarak görüntülenmiştir. Hata çubukları iki biyolojik çoğaltır standart sapmasını yansıtır. (C) Metagene analizi etrafında başka genomik bölge ve gen organları içinde promotör göreceli bağlama tercihli zVegT (etiketler 25 bp üzerinde binned) gösterir. (D) Heatmap k-ortalama gösterir kümelenmiş (k = 5), DNA doluluk seviyeleri gastrula aşamasında tüm zVegT bağlı bölgelere zVegT ve Smad2 / Smad3 (ChIP-Dizi veriler 31) göreceli (etiketler 25 bp üzerinde binned). heatmap De novo motif analizi zVegT-% 38'inde motifi bağlayıcı kanonik T-box transkripsiyon faktörü keşfeder 2 tabanlı ve bp başına 5 etiketleri merkezli. (E) logaltta yatan motifi mı arka dizileri,% 5 bulma oranı normalize ise, bağlı bölgeleri. kanonik Smad2 / Smad3 bağlayıcı motifi pek zenginleştirilmiş ise yoğunluk haritası, zVegT bağlanma sitelerin merkezinde T-box motifi için en yüksek zenginleştirme gösterir. (F) Histogram, her hedef genin hesaplanır zVegT DNA doluluk seviyelerini gösterir [-] tüm zirveleri (+/- 200 bp) 5 kb yukarı arasından. transkripsiyon başlangıç siteleri gelen ve 1 kb mansap [+] (G) yüksek DNA doluluk içinde -5 kb düzeyleri ve 1 kb with Top 300 genlerin Erken embriyonik gelişim biyolojik süreçleri için zenginleştirilmiş. Bu terimler zVegT varsayımsal fonksiyonu ile uyumludur GO. FDR iki kuyruklu Fisher testine göre ve çoklu test için düzeltilir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
Tartışmalar
Bizim protokol yapmak ve Xenopus embriyolar genom kromatin profillerini analiz nasıl özetliyor. Bu silico zenginleştirilmiş genomik siteleri temsil okur milyonlarca işlem, in vivo olarak, endojen mahallere çapraz bağlama proteinlerinden her adımı kapsar. Genom taslaklarının artan sayıda mevcut olduğundan, bu protokol diğer model olan ve olmayan model organizmalar için geçerli olmalıdır. Önceki çalışma 8,31,33,34 dışında bu protokol belirler en önemli deney bölümünde, çapraz-bağlanmış çekirdekleri elde etmek sonrası tespit işlemdir. Verimli kromatin çözünmesini ve kesme ve kolay upscaling kolaylaştırır. Birlikte kütüphane hazırlık geliştirilmiş verimlilik ile bu protokol yarısından itibaren faiz kromatin-ilişkili epitopu ifade iki milyona hücrelerine yüksek karmaşıklık ChIP-Seq kütüphaneler yapıyı sağlar. ChIP-QPCR deneyler için, bu hücrelerin birkaç on bin normal olarak yeterlibelki altı farklı genomik DNA loci zenginleştirme kontrol etmek için. Bu numaralar tahminlerdir, ancak protein ifadesi seviyesi, antikor kalitesine bağlı olarak etkinliği ve epitop ulaşmak çapraz bağlama olarak değişiklik gösterebilir. Bir rehber olarak, tek bir Xenopus embriyo orta Blastula aşama (Nieuwkoop ve Faber 29 den sonra 8.5) yaklaşık 4000 hücreleri içeren geç gastrula aşama (12) de 40,000 hücre ve erken tailbud aşama (20) de 100.000 hücre.
etkin immüno-çökeltme için kesin sabitleme süresi ChIP-qPCR (bölüm 10) tarafından empirik olarak tespit edilmesi gerekmektedir. Deney X içeriyorsa Genel olarak, uzun tespit süreleri gereklidir embriyolar, erken gelişim aşamalarını ve zayıf (ya da dolaylı) DNA bağlama özelliklerini laevis. Bununla birlikte, kromatin kesme daha az etkili hale gelir, 40 dakikadan daha uzun bir Xenopus embriyolar sabitleme ya da belirtilmedikçe, (bölüm 3) daha fazla embriyolar işlem tavsiye edilmez. Bu önemli değildirçok zor sarısı zengin embriyolardan nükleer çıkarma yapabilir formaldehit söndürülmesi için bu ortak adım olarak tespit sonrasında herhangi bir glisin kullanmak için. Şu anda, bunun nedeni bilinmemektedir. Formaldehit-glisin ilave maddesi, ayrıca aşağıdaki amino grupları ya da arginin artığını 35 N-terminali ile reaksiyona düşünülebilir.
Antikor herhangi bir çip deney anahtarıdır ve yeteri kadar kontrol (Landt ve ark. 36 ile kılavuza bakınız) ilgi epitopu için spesifikliğini göstermek amacıyla gerekmektedir. Hiçbir ChIP dereceli antikor varsa bu proteinler işgal endojen bağlayıcı siteleri 37 gibi, epitop füzyon proteinleri karşılık gelen giriş meşru bir alternatif olabilir. Bu durumda, enjeksiyon yapılmamış embriyolar bir negatif kontrol yerine non-spesifik serum ile bir çip olarak kullanmak en iyisidir. İlgi dahilindeki protein Enri zayıf kazanımı ile sonuçlanan, düşük seviyelerde ifade edilir, bu strateji de tatbik edilebilirched DNA.
Çünkü kullanımda DNA düşük miktarda ChIP-Seq kütüphaneler, yapım gelince, bu temizlik adımların sayısını azaltmak ve en azından DNA kaybı tutmak tepkileri birleştirmek işlemleri için tercih edilmesi önerilir. adaptörleri ve primerler (Belirli Malzemeleri / Ekipman Tabloya bakınız) multipleks sıralama ve NGS platformu ile uyumlu olması gerekir. (Uzun tek sarmallı silah içeren) Y-adaptörler kullanarak, bu boyut seçerek DNA ekler önce PCR 3-5 mermi ile ön yükseltmek kütüphane için kritik (örn., 100 bp 300) jel elektroforezi ile. Tek iplikli uçları, DNA fragmanları, heterojen geçirilecek neden olur. Deneme PCR döngüsü sayısını belirlemek için tavsiye edilir giriş DNA (örneğin, 0.1, 0.5, 1, 2, 5, 10 ve 20 ng) farklı miktarlarda (daha az veya 18 döngü eşit) bir boyuta getirmek için gerekli olan 100 ila 200 ng -seçilmiş kütüphanesi. PCR döngü sayısını azaltmak Redu sekanslanmasını vermektedirndant daha az olasıdır okur. Katı faz geri dönüşümlü immobilizasyon boncuklar verimli ilgi DNA kurtarmak ve güvenilir ligasyon ve PCR reaksiyonları herhangi bir serbest adaptörleri ve dimerlerini kaldırmak için reaktifler temizlik iyi.
Sayı, tip ve uzunluk açısından civarında 20-30.000.000 tek uç 36 bp yeterli derinliğe sahip tüm Xenopus genomunu kapsayacak şekilde en ChIP-Seq deneyler için yeterli okur, okur. En yaygın NGS makineleri bu kriterleri karşılama rutin yeteneğine sahiptir. Bununla birlikte, okuma geniş bir dağılımı beklenen ise okur oldukça keskin yükselme daha histon modifikasyonları ile gözlemlendiği üzere, sayısını artırmak için yararlı olabilir. Birçok ChIP-Dizi deneyler için, 4-5 farklı dizin kütüphaneleri birleştirilebiliyor ve yüksek performanslı NGS makinesi kullanılarak bir akış hücresi şeritte dizilenmiştir. Bazen w mappability artırmak için okuma uzunluğu ve diziyi DNA örneğinin iki ucu (paired-end) genişletmek için de tavsiye edilirtekrarlayan genomik bölgelerde kromatin tavuk analiz.
Bu protokol, kopyalama faktörleri, bu sinyal aracı ve post-translasyonel modifikasyonları histon olarak kromatin özelliğin çok çeşitli başarı ile uygulanmıştır. Onlar geliştirmek ve kromatin profilleri yorumlamak zor hale Ancak, embriyolar hücresel heterojenite artan derecesi elde. Ümitli adımlar hücre tipine özgü çekirdek 38,39 ayıklanması doku-spesifik profil kromatin manzara Arabidopsis ve Drosophila yapılmıştır. Bizim protokol, diğer embriyolar doku-spesifik ChIP-Sek için önünü açabilecek bir nükleer çıkarma adımı içerir.
Açıklamalar
The authors declare that they have no competing financial interests.
Teşekkürler
We thank Chris Benner for implementing the X. tropicalis genome (xenTro2, xenTro2r) into HOMER and the Gilchrist lab for discussions on post-sequencing analysis. I.P. assisted the GO term analysis. G.E.G and J.C.S. were supported by the Wellcome Trust and are now supported by the Medical Research Council (program number U117597140).
Malzemeler
| Name | Company | Catalog Number | Comments |
| 1/16 inch tapered microtip | Qsonica | 4417 | This microtip is compatible with Sonicator 3000 from Misonix and Q500/700 from Qsonica. |
| 8 ml glass sample vial with cap | Wheaton | 224884 | 8 ml clear glass sample vials for aqueous samples with 15-425 size phenolic rubber-lined screw caps. |
| Adaptor | e.g., IDT or Sigma | NA | TruSeq universal adaptor,
AATGATACGGCGACCACCGAG ATCTACACTCTTTCCCTACAC GACGCTCTTCCGATC*T. TruSeq indexed adaptor, P-GATCGGAAGAGCACACGTC TGAACTCCAGTCAC ‐NNNNNN‐ ATCTCGTATGCCGTCT TCTGCTT*G. *, phosphorothioate bondphosphate group at 5' end. NNNNNN, index (see TruSeq ChIP Sample Preparation Guide for DNA sequence). Order adaptors HPLC purified. Adaptors can be prepared by combining equimolar amounts (each 100 µM) of the universal and the indexed adaptor and cooling them down slowly from 95 °C to room temperature. Use 1.5 pmol per ng of input DNA. Store at -20 °C. |
| b2g4pipe (software) | Blast2GO | non-commercial | http://www.blast2go.com/data/blast2go/b2g4pipe_v2.5.zip |
| BLAST+ (software) | Camacho et al. | non-commercial | http://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE=BlastDocs& DOC_TYPE=Download |
| Bowtie (software) | Langmead et al. | non-commercial | http://bowtie-bio.sourceforge.net/index.shtml |
| cisFinder (software) | Sharov et al. | non-commercial | http://lgsun.grc.nia.nih.gov/CisFinder/ |
| Chip for capillary electrophoresis | Agilent Technologies | 5067-1504 | Load this chip with 1 µl DNA for library quality control. Store at 4 °C. |
| Chip-based capillary electrophoresis system | Agilent Technologies | G2940CA | The Agilent 2100 BioAnalyzer is used to check the quality of ChIP-Seq libraries. Keep reagents at 4 °C. |
| ChIP-Seq library preparation kit (KAPA Hyper Prep Kit) | Kapa Biosystems | KK8504 | Kit contains KAPA end repair and A-tailing enzyme mix, end Repair and A-tailing buffer, DNA ligase, ligation buffer, KAPA HiFi HotStart ReadyMix (2X), and KAPA library amplification primer mix (10X) (see also PCR primers). Adaptors are not included. Store at -20 °C. |
| ChIP-Seq library preparation kit (alternative, ThruPLEX-FD Prep Kit) | Rubicon Genomics | R40048 | Kit uses their own stem-loop adaptors and primers. This kit eliminates intermediate purification steps and is as sensitive as the KAPA Hyper Prep Kit. Store at -20 °C. |
| Cluster3 (software) | de Hoon et al. | non-commercial | http://bonsai.hgc.jp/~mdehoon/software/cluster |
| FastQC (software) | Simon Andrews | non-commercial | http://www.bioinformatics.babraham.ac.uk/projects/fastqc |
| Fluorometer | life technologies | Q32866 | Qubit 2.0 Fluorometer |
| Fluorometer reagents | life technologies | Q32851 | The kit provides concentrated assay reagent, dilution buffer, and pre-diluted DNA standards for the Qubit fluorometer. Store DNA standards at 4 °C, buffer and dye at room temperature. |
| Formaldehyde | Sigma | F8775-4X25ML | Formaldehyde solution, for molecular biology, 36.5-38% in H2O, stabilised with 10-15% methanol. Store at room temperature. CAUTION: Formaldehyde is corrosive and highly toxic. |
| Gel (E-Gel EX agarose , 2%) | life technologies | G4010 | Pre-cast gel with 11 wells, openable format. Leave one lane between ladder and library empty to avoid cross-contamination. Store gels at room temperature. |
| Gel electrophoresis system | life technologies | G6465 | E-Gel iBase and E-Gel Safe Imager combo kit for size-selecting ChIP-Seq libraries. |
| Gel extraction kit | Qiagen | 28706 | Store all reagents at room temperature. Use 500 µl of QG buffer per 100 mg of 2% agarose gel slice to extract DNA. Use MinElute columns (from MinElute PCR purification kit) to elute DNA twice. |
| HOMER (software) | Chris Benner | non-commercial | http://homer.salk.edu/homer/index.html |
| Hybridization oven | Techne | FHB1D | Hybridizer HB-1D |
| IGV (software) | Robinson et al. | non-commercial | http://www.broadinstitute.org/igv/home |
| Illumina CASAVA-1.8 quality filter (software) | Assaf Gordon | non-commercial | http://cancan.cshl.edu/labmembers/gordon/fastq_illumina_filter |
| Java TreeView (software) | Alok Saldanha | non-commercial | http://jtreeview.sourceforge.net |
| Laboratory jack | Edu-Lab | CH0642 | This jack is used to elevate sample in sound enclosure for sonication. |
| Ladder, 100 bp | New England BioLabs | N3231 | Keep 1x solution at room temperature. Store stock at -20 °C. |
| Ladder, 1 kb | New England BioLabs | N3232 | Keep 1x solution at room temperature. Store stock at -20 °C. |
| Low-retention 1.5-ml microcentrifuge tubes | life technologies | AM12450 | nonstick, RNase-free microfuge tubes, 1.5 ml |
| MACS2 (software) | Tao Liu | non-commercial | https://github.com/taoliu/MACS |
| Magnetic beads | life technologies | 11201D | These Dynabeads are superparamagnetic beads with affinity purified polyclonal sheep anti-mouse IgG covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 11203D | These Dynabeads are superparamagnetic beads with affinity purified polyclonal sheep anti-rabbit IgG covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 10001D | These Dynabeads are superparamagnetic beads with recombinant protein A covalently bound to the bead surface. Store at 4 °C. |
| Magnetic beads | life technologies | 10003D | These Dynabeads are superparamagnetic beads with recombinant protein G covalently bound to the bead surface. Store at 4 °C. |
| Magnetic rack | life technologies | 12321D | DynaMag-2 magnet |
| MEME | Bailey et al. | non-commercial | http://meme.nbcr.net/meme/ |
| Na3VO4 | New England BioLabs | P0758 | Sodium orthovanadate (100 mM) is a commonly used general inhibitor for protein phosphotyrosyl phosphatases. Store at -20 °C. |
| NaF | New England BioLabs | P0759 | Sodium fluoride (500 mM) is commonly used as general inhibitor of phosphoseryl and phosphothreonyl phosphatases. Store at -20 °C. |
| NGS machine | Illumina | SY-301-1301 | Genome Analyzer IIx |
| NGS machine (high performance) | Illumina | SY-401-2501 | HiSeq |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2028 | Use as control for goat polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2025 | Use as control for mouse polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Normal serum (antibody control) | Santa Cruz Biotechnology | sc-2027 | Use as control for rabbit polyclonal IgG antibodies in ChIP-qPCR experiments. Store at 4 °C. |
| Nucleic acid staining solution | iNtRON | 21141 | Use RedSafe nucleic acid staining solution at 1:50,000. Store at room temperature. |
| Orange G | Sigma | O3756-25G | 1-Phenylazo-2-naphthol-6,8-disulfonic acid disodium salt. Store at 4 °C. |
| PCR primers | e.g., IDT or Sigma | Primers to enrich adaptor-ligated DNA fragments by PCR: AATGATACGGCGACCACCGA*G and CAAGCAGAAGACGGCATACGA*G, phosphorothioate bond. Primers designed by Ethan Ford. Combine primers at 5 µM each. Use 5 µl in a 50 µl PCR reaction. Store at -20 °C. | |
| MinElute PCR purification kit | Qiagen | 28006 | for purification of ChIP-qPCR and shearing test samples. Store MinElute spin columns at 4 °C, all other buffers and collection tubes at room temperature. |
| Phenol:chloroform:isoamyl alcohol (25:24:1, pH 7.9) | life technologies | AM9730 | Phenol:Chloroform:IAA (25:24:1) is premixed and supplied at pH 6.6. Use provided Tris alkaline buffer to raise pH to 7.9. Store at 4 °C. CAUTION: phenol:chloroform:isoamyl alcohol is corrosive, highly toxic and combustible. |
| Primer3 (software) | Steve Rozen & Helen Skaletsky | non-commercial | http://biotools.umassmed.edu/bioapps/primer3_www.cgi |
| Protease inhibitor tablets | Roche | 11836170001 | cOmplete, Mini, EDTA-free. Use 1 tablet per 10 ml. Store at 4 °C. |
| Protease inhibitor tablets | Roche | 11873580001 | cOmplete, EDTA-free. Use 1 tablet per 50 ml. Store at 4 °C. |
| Proteinase K | life technologies | AM2548 | proteinase K solution (20 µg/µl). Store at -20 °C. |
| RNase A | life technologies | 12091-039 | RNase A (20 µg/µl). Store at room temperature. |
| Rotator | Stuart | SB3 | Rotator SB3 |
| SAMtools (software) | Li et al. | non-commercial | http://samtools.sourceforge.neta |
| Solid phase reversible immobilisation beads | Beckman Coulter | A63882 | The Agencourt AMPure XP beads are used to minimise adaptor dimer contamination in ChIP-Seq libraries. Store at 4 °C. |
| Sonicator 3000 | Misonix/Qsonica | Newer models are now available. Q125, Q500 or Q700 are all suitable for shearing crosslinked chromatin. | |
| Sound enclosure | Misonix/Qsonica | optional: follow the manufacturer's recommendation to obtain the correct sound enclosure. | |
| Thermomixer | eppendorf | 22670000 | Thermomixer for 24 x 1.5 mL tubes. Precise temperature control from 4 °C above room temperature to 99 °C. |
Referanslar
- Gilmour, D. S., Lis, J. T. Detecting protein-DNA interactions in vivo: distribution of RNA polymerase on specific bacterial genes. Proc Natl Acad Sci U S A. 81 (14), 4275-4279 (1984).
- Gilmour, D. S., Lis, J. T. In vivo interactions of RNA polymerase II with genes of Drosophila melanogaster. Mol Cell Biol. 5 (8), 2009-2018 (1985).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Ren, B., et al. Genome-wide location and function of DNA binding proteins. Science. 290 (5500), 2306-2309 (2000).
- Johnson, D., Mortazavi, A., Myers, R., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Hellsten, U., et al. The genome of the Western clawed frog Xenopus tropicalis. Science. 328 (5978), 633-636 (2010).
- Lee, T. I., Johnstone, S. E., Young, R. A. Chromatin immunoprecipitation and microarray-based analysis of protein location. Nature Protocols. 1 (2), 729-748 (2006).
- Blythe, S. A., Reid, C. D., Kessler, D. S., Klein, P. S. Chromatin immunoprecipitation in early Xenopus laevis embryos. Dev Dyn. 238 (6), 1422-1432 (2009).
- Gentsch, G. E., Smith, J. C. Investigating physical chromatin associations across the Xenopus genome by chromatin immunoprecipitation. Cold Spring Harb Protoc. 2014 (5), (2014).
- Ubbels, G. A., Hara, K., Koster, C. H., Kirschner, M. W. Evidence for a functional role of the cytoskeleton in determination of the dorsoventral axis in Xenopus laevis eggs. J Embryol Exp Morphol. 77, 15-37 (1983).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Heinz, S., et al. Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities. Mol Cell. 38 (4), 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9 (9), R137 (2008).
- Robinson, J. T., et al. Integrative genomics viewer. Nat Biotechnol. 29 (1), 24-26 (2011).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Brief Bioinform. 14 (2), 178-192 (2013).
- Imoto, S., Nolan, J., Bioinformatics Miyano, S. . 20 (9), 1453-1454 (2004).
- Camacho, C., et al. BLAST+: architecture and applications. BMC Bioinformatics. 10, 421 (2009).
- Conesa, A., et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research. Bioinformatics. 21 (18), 3674-3676 (2005).
- Sive, H., Grainger, R., Harland, R. . Early development of Xenopus laevis: A laboratory manual. , (2000).
- Chen, Y., et al. Systematic evaluation of factors influencing ChIP-seq fidelity. Nat Methods. 9 (6), 609-614 (2012).
- Sharov, A. A., Ko, M. S. H. Exhaustive search for over-represented DNA sequence motifs with CisFinder. DNA Res. 16 (5), 261-273 (2009).
- Bailey, T. L., et al. MEME SUITE: tools for motif discovery and searching. Nucl Acids Res. 37 (2), W202-W208 (2009).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10 (10), 669-680 (2009).
- Gentsch, G. E., et al. In vivo T-box transcription factor profiling reveals joint regulation of embryonic neuromesodermal bipotency. Cell Rep. 4 (6), 1185-1196 (2013).
- Barabasi, A. L., Oltvai, Z. N. Network biology: understanding the cell's functional organization. Nat Rev Genet. 5 (2), 101-113 (2004).
- Biggin, M. D. Animal transcription networks as highly connected, quantitative continua. Dev Cell. 21 (4), 611-626 (2011).
- Nieuwkoop, P. D., Faber, J. . Normal table of Xenopus laevis (Daudin): a systematical and chronological survey of the development from the fertilized egg till the end of metamorphosis. , (1994).
- Akkers, R. C., et al. A hierarchy of H3K4me3 and H3K27me3 acquisition in spatial gene regulation in Xenopus embryos. Dev Cell. 17 (3), 425-434 (2009).
- Yoon, S. J., Wills, A. E., Chuong, E., Gupta, R., Baker, J. C. . HEB and E2A function as SMAD/FOXH1 cofactors. Genes Dev. 25 (15), 1654-1661 (2011).
- Jallow, Z., Jacobi, U. G., Weeks, D. L., Dawid, I. B., Veenstra, G. J. Specialized and redundant roles of TBP and a vertebrate-specific TBP paralog in embryonic gene regulation in Xenopus. Proc Natl Acad Sci U S A. 101 (37), 13525 (2004).
- Buchholz, D. R., Paul, B. D., Shi, Y. -. B. Gene-specific changes in promoter occupancy by thyroid hormone receptor during frog metamorphosis. Implications for developmental gene regulation. J Biol Chem. 280 (50), 41222-41228 (2005).
- Wills, A. E., Guptaa, R., Chuonga, E., Baker, J. C. Chromatin immunoprecipitation and deep sequencing in Xenopus tropicalis and Xenopus laevis. Methods. 66 (3), 410-421 (2014).
- Metz, B., et al. Identification of formaldehyde-induced modifications in proteins: reactions with model peptides. J Biol Chem. 279 (8), 6235-6243 (2004).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Res. 22 (9), 1813-1831 (2012).
- Mazzoni, E. O., et al. Embryonic stem cell-based mapping of developmental transcriptional programs. Nat Methods. 8 (12), 1056-1058 (2011).
- Deal, R. B., Henikoff, S. A simple method for gene expression and chromatin profiling of individual cell types within a tissue. Dev Cell. 18 (6), 1030-1040 (2010).
- Bonn, S., et al. Tissue-specific analysis of chromatin state identifies temporal signatures of enhancer activity during embryonic development. Nat Genet. 44 (2), 148-156 (2012).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır