Method Article
Genom düzenlenmiş İnsan Pluripotent Kök Hücre Hatları oluşturulması: İzolasyon hedefleyen Gönderen
Bu Makalede
Özet
Genome editing of human pluripotent stem cells (hPSCs) can be done quickly and efficiently. Presented here is a robust experimental procedure to genetically engineer hPSCs as exemplified by editing the AAVS1 safe harbor locus to express EGFP and introduce antibiotic resistance.
Özet
Insan pluripotent kök hücreler (hPSCs) Genomu düzenleme insani gelişmeyi anlamak ve hastalığın patofizyolojisi araştırmak için hangi genetik olarak kontrol ve klinik olarak anlamlı bir platform sağlar. Genom düzenleme için siteye özgü nükleazlardır (Sosyal Güvenlik Numaralarını) kullanılarak, bir başka izogenik ortamda belirli genetik değişikliklerin barındıran yeni hPSC hatlarının hızlı türetme mümkün olur. Çinko parmak nükleazlar (ZFNs), transkripsiyon aktivatörü gibi efektör nükleazlar (Talens) ve kümelenmiş düzenli interspaced kısa palindromic tekrarlar (CRISPR) / Cas9 en sık kullanılan SSNs vardır. Bu nükleazlara her böylece bir genomik lokusta kesin gen düzenleme teşvik, belirli bir sahada iki iplikçikli bir DNA kırılmasının katılmasıyla işlev görürler. SSN-meditasyon genom düzenleme ekleme / silme mutasyonlar veya alt tanıtmak ya hücrenin endojen DNA onarım mekanizmaları, homolog olmayan uç (NHEJ) katılmadan ve homoloji yönettiği onarım iki (HDR), sömürençift sarmallı mola yerinde bir homolog onarım şablonu kullanarak genom er. HPSCs Elektroporasyon gibi floresan gazetecilere ve antibiyotik direnç kasetleri olarak transgenlerin dahil Sosyal Güvenlik Numaralarını ve onarım şablonları transfecting etkin bir yoludur. Elektroporasyondan sonra, antibiyotik direnç seçilerek, tamir yapısı dahil yalnızca hPSCs izole etmek mümkündür. Mekanik hPSC koloniler alınır ve genotipleme boyunca hedef yerde uygun bir entegrasyon teyit doğru hedef ve genetik olarak homojen bir hücre hatlarının izolasyonu sağlar. Bu protokolün geçerliliği EGFP ve puromycin direnç insan pluripotent kök hücreleri AAVS1 güvenli liman mahaline içine inşa dahil her üç SSN platformları kullanarak burada gösterilmektedir.
Giriş
Genom düzenleme teknolojileri hızla moleküler ve hücre biyolojisi 1 için standart araçları haline gelişmektedir. HPSCs genetik olarak sağlam primer insan hücreleri, bir kendi kendini yenileyen kaynağı temsil insan pluripotent kök hücreler (hPSCs) genetik mühendisliği, özellikle ilgi çekicidir. hPSCs hastalığı modelleme veya transplantasyon terapisi 2,3 için bir kaynak gibi çeşitli hücre tiplerine ayırt edilebilir. AAVS1 mahalinde bir raportör yapısının hedeflenen entegrasyonu için endojen DNA tamir mekanizmaları ile birlikte siteye özgü nükleazlardır (SSNs) üç farklı tipte kullanan bir protokoldür burada gösterdi. HPSCs içine SSNs transfeksiyonu sonra, muhabir barındıran izojenik hücre popülasyonlarının izole etmek için nasıl gösterilmektedir.
Özellikle pluripotent hücre genomları kök, genom manipüle etme yeteneği kullanarak SSNs çinko parmak nükleazlardır (ZFNs) ve transkripsiyon activat yararı olarak yeni bir fenomen değil,gen düzenleme için veya benzeri bir efektör nükleazlar (Talens) birkaç yıl önce 4-10 gösterilmiştir. Ancak, S. pyogenes CRISPR gelişiyle / Cas9 teknolojisiyle 11-13 ile gen düzenleme 14 yaygın olarak erişilebilir hale gelmiştir. Tüm SSNs homolog olmayan uç birleştirme (NHEJ) ya da homoloji yönettiği onarım (HDR) 15 kullanılarak endojen hücresel mekanizmalar tarafından tamir edilir belirtilen hedef sitenin 1,4,5,11 bir çift sarmallı DNA kırılmasını (DSB) tanıtmak. NHEJ hata eğilimli ve yeni elemanlar SSN sahip bir tamir şablonunun ko-transfeksiyon yoluyla uygulanabilir için HDR izin verirken, gen fonksiyonunun kaybıyla sonuçlanan çerçeve kayma mutasyonları neden olabilir. Gen düzenleme kolaylaştırmak DNA tamiri altında yatan ilkeler, her SSN büyük ölçüde aynı olduğu düşünülen edilirken, platformlar arasında bazı farklılıklar dikkat edilebilir. De ZFNs ve novo tasarım esnekliği ve nukleaz optimizasyonu 16 alenen bununla birlikte kullanılmasını sağlarBireysel ZFNs tasarım mevcut montaj kütüphaneleri ve tarama araçları zaman alıcı olabilir. ZFN aracılı hedefleme için istenen lokus belirlendikten sonra, ZFN çiftleri online bir araç ZiFit 17 ile tasarlanabilir. Tasarımı sonra ZFNs modüler plasmid klonlama 18 birkaç tur ile monte edilebilir. Alternatif olarak, birçok ticari olarak temin edilebilen, önceden onaylanmış ZFNs 19 vardır. TALE nükleazlar aynı zamanda online araçları ve kamuya açık bileşenler 17,20 kullanılarak dizayn edilebilir. Örneğin, hızlı bir şekilde TALENS FLASH düzeneğinin 21 veya PCR bazlı bir hiyerarşik Altın Kapı düzeneği 22 kullanılarak yoluyla beş TACE tekrarlarının bloklarından monte edilebilir. SSN tasarımı ve CRISPR / Cas9 kullanarak inşaat hızının kolaylığı yaygın erişilebilir düzenleme aracı genom yaptık. Kılavuz RNA'lar çoklama tek yapı 14 ile birkaç loci hedef için CRISPR kısa kılavuz RNA aracılı hedefleme / Cas9 da izin verir. DizaynHedef mahaline proksimal gen düzenleme için Cas9 bir protospacer bitişik motif (S. için bir NGG trinucleotide Cas9 pyrogenes PAM) sadece kimlik gerektirir. Px330 plasmid 14 içine PAM 20 baz çifti 5 'e karşılık gelen bir oligonükleotid sokulmasıyla, yapı, bir klonlama adımında birleştirilebilir. S. ek olarak pyogenes Cas9, N arasından Cas9 5'-NNNNGATT-3 '(PAM) tanır meningitidis (NmCas9) hPSCs 23 verimli gen düzenleme için izin vermek için gösterilmiştir.
SSK tasarım kolaylığı farklılıklara ek olarak, her bir platform belirli özelliklere sahiptir. Örneğin, ZFNs ve TALENS Cas9 çok oluşturmak uçlu DSBs künt düşünülmektedir ise dört nükleotid 5 'çıkıntı 24 oluşturur Foki nükleaz etki kullanmaktadır. ZFNs, TALENS ve Cas9, açma-kapama hızı hedef DNA, protein stabilitesi açısından farklılık ve DNA tarama modu her biri cteorik düzenleme sonucu 1 küçük farklılıklara neden ould. Ileri çalışmalara tam bu farklılıkların sonuçlarını anlamak için gerekli olacak, biz burada üç platformlarda son derece sağlam ve kolaylıkla genetiği değiştirilmiş hPSCs oluşturmak için kullanılabilecek bir protokol açıklar.
Ne olursa olsun SSN seçim, elektroporasyon hPSCs 25 içine Sosyal Güvenlik Numaralarını ve benzerlik onarım şablonları transfect sağlam bir işlemdir. Antibiyotik direnci için seçimden sonra koloniler hayatta sayısı lokus spesifik parametreler ve düzenleme stratejisi (transgenik ekin ve seçim modu, örneğin boyutu) bağlıdır. Burada açıklanan protokol genellikle yaklaşık 150-400 tek hücreli türetilmiş koloniler halinde sonuçlanır.
Gen-düzenleme, bu protokolü kullanarak AAVS1 loküsündeki önce SSNs 4,5 etkinliğini göstermek için kullanılır olmuştur. AAV-CAGGS EGFP onarım şablonu bir gen tuzak str kullanırategy locus spesifik bir şekilde puromisin direnç kazandırmak için. Kısaca, tamir şablonu yükselticisiz puromisin direnç kasetinin üst kısmında bulunan bir bağlayıcı alıcı alanı ihtiva eder. AAVS1 lokusundaki PPP1R12C geninin ilk intronu içine doğru entegrasyon üzerine, direnç kaseti düzenlenmiş genin promoterinden ifade edilir. Bu özel AAVS1 tahlil sağlamlığı bize her SSN platformunun etkinliğini karşılaştırmak için izin verir.
Sosyal Güvenlik Numaralarını kullanarak Gen düzenleme bozabilir ve / veya teorik herhangi geni değiştirmek için yeteneği verilen güçlüdür. HPSCs bu stratejiyi uygulanması hPSCs daha sonra, nöronlar 26 gibi insan hücre tiplerinin bir çok ayrışmıştır edilebilir, çok yönlü ve çok 27 hepatositlerin ve 28 kardiyomiyositlerin. Buna ek olarak, bir hastada elde edilen uyarılmış pluripotent kök hücrelerin kullanımı, bir hastaya özgü genetik temelinde 29 onarım ya da bilinen hastalığa neden olan mutasyonlar devreye girmesine olanak , Hastalık mekanizmaları ve hastanın kendi hücrelerini kullanarak test 30 terapötikleri araştırmak için bir platform sağlar. Özetle, hPSCs gen düzenleme insan gelişimi ve hastalığın 31 temel biyoloji araştırmak için etkili ve çok yönlü bir yaklaşımdır.
Protokol
Bu yazıda anlatılan prosedürleri gözden ve UC Berkeley Kök Hücre Araştırma Gözetim Komitesi tarafından kabul edildi.
1. Düzenleme için Kök Hücreler hazırlayın
- Büyüme ve mitomisin C ile pasifleştirilmiş fare embriyonik fibroblast 2,4 x 10 6 hücre / levha ihtiva eden bir 6-yuvalı plaka kültür insan pluripotent kök hücreleri (hPSCs) (MEF) besleyiciler jelatin 32 üzerinde büyümüş. Oyuk (HESC ortamı) başına insan embriyonik kök hücre ortamının 3 ml hPSCs korumak ve 37 büyümesini % 3 O 05/02% CO2 ile ° C inkübatör.
Not: Bu protokolün başarısına için, düşük oksijenli bir inkübatör hPSCs korumak için gerekli değildir; Ancak bu kat ve hücre sayıları buna göre ayarlanması gerekir, böylece hPSCs yüksek O 2 ortamında hızlı çoğalan dikkat etmek önemlidir. Ayrıca, düşük oksijen muhafaza hPSCs kendiliğinden farklılaşma 33 alt fiyat sahip olduğuna dikkat edilmelidir.- TO 500 mi HESC ortam 380 ml DMEM / F12, 75 mi Fetal Sığır Serumu (FBS) kombine olmak, 25 mi nakavt Serum Replacement (KSR). Glutamin (1 mM nihai konsantrasyon), 5 ml 100x, esansiyel olmayan amino asitler, 100 birim / ml Penisilin-Streptomisin (P / S), bazik Fibroblast Gelişim Faktörü (bFGF) (4 ng / ml nihai konsantrasyon), ve 2- ekleme merkaptoetanol (5.5 uM nihai konsantrasyon).
- Medya (3 ml) tüm hacmini çıkarmadan bir cam pipet ve vakum kullanarak ortamı değiştirin. 3 ml ılık hESC medya serolojik bir pipet kullanarak değiştirin. HPSCs yaklaşık% 50 konfluent (Gün -1) kadar tekrarlayın medya her gün değiştirin.
- Bir gün (Gün -1) hedefleme önce, eski medya kaldırılması ve 10 uM Y-27632 ile desteklenmiş sıcak hESC medya ekleyerek, HESC ortamı değiştirmek.
- Ayrıca Gün -1 üzerine, DR4 farelerinin (2.4 x 10 6 hücre / plaka) 34 ilaç dirençli MEF besleyici hücreler iki 6-kuyu veya 10 cm plakalara birini hazırlamak.
Not: Genel olarak, 6 kuyu plakalar reklam vardıravantajdır üzerinde 10 cm'lik tabak, onlar daha çok medyayı ağırlayacak olarak. 6 kuyu plakaları da farklı kuyulardan farklı klonlar bağımsız olmasını sağlamak. Bununla birlikte, 10 cm plaka, bu işlem için uygun bir mikroskop bağlı olarak, daha kolay toplama sağlayacaktır.
2. Düzenleme Pluripotent Kök Hücreler
- 1.5 ml tüp içine CRISPR / Cas9 px330 kodlayan plazmid (Şekil 1) ZFN 1 ve 2, TALEN, 1 ve 2 ya da 15 ug 5 ug her bir pipetleme transfeksiyon çözelti hazırlayın. Pipet 30 de bu tüpün içine onarım plazmid mikrogram. Son olarak, tüpe pipet 1x Fosfat Tamponlu Tuz (PBS) 300 ul hacmi getirmek için.
Not: 300 ul transfeksiyon altında çözeltisinin hacmi tutmak için 300 ng / ul'lik bir konsantrasyona sahip en az (herhangi bir kit uygundur) bir Midiprep plazmidler hazırlayın. Bu plazmid düz hale getirmek üzere, ya da endoto kullanmak için, fenol / kloroform ekstraksiyonu gerçekleştirmek için gerekli değildirXin ücretsiz plazmid hazırlık kiti. - Cam bir pipet ve vakum kullanılarak hPSCs ortamı çıkarın. Her bir kuyunun içine Pipet 2 ml ılık 1x PBS hücrelerini yıkamak için.
- Hemen bir cam pipet ve vakum kullanılarak PBS çıkarın. Her bir gözdeki hücrelerin doğrudan 0,5 ml% 0.25 tripsin-EDTA çözeltisi ekleyin. Yaklaşık 10 dakika boyunca bir inkübatör (37 ° C /% 5 CO 2/3% O 2) yerleştirin. ya da besleyici tabaka plakasını kaldırın başlayana kadar.
- Tripsin reaksiyonu durdurmak için her bir oyuğa, 2 ml sıcak esWash ortamı (470 ml DMEM / F12, 25 mi FBS, 100 ünite / ml P / S) ekleyin.
- Bir yaprak gibi dökülmek bu besleyici hücreler olun. Tüm kuyuları birleştirerek, iyi bir tek 50 ml'lik konik tüp içine her içeriğini pipetle. 10 ml'lik serolojik pipet kullanarak triturate hücreleri. Bu besleyici tabaka kırmak için gerekli değildir; hPSCs dikkatlice toz haline getirilerek besleyici tabakanın ayrılırlar. Hücre süspansiyonunun yaklaşık 15 ml olmalıdır.
- VOLU getirmek için tüp esWash ortam 25 ml ilave edilirBeni en fazla 40 ml toplam. Büyük besleyici topakları 1-2 dakika boyunca tüpün dibinde yerleşmek için izin verin. Yeni 50 ml konik tüp içine bir serolojik pipet ve mevduat kullanarak tüpten (~ 38 ml) çıkarın.
- 190 x g'de 5 dakika aşağı doğru döndürün. Cam bir pipet ve vakum kullanılarak tüpten çıkarın. Hücre pelet rahatsız değil emin olun. PBS 1x 500 ul hücrelerin tekrar. Daha önce hazırlanan plazmid transfeksiyon solüsyonu ile birleştirin. Bu aşamada hücreleri saymak. Elektroporasyon başına 5-10 milyon hücre kullanın.
- , 4 mm'lik bir elektroporasyon küveti içine tüm 800 ul süspansiyon Pipet 3-5 dakika buz üzerine yerleştirin. 250 V, 500 iF, ∞ direnci ve 4 mm küvet boyutu: elektroporasyon aşağıdaki parametrelerle sistem üzerinde üstel programı kullanarak electroporate hücreleri. Elektroporasyondan sonra, 3 dakika boyunca geri buz üzerine küvet kullanır.
Not: elektroporasyon sistemi üzerinde elektroporasyon sabit süresine uyulmalıdır. Zaman sabitihücre sayısı ve DNA saflığı ile değişir. Listelenen koşulları ve gen üreteci II kullanırken Başarılı nakiller genellikle 10-14 msn arasında sabit bir zaman var. Zaman sabitleri bu değerlerden değişir daha düşük elektroporasyon verimleri oluşabilir. - Sıcak HESC Medya 10 uM Y-27632 ile desteklenmiş 18 ml electroporated hücreleri tekrar süspansiyon. Plaka 3 DR4 besleyici hücreler ile bir 6 yuvalı plaka her oyuğuna, bu, tek bir hücre süspansiyonunun mi. Inkübatör (37 ° C /% 5 CO 2 /% 3 O 2) geri dönün.
Pozitif Kolonilerin 3. Seçim
- Gün 2, bir cam pipet ve vakum kullanılarak tüm medyayı çıkarın. 10 uM Y-27632 ile takviye edilmiş, sıcak HESC ortamının 3 ml değiştirin. 3. Gün, cam pipet ve vakum kullanılarak tüm medyayı çıkarın. Y-27632 de dahil olmak üzere olmadan sıcak HESC ortamının 3 ml değiştirin. 4. Gün, cam pipet ve vakum kullanılarak tüm medyayı çıkarın. 3 ml selecti antibiyotik ile takviye edilmiş, sıcak HESC ortamı değiştirinüzerinde. Inkübatör (37 ° C /% 5 CO 2 /% 3 O 2) geri dönün.
Not: direnç kaseti bağlıdır kullanılan antibiyotik türü tamir şablonunda dahil. WIBR 3. hPSCs (NIH kayıt 0079), 0.5 ug / ml puromisin, 70 ug / ml G418 (genetisin) ve 35 ug / ml higromisin çalışırken başarılı bir şekilde kullanılmıştır. Seçimi için konsantrasyonlar yaklaşık bir hafta içinde, vahşi tipli hücreleri öldürmek için gereken antibiyotiğin asgari konsantrasyon kurarak ampirik olarak belirlenmelidir. - Gün 5-12, antibiyotikler ile desteklenmiş sıcak hESC medya ile eski Ortam her zaman yerine günlük ortamı değiştirmek.
Not: Hücre ölümü büyük miktarda bekleyin. Bireysel koloniler günde 8-10 civarında anlaşılacaktır. Antibiyotiksiz düzenli HESC ortam sürekli seçimi 12-14 gün sonra kullanılabilir. Hücre yoğunluğu yüksek ya da hücre ölümü yavaş ise, ortam asitleştirilmesi kaçınılmalı ve bu ortam VOLU arttırmak gerekli olabilirSeçimin ilk birkaç gün boyunca beni (4-5 ml).
4. Toplama Seçilen koloniler (Gün 12-14)
- Günde 12-14 koloniler gözlemleyin. Diseksiyon mikroskobu üzerinde almak ve onlar ayırt başlıyor hücreleri içermediğinden emin olmak için hazırız koloniler gözlemleyin. Yaklaşık boyutu 800-1,200 uM olmalıdır. Koloniler günde 12 önce bu büyüklüğe ulaşmak ise, daha sonra çekilmesi gerektiği önerilmektedir.
Not: Her hedefleme deney için, istenilen genotip izole sağlamak için gerektiği kadar koloniler seçin. Bir örnek olarak, AAV-CAGGS EGFP onarım bir şablonla AAVS1 düzenleme sürekli olarak sağlam bir entegrasyon gösterilen ve sadece 12-24 koloniler, yaklaşık 5-10 heterozigotik hedeflenen klonlar elde etmek üzere alınmayı gerektiren gelmiştir. Daha fazla koloni gerektirebilir diğer hedefleme deneyleri (Tablo 1) çekilecek. Doğru hedefleme olaylarının sıklığı gibi SS verimliliği gibi faktörlere bağlıdırN DSB, insert büyüklüğü ve seçim stratejisi tanıtmak. Doğru düzgün hedeflenmiş bir klon elde edilmesi için gereken koloni sayısını azaltarak, bir hedef bölgeye entegre olduğunda burada sunulan AAVS1 lokusu için gen trap yaklaşım durumunda, seçim marker yalnızca ifade edilir. - (Her koloni aldı doları / plaka 2.4 x 10 6 hücre, bir de kullanılır) Bir gün almadan önce MEF'ler 12-kuyu plakaları hazırlamak.
- Toplama gününde, bir cam pipet ve vakum kullanılarak 12 de MEF plakaları tüm medya kaldırmak ve 1 ml hESC medya ile değiştirin. Ayrıca toplama gününde, seçilmiş olacak 6 kuyu plakalar üzerinde hESC ortamı değiştirmek.
- Besleyici katmandaki tek tek kolonilerin mekanik ayrışma için cam pipetler çekin. Bir in-kaputu Bunsen beki üzerinde pipetler indirin ve cam dövülebilir kadar genişleme noktasında yumuşadı. Hızla alev kaldırmak ve pipet ayrı bir açısal noktası oluşturma çekin. Kırındar bir kanal bırakarak eğilmiş eksene yaklaşık 2 cm etmektedir. 1-2 saniye alev teşhir ederek kanalın sonuna lehçe.
Not: İsteğe bağlı, ancak bu kültürün klonalite azaltabilir, koloniler bir p20 pipet ucu almak, donuk ucu potansiyel sonraki avanta kirletici, ortama her koloninin hücreleri büyük miktarlarda çıkarmak olabilir. - P1000 filtre pipet alarak ve dar ucuna bir emme ampul takarak cihazı toplama birleştirin. Geniş sonunda çekti cam pipet yerleştirin.
- HPSCs bir 6-deney levhası, bir doku kültürü kaputu monte edilen bir diseksiyon mikroskobu aşamasında alınmayı yerleştirin. Toplama cihazının ampul sıkıştırın ve yavaşça ortama parçaları serbest bırakmak için özen tüketim ve 10-20 eşit büyüklükte parçalar halinde bireysel koloni kesti. Ampulü bırakarak pipet içine koloni kesilmiş hPSC parçalarını alın. Aktarım sırasında mümkün olduğunca az medyayı almaya çalışın.
- Transfer 12 kuyu MEF plaka tek bir kuyudan, şimdi kırık koloni bırakmadan doğrudan kuyuya tekrar ampulü sıkıştırarak bireysel koloni. Tek hücreden türeyen klonlann belirlenmesine olanak sağlamak için eşsiz bir Her çukurun etiketleyin. Cam pipet her zaman değişen gerektiği gibi birçok koloniler için tekrarlayın.
Not: Toplama koloniler öğrenmek için biraz zaman alabilir. Bu hedeflenen koloniler izole etmeye çalışırken önce bazı kontrol hücreleri üzerinde deneyci uygulaması tavsiye edilir. - Hafifçe her bir orta hücre birikimini önlemek için birinci plaka, sallanan kuluçka makinesi (37 ° C /% 3 O 05/02% CO2) 12 gözlü levha üzerinde döndürür.
- Bir sonraki gün, her takip eden gün bir cam pipet ve vakum kullanılarak ortamın hacmini çıkarın ve hücreleri, yaklaşık% 50 konfluent kadar ılık HESC ortamı 1,5 ml ile değiştirin (bu genellikle 10-12 gün sürer).
- 10-12 gün sonra, her bir iki koloni birini seçmek ve yeniden yeni 12-kuyu MEF plakaları transferpeating bir yineleme plaka üretmek için 4.1-4.8 adımları tekrarlayın.
- Orijinal MEF plakaların her kalan koloniler DNA (aşağı bakınız) ekstrakte edin.
- Cam bir pipet ve vakum kullanılarak tüm kuyulardan bulunan ortamı çıkarın. Iyi hücreleri yıkamak için her üzerine PBS 1x Pipet 1 ml. Cam pipet ve vakum kullanılarak PBS çıkarın. Pipet hücre parçalama tamponu 250 ul (H2O içinde nihai konsantrasyonlar: 10 mM Tris HCI, 5 mM EDTA,% 0.2 SDS, 200 mM NaCI, 0.08 mg / ml proteinaz-K) her üzerine yerleştirin. Inkübatör Yeri (37 ° C /% 3 O 2 /% 5 CO 2) O / N.
- Sonraki gün, pipet Her oyuğun içeriği bireysel 1.5 mi borular. Pipet Her tüpe izopropil alkol 250 ul DNA çökeltildi. Kuvvetlice tüpü çalkalayın. Beyaz bir çökelti görünür olmalıdır.
- Bir masa üstü santrifüjü içinde 13,000 rpm'de 3 dakika için her bir tüp dönerler. Spin aşağı sonra sıvı atık haline boşaltılarak süpernatant atın. Bir küçük DNA pelet altına sıkışmış kalmalıdırtüp. DNA pelet yıkamak için her bir tüp içine% 70 etanol içinde 250 ul Pipet. Kuvvetlice sallayın. Bir masa üstü santrifüjü içinde 13,000 rpm'de 3 dakika için her bir tüp dönerler.
- Aşağı iplik sonra, sıvı atıklar içine boşaltılarak süpernatant atın. Küçük bir DNA pelleti, tüpün altına takılıp kalması gerekir. Tüp sol herhangi bir sıvı yoktur yani süpernatant geri kalanını pipetle. 5-10 dakika masa üstü kuruması için açık tüp bırakın. Kurutma işleminden sonra, 250 ul TE tamponu içinde yeniden süspanse DNA. DNA, çözünmesi için 6 saat süre ile 37 ° C 'de yerleştirin.
- PCR optimize önceden veya güney leke strateji kullanarak her bir örnek Genotip.
Not:.. AAVS1 AAV-CAGGS EGFP onarım şablonu kullanan hedefleme için, Güney benek stratejisi ve arkadaşları, 2009 4 kapsamlı bir güney lekeleme protokolü Southern bulunabilir Hockemeyer bulunabilir 2006 35 arasında çoklu PCR genotipleme için. Aynı deney, astar ve koşullar olabilirTablo 2 36 bulunabilir. - Doğru hedeflenmiş olmayan kuyular atın ve her gün doğru hedeflenmiş hücreler üzerinde HESC medya değiştirerek devam edin. Bunların yaklaşık% 50 konfluent olduğunda, hücreleri aşağı Freeze protokolü dondurma için aşağıya bakınız.
- Bir gün dondurmadan önce, eski medya kaldırılması ve 10 uM Y-27632 ile desteklenmiş sıcak hESC medya ekleyerek, HESC ortamı değiştirmek.
- Donma günde iki 15 ml konik tüp içinde dondurulur olan bir 12 oyuklu plakanın her bir kuyucuğu çözelti A ve çözelti B, 0.5 ml, her hazırlayın. Buz üzerinde yerleştirin çözümleri. Çözelti A:% 50 hESC medya ve% 50 FBS; Çözelti B:% 80 FBS ve% 20 dimetil sülfoksit (DMSO).
- Dondurmadan önce tek hücre içine hPSC kolonileri ayırmak. Buna göre hacimleri ölçekleme, bunu yapmak için adımlar 2,2-2,8 izleyin. Çözelti A'nın 0.5 ml tam tekrar süspansiyon hücre topağı
- Hücre süspansiyonu, pipet up 0.5 ml solüsyon B ekleyin ve aşağı hücre süspansiyonu homojen hale getirmek için. Take 2 ml cryotube hücre süspansiyonu (~ 1 mi) ve yatağın toplam hacim. Kapağı sıkıca kapatılır.
- -80 ° C dondurucu O / N yerleştirin cryotube. Dondurma gün sonra, -80 ° C derin dondurucuda dondurulmuş hücreleri çıkarmak ve hemen uzun süreli depolama için sıvı azot tankına yerleştirin.
Sonuçlar
Burada Genetiği hPSC satırları oluşturmak için üç farklı SSN platformları ile uyumlu bir protokol göstermektedir. Biz EGFP muhabiri ve bir puromycin direnç kaset 4 tanıtan bir onarım şablonu kullanarak daha önce yayınlanmış ZFNs 4, Talens 5 ve CRISPR / Cas9s 37 kullanılarak AAVS1 loküsündeki WIBR 3. insan embriyonik kök hücrelerini hedef aldı.
Biz farklılaşmamış hPSCs bakım ve genişletme izin veren bir hücre kültürü iş akışı (Şekil 2) için MEF'ler bizim hPSCs kültürlü, o da etkili ve ölçeklenebilir maliyetidir. Hücreler gerekenden daha uzun büyüdüklerinde transfekte pluripotent hücrelerin sayısını ve dolayısıyla elde edilen doğru hedef hPSC koloni sayısını azaltarak, artan farklılaşma riski vardır.
Bu electropSSN platformu başına 5.0 x 10 6 hücre orated ve DR4 MEF'lerin tek 6 plaka üzerine her hedef hücreleri kaplı. Seçimden sonra, her platform EGFP-pozitif koloniler (Şekil 3) ve hedefsiz homozigotik ve heterozigotik olarak hedef hedef klonlar (; Tablo 3 Şekil 4A, B) bir kombinasyonu ile sonuçlanmıştır. Burada yer alan şartlar altında, AAVS1 TALENS en EGFP-pozitif klonlar neden olduğunu bulmak. Bu deney için kullanılan onarım şablonu EGFP raportör ve puromisin direnç kasetinin üst kısmında bulunan bir bağlayıcı alıcı alan oluşur. Bu "gen trap" stratejisi (Şekil 1) kullanılarak, yapı puromisin dirençli bir kasetin ekspresyonunu tahrik etmek için endojen promotör ile, AAVS1 lokusunun ilk intronu eklemek gerekir. Ekspresyonu engellemelidir onarım şablonu içinde puromisin direnç geni ekspresyonunu tahrik eden promoterden olmaması rastgele entegrasyon olay.
Bu nedenle, tüm puromycin dirençli klonlar AAVS1 sitesinde hedef olacağını bekliyoruz. Bu ancak en PCR stratejileri (Şekil 1, Şekil 4C) ile klonların bir alt kümesi, bir iç probu kullanılarak Southern blot ile tespit edilebilir AAVS1 lokusunda olarak anormal entegrasyonlar taşıdığına işaret edilmelidir 4. Bu entegrasyon olaylar büyük olasılıkla donör plazmid 4 çoklu entegrasyonlar yol açan heterolog hedef olayların sonucudur. Her SSN deneyinden 24 koloniler aldı ve tüm platformlar çok yüksek hedefleme verimliliği vardı ve sadece çok az farklılıklar gösterdiğini gördük. PCR tarafından sorguya gibi TALEN platformu en homozigotik hedeflenen klonlar (Tablo 3) varken, CRISPR / Cas9 en doğru hedeflenmiş klonları elde edilmiştir.
/files/ftp_upload/53583/53583fig1highres.jpg "width =" 700 "/>
Gen Şekil 1. şematik AAV-CAGGS EGFP onarım şablonu. Dan Hockemeyer ve ark. Modifiye kullanarak AAVS1 lokusunu düzenlenmiş, 2009. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
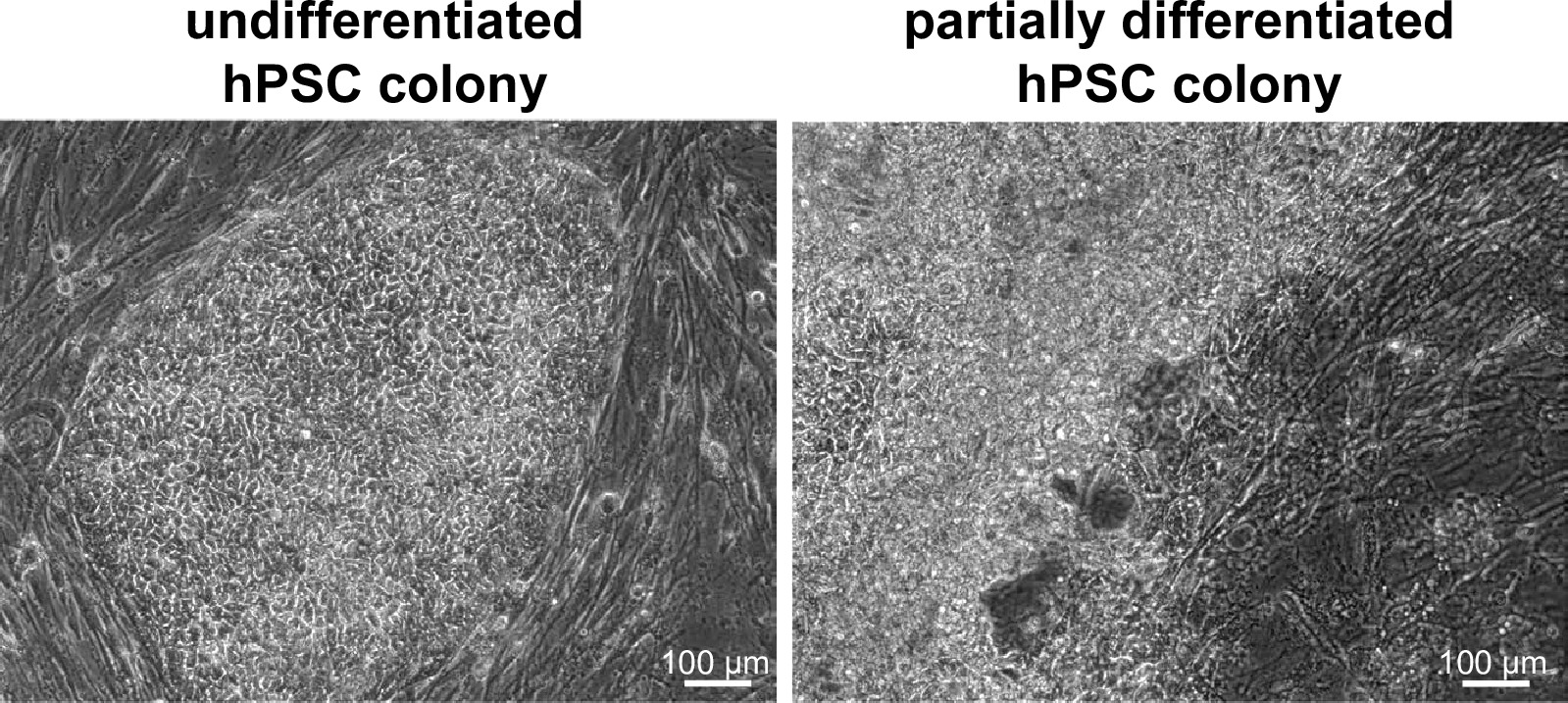
WIBR 3. hücrelerinin Şekil 2. kolonileri. 3 insan embriyonik kök hücreleri önce hedeflemesine WIBR # kolonileri Temsilcisi parlak bir alan görüntüler. Farklılaşma eksikliği ve ideal olmayan bir (sağ) farklı olarak ideal bir kolonisi (solda) besleyici tabakadan net bir ayrım unutmayın. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
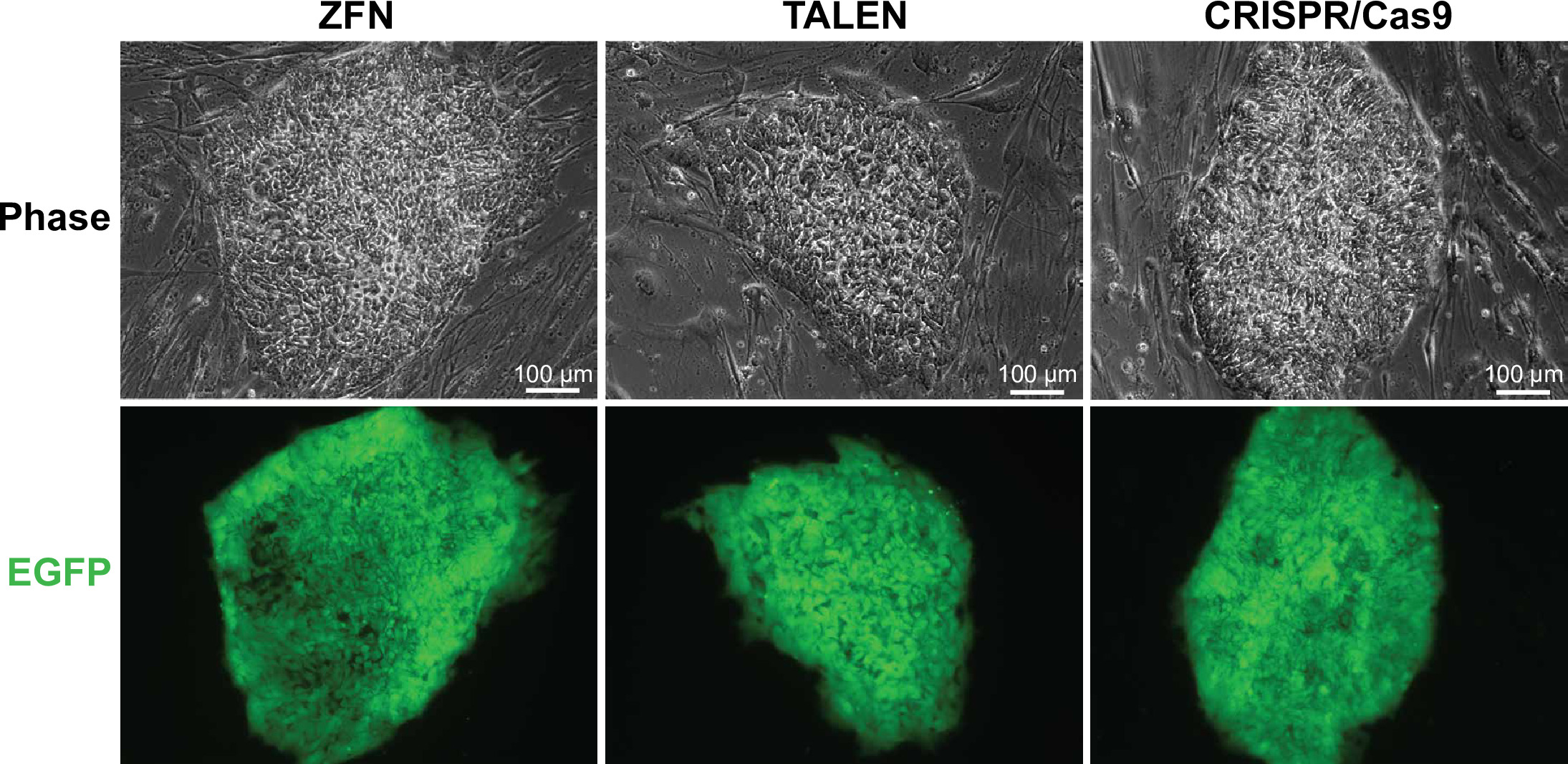
Şekil 3. EGFP-pozitif WIBR 3. hücreleri. WIBR # AAVS1 mahalinde bir EGFP-ifade onarım şablonu ile hedeflenen 3 hücrelerinin Temsilcisi görüntüler. ZFNs düzenlenmiş temsilci kolonilerin Görüntüler, Talens ve CRISPR / Cas9 gösterilmiştir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
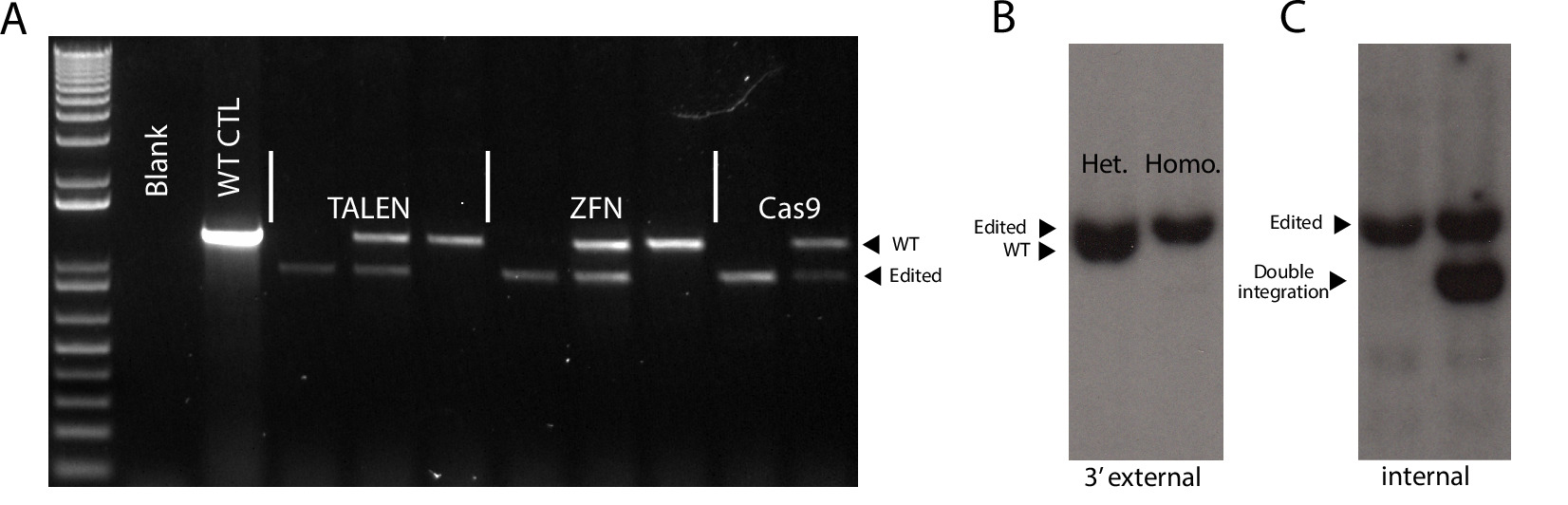
Şekil 4. Genotipleme stratejileri doğru hedefleme onaylayın. (A) Üç SSN platformlarda, hedefsiz heterozigot ve homozigot hedeflenen klonlar gösteren Temsilcisi PCR genotipleme sonuçları. WT CTL yabani tip kontrolü =. (B) Repreheterozigot hedeflenen klon ve 3 'dış prob ile tespit edilen bir homozigot hedeflenen klon gösteren silci Southern blot sonuçları. Fragment boyutları: WT-6.5 kb, Düzenlendi-6.9 kb. (C), uygun şekilde düzenlenmiş bir klon ve rastlantısal olmayan çift entegrasyonu ile heterozigot klon gösteren Örnek Southern blot sonuçları. Fragment boyutları:. Düzgün düzenlenmiş-6.9 kb, anormal ek entegrasyon-5 kb bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
| Çalışma | Gen hedefli | Platform | Onarım şablon türü | Klonların # aldı | Hedefleme verimliliği |
| Sexton ve diğ., 2014 | TPP1 | ZFN | GFP-Puro | bildirilmemiş | bildirilmemiş |
| Sexton ve diğ., 2014 | TERT | ZFN | Higromisin | bildirilmemiş | bildirilmemiş |
| Hockemeyer ve diğ., 2009 | POU5F1 | ZFN | GFP-Puro | 31 | 39.0% |
| Hockemeyer ve diğ., 2009 | PITX3 | ZFN | GFP-Puro | 74 | % 14,9 |
| Hockemeyer ve diğ., 2011 | POU5F1 | TALEN | GFP-Puro | 68 | % 91.0 |
| Hockemeyer ve diğ., 2011 | PITX3 | TALEN | GFP-Puro | 96 | % 13,0 |
| Merkle ve diğ., 2015 | VASA | Cas9 | Muhabir-Geneticin | 139 | % 94.0 |
| Merkle ve diğ. </ em>, 2015 | CRH | Cas9 | Muhabir-Geneticin | 30 | % 93.0 |
| Merkle ve diğ., 2015 | Hcrt | Cas9 | Muhabir-Geneticin | 154 | % 92.0 |
| Merkle ve diğ., 2015 | HMX2 | Cas9 | Muhabir-Geneticin | 11 | % 45.0 |
| Forster et al., 2014 | LRG5-Nterm | ZFN | GFP-Puro | bildirilmemiş | % 30.0 |
| Forster et al., 2014 | LRG5-Cterm | ZFN | GFP-Puro | bildirilmemiş | % 14.0 |
| Soldner ve diğ., 2011 | SNCA | ZFN | Puro | 96 | % 1.0 |
Tablo diğer genlerin 1. Tanım Corre olan bu yöntem kullanılarak hedeflenensponding daha önce yayınlanmış çalışmalar alınan verim hedefleme. 4,5,38-41
| Locus | Sekans | Notlar |
| AAVS1-F astar | CTCTAACGCTGCCGTCTCTC | PCR koşulları: Tm = 57 ° C, 35 döngü |
| AAVS1-WT-R primer | GCTTCTCCTCTTGGGAAGTG | WT bandı: 1273 bp |
| AAVS1 Hedefli-R astar | CGTCACCGCATGTTAGAAGA | Hedeflenen bant: 992 bp |
| T2-Cas9-rehber | GGGCCACTAGGGACAGGAT | Mali ve diğ., 2013 |
| AAVS1-ZFN Sağ | TAGGGACAGGAT | Hockemeyer kaynaktan ve diğ., 2009 |
| AAVS1-ZFN-Sol | TGGGGTGTCACC | Hockemeyer kaynaktan ve diğ., 2009 |
| AAVS1-TALEN Sağ | TCCTAACCACTGTCTTT | Hockemeyer kaynaktan ve diğ., 2011 |
| AAVS1-TALEN-Sol | CCCCTCCACCCCACAGT | Hockemeyer kaynaktan ve diğ., 2011 |
Astar ve SSN hedef dizilerin Tablo 2. listesi.
| Konstrukt Hedefleme | EGFP + Kolonileri sayısı | Hedeflenen aldı klonlar (PCR doğrulanmadı) | (Güney blot) Önceki Bildiren Doğru Hedefleme Verimliliği |
| ZFN | 150 | % 86.9 (% 73.9 het /% 13,0 homo) | % 56 (% 50 het /% 6 homo) |
| TALEN | 412 | % 91.3 (% 47.8 het /% 39,1 homo) | % 47 (% 37.5 het /% 9,3 homo) |
| CRISPR-Cas9 | 235 | % 95.7 (% 69.5 het / 2% 6,3 homo) | bildirilmemiş |
Tablo 3. Karşılaştırmalı EGFP-pozitif sayılar ve Talen, ZFN hedeflenmiş ve CRISPR / Cas9 insan kök hücre kolonileri. PCR önceki deneylerde 4,5 uygun tek entegrasyonlar doğrulanmış Güney lekesi ile karşılaştırıldığında bu deney için AAVS1 loküsündeki entegrasyonlar doğrulandı.
Tartışmalar
Gen-düzenlenmiş insan pluripotent kök hücrelerin homojen popülasyonları izole etmek için burada sunulan yöntem hedeflenen loküsündeki sadece farklı izojenik hPSC hatları oluşturmak için güçlü bir yaklaşımdır. Bu hücreler insan hücre farklılaşması ve gelişimi mekanizmaları prob hem de kontrollü bir genetik ortamda monogenik hastalıkların patofizyolojisi anlamak için ideal bir sistem bulunmaktadır. Burada gösterildiği gibi, bu AAVS1 lokusuna yönelik entegrasyonu sağlamak için üç bağımsız SSK tasarım stratejileri (ZFNs, TALENS ve CRISPR / Cas9) kullanmak mümkündür. Bu yöntemlerin her biri kendi avantajları ve dezavantajları vardır. Potansiyel ZFNs bir avantajı, bir dereceye kadar, TALENS bağımsız nükleaz 42 bağlanma domenlerinin DNA geliştirmek amacıyla yinelemeli mühendisliği sağlar tasarım esnekliği vardır. Bu nukleaz optimizasyonu CRI ile başarılabilir ötesinde ZFNs ve Talens özgünlüğünü artırabilirSPR / Cas9 sistemi. Bu tür seçiciliği, hedef yüksek bir spesifite derecesi gerektiren klinik uygulamalar için önemli olabilir. CRISPR / Cas9 sisteminin en önemli avantajı kullanım kolaylığı olduğunu. TALEN ve ZFN yapım setleri (Addgene 21 aracılığıyla, yani,) kamuya olmasına rağmen sadece gerekli özelleştirme bir 20 baz çifti oligonükleotid olarak px330 plazmidi kullanıldığında, CRISPR / Cas9 tabanlı SSNs (oluşturmak için önemli ölçüde daha kolay Tasarım 14). Bu basitlik kendi çalışmalarında genom düzenleme dahil etmek isteyen araştırma laboratuarları için avantajlı olduğunu kanıtlamaktadır.
Gen-düzenlenmiş hPSC satırları oluşturmak için nucleofection 43, dahil olmak üzere alternatif transfeksiyon teknikleri vardır; Ancak elektroporasyon, tutarlı ve düşük maliyetli 4,5,25 olduğu gösterilmiştir. Nucleofection SSK etkinliği yükselen doğrudan çekirdeğin içine Cas9-kılavuz RNA ribonükleoprotein kompleksleri transfekte etmek için kullanılabilird sadakat 44. MEF'lerden üzerinde hPSCs Büyüyen aşırı farklılaşma olmadan bir pluripotent devlet hPSCs korumak için sağlam ve ucuz bir yöntemdir. Buna ek olarak, bu genetik olarak özdeş kolonilerin kolay izole edilmesini sağlar. Alternatif olarak, bununla birlikte, bu kültür koşulları besleyici göre kültürler daha pahalı olabilir MEF'ler içermeyen kültür hPSCs mümkündür. Bundan başka, tüm süreç çok nadir düzenleme etkinlikleri izolasyonu veya paralel düzenleme deneylerinde, birçok farklı hücre serilerinin jenere edilmesi için izin ölçeklenebilir.
Burada açıklanan protokol sağlamdır; Ancak doğru düzenlenmiş klonlar elde edilebileceği ile verimliliği etkileyen birçok önemli adımlar vardır. Bu yöntem için en kritik bileşeni yüksek kaliteli MEFS ve ilaca dirençli DR4 MEFS yaşıyor. Tek hPSCs sağkalım ince ve düşük kaliteli MEF'ler farklılaşmamış hPSC hatlarının izolasyonu engel olacak. İkinci olarak, Y-27632 kullanımı daanormal karyotip 45 olan hücreler için selektif bir basınç oluşturmadan tek hücre hayatta kalmasını garantileyen, anahtar. Üçüncü olarak, iyi aralıklı koloniler çekme türetilen hücre çizgilerinin genetik homojenlik sağlar. Son olarak, bu açılış birçok küçük parçalar halinde koloni kırmak için yeterince küçük olacak şekilde birden fazla koloniler yeni kuyuda büyüyecek sağlanması, cam pipetler hazırlamak için önemlidir. Bir kopya plaka genotiplemeye izole kolonilerin orijinal plaka bırakarak, kültürde var Bu iyi aralıklı alt-klon toplama izin verir. Bu protokolde zorlu kısmı cam pipetler çekerek manuel; Bu daha önce uygulanmalıdır. Bir cam pipet gerektirmeyen klonu toplama birden çok teknik vardır olduğuna dikkat edilmelidir. Deneyci kendileri için en uygun olanı bulmak için teşvik edilir.
Basit modifikasyonlarla aşılabilir, bu protokole sınırlamalar vardır. O amaçlayan bir deneynly kimin tamir şablonu seçme kaseti içermiyor, düzenlendiği hücrelerin zenginleştiren başka bir yöntem kullanmanız gerekir tamir veya biri olmadan ilgi odağı bozabilir. Pozitif bir klon bulmak için çekilmesi gereken koloni sayısı, seçim yapılmadan büyük ölçüde artırır unutmayın. Verimliliğini artırmak için bir strateji, bir flüoresan proteini eksprese eden bir bütünleşmeyen plazmid birlikte transfekte etmektir. Hücreler iki gün boyunca geri bırakıldıktan sonra, hedef hücreler, flüoresanslı etkinleştirilmiş hücre tasnif kullanılarak pozitif floresans kriteri ve 29 replated olabilir. Bu işlem, elektroporasyon ile plazmit ile transfekte edilmiş hücreler için zenginleştirir ve böylece hücre içinde aynı anda düzenleme olayın olasılığını arttırır.
Burada açıklanan teknikler aynı anda birkaç loci 14,46,47 hedef için birden fazla rehber RNA'ların kullanmak için uzatılabilir. Birçok protokol hPSCs ayırt etmek için kurulmuşturfarklı hücre tiplerine, ilgi 30 hücre tiplerinde çeşitli genetik manipülasyonlar izin. Genel olarak, biz ne olursa olsun SSN seçim hPSCs etkili genom düzenleme potansiyelini ortaya koymuştur. Bu teknik, herhangi bir genomik loküsündeki gen düzenlenmiş olan izojenik hPSC satırları oluşturmak için adapte edilebilir öneriyoruz.
Açıklamalar
Yazarlar hiçbir rakip mali çıkarlarını beyan ederim.
Teşekkürler
Bu çalışma Helen Bateup bir Beyin Araştırmaları Vakfı Tohum Grant (BRFSG-2014-02) tarafından desteklenmiştir. Dirk Hockemeyer Ellison Tıp Vakfı Yaşlanma Yeni Bir Scholar ve Glenn Vakfı yanı sıra Shurl ve Kay Curci Vakfı tarafından desteklenmektedir. DH ayrıca NIH hibe 1R01CA196884-01 tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| DMEM/F12 | Life Technologies | 11320082 | |
| Fetal Bovine Serum (HI) | Life Technologies | 10082-147 | |
| Knockout Serum | Life Technologies | 10828-028 | |
| Fibroblast Growth Factor - basic | Life Technologies | PHG0261 | |
| Pen/Strep | Life Technologies | 15140-122 | |
| Glutamine | Life Technologies | 25030-081 | |
| MEM NEAA | Life Technologies | 11140-050 | |
| 2-mercaptoethanol | Life Technologies | 21985-023 | |
| Y-27632 | Calbiochem | 688000 | |
| 6-well plates | Corning | 3506 | |
| 12-well plates | Corning | 3512 | |
| 4 mm Electroporation cuvettes | Bio-rad | 165-2081 | |
| X-cel gene pulser II | Bio-rad | 165-2661 | |
| 0.25% Trypsin-EDTA | Life Technologies | 25200-056 | |
| 10× Phosphate buffered saline (PBS) pH7.4 | Life Technologies | 70011-044 | |
| Puromycin | Life Technologies | A11138-02 | |
| Pasteur pipettes, plugged | VWR | 14672-412 | |
| Tris-HCl | Sigma-Aldrich | S5941 | |
| NaCl | Sigma-Aldrich | S9888 | |
| SDS | Sigma-Aldrich | L3771 | |
| EDTA | Sigma-Aldrich | E5134 | |
| Proteinase-K | Life Technologies | AM2544 | |
| Ethanol | VWR | TX89125-172SFU | |
| Isopropyl Alcohol | VWR | MK303216 | |
| TE Buffer | Life Technologies | 12090-015 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| 1.8 ml Cryotubes | ThermoScientific | 377267 |
Referanslar
- Carroll, D. Genome engineering with targetable nucleases. Annu Rev Biochem. 83, 409-439 (2014).
- Lancaster, M. A., Knoblich, J. A. Organogenesis in a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Matsa, E., Burridge, P. W., Wu, J. C. Human stem cells for modeling heart disease and for drug discovery. Sci Transl Med. 6 (239), 239ps6 (2014).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat Biotechnol. 27 (9), 851-857 (2009).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat Biotechnol. 29 (8), 731-734 (2011).
- Bozas, A., Beumer, K. J., Trautman, J. K., Carroll, D. Genetic analysis of zinc-finger nuclease-induced gene targeting in drosophila. Genetics. 182 (3), 641-651 (2009).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Urnov, F. D., et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature. 435 (7042), 646-651 (2005).
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., Charpentier, E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jinek, M., East, A., Cheng, A., Lin, S., Ma, E., Doudna, J. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Doudna, J. A., Charpentier, E. Genome editing. the new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/cas systems. Science. 339 (6121), 819-823 (2013).
- Jasin, M. Genetic manipulation of genomes with rare-cutting endonucleases. Trends Genet. 12 (6), 224-228 (1996).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat Rev Genet. 11 (9), 636-646 (2010).
- Sander, J. D., Maeder, M. L., Reyon, D., Voytas, D. F., Joung, J. K., Dobbs, D. ZiFiT (zinc finger targeter): An updated zinc finger engineering tool. Nucleic Acids Res. 38, W462-W468 (2010).
- Carroll, D., Morton, J. J., Beumer, K. J., Segal, D. J. Design, construction and in vitro testing of zinc finger nucleases. Nat Protoc. 1 (3), 1329-1341 (2006).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39 (12), e82 (2011).
- Reyon, D., Tsai, S. Q., Khayter, C., Foden, J. A., Sander, J. D., Joung, J. K. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol. 30 (5), 460-465 (2012).
- Sanjana, N. E., Cong, L., Zhou, Y., Cunniff, M. M., Feng, G., Zhang, F. A transcription activator-like effector toolbox for genome engineering. Nat Protoc. 7 (1), 171-192 (2012).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from neisseria meningitidis. Proc Natl Acad Sci U S A. 110 (39), 15644-15649 (2013).
- Podhajska, A. J., Szybalski, W. Conversion of the FokI endonuclease to a universal restriction enzyme: Cleavage of phage M13mp7 DNA at predetermined sites. Gene. 40 (2-3), 175-182 (1985).
- Costa, M., et al. A method for genetic modification of human embryonic stem cells using electroporation. Nat Protoc. 2 (4), 792-796 (2007).
- Chambers, S. M., Fasano, C. A., Papapetrou, E. P., Tomishima, M., Sadelain, M., Studer, L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Cai, J., et al. Directed differentiation of human embryonic stem cells into functional hepatic cells. Hepatology. 45 (5), 1229-1239 (2007).
- Kehat, I., et al. Human embryonic stem cells can differentiate into myocytes with structural and functional properties of cardiomyocytes. J Clin Invest. 108 (3), 407-414 (2001).
- Soldner, F., et al. Parkinson's disease patient-derived induced pluripotent stem cells free of viral reprogramming factors. Cell. 136 (5), 964-977 (2009).
- Wen, Z., et al. Synaptic dysregulation in a human iPS cell model of mental disorders. Nature. 515 (7527), 414-418 (2014).
- Chiba, K., Hockemeyer, D. Genome editing in human pluripotent stem cells using site-specific nucleases. Methods Mol Biol. 1239, 267-280 (2015).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of mouse embryonic fibroblast cells suitable for culturing human embryonic and induced pluripotent stem cells. J Vis Exp. , (2012).
- Ezashi, T., Das, P., Roberts, R. M. Low O2 tensions and the prevention of differentiation of hES cells. Proc Natl Acad Sci U S A. 102 (13), 4783-4788 (2005).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25 (18), 3745-3746 (1997).
- Southern, E. Southern blotting. Nat Protoc. 1 (2), 518-525 (2006).
- Henegariu, O., Heerema, N. A., Dlouhy, S. R., Vance, G. H., Vogt, P. H. Multiplex PCR: Critical parameters and step-by-step protocol. BioTechniques. 23 (3), 504-511 (1997).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Sexton, A. N., et al. Genetic and molecular identification of three human TPP1 functions in telomerase action: Recruitment, activation, and homeostasis set point regulation. Genes Dev. 28 (17), 1885-1899 (2014).
- Merkle, F. T., et al. Efficient CRISPR-Cas9-mediated generation of knockin human pluripotent stem cells lacking undesired mutations at the targeted locus. Cell Rep. 11 (6), 875-883 (2015).
- Soldner, F., et al. Generation of isogenic pluripotent stem cells differing exclusively at two early onset parkinson point mutations. Cell. 146 (2), 318-331 (2011).
- Forster, R., et al. Human intestinal tissue with adult stem cell properties derived from pluripotent stem cells. Stem Cell Reports. 2 (6), 838-852 (2014).
- Miller, J. C., et al. Improved specificity of TALE-based genome editing using an expanded RVD repertoire. Nat Methods. , (2015).
- Hohenstein, K. A., Pyle, A. D., Chern, J. Y., Lock, L. F., Donovan, P. J. Nucleofection mediates high-efficiency stable gene knockdown and transgene expression in human embryonic stem cells. Stem Cells. 26 (6), 1436-1443 (2008).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nat Biotechnol. 25 (6), 681-686 (2007).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Sci Rep. 4, 5400 (2014).
- Ousterout, D. G., Kabadi, A. M., Thakore, P. I., Majoros, W. H., Reddy, T. E., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome editing for correction of dystrophin mutations that cause duchenne muscular dystrophy. Nat Commun. 6, 6244 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır