Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Çok renkli yerelleştirme mikroskobu canlı memeli hücre organelleri tek membran proteinlerin
Bu Makalede
Özet
Burada, içinde canlı hücre organelleri tek zar proteinleri çok renkli yerelleştirme için bir iletişim kuralı mevcut. Fluorophores eklemek için kendi kendine etiketleme protein kullanılır. Aynı organel farklı membranlar bölmeleri içinde bulunan proteinler, ~ 18 hassasiyetle lokalize nm.
Özet
Proteinlerin hücresel subcompartments lokalizasyonu hakkında bilgi belirli kendi işlevini anlamak çok önemlidir. Burada, yerelleştirme üreten ve haritalar bu proteinlerin izleme proteinler için erişilebilir microcompartments belirlenmesi için izin veren bir süper çözünürlük teknik mevcut. Ayrıca, çok renkli yerelleştirme mikroskobu tarafından yerelleştirme ve profilleri farklı subcompartments proteinlerin izleme aynı anda elde edilir. Teknik canlı hücreler için özgüdür ve tek mobil zar proteinleri tekrarlayan görüntüleme üzerinde temel alır. Proteinler ilgi genetik olarak belirli, sözde Self Etiketleme etiketleri ile erimiş. Bu etiketleri bir substrat ile kovalent bir şekilde tepki enzimlerdir. Bu yüzeyler için Birleşik floresan boyalar vardır. Reaksiyon ile floresan enzim öğesini proteinlerin etiketli yüzeylerde sonuçlar etiketli proteinler. Burada, Tetramethylrhodamine (TMR) ve silikon rodamine (sır) Floresan boyalar enzimler yüzeyler için bağlı olarak kullanılır. Substrat konsantrasyonu nM Aralık pM içinde kullanılarak, alt stokiometrik etiketleme farklı sinyaller de sonuçları elde edilir. Bu sinyaller ~ 15-27 ile yerelleştirilmiştir nm hassas. Sayede renk sayısını sınırlı kullanılabilir membran geçirgen boyalar ve enzimler Self Etiketleme repertuar tek moleküllerin çok renkli görüntüleme için tekniği sağlar. Biz kalite kontrol enzimi (Pten) lokalizasyonu belirlenerek teknik fizibilite göstermek-kinaz 1 (PINK1) ile ilgili olarak diğer membran proteinlerinin işleme sırasında farklı mitokondrial bölmeleri indüklenen. Farklı etiketlenmiş tek protein molekülünü perde veya ortak izleme arasındaki gerçek fiziksel etkileşim için test olsa da, düşük etiketleme derece aynı zamanda etiketli iki bitişik protein sahip olma olasılığını azaltmak için sınırlıdır. Teknik proteinler membran bölmeleri görüntüleme için güçlü olmakla birlikte, çoğu durumda son derece mobil çözünür proteinler lokalizasyonu belirlemek uygun değil.
Giriş
Bu iletişim kuralının amacı yerelleştirilmesine ve tek zar proteinleri canlı hücreleri içinde izlemek için bir görüntüleme yöntemi sağlamaktır. Bu yöntem1,2izleme ve yerelleştirme mikroskobu (TALM) diyoruz. Stokastik optik imar mikroskobu (fırtına)3 ve floresan Photoactivation yerelleştirme mikroskobu ((F) PALM)4,5gibi TALM bir tek molekül tabanlı floresans yerelleştirme tekniktir. Ancak, zar proteinleri birlikte hareketlilik ile aynı yinelenen görüntüleme için mobil protein erişilemez microcompartment molekül farklı pozisyonlar ortaya çıkarır, etiketli şekilde farklıdır. Başka bir deyişle, protein mümkün yerelleştirmeler organel mimarisini ve protein1hareketliliğini tarafından ayarlanır. Yerelleştirme ve yörünge haritaları görüntüleme mobil proteinler tarafından ortaya çıkarır çünkü çeşitli diğer süper çözünürlük teknikleri6,7,8 ' e tamamlayıcı yöntemdir. Etiketleme başına sigara floresan genetiği füzyon protein kullanarak dayanmaktadır. Bu füzyon proteinler kovalent bir boya için Birleşik bir substrat ile reaksiyona enzimler Self Etiketleme. Bu yordam etiketleme derecesi olabilir avantajı vardır ekledi substrat miktarı kontrollü. Ayrıca, floresan, bağlı olarak seçilen konjuge boya rengini değiştirmek için izin verir. Birkaç kendini etiketleme enzim-mevcut9etiketlerdir. Tam olarak onlar ağartılmış kadar enzim-Etiketler Self Etiketleme, konjuge boyalar genellikle daha istikrarlı ve floresan proteinler1 ve bireysel proteinler bu nedenle artık kaydedilebilir daha parlak ve daha fazla kullanmanın bir diğer avantajı. Bu yörüngeleri mobil proteinlerin kaydı ve difüzyon katsayıları10,11çıkarılması için sağlar.
Burada, biz TALM fizibilite mitokondri zar proteinleri ile göstermek, ama aynı zamanda farklı hücre türleri12,13de dahil olmak üzere diğer içi ve ekstra cellular membran proteinlerinin için uygulanabilir. Biz çok renkli TALM daha da proteinler tamamlayıcı için varolan Süper çözünürlük floresans mikroskobu teknikleri14,15, içinde farklı subcompartments arasında eşzamanlı ayrım sağlar göstermek 16. TALM17Imaging canlı hücre ile uyumludur. Fotoğraf-fizik seçilen rhodamines Tetramethylrhodamine (TMR) ve silikon-Rhodamien (sır), özellikle onların parlaklık ve istikrar, sağlar kayıt tek zar proteinleri için yerelleştirme (ve yörünge) haritalar sağlayan birden çok çerçeve. Ancak, TALM hareket bulanıklığı çok yüksek olduğu ve kare başına toplanan fotonlar uygun yerelleştirme için çok düşük çözünür proteinler yüksek difüzyon katsayıları ile lokalizasyonu için sınırlıdır. Ayrıca, TALM Fototoksik etkileri azaltmak örneğin fırtına veya uyarılmış emisyonu tükenmesi (STED) mikroskopi6,7, daha az uyarma güç gerektirir. Fototoksik stres organellar morfoloji18 ve böylece mobilite analiz19kez etkiler bu yana bu önemlidir. Özetle, biz yerelleştirme mikroskobu yöntemleri fırtına/STED arasında bir boşluk doldurur bir teknik olarak çok renkli TALM canlı hücreler içinde mevcut / (F) PALM ve protein hareketlilik floresans kurtarma gibi photobleaching sonra analiz teknikleri (sıkı bağlamak)20 ,21, floresans korelasyon spektroskopisi (FCS)22ve floresan çapraz korelasyon spektroskopisi (FCCS)11,23.
Access restricted. Please log in or start a trial to view this content.
Protokol
Aşağıdaki iletişim kuralı yerel kurum Araştırma Etik Komitesi kuralları izler.
1. yöntemleri
-
Hücre kültürü
- Hücreleri, örneğin HeLa hücreleri (insan serviks Karsinomu), 37 ° C ve % 5 CO2büyüme ortamının 5 mL içeren bir T25 hücre kültür şişesi yetiştirmek.
Not: Görüntüleme için hazırlanmış coverslips (1.3 ve 1.4 adımlara bakın) üzerine hücreleri bölme ve orta Imaging'de tutun.
- Hücreleri, örneğin HeLa hücreleri (insan serviks Karsinomu), 37 ° C ve % 5 CO2büyüme ortamının 5 mL içeren bir T25 hücre kültür şişesi yetiştirmek.
-
Hücre transfection
Not: stabil tagged proteinler mümkün olduğunca hızlı hücre satırlarını kullanın güçlü overexpression önlemek için24 . Plazmid DNA transfection için kullanılan miktarını geçici transfection için uyum. Örneğin, CA2 + fosfat transfection25 kullanıldığında, 2,5 – 5 µg, plazmid DNA ile 3.5 cm hücre kültür tabağına hücreleri (% 80 – 90 confluency) transfect. Çift Kişilik transfection yerine getirirken, her plazmid yapı başına 2,5 µg kullanın.- Çift renk deneyler, bir kendi kendine etiketleme protein kararlı bir ifadeyle bir hücre kültürünü kullanın ve geçici diğer kendi kendine etiketleme protein17kodlama plazmid ile transfect.
Not: Burada, Çift renk deneyler için HeLa hücreleri stabil Self Etiketleme proteinler PINK1 Halo etiketi ve Tom20-fSNAP-etiketi ifade kullanılmıştır.
- Çift renk deneyler, bir kendi kendine etiketleme protein kararlı bir ifadeyle bir hücre kültürünü kullanın ve geçici diğer kendi kendine etiketleme protein17kodlama plazmid ile transfect.
-
Coverslips temizlik
- Yer coverslips bir ölçek. 30 mL H2O coverslips içeren kabı ekleyin ve onların yüzeyindeki toz çıkarmak için yavaşça sallayın.
- Coverslips cımbızla toplamak ve onları azot bir akışı ile kurutun.
- Örneğin, coverslips yüzeyinde herhangi bir organik kirlenme plazma temizleyerek kaldırın.
Not: daha fazla cam malzemenin kirlenmesini önlemek için coverslips işleme sırasında eldiven giymek.
Dikkat: ne zaman coverslips plazma temizlik tarafından temizlenir, coverslips sadece üst tarafı temizlenir; Bu yan kaplama ile poli-L-lizin-Polietilen glikol-arginin-glisin-aspartat (PLL-PEG-RGD) (Bölüm 1.4) ve (Bölüm 1.5) tohum hücre için kullanın.
-
Coverslip kaplama PLL-PEG-RGD ile
Not: PLL-PEG-RGD Polietilen glikol (3.000 Da) ve cysteine-glycine-arginine-glycine-aspartate-serine (CGRGDS) peptid ile bağlı bir poli-L-lizin (PLL) türevidir. PLL olumsuz şarj edilmiş cam yüzeye bağlar ve PEG fırça oluşturur. Bu büyük ölçüde belirsiz bağlamasını şarj edilmiş floresan boyalar da azaltır. Buna ek olarak, RGD motifi sinyal peptid integrin reseptör taklit eder ve böylece integrin aracılı bağlılık yoksa değil kolayca uygun hücre teşvik etmektedir.- PLL-PEG-RGD daha önce açıklandığı26hazırlayın. Kısacası, PLL-PEG-RGD 0,8 mg 1 ml PBS çözülür. PLL-PEG-RGD çözüm 10 µL temiz bir coverslip üst tarafta ekleyin.
- İkinci bir coverslip alıp temiz yüzeyi ile baş aşağı (Bu PLL-PEG-RGD düşüş üstüne var) ilk coverslip üzerine yerleştirin; Bu iki coverslips arasında PLL-PEG-RGD çözüm sandviç sonuçlanır.
- Dikkatle sandviç coverslips bir ölçek içinde yerleştirin ve tozsuz kuru bir ortamda oda sıcaklığında 1 h için kuluçkaya.
- 1 saat sonra tamamen su ile coverslips kapsayacak şekilde kabı H2O 30 mL ekleyin.
- Coverslips birbirinden ayırmak kadar kabı hafifçe sallayın.
- Sudan coverslips toplamak ve onları azot gazı akışı altında kuru için cımbız kullanın.
Not: Kaplı coverslips bir kuru, steril cam Petri kabına kapak ile birkaç gün saklanabilir.
-
Görüntüleme için örnek hazırlanması
- Tek kaplı coverslips yukarı bakacak şekilde PLL-PEG-RGD kaplı yüzey ile bir 35 mm hücre kültür tabak içine aktarmak ve orta üst görüntüleme 2 mL ekleyin.
- 2 hücre kültür çanak ile kaplı coverslip ortamda Imaging ml ilgili zar proteinleri, kendi kendine etiketleme Etiketler hızlı ~ 500.000 trypsinized hücreleri (200-500 µL) ekleyin. Shake yavaşça elle elde etmek için hücreleri homojen bir dağılım sağlamak için bir düzgün hücre katmanı.
- % 80 confluency ulaşıncaya kadar hücreleri 37 ° C ve % 5 CO2 kuluçkaya.
Not: Hücre örnekleri 3 gün önce görüntüleme ve 1 gün önce transfection numaralı seribaşı. Stabil faiz protein Hızlı, hücreleri, 2 gün önce görüntüleme numaralı seribaşı. Daha sonra yalnızca coverslip üzerinde yetiştirilen hücreleri görüntüsü.
-
Tagged proteinlerin etiketleme
Not: En floresan yüzeylerde su içermeyen DMSO içinde çözünmüş gerekir. Biz nihai etiketleme konsantrasyon 0,2 – 30 nM17olduğunda 1 µM floresan substrat hisse senedi çözümleri kullanmanızı tavsiye ederiz. Membran proteinlerinin hücreleri içinde görüntüleme için membran geçirgen floresan yüzeylerde kullanın.- Görüntüleme orta olarak 37 ° C su banyosunda ısıtın.
- Önceden ısıtılmış görüntüleme orta 1 mL kapaklı 2 mL tüp içine pipet. 0,2-30 µL floresan yüzeylerde, nihai etiketleme çözüm hazırlamak için 1 µM hisse senedi çözümlerinden ekleyin (son konsantrasyonu: 0,2 – 30 nM).
- Girdap etiketleme çözüm için 10 s.
- Bir coverslip sayfasındaki hücreleri ile 35 mm Kültür çanak orta yerine (bkz. Adım 1.5) tarafından 1 mL hazırlanmış etiketleme çözüm.
- 37 ° C ve % 5 CO2 20-30 dk için etiketleme çözüm hücrelerde kuluçkaya.
- PBS 2 mL bir kez o zaman orta iki kez görüntüleme 2 mL hücreleri yıkayın. Son olarak, hücre yemek için taze görüntüleme orta 1 mL pipet ve örnek için en az 1 h 37 ° C ve % 5 CO2 Kombine Kuluçka makinesi içine koy. Görüntüleme daha önce bir kez daha görüntüleme orta değişimi.
Not: deney ilk kez çalıştırırken, doğru organellar membranlar için kendi kendine etiketli proteinlerin organelleri piyasada bulunan organel özel boyalar27,28ile boyama tarafından hedefleme onaylamak. Bu durumda, ayrıca substrat Self Etiketleme enzimler için 100-200 nM güçlü sinyaller üretmek için kullanın.
-
Bir floresan boncuk örnek hazırlanması
Not: çok renkli floresan boncuk (0,1 µm) optik drift belirlemede ve farklı kanal görüntüleri hizalamak için kullanılır. Kaydedilen görüntüleri ile bir homojen koordinatlar matrisini iki emisyon kanallar için oluşturulur.- Sulandırmak boncuk saf H2O. % 1 için çözüm
- Yer 5 damla temizlenmiş bir coverslip üzerinde beş farklı pozisyonlarda floresans boncuk ile hazırlanan çözeltinin (bkz. Adım 1.3).
- Temiz bir bankta kuru floresan boncuk örnek ver.
Not: Örnek yeniden kullanılabilir; Bu nedenle, örnek kirlenmesini önlemek ve 4 ° C'de tutmak için alüminyum folyo ile kapak
2. mikroskobu
-
Deneysel Kur
Not: Çift renkli tek molekül görüntüleme için bir temel mikroskobu sistemi ters bir mikroskobunun dayanmaktadır: o iki lazerler multi-mode-optik bir kutuplaşma monomode fiber bir tek toplam iç yansıma (tır) bakımı ile birleştiğinde ile donatılmıştır kondansatör, TIRF, polyband emisyon filtreleri, bir resim splitter ve çok hassas kamera (Şekil 1) için tasarlanmış bir petrol daldırma amaç. Bir tır kondansatör sağlayan sürekli olay açısını ayarlamak için epi son derece eğimli-arasında geçiş yapmak için gerekli değildir ve lamine optik levha (son derece eğimli ince aydınlatma (HILO)29) ve TIRF uyarma modu ile en iyi duruma getirilmiş penetrasyon derinliği. Görüntüleri bir çok hassas soğutmalı dedektör sistemi ile elde, Örneğin, bir arka ışıklı elektron çarparak hesaplanır eşleşmiş cihaz (EMCCD) kamera (kuantum verimliliği QE > % 90) veya bir sCMOS kamera (QE > % 80-90).- Optik drift (bkz. Adım 2.2) Floresan boncuk bu deney için Örneğin, 10.000 çerçeveler denemede kaydedilen zaman daha sonra kullanılacak olan, ayrıca 10.000 çerçevelerle boncuk örnek kayıt olarak aynı koşullarda Imaging tarafından belirler. Optik drift belirlenmesi için ilk kare ve son alınan kare (Şekil 1B) boncuk konumunu karşılaştırın. Gerekirse, daha sonra görüntü serisi için optik drift30 düzeltmek ve/veya drift istikrarlı ortamları kullanın.
- Filtre küp ile uygun dikroik kiriş kırık, Örneğin, turuncu ve kırmızı floresans artı turuncu floresans ve kırmızı floresans için yeterli emisyon filtreler için donatmak. Resim splitter uygun süzgeçleri ile donatmak. Bir kanal sinyalleri olası sızıntı için her iki kanal (Şekil 1 c) tek renk örnekleri kaydederek diğer kanal kontrol edin.
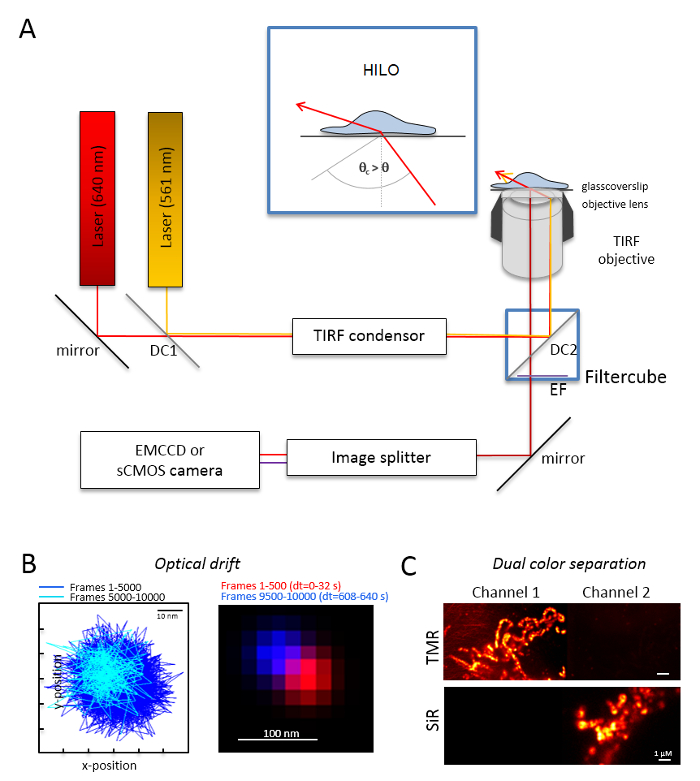
Resim 1 : Çok renkli izleme ve yerelleştirme mikroskobu (TALM) ile turuncu ve kırmızı yayıcılar için optik düzen. (A)Inverted mikroskop Kur ile en az iki uyarma lazerler, TIRF Kondenser, TIRF uygun amaç, bir resim splitter ve hassas bir fotoğraf makinesi. İç metin: organelleri hücrelere heyecanlandırmak için olay Işın açısı elde TIRF için kritik açı eğimli ve optik sayfası aydınlatma (HILO) lamine daha küçük olarak ayarlanmalıdır. DC1: Dikroik ayna 1; DC2: Dikroik ayna 2. EF: emisyon filtre. Floresan boncuk aynı kare hızı itibariyle aşağıdaki deneyler ile 10.000 çerçeveleri için konumlarını görüntüleme tarafından optik drift (B) Test (burada: 15 Hz). İlk 500 çerçeveleri ve son 500 çerçeveleri bağlı pozisyonların drift göster. Ayrıca, birleştirilen görüntünün konumunu ilk ve son kareyi kırmızı ve mavi ile en az bir drift göster. Drift sinyallerin merkezi arasındaki mesafe toplam kayıt zaman, burada 125 pm/s. (C) kontrol sinyalleri, burada TMR ve efendim açık ayrılması bölünmüş olmasıdır. Her iki kanal için kümülatif toplamı imge--dan 3000 çerçeveler (Kanal 1 TMR) ve Kanal 2'deki efendim üretildi. EfendimHTL Tom20-HaloTag ve OxPhos karmaşık V-HaloTag için TMRHTL bağlıydı. Renkler yanlış renktir. Ölçek çubukları = 100 nm (B) ve 1 µm (C). Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
-
Fiziksel görüntü splitter hizalamasını görüntüleri oluşturulan
Not: bir coverslip üzerinde hazırlanan numune montaj için kendi kendine yapılan bir örnek sahibi olabilir (Şekil 2A) kullanılır. Toz, vb örnek düşen önlemek için kültür çanak kapağı gevşek odası üstüne monte durumdayken yerleştirin. Aynı örnek sahibi coverslip floresan boncuk veya hücreleri ile bağlamak için kullanılabilir; hücre görüntüsü, 0,5-0,8 ekleyin mL orta görüntüleme. Resim splitter görüntü ikiye böler ya da daha fazla hayalice kanalları ayrılmış ve yan yana aynı kamera projeler. Bu işlem potansiyel olarak farklı optik yolları geçiş nedeniyle kanallar arasında sistematik çarpıtmalara tanıtır ve doğrudan colocalization analiz engellenmemesine. Bu nedenle, ilk fiziksel hizalama ve ikinci, gerçekleştirmek bir dönüştürme matrisini hizalamayla sonrası düzeltme. Her iki hizalama işlemleri için floresan boncuk rastlanılmaması görüş alanı Dağıtılmış.- Örnek-tutucu politetrafloroetilin (PTFE) arasında floresan boncuk ile hazırlanmış örnek mount-yüzük ve kırmızı Kauçuk halka (Şekil 2A).
- Mikroskop, tüm donanım bileşenleri ve Mikroskopi için gerekli tüm yazılım başlatın.
- Amaç ve coverslip alt isopropanol ile ıslak bir hav bırakmayan dokusu bezle temizleyin. Sonra her iki öğe taze bir hav bırakmayan dokusu ile kuru. Bir damlacık Daldırma yağ objektif lens öğrenci üzerinde yerleştirin.
- Petrol coverslip altındaki kişiler ile mikroskop sahne alanı'na boncuk örnek örnek sahibi yerleştirin. Üstündeki iletim ışık veya lazer çizgi kullanarak odak.
- İki uyarma lazer güç iki floresan kanal benzer sinyal yoğunluğu elde etmek için ayarlayın. Birçok farklı floresan sinyalleri ile bir alanı arayın.
- Kamera kontrol yazılımı kullanarak floresan kanalları birleştirilmiş bir görünümünü oluşturun. Daha sonra el ile iki floresan kanallardan (Şekil 2B) sinyallerin en iyi yerleşimi elde etmek için görüntü splitter iç Aynalar yatırmak için resim splitter vidaları kullanın.
Not: dikkat! Kameranın dinamik alanı fazla olamaz.
-
Hizalama kayma dönüştürme gerçekleştiren yazılım tarafından hayalice ayrı kanal
Not: Bizim yazılım eklentisi (istek üzerine) ile ilgili sonrası düzeltme hizalama ve yerelleştirme aşağıdaki bölümü gösterir.- Yazılım kontrol TIRF mikroskop başlangıç ve tek tek kanalları Canlı akışı modunda görüntülemek. Bir anlık görüntü görüntü heyecan verici floresans tüm kanallar (Şekil 2C) almak.
- Bu anlık görüntü görüntü dönüştürme matrisini üretmek için kullanın (bkz. Şekil 2).
Not: Dönüştürme matrisini kayma dönüştürme, çeviri (sapma iki kanal arasında tek bir nokta kaynaktan sinyallerin) düzeltir bir afin birine genellikle, kullanılıyor. - Yazılım analiz eklentisi başlatmak (bizim lab gelen istek üzerine temin edilebilir, Şekil 2Cbakınız).
- (Bkz. Adım 2.2) önceden kaydedilmiş çift renkli resimler floresan boncuk belgili tanımlık bilgisayar yazılımı yükleyin. Floresan kanalları kullanılan yönünü seçin. Sonra 'Evet' 'görüntüleri ayarlamak için' sorulduğunda'i tıklatın ve daha önce alınmış anlık görüntü seçin.
- Açık belgili tanımlık "birim birim dönüştürme faktörlerinin (piksel boyutunu, kare oranı, foton dönüştürme faktörü) tanımlamak için müdür".
- Açık belgili tanımlık "Yerelleştirme müdür". İlk işlev (PSF) noktası yayıldı belirlemek. Sıkıştırma belgili tanımlık düğme: "PSF radius". Açılan "PSF Tahmincisi" penceresine sayısal diyafram ve maksimum emisyon tanımlayın. Başlat "tahmini PSF RADIUS" tıklayarak. Elde edilen deneysel PSF tanımlamak değerlendirmesi kutusunu kabul, deflasyon döngü sayısını ve bilgisayarın kaç çekirdek hesaplamada kullanılır. "Tek parçacık yoğunluğu dağılımı bir 2D simetrik Gauss işlev tarafından (Şekil 2C) uygun başlatmak için yerelleştirilmesine" tuşuna basın.
- "Değerlendirmesi kutusunu kabul elde edilen deneysel PSF tanımlamak", deflasyon döngü sayısını ve bilgisayarın kaç çekirdek hesaplamada kullanılır. "Tek parçacık yoğunluğu dağılımı bir 2D simetrik Gauss işlev tarafından (Şekil 2C) uygun başlatmak için yerelleştirilmesine" tuşuna basın.
- Açık belgili tanımlık "kalibrasyon müdür". İki kanal işlenmiş birleştirilen görüntüyü orijinal sinyalleri ve yerelleştirilmiş merkezleri gösterilir. "Afin" modu seçin. El ile ortaya çıkmış iki kanal yerelleştirilmiş merkezlerinde karşılık gelen çiftlerini bir bağlantı çizgisi çizerek aynı floresan boncuk bağlayın.
- Tüm görüş alanı dağıtılan karşılık gelen sinyalleri bağlayın. Bundan sonra "kabul" düğmesine basın. Kalibrasyon kaydedin.
Not: Kayma dönüştürme floresan her boncuk örneklenmiş ve arasında interpolasyon. Ayıklanan dönüştürme işlevi böylece onlar içinde onların yerelleştirme hassas yerleşimi daha sonra deneysel çift renkli tek molekül yerelleştirmeler düzeltmek için kullanılan bir deplasman alan Δr(x,y) temsil eder. Kayma dönüştürme matrisini genellikle çevirme, ölçekleme ve döndürme nanometre doğruluk ile kanallar arasında düzeltir bir afin biridir ve bu el ile bire bir eşleştirme (Şekil 2C) varılabilir.
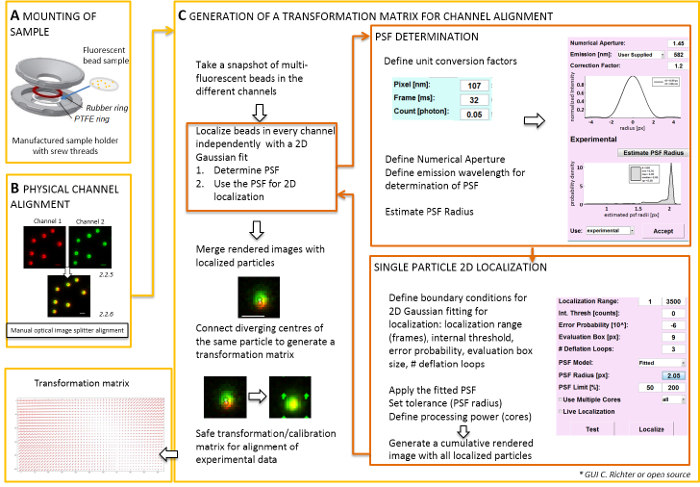
Resim 2 : İş akışı Çift renk hizalama için. (A) Floresan boncuk ile coverslip bir örnek tutucu bir PTFE ve kauçuk halka arasında monte edilmiş. Sonra üst ve alt kısmına odasının cıvatalı birlikte. (B) resim splitter tarafından oluşturulan kanal sayısı fiziksel hizalaması. Boncuk (0,1 µm) Floresan sinyallerini iki kanal içinde kaydedilen (yeşil ve kırmızı, yanlış renkler) birleştirilir. En iyi yer paylaşımı farklı sinyaller elde kadar karşılık gelen optik görüntü splitter, el ile vidalar (sarı renk, alt panel). Bir dönüştürme matrisini (C) nesil post-processive kanal hizalama için. Bir parçacık tam yerelleştirme için noktası işlevi (PSF) emisyon dalga boyu ve amacın sayısal diyafram olan bağımlılığı yayılır belirlemek gereklidir. Bir PSF merkezinde uygun bir simetrik iki boyutlu Gauss tarafından analiz onun yoğunluğu profili tarafından belirlenir. Sinyal tepe elde edilen yerelleştirme sonra özgün, bulanık sinyalleri öngörülmektedir. Birleştirilmiş bir görüntüde, daha sonra deneysel veri post-processive hizalama için kullanılan bir dönüştürme matrisini oluşturmak için iki kanal gelen sinyalleri yerelleştirilmiş merkezlerinden bağlı. Ölçek çubukları 1 µm (B, C) =. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
-
Mitokondri zar proteinleri tek molekül görüntüleme
Not: Oda sıcaklığında tüm deneyler yapılmaktadır. T-hücreleri veya yapışık olmayan hücreleri özel içinde31Imaging önce immobilize gerekir.- Numune yapışık hücreleri ile coverslip arasında kauçuk ve PTFE halkalar (Şekil 3A) bağlama. Odası 0.5 – 0.8 ile doldurmak orta Imaging mL.
- Adımları 2.2.2–2.2.5 tekrarlayın.
- Kritik açı belirli bölge bir HILO sayfa32 (HILO modu, Şekil 1A) ile ilgi heyecanlandırmak tır modu için daha küçük bir olay açısı oluşturmak için yazılım kontrol aydınlatma açısı TIRF mikroskop ayarlayın.
Dikkat: lazer ışını ile doğrudan göz temasından sakının! - EM kazanç ve yeterli fotonlar kare başına toplar deneme için uygun bir çekim hızı seçin.
- Lazer güç yerelleştirme hassas doğrudan S/N33 ' e (Şekil 3 c) karşılık gelen bu yana (S/N) oranı (Şekil 3B), gürültü yüksek sinyal elde etmek için ayarlayın.
- Hücre çevre ile örtüşmeyen, uzamış mitokondri ve tek molekül sinyalleri (3D şekil; bir alan bulmak Takıma giren Video 1). Yok tek molekül sinyalleri görünür durumdaysa, görünüşte sonuçlarını tek molekül sinyalleri (3E rakam) Ağartma kadar bekleyin.
- Kayıt sinyalleri sayısı kadar makul devamı (genellikle 1.000-10.000 çerçeveler floresan boyalar, Şekil 3Fbeyazlatma davranışını bağlı olarak) için çok düşüktür.
- Görüntü işleme yazılımı ve onay mitokondrial yapıları için en az 1.000 kaydedilen çerçeveleri (Şekil 3 g) toplu işlenen toplam görüntüsünü oluşturarak başlayın.
Not: En hızlı mümkün kare hızı okuma alanına göre belirlenir. Görüş alanı bir kanal için bir çift renkli resim splitter (512 x 512 piksel) 256 x 512 piksel ve dört renk 256 x 256 piksel için azalır. Böylece, bir resim splitter için iki renk kullanmak için bu en düşük olası okuma zaman elde etmek için 30 Hz. çerçeve aktarım modunu kümesidir. - Yazılım analiz eklenti ve yük ham veri başlatın. Seçme belgili tanımlık kanal yönlendirme ve yük imge. Adım "Ayarla'ya görüntülerde" sorulduğunda 2.3.9 dönüştürme matrisini kullanın. Kanalları ayrı ayrı görüntülenir.
- "Birim Yöneticisi" daha önce olduğu gibi her kanal için birim dönüştürme faktörleri tanımlamak için açın. Her kanal için "Yerelleştirme Yöneticisi" açın. Sonra değerlendirme kutusunda, deflasyon döngü sayısını tanımlamak için kullanılan ve bilgisayarın kaç çekirdek hesaplaması için kullanılan küme koşulları teorik PSF ekleyin. Nihayet, yerelleştirilmiş tek parçacıklar (Şekil 3 H; almak için "yerelleştirilmesine" tuşuna basın Takıma giren Video 2).
- Not program son olarak gösterilen bir toplu superresolution görüntü oluşturmak tüm parçacıklar (Şekil 3I) Lokalize.
- Örneğin, açık kaynak yazılım veya istek üzerine yazılım tarafından çözümlemesi gerçekleştirin.
- Örneğin, çoklu hedef izleyici10 ile yerelleştirilmiş her iki kanal tek molekülleri izlemek
Not: Adım 2.4.13 sınır koşulları doğru ayarlanmış ilgi proteinlerin diffusibility hakkında ön (deneysel) bilgi ihtiyacı var. Genellikle, doğru sınır koşulları bulma yinelemeli bir işlemdir.
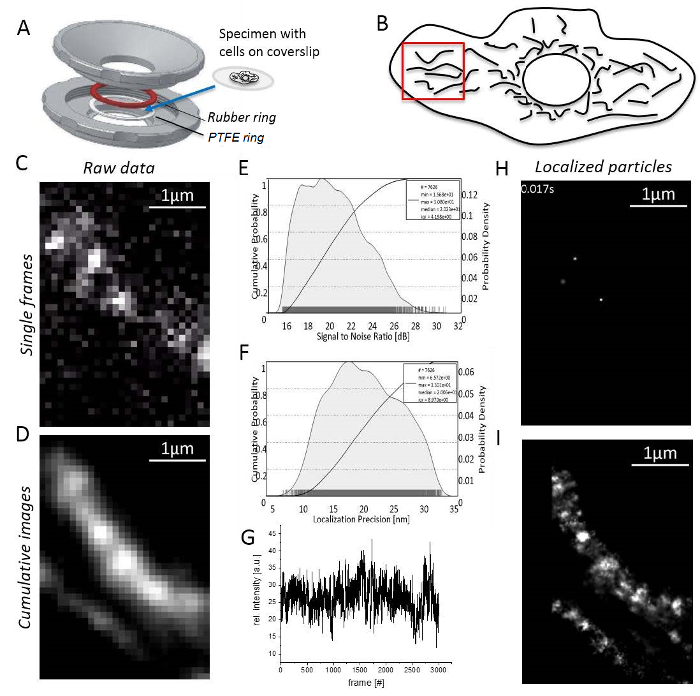
Şekil 3 : Adımları sırasında tek molekül yerelleştirme mikroskobu. (A) A coverslip örnek ile üst ve alt parçası arasında (gri) (J. Bereiter-Hahn tarafından tasarlanmış) ev yapımı örnek sahibi monte edilmiş. Örnek-tutucu parça cıvata biraraya geldiğimizde bir kauçuk halka (kırmızı) ve PTFE ring (beyaz) sistemi yukarıda ve aşağıda coverslip kapatın. (B) sinyal gürültü oranı TMR sinyal. (C) hesaplanan tüm yerelleştirilmiş parçacıklar için yerelleştirme hassas çubuk. (D) seçimi, burada, hücre çevre ile açıkça ayrılmış mitokondri görüntüleme için makul bir bölge. (E) kayıt ve görüntü işleme: ayrı tek molekül sinyalleri ile tek bir kare görüntülenir (burada, CV-HaloTag/TMRHTL tek molekülleri kaydedildi). (F) yoğunluk TMR kayıt zamanla. (G) kümülatif toplamı görüntü 3000 kare, işlenmemiş. (H) parçacıklar, CV-HaloTag/TMR tek bir kare bir 2D Gauss işlev ile yerelleştirilmişHTL . (Ben) toplu, işlenen tüm yerelleştirilmiş CV-HaloTag/TMRHTL parçacıkları 3000 kare gösterilen toplam görüntü. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Çok renkli görüntüleme ve colocalization analiz proteinlerin alt organellar yerelleştirme belirlemek için yardımcı olabilir. Biz bu daha önce sitozolik fosfataz ve tensin homologue, PINK1, mitokondrial proteaz17tarafından işleme nedeniyle farklı alt mitokondrial konumları olan ile gösterilmiştir. PINK1 mitokondriyal işlev34,35güvence altına alınması önemli bir faktördür. PINK1 sıras?...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Burada, mobil zar proteinleri çift renk tek molekül yerelleştirme için bir teknik sunuldu. Aşağıdaki iletişim kuralı, membran proteinlerinin erimiş TMR ve onların anılan sıraya göre yüzeylerde Birleşik efendim rodamine boyalar ile reaksiyona proteinler Self Etiketleme için. Rodamine are parlak ve photostable boya ve böylece yinelenen görüntüleme1için izin verir. Başarılı performans için çeşitli koşullar ve kritik konular akılda tutulması gerek.
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Yazarlar Biyofizik grup ve Jacob Piehler Üniversitesi Osnabrück sürekli desteği, teknik yardım ve malzeme hazırlanması için Wladislaw Kohl ve mikroskoplar kullanmak için sağlamak için CellNanOs yönetim kurulu için teşekkür etmek istiyorum. Proje SFB 944 tarafından finanse edildi.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| (2-(4-(2-hydroxyethyl)-1-piperazinyl)-ethanesulfonic acid, 1 M) (HEPES) | Biochrom | #1104E | |
| DC1: Dichroid beam splitter | Chroma | 640 dcxr | NC506031 |
| DC2: Polychroic Mirror, beamsplitter | Chroma | zt405/488/561/640rpc | discontinued |
| Dulbecco´s Phosphate-Buffered Saline (PBS) 1x (w/o Ca & Mg) | Sigma-Aldrich & Co. | #RNBF8311 | |
| Earle´s MEM without phenol red, without L-Glutamine and without NaHCO3 containing 1% FBS, 0.1% HEPES, 0.1% NEAA, 0.1% Alanyl-L-Glutamine and 34.78% sodium hydrogen carbonate (NaHCO3 0.75g/l) | Imaging medium | ||
| Earle´s minimum essential medium (MEM) with phenol red, containing 1% Fetal Bovine Serum Superior (FBS), 0.1% HEPES (2-(4-(2-hydroxyethyl)-1-piperazinyl)-ethanesulfonic acid, 1 M), and 0.1% non-essential amino acids (NEAA) | Growth medium | ||
| EF: Emission filter quadbandpass | AHF analysentechnik | F72-866 | Brightline HC 446 nm/523 nm/600 nm/677 nm |
| EMCCD camera | Andor | Andor iXON 897 | EMCCD camera |
| Emission filter QuadView filter cubes, orange | AHF analysentechnik | F39-637 | bandpass 582 - 619 nm |
| Emission filter QuadView filter cubes, red | Chroma | bandpass 655 - 725 nm (HQ 690/70) | |
| FBS (Fetal bovine serum) superior | Biochrom | S0615 | |
| Fluorescent beads: TetraSpeck™ Microspheres, 0.1 µm, fluorescent blue/green/orange/dark red | Thermo Fisher Scientific | T7279 | fluorescent microspheres |
| Glutamine | Biochrom | #0951C | |
| HeLa cells | DSMZ | ACC-57 | Cervical carcinoma cells from patient Henrietta Lacks |
| Hela cells CI::paGFP, stable | Muster et al., PLOSOne 2010 | ||
| Hela cells CV g::Halo7-Tag, stable | Appelhans et al., NanoLett 2012 | ||
| Hela cells Tom20::Halo7-Tag, stable | Appelhans et al., NanoLett 2012 | ||
| Image splitter | Photometrics | Dual-View QV2 | image splitter emission |
| Imaging processing software | ImageJ2 / Fiji | freeware | |
| Immersion Oil - ImmersolTM 518 F (ne = 1.518, ve = 45) | Carl Zeiss Jena GmbH | 444960-0000-000 | |
| Inverted epifluorescence microscope | Olympus IX-71/73/83 | ||
| Laser 561 nm, 200 mW | CrystaLaser | CL-561-200 | 561 nm emission |
| Laser 642 nm, 140 mW | Omicron | Luxx-642-140 | 642 nm emission |
| MATLAB | MathWorks | version R2013a | |
| MEM with Earle's Balanced Salt Solution 2.2 g/L NaHCO3, stable glutamine w/o PR | Biochrom | FG-0385 | |
| MEM with Earle's Balanced Salt Solution with 2.2 g/L NaHCO3, stable glutamine, Phenolred | Biochrom | FG-0325 | |
| MitoTracker® Deep Red FM | Thermo Fisher Scientific | M22426 | dye |
| MitoTracker® Green FM | Thermo Fisher Scientific | M7514 | dye |
| Multi-mode-optical polarization maintaining monomode fiber | Pointsource/Qioptiq | KineFLEX | |
| NHS-PEG-MAL, Rapp Polymer | Rapp Polymere GmbH Tübingen | coverslip coating | |
| non-essential amino acids (NEAA) | Biochrom | #0802E | |
| PEG 800 (Polyethylene glycol) 10 % | Carl Roth GmbH | Art No. 0263.1 | coverslip coating |
| Penicillin/Streptomycin | Biochrom | #0122E | |
| Plasmid for PINK1-Halo7-Tag expression | Beinlich et al., ACS Chemical Biology 2015 | ||
| Poly-L-lysine (1.2 mg/ml) | Sigma-Aldrich & Co. | Cat. No.P9155 | coverslip coating |
| RGD Peptide (Ac-CGRGDS-COOH) | Coring System Diagnostix GmbH, Gernsheim | coverslip coating / Intergrin receptor motif | |
| Silicon Rhodamine linked to HaloTag®-Ligand (SiRHTL) | personal gift from Kai Johnson | dye | |
| Software analysis plugin | self-written C. P. Richter, Biophysik Osnabrück | SLIMFAST 16g | |
| Tetramethylrhodamine / SNAP-Cell® TMR-Star linked to SNAP-Ligand (TMRstar) | New England Biolab® | S9105S | dye |
| Tetramethylrhodamine linked to HaloTag®-Ligand (TMRHTL) | Promega | G8251 | dye |
| TIRF condensor | Olympus | Cell^TIRF MITICO System | TIRF condensor |
| TIRF microscope controlling software | Olympus cellSens 1.12 | ||
| TIRF objective | Olympus | 150x oil objective (N.A. 1.45; Olympus UAPO) | |
| Trypsin/EDTA 10x | Biochrom | #0266 | |
| Water H2O 99,5 % Rotipuran® Low organic | Carl Roth GmbH | Art. No. HN57.1 |
Referanslar
- Appelhans, T., Richter, C., Wilkens, V., Hess, S., Piehler, J., Busch, K. Nanoscale organization of mitochondrial microcompartments revealed by combining tracking and localization microscopy. Nano Letters. 12 (2), 610-616 (2012).
- Appelhans, T., Busch, K. Single Molecule Tracking and Localization of Mitochondrial Protein Complexes in Live Cells. Methods Mol Biol. 1567, 273-291 (2017).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Methods. 3 (10), 793-795 (2006).
- Gould, T. J., Verkhusha, V. V., Hess, S. T. Imaging biological structures with fluorescence photoactivation localization microscopy. Nat Protoc. 4 (3), 291-308 (2009).
- Pennacchietti, F., Gould, T. J., Hess, S. T. The Role of Probe Photophysics in Localization-Based Superresolution Microscopy. Biophys J. 113 (9), 2037-2054 (2017).
- Wegel, E., Göhler, A., Lagerholm, B. C., Wainman, A. Imaging cellular structures in super-resolution with SIM, STED and Localisation Microscopy: A practical comparison. Scientific reports. , Available from: https://www.nature.com/articles/srep27290?WT.feed_name=subjects_physical-sciences (2016).
- Pellett, P., et al. Two-color STED microscopy in living cells. Biomedical Optics Express. 2 (8), (2011).
- Ishigaki, M., et al. STED super-resolution imaging of mitochondria labeled with TMRM in living cells. Mitochondrion. 28, 79(2016).
- Liss, V., Barlag, B., Nietschke, M., Hensel, M. Self-labelling enzymes as universal tags for fluorescence microscopy, super-resolution microscopy and electron microscopy. Scientific Reports. 5, 17740(2015).
- Sergé, A., Bertaux, N., Rigneault, H., Marguet, D. Dynamic multiple-target tracing to probe spatiotemporal cartography of cell membranes. Nat Methods. 5 (8), (2008).
- Appelhans, T., Busch, K. B. Dynamic imaging of mitochondrial membrane proteins in specific sub-organelle membrane locations. Biophysical reviews. 9 (4), 345-352 (2017).
- Wilmes, S., et al. Triple-color super-resolution imaging of live cells: resolving submicroscopic receptor organization in the plasma membrane. Angewandte Chemie. 51 (20), 4868-4871 (2012).
- Niewidok, B., et al. Single-molecule imaging reveals dynamic biphasic partition of RNA-binding proteins in stress granules. J Cell Biol. , (2018).
- Wurm, C. A., Jakobs, S. Differential protein distributions define two sub-compartments of the mitochondrial inner membrane in yeast. FEBS Lett. 580 (24), 5628-5634 (2006).
- Schmidt, R., Wurm, C. A., Punge, A., Egner, A., Jakobs, S., Hell, S. W. Mitochondrial cristae revealed with focused light. Nano Lett. 9 (6), 2508-2510 (2009).
- Kukat, C., Wurm, C. A., Spahr, H., Falkenberg, M., Larsson, N. G., Jakobs, S. Super-resolution microscopy reveals that mammalian mitochondrial nucleoids have a uniform size and frequently contain a single copy of mtDNA. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13534-13539 (2011).
- Beinlich, F., Drees, C., Piehler, J., Busch, K. Shuttling of PINK1 between Mitochondrial Microcompartments Resolved by Triple-Color Superresolution Microscopy. ACS chemical biology. 10 (9), 1970-1976 (2015).
- Shim, S., et al. Super-resolution fluorescence imaging of organelles in live cells with photoswitchable membrane probes. Proceedings of the National Academy of Sciences. 109 (35), 13978-13983 (2012).
- Sbalzarini, I., Mezzacasa, A., Helenius, A., Koumoutsakos, P. Effects of Organelle Shape on Fluorescence Recovery after Photobleaching. Biophysical Journal. 89 (3), 1482-1492 (2005).
- Reits, E., Neefjes, J. From fixed to FRAP: measuring protein mobility and activity in living cells. Nature Cell Biology. 3 (6), E145-E147 (2001).
- Goehring, N., Chowdhury, D., Hyman, A., Grill, S. FRAP Analysis of Membrane-Associated Proteins: Lateral Diffusion and Membrane-Cytoplasmic Exchange. Biophysical Journal. 99 (8), 2443-2452 (2010).
- Bacia, K., Haustein, E., Schwille, P. Fluorescence correlation spectroscopy: principles and applications. Cold Spring Harbor protocols. 2014 (7), 709-725 (2014).
- Sukhorukov, V., Dikov, D., Busch, K., Strecker, V., Wittig, I., Bereiter-Hahn, J. Determination of protein mobility in mitochondrial membranes of living cells. Biochimica et biophysica acta. 1798 (11), 2022-2032 (2010).
- Kim, T. K., Eberwine, J. H. Mammalian cell transfection: the present and the future. Anal Bioanal Chem. 397 (8), 3173-3178 (2010).
- Graham, F. L., van der Eb, A. J. A new technique for the assay of infectivity of human adenovirus 5 DNA. Virology. 52 (2), 456-467 (1973).
- Wedeking, T., et al. Spatiotemporally Controlled Reorganization of Signaling Complexes in the Plasma Membrane of Living Cells. Small. 11 (44), 5912-5918 (2015).
- Mironov, S. L., Ivannikov, M. V., Johansson, M. [Ca2+]i signaling between mitochondria and endoplasmic reticulum in neurons is regulated by microtubules. From mitochondrial permeability transition pore to Ca2+-induced Ca2+ release. J Biol Chem. 280 (1), 715-721 (2005).
- Poot, M., et al. Analysis of mitochondrial morphology and function with novel fixable fluorescent stains. J Histochem Cytochem. 44 (12), 1363-1372 (1996).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells (vol 5, pg 159). Nature Methods. 5 (5), 455(2008).
- Elmokadem, A., Yu, J. Optimal Drift Correction for Superresolution Localization Microscopy with Bayesian Inference. Biophys J. 109 (9), 1772-1780 (2015).
- Barlag, B., et al. Single molecule super-resolution imaging of proteins in living Salmonella enterica using self-labelling enzymes. Sci Rep. 6, 31601(2016).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nature Methods. 5 (2), 159-161 (2008).
- Mortensen, K. I., Churchman, L. S., Spudich, J. A., Flyvbjerg, H. Optimized localization analysis for single-molecule tracking and super-resolution microscopy. Nat Methods. 7 (5), 377-381 (2010).
- Jin, S., Youle, R. J. PINK1- and Parkin-Mediated Mitophagy at a Glance. Journal of Cell Science. 125, 795-799 (2013).
- Yamano, K., Youle, R. J. PINK1 is degraded through the N-end rule pathway. Autophagy. 9 (11), 1758-1758 (2013).
- Wiedemann, N., et al. Machinery for protein sorting and assembly in the mitochondrial outer membrane. Nature. 424 (6948), 565-571 (2003).
- Thompson, R., Larson, D., Webb, W. Precise Nanometer Localization Analysis for Individual Fluorescent Probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır