Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Tasarım ve Reconfigurable DNA akordeon raf sentezi
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Biz tasarım, simülasyon, ıslak-laboratuvar deneyleri ve analiz için 6 6 kafesleri reconfigurable bir DNA akordeon raf için detaylı iletişim kuralı tanımlamak.
Özet
DNA nanostructure tabanlı mekanik sistemler veya 2D ve 3D karmaşık nano çekimde nanometre ångström çözünürlük için üretmek, DNA nanomachines büyük bir potansiyel nanoteknoloji ilaç dağıtım moleküler reaktörler gibi çeşitli alanları göster, ve nanoplasmonic sistemleri. Hangi topluca elemanları, 2D veya 3D nano ağ içinde birden çok sahne DNA girişlerine yanıt işleyebilirsiniz, reconfigurable DNA akordeon rack, açıklanmıştır. Platform DNA nanomachines bir ağ ölçeği ile birden fazla yeniden yapılandırılması aşamaları için birkaç öğeleri denetleyebilirsiniz öğelerin sayısını artırmak için bir potansiyele sahiptir.
Bu protokol için deneysel işlemin tamamı 6 6 kafesleri reconfigurable DNA akordeon raf açıklar. Protokol yapıları ve ıslak-Laboratuvar deney sentezi ve yeniden yapılandırılması için tasarım kuralı ve simülasyon yordam içerir. Buna ek olarak, TEM (transmisyon elektron mikroskobu) ve perde (floresan rezonans enerji transferi) kullanarak yapı analizi iletişim kuralında dahildir. Bu protokol için yeni tasarım ve simülasyon Yöntemler araştırmacılar DNA akordeon raf daha fazla uygulamaları için kullanmak için yardımcı olacaktır.
Giriş
2D ve 3D karmaşık nano çekimde nanometre için ürettikleri çünkü DNA nanoyapıların veya DNA nanomachines1,2,3,4,5 temel mekanik sistemler benzersizdir Ångström çözünürlük, göre çeşitli biyomoleküler uyaranlara2,3,6. Bu yapılar üzerinde fonksiyonel malzemeleri ekleyerek ve konumlarını kontrol, bu yapıların çeşitli bölgelerine uygulanabilir. Örneğin, DNA nanomachines moleküler reaktör7, uyuşturucu teslim8ve nanoplasmonic sistemleri9,10için önerilmiştir.
Daha önce biz hangi öğeleri11 (Şekil 1A) 2D veya 3D nano ağ işleyebilirsiniz reconfigurable DNA akordeon rack, tanıttı. Sadece bir kaç elemanları kontrol diğer DNA nanomachines farklı olarak, platform topluca düzenli aralıklarla dizilmiş 2D veya 3D öğeleri çeşitli aşamalarında üzerinde değişiklik yapabilir. Biz bir programlanabilir biyolojik ve kimyasal reaksiyon ağ veya moleküler bilgi işlem sistemi sistemimiz kontrol edilebilir öğelerin sayısını artırarak inşa olabilir tahmin. DNA akordeon raf birden fazla DNA kirişler ağ tek iplikçikli DNA'sını (Şekil 1B) oluşan eklemlere bağlandığı bir yapıdır. DNA kiriş tarafından üretilen akordeon raf kirişler yapışkan parçası melezlemek ve kilitleri (kilitli durum) köprüleme parçası uzunluğuna göre kiriş arasındaki açıyı değiştirmek DNA kilitleri tarafından yapılandırılır. Buna ek olarak, çok adımlı yeniden yapılandırılması DNA kilitleri çıkıntı tabanlı strand deplasman12,13arası ayırma tarafından yeni kilitleri serbest devlet oluşumu sonra ekleyerek gösterilmiştir.
Bu protokol için biz tüm tasarım ve sentez reconfigurable DNA akordeon raf süreci açıklanmaktadır. Protokol tasarım, simülasyon, ıslak-laboratuvar deneyleri ve analiz 6 6 kafesler DNA akordeon raf ve bunlar yeniden yapılanma sentezi için içerir. Protokol kapsamında yapısı önceki araştırma11 temel model ve 65 nm tarafından 65 nm boyutunda 14 demeti oluşan. Tasarım ve simülasyon açısından akordeon raf yapısal tasarımı geleneksel DNA origami14,15 (sıkı paketlenmişyani ) farklıdır. Bu nedenle, tasarım kural ve Moleküler Simülasyon geleneksel yöntemleri değiştirilmiştir. Göstermek için biz caDNAno14 tarihinde yaklaşım ve akordeon raf oxDNA16,17 ile ek komut dosyalarını kullanma simülasyonu kullanarak tasarım tekniği göstermek. Son olarak, yapılandırılmış akordeon raf yapıları çözümlenmesi için her iki iletişim kurallarını-TEM ve perde açıklanmıştır.
Protokol
1. tasarım 6 6 DNA akordeon raf caDNAno14 ile tarafından
- Bir DNA akordeon raf tasarımı için caDNAno 2.0 yazılım14 yükleyip (caDNAno 2.5 mevcuttur ayrıca https://github.com/cadnano/cadnano2.5 üzerinde). CaDNAno14 ' ü aç ve Kare aracı kare bir kafes ile yeni bir bölüm eklemek için tıklatın.
- Her ışın akordeon raf numarası ve caDNAno14 (Şekil 2) sol kafes paneli üzerindeki çizin.
- Kalem aracını tıklatın ve doğru Düzenleme panelindeki caDNAno14her ışın çizin. Mola kirişler her 32 bp, bitişik kirişler arasında eklem içindir. Zımba kesişim eklem aynı pozisyonda yerleştirin. Ekle aracı ve Kurşun kalem aracı caDNAno14 ' te ek tek iplikçikli kesişim var eklem bildirmek için kullanın.
- Kurşun Kalem aracını tıklatın ve eklem bağlayın. Her ışını yedi eklem vardır.
- İskele algoritması11yönlendirme daha önce raporlanmış iskele kullanarak tek döngü içine birleştirmek için iskele kesişim oluşturmak. 8 olmak en az bağlayıcı etki iskele ve elyaf iplikleri arasında izin vermeyin bp (Şekil 3).
- Kullanılmayan iskele akordeon raf karşı taraflarda bulunan köşeleri toplantıda Şekil 3' te gösterildiği gibi yerleştirin.
- Aracı Break' ı tıklatın. Dairesel veya 60'dan daha uzun nerede zımba iplikçikleri ayrılmaktadır kırmak bp.
- DNA kilit dizilerini tasarlayın.
- Aracı Break' ı tıklatın. Yapışkan bir parçası yapmak ve 8 silmek için bir elyaf DNA bölgesinin sonu 8 bp bp zımba DNA bölge. 6 6 akordeon raf 18 yapışkan bölümden (Şekil 1) vardır.
- Ters sıralarını kilit ayrılmaktadır her iki ucunda yapışkan bölümlerine tamamlayıcı yere takıp onları istenilen uzunlukta (Şekil 1B) poli T ipliklerini oluşan bir köprüleme bölgeye göre bağlayın.
- Yeniden yapılandırılması için 8 add bp DNA sonunda çıkıntı sıralarının kilitler için strand çıkarma. Kullanılan çıkıntı Tablo 2' de dizisidir.
- Köprüleme bölgesine tamamlayıcı ters poli A iplikçikleri hazırlayın.
- Yeniden yapılandırılması deney DNA kilitlerine için tamamlayıcı ters tasarım ayrılmaktadır.
- Sıra aracını tıklatın ve iskele DNA'ı tıklatın. Standart M13mp18. Verme aracı tıklatın ve sıra (Tablo 1) csv biçiminde kaydedin iskele seçin.
2. oxDNA yapısıyla simülasyonu
- Karşıdan yükleyip oxDNA16,17. En son kaynak kodu https://sourceforge.net/projects/oxdna/files/ üzerinde kullanılabilir.
- Python komut dosyası 'cadnano_interface.py' oxDNA16,17 paketinde sağlanan, kullanarak caDNAno14 dosyasını başlangıç yapılandırma dosyalarından olun. Kullanımı aşağıdaki gibidir: 'python cadnano_interface.py cadnano_file.json sq'. Topoloji dosya ve yapılandırma dosyası şimdi oluşturulur.
Not: Kaç iplikçikleri topoloji dosya içerir ve nükleotid yapısı ve omurga-omurga nükleotitler arasındaki bağları ile ilgili bilgi bulunmaktadır. Yapılandırma dosyası timestep, enerji ve kutusu boyutu gibi genel bilgileri içerir. Oryantasyon pozisyon vektör, omurga tabanlı vektör, normal vektör, hız ve nükleotid açısal hızı gibi de dahil (Şekil 4) bilgilerdir. - Topoloji ve yapılandırma dosyanızdaki akordeon raf çok yapısal bilgileri yansıtacak olmaları için caDNAno14 değiştirin. CaDNAno14 topoloji ve yapılandırma dosyaları görüntülenir zaman tüm kirişler paralel olarak düzenlenmiştir. Ancak, akordeon raf bir kafes yapısı bu nedenle gümrüklü nükleotitler arasındaki mesafe kadar simülasyon (Şekil 5).
- Döndürme ve istenen kafes yapısı her ışın taşıyın. Yapılandırma dosyasında soldaki dokuz sütun konumu vektör, omurga tabanlı vektör ve normal vektör (Şekil 4) vardır. Bir ışın döndürmek için tüm konum, omurga-base ve normal vektörel çizimler dönme dönüştürme kullanarak döndürün. O zaman bir ışın Şekil 5' te gösterildiği gibi bulmak için konum vektör değiştirerek taşıyın.
- OxDNA paketinde sağlanan komut dosyasını kullanarak yapı sakin ol ($oxDNA örneğe bakın/örnekler/RELAX_INITIAL_CONFIGURATION için daha fazla bilgi).
- Moleküler dinamiği simülasyon rahat yapılandırma dosyası kullanarak on milyon adımları için çalıştırın. Kullanımı aşağıdaki gibidir: '. / oxDNA < giriş >' veri her 5000 veya 10000 adımları kaydetmek.
- Görselleştirme
Not: Cogli kullanarak yapıları görüntülenir.- Download ve cogli (https://sourceforge.net/projects/cogli1/) en son sürümünü yükleyin.
- Cogli topoloji ve yapılandırma dosyaları ile oxDNA benzetim çalıştırın. Kullanımı aşağıdaki gibidir: '. / cogli1 -t < topoloji Dosya >< yapılandırma dosyası >'.
- Btuşuna basarak kutusunu gizlemek.
3. yapı sentezi
Not: Sentez yöntemi önceki iletişim kuralı15,18uyarlanmıştır.
- Tasarlanmış DNA zımba bir oligonükleotid sağlayıcısından satın.
- Bu DNA zımba 100 mikron nükleaz ücretsiz su kullanarak için konsantrasyon ayarlayın.
- 'Devlet ücretsiz' yapısı bir tüp oluşturan her DNA strand havuz ve konsantrasyon için her iplikçik 2 mikron için ayarlayın.
- Havuzu DNA kilit uzunluğu ve kilit sitelerin sayısı tarafından tüpler içine ayrılmaktadır ve konsantrasyon için her iplikçik 2 mikron için ayarlayın. 18, 9 ve 4 kilit siteleri kullanılır. Aynı konsantrasyonu, köprüleme bölgesine tamamlayıcı poli A iplikçikleri ekleyin.
- Tamamlayıcı DNA için ters havuzu iplikçikleri ayrılmaktadır boyla tüpler içine kilitle ve konsantrasyon için her iplikçik 2 mikron için ayarlamak.
- MgCl2 çözüm 300 hazırlamak 70 μL nükleaz ücretsiz su ve 1 mikron MgCl2 çözüm 30 μL karıştırma tarafından nM. 5 Tris EDTA çözüm x 95 μL nükleaz ücretsiz su ve 100 x Tris EDTA çözüm 5 μL karıştırarak hazırlayın.
- Zımba DNA, MgCl2 çözüm 1.1 μL 2 μL, Tris EDTA çözüm, nükleaz ücretsiz su 7.6 μL ve konsantrasyonu 110 olduğu iskele DNA 7.3 μL 2 μL ekleyin nM 20 μL karışık stok yapmak. İskele DNA son konsantrasyonu 40'a ayarla nM, DNA zımbala 200 nM, MgCl2 ' ye 16 mM ve Tris EDTA çözüm 0.5 x.
- Hızla 4 ° c ° C. başına 40 dk hızında 60 ° c ile 60 ° C ° C ve serin başına 4 dk hızında 80 ° c termal cycler ve serin karışık hisse senedi çözümde ısı
4. arıtma yapısı
Not: Tüm yapıları örnekleri analiz daha önce saf. Bu bölümde, önceki edebiyat19uyarlanmıştır PEG arıtma protokolü açıklar. Örnek ayrıca jel elektroforez tarafından önceki edebiyat15,18' açıklandığı gibi saf.
- 5 M NaCl ve 100 x Tris-EDTA hazırlayın.
- Yağış-tampon 8000, PEG 500 μL Tris EDTA x 100 ve 101 μL 5 M NaCl ve 249 μL nükleaz ücretsiz su 150 μL karıştırarak hazırlayın.
- Hedef-tampon Kısım 3.3 300 nM MgCl2 çözümden 5.5 μL, 5 x Tris EDTA çözümden Kısım 3.3 10 μL ve 84,5 μL nükleaz ücretsiz su karıştırılarak hazır olun.
- Bölüm 3 ve yağış-Bölüm 4.2 arabelleğinden 20 μL sentezlenmiş yapısının Mix 20 μL. Karışık hisse senedi 16000 x g 4 ° C'de te spin Süpernatant kaldırmak ve Pelet Bölüm 4.3 hedef arabelleğinden içinde çözülür.
5. yeniden yapılanma 'Devlet kilitli' için bir 'devlet' akordeon raf
- DNA kilitleri yapılandırma deneme için olmadan yapı sentez.
- DNA kilit dizilerini Bölüm 3 hazırlayın.
- İstenilen uzunlukta DNA kilit iplikçiklerinin 2 μL sentezlenmiş yapısı 20 μL ekleyin. DNA kilit dizilerini konsantrasyon yapısı beş kat daha yüksek.
- Örnek 0, 10, 25, 50 için kuluçkaya veya ne kadar hızlı yeniden yapılandırılması görmek için 100 dakika oluşur.
- 100 dakika kuluçka için örnek 50 ° C'de 30 dakika boyunca kuluçkaya ve 25 ° C de 0,33 ° C/dakika'dan başlayarak bir oranda aşağı yavaş yavaş serin.
- 50 dakika kuluçka için örnek 50 ° C'de 15 dakika boyunca kuluçkaya ve 25 ° C de 0,66 ° C/dakika'dan başlayarak bir oranda aşağı yavaş yavaş serin.
- 25 dakika kuluçka için örnek 50 ° c için 7.5 dakika kuluçkaya ve 25 ° C de 1.32 ° C/dakika'dan başlayarak bir oranda aşağı yavaş yavaş serin.
- 10 dakika kuluçka için örnek 50 ° c için 1 dakika kuluçkaya ve 25 ° C de 3.3 ° C/dakika'dan başlayarak bir oranda aşağı yavaş yavaş serin.
- DNA kilitleri dizilerini eklendikten sonra 0 dakika kuluçka için örnek 4 ° C sağ saklayın.
- Şu takma adım sonra hızla örnek ile 4 ° C arasında istenmeyen denatürasyon önlemek için sakin.
6. yeniden yapılanma bir 'devlet bir 'devlet için' kilitli' dan akordeon raf
- DNA kilitleri yapılandırma deneme için istenilen uzunlukta yapısıyla sentez.
- Bölüm 3 ters tamamlayıcı iplikçikleri hazırlayın.
- İstenilen uzunlukta kilit ipliklerini için tamamlayıcı ters sentezlenmiş yapısı 20 μL iplikçiklerinin 2 μL ekleyin. DNA kilit dizilerini konsantrasyon yapısı beş kat daha yüksek.
- Örnek 0, 12, 60, 120 için kuluçkaya, ne kadar hızlı yeniden yapılandırılması görmek için 240 dakika oluşur.
- 12, 60, 120, 240 dakika kuluçka, hızla örnek 40 ° c ısı ve yavaş yavaş her birine karşılık gelen kez aşağı 20 ° C serin. Şu ayırmayı adımdan sonra hızla örnek ile 4 ° C arasında istenmeyen denatürasyon önlemek için sakin.
- Geriye doğru tamamlayıcı iplikçikleri eklendikten sonra 0 dakika kuluçka için örnek 4 ° C sağ saklayın.
7. TEM görüntüleme
Not: TEM görüntüleme Protokolü önceki edebiyat18,20uyarlanmıştır.
- 1.25 M NaOH çözüm 87,5 μL nükleaz ücretsiz su ve 10 M NaOH çözüm 12.5 μL karıştırarak hazırlayın.
- 1.25 M NaOH çözüm 1 μL % 2 uranyl Format çözüm 50 μL için ekleyin.
- Girdap 3 dakika ve 2 dakika için maksimum hızda santrifüj için çözüm. Arıtılmış örnek 3 μL kızdırma taburcu TEM ızgara üzerinde 3 dakika boyunca mevduat ve hızlı bir şekilde filtre kağıdı ile yıkayın.
- Hazır uranyl Format çözüm 7 μL 30 saniyeliğine mevduat ve hızlı bir şekilde filtre kağıdı ile yıkayın.
- Uzunluk ve açı tarafından TEM görüntüsü akordeon yapısının ölçmek.
8. perde Analizi
- ATTO 550 ve Förster mesafe 6.5 olduğu sınav 647N boya kullanın nm. Zımba 58 ve elyaf 117 Tablo 1 ' deki fluorescently etiketli lifler ile değiştirin. O zaman fluorescently etiketli iplikçikleri yapısıyla Bölüm 3'te açıklanan yöntemi tarafından sentez.
- Arıtılmış örnek konsantrasyonu ölçmek.
- 10 nM ve yük 50 μL 384 Mikroplaka için örneğine de normalleştirmek.
- Donör ve alıcısı boyalar 550 nm ve ölçü birimi ile örnek heyecanlandırmak floresans spektrum üzerinden 570 nm 800 nm bir yer ile.
- Sadece donör örnek floresans spektrumu aynı şekilde ölçün.
- Örneği 650'boyalar heyecanlandırmak nm ve floresan spektrum ve 670 ölçü nm 800 nm. Alıcısı konsantrasyonunu ölçmek için bu.
- Standart sapmalar sentezlenmiş ve ayrı ayrı saf üç örnekleri ile aynı deneyi tekrar ederek elde edilir.
- PERDE verimlilik oranı21aşağıda denklemi tarafından açıklandığı gibi bir yöntem ile hesaplayın.
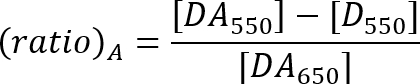
DA550: donör ve 550 nm uyarma, alıcısı ile örnek alıcısı tepe floresan yoğunluğu.
D550: floresan yoğunluğu salt donör örneği 550 nm uyarma'alıcısı emisyon mesafeden.
DA650: alıcısı tepe floresan yoğunluğu ile donör ve alıcısı 650 nm uyarma, örnek.
Sonuçlar
Tarafından tasarlanmış 6 6 DNA akordeon raf oxDNA16,17 benzetimli ve sonuçları Şekil 6' da gösterilmiştir. Simülasyon sonucu o amaçlanan yapısı yapısı bozulma oluşturulur doğrulandı.
Şekil 7 TEM Albümdeki yapılandırılmış yapıları ile 2, 8, 13 ve 20 bp kilit uzunluğunu görüntülerdir....
Tartışmalar
Bu iletişim kuralı tüm süreç tasarımı, simülasyon, sentez ve analiz temel 2D DNA akordeon raf tanıtır. Standart DNA origamisi, tasarım kuralı farklıdır çünkü DNA akordeon raf kesişim esneklik14,15, ek nükleotit yok modifiye tasarım ve simülasyon kuralları tarif edilmistir. Bu, protokol DNA akordeon raflar kullanarak çeşitli araştırmalar hızlandırabilir bekliyoruz. Buna ek olarak, açıklanan protokolü de standart DNA origamisi yerine ...
Açıklamalar
Yazarlar ifşa yoktur
Teşekkürler
Bu araştırma kısmen küresel araştırma geliştirme merkezi Program aracılığıyla Ulusal Araştırma Vakfı, Bilim Bakanlığı ve ICT (MSIT) (2015K1A4A3047345) ve Nano· tarafından finanse edilen Korea(NRF) tarafından desteklenmiştir Malzeme teknolojisi geliştirme programı aracılığıyla Ulusal Araştırma Vakfı, Kore (Bilim Bakanlığı ve ICT (MSIT) (2012M3A7A9671610) tarafından desteklenen NMK). Seul Ulusal Üniversitesi'nde Mühendislik Araştırma Enstitüsü araştırma tesisleri bu iş için sağlanan. Yazarlar Tae-genç Yoon (Biyolojik Bilimler, Seul Ulusal Üniversitesi) doğru perde analizi için şükran floresans spektroskopisi ile ilgili olarak kabul.
Malzemeler
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
Referanslar
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -. G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127 (2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102 (2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101 (2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272 (2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486 (2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446 (2018).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır