Method Article
Stres pozlama ile ilişkili epigenetik değişiklikleri tanımlamak için bir fare metil-Seq Platform
Bu Makalede
Özet
Burada, protokolü ve metil-Seq, kronik stres pozlama ile ilişkili epigenetik değişiklikleri tanımlamak için bir fare modeli kullanarak bir epigenomic platform uygulanması açıklanmaktadır. Sonuçları sıçan metil-Seq platformu sıçanlarda stres maruz kaynaklanan metilasyonu farklılıklar tespit kapasitesine sahip olduğunu göstermektedir.
Özet
Hayvanlar daha çeşitli genleri kullanıma sunulduğunda bu hayvan modellerinde dinamik epigenetik değişiklikleri yakalayabilir araçlar için artan bir ihtiyaç vardır. Sıçan nerede epigenetik bir araç anlayışlı mekanik bilgi sağlamak için birçok farmakolojik ve davranışsal çalışma tamamlayıcı bir modelle hayvandır. Bu amaçla, DNA metilasyonu düzeyleri arasında fare genom değerlendirebilir fare için SureSelect hedef yakalama (metil-Seq adlandırılır) sistemi adapte. Sıçan Tasarım Rehberleri, CpG adaları, ada sahilleri ve tüm RefSeq genler GC-zengin bölgelerden hedef.
Platform bir sıçan deney uygulamak için erkek Sprague Dawley rat için 3 hafta sonra genomik DNA ekstraksiyon için kan örnekleri toplanmıştır, kronik değişken strese maruz bırakıldı. Metil-Seq kitaplıkları sıçan DNA örneklerinden kesme, bağdaştırıcı ligasyonu, hedef zenginleştirme, bisülfit dönüşüm ve çoğullama tarafından inşa edildi. Kitaplıkları bir nesil sıralama platformunda sıralı ve sıralı okuma DMRs DNA vurguladı ve vurgusuz sıçan arasında tanımlamak için analiz edildi. En iyi aday DMRs bağımsız olarak platform sağlamlık onaylamak için bisülfit pyrosequencing tarafından onaylanmış.
Sıçan metil-Seq platformu strese maruz tarafından indüklenen metilasyonu değişiklikleri yakalayabilir yararlı bir epigenetik araç olduğundan sonuçlar gösterilmektedir.
Giriş
Yüksek işlem hacmi sıralama gelişmeler genom dizileri model ve model organizmalar için çok sayıda yol açmıştır. Böyle dizileri kullanılabilirliğini genetik, karşılaştırmalı genomik ve transcriptomics araştırma kolaylaştırdı. Örneğin, kullanılabilir genom dizileri ile histon değişiklikler1veya bisülfit sıralama, dernek göre DNA zenginleştirmek ChIP-Seq deneyler sıralama verileri hizalama için son derece yararlı olan DNA metilasyonu tarafından ölçer urasil algılama bazlar cytosines2bisülfit dönüşümden kurdu. Ancak, kendi tasarım gen işlevini etkileyebilir species-specific düzenleyici sıralarının ek açıklama eklenen veri eksikliği nedeniyle kullanılabilir genomik sıralama veri epigenomic platformlarının uygulamasında gecikmeler olmuştur.
Özellikle, DNA metilasyonu methylomic platformu oluşturmak için kullanılabilir genomik veri kullanan bir DNA üzerinde en çok çalışılmış epigenetik değişikliklerin biridir. Böyle bir örnek için yaygın olarak çeşitli disiplinlerde Onkoloji üzerinden psikiyatri4,5kullanılan insan methylome3dizi tabanlı bir platformdur. Neredeyse genomik sıra kendi ilk tasarım yararlanmaktadır hiç yaygın olarak kullanılan platformlar gibi ne yazık ki, insan olmayan hayvan modelleri için benzer platformlar, azdır.
İnsan olmayan hayvan modellerin methylomic manzara değerlendirmek için kullanılan genel yöntem azaltılmış temsil bisülfit (RRBS)6sıralama var. Bu yaklaşım sağlayan, kapsamlı methylomic manzara sağlarken daha düşük okuma derinlemesine kapsama maliyet ve genom2 büyük gen-yoksul bölgelerde sınırlı fonksiyonel bilgi nedeniyle bütün-genom bisülfit sıralama maliyetini üstesinden gelir. . RRBS kısıtlama Özet ve boyutu-son derece GC-zengin serileri gibi yaygın olarak gen Rehberleri buldum ve gen Yönetmeliğin7' bir rol oynamaya düşündüm CpG adaları için zenginleştirmek için genomik DNA yelpazesi içerir. RRBS yöntemi önemli çalışmalar bir dizi olarak kullanılan iken enzimleri üzerindeki güven önemli sorunlar ve sınırlamalar değildir. Örneğin, zenginleştirme GC zengini diziler RRBS içinde tamamen belirli dizileri Restriksiyon enzimi ve sonraki boyutu seçimi tarafından Elektroforez tarafından tanınan varlığı bağlıdır. Böylelikle bu kısıtlama siteler içermeyen birisi genomik alan boyutu seçimi sırasında hariç tutulur. Aynı kısıtlama siteleri farklı türler arasında aynı loci mevcut sürece Ayrıca, türler arası karşılaştırmalar zorlu.
RRBS sınırlamaları aşmak için bir platform tasarımında yayımlanmış genomik sırayı yararlanan bir zenginleştirme yöntemi kullanmaya yaklaşımdır. Dizi tabanlı insan platformu astar probları alleli özgü (CG TG bisülfit dönüştürme sonra) hedef tavlama ve astar uzantısı için belirli CpGs karşı tasarlanmış kullanır. Tasarım sadece mevcut insan genom dizisi, ancak düzenleyici bölgeleri deneysel olarak doğrulanmadı soruşturma, kodla ve ENSEMBL8gibi birden fazla satır elde yansıtır. İnsan methylomic araştırmalarda geniş kullanımı rağmen benzer bir platform modeli hayvanlar için mevcut değil. Ayrıca, dizi tabanlı biçimi önemli kısıtlaması sonda yerleşim için kullanılabilir yüzey alanı yerleştirir. Son birkaç yıl içinde çabaları yakalama sonda tasarım ve yeni nesil sıralama yüksek üretilen iş özelliği tarafından tanınan hedef-özgüllük birleştirmek için yapılmıştır. Böyle bir çaba metilasyonu9,10beyin özgü veya glukokortikoid kaynaklı farklılıkları tanımlamak için kullanılan fare genom (fare metil-Seq) için hedef dayandırılmış zenginleştirme sistemi sonuçlandı. Diğer model ve model olmayan hayvanlar için benzer platformlar epigenomic araştırma bu hayvanlarda kolaylaştırmak için ihtiyaç vardır.
Burada, fareyi üzerinde methylomic analizi yapmak için bu yeni platform uygulanması gösterilmektedir. Fareyi Farmakoloji, metabolizma, Nöroendokrinoloji ve davranış önemli bir hayvan model olarak hizmet vermiştir. Örneğin, ilaç zehirlenmesi, obezite, stres tepkisi veya uyuşturucu bağımlılığı ortaya çıkmasına temel mekanizmaları anlamak için artan bir ihtiyaç vardır. Bu koşullar ile ilişkili methylomic değişiklikleri esir alma yeteneğine sahip bir yüksek-den geçerek platform mekanizmaları anlayışımızı artış olacaktır. Sıçan genom hala düzenleyici bölgeleri için ek açıklama yoksun olduğundan, biz dahil olmayan rehberleri, CpG adaları, ada sahilleri11ve GC zengini diziler sıçan metil-Seq platformu12içine daha önce tanımlanmış.
Başarılı tasarımı ve uygulanması için fare genom SureSelect hedef zenginleştirme (genel olarak metil-Seq da adlandırılır) platform değerlendirmek için bir fare modeli differentially metillenmiş tanımlamak için kronik değişken stres (CVS)13 istihdam bölgeler arasında vurgusuz ve stresli hayvanlar. Bizim platform tasarımı, protokolü ve uygulama bir kapsamlı ve tarafsız epigenetik kimin genomik dizisi zaten mevcuttur ancak kötü açıklamalı kalır bir organizma üzerinde araştırma isteyebilirsiniz müfettişler için yararlı olabilir.
Protokol
Tüm deneyler uygun olarak ve kurumsal hayvan bakım ve kullanım komite toplantısında Johns Hopkins Tıp Fakültesi de dahil olmak üzere tüm ilgili yasal ve kurumsal yönergeleri, uyum içinde tamamlandı.
1. hayvanlar
- Erkek ergen Sprague-Dawley rat 4 haftalık edinin. Bir sıcaklık Polikarbonat sıçan kafeslerde hayvanlar ev- ve nem kontrollü oda bir 12 h ışık, 12 h karanlık döngü ile 0600 h., ışık başlangıçlı üzerinde ad libitum access su hayvanlarla sağlamak.
- Fareler ile ilgili stres azaltmak 1 hafta alışmana izin. Çift-ev hayvanları (N = 16) yalıtım stres engel ve kronik değişken 5 hafta-in yaş, başlamak için 3 hafta boyunca (CVS) rejimi stres.
2. kronik değişken stres
- CVS rejimi sabah (9-11: 00) bir kez ve bir kez öğleden sonra (1-3 PM) düzensiz zaman rutin öngörülemeyen tutmak için yönetmek. Gecede hafif stres dahil. CVS rejimi içerir: 1) 3 h bir kısıtlama silindir; 2) 10 dk Yüzme; 3) 3 h kafes eğimli 4) 1 h platformu; sallayarak yavaş ve 5) 4 ° C soğuk odada 1 h.
Not: Gece stresler (5-kafes) sosyal dışlama etkisi, sosyal izolasyon, ıslak yatak, Gıda kısıtlama içerir ve ışıklar açıkken. Stres rejimi bir normal haftalık zamanlama Tablo 1' de verilmiştir.
3. endokrin deneyleri
-
Kuyruk kan (~ 50 mL) dağıtımları kullanarak corticosterone (CORT) düzeyleri temel hormon düzeyleri (0 gün), bir kez haftalık CVS ortasında sırasında kurmak için önceden CVS rejimi için aynı zamanda (9 AM) deneme boyunca haftada iki kez toplanan belirlemek (gün 4,11 ve 18), CVS her 7 gün sonra (gün 7 ve 14) ve CVS (21 gün) ve sonuç. Günlük stres alay önce kan örnekleri toplamak.
- Bir son gövde kan örneği ötenazi (gün 25) sırasında RIA ve genomik DNA ekstraksiyon için toplamak.
- Tüm kan örnekleri (600 x g, 4 ° C, 10 dk) plazma kan hücrelerinden ayırmak için santrifüj kapasitesi. Damlalıklı plazma (süpernatant) dışarı ve örnekleri-80 ° C'de depolayın
- Çözülme ve plazma radioimmunoassay (RIA) tarafından CORT düzeylerini belirlemek için kullanın. 3 hafta plazma CORT düzeyleri stres rejimi sağlamlık doğrulamak için stresli hayvanlarda yüksek emin olun.
4. davranış
- CVS rejimi sonra (gün 23-24), her hayvan için yüksek anksiyete gibi davranış değerlendirmek artı labirent (EPM)14.
- Bir video kamera kaydı ile hayvanlar üzerinde 300 s ve zaman ortasında, harcanan puanı için EPM aleti silah kapalı ve açık kollar açık.
5. fare metil-Seq tasarımını
- UCSC genom tarayıcı kullanarak, yedek genomik koordinatları (sıçan Kasım 2004 rn4 derleme) CpG adaları ve ada sahilleri (± 1 CpG adaları kanat kb), organizatör (± 1 kb her TSS ') her RefSeq gen ve üzerinden kullanılabilir olan diğer sahneleri için elde ilgili belgeleri.
Not: fare metil-Seq için önceki bir dizi tabanlı metilasyonu platform üzerinden ek GC zengini diziler eklendi12. 5 kbps büyük bölgeleri için 500 bps alternatif bölgelerinde 1 kbps tarafından atlandı takip örneklenmiş. Son fare metil-Seq tasarım 111 Mbps, 2.3 milyon CpGs oluşur; ve 594 bps bir ortalama bölge boyutu. 228,800 benzersiz loci hedefler. - Genomik koordinatları derlenmiş bir liste için uygun prob tasarım bir piyasada bulunan hedef yakalama tasarım yazılımı girin.
6. genomik DNA sıçan metil-Seq kitaplığından inşaatı
Not: toplu iş etkileri ortadan kaldırmak için aynı anda birden fazla örnekleri işleme ve buna göre ana karışımları kadar ölçek. Piyasada bulunan bir DNA ekstraksiyon kiti kullanılarak DNA ayıklayın. Yöntemleri her iki sütun veya yağış alan verim yüksek kaliteli genomik DNA (260/280 oranı ~ 1.8). Fenol dayalı yöntemlerin kullanımı tavsiye edilmez. Elute veya düşük TE arabellekte (10 mM TE, 0,1 mM EDTA, pH 8.0) DNA resuspend.
- Numune hazırlama
Not: DNA'ya bağlanıcı kullanarak her adım için manyetik boncuklar, boncuklar için oda sıcaklığında en az 30 dk için acclimated ve iyi karışık kullanmadan önce emin olun.- DNA kesme
- Bir yer her örneğinin ilk çift iplikçikli DNA konsantrasyonu belirlemek için kullanın. Seyreltik > gDNA düşük TE arabelleği (10 mM TE, 0,1 mM EDTA, pH 8.0) düşük DNA'ya bağlanıcı microcentrifuge tüpler ile 50 µL için 1 µg.
- İzotermal bir sonicator kullanarak örnekleri kesme (% 10 iş hacmi, 5 yoğunluğu, 200 devir-veri bloğu, 6 döngüleri 60 s, frekans süpürme, 4 ° C).
- DNA boyutu ve miktar ölçer Elektroforez tabanlı sistemi kullanılarak DNA kalitesini değerlendirmek.
Not: önerilen DNA 1 µg veya 3 µg tutardır. Malzeme başlayan sınırlı varsa, en düşük giriş tutarı olmalıdır > 500 ng, daha düşük miktarlarda olumsuz etkiler miktar ve kalitesini oluşturulan kitaplıkları.
- Onarım DNA biter.
- Buz üstünde son-onarım Master Mix hazırlamak için fareyi metil-Seq seti kullanın. Her örnek için 52 µL karışımı ekleyin ve ısıtmalı kapak (için 30 dk, 4 ° C tutun 20 ° C) olmadan bir termal cycler kuluçkaya.
Son onarım Master Mix (örnek) başına:
Su 35.2 µL
Son onarım arabelleği 10 µL (10 x)
DNTP Mix 1.6 µL
T4 DNA polimeraz 1 µL
Klenow DNA polimeraz 2 µL
T4 polinükleotit kinaz 2.2 µL - 180 µL DNA'ya bağlanıcı manyetik boncuklar ve taze hazırlanmış % 70 etanol örnek başına 400 µL kullanarak örnekleri arındırmak. Her örnek için 180 µL boncuk ekleyin ve oda sıcaklığında 5 min için kuluçkaya. Boncuk cips, süpernatant kaldırmak ve Pelet % 70 etanol 200 µL içinde resuspend. Etanol kaldırmak ve yıkama bir kez yinelenir.
- Manyetik bir tabak boncuk cips ve mümkün olduğunca çok etanol kaldırmak için kullanın. Kuru bir 37 ° C olarak heatblock 3-5 dk kadar boncuk Pelet tamamen kuru. Nükleaz ücretsiz su 44 µL içinde resuspend ve yaklaşık 42 µL süpernatant ile toplamak.
Durdurma noktası: tamir DNA sona erdikten sonra örnekleri olabilir mühürlü ve -20 ° C'de depolanan
- Buz üstünde son-onarım Master Mix hazırlamak için fareyi metil-Seq seti kullanın. Her örnek için 52 µL karışımı ekleyin ve ısıtmalı kapak (için 30 dk, 4 ° C tutun 20 ° C) olmadan bir termal cycler kuluçkaya.
- Adenilat 3' biter.
- Adenylation Master Mix buzda hazırlayın. Her örnek için 9 µL karışımı ekleyin ve ısıtmalı kapak (37 ° C 30 dk, 4 ° C tutmak için) olmadan bir termal cycler kuluçkaya.
Adenylation Master Mix (örnek) başına:
Klenow arabellek 5 µL
DATP 1 µL
Klenow DNA polimeraz 3 µL - 90 µL DNA'ya bağlanıcı manyetik boncuklar ve taze hazırlanmış % 70 etanol örnek başına 400 µL kullanarak örnekleri arındırmak. Her örnek için 90 µL boncuk ekleyin ve oda sıcaklığında 5 min için kuluçkaya. Boncuk cips, süpernatant kaldırmak ve Pelet % 70 etanol 200 µL içinde resuspend. Etanol kaldırmak ve yıkama bir kez yinelenir.
- Manyetik bir tabak boncuk cips ve mümkün olduğunca çok etanol kaldırmak için kullanın. Kuru bir 37 ° C olarak heatblock 3-5 dk kadar boncuk Pelet tamamen kuru. Nükleaz ücretsiz su 35 µL içinde resuspend ve yaklaşık 33,5 µL süpernatant ile toplamak.
- Adenylation Master Mix buzda hazırlayın. Her örnek için 9 µL karışımı ekleyin ve ısıtmalı kapak (37 ° C 30 dk, 4 ° C tutmak için) olmadan bir termal cycler kuluçkaya.
- Metillenmiş bağdaştırıcı ligate.
- Tüp ligasyonu Master Mix buz üzerinde hazırlamak ve her örnek için 16,5 µL karışımı ekleyin. Isıtmalı kapak (15 dakika, 4 ° C tutun 20 ° C) olmadan bir termal cycler kuluçkaya.
(Örnek) başına ligasyonu Master Mix:
Su 2.5 µL
Metil-Seq 2.5 µL bağdaştırıcısı metillenmiş
10 µL T4 DNA ligaz arabelleği (5 x)
T4 DNA ligaz 1,5 µL - 90 µL DNA'ya bağlanıcı manyetik boncuklar ve taze hazırlanmış % 70 etanol örnek başına 400 µL kullanarak örnekleri arındırmak. Her örnek için 90 µL boncuk ekleyin ve oda sıcaklığında 5 min için kuluçkaya. Boncuk cips, süpernatant kaldırmak ve Pelet % 70 etanol 200 µL içinde resuspend. Etanol kaldırmak ve yıkama bir kez yinelenir.
- Manyetik bir tabak boncuk cips ve mümkün olduğunca çok etanol kaldırmak için kullanın. Kuru bir 37 ° C olarak heatblock 3-5 dk kadar boncuk Pelet tamamen kuru. Nükleaz ücretsiz su 22 µL içinde resuspend ve yaklaşık 22 µL süpernatant ile toplamak. Bir bioanalyzer kullanarak kalitesini değerlendirmek.
Not: DNA'ın toplam tutarı 500 ng, yamultma ve işlem ek DNA sonraki adımlara geçmeden önce olup olmadığını. Ortalama DNA boyutu daha--dan 30 bps tarafından artırmaz, reaktifleri yeni, T4 DNA polimeraz, Klenow ve/veya T4 ligaz eski olabilir emin olmak için denetleyin.
Durdurma noktası: metillenmiş bağdaştırıcısı'birleştirilmesi (ligasyon) sonra örnekleri olabilir mühürlü ve -20 ° C'de depolanan
- Tüp ligasyonu Master Mix buz üzerinde hazırlamak ve her örnek için 16,5 µL karışımı ekleyin. Isıtmalı kapak (15 dakika, 4 ° C tutun 20 ° C) olmadan bir termal cycler kuluçkaya.
- DNA kesme
- Hibridizasyon
- Örnekleri düşük DNA'ya bağlanıcı microcentrifuge tüpler için transfer edecek ve ısıtmalı vakum yoğunlaştırıcı az 3.4 µL. Reconstitute örnekleri 3.4 µL için numune hacmi azaltmak için.
Not: Örnekleri örnekleri tüm sıvı önce vakum yoğunlaştırıcı kaldırılır emin olmak için yaklaşık ~ 3 µL için konsantre buharlaşır. - Hibridizasyon arabellek oda sıcaklığında ve metil-Seq blok Mix buzda hazırlayın. Her örnek için 5.6 µL metil-Seq blok karışımı ekleyin ve termal cycler (95 ° C 5 min, 65 ° C için 2 dk, 65 ° C tutmak için) kuluçkaya.
Hibridizasyon arabellek (örnek) başına:
Metil-Seq Hyb 1 6.63 µL
Metil-Seq Hyb 2 0,27 µL
Metil-Seq Hyb 3 2,65 µL
4 metil-Seq Hyb 3.45 µL
Metil-Seq blok Mix (örnek) başına:
Metil-Seq dizin oluşturma blok 1 2.5 µL
Metil-Seq blok 2 2.5 µL
Metil-Seq blok 3 0.6 µL - RNase blok Mix ve Kütüphane yakalama hibridizasyon karışımı hazırlayın. Her örnek için 20 µL yakalama Kütüphane hibridizasyon karışımı ekleyin ve 65 ° c en az 16 h için kuluçkaya.
RNase blok Mix (örnek) başına:
RNase blok 0.5 µL
Su 1,5 µL
Kütüphane hibridizasyon Mix (örnek) yakalama:
Hibridizasyon arabelleği 13 µL
RNase blok Mix 2 µL
Sıçan metil-Seq 5 µL yakalama Kütüphane
Not: Non-spesifik bağlama önlemek için hibridizasyon Mix eklerken reaksiyonlar 65 ° C'de tutmak . - Aliquot 50 µL streptavidin manyetik boncuklar örnek yeni bir 8-şey şerit tüp içine başına. Boncuk 200 µL metil-Seq bağlama arabellek ile yıkayın. Manyetik plaka boncuk cips ve süpernatant 3 yıkama olmak üzere toplam her yıkama arasında kaldırmak için kullanın. Son yıkama sonra streptavidin boncuk metil-Seq bağlama arabellek 200 µL içinde resuspend.
- Yıkanmış streptavidin manyetik boncuklar 200 µL için örnekleri ekleyin ve 30 dk dönen bir mikser kullanarak için oda sıcaklığında kuluçkaya. Karıştırma, metil-Seq yıkama tampon 2 aliquot 200 µL 96-şey plaka başına örnek ve 65 ° C'ye önceden ısıtmak için bir termal cycler yerde onaylatılacak kuyu içine süre
- Streptavidin manyetik boncuklar Manyetik plaka kullanarak kuluçka sonra cips ve 200 µL boncuk resuspend metil-Seq yıkama tampon 1. 15 dakika oda sıcaklığında kuluçkaya. Cips ve süpernatant atmak için manyetik bir tabak kullanın.
- Boncuk metil-Seq yıkama tampon 2 ile 3 kez yıkayın: arabellek (adım 6.2.5 önceden ısındı.) 2 yıkama 200 µL boncuk Pelet resuspend, termal cycler (65 ° C, 10 dk) boncuklar kuluçkaya ve cips boncuk. Süpernatant manyetik bir tabak kullanarak her yıkama sonra atın.
Not: hibridizasyon reaksiyonlar 65 ° C'de yıkama tampon 2 non-spesifik bağlama önlemek için eklerken korumak. - Metil-Seq elüsyon arabelleği 20 µL yıkanmış boncuk ekleyin ve 20 dk. kullanım Pelet boncuk ve yeni bir şerit tüp süpernatant aktarmak için manyetik bir tabak için oda sıcaklığında kuluçkaya. Boncuk atmak.
Not: kuluçka sırasında bisülfit dönüşüm reaktif hazırlayın.
- Örnekleri düşük DNA'ya bağlanıcı microcentrifuge tüpler için transfer edecek ve ısıtmalı vakum yoğunlaştırıcı az 3.4 µL. Reconstitute örnekleri 3.4 µL için numune hacmi azaltmak için.
- Bisülfit dönüştürme
Not: uygun reaktifler ve ticari olarak mevcut bisülfit dönüşüm kiti yönergeleri kullanarak eluted ssDNA bisülfit dönüştürme gerçekleştirmek.- 130 µL hazırlanan bisülfit dönüşüm reaktif süpernatant için önceki adımı ekleyin. Her biri 150 µL reaksiyonlar eşit iki kuyu içine bölün. Termal cycler (2,5 h, 4 ° C tutun 64 ° C) kuluçkaya.
Not: 150 µL tepki homojen sıcaklık emin olmak için iki ayrı kuyu eşit olarak bölünür. 2,5 h için kuluçka sonra hemen bir sonraki adıma geçin. - Sütun bağlama arabellek 600 µL ekleyerek spin ve bir kez yıkama arabellek 100 µL ile yıkamak için örnekleri bağlayın. Sütunlar (15.000 x g, 1 dk) tüm bisülfit dönüşüm adımları arasında santrifüj kapasitesi ve akışı aracılığıyla atın.
- 200 µL Desulphonation arabelleği sütun ekleyerek Desulphonate örnekleri. 15-20 dk. tekrar Santrifüjü için oda sıcaklığında kuluçkaya ve akışı aracılığıyla atın.
- Yıkama arabelleği 200 µL sütunlarla iki kez yıkayın. Her örnek için oda sıcaklığında 3 dk kuluçka ve centrifuging sütun için (15.000 x g, 1 dk) 10 µL elüsyon arabelleği ekleyerek elute. Toplamda 20 µL elüsyon adımı yineleyin.
- PCR reaksiyon Master Mix 1 buzda hazırlayın. 82 µL karışımı her örnek için ekleyin. Aşağıdaki programı ile termal cycler kuluçkaya.
PCR reaksiyon Master Mix 1 (örnek) başına:
Su 30 µL
Metil-Seq PCR Master Mix 50 µL
Metil-Seq PCR1 astar F 1 µL
Metil-Seq PCR1 astar R 1 µL
Termal Cycler Program:
Sahne 1, 1 döngüsü: 95 ° C 2 dk
Aşama 2, 8 devir: 95 ° C 30 s, 60 ° C 30 s, 72 ° C 30 s
Sahne 3, 1 döngüsü: 72 ° C 7 dk
Evre 4, 1 döngüsü: 4 ° C tutun - 180 µL DNA'ya bağlanıcı manyetik boncuklar ve taze hazırlanmış % 70 etanol örnek başına 400 µL kullanarak örnekleri arındırmak. Her örnek için 180 µL boncuk ekleyin ve oda sıcaklığında 5 min için kuluçkaya. Boncuk cips, süpernatant kaldırmak ve Pelet % 70 etanol 200 µL içinde resuspend. Etanol kaldırmak ve yıkama bir kez yinelenir.
- Manyetik bir tabak boncuk cips ve mümkün olduğunca çok etanol kaldırmak için kullanın. Kuru bir 37 ° C olarak heatblock 3-5 dk kadar boncuk Pelet tamamen kuru. Nükleaz ücretsiz su 21 µL içinde resuspend ve yaklaşık 19.5 µL süpernatant ile toplamak.
- 130 µL hazırlanan bisülfit dönüşüm reaktif süpernatant için önceki adımı ekleyin. Her biri 150 µL reaksiyonlar eşit iki kuyu içine bölün. Termal cycler (2,5 h, 4 ° C tutun 64 ° C) kuluçkaya.
- Dizin oluşturma
- PCR reaksiyon Master Mix 2 buz üzerinde hazırlamak. 25,5 µL ekleme Master Mix 2 her örnek için. 5 µL astar bireysel örnekleri için dizin oluşturma ticari ekleyin ve bir termal cycler kuluçkaya.
PCR reaksiyon Master Mix 2 (örnek) başına:
25 µL metil-Seq PCR Master Mix
0.5 µL metil-Seq ortak dizin oluşturma astar
Termal Cycler Program:
Sahne 1, 1 döngüsü: 95 ° C 2 dk
Aşama 2, 6 döngüsü: 95 ° C 30 s, 60 ° C 30 s, 72 ° C 30 s
Sahne 3, 1 döngüsü: 72 ° C 7 dk
Evre 4, 1 döngüsü: 4 ° C tutun
Not: Ek döngü (2-3) başlangıç DNA toplama önerilen değerler ise gerekli olabilir. - 90 µL DNA'ya bağlanıcı manyetik boncuklar ve taze hazırlanmış % 70 etanol örnek başına 400 µL kullanarak örnekleri arındırmak. Her örnek için 90 µL boncuk ekleyin ve oda sıcaklığında 5 min için kuluçkaya. Boncuk cips, süpernatant kaldırmak ve Pelet % 70 etanol 200 µL içinde resuspend. Etanol kaldırmak ve yıkama bir kez yinelenir.
- Manyetik bir tabak boncuk cips ve mümkün olduğunca çok etanol kaldırmak için kullanın. Kuru bir 37 ° C olarak heatblock 3-5 dk kadar boncuk Pelet tamamen kuru. Nükleaz ücretsiz su 24 µL içinde resuspend ve yaklaşık 24 µL süpernatant ile toplamak.
- Konsantrasyon ve bp boyutu üzerinde bir bioanalyzer yüksek-duyarlı DNA algılama reaktifler kullanarak değerlendirmek.
Not: Kütüphane DNA olup olmadığını belirlemek bioanalyzer başarısız olursa, ek DNA ile hazırlık adımları yineleyin.
Nokta durdurma: arıtma sonra Dizin oluşturulmuş örnekleri olabilir mühürlü ve -20 ° C'de depolanan - Örnekler uygun yeni nesil sıralama platformu için kullanılan havuz.
- DNA derişim Kütüphane boyutu ve miktarı verilen birime göre belirler, bioanalyzer, konsantrasyon verileri kullanarak düşük TE arabellek (6.1.1.1) ile sulandırmak ve 15 son bir konsantrasyon için tüm örneklerini birleştirmek pM.
Not: Kütüphane miktarının bir daha duyarlı ve birleştirilmiş bağdaştırıcıları hedef primerler kullanılarak nicel gerçek zamanlı PCR tarafından yöntemdir. - Havuza alınan örnekleri yeni nesil sekanslama Lane'de başına 4 örnek için yeterli şerit sayısı çalıştırın.
Not: 16 kitaplık örnekleri benzersiz dizine ve kombine varsa, örneğin, üzerinde 4 şeritli, eşdeğer 4 örnek lane / kütüphaneler çalıştırın.
- DNA derişim Kütüphane boyutu ve miktarı verilen birime göre belirler, bioanalyzer, konsantrasyon verileri kullanarak düşük TE arabellek (6.1.1.1) ile sulandırmak ve 15 son bir konsantrasyon için tüm örneklerini birleştirmek pM.
- PCR reaksiyon Master Mix 2 buz üzerinde hazırlamak. 25,5 µL ekleme Master Mix 2 her örnek için. 5 µL astar bireysel örnekleri için dizin oluşturma ticari ekleyin ve bir termal cycler kuluçkaya.
7. yeni nesil sekanslama üzerinde sıralama
- Bir yeni nesil sıralama makine üzerinde sıralama tarafından takip metil-Seq Kütüphane, kümeleme için kurumsal sıralama çekirdek örnekleri gönderin.
8. DMRs belirlemek için çözümlemesi
- Bismark15bisülfit dönüştürülür, artı-strand genom ham giriş okuyan hizalamak için bir iç sıra aligner16,17olarak, papyon 2.0 çağırır, uygulamak. Hizalama, Bismark_methylation_extractor kalite kontrol gerçekleştirmek ve her CpG bir tahmini metilasyonu değeri atamak için kullanın.
- DMRs bir listesini BS-Seq paket18 ile Bioconductor içinde oluşturun. Filtre DMRs temel 3 ardışık CpGs ve P-değeri büyük olan < 0,05.
Not: genomik koordinatları, en yakın RefSeq gen Uzaklik, CpGs içinde her DMR, ortalama % CpG metilasyonu değeri arasında iki karşılaştırma grupları (vs vurgusuz vurguladımesela ), P-değeri, DMR sayısını içeren bir DMR listesi oluşturmak ve FDR (yanlış bulma oranı) değeri. DMR listesini, i.ekullanın., pyrosequencing astar doğrulama için tasarlamak için genomik koordinatları.
9. bisülfit Pyrosequencing doğrulanması
-
Astar tasarım
- Tasarım bisülfit PCR için astar ve pyrosequencing. İç içe geçmiş PCR bir DMR 150-400 bps yükseltmek PCR astar (dış ve iç içe geçmiş) iki kümesi tasarlayın.
Not: genel olarak, tasarlanmış astar en az 24 üsleri vardır. uzun en az 4-5 ardışık olmayan G'nin (C ters astar için) ile hesabınıza sıra karmaşıklığı kaybı sıcaklığından tavlama azaltılmış. Bir iç içe geçmiş astar biotin etiketli ve HPLC saf olacak. Ancak, standart astar ilk tepkiler üzerinde bir özel jel çözülerek PCR adım optimize etmek için sipariş.- Pyrosequencing tahlil astar tamamlayıcı biotinylated hedefleyen böylece tasarım yolda sadece 1-2 üsleri akıntıya karşı denetlesinler için CpGs. Her pyrosequencing astar güvenilir 30 bps aşağı tahlil gibi birden çok pyrosequencing astar gerektiği şekilde tasarlayın.
- Rt1-m4 için aşağıdakileri kullanın:
rRT1M4 dışında-F TGTAYGATTTTGGTTATYGTAAAT
rRT1M4 dışında-R AACTTACAAATTTCACCAACTCA
rRT1M4 Nested-F GTGGGTTAYGTGGATAATATATAG
rRT1M4 Nested-R AATCACTTACCATTCTCTCTCTAACTA
rRT1M4 Pyro1 TAYGTGGATAATATATAGAT
rRT1M4 Pyro2 GATAGTTATTTGGYGAGTTAG
rRT1M4 Pyro3 GAGTATTTGGAGGAGTTGAT
rRT1M4 Pyro4 GGATTTTAATATTTGGT
- Tasarım bisülfit PCR için astar ve pyrosequencing. İç içe geçmiş PCR bir DMR 150-400 bps yükseltmek PCR astar (dış ve iç içe geçmiş) iki kümesi tasarlayın.
-
Piyasada bulunan bir kit fare kan gDNA bisülfit dönüşüm için kullanın.
Not: Aşağıdaki değişiklikler ile piyasada bulunan kitinden bisülfit dönüşüm adımları uyarlanmıştır: adım 1, kan gDNA 50-100 ng ekleyin ve 20 µL su ile oranında seyreltin. 9. adımda örnek başına 20 µL elute.- Bisülfit dönüşüm reaktif üreticinin protokolüne göre hazırlamak ve sulandırılmış gDNA ile birleştirir. Termal cycler (2,5 h, 4 ° C tutun 64 ° C) kuluçkaya.
- Bağlama arabellek dönüştürülmüş gDNA spin sütunlarda ve santrifüj (15.000 x g, 1 dk) ekleyin. Yıkama sütunları bir kez daha sonra Desulphonation arabellek için sütunları ekleyin ve 15 dakika oda sıcaklığında kuluçkaya. Santrifüj (15.000 x g, 1 dk.).
- Sütun yıkama tamponu ve santrifüj (15.000 x g, 1 dk) ile yıkayın. Santrifüjü (15.000 x g, 2 dk) ile yıkama adımı yineleyin. 20 µL eklemek elüsyon tampon ve santrifüj (15.000 x g, 1 dk) elute.
-
PCR güçlendirme
- Dış PCR Master Mix hazırlayın. 3.5 µL bisülfit dönüştürülmüş gDNA için Master Mix 21,5 µL ekleyin ve termal cycler programını çalıştırın.
Dış PCR Master Mix:
Su 16.25 µL
Polimeraz arabelleği 2.5 µL [10 x]
0.5 µL dNTP [10 mM]
İleri astar 1 µL [0,1 µM]
Ters astar 1 µL [0,1 µM]
Taq DNA polimeraz 0,25 µL [5000 U/mL].
Termal cycler programı:
Sahne 1, 1 döngüsü: 94 ° C 4 dk
Aşama 2, 47 devir: 94 ° C 1 dakika, 53 ° C 30 s, 72 ° C 1 dk
Sahne 3, 1 döngüsü: 72 ° C 8 dk, 4 ° C tutun - İç içe geçmiş PCR Master Mix hazırlayın. Master Mix 23 µL örnek 2 µL için dış PCR ekleyin ve dış PCR termal cycler programı yineleyin. PCR ürün kalitesi jel elektroforez (1 x TAE arabellek, % 1'özel jel) aracılığıyla değerlendirmek.
İç içe geçmiş PCR Master Mix:
Su 17,75 µL
Polimeraz arabelleği 2.5 µL [10 x]
0.5 µL dNTP [10 mM]
İleri astar 1 µL [0,1 µM]
Ters astar 1 µL [0,1 µM]
Taq DNA polimeraz 0,25 µL [5000 U/mL]
Not: iç içe geçmiş PCR için ileri veya geriye doğru astar biotinylated olması gerekir.
- Dış PCR Master Mix hazırlayın. 3.5 µL bisülfit dönüştürülmüş gDNA için Master Mix 21,5 µL ekleyin ve termal cycler programını çalıştırın.
-
Pyrosequencing
- 38 µL bağlama arabellek, su 35 µL ve streptavidin kaplı sepharose boncuk örnek başına 2 µL içeren bir ana karışım yapmak. Bir 96-şey tabak içinde 75 µL ana mix ve iç içe geçmiş PCR ürününün 5 µL ekleyin. 15-60 dakika için bir plaka shaker sallamayın.
- Sallayarak süre, astar (0.5 µM, seyreltilmiş yılında TAV arabellek) 12 µL pyrosequencing tahlil plaka kuyu ekleyin.
- Sallayarak sonra bağlama tepki yıkama arabellekleri kullanarak yıkama adımları gerçekleştirin. Çukur yer vakum aracında su sonra plaka örnekler toplamak. Vakum % 70 etanol, NaOH (0.2 M) ve Tris asetat arabellek (10 mM, pH 7,4) içeren yarı dolu kaplar aracında daldırın. HS tahlil plaka boncuk aktarmak için vakum aracında vakum ve yer bağlantısını kesin.
- 2 dk. 5 min için serin sonra pyro programına başlamak için izin ver plaka için 80 ° C'de kuluçkaya ve plaka ısı blokta yer.
Sonuçlar
Sıçan metil-Seq platformu başarılı bir uygulama çeşitli ölçütlere bağlıdır. Şekil 1 çalışmanın genel iş akışı gösterilmektedir ve ileri taşımadan önce gerekli olan belirli kalite kontrol (QC) adımlar vurgular. Dikkat edilmesi gereken ilk faktör biridir methylome arasında oluşan epigenetik değişiklikleri büyüklüğünü belirlemek hayvan modeli ve stres rejimi sağlamlık. Hayvan çalışmalarımız corticosterone (CORT) maruz kalma DNA metilasyonu19,20değişikliklere neden olabilir bizim önceki gözlem esas beri bizim kronik değişken stres (CVS) rejimi üretmek için yeterli rigor gerekiyordu yüksek plazma CORT düzeyleri ile Rats vurguladı. Tipik bir haftalık CVS rejimi Tablo 1 ' de gösterilen ve günlük stres sabah, öğleden sonra oluşuyordu ve gecede sürekli habituation önlemek için değiştirilir ve stres tepkisi azalmış. 3 haftalık rejim stresli hayvanlar ortalama plazma CORT önemli ölçüde yüksek seviyede sergilenen [gün 4 – 21, kontrol: 32.7 3.7 ng/mL, stres: 103.0 11,9 ng/mL (ortalama SEM), P 2.2 x 10-4, Şekil 2A=] Bu olarak vurgusuz, hayvan kontrol. Sürekli olarak, bu hayvanlar da yükseltilmiş durumda büyük anksiyete gibi davranış artı labirent (EPM), EPM ve kollarını açarak (2B rakam) zamanında daha az kapalı kollarında geçirdiğim önemli ölçüde daha fazla gösterildiği gibi gösterdi. Bu sonuçlar CVS pozlama önemli endokrin ve bize bu değişiklikler belirli DNA metilasyonu imzaları ile ilişkili olup olmadığını araştırmak için önde gelen davranış değişiklikleri için yol göstermektedir.
Biz metil-Seq Kütüphane başarılı yapımı için çok önemli birkaç denetim noktaları vurgulamak. Yeterli miktarda DNA ile başlayan gerekli, sonication, birden fazla yıkama/arıtma, hedef zenginleştirme ve bisülfit dönüşüm adımları sırayla DNA bitmiş kütüphanede bulunan miktarı azaltın. Her ne kadar birçok PCR güçlendirme adımları DNA şablon kaybı hafifletmek, aşırı PCR döngüsü numaraları yüksek yinelenen okuma neden olabilir. Sıçan metil-Seq çalışmada için kan gDNA fare başına 2 g kullanıldı. Biz metil-Seq kitaplıkları DNA miktarı düşük 500 olarak başlayan ile yapılabilir notu ng. Sonraki sıralama için kitaplıklarına yetersiz miktarda üreten riski olsa daha küçük malzeme başlayan (floresan aktif hücre sıralama) FACS tarafından izole DNA kitaplıkları oluşturmak veya yumruk, iğne kullanıcılara izin verir. QC Elektroforez örneği DNA molekül ağırlığı, miktar ve derişim sağlar bir bioanalyzer üzerinde 1 m tarafından gerçekleştirilir. Bioanalyzer kullanımını gerektiren üç kritik adım vardır: 1) sonication adım yeterli DNA'sı (~ 170 bp, kırmızı, Şekil 3); kesme emin olmak için takip 2) bir vardiyada yamultulmuş DNA (~ 200 bp, mavi, Şekil 3) onların sonraki amplifikasyon PCR tarafından; emin olmak için Ortalama büyüklüğü bağdaştırıcısı ligasyonu adım takip simgesiyle ve 3) miktar ve sıralama için Kütüphane boyutu sağlamak için son Kütüphane arıtma adım takip.
R-paketleri BSSeq ve BSmooth Bioconductor içinde veri18sıralama bisülfit analiz etmek için kullanılmıştır. Onlar araçları ve yöntemleri, kalite kontrol, gerçekleştirme sırası okur hizalamak için içerir ve differentially tanımlayan metillenmiş bölgeleri (DMRs). BSmooth yazılım papyon 2.016,17 CpG düzeyi ölçüm özetleri, hizalama genomik sıralarını bisülfit dönüştürülmüş ham giriş okur tarafından elde etmek için bir iç sıra aligner olarak çağırır. Hizalanmış okuma sonra sistematik sıralama ve aşağı akım analizleri çarpık Bankası-arama hataları tanımlamak için aramak sıkı kalite kontrol işlemleri yoluyla filtre uygulanır. Araziler bir dizi görsel olarak filtreleme bu süreç içinde yardım etmek için oluşturulur. Sıralama ölçütleri de belge numarası hizalanmış okur, % hedef ve diğerleri (Tablo 2) CpG kapsama başına gibi ilgili bilgileri için oluşturulur. Bir kez veri süzülür, yumuşatma/normalleştirme algoritması gerçekleştirildiğinde, nerede her CpG tüm QC dayalı bir tahmini metilasyonu değer atanmış her örnekten okur ve CpGs metilasyon daha doğru arama emin olmak için komşu tahminleri durumu bile sıra kapsamı düşük olduğu durumlarda. Bu değer her CpG sitede metilasyonu olasılığının Düzgünleştirilmiş bir tahmin sağlar. Sıralaması için en azından en önemli ölçüde farklı genomik bölgelerine ve iki tedavi grupları arasında her örneğinin düzgünleştirilmiş metilasyonu tahminleri ortalaması karşılaştırarak, DMRs listesi oluşturulur (Tablo 3).
DMR vurguladı ve vurgusuz gruplar arasında fare MHC gen Rt1-m4, organizatörü ile yer oldu üst vurgusuz hayvanların (Şekil 4A) daha tüm CpGs arasında metilasyon katlara sergilenmesi hayvanlar vurguladı. Metil-Seq platformu ve veri analizi başarılı uygulanması onaylamak için astar DMR ve kan DNA metilasyonu düzeyleri ve vurgusuz stresli hayvanlar (metil-Seq tarafından sıralı 8 ve 8 değil sıralı) tüm kohort karşı tasarlanmıştır bisülfit pyrosequencing tarafından değerlendirildi. Sonuçları 10 dışarı-in denetlesinler 12 CpGs arasında DNA metilasyonu önemli artış göstermektedir (% metilasyonu, P 5.1-10,4 değişiklik < 0,037, Şekil 4B). KEGG yolu analiz tüm sözde önemli DMRs stres ile ilgili yollar tanımlamak için gerçekleştirildi. Sürekli olarak, DMR ilişkili yolları hastalıkları kronik stres maruz kalma, diyabet, kardiyovasküler hastalık ve kanser (Tablo 4) gibi ilişkili karıştığı. 21 , 22 , 23 , her hayvan için ortalama 3 haftalık CORT düzeylere KSY-10 düzeylerinde metilasyonu strese maruz kalma derecesi ve epigenetik veri arasındaki ilişkiyi göstermek için karşılaştırıldı. Sonuçlar gösterdi endokrin ve metilasyonu veri arasında mütevazı bir korelasyon (R2= 0.54, P = 0.001, Şekil 5).
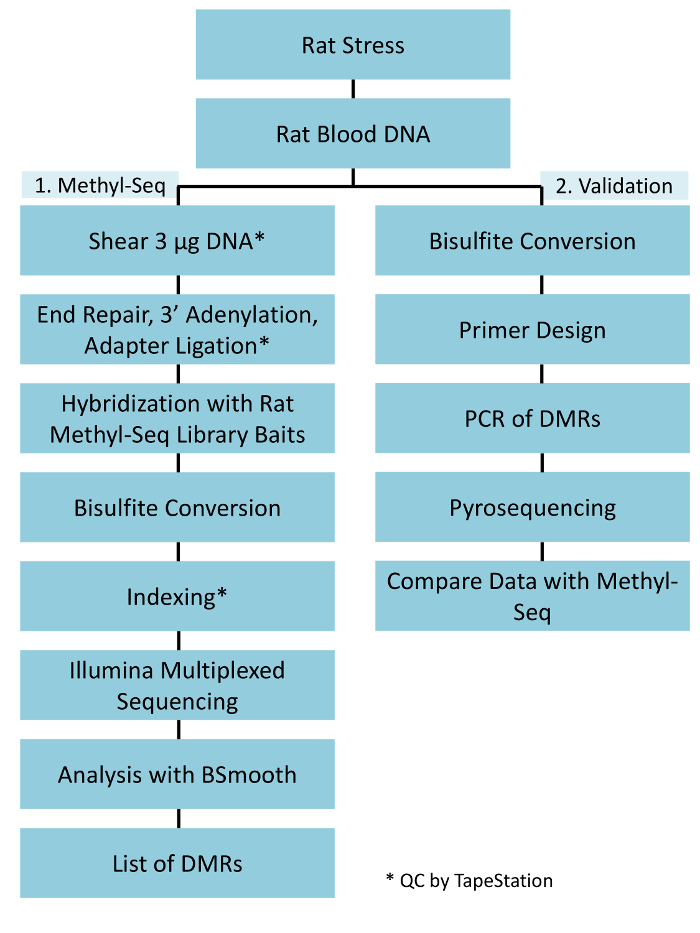
Şekil 1: genel olarak şematik iş akışı sıçan metil-Seq platformu için. Bir g kandan ayıklanan genomik DNA'ın vurguladı ve kontrol fareler ilk sıralama, analiz ve hedef tanımlama için metil-Seq kitaplıkları oluşturmak için işlenmiş. Başka bir 100 ng DNA'ın bisülfit pyrosequencing tarafından tanımlanan epigenetik hedefleri bağımsız doğrulamak için kullanılır. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
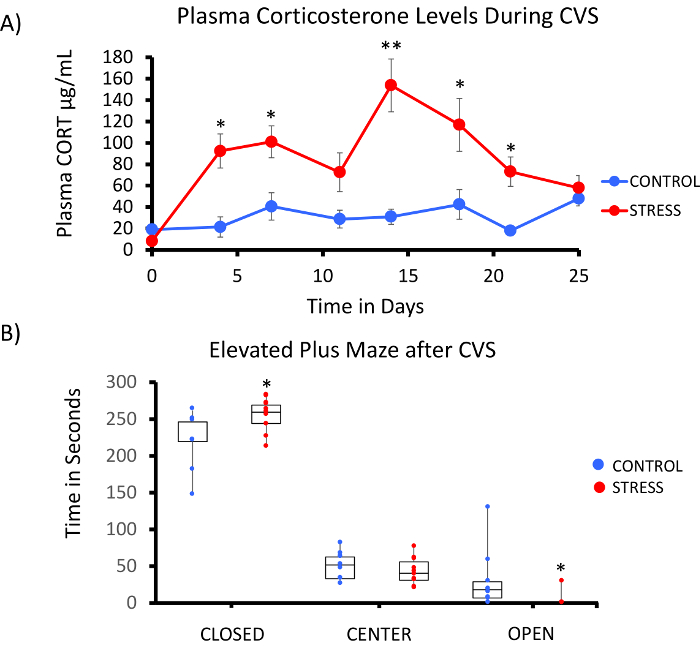
Şekil 2: kronik değişken stres (CVS) maruz açar sıçanlarda endokrin ve davranış değişiklikleri için. (A)corticosterone (CORT) birden fazla örnekleme göstermek sağlamlık 3 hafta CVS rejimi. Kan örnekleri günlük stres alay önce sabahları toplanmıştır. (B) vurguladı hayvanlar kapalı silah ve yükseltilmiş kollarını açarak zamanında artı labirent (EPM) daha az daha fazla zaman geçirdim. Öğretici Boxplots her hayvan için veri noktası ile gösterilir. Öğrenci T-testi için istatistiksel anlamlılık gerçekleştirildi. * P < 0,05, ** P < 0,01, ve *** P < 0,001. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
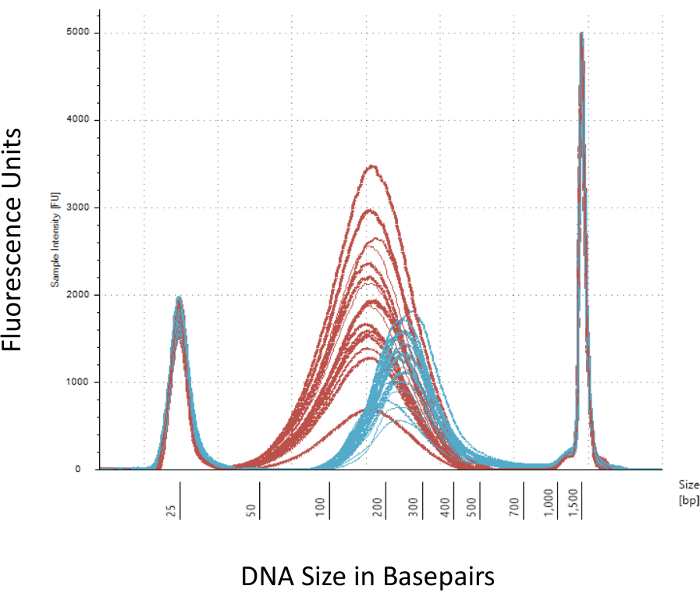
Şekil 3: Nefelometri yamultulmuş ve bağdaştırıcı bakmaksızın sıçan DNA üzerinde bir bioanalyzer. Kırmızı ve mavi eğrileri miktar ve genomik DNA (kırmızı) bir izotermal sonicator ve bağdaştırıcı ligasyonu, sırasıyla kesme takip boyutunu gösterir. Her satır bir örnek ve kırmızı temsil eder ve mavi eğrileri DNA ' (bitiş-tamir, 3'-adenylation ve örnek temizleme) birkaç adım sırasında her iki kaybı yansıtmak ve bağdaştırıcıları ligasyonu nedeniyle bp boyutunda artırmak. Keskin doruklarına 25 bp ve 1500 bp yükleme arabelleğe eklendi standart işaretleri vardır. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
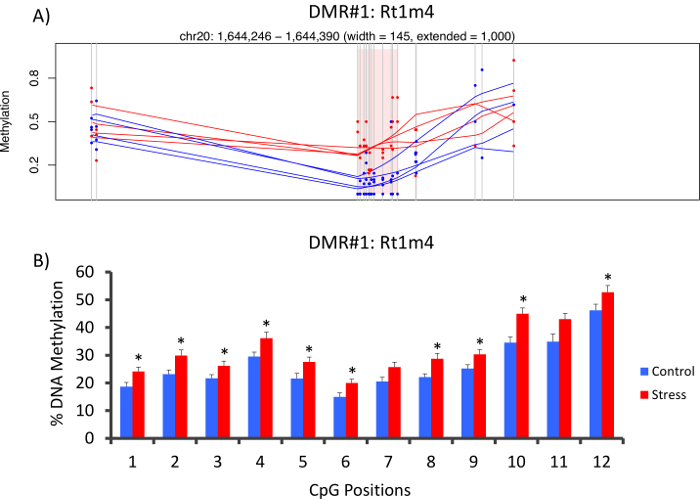
Şekil 4: epigenetik değişiklikleri CVS kaynaklı sıçan metil-Seq. tarafından tespit Sıçan metil-Seq veri analizi(a)karıştığı gen Rt1m4 organizatörü differentially metillenmiş bölgesine (DMR) stresli (kırmızı) ve denetim (mavi) fareler arasında. Grafik çıktı Rt1m4 DMR (pembe gölgeli bölgesi) için her CpG (dikey gri çizgi), dört örnekleri her grup (kırmızı veya mavi çizgiler) ve her hayvan (kırmızı veya mavi nokta) için % metilasyonu düzeylerini görüntüler. (B) on iki CpGs içinde DMR bisülfit pyrosequencing tarafından onaylanmış. Çubuk grafikler SEM demek olarak temsil edilir ve bir öğrenci T-testi için istatistiksel anlamlılık gerçekleştirildi. * P < 0,05. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
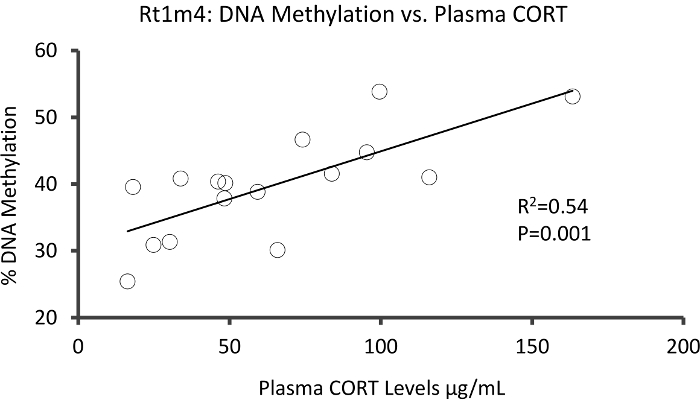
Şekil 5: doğrusal regresyon çözümlemesi gösterdi mütevazı bir korelasyon arasında % DNA metilasyonu KSY-10 / Rt1m4 ve 3 hafta plazma CORT düzeyleri vurguladı her ikisi anlamına gelir ve kontrol hayvanlar (N = 16). Stresli hayvanlar verilerden kırmızı daireler tarafından temsil edilen. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
| Hafta | Gün 1 | 2. gün | 3. gün | 4. gün | Gün 5 | Gün 6 | Gün 7 |
| AM | Kısıtlama | Yüzmek | Soğuk Oda | Yüzmek | Kısıtlama | Shaker | Yüzmek |
| PM | Shaker | Kafes Tilt | Kısıtlama | Shaker | Soğuk Oda | Kısıtlama | Soğuk Oda |
| Bir gecede | Gıda kısıtlamak | Odada mevcut yataklar kullanılıyorsa ıslak | Yalıtım | Işık | Kalabalık | Işık | Odada mevcut yataklar kullanılıyorsa ıslak |
Tablo 1: Bir normal haftalık zamanlama, kronik değişken stres rejimi (CVS).
| Sıralama ölçütleri | Stres1 | Kontrol1 |
| (n = 4) | (n = 4) | |
| Son Okunma (başı) eşleştirilmiş | 89,290,397 | 80,165,674 |
| Benzersiz olarak eşlenen eşleştirilmiş sonunda (UMPER) okur | 39,200,255 | 35,013,406 |
| Hizalama oranı/eşleme verimlilik (UMPER / başı) | % 44 | % 44 |
| Yinelenen okuma (UMPER %) | % 73 | % 65 |
| Deduplicated UMPER | 10,481,031 | 12,306,018 |
| Ortalama okuma derinlik kapsama (x) (ARDC) | 6 x | 6 x |
| CpGs (N) | 12,056,878 | 12,056,878 |
| ARDC (x) CpGs | 2 x | 2 x |
| En az 10 okuma (N) ile CpGs | 481,383 | 595,850 |
| ARDC (X) CpGs ile en az 10 defa okundu | 19 | 19 |
| Hedef CpGs (sonda hedef bölgeleriyle tam örtüşme) | 1,923,872 | 2,007,638 |
| Hedef ARDC (x) CpGs | 7 x | 8 x |
| Hedef CpGs üzerinde en az 10 okuma (N) ile | 428,249 | 531,419 |
| Hedef ARDC (x) CpGs en az 10 ile okur | 18 x | 18 x |
| Hedef (hedef bölgeleriyle Probe 1 veya daha fazla baz çifti örtüşme ile PER) (UMPER) | 8,277,715 | 9,369,523 |
| Hedef (Deduplicated UMPER) Toplam % | % 78 | % 77 |
| Hedefe (Toplam üsleri eşlenen) Mb | 125 mb | 128 mb |
| Açık hedef ortalama okuma derinlik kapsama (x) (ARDC) | 9 x | 10 x |
| 1 Her grup bireylerde genelinde ortalamalar göre sıralama ölçütleri |
Tablo 2: fare metil-Seq platformu elde edilen ölçümler sıralanıyor.
| Chr | Başlat | bitiş | Gen | mesafe | areaStat | meanDiff | stres | Denetim | Yön |
| chr20 | 1,644,246 | 1,644,390 | RT1-M4 | in_gene | 93.03 | 0,22 | 0,33 | 0,11 | kazanç |
| chr5 | 160,361,352 | 160,361,564 | LOC690911 | in_gene | -70.75 | -0.19 | 0,72 | 0,91 | kaybı |
| chr3 | 61,138,281 | 61,138,330 | RGD1564319 | 265569 | 61.79 | 0.21 | 0,94 | 0,72 | kazanç |
| chr2 | 143,064,811 | 143,065,010 | Ufm1 | 8569 | -59.48 | -0.11 | 0,13 | 0,24 | kaybı |
| chr7 | 30,764,111 | 30,764,284 | Ntn4 | in_gene | 57.04 | 0.21 | 0,94 | 0,73 | kazanç |
| chr17 | 12,469,112 | 12,469,218 | Idnk | 41996 | -50.91 | -0.13 | 0,74 | 0,88 | kaybı |
| chr7 | 47,101,725 | 47,101,930 | Pato | in_gene | -50.54 | -0.12 | 0.64 | 0,76 | kaybı |
| chr5 | 76,111,248 | 76,111,822 | Txndc8 | 151703 | -50.38 | -0.11 | 0.85 | 0,96 | kaybı |
| chr11 | 80,640,132 | 80,640,356 | Dgkg | in_gene | -50.07 | -0.16 | 0,73 | 0,89 | kaybı |
| chr8 | 71,759,248 | 71,759,411 | Mir190 | 210226 | -47.84 | -0.17 | 0,58 | 0,75 | kaybı |
Tablo 3: en iyi 10 differentially bölgeleri metillenmiş. Her DMR çıkış tablosu için sağ sütun soldan gösterir: kromozom konumu (chr), koordinatları (başlangıç/bitiş), gen adı, transkripsiyon mesafeden sitesini başlatın, vurguladı ve kontrol grupları (areaStat) arasındaki fark alanı istatistikleri, demek Ortalama metilasyon düzeyleri için her DMR arasında fark metilasyonu (meanDiff), vurguladı ve kontrol grubu (stres/denetimi) ve metilasyonu yönünü denetimlerden değiştirin.
| KEGG yol şartları | Gen sayısı | % | P-değeri | Benjamini |
| Diyabet | ||||
| Tip II diyabet | 12 | 0,1 | 3.6 x 10-4 | 9,8 10-3 x |
| Kardiyovasküler hastalık | ||||
| Vasküler düz kas kasılma | 18 | 0,1 | 1.6 x 10-3 | 3.6 x 10-2 |
| Aritmojenik Sağ ventrikül kardiyomiyopati (R.V.C) | 13 | 0,1 | 4.0 x 10-3 | 7.1 x 10-2 |
| Dilate kardiyomiyopati | 14 | 0,1 | 7,6 10-3 x | 1.2 x 10-1 |
| Nöron işlevi | ||||
| Uzun vadeli kullanılmasının muhtemelen | 11 | 0,1 | 1.5 x 10-2 | 1.4 x 10-1 |
| Sinyal verme | ||||
| MAPK sinyal yolu | 35 | 0,2 | 2.4 x 10-4 | 10-3 x 9,9 |
| Kalsiyum sinyal yolu | 22 | 0,1 | 1.2 x 10-2 | 1.4 x 10-1 |
| Kemokin sinyal yolu | 21 | 0,1 | 1.2 x 10-2 | 1.3 x 10-1 |
| Kanser | ||||
| Yolları kanser | 42 | 0,3 | 4.1 x 10-5 | 3.4 x 10-3 |
| Tümörü | 15 | 0,1 | 4.4 x 10-5 | 2.4 x 10-3 |
| Sigara-küçük hücreli akciğer kanseri | 10 | 0,1 | 7,9 10-3 x | 10-1 x 1.1 |
| Kolorektal kanser | 13 | 0,1 | 8,4 10-3 x | 10-1 x 1.1 |
| Kronik miyeloid lösemi | 12 | 0,1 | 1.2 x 10-2 | 1.3 x 10-1 |
Tablo 4: KEGG yolu analizini sıçan metil-Seq. tespit DMRs
Tartışmalar
Bu çalışmada tasarlanmış ve metil-Seq platform sıçan genom için uygulanmaktadır. Yararını özellikli bir sıçan stres göstererek, deneysel ve analitik boru hattı differentially metillenmiş bölgeler arasında iki karşılaştırma grupları sağlayabilir gösterdi.
Başarılı bir uygulama platformu sağlamak için uyması gereken birkaç önemli adımlar gerekir. İlk, ilk DNA kalite ve miktar kalite ve son metil-Seq Kütüphane miktarı üzerinde önemli bir etkisi vardır. Spektrofotometre, yerine bir yer bizim DNA ölçüm çift iplikçikli DNA mevcut miktarı yansıyan sağlamak için kullanılır. Bioanalyzer miktar DNA kesme takip ve sonra bağdaştırıcı ligasyonu ve moleküler boyutu ölçmek için kullanıldı. Moleküler boyutu arasındaki adımları "kayma" doğrulama bağdaştırıcısı bağdaştırıcı aracılı PCR sonraki adımda geçirecek her DNA parçasının ucunda varlığını doğrulamak çok önemlidir. Bağdaştırıcı ligasyonu adım kalan DNA miktarı da en az 100 beri önemlidir ng Kütüphane ürünün bu adımda yeterli miktarda hedef zenginleştirme ve bisülfit dönüşüm adımları sonra kullanılabilir olduğundan emin olmak için gerekli. Böylece sonraki yeni nesil sekanslama kümeleme için Kütüphane düzgün seyreltilmiş son bir yüksek-duyarlı ölçüm üzerinde inşa metil-Seq Kütüphane gerçekleştirildi. Son olarak, bisülfit pyrosequencing analitik boru hattı doğruluğunu değerlendirmek için son derece nicel, bağımsız bir yöntem istihdam edildi. Orijinal örnekleri ve ek hayvan kullanarak çoğaltma kullanarak son doğrulama deneme DNA metilasyonu biyolojik olarak önemli değişiklikleri algılayabilir emin olmak için çok önemli adımlar vardır.
Biz de protokol sapma veya karşılaşılan sorunlar eğer durumunda birkaç yönergeleri içerir. İlk olarak, son-onarım, bağdaştırıcı ligasyonu veya manyetik boncuk arıtma adımları sırasında çok fazla DNA kaybetmek mümkündür. Alternatif olarak, DNA miktarda başlayan küçük olabilir (< 200 ng) sınırlı doku/DNA kullanılabilirlik veya floresan aktif hücre sıralama gibi çeşitli zenginleştirme yöntemleri uygulanması nedeniyle. Döngüsü iki kütüphane amplifikasyon adımları sırasında artırıldığında DNA aşırı kaybı için telafi etmek mümkün veya düşük DNA miktar Kütüphane inşaat Protokolü boyunca başlangıç olabilir. Aşırı şablon amplifikasyon sıralı yinelenen okuma sayısı artışa yol muhtemeldir gibi ancak, hayır daha fazla ek 2 – 3 döngüleri, tavsiye edilir. Bu çoğaltmaları yüzde metilasyonu hesaplamalarda önyargı önlemek için hizalama adım sırasında hariç tutulur. Ortalama DNA boyutu daha--dan 30 bps tarafından artırmaz, ikincisi, reaktifleri yeni, T4 DNA polimeraz, Klenow ve/veya T4 ligaz eski olabilir emin olmak için denetleyin. Piyasada bulunan yedek reaktifler kullanılabilir.
Ayrıca, öngörülen DMRs nerede DNA metilasyonu farkları yok veya daha az bu analiz tarafından tahmin daha pyrosequencing tarafından doğrulamayabilir mümkündür. Zavallı doğrulama aday bölgeler pyrosequencing sonuçları ne zaman fark metilasyonu teyit etmez gibi birçok genom çapında analizleri için çok yaygın bir sorun veya etkisi boyutu göre analiz öngörülen çok daha küçüktür. BSmooth bir analitik "düzgünleştirir" metilasyon düzeyi birden çok CpGs bir pencere boyunca paketidir. Şu anki deneme için BSmooth olan metilasyonu düzeyleri bisülfit pyrosequencing tarafından doğrulanmış DMR karıştığı. Ancak, büyük olasılıkla BSmooth tarafından öngörülen metilasyonu düzeyleri ve bu pyrosequencing tarafından doğrulanmadı arasında tutarsızlıklar olacaktır. Tüm CpGs içinde bir DMR ardışık DNA metilasyonu fazla % 50 oranında farklı olabilir CpGs veya metilasyonu değerleri nedeniyle dışlandı CpGs de dahil olmak üzere, ortalama metilasyon değerleri tahminleri düzeltme işlevi tutarsızlıklar ortaya alt eşik derinlik okuyun. R-paketleri gibi MethylKit24 CpGs veya hatta tek CpGs olan metilasyonu düzeyleri güçlü olanlar pyrosequencing tarafından doğrulanmış ile ilişkilendirmek küçük pencereler tanımlamak için kullanılabilir. Uygulama farklı paketleri ve onların tahmin edilen bölgeler ya da CpGs fark metilasyon pyrosequencing tarafından test veri sağlamlığı sağlayacaktır. Alternatif olarak, özgün metil-Seq kütüphaneler resequenced ve okuma arttırmak için okuma dosyaları eklendi. Metilasyon düzeyi tayini beri yarı nicel ve dikte edilen okuma sayısı [(# of CpGs) / (TpGs + CpGs #)], verilen CpG yüzde metilasyonu değerini doğruluğunu artırmak için okuma derinliği artan. Bu çalışmada, ancak metilasyonu değerleri en az on okuma tarafından tespit edildi ve bir genel okuma kapsama alanı 19 x her CpG için elde CpGs olarak.
Sıçan metil-Seq platform sınırlamaları değildir. Bütün-genom bisülfit sıralama daha daha maliyetli olsa da, diğer yöntemlerden daha önemli ölçüde daha pahalıdır. Yine de, en maliyet şerit sequencer üzerinde satın alma ve yakalama sistemi için değil oldu. Okuma derinlik gerekli, büyük (25-%70) farklılıklar12 DNA metilasyonu'nedeniyle daha az gerektiren çapraz-doku karşılaştırmalar ile bağlı olarak, maliyeti daha fazla örnek lane / çoğullama ve daha yüksek kapasiteli platform kullanılarak azaltılabilir. Ayrıca, numune hazırlama diğer yöntemlerden daha fazla zaman alır. Benzer olsa da yeni nesil sıralama dahil diğer açılan yaklaşımlar, iş yükü için eklenen bisülfit dönüşüm ve arıtma adımları ekleyebilirsiniz. Genel olarak, metil-Seq platform bütün-genom sıralama için düşük maliyetli bir alternatif ve 2,3 milyondan fazla CpGs, çözünürlükte baz çifti olan miktardan çok daha fazla bu Mikroarray tabanlı platformlar tarafından denetlesinler daha sağlar. Bugüne kadar ticari olarak mevcut insan ve fare metil-Seq platformlar makak beyin25,26alkol bağımlı değişiklikleri, fare beyin9ve kan-beyin nörogelişimsel genlerinde belgelemek için kullanılmıştır glukokortikoid10hedefler. Ayrıca, sıra tanıma ne olursa olsun belirli bölgeler enzimleri tarafından hedef yeteneği türler arası karşılaştırmalar için ideal bir platform yapar. Bu çalışmada, metil-Seq platformu birçok farmakolojik, metabolik ve davranışsal deney genom-wide methylomic Aracı parası olmadan gerçekleştirildiği fare için tasarlanmış. Bizim veri DMRs bir stres sıçan modelinde algılamak için kullanılan ve genel plazma CORT düzeyleri gibi fizyolojik diğer parametreleri korelasyon gösterir.
Metil-Seq, düzenleyici bölgeleri belgeleyen kadar deneysel kanıt olmayabilir sıralı genleri ile hayvanlarda epigenetik deneyler için ideal bir platformdur. Gibi bölgelerden kullanılabilir yapıldığında, ek bölgeleri özel olarak tasarlanmış ve geçerli sürüme bağlı. Ayrıca, hedef zenginleştirme Restriksiyon enzimi tanıma tarafından sınırlı değildir bu yana platform karşılaştırmalı genomik için idealdir. Örneğin, herhangi bir gen ilgi organizatörü bölgesi olup belirli kısıtlama sitesi ne olursa olsun limanlar yakalanabilir. Benzer şekilde, bu fare ya da ilgi genom korunmuş insana ilişkin tespit gibi herhangi bir yasal bölgelerde, yakalanabilir.
Açıklamalar
El yazması bir yarışma Ödülü AGILENT Technologies bir parçasıdır.
Teşekkürler
Bu çalışmada NIH grant MH101392 (RSL) ve destek aşağıdaki Ödülleri ve vakıflar tarafından finanse edildi: NARSAD genç araştırmacı Ödülü, Margaret Ann Fiyatı araştırmacı Fonu, James Wah duygudurum bozuklukları bilim adamı Fonu Charles T. Bauer Foundation, Baker ile Vakfı ve proje maç Vakfı (RSL).
Malzemeler
| Name | Company | Catalog Number | Comments |
| Radioimmuno assay (RIA) | MP Biomedicals | 7120126 | Corticosterone, 125I labeled |
| Master Pure DNA Purification Kit | Epicentre/Illumina | MC85200 | |
| Thermal-LOK 2-Position Dry Heat Bath | USA Scientific | 2510-1102 | Used with 1.5 mL tubes |
| Vortex Genie 2 | Fisher | 12-812 | Vortex Mixer |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023 | 100% Ethanol, molecular grade |
| Centrifuge 5424 R | Eppendorf | - | Must be capable of 20,000 x g |
| Qubit 2.0 | ThermoFisher Scientific | Q32866 | Fluorometer |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32850 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32851 | High sensitivity DNA detection reagents |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| SureSelectXT Rat Methyl-Seq Reagent Kit | Agilent Technologies | G9651A | Reagents for preparing the Methyl-Seq library |
| SureSelect Rat Methyl-Seq Capture Library | Agilent Technologies | 931143 | RNA baits for enrichment of rat targets |
| IDTE, pH 8.0 | IDT DNA | 11-05-01-09 | 10 mM TE, 0.1 mM EDTA |
| DNA LoBind Tube 1.5 mL | Eppendorf | 22431021 | |
| Covaris E-series or S-series | Covaris | - | Isothermal sonicator |
| microTUBE AFA Fiber Pre-Slit Snap-Cap 6x16mm (25) | Covaris | 520045 | |
| Water, Ultra Pure (Molecular Biology Grade) | Quality Biological | 351-029-721 | |
| Veriti 96 Well-Thermal Cycler | Applied Biosystems | 4375786 | |
| AMPure XP Beads | Beckman Coulter | A63880 | DNA-Binding magnetic beads |
| 96S Super Magnet | ALPAQUA | A001322 | Magnetic plate for purification steps |
| 2200 TapeStation | Agilent Technologies | G2965AA | Electrophresis-based bioanalyzer |
| D1000 ScreenTape | Agilent Technologies | 5067-5582 | |
| D1000 ScreenTape High Sensitivity | Agilent Technologies | 5067-5584 | |
| D1000 Reagents | Agilent Technologies | 5067-5583 | |
| D1000 Reagents High Sensitivity | Agilent Technologies | 5067-5585 | |
| DNA110 SpeedVac | ThermoFisher Scientific | - | Vacuum Concentrator |
| Dynabeads MyOne Streptavidin T1 magnetic beads | Invitrogen | 65601 | Streptavidin magnetic beads |
| Labquake Tube Rotator | ThermoFisher Scientific | 415110Q | Nutator Mixer is also acceptable |
| EZ DNA Methylation-Gold Kit | Zymo Research | D5006 | Bisulfite conversion kit. Contains Binding, Wash, Desulphonation, and Elution buffers |
| Illumina Hi-Seq 2500 | Illumina | - | Next-generation sequencing machine |
| PCR and Pyrosequencing Primers | IDT DNA | Variable | |
| Taq DNA Polymerase with ThermoPol Buffer - 2,000 units | New England BioLabs | M0267L | |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | |
| Pyromark MD96 | QIAGEN | - | Pyrosequencing machine |
| Ethyl Alcohol 200 Proof | Pharmco-Aaper | 111000200 | 70% Ethanol solution |
| Sodium Hydroxide Pellets | Sigma-Aldrich | 221465 | 0.2 M NaOH denature buffer solution |
| Tris (Base) from J.T. Baker | Fisher Scientific | 02-004-508 | 10 mM Tris Acetate Buffer wash buffer solution |
| PyroMark Gold Q96 Reagents (50x96) | QIAGEN | 972807 | Reagents required for pyrosequencing |
| PyroMark Annealing Buffer | QIAGEN | 979009 | |
| PyroMark Binding Buffer (200 mL) | QIAGEN | 979006 | |
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-01 | Streptavidin-coated sepharose beads |
| PyroMark Q96 HS Plate | QIAGEN | 979101 | Pyrosequencing assay plate |
| Eppendorf Thermomixer R | Fisher Scientific | 05-400-205 | Plate mixer. 96-well block sold separately (cat. No 05-400-207) |
| SureDesign Website | Agilent Technologies | - | Target capture design software (https://earray.chem.agilent.com/suredesign/) |
| UCSC Genome Browser | University of California Santa Cruz | - | rat Nov 2004 rn4 assembly |
| Agilent Methyl-Seq Protocol | Agilent Technologies | - | https://www.agilent.com/cs/library/usermanuals/public/G7530-90002.pdf |
Referanslar
- Barski, A., et al. High-resolution profiling of histone methylations in the human genome. Cell. 129 (4), 823-837 (2007).
- Meissner, A., et al. Genome-scale DNA methylation maps of pluripotent and differentiated cells. Nature. 454 (7205), 766-770 (2008).
- Bibikova, M., et al. High density DNA methylation array with single CpG site resolution. Genomics. 98 (4), 288-295 (2011).
- Naumov, V. A., et al. Genome-scale analysis of DNA methylation in colorectal cancer using Infinium HumanMethylation450 BeadChips. Epigenetics. 8 (9), 921-934 (2013).
- Wockner, L. F., et al. Genome-wide DNA methylation analysis of human brain tissue from schizophrenia patients. Translational Psychiatry. 4, e339 (2014).
- Meissner, A., et al. Reduced representation bisulfite sequencing for comparative high-resolution DNA methylation analysis. Nucleic Acids Research. 33 (18), 5868-5877 (2005).
- Smith, Z. D., Gu, H., Bock, C., Gnirke, A., Meissner, A. High-throughput bisulfite sequencing in mammalian genomes. Methods. 48 (3), 226-232 (2009).
- Slieker, R. C., et al. Identification and systematic annotation of tissue-specific differentially methylated regions using the Illumina 450k array. Epigenetics Chromatin. 6 (1), 26 (2013).
- Hing, B., et al. Adaptation of the targeted capture Methyl-Seq platform for the mouse genome identifies novel tissue-specific DNA methylation patterns of genes involved in neurodevelopment. Epigenetics. 10 (7), 581-596 (2015).
- Seifuddin, F., et al. Genome-wide Methyl-Seq analysis of blood-brain targets of glucocorticoid exposure. Epigenetics. 12 (8), 637-652 (2017).
- Irizarry, R. A., et al. The human colon cancer methylome shows similar hypo- and hypermethylation at conserved tissue-specific CpG island shores. Nature Genetics. 41 (2), 178-186 (2009).
- Lee, R. S., et al. Adaptation of the CHARM DNA methylation platform for the rat genome reveals novel brain region-specific differences. Epigenetics. 6 (11), 1378-1390 (2011).
- Jankord, R., et al. Stress vulnerability during adolescent development in rats. Endocrinology. 152 (2), 629-638 (2011).
- Pellow, S., Chopin, P., File, S. E., Briley, M. Validation of open:closed arm entries in an elevated plus-maze as a measure of anxiety in the rat. Journal of Neuroscience Methods. 14 (3), 149-167 (1985).
- Krueger, F., Andrews, S. R. Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications. Bioinformatics. 27 (11), 1571-1572 (2011).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), R25 (2009).
- Hansen, K. D., Langmead, B., Irizarry, R. A. BSmooth: from whole genome bisulfite sequencing reads to differentially methylated regions. Genome Biology. 13 (10), R83 (2012).
- Lee, R. S., et al. Chronic corticosterone exposure increases expression and decreases deoxyribonucleic acid methylation of Fkbp5 in mice. Endocrinology. 151 (9), 4332-4343 (2010).
- Lee, R. S., et al. A measure of glucocorticoid load provided by DNA methylation of Fkbp5 in mice. Psychopharmacology. , (2011).
- Bose, M., Olivan, B., Laferrere, B. Stress and obesity: the role of the hypothalamic-pituitary-adrenal axis in metabolic disease. Current Opinion in Endocrinology, Diabetes, and Obesity. 16 (5), 340-346 (2009).
- Brydon, L., Magid, K., Steptoe, A. Platelets, coronary heart disease, and stress. Brain, Behavior, and Immunity. 20 (2), 113-119 (2006).
- McKlveen, J. M., et al. Chronic Stress Increases Prefrontal Inhibition: A Mechanism for Stress-Induced Prefrontal Dysfunction. Biological Psychiatry. 80 (10), 754-764 (2016).
- Akalin, A., et al. methylKit: a comprehensive R package for the analysis of genome-wide DNA methylation profiles. Genome Biology. 13 (10), R87 (2012).
- Cervera-Juanes, R., Wilhelm, L. J., Park, B., Grant, K. A., Ferguson, B. Alcohol-dose-dependent DNA methylation and expression in the nucleus accumbens identifies coordinated regulation of synaptic genes. Translational Psychiatry. 7 (1), e994 (2017).
- Cervera-Juanes, R., Wilhelm, L. J., Park, B., Grant, K. A., Ferguson, B. Genome-wide analysis of the nucleus accumbens identifies DNA methylation signals differentiating low/binge from heavy alcohol drinking. Alcohol. 60, 103-113 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır