Method Article
Fisyon mayası Schizosaccharomyces pombe derin sıralama yoluyla, kendiliğinden bastırıcı ekran
Bu Makalede
Özet
Biz Maya fizyon basit bastırıcı ekran protokolünde mevcut. Bu yöntem etkili, alkil-Alerjik ve kez tek bir genomik locus meydana gelen mutasyonlar için seçici değildir. Protokol bir mutasyon veya bir uyuşturucu nedeniyle büyüme kusurları sıvı kültüründe hafifletmek bastırıcılarının yalıtmak için uygundur.
Özet
Bir mutasyon kaynaklanan fenotipik kusurlar bastırmak mutant gen için genetik bir ekran yakından ilgili biyokimyasal yolları ait genlerin tanımlamak için güçlü bir yaklaşımdır. Önceki yöntemler sentetik genetik dizi (SGA) analiz ve ultraviyole (UV) veya etil methanesulfonate (EMS) veya N-etil-N-nitrosourea (TRK), gibi kimyasallar kullanarak rastgele mutagenesis teknikleri gibi yaygın olarak kullanılan ancak çoğu kez pahalı ve zahmetli. Ayrıca, bu alkil tabanlı tarama yöntemleri sık bastırıcılarının ayırma karmaşıklığına ek birden çok mutasyonlar inducing organizma üzerinde ciddi yan etkileri ile ilişkilidir. Burada, hangi Schizosaccharomyces pombebir büyüme üründe görüşmek mutantlar mutasyonların bastırıcı tanımlamak için basit ve etkili bir iletişim kuralı mevcut. Fitness standart zengin sıvı veya sentetik sıvı ortamlardan bir büyüme eksikliği içeren hücrelerin üzerinde uzun bir süre bir otomatik 96-şey plaka okuyucu kullanarak kurtarma için izlenebilir. Bir hücre kültürü bir bastırıcı mutasyon satın aldı sonra alt öğelerin bu ebeveyn hücre kul. Ebeveyn hücreleri üzerinde bir rekabetçi büyüme avantaj içeren kurtarılan hücreleri izole ve ebeveyn hücreleri ile backcrossed. Bastırıcı mutasyon sonra bütün-genom sıralama kullanılarak tanımlanır. Bu yaklaşımı kullanarak, başarılı bir şekilde Elf1, bir AAA + aile nükleer mRNA taşıma ve genomik istikrar bakımından önemlidir ATPaz kaybı nedeniyle ciddi büyüme kusurları hafifletmek birden çok bastırıcılarının izole ettim. Sitemizde şu anda 400 genler üzerinde S. pombe içinde büyüme hata conferring mutantlarla. Bu genlerin uncharacterized çoğu gibi bizim yöntemi kullanıcı dostu, yüksek-den geçerek bu yaklaşımla roman fonksiyonel etkileşimleri tanımlaması acele edecektir öneriyorum.
Giriş
Genler arasındaki fonksiyonel bağlantılar anlayışının temelini hangi tarafından çeşitli fenotipleri1üretmek için karmaşık genetik özellikleri sapmak moleküler mechanism(s) tanımlama yeteneği dayanır. Fisyon mayası, Schizosaccharomyces pombe (S. pombe), protein kodlayıcı genlerin çoğunluğu canlılık2için gereksiz. Bu sonuç, bu genlerin önemsizlik ama oldukça karmaşık telafi edici mekanizmaların böyle genler ait biyokimyasal yolları temel konuşmuyor. Bu telafi edici mekanizmaların dissekan kapsamlı genetik etkileşimler ele geçen ve fonksiyonel biyokimyasal yolları3,4anlayışımızı genişletti epistasis haritalar üretti.
Yüksek üretilen iş yöntemleri (örneğin, sentetik genetik dizi analizi veya ÖYD) mayası genom genelindeki genetik etkileşimler tanımlamak ve fisyon mayası5,6kullanmak için genişletilmiş için geliştirilmiştir. Bu tür yaklaşımlar, çoğunlukla tüm uygun tek protein kodlayıcı gen silme içeren suşları içeren bir kitaplık kullanır (kapsayan yaklaşık 3300 haploit silme mutantlar fisyon mayası genom üzerinde % 92) ve genetik haçlar arasında gerçekleştirmek için bir robot kol gerektirir ilgi ve library6 tüm olası suş strain. Ayrıca, SGA teknikleri doğru ve verimli çiftleşme yeteneğimizi Kütüphane suşlarının üzerinde bağlıdır, şu anda 444 için anormal bir fenotip S. pombe2genler ile karakterizedir.
Genetik etkileşimler karmaşıklığına rağmen büyük bir yük için bireysel mutasyonlar her gen taşıyan iki suşlar fenotip iki genlerdeki mutasyonlar taşıyan fenotip karşılaştırma birinde iki önemli sonucu olabilir: 1) Çift Kişilik mutant fenotip olduğunu beklenen çarpımsal ebeveyn fenotipleri hastalık şeklinde veya en aşırı durumda, ölümcül daha kötü. Bu, negatif bir genetik etkileşim adlandırılır ve genellikle iki gen paralel biyolojik yollar hareket bir işarettir. 2) Çift Kişilik mutant fenotip ebeveyn fenotipleri, olarak da bilinen olumlu bir genetik etkileşim beklenen kombinasyonu iyidir. Olumlu bir genetik etkileşim özellikle ilginç çünkü bu genlerin aynı işlemde işlev gösterir. İki olumlu etkileşen genlere sahip üç olası ilişkileri: bir mutant gen up-paralel bir yol diğer gen ifadesinin düzenleyen, iki gen birbirine aynı yolu aşağı içinde konserde çalışabilir veya iki gen kodlamak birbirleriyle doğrudan etkileşim proteinler. Bu nedenle, pozitif genetik etkileşimler gen düzenleyici düğümleri eşleme ve biyokimyasal yolları7,8uncharacterized genlerinde sınıflandırmak için kullanılabilir.
Bir bastırıcı genellikle iki gen9,10arasında pozitif bir genetik etkileşim temsil eden başka bir gen mutasyon hastalık fenotip hafifletmek olabilir bir mutasyon var. Onlar bastırmak mutasyon bundan farklı bir odağı üzerinde baskılayıcı mutasyonlar extragenic bastırıcılarının bilinir. Onlar sentetik öldürücü fenotip (Lazarus etkisi olarak da bilinir)11tahlisiye tarafından uygun olmayan Genetik mutasyonlar okumak özellikle değerlidir. Onlar da kalıtsal hastalıklar12,13tedavisinde potansiyel terapötik uygulamalar var.
Tüm bu nedenlerle, çeşitli model organizmalar mutasyonların bastırıcı tanımlaması yaygın olarak çeşitli biyokimyasal yolları14,15,16anlayışımızı kolaylaştırmak için kullanılmıştır. Tarama için bastırıcılarının genellikle söz konusu mutasyon fenotip üzerinde temel alır ve fenotip hafifletmek mutasyonlar yalıtmak için rastgele mutagenesis iletken gerektirir. Hemen hemen tüm canlılar rasgele mutagenesis yöntemleri kurduk. Örneğin, N-etil-N-nitrosourea (TRK) ve ethylmethanesulfonate (EMS), nokta mutasyonlar DNA inducing Kabil, yaygın olarak çeşitli bakteri fareler17,18,19 modelleri ve istihdam edilmektedir iki değiştirme . Buna ek olarak, mangan klorür uzun Mayalar manganez katyon yetenek DNA onarım yolları20inhibe için kullanılmıştır. Başka bir ortak yaklaşım genom çapında mutajenik pirimidin dimer21,22oluşturur UV kaynaklı mutagenesis var.
Bastırıcı mutasyonlar tanımlamak için kimyasal mutagenesis kullanımı popüler olmasına rağmen birçok sakıncaları, tehlikeli kimyasallar, son derece değişken başarı oranları ve giriş ekstra karıştırıcı değişkenlerin kullanımı da dahil olmak üzere yöntemi vardır birden çok hücresel23,24alkil olumsuz yan etkileri tarafından sunulan. Ayrıca, kimyasal mutagenesis kez birden çok karmaşıklığı kullanarak genetik ve organizma25bastırıcı fenotip haiz tam mutasyonu tanımlamak için sıralama teknikleri ekler genom mutasyonların neden olmaktadır.
Güncel mutagenesis yaklaşımları eksiklikleri gidermek için herhangi bir değiştirme veya silme kitaplıkta dayanmaz fisyon mayası spontan bastırıcı mutasyonlar için ekran için bir yöntem mevcut. Yöntem bastırıcılarının pozitif seçim tahlil ile izole ediyor. Bu yöntem prensibi bir otomatik plaka okuyucu tarafından izlenebilir sıvı kültüründe mutasyona uğramış bastırıcı subpopulation büyüme avantajı temel alır. Sadece bir genetik arka plan kadar temiz ya da monogenic allelleri bastırıcılarının bütün-genom sıralama önce varlığını doğrulamak istiyorsanız çiftleşme ve mayoz kullanılır. Bastırma fenotip tek bir mutasyon neden olup olmadığını, bastırıcı fenotip 2:2 ebeveyn suşları ile backcrossing sonra ayırmak. Bastırıcı mutasyon sonra bütün-genom sıralama kullanılarak belirlenebilir. Önerdiğimiz bu yöntem büyük bir nüfus için sıvı kültüründe büyüyebilir tüm mikroorganizmaların bastırıcılarının eleme için geçerlidir.
Protokol
1. inşaat ve hazırlık süzüyorlar
- Bir mutasyon veya silme işlemini gen (yfm, senin favori mutasyon)26daha önce açıklandığı gibi standart site yönettiği mutagenesis (SDM) kullanarak oluşturun.
- Ekranın başlamadan önce mutant suşları ile genetik arka plan temiz ve taze d. mutant hücreler ebeveyn suşları olarak oluşturmak için bir vahşi tipi yük (en iyi şekilde) backcross. Çizgi ebeveyn zorlanma bireysel kolonileri için standart zengin medya plakaları. Rastgele plaka okuyucu tahlil için istenen mutasyonlar ile 8-16 bağımsız koloniler (biyolojik çoğaltır) seçin (bkz: 3.1).
Not: Bu protokol ebeveyn suşları sıvı ortamda (az ya da zengin, ile veya olmadan uyuşturucu veya büyüme kusur neden sıcaklık vardiya ile) defect bir büyüme olduğunda etkilidir. Tüm ebeveyn suşları haploit ve böylece genetik olarak tamamlayıcı bir çiftleşme türüyle haploit diğer suşları ile geçti mümkün olmalıdır.
2. plaka okuyucu tahlil
- Steril bir aplikatör ile her (yok tam olarak ne kadar başlangıç aşılamak gerekli kültür) adımda 1.1 hazırlanan kolonileri az miktarda alıp bir 96-şey polistren Mikroplaka yerleştirin. Her biri 200 µL uygun sıvı medya (zengin ya da çok az, ya da uyuşturucu olmadan) kolonilerde askıya alma. Her satır içeren 200 µL aynı medya (hücre) tepside için boş bir kuyu bulunmaktadır.
- Bir plaka okuyucu algılama yazılımı aşağıdaki protokol bağlı bir otomatik Mikroplaka okuyucu için Çalıştır: sürekli hızlı yörünge sallayarak (425 BGBM, 3 mm genlik) ile 30 ° c 24 saat ve sıcaklık için Kinetik bir program ayarla. Bir dalga boyu 600nm optik yoğunluk için IR ışık saçma ölçmek için optik okuma ve plaka 2 dk (iyi başına 24 Saat süre içinde toplam 721 okuma) bir okuma frekansta aşağıdan okumak için ışığı ayarlayın.
- = 0.1 sonra 24 h, son beyaz optik yoğunluk ölçümleri (beyaz OD600) kaydetmek ve birimin her doz aşağı örneklerinin sulandırmak için gerekli belirlemek için aşağıdaki formülü kullanın:
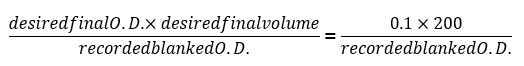
Not: plaka okuyucu yazılım verileri verme ve toplu bir işleve işlemek seyreltme birim deneysel her kuyudan kullanılmak üzere yukarıdaki formül eklemek için bir elektronik tablo yazılımı kullanın. - Her 24 saat, her gün doz 0 olarak aynı ortamı kullanarak örnekleri seyreltik = 0.1 (yaklaşık 1.5 x 106 hücre/mL) formülü kullanarak belirtilen adım 2.3. Tüm büyüme eğrileri günlük oluşturulan ve kohort aynı genetik geçmişi olan diğer önemli ölçüde daha yüksektir bir son doz veya için benzer bir büyüme eğrisi tarafından değerlendirilecektir bir artan büyüme oranı gösterir herhangi bir bireysel koloni Not kaydetme vahşi-türü koloniler.
Not: Bu tahlil genellikle yaklaşık 7-14 gün sürer. Steril koşullar altında tüm adımları gerçekleştirin.
3. fenotip ile bastırıcı kolonileri ve seçimi.
- Plaka okuyucu tahlil (Adım 2.4) son günden itibaren ebeveyn mutasyon fenotip hafifletmek olabilir bir bastırıcı mutasyon kazanıyor tarafından belirgin bir şekilde kurtarılan büyüme oranı, muhtemelen var sıvı kültürleri kaydedin. Aktarım ve 250 µL sıvı kültür 250 µL % 50 gliserol içeren bir cryotube için karıştırın. Flaş hücreler sıvı azot içinde dondurmak ve suşların 80oC-süresiz olarak kaydedin.
- Bastırıcı mutasyon genetik kalıtsal bir öğe olduğunu doğrulamak için yfm P çapraz için standart genetik geçiş yöntemleri kullanın (için ebeveyn, zorlanma kullanılan plaka okuyucu tahlil başında) yfm S (için bastırıcı, vasıl belgili tanımlık son kaydedilen zorlanma ile plaka okuyucu tahlil). Bastırıcı mutasyon gerçekten bir genetik kalıtsal unsur ise, yfm P × yfm S iki kolonileri hastalık fenotip ebeveyn baskı var ve iki kolonileri bastırıcı kurtarılan büyüme oranına sahip tetrads verim zorlanma.
- Adım 3.2 çapraz bastırıcı fenotip (S zorlanma) ile üç kolonileri almak ve aynı genetik ebeveyn fenotip (P zorlanma) ile üç koloniler (her biri için 3 Biyolojik çoğaltır) çapraz ve genomik DNA ekstraksiyon ile devam edin ve sıralama aşağıdaki adımları.
Not: Adım 3.2 ve 3.3 kesinlikle önerilir, ancak gerekli değildir. Alternatif olarak, bir 3.1 zengin medyada tek koloniler toplanan kurtarılan sıvı kültür yayıldı, o zaman rasgele olarak daha fazla genetik onay olmadan bütün-genom sıralama için biyolojik triplicates üç kolonileri almak. Bu durumda, üç biyolojik triplicates ebeveyn baskı genomik sıralama karşılaştırma için kullanılmalıdır.
4. genomik DNA ekstraksiyon, Kütüphane üretim ve sıralama.
- DNA ekstraksiyon, kitaplık hazırlama ve sıralama, rastgele seç yfm P zorlanma başına üç biyolojik çoğaltır ve üç biyolojik çoğaltır her tek tek ortaya yfm S suşların genetik haçlar (Adım 3.2) veya gelen Tek kolonileri S baskı (Adım 3.3 Not) elde etmek için yaymak levha.
- Suşlarının 10 mL kültürlerde zengin medya orta log fazı için büyümek (OD = 0.5 – 0.8, 0,75-1.2 x 107 hücre/mL hakkında) ve sürekli 250 devir / dakikada sallayarak ile sıvı kültürleri 30 ° C'de büyümeye titreyen bir kuluçka makinesi kullanın. Santrifüjü 1000 x gde 5 min için 4 ° C'de tarafından hücreleri toplamak.
- Pelleted hücrelerde DNA ekstraksiyon arabelleği 400 µL askıya alma (%2 Triton X-100, % 1 SDS, 100 mM NaCl, 10 mM Tris-Cl (pH 8.0), 1 mM Na2-EDTA), daha sonra 400 µL cam boncuk ve 400 µL 25:24:1 fenol: kloroform: izoamil alkol ekleyin. 4 ° C'de 2 min için şiddetle girdap
- Birkaç kez ters çevirme tarafından ek bir 200 µL DNA ekstraksiyon arabellek ve karışımı ekleyin. Santrifüj 20.000 x gde 4 ° C'de 5 min için.
- Sulu faz için temiz bir tüp transfer 20 µg RNase A/T1 karışımı ekleyin ve 15 dakika 37 ° C'de kuluçkaya.
- 25:24:1 fenol: kloroform: izoamil alkol, spin, 20.000 x g, 4 ° C'de 5 min için eşit bir birim eklemek sonra temiz bir tüp sulu faz transfer.
- Kloroform eşit bir birim eklemek, birkaç kez, ters çevirme karıştırın sonra 20.000 x gde 4 ° C'de 5 min için spin sonra sulu faz için temiz bir tüp aktarmak.
- % 100 etanol iki birim artı % 10-20 ° c için en az 2 h, 3 M NaOAC (pH 4,3) hacmi ile acele DNA sonra 20.000 x g ve toplamak Pelet 4 ° C'de 5 min için spin.
- Pelet (çöktürülmüş DNA) iki kez soğutulmuş % 70 etanol (santrifüj 20.000 x g, 5 dk, 4 ° C) ile yıkama ve Pelet 10 mM Tris arabellek (pH 7,4) 50 µL içinde askıya alma.
- Bir kütüphane hazırlık Kiti ( Tablo malzemelerigörmek) bütün-genom sıralama kitaplığı hazırlamak için üreticinin önerilerini kullanın.
Not: PCR güçlendirme sırasında oluşturulan hata mutasyonlar en aza indirir PCR amplifikasyon olmadan genomik Kütüphanesi inşaatı verdiğinden Malzemeleri tabloda listelenen kiti tavsiye ediyoruz. Buna ek olarak, genomik Kütüphanesi hazırlık sırasında boncuk için izin verme zamanı 1-2 dk kurutma boncuk kısalma tarafından tamamen kuru. - Kütüphane hazırlık sırasında parametreleri yamultma için odaklanmış bir sonicator kullanın (bkz. Tablo reçetesi) ve % 20, en yüksek güç için 175 W, patlama, başına 200 döngü ile görev faktör ayarlayın ve frekans süpürme modu 5.5oC-6 ° c 45 için alternatif olarak s., kullanmak bir DNA ve sistem kesme chomatin ( Tablo malzemelerigörmek) aşağıdaki ayarlarla: pulse modu, 15 ile 4 ° C'de % 50 genlik s 20 dk bir toplam işlem süresi ile 10 min için kapalı açık ve 15 s.
- İtina ile bu adımda kullanılan tehlikeli maddeleri işlemek için gereklidir. Uygun malzeme güvenlik bilgi formları ve kurumun çevre sağlığı ve güvenliği Office NaOAC, etanol, 25:24:1 fenol: kloroform: izoamil alkol ve kloroform işlemek için başvurun.
- Elde edilen genomik kitaplıkları sıra. Tüm sıralama okuma en az üç kez bir tek nükleotid menzil çözünürlüğü ile tüm genomunun kapsamalıdır. Sıralama eşleştirilmiş sona erdi (veya son teknoloji) önerilir.
5. Biyoinformatik analizi bastırıcı mutasyonlar tanımlanması için
- Sürekli olarak ebeveyn arasında tanımlanır genomik değişiklikleri odaklanmak için Biyoinformatik analizi yapmak ve tüm biyolojik bastırılmış yfm suşları çoğaltır.
Not: Tam potansiyel satış işlemi aşağıda açıklanmıştır ama buna ek olarak, iki düz metin BASH komut dosyalarını, fastq_to_vcf.sh ve vcfprocess.sh, örnekler okur işleme iş akışı VCF değişken göstermek için ek malzeme olarak eklenir dosyaları ve işleme ve VCF kesişimi dosyaları anılan sıraya göre. - Trim kısa-Okunma: komut satırları (tüm diğer seçenekleri varsayılan) takip kesme (https://github.com/jbpease/shear) kullanarak
Shear.py--fq1 $FASTQ1 - fq2 $FASTQ2--out1 $OUTFQ1--out2 $OUTFQ2 \
-barcodes1 $BARCODE--platform TruSeq--trimqual 20:20 \
--trimpolyat 0--trimambig--filterlength 50--filterunpaired - Harita, BWA v0.7.1527kullanarak PomBase (ftp://ftp.ebi.ac.uk/pub/databases/pombase/pombe/Chromosome_Dumps/fasta/) elde edilen S. pombe başvuru genom v2.30 okur. (Tüm diğer seçenekleri varsayılan) aşağıdaki komut satırını kullanın:
BWA mem -t 8 $GENOME $OUTFQ1 $OUTFQ2 > $SAM1 - Hizalama SAM dosyaları GATK koymak en iyi yöntemler28 varyant Aradığınız GATK v3.629, PicardTools v2.5.0 (http://broadinstitute.github.io/picard) ve SAMtools v1.3.130kullanarak için boru hattı. Aşağıdaki komut satırlarını ve parametreleri (tüm diğer seçenekleri varsayılan) kullanın:
Java-Xmx30g-picard.jar AddOrReplaceReadGroups giriş kavanoz $SAM1 = \
ÇIKIŞ $BAMMARKED RGID = 1 RGLB = lib01 RGPL = illumina = \
RGPU $BARCODE RGSM = $SAMPLENUMBER =
samtools fixmate - O bam $BAMMARKED $BAMFIXED
-O bam -o $BAMSORTED -T /home/peasejb/tmp $BAMFIXED samtools sıralama
samtools dizin $BAMSORTED
Java-Xmx30g-GenomeAnalysisTK.jar -T kavanoz HaplotypeCaller \
-R $GENOME-ı $BAMSORTED..... genotyping_mode bulma \
-stand_emit_conf 10 - stand_call_conf 30 -o $VCFRAW - Tabix kullanarak sıkıştırmak ve dizin VCF dosyaları:
bgzip $VCFRAW.vcf
tabix $VCFRAW.vcf.gz - Ebeveyn arasında VCF dosyaları karşılaştırmak ve bastırıcı suşları BCFtools v1.3.127kullanarak sıralama çoğaltır. Aşağıdaki komut satırlarını ve parametreleri (tüm diğer seçenekleri sol varsayılan) kullanın:
bcftools isec - n + 1 $VCFPARENTAL1.gz $VCFPARENTAL2.gz $VCFPARENTAL3.gz \
$VCFMUTANT1.gz $VCFMUTANT2.gz $VCFMUTANT3.gz > common_variants.list
Not: Bu komut nerede ilk mutant yalnızca görünen sıra türevleri "000100", "000010", "000111," olarak tüm üç mutantlar olarak sadece ikinci mutant olarak ikili kodlanmış ikili desenleri ile kodlanmış bir dosya vermiştir vs. Bu dosyalar ebeveyn her kümesi için oluşturulan ve mutant VCF dosyaları çoğaltır. - Varyant kavşak listesi dosyalarını UNIX grep komutunu kullanarak her satır için eklenen dosya adıyla birlikte derlemek:
GREP "." *.list > all.list - Özel bir Python komut dosyası (variant_characterize.py) kullanarak tam değişken listesi geçerli GFF3 ek açıklama dosya (ftp://ftp.ebi.ac.uk/pub/databases/pombase/pombe/Chromosome_Dumps/gff3/schizosacchar omyces_pombe.chr.gff3) ile çapraz başvuru protein kodlayıcı bölgeler (eş anlamlı ve eşanlamlı sigara), 5've 3'utrs ve ncRNA tutarlı SNP siteleri tanımlamak için.
python3 variant_characterize.py--listesi common_variants.list \
--gff schizosaccharomyces_pombe.chr.gff3 \
--fasta Schizosaccharomyces_pombe. ASM294v2.30.DNA.Genome.FA \
--000100--all.list.filter.000100 dışarı desen
Bu komut dosyası değiştirme tekrar--desen ve sonek çıktı dosyasının (--dışarı) ikili kullanma
desenler: 000010, 000001, 000110, 000011, 000101 ve 000111 - Bir elektronik tablo olarak görüntülenmek üzere bir sekmeyle ayrılmış dosya, tüm bu komut dosyası çalışır çıktısı birleştirin. Ek açıklama eklenen tablo versiyonlarının görünen bir veya her iki mutant strain(s) arka göre içerir. Bir tek mutant suşu (000100, 000010, 000001), her iki görünüm ikili bayrak alanını gösterir iki mutant suşları (000011, 000101, 000110) veya tüm üç mutant suşları (000111).
- Açıklamalı çıkış listeleri ebeveyn örneklerinde bulunamadı ama bir, iki veya tüm üç mutant örnekleri buldum versiyonlarının analiz. Ek açıklamayı sınıf Variant (eşanlamlı/sigara-eş anlamlı bir kodlama bölgesi, 3' / 5' UTR, kodlamayan, vb.) ve genomik konumunu gösterir. Aday mutasyonlar bu listeden, örnek olarak bir şiddetle ilgili adayların sürekli tüm üç suş görünme eş anlamlı olmayan bir kodlama değişken olabilir. Güçlü aday, başka tür bir birikimi birbirine yakın veya aynı gen içinde görülen mutant suşları çeşitli eşanlamlı sigara veya sözde düzenleyici mutasyonlar olur.
Sonuçlar
Yavaş büyüyen mutantlar fenotipik kurtarma sıvı kültüründe göster.
Biz üç mutantlar dahil ile hasta, yavaş büyüyen, fenotip biyolojik yollar çeşitli seçilmiş: AAA aile ATPaz Elf1, Histon Deacetylase Clr6 ve Exon Junction karmaşık bileşen Fal1. Bir vahşi tipi yük ve vahşi-türü suşları ile backcrossed bu üç gen mutasyon taşıyan suşları için bireysel kolonileri çizgili ve 16 tek koloniler olarak 96-şey plaka kullanarak zengin sıvı ortamda kültürlü olmak rastgele seçilmiştir yukarıda açıklanan. Büyüme eğrileri bireysel kolonileri ilk zamanda noktada (0 gün) ve 6 gün boyunca sürekli izlenmesi plaka okuyucu kullanarak kaydedildi. Beklendiği gibi vahşi tipi Koloni yok göze çarpan değişiklikler onların büyüme eğrileri boyunca deneme31 (şekil 1) gösterir. Özellikle, elf1∆ arka plan ile dört kolonileri ve bir fal1∆ koloni dramatik bir değişim yavaş büyüyen büyüme bazı düzeylerde büyüme için benzer veya bu vahşi tipi kolonileri yakın gösterir. Önemli ölçüde, tüm clr6-1 mutantlar tahlil31 (şekil 1) sonuna daha hızlı bir oranda büyüyen bir tutarlı fenotipik kurtarma göster. Farklı fenotipleri karakterize etmek için yavaş "P suşları" (ya da ebeveyn suşları) olarak büyüyen özgün suşları için ve fenotipik kurtarma "S suşları" (ya da bastırılmış suşları) olarak gösterilen suşları için başvurun. Unutmayın Resim 1 tarama deneme bir tur, bir örnektir ve toplam tamamlayıcı bastırıcılarının tespit ve aşağıdaki temsilcisi sonuçlarında sıralı temsil etmiyor.
Fenotipik kurtarma için kalıtsal özellikleri atfedilen
S. pombe haploid zengin medya ama tamamlayıcı çiftleşme türleri dostum altında azot açlık ile iki haploit suşları olarak büyüyebilir. Fisyon mayası mayoz hücre bölünmesi iki tur tarafından takip çoğaltma bir tur gerektirir. Cinsel döngüsü ebeveyn suşu genetik malzeme ile genetik özellikleri klasik Mendel genetik (şekil 2A) kurallarına ayrılığı 2:2 taşıyan dört haploit sporlar oluşumu sonuçlanır. Arka-crossing tüm suşları (S suşları) 2 küçük (büyüme kusur) ve 2 büyük (bastırıcı fenotip) yılında onların ebeveyn suşları (P suşları) ile bastırılır zaman zaman zaman aynı miktarda için aynı plaka üzerinde büyüdü, 2:2 segregasyon doğruladık koloniler. Bastırılmış elf1∆, clr6-1 ve fal1∆ hücreler için tek tek örnekler şekil 2Biçinde gösterilir. Biz tüm izole S suşları onların P suşlarının (veri gösterilmez) yavaş büyüyen fenotip bastırır monogenic genetik öğe taşımak doğruladı.
Bütün-genom sıralama başarıyla bastırıcı mutasyonlar tanımlar
Örnek olarak, elf1∆ S suşları fenotipik kurtarma sorumlu genetik öğeleri tanımlamak için eşleştirilmiş uç tüm genom sıralama kullanılmıştır. Veri çözümleme hakkında daha ayrıntılı açıklamalar mevcut online31' dir. Kısaca, iki bağımsız olarak yaratılan elf1∆ P suşlarının biyolojik triplicates ve biyolojik elf1∆ S suşları, her biri farklı bastırıcılarının içeren beş tamamlayıcı grupları kopyası istihdam. Biyoinformatik analizi (6.1-10), ek açıklama eklenen varyantları listesini elde sonra bazı sınıflar analiz için ilgili versiyonlarının öncelik. Biz onların ebeveyn elf1∆ P suşları ile (şekil 3 ve ek tablolar 1-4 göre bireysel elf1∆ S suşlarının biyolojik çoğaltır, tümünde aynı tutarlı genomik değişiklikler tanımlaması üzerinde duruldu ). Rli1 +, SPBPJ4664.02, cue2 + ve rpl2702 +da dahil olmak üzere tüm beş farklı elf1∆ S suşları, CD bölgelerde, beş nonsynonymous değişiklikleri tespit edilmiştir. Mutasyonlar farklı amino asitler rastlanmasa da S-A1 ve S-A2 mutasyona uğramış SPBPJ4664.02, içerir. SPBPJ4664.02 ile yüz-in tekrarlar uzun gen (11,916 nukleotid) olduğundan, mutasyonlar sıralama tarafından takip PCR gerçekleştirerek onaylanması koyamadık. S-A3 rli1 her iki biyolojik çoğaltmaları tutarlı olarak silinmesi mutant içerir. Ancak, mutant elf1∆ arka planda S fenotip ile birlikte ayırmak değil. Biz bir cue2 mutant (cue2-1) S-B1, amino asitler ile 396-400 eksik tespit. S-B2 pozisyonda 45 amino asit glisin aspartat31olarak değiştirir bir rpl2702 mutant (rpl2702-1) içerir. Cue2-1 ve rpl2702-1 elf1∆ bastırıcılarının aşağıda gösterildiği gibi teyit edildi.
Tanımlanan bastırıcı mutasyonların genetik onay kurtarma fenotip kalıtsallığı doğrular
İki tespit nonsynonymous değişiklikler, cue2-1 ve rpl2702-1, site yönettiği mutagenesis için standart protokolleri kullanarak laboratuvarda yeniden. Çift Kişilik mutant suşları cue2-1 elf1∆ P ve rpl2702-1 elf1∆ P ücretsiz elf1∆ P zorlanma31 ile (şekil 4) kesişti. Bu ekran üzerinden tespit nonsynonymous mutasyonlar elf1∆ Pbastırmak için yeterli olsaydı, o zaman elde edilen tetrads 2:2 her dörtlü 4 sporlar kaynaklanan sömürgelerinde büyük oranı küçük gösterir. Gerçekten de, genetik geçiş tespit bastırıcı mutasyonlar elf1∆ p yavaş büyüyen fenotip baskılayarak başarılı ve kalıtsal gösterdi.

Şekil 1: fenotipik kurtarma izlenen bir plaka okuyucu olarak büyüme eğrileri kaydederek. On altı tek kolonileri vahşi-türü (WT), elf1∆, clr6-1ve fal1∆ bir 96-şey tabağına yerleştirildi. Büyüme eğrileri 24 Saat süre içinde kaydedildi ve koloniler zengin medya günlük yeniden seyreltilmiş. Büyüme kusur gün 0 24 saat döneminin sona düşük Absorbans (OD) tarafından belirgindir. Phenotypically kurtarılan suşları için görüntüleyen bir büyüme eğrisi benzer, ya da yakın, o vahşi-türü 24 Saat süre günde 6. Elf1∆, fal1∆bir kolonisi ve clr6-1 ' in tüm kolonileri dört kolonileri fenotipik kurtarma çeşitli düzeylerde 6 gün sonra gösterdi. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
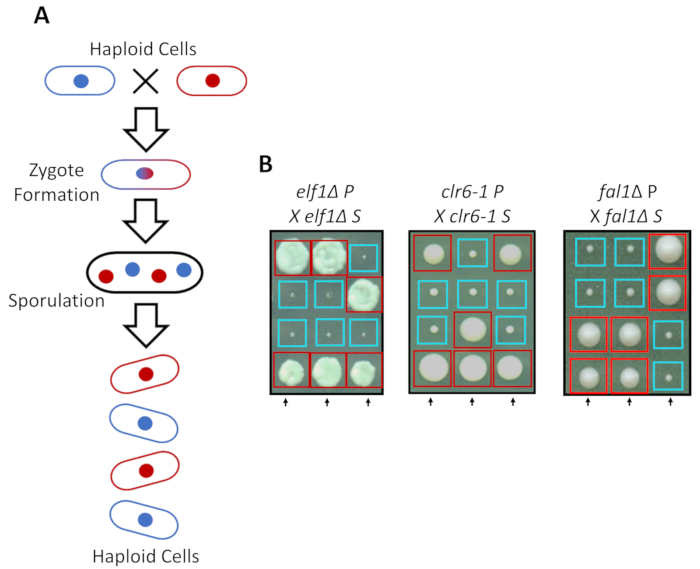
Şekil 2: genetik geçiş fenotipik kurtarma için tek bir kalıtsal alleli atfedilir onaylayabilir. (Fisyon mayası hücreleri azot açlık, tamamlayıcı çiftleşme türü olan iki haploit hücreleri tabi 4 sporlar dörtlü oluşturmak için sporulates bir zigot oluşturduğunuzdaA). Ebeveyn genetik malzeme Mendel genetik kurallarına mayoz sırasında ayırmak. (B) Phenotypically kolonileri kurtarılan (etiketli S, bastırılmış için) ebeveyn genotip ücretsiz ebeveyn kolonilerine ile geri çarpı işareti ile gösterilir (hangi gösterir için P, etiketli fenotipik hiçbir kurtarma ebeveyn). 2:2 küçük gösterilen genetik haçlar (zavallı fitness) büyük (kurtarılan fitness) koloniler için fenotipik kurtarma kalıtsal ve tek bir genetik öğeye bağlanabilir göstermektedir. Kırmızı kutuları bastırıcı alleli taşıyan koloniler, ve mavi kutu ebeveyn alleli taşıyan koloniler. Bu rakam üzerinden Marayati ve ark., 201831değiştirildi. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
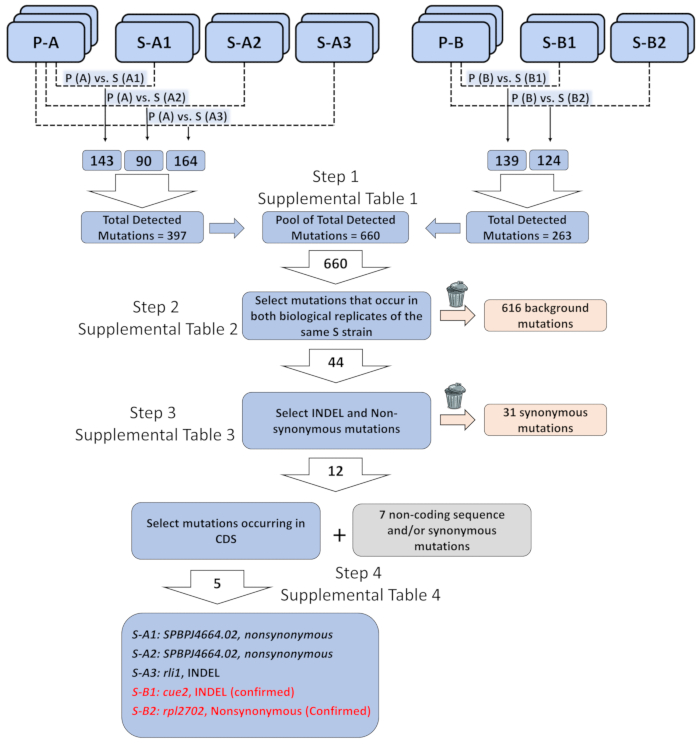
Şekil 3: genom çapındaki sıralama veri analizi genetik öğeleri fenotipik kurtarılması sorumlu tanımlamak için. Üç biyolojik çoğaltır iki ebeveyn "P" soyu (P-A ve P-B), ve beş phenotypically kurtarılan iki biyolojik çoğaltır açık "S" suşları (S-A1, S-A2 ve A3 S - kurtarılan P-A; S-B1 ve S-B2 P-b), sıralı ve mutasyonlar kurtarılan zorlanma elde edilen (örneğin, P-A S-A1, vb.) ebeveyn zorlanma genom karşılaştırıldığında her mutasyon listesi olarak organize edilmiştir. Böyle ikili karşılaştırmalar algılanan mutasyonlar tüm genom genelinde toplam sayısı 660 yapıldı. Toplam 44 mutasyonların aynı "S" zorlanma her iki biyolojik çoğaltır içinde oluşan tek mutasyonlar seçildiğinde tespit edildi. 44 mutasyonlar dışında 12 mutasyonlar ekleme/silme (INDEL) veya non - eş anlamlı mutasyonlar vardı. 12 dışarı INDEL veya eş anlamlı olmayan mutasyonlar, beş sıra kodlama protein oluştu. Beş mutasyonlar potansiyel olarak phenotypically kurtarılan suşları için sorumlu tek genetik öğe ile ilişkili: S-A3, cue2 INDEL INDEL rli1 içinde bulunan S-A1 ve S-A2, SPBPJ4664.02 eş anlamlı olmayan bir mutasyon bulunan S-B1 ve eşanlamlı S-B2 içinde bulunan rpl2702 mutasyonların bulundu. Detaylı sırası bilgi mutasyonlar ve filtre uygulanmış arka plan üzerinde ek tablolar 1-4' e eklenmiştir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
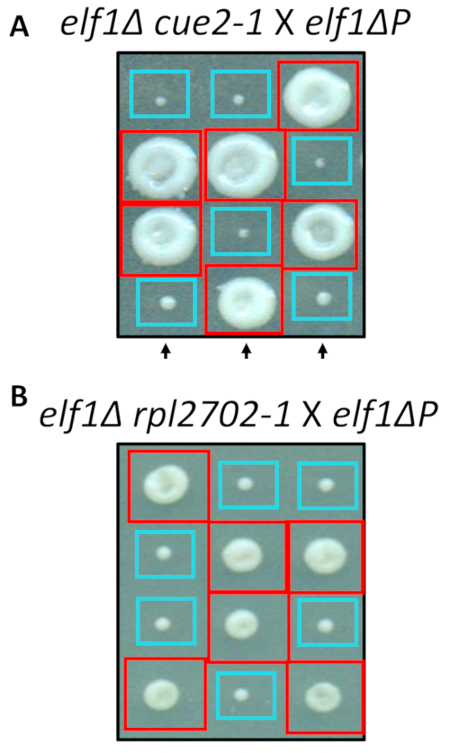
Şekil 4: Bütün-genom sıralama yoluyla tespit bastırıcılarının onayı. Bütün-genom sıralama sonuçlarından bağımsız olarak mutasyonlar üreten ve gerçekleştirme fenotipik kurtarma bir elf1∆ P zorlanma ve elf1∆ bir elf1∆ cue2-1 türe geçiş tarafından onaylamak için genetik geçer doğrulandı rpl2702-1 elf1∆ P zorlanma ile. Üç temsilcisi dikey tetrads gösterilir. Kırmızı kutuları vardır Çift-mutant koloniler (elf1 cue2-1veya elf1 rpl2702-1); Mavi kutuları elf1∆ kolonileri vardır. Bu rakam üzerinden Marayati ve ark., 201831değiştirildi. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Ek tablo 1. Bu tabloyu indirmek için buraya tıklayınız.
Ek tablo 2 . Bu tabloyu indirmek için buraya tıklayınız.
Ek tablo 3 . Bu tabloyu indirmek için buraya tıklayınız.
Ek tablo 4 . Bu tabloyu indirmek için buraya tıklayınız.
Ek dosyaları kodlama . Dosyaları indirmek için buraya tıklayınız.
Tartışmalar
Burada açıklanan Protokolü temsil eden bir roman ve spontan bastırıcı mutasyonlar fisyon mayası, bir fenotip S. pombe, 400'den fazla genlerin karakteristik yavaş büyüme veriyor mutasyonların fenotipik kurtarma yoluyla tespit için basit ekran Bunların çoğu fonksiyonun bilinmeyen2,32kalır. Önceki yöntemler mikroorganizmaların mutajen21kullanımı veya uygulamanın bir sıcaklık değişimi sıcaklığa duyarlı mutant arka planlar33olarak da dahil olmak üzere, mutasyonların bastırıcı için ekran için diğer yaklaşımlar almış. Buna ek olarak, bu iletişim kuralını fenotipik kurtarma ek çevre/kimyasal sorunsuz oluşur ve sıvı içinde kullanılabilir kaynakları sonunda devralarak bastırma mutasyonlar yükselişi fitness avantajı vurgular gösterir Kültür. Elf1∆ veya fal1∆ gibi her iki işlev kaybı mutasyonlar ve nokta mutasyon gibi clr6-1, mutantlar olarak sürece etkilidir çünkü bu ekran bypass bastırıcılarının veya etkileşim bastırıcılarının yalıtım sağlar Fitness kusurları sıvı kültüründe göstermektedir.
Şimdiye kadar biz araştırdık tüm kurtarılan S suşları fenotipik kurtarma değişen derecelerde göstermiştir. Genetik geçiş yoluyla tespit gibi kurtarılan fenotip tek genetik öğe atfedilebilecek olup ( Şekil 2' de gösterilen kalıtsal örnekler). Bu yöntem genellikle birden fazla genomik loci hedef kimyasal veya UV tabanlı bastırıcı ekranlara kıyasla en önemli avantajlarından biri bu. 16 kolonileri dışında/soy (%10) kurtarılan bir ya da iki kolonileri gözlemlemek için ortaktır bir hafta içinde. Ancak, biz kaybı-in-işlevini hiç bulunamadı neredeyse vahşi türü büyüme oranı Rrp6, nükleer özgü eksozom alt birim gibi belirli mutantlar elf1Δ hücreleri31içinde gözlenen fark ettim. Rrp6 fonksiyonu yalnızca kısmen bastırıcılarının, önemli işlevi aracılığıyla ağır segregasyonun kusuru düzenleyici içinde neden için gösterilen fal1∆, dahil olmak üzere test, diğer mutantları işlevinin aksine tarafından telafi ki muhtemelen 34eklemek. Yfg benzersiz, değiştirilebilir roller hücre büyüme olduğunda eleme yöntemleri bu alternatif bastırıcı aynı sorun tabi olacağına inanıyorum.
Genomik sıralama gerçekleştirmeden önce arka çapraz plaka-okuyucu, genetik arka plan temizlemek ve biyolojik çoğaltır elde etmek için ebeveyn suşları ile tanımlanan phenotypically kurtarılan kolonileri için en iyi yöntemdir. Buna ek olarak, derin bütün genom sıralama tek nükleotid değişiklikleri olan en küçük ilgilendikleri için tarama biyolojik çoğaltır arasında özdeş değil, yüzlerce tanımlar. Örneğin, iki elf1Δ P ve beş farklı S suşları (şekil 3) arasındaki tüm üç kromozomlar arasında Toplam 660 genomik değişikliklerini bulduk. Biz yaygın olarak sıralı biyolojik arasında özdeş mutasyonlar çoğaltır her tür yeni mutasyonlar genomik Kütüphanesi inşaat önce elf1Δ hücreleri kültür sırasında çıkabilecek veya rasgele hatalar olabilir düşündüren gözlemleme Kütüphane inşaat ve sıralamanın sırasında tanıttı. Bu nedenle, biyolojik çoğaltır tutarlı mutasyonlar yalıtma bastırıcılarının bütün-genom sıralama kullanarak başarılı tanımlaması içinde önemli bir yönü var.
Tespit ve beş sıralı S suşları CD bölgelerde iki bastırıcılarının doğruladı. SPBPJ4664.02 mutasyonlar S-A1 ve S-A2 suşları tespit edildi, SPBPJ4664.02 geçerli bir bastırıcı çünkü onlar () ile birbirlerini tamamlayıcı değildir olarak S-A1 ve S-A2 bir bastırıcı aynı gen içermez olası olsa da veri) gösterilmez. Ayrıca rli1 elf1Δile backcrossed S fenotip ile birlikte ayırmak değil S-A3'doğruladı değil. Alternatif olarak, belirli mutasyonlar kodlamayan bölgeler S-A1, S-A2 ve S-A3 bulduk. Bu değiştirilmiş bu kodlamayan genomik bölgeler bizim gelecekteki çalışmalarda ele alınacaktır elf1Δ fenotip hafifletmek mümkündür. Gibi bir genetik mutasyon eşlemek için yıl sürebilir, bir bağlantı yöntemi, geleneksel yöntemlerle karşılaştırıldığında biz monogenic bir öğe S fenotip neden teyit sonra iki ay içinde iki bastırıcılarının tespit. Bütün-genom sıralama teknolojinin hızlı gelişimi ile bu yöntem tutarlı Genetik mutasyonlar öngörülebilir gelecekte tanımlamak için daha verimli olacaktır iyimser.
Özet olarak, bu iletişim kuralı başarıyla herhangi bir gen bastırıcı mutasyonlar sıvı kültüründe yavaş büyüyen bir kusur ile ilgi tanımlamak için adım adım yönergeler sağlar. Bu tahlil sadeliği birden fazla genetik arka plan ilgi küçük uygulamalı eğitim ile büyük ölçekli tarama sağlar. Daha fazla işlem günlük dilutions gerçekleştirmek için bir sıvı işleme robot kullanarak otomatik hale getirmek için oda var. Laboratuvar manipülasyon mikroorganizmaların kaçınılmaz büyüme sıvı kültüründe, uygunluk, doğal olarak seçici bir işlemde gerektirdiğinden önerdiğimiz bu protokol geniş bakteri gibi diğer büyük nüfus model organizmalar için uygulanabilir ve diğer Maya türler.
Açıklamalar
Yazarlar bu yöntem ve hiçbir rakip mali çıkarlarının kullanılan aletleri üreticileri tarafından hiçbir onay bildirimini.
Teşekkürler
Bu eser tarafından Ulusal Genel tıbbi Bilimler Enstitüsü, hibe 1R15GM119105-01 K.Z. için destek verdi Bütün yorumcular anlayışlı yorum için teşekkür ederiz. Biz de James Tucker, Alicia Anderson, Elizabeth siyah ve Glen Marrs tartışma ve bu el yazması üzerine yorumlar için teşekkür ederim.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Adenine, Powder | Acros Organics | 147441000 | Use at 75 mg/L to make liquid and solid rich media (YEA) |
| Bacteriology Petri Dish | Corning, Falcon | C351029 | 100 ×15 mm, use to grow strains to single colonies on solid rich media |
| D-Glucose Anhydrous, Powder | Fisher Chemical | D16-1 | Use at 30 g/L to make liquid and solid rich media (YEA) |
| Difco Agar, Granuated | Becton, Dickinson and Co. | 214530 | Use at 20 g/L to make solid rich media (YEA) |
| DNA extraction buffer | 2% Triton X-100, 1% SDS, 100 mM NaCl, 10 mM Tris-Cl (pH 8.0), 1mM Na2-EDTA | ||
| Focused-ultrasonicator | Covaris Inc. | S220 | Alternatively, use QSonica Q800R sonicator/DNA and chromatin shearing system |
| Gen5 Data Collection and Analysis Software | Biotek, Inc. | GEN5SECURE | Or equivalent, must be compatible with the micro-plate reader, use to export data readings from the micro-plate reader |
| Hydrochloric Acid 1N, Liquid | Fisher Chemical | SA48-4 | Use to adjust pH to 5.5 in liquid and solid rich media |
| Liquid Rich Media (liquid YEA) | 30 g/L D-Glucose, 5 g/L Yeast Extract, 75 mg/L Adenine, pH adjusted to 5.5 with 1 M HCl | ||
| Microplate Reader, Synergy H1 Hybrid Multi-Mode Reader | Biotek, Inc. | BTH1MG | Or equivalent, must read visible light at 600 nm wavelength range |
| Rich Media agar plates (YEA plates) | 30 g/L D-Glucose, 5 g/L Yeast Extract, 75 mg/L Adenine, 20 g/L Agar, pH adjusted to 5.5 with 1 M HCl. | ||
| RNase A/T1 mix | Thermo Fisher Scientific | EN0551 | Use according to manufacturer recommendation |
| Sterile Polystyrene Inoculating Loop | Corning, Inc. | OS101 | Or equivalent, use to transfer colonies from agar plates to 96-well plate |
| Sterile workspace and burners | |||
| Tissue Culture Plate, 96-well Optical Flat Bottom with Low Evaporation Lid | Corning, Falcon | C353072 | Or equivalent, must have optical flat bottom for micro-plate ready |
| TruSeq DNA PCR-Free LT/HT Library Prep Kit | Illumina, Inc. | 20015962 | Use to prepare the whole-genome sequencing library |
| Yeast Extract, Powder | Fisher Chemical | BP1422-500 | Use at 5 g/L to make liquid and solid rich media (YEA) |
Referanslar
- McKay, J. K., Latta, R. G. Adaptive population divergence: Markers, QTL and traits. Trends in Ecology and Evolution. 17 (6), 285-291 (2002).
- Wood, V., Harris, M. A., et al. PomBase: A comprehensive online resource for fission yeast. Nucleic Acids Research. 40 (D1), (2012).
- de Visser, J. A. G. M., Cooper, T. F., Elena, S. F. The causes of epistasis. Proceedings of the Royal Society B: Biological Sciences. 278 (1725), 3617-3624 (2011).
- Sailer, Z. R., Harms, M. J. Detecting high-order epistasis in nonlinear genotype-phenotype maps. Genetics. 205 (3), 107911088 (2017).
- Kuzmin, E., Costanzo, M., Andrews, B., Boone, C. Synthetic genetic arrays: Automation of yeast genetics. Cold Spring Harbor Protocols. 2016 (4), 326-332 (2016).
- Tong, A. H. Y., Boone, C. Synthetic genetic array analysis in Saccharomyces cerevisiae. Methods in Molecular Biology. 313 (1), 171-192 (2006).
- Dixon, S. J., Costanzo, M., Baryshnikova, A., Andrews, B., Boone, C. Systematic Mapping of Genetic Interaction Networks. Annual Review of Genetics. 43 (1), 601-625 (2009).
- Boone, C., Bussey, H., Andrews, B. J. Exploring genetic interactions and networks with yeast. Nature Reviews Genetics. 8 (6), 437-449 (2007).
- Bai, X., Yang, Z., Jiang, H., Lin, S., Zon, L. I. Genetic suppressor screens in haploids. Methods in Cell Biology. , 129-136 (2011).
- Manson, M. D. Allele-specific suppression as a tool to study protein-protein interactions in bacteria. Methods. 20 (1), 18-34 (2000).
- Motter, A. E., Gulbahce, N., Almaas, E., Barabási, A. L. Predicting synthetic rescues in metabolic networks. Molecular Systems Biology. 4, 168 (2008).
- Peterson, R. T., Shaw, S. Y., et al. Chemical suppression of a genetic mutation in a zebrafish model of aortic coarctation. Nature Biotechnology. 22 (5), 595-599 (2004).
- Giorgini, F., Guidetti, P., Nguyen, Q., Bennett, S. C., Muchowski, P. J. A genomic screen in yeast implicates kynurenine 3-monooxygenase as a therapeutic target for Huntington disease. Nature Genetics. 37 (5), 526-531 (2005).
- Forsburg, S. L., Patton, E., et al. The art and design of genetic screens. Nature reviews. Genetics. 2 (9), 659-668 (2001).
- Johnston, D. S. The art and design of genetic screens. Genetics. 3 (March), 176-188 (2002).
- Jorgensen, E. M., Mango, S. E. The art and design of genetic screens: Caenorhabditis elegans. Nature Reviews Genetics. 3 (5), 356-369 (2002).
- Gocke, E., Müller, L. In vivo studies in the mouse to define a threshold for the genotoxicity of EMS and ENU. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 678 (2), 101-107 (2009).
- Suzuki, T., Hayashi, M., et al. A comparison of the genotoxicity of ethylnitrosourea and ethyl methanesulfonate in lacZ transgenic mice (Muta(TM)Mouse). Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 395 (1), 75-82 (1997).
- Uttam, J., Alberico, C., De Stasio, E. ENU Mutagenesis. International C. elegans Meeting. , (1995).
- Putrament, A., Baranowska, H., Ejchart, A., Prazmo, W. Manganese Mutagenesis in Yeast. Methods in Cell Biology. 20, 25-34 (1978).
- Bose, J. L. Chemical and UV mutagenesis. Methods in Molecular Biology. 1373, 111-115 (2016).
- Ikehata, H., Ono, T. The Mechanisms of UV Mutagenesis. Journal of Radiation Research. 52 (2), 115-125 (2011).
- Shrivastav, N., Li, D., Essigmann, J. M. Chemical biology of mutagenesis and DNA repair: cellular responses to DNA alkylation. Carcinogenesis. 31 (1), 59-70 (2010).
- De Stasio, E. A., Dorman, S. Optimization of ENU mutagenesis of Caenorhabditis elegans. Mutation Research - Genetic Toxicology and Environmental Mutagenesis. 495 (1-2), 81-88 (2001).
- Probst, F. J., Justice, M. J. Mouse mutagenesis with the chemical supermutagen ENU. Methods in Enzymology. 477 (C), 297-312 (2010).
- Bähler, J., Wu, J. Q., et al. Heterologous modules for efficient and versatile PCR-based gene targeting in Schizosaccharomyces pombe. Yeast. 14 (10), 943-951 (1998).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows – Wheeler transform. Bioinformatics. 25 (14), 1754-1760 (2009).
- Van der Auwera, G. A., Carneiro, M. O., et al. From fastQ data to high-confidence variant calls: The genome analysis toolkit best practices pipeline. Current Protocols in Bioinformatics. 43, (2013).
- Mckenna, A., Hanna, M., et al. The Genome Analysis Toolkit: A MapReduce framework for analyzing next-generation DNA sequencing data. Genome Research. 20, 1297-1303 (2010).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Marayati, B. F., Drayton, A. L., et al. Loss of Elongation-Like Factor 1 Spontaneously Induces Diverse, RNase H-Related Suppressor Mutations in Schizosaccharomyces pombe. Genetics. 209 (4), 967-981 (2018).
- Harris, M. A., Lock, A., Bähler, J., Oliver, S. G., Wood, V. FYPO: The fission yeast phenotype ontology. Bioinformatics. 29 (13), 1671-1678 (2013).
- Xu, X., Wang, L., Yanagida, M. Whole-Genome Sequencing of Suppressor DNA Mixtures Identifies Pathways That Compensate for Chromosome Segregation Defects in Schizosaccharomyces pombe. G3: Genes|Genomes|Genetics. 8 (3), 1031-1038 (2018).
- Marayati, B. F., Hoskins, V., et al. The fission yeast MTREC and EJC orthologs ensure the maturation of meiotic transcripts during meiosis. RNA. 22 (9), 1349-1359 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır