Method Article
Hidrolik Çatlatmanın Akışlar Üzerindeki Etkisini Mikrobiyal Moleküler İmzalar Kullanarak Değerlendirmek
Bu Makalede
Özet
Burada, hidrolik kırılmanın su ve tortu mikrobiyal topluluklarını analiz ederek yakındaki akarsular üzerindeki etkilerini araştırmak için bir protokol sunuyoruz.
Özet
Genellikle "fracking" olarak adlandırılan hidrolik kırılma (HF), kayaları parçalamak, petrol ve gaz salmak için yüksek basınçlı su, kum ve kimyasalların bir karışımını kullanır. Bu süreç, daha önce ulaşılamaz olan kaynaklara erişim verdiği ve şimdi ABD'deki toplam doğal gazın üçte ikisini ürettiği için ABD enerji endüstrisinde devrim yaptı. Fracking'in ABD ekonomisi üzerinde olumlu bir etkisi olmasına rağmen, çeşitli çalışmalar zararlı çevresel etkilerini vurgulamıştır. Özellikle endişe verici olan, fracking'in tüm su havzasının sağlığı üzerindeki orantısız büyük etkileri nedeniyle özellikle önemli olan baş suyu akışları üzerindeki etkisidir. Bu akarsulardaki bakteriler, akış sağlığının göstergeleri olarak kullanılabilir, çünkü bakterilerin mevcut olması ve rahatsız bir akıştaki bolluklarının, aksi takdirde karşılaştırılabilir ancak bozulmamış bir akıştakilerden farklı olması beklenir. Bu nedenle, bu protokol, akışların fracking'den etkilenip etkilenmediğini belirlemek için bakteri topluluğunu kullanmayı amaçlamaktadır. Bu amaçla, fracking yakınındaki akarsulardan (potansiyel olarak etkilenen) ve yukarı akıştan veya farklı bir fracking aktivitesi havzasında (engellenmemiş) tortu ve su örnekleri toplanmalıdır. Bu örnekler daha sonra mikrobiyal topluluk kompozisyonunu araştırmak için nükleik asit ekstraksiyonuna, kütüphane hazırlığına ve dizileme işlemine tabi tutulur. Korelasyonel analiz ve makine öğrenimi modelleri daha sonra hangi özelliklerin toplumdaki varyasyonu açıklayıcı olduğunu belirlemek ve fracking'in etkisi için tahmine dayalı biyobelirteçlerin tanımlanması için kullanılabilir. Bu yöntemler, fracking'e yakınlığına dayanarak, baş suyu akışları arasındaki mikrobiyal topluluklarda çeşitli farklılıkları ortaya çıkarmakta ve fracking faaliyetlerinin çevresel etkileri hakkında gelecekteki araştırmalar için bir temel görevi görebilebilir.
Giriş
Hidrolik çatlatma (HF) veya "çatlama", fosil yakıtlara olan talep artmaya devam ettikçe giderek yaygınlaşan bir doğal gaz çıkarma yöntemidir. Bu teknik, genellikle sıkışmış gazları serbest bırakmak için metan bakımından zengin şist yataklarına su, kum ve kimyasalların bir karışımını enjekte etmek için yüksek güçlü sondaj ekipmanları kullanmaktan oluşur1.
Bu alışılmadık hasat teknikleri nispeten yeni olduğundan, bu tür uygulamaların yakındaki su yolları üzerindeki etkilerini araştırmak önemlidir. Fracking faaliyetleri, ekipman taşımacılığı ve kuyu ped yapımı için büyük arazi alanlarının temizlenmesini zorunlu kmaktadır. Her kuyu ped 2 için yaklaşık 1,2-1,7hektar arazi temizlenmelidir, sistemin kaçak ve su kalitesini potansiyel olarak etkiler3. Hangi biyositlerin kullanıldığı da dahil olmak üzere fracking sıvısının tam kimyasal bileşimini çevreleyen şeffaflık eksikliği vardır. Ek olarak, fracking atıksu yüksek tuzlu su olma eğilimindedir2. Ayrıca, atık su metaller ve doğal olarak oluşan radyoaktif maddeler içerebilir2. Bu nedenle, insan hatası veya ekipman arızası nedeniyle fracking sıvısının sızma ve dökülme olasılığı söz ile ilgilidir.
Akarsu ekosistemlerinin çevredeki manzaralardaki değişikliklere karşı çok hassas olduğu bilinmektedir4 ve biyoçeşitliliği korumak için önemlidir5 ve tüm su havzası içinde uygun besin döngüsü6. Mikroplar tatlı su akışlarındaki en bol organizmalardır ve bu nedenle besin döngüsü, biyobozunurluk ve birincil üretim için gereklidir. Mikrobiyal topluluk kompozisyonu ve işlevi, perturbanceye duyarlılıkları nedeniyle ekosistem hakkında bilgi edinmek için harika araçlar olarak hizmet eder ve son araştırmalar, fracking aktivitesine yakınlığa dayanan gözlemlenen bakteri derlemelerinde belirgin kaymalar göstermiştir7,8. Örneğin, Beijerinckia, Burkholderiave Methanobacterium fracking yakınındaki akarsularda zenginleştirilmiş olarak tanımlanırken, Pseudonocardia, Nitrospirave Rhodobacter fracking7'yeyakın olmayan akarsularda zenginleştirilmiştir.
16S ribozomal RNA (rRNA) geninin yeni nesil dizilimi, tüm genom dizileme yaklaşımlarından daha hızlı ve daha ucuz olan bakteriyel topluluk bileşimini belirlemek için uygun fiyatlı bir yöntemdir9. Moleküler ekoloji alanında yaygın bir uygulama, 16S rRNA geninin son derece değişken V4 bölgesini, genellikle geniş bir tanımlama9kapsamına sahip cins seviyesine kadar sıralama çözünürlüğü için kullanmaktır , çünkü öngörülemeyen çevresel örnekler için idealdir. Bu teknik, yayınlanan çalışmalarda yaygın olarak uygulanmıştır ve kırma işlemlerinin su ortamları üzerindeki etkisini belirlemek için başarıyla kullanılmıştır7,8. Bununla birlikte, bakterilerin tespit edilen bolluklarını etkileyen 16S rRNA geninin değişen kopya numaralarına sahip olduğunu belirtmek gerekir10. Bunu hesaba katmak için birkaç araç var, ancak etkinliği şüpheli10. Yaygınlığı hızla büyüyen ve bu zayıflıktan yoksun olan bir başka uygulama, tüm RNA'ların sıralı olduğu metatranscriptomic dizilemedir ve araştırmacıların hem aktif bakterileri hem de gen ifadelerini tanımlamalarına izin verir.
Bu nedenle, daha önce yayınlanan 7 , 8,11,12çalışmalarındaki yöntemlerin aksine, bu protokol mikrobiyal topluluk işlevini (metatranscriptomics) araştırmak için örnek toplama, koruma, işleme ve analizi de kapsamaktadır. Burada ayrıntılı olarak açıklanan adımlar, araştırmacıların, eğer varsa, fracking'in antimikrobiyal direnç genleri de dahil olmak üzere akışlarındaki mikroplar tarafından ifade edilen genler ve yollar üzerinde nasıl bir etkisi olduğunu görmelerini sağlar. Ayrıca, numune toplama için sunulan ayrıntı düzeyi geliştirilmiştir. Adımların ve notların birkaçı deneyimli araştırmacılar için açık görünse de, araştırmaya yeni başlayanlar için paha biçilmez olabilir.
Burada, laboratuvarlarımızın birkaç yıllık deneyimine dayanarak fracking'in yakındaki akışlar üzerindeki etkisini araştırmak için bakteriyel genetik veriler üretmek için örnek toplama ve işleme yöntemlerini açıklıyoruz. Bu veriler, fracking durumuna karşılık gelen farklılıkları tanımlamak için aşağı akış uygulamalarında kullanılabilir.
Protokol
1. Nükleik asit ekstraksiyonu için tortu örneklerinin toplanması
- Steril bir 50 mL konik tüpü dere suyuna batırın. İstenmeyen insan kontaminasyonini önlemek için numune toplama sırasında eldiven giyin. Bu adımı kıyıdan veya sudaysa yukarı akışa bakacak şekilde gerçekleştirin.
- Konik tüp suya batırılırken, kapağı çıkarın ve konik boruya 1 ila 3 cm derinlikten yaklaşık 3 mL tortu kepçelemek için kullanın.
- Konik tüpü sudan çıkarın ve tortu örneğini (yaklaşık 1 mL) kaplayan ince bir tabaka hariç tüm suyu dökin.
- 1000 μL pipet ve uygun pipet uçları kullanarak, toplanan örneğe 3 mL DNA/RNA koruyucu (koruyucu spesifikasyonlar için malzeme tablosuna bakın) ekleyin. Pipet uçlarını steril bir pipet uç kutusunda tutun ve sadece kullanmadan hemen önce takın ve kullanımdan sonra atın. Koruyucu ve numunenin iyice karıştırılmasını sağlamak için kapaklı konik tüpü 10 kez ters çevirin.
NOT Adım 1.4 gerekli değildir, ancak RNA'nın daha sonra çökeltilerden çıkarılması şiddetle önerilir. - Numunelerin geri kalanı için numuneleri buza yerleştirin. Toplama işleminden döndükten sonra, numuneler 16S analizi (DNA) için kullanılacaksa -20 °C veya metatranscriptomics analizi (RNA) için kullanılacaksa -70 °C'de bir dondurucuda saklayın.
2. Nükleik asit ekstraksiyonu için filtre toplama
- Steril bir 1 L şişenin kapağını çıkarın. Yukarı veya kıyıdan bakarken, şişeyi dere suyuyla doldurun ve sonra atın. Şişeyi koşullamak için bu işlemi iki kez daha tekrarlayın. Tüm şişeyi dördüncü kez doldurun ve kaplayın.
NOT: 1 L şişeyi yeniden kullanıyorsanız, 2 dakika boyunca% 10 çamaşır suyu ile durulanarak sterilize edilebilir, ardından deiyonize su ile üç kez durulanabilir ve ardından% 70 etanol ile bir kez durulanabilir ve son olarak ayarlarla otomatik olarak kapatılabilir: 121.1 ° C'de 30 dk maruz kalma süresi ve 15 dakika kurutma süresi. Otomatik kapatma sırasında, şişenin işlem sırasında sıkıştırılmasını önlemek için şişenin üzerindeki kapak çok gevşek olmalıdır. - Sabit bir yüzeye çıktıktan sonra steril bir Luer kilit şırınnası kullanın ve tam bir hacim çizin. Daha sonra şırınnayı 0,22 μm gözenek boyutuna sahip steril ve DNA/RNA içermeyen 1,7 cm çapında poliethersülfon filtresine bağlayın ve pistonu tamamen aşağı bastırarak tüm hacmi filtreden geçirin. Şişede toplanan toplam hacim (1 L) filtreden itilene kadar bu işlemi tekrarlayın.
NOT: Şırınnanın hacmi değişken olabilir, eğer filtreden itilen toplam su miktarı izlenirse. Ancak genel olarak 60 mL tercih edilir. 1 L ideal olsa da, anekdotsal olarak, en az 200 mL'lik bir hacim muhtemelen DNA ve RNA'nın çıkarılması için yeterli biyokütle (mL başına ~ 20.000 hücre varsayılarak) toplayacaktır. - Şırındın içine kabaca 20 mL değerinde hava çekerek ve filtreden geçirerek filtreden fazla suyu çıkarın.
NOT: Bu, adım 2.4 gerçekleştirilirse koruyucunun kaybını önlemeye yardımcı olacaktır. - P1000 mikropipette kullanarak, filtreyi yatay olarak tutarken filtrenin daha büyük açıklığı (şırıngaya bağlı olduğu yer) içinden boşaltarak 2 mL DNA/RNA koruyucu ekleyin. Koruyucunun filtreye girdiğinden emin olmak için pipet bastırıldığında pipet ucu filtrenin namlusunun içinde olmalıdır. Her kullanımdan sonra ucu değiştirin.
NOT: Tortu toplamada olduğu gibi, bu adım gerekli değildir, ancak daha sonra özellikle RNA için nükleik asit veriminin artması şiddetle tavsiye edilir. - Bir kare parafin filmini soyun ve mühürlemek için filtrenin her bir açıklığının / ucunun etrafına sıkıca sarın. Parafin filmi sarılmış filtreyi steril bir numune torbasına yerleştirin ve toplama sırasında tüm torbayı buza yerleştirin.
NOT: Filtreyi sarmak için kullanılan tarafın steril olduğundan, yani daha önce ortama maruz kalmadığından emin olun. - Örneklemeden döndükten sonra filtreleri 16S için -20 °C veya meta-transkriptomik için -70 °C'de saklayın.
3. Nükleik asit ekstraksiyonu ve nicelleştirme
- Numune transfere başlamadan önce çalışma alanını %10 Çamaşır Suyu ve %70 Etanol ile temizleyin.
- Tortu için (adım 1.5'ten itibaren), genellikle ~0.25 g örnek kullanın. Alev, metal bir aleti % 70 etanol kabına batırarak ve numuneler arasında etanol yakarak sterilize eder.
- Filtreler için (adım 2.6'dan itibaren), filtre kağıdını ekstraksiyon için steril bir tüpe taşıyın. Bunu yapmak için aşağıdaki adımları izleyin.
- Alüminyum folyoyu katlayarak, kıvrımın iç kısmını dış ortama maruz kalmaması için steril, DNA ve RNA serbest yüzeyi oluşturun ve katlanmış parçayı ayarlarla otomatikleştirin: 121,1 °C ve 5 dakika kuruma süresi.
- %70 etanol ve açık alev ile mengene kavramayı sterilize edin. Ardından steril yüzeydeki filtre muhafazasını kırmak ve çekirdeği kasadan çıkarmak için mengene tutuşunu kullanın.
- Filtre kağıdını üst ve altta ve ardından dikiş boyunca dilimleyarak çekirdekten kesmek için steril bir neşter kullanın. Filtre kağıdını steril cımbız kullanarak katlayın ve ardından neşteri kullanarak filtreyi küçük parçalara bölün.
- Filtre parçalarını ekstraksiyon için bir mikrosantrifüj tüpüne yerleştirin. Filtre kağıdının sterilize edilmemiş veya nükleik asit bulunabilecek yüzeylerle temas etmediğinden emin olun, çünkü bu numunenin istenmeyen kirlenmesine yol açacaktır.
- DNA yalıtımını daha önce açıklandığı gibi13 veya piyasada bulunan sütun tabanlı bir kit kullanarak gerçekleştirin (bkz. Malzeme Tablosu). Listelenen ticari kit için adımlar aşağıda kısaca açıklanmıştır.
- Numune içindeki hücreleri bir boncuk tüpüne aktararak ve en az 5 dakika boyunca yüksek hızda bir hücre bozucuya maruz kalarak lyse. Santrifüj ve steril bir mikrosantrifüj tüpüne süpernatant aktarın.
- Üst noktaya (1:1 birim) lizis arabelleği ekleyin ve sağlanan filtreye (sarı) aktarın. Filtreyi santrifüjleyin.
- Filtreyi yeni bir steril mikrosantrifüj tüpüne aktarın. Hazırlık tamponu (400 μL), santrifüj ekleyin ve akışı atın.
- Yıkama tamponu (700 μL), santrifüj ekleyin ve akışı atın. Ardından yıkama tamponu (400 μL), santrifüj ekleyin ve akışı tekrar atın.
- Filtreyi yeni bir steril mikrosantrifüj tüpüne aktarın. 50 μL DNase/RNase içermeyen su ile elute edin ve santrifüjlemeden önce oda sıcaklığında 5 dakika bekletin.
- Bu kübasyon döneminde, III-HRC filtresini bir toplama tüpüne yerleştirerek ve içine HRC hazırlık çözeltisini (600 μL) ekleyerek hazırlayın, ardından 8.000 x g'da3 dakikalık bir santrifüjleme adımı .
- Hazırlanan filtreyi steril bir mikrosantrifüj tüpüne taşıyın. Elde edilen DNA'yı adım 3.4.5'ten bu filtreye aktarın ve 3 dakika boyunca 16.000 x g'da santrifüjleyin. Akışta çıkarılan DNA var.
- DNA özlerini hem çökeltiler hem de filtreler için -20 °C'de saklayın.
NOT: DNA özleri- 20 ° C'de yaklaşık 8 yıl boyunca saklanabilir, sabit sıcaklık, sınırlı ışığa maruz kalma ve zararlı kirleticiler olmadığı varsayılır14. - Üreticinin protokolüne göre RNA yalıtımı gerçekleştirin. RNA özlerini -80 °C'de saklayın.
- Numune içindeki hücreleri bir boncuk tüpüne aktararak ve en az beş dakika boyunca yüksek hızda bir hücre bozucuya maruz kalarak Lyse. Santrifüj ve steril bir mikrosantrifüj tüpüne süpernatant aktarın.
- Üst noktaya (1:1 birim) lizis arabelleği ekleyin ve sağlanan sütuna (sarı) aktarın. Sütunu santrifüjle.
- Akışa% 95-100 etanol eşit hacim ekleyin ve beş kez yukarı ve aşağı pipetleme yaparak karıştırın.
- IICG Sütununu (yeşil) steril bir mikrosantrifüj tüpüne yerleştirin. Karışık çözeltiyi sütuna ve santrifüje aktarın.
- Yıkama tamponu (400 μL), santrifüj ekleyin ve akışı atın.
- Kolona 5 μL DNase I ve 75 μL DNA sindirim tamponu ekleyin ve oda sıcaklığında 15 dakika kuluçkaya yatırın.
- Hazırlık tamponu (400 μL), santrifüj ekleyin ve akışı atın.
- Yıkama tamponu (700 μL), santrifüj ekleyin ve akışı atın. Ardından yıkama tamponu (400 μL), santrifüj ekleyin ve akışı tekrar atın.
- Sütunu yeni bir steril mikrosantrifüj tüpüne aktarın. 50 μL DNase/RNase içermeyen su ile elute edin ve santrifüjlemeden önce 5 dakika bekletin.
- Bu kuluçka süresi boyunca, III-HRC filtresini bir toplama tüpüne yerleştirerek ve içine HRC hazırlık çözeltisini (600 μL) ekleyerek hazırlayın, ardından 8.000 x g'da3 dakikalık bir santrifüjleme adımı .
- Hazırlanan filtreyi steril bir mikrosantrifüj tüpüne taşıyın. Eluted RNA'yı adım 3.6.9'dan bu filtreye aktarın ve 3 dakika boyunca 16.000 x g'da santrifüjleyin. Akış ayıklanan RNA'yı içerir.
NOT: RNAözleri, 15'ibozmaya başlamadan önce yalnızca bir yıl saklanabilir. Hem DNA hem de RNA özleri tekrarlanan donma-çözülme ile bozulur. Bazı protokoller aynı örnekten hem DNA hem de RNA çıkarılmasına izin verir16,17.
- Çıkarılan DNA ve RNA örneklerini bir florometre veya spektrofotometre kullanarak ölçün. Florometre DNA konsantrasyon değerleri gibi Tablo 1'e bakın. Örnek bir spektrofotometre nicelleştirmeprotokolü için, bkz. Malzeme Tablosunda listelenen kit ile tortu DNA konsantrasyon değerleri genellikle 1 ila 40 ng/μL arasında değişirken, filtre DNA konsantrasyon değerleri 0,5 ila 10 ng/μL arasında değişir. tipik olarak 0,5 ila 5 ng/μL arasında değişen.
4. 16S rRNA kütüphanesi oluşturma
- Çalışma alanını %10 Çamaşır Suyu ve %70 Etanol ile temizleyin. Çalışma alanı, laminer akış koşulları (laminar akış davlumbaz) üretebilen kapalı bir alan olmalıdır.
- DNA özlerini kullanın (adım 3.5'ten itibaren) ve 16S rRNA amplicon dizilimini standart bir PCR protokolü ile hazırlayın, örneğin Dünya Mikrobiyomunun web sitesinde açıklanan ve laminer akış koşulları altında 16S rRNA19'un V4 hipervariable bölgesini yükselten.
- Daha önce açıklandığı gibi% 2 agarose jel hazırlayın ve katılaştırmasına izin verin17. 7 μL PCR ürünü ve 13 μL DNase içermeyen suyu karıştırın. 1x'lik son konsantrasyona jel yükleme boyası ekleyin. Agarose katılaştıktan sonra, bu PCR ürünleri karışımını% 2 agarose jel üzerine yükleyin.
NOT: Alternatif olarak, bu jeller daha hızlı koştukça ve önceden yapılmış olarak geldiği için bunun yerine bir ön döküm jel kullanılabilir. - Dünya Mikrobiyomunun protokolünü kullanarak 16S rRNA V4 amplicons için başarılı amplifikasyon olarak 386 bant boyutunu kontrol etmek için jeli 60-90 dakika boyunca 90 V'ta çalıştırın.
5. DNA 16S rRNA kütüphanesi saflaştırma
- Parlak bantlar veren numuneler için 10 μL, uygun büyüklükte steril mikrosantrifüj tüpünde soluk bantlar veren numuneler için 13 μL PCR ürünleri havuzu.
- Bir florometre veya spektrofotometre kullanarak elde eden havuzun konsantrasyonunu kontrol edin ve daha önce olduğu gibi% 2 agarose jel hazırlayın. İdeal olarak, havuz en az 10 ng / μL konsantrasyona sahip olmalıdır ve çoğu numune yaklaşık 25 ng / μL konsantrasyona sahip olmalıdır.
- Konsantrasyon ve hacim izin veriyor, % 2 agarose jel kuyusunda yaklaşık 150-200 ng yükleyin.
- Jeli 90 voltta 60-90 dakika çalıştırın.
- %2'lik bir agarose jel çalıştırarak havuzalanmış kütüphaneyi arındırın.
- Jelden 386 bp DNA bandını kesin ve daha önce açıklandığı gibi ticari olarak mevcut bir kit kullanarak havuza alınan kütüphaneyi arındırın20. Saflaştırılmış DNA'ya 30 μL 10 mM Tris-Cl (pH 8.5) sürün. Jelin kesilmesi PCR ampliconlarını hem deneyciye hem de çevreye yayacağından, gelecekteki kirlenmeyi önlemek için DNA veya RNA ekstraksiyonundan farklı bir alanda bu adımı gerçekleştirin.
- Florometre veya spektrofotometre kullanarak saflaştırılmış havuzun konsantrasyonunu kontrol edin. Arınma iyi giderse, konsantrasyonu teşvik edilmemiş havuzun en az yarısı olmalıdır. Genellikle, son konsantrasyon 5 ila 20 ng / μL arasında değişmelidir.
- Yeni nesil sıralama için saflaştırılmış kitaplıkları gönderin. Nakliye kabına kuru buz dahil larak nakliye sırasında soğuk tutulmalarını sağlayın.
6. RNA kütüphanesi oluşturma ve temizleme
- RNA kütüphaneleri oluşturmak için çeşitli ticari kitler kullanılabilir. Hangisi kullanılırsa kullanılsın, steril bir laminer akış ortamında çalışırken yazıldığı gibi üreticinin protokolünü izleyin. Malzeme Tablosunda kit protokolünün çok özetlenmiş bir versiyonu21'inaltında sunulmuştur.
- İlk iplikçik cDNA sentez ana karışımını (8 μL çekirdeksiz su ve 2 μL first strand sentez enyzme mix) yapın ve örneğe ekleyin. Numuneyi protokolde belirtilen koşullarla termostaste yerleştirin.
- İkinci iplikçik cDNA sentez ana karışımını (8 μL İkinci Iplikçik Sentez Reaksiyon Tamponu, 4 μL İkinci Iplikçik Sentez Enzim Karışımı ve 48 μL çekirdeksiz su) buz üzerinde yapın ve örneğe ekleyin. Bir saat boyunca 16 °C'ye ayarlanmış bir termosikleyiciye yerleştirin.
- Sağlanan boncukları (144 μL) ekleyerek ve iki adet %80 etanol yıkama (200 μL) gerçekleştirerek reaksiyonun saflaştırılması.
- Sağlanan TE tamponu (53 μL) ile elute edin ve süpernatant 50 μL'yi temiz bir PCR tüpüne aktarın. PCR tüpünü buza yerleştirin.
- Buz üzerinde son hazırlık ana karışımını (7 μL Uç Hazırlık Reaksiyon Tamponu ve 3 μL Uç Hazırlık Enzim Karışımı) yapın ve PCR tüpüne ekleyin. PCR tüpünü protokolde belirtilen koşullara sahip bir termostale yerleştirin.
- Seyreltilmiş Adaptör (2,5 μL), Ligasyon Ana Karışımı (30 μL) ve Ligasyon Arttırıcı (1 μL) çözeltilerini buz üzerinde karıştırın. Karışık çözeltileri numuneye ekleyin ve 20 °C'de 15 dakika boyunca bir termoskope yerleştirin.
- Sağlanan boncukları (87 μL) ekleyerek ve daha önce olduğu gibi etanol yıkama (200 μL) ve elüasyon yaparak reaksiyondan arındırın, ancak sadece 17 μL TE ekleyin.
- Endeksleri (10 μL) ve Q5 Master Mix (25 μL) çözeltisini ekleyin ve protokolde açıklanan koşullarla bir termosiklete yerleştirin.
- Sağlanan boncukları (45 μL) ekleyerek ve ilave iki etanol yıkama (200 μL) ve 23 μL TE ile elüt yaparak reaksiyonun arındırın. 20 μL'yi temiz bir PCR tüpüne aktarın.
- Biyoanalyzer, florometre veya spektrofotometre kullanarak kütüphanelerde tespit edilebilir RNA konsantrasyonları olup olmadığını kontrol edin.
- Metatranscriptomic kitaplıkları kabaca eşitlikçi bir oranda bire birler.
- 250 ile 400 bp arasındaki tüketim parçaları dışında, 16S kitaplık saflaştırması için aynı protokolü izleyen kitaplığı arındırın. 16S kütüphanesinin güçlendirilmiş bölgeyi temsil eden ayrı bir bandı varken, buradaki sonuç bir lekedir.
- Arınmış kütüphanenin konsantrasyonlarını daha önce olduğu gibi kontrol edin.
- Arıtılmış kütüphaneyi kuru buzla bir sıralama tesisine gönder.
NOT: Alternatif olarak, RNA özleri kütüphane hazırlama ve sıralama için bir üniversiteye veya özel şirkete gönderilebilir.
7. Mikrobiyal topluluk analizi
- Sıralama tamamlandıktan sonra örnek verilere erişin. Kullanılabilir bir bilgisayara indirin.
NOT: İdeal olarak, cihazda en az 16 gigabayt RAM olmalıdır. Bilgi işlem gereksinimlerinin (Qiime2 için) tartışılması için https://forum.qiime2.org/t/recommended-specifications-to-run-qiime2/9808 bakın. - 16S rRNA verilerini çözümlemek için mothur, QIIME2 ve R gibi yazılımları kullanın. Örnek bir QIIME2 16S analiz öğreticisi için https://docs.qiime2.org/2020.11/tutorials/moving-pictures/ buraya bakın.
- Metatranscriptomics (RNA) verileri için, örneklerde hangi genlerin ve yolların bulunduğunu belirlemek için HUMAnN2 ve ATLAS kullanın.
NOT: Ek Bilgi dosyasında çeşitlilik ve rastgele orman analizi ile sonuçlanan bir örnek metatranscriptomics ardışık düzeni sunulmuştur. Tüm komutlar komut satırı üzerinden çalıştırılır, örneğin Mac için Terminal kullanıcıları.
Sonuçlar
DNA ve RNA ekstraksiyonlarının başarısı çeşitli ekipman ve protokoller kullanılarak değerlendirilebilir. Genel olarak, herhangi birinin tespit edilebilir konsantrasyonu, ekstraksiyonun başarılı olduğu sonucuna varmak için yeterli kabul edilir. Tablo 1 incelendiğinde, biri hariç tüm ekstraksiyonlar başarılı olarak adlandırılacaktır. Bu adımdaki başarısızlık genellikle düşük başlangıç biyokütlesi, zayıf numune koruması veya ekstraksiyon sırasında insan hatasından kaynaklanır. Filtreler söz konusu olduğunda, konsantrasyon algılamanın altında olsa bile ekstraksiyon başarılı olmuş olabilir. Bu özler PCR için bantlar (16S yapıyorsanız) veya kütüphane hazırlığından sonra tespit edilebilir bir konsantrasyon (metatranscriptomics) vermezse, muhtemelen gerçekten başarısız oldular.
16S protokolüne uyulmuşsa, Şekil 1'deki4 ve 6 kuyularında görüldüğü gibi PCR amplifikasyonu izleyen parlak bantlar başarıyı gösterirken, üst sıradaki diğer kuyularda görüldüğü gibi bant eksikliği başarısızlığı gösterir. Ayrıca, jel şeridinde negatif PCR kontrolü içeren parlak bir bant da bir arızaya işaret edecektir, çünkü negatif kontrolleri etkileyen kontaminasyonun numuneleri etkilemediğini varsaymak riskli olacaktır.
Hem 16S hem de metatranscriptomics için, sıralamanın başarısı elde edilen sıra sayısına bakılarak değerlendirilebilir (Şekil 2). 16S örnekleri en az 1.000 diziye sahip olmalı ve en az 5.000 ideal olmalıdır (Şekil 2A). Aynı şekilde, metatranscriptomics örnekleri en az 500.000 diziye sahip olmalı ve en az 2.000.000 ideal olmalıdır (Şekil 2B). Bu minimumlardan daha az diziye sahip numuneler, bakteri topluluklarını doğru bir şekilde temsil etmeyebileceğinden analizler için kullanılmamalıdır. Bununla birlikte, minimum ve ideal arasında kalan örnekler hala kullanılabilir, ancak birçok örnek bu aralıkta düşerse sonuçlar daha dikkatli yorumlanmalıdır.
Sonraki aşağı akış analizinin başarısı, yalnızca beklenen çıktı dosyalarının elde edilip edilmediğine bağlı olarak belirlenebilir. Her halükarda, QIIME2 ve R (Şekil 3) gibi programlar, bakteri toplulukları arasındaki potansiyel önemli farklılıkların fracking'e dayalı olarak değerlendirilmesine izin vermelidir. Şekil 3 verileri, 16S ve metatranscriptomics analizi için on üç farklı akışta yirmi bir farklı bölgeden tortu örnekleri toplanarak elde edildi. Bu yirmi bir siteden on ikisi fracking aktivitesinin aşağı akışıydı ve HF + olarak sınıflandırıldı ve dokuzu ya fracking aktivitesinin yukarı akışında ya da fracking'in gerçekleşmediği bir su havzasındaydı; bu akışlar HF- olarak sınıflandırıldı. Fracking aktivitesinin varlığının yanı sıra, akarsular başka türlü karşılaştırılabilirdi.
Bu farklılıklar, fracking durumuna göre tutarlı kompozisyon kaymaları şeklinde olabilir. Bu durumda, HF+ ve HF- örneklerinin Şekil 3A ve Şekil 3B'de olduğu gibi bir PCoA grafiğinde birbirinden ayrı kümelenmesi beklenir. Bu belirgin kaymaların sadece ordinatasyon yönteminin bir eseri olmadığını doğrulamak için daha fazla istatistiksel analize ihtiyaç vardır. Örneğin, Şekil 3A ve Şekil 3B'nin dayandığı mesafe matrisi üzerinde yapılan permanova 22 testi, fracking durumuna göre önemli kümelenmeyi ortaya çıkardı, yani arsada gözlenen ayrım, bir ordinatlama eseri yerine örneklerin bakteri toplulukları arasındaki farklılıklarla tutarlıdır. Önemli bir PERMANOVA veya ANOSIM sonucu, HF+ ve HF- numuneleri arasındaki tutarlı farklılıkların güçlü bir göstergesidir, bu da HF+ örneklerinin kırılmadan etkilendiğini gösterirken, yüksek p değeri numunelerin etkilenmediğini gösterir. Metatranscriptomic veriler de aynı yöntemler kullanılarak görselleştirilebilir ve değerlendirilebilir.
Diferansiyel özelliklerin (mikroplar veya işlevler) incelenmesi, örneklerin de etkilendiğine dair kanıtlar ortaya çıkabilir. Diferansiyel özellikleri belirlemenin bir yöntemi rastgele bir orman modeli oluşturmaktır. Rastgele orman modeli, örneklerin fracking durumunun ne kadar iyi sınıflandırılabileceğini görmek için kullanılabilir. Model şans eseri beklenenden daha iyi performans gösterirse, bu, fracking durumuna bağlı farklılıkların ek kanıtı olacaktır. Ayrıca, en önemli tahminciler, örnekleri doğru bir şekilde ayırt etmek için hangi özelliklerin en önemli olduğunu ortaya çıkarır (Şekil 3C). Bu özellikler de o zaman fracking durumuna göre sürekli olarak farklı değerlere sahip olurdu. Bu diferansiyel özellikler belirlendikten sonra, literatür daha önce fracking ile ilişkili olup olmadıklarını görmek için gözden geçirilebilir. Bununla birlikte, çoğu sadece 16S rRNA kompozisyon verilerini kullandığı için diferansiyel fonksiyonları belirleyen çalışmalar bulmak zor olabilir. Bu nedenle, diferansiyel fonksiyonların etkilerini değerlendirmek için, olası bir yöntem, daha önce fracking sıvısında yaygın olarak kullanılan biyositlere karşı potansiyel dirençle ilişkili olup olmadıklarını veya yüksek tuzlu koşulların tolere edilmesine yardımcı olup olmadıklarını görmek olacaktır. Ayrıca, bir ilgi taksonunun işlevsel profilini incelemek fracking'in etkisinin kanıtlarını ortaya çıkarabilir (Şekil 3D). Örneğin, bir takson rastgele orman modeli tarafından diferansiyel olarak tanımlanırsa, HF+ örneklerindeki antimikrobiyal direnç profili HF örneklerindeki profiliyle karşılaştırılabilir ve büyük ölçüde farklılık gösterirse, bu, biyosit içeren fracking sıvısının akışa girdiğini gösterebilir.
| Örnek Kimliği | Konsantrasyon (ng/μL) |
| 1 | 1.5 |
| 2 | 1.55 |
| 3 | 0.745 |
| 4 | 0.805 |
| 5 | 7.82 |
| 6 | 0.053 |
| 7 | 0.248 |
| 8 | 0.945 |
| 9 | 1.82 |
| 10 | 0.804 |
| 11 | 0.551 |
| 12 | 1.69 |
| 13 | 4.08 |
| 14 | Below_Detection |
| 15 | 7.87 |
| 16 | 0.346 |
| 17 | 2.64 |
| 18 | 1.15 |
| 19 | 0.951 |
Tablo 1: Florometre 1x DS DNA yüksek hassasiyetli teste dayalı örnek DNA konsantrasyonları. 14 hariç tüm bu örneklerin ekstraksiyonları, tespit edilebilir miktarda DNA'ya sahip olması nedeniyle başarılı olarak kabul edilecektir.
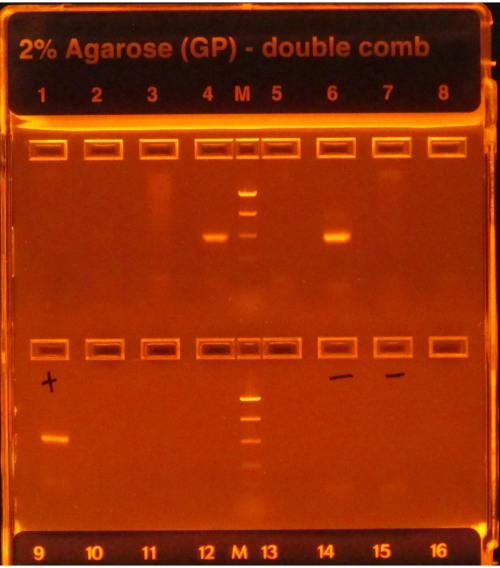
Şekil 1: PCR ürünleri ile örnek e-jel. Jel önceden boyanmış ve UV ışığı altında görselleştirilmiş ve üzerinde herhangi bir DNA bulunmasına neden olmuş. PCR, ilk sıradaki 4 ve 6 numaralı kuyulardaki numuneler için çalıştı, çünkü her ikisi de beklenen boyutta tek bir parlak banda sahipti (merdivene göre). Diğer altı kuyudaki numuneler için PCR başarısız oldu, çünkü herhangi bir bant üretmediler. Pozitif kontrol (birinci kuyu, ikinci sıra) PCR'nin düzgün yapıldığını gösteren parlak bir banta sahipti ve negatif kontroller (kuyular 6 ve 7, ikinci sıra) herhangi bir bant yoktu, bu da örneklerin kontamine olmadığını gösteriyordu. Negatif bir bant numuneler kadar parlak olsaydı, PCR bir başarısızlık olarak kabul edilirdi, çünkü numunelerin sadece kontaminasyonun sonucu olmayan ampliconlara sahip olduğunu varsaymak riskli olurdu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
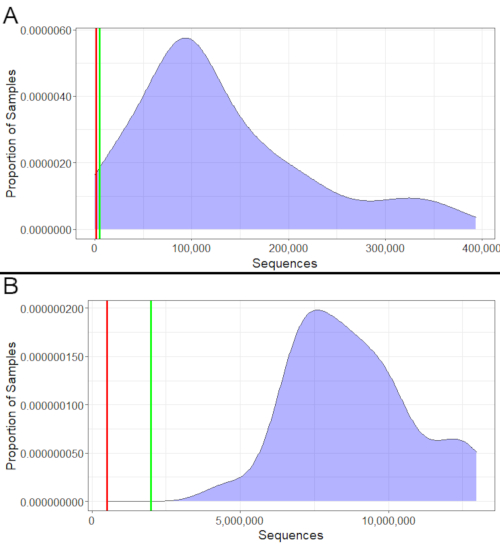
Şekil 2: Örnek sıra sayıları. (A) 16S örnek sıra sayıları. Bu 16S örneklerin neredeyse hepsi 1.000'den fazla diziye sahipti. 1.000'den az diziye sahip olan çok az sayıdaki dizi, bakteri topluluklarını doğru bir şekilde temsil etmek için yetersiz dizilere sahip oldukları için aşağı akış analizlerinin dışında tutulmalıdır. Birkaç dizi 1.000 ila 5.000 diziye sahipti; ideal olmasa da, çıplak minimumu aştıkları için hala kullanılabilirler ve örneklerin çoğu ideal minimum 5.000'i de aşıyor. (B) Metatranscriptomics örnek sayılır. Tüm örnekler hem minimum (500.000) hem de ideal minimum (2.000.000) dizi sayısını aştı. Bu nedenle, sıralama hepsi için başarılı oldu ve hepsi aşağı akış analizinde kullanılabilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
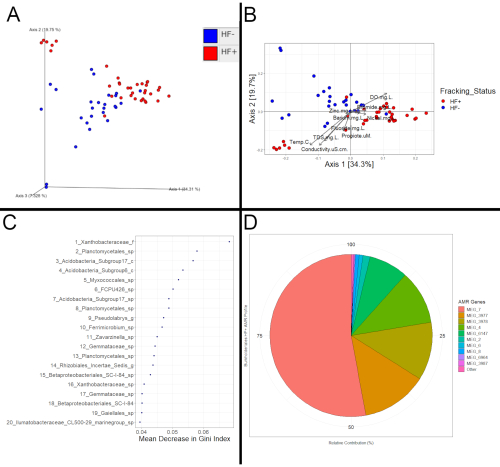
Şekil 3: Örnek analiz. (A) QIIME2 aracılığıyla oluşturulan ve görselleştirilen Ağırlıklı Unifrac mesafe matrisi ile hesaplanan koordinatlara dayalı PCoA çizimi. (B) QIIME2'den dışa aktarılan Ağırlıklı Unifrac mesafe matrisi ile hesaplanan koordinatlara dayalı PCoA çizimi. Koordinatlar Phyloseq kullanılarak görselleştirildi ve R. Meta veri vektörlerindeki ggplot2 paketleri Vegan paketi kullanılarak arsaya takıldı. Her nokta, daha benzer topluluk kompozisyonlarını gösteren daha yakın noktalar ile bir örneğin bakteri topluluğunu temsil eder. Bu 16S tortu örnekleri için fracking durumuna göre kümelenme gözlendi (PERMANOVA, p=0.001). Ayrıca, vektörler HF+ örneklerinin, HF örneklerine kıyasla farklı bakteri topluluk kompozisyonuna karşılık gelen daha yüksek Baryum, Bromür, Nikel ve Çinko seviyelerine sahip olma eğiliminde olduğunu ortaya koymaktadır. (C) Numuneler arasında fracking durumunu tahmin etmek için bakteriyel bollukların nerede kullanılabileceğini test eden rastgele bir orman modeli için en iyi tahmincilerin grafiği. Rasgele orman modeli, randomForest paketi kullanılarak R aracılığıyla oluşturuldu. İlk 20 tahminci, numuneleri ayırmak için kullanıldığında Gini Endeksi'nde Ortalama Düşüş şeklinde safsızlıktaki (HF+ ve HF- numune sayısının birlikte gruplandırıldığı ölçü) düşüşlerin yanı sıra gösterilmiştir. (D) Metatranscriptomic verilere dayalı Burkholderiales profilinin antimikrobiyal direnç profilini gösteren pasta grafik. Diziler ilk olarak Kraken2 ile hangi taksona ait olduklarını belirlemek için açıklamalı olarak belirlendi. BLAST daha sonra hangi antimikrobiyal direnç genlerinin ("MEG_#" şeklinde) aktif olarak ifade edildiğini belirlemek için bu açıklamalı diziler ve MEGARes 2.0 veritabanı ile kullanıldı. Burkholderiales üyeleri tarafından ifade edilen antimikrobiyal direnç genleri daha sonra bu takson arasında en yaygın olanları görmek için çıkarıldı. Metatranscriptomics, daha maliyetli ve zaman alıcı olsa da, 16S verileriyle yapılamayan bu gibi işlevsel analizlere izin verir. Özellikle, kraken2 bu örnek analiz için kullanıldı, YERINE HUMAnN2. Kraken2, HUMAnN2'den daha hızlıdır; bununla birlikte, humann2 gibi kompozisyon, katkı ve işlevler (genler) ve yollar yerine yalnızca kompozisyon bilgilerini verir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tamamlayıcı Dosya: Örnek bir metatranscriptomics ardışık düzeni. Bu dosyayı indirmek için lütfen tıklayınız.
Tartışmalar
Bu makalede açıklanan yöntemler, grubumuz tarafından 2014 ve 20187,8,10 yılları arasında yayınlanan çeşitli çalışmalar boyunca geliştirilmiş ve rafine edilmiş ve yakında yayına sunulacak üç yıllık bir projede fracking'in su toplulukları üzerindeki etkilerini araştırmak için işbirliğine dayalı bir projede başarıyla istihdam edilmiştir. Bu yöntemler projenin geri kalanı boyunca kullanılmaya devam edecektir. Ayrıca, fracking'in akışlar ve ekosistemler üzerindeki etkisini araştıran diğer güncel literatür, örnek toplama, işleme ve analiz 7 , 8,10,11için benzer yöntemleri açıklar. Bununla birlikte, bu makalelerin hiçbiri metatranscriptomik analiz kullanmadı ve bu makaleyi, fracking'in yakındaki akışlar üzerindeki etkisini ortaya çıkarmak için bu analizlerin nasıl kullanılabileceğini açıklayan ilk makale haline getirdi. Ayrıca, numune toplama için burada sunulan yöntemler ve kontaminasyonu önlemek için atılan adımlar daha ayrıntılıdır.
Protokolümüzün en önemli adımlarından biri ilk numune toplama ve korumadır. Toplama sırasında aseptik veya steril bir ortamı korumak zor olabileceğinden, alan örneklemesi ve toplanması belirli zorluklarla birlikte gelir. Bu adım sırasında, numunelerin kirlenmesini önlemek çok önemlidir. Bunu yapmak için eldiven giyilmeli ve sadece steril kapların ve aletlerin numunelerle temas etmesine izin verilmelidir. Nükleik asit bozulmasını azaltmak için numuneler de toplamadan hemen sonra buza yerleştirilmelidir. Toplama üzerine ticari bir nükleik asit koruyucu eklemek de nükleik asit verimini artırabilir ve numunelerin toplamadan sonra daha uzun süre saklanmasını sağlayabilir. Nükleik asit ekstraksiyonu her gerçekleştirilirse, uygun miktarda numunenin kullanılması önemlidir, ekstraksiyon için kullanılan spin filtrelerini çok fazla tıkayabilir (bunlardan faydalanan protokoller için) ancak çok az verim düşük verime neden olabilir. Hangi kit kullanılırsa kullanılanın talimatlarına uyduklarına emin olun.
Alan toplama işlemine benzer şekilde, özellikle düşük nükleik asit verim örnekleri (çok miktarda çakıl veya kaya içeren numuneler) veya su örnekleri gibi düşük nükleik asit verim örnekleriyle çalışırken, nükleik asit ekstraksiyonu ve numune hazırlama sırasında kontaminasyonu önlemek veya en aza indirmek de önemlidir. Bu nedenle, numune toplamada olduğu gibi, kontaminasyonu azaltmak için tüm bu adımlar sırasında eldiven giyilmelidir. Ek olarak, laboratuvar prosedürleri sırasında kullanılan tüm çalışma yüzeyleri önceden% 10 çamaşır suyu çözeltisi ve ardından% 70 etanol çözeltisi ile silinerek sterilize edilmelidir. Pipetleme adımları (3-6) için, pipet nedeniyle kirlenmeyi önlemek için filtre uçları kullanılmalıdır, steril olmayan bir yüzeye her dokunduklarında uçlar değiştirilmelidir. Pipetler de dahil olmak üzere laboratuvar çalışmaları için kullanılan tüm aletler çamaşır suyu ve etanol çözeltileri ile önce ve sonra silinmelidir. Kontaminasyonu değerlendirmek için, her nükleik asit ekstraksiyonu ve PCR reaksiyonları sırasında ekstraksiyon boşlukları ve negatifleri (steril sıvı) dahil edilmelidir. Ekstraksiyonlardan sonra yapılan niceleme negatiflerde sapkabilme miktarda DNA/RNA ortaya çıkarsa, yeterli örnek kalmışsa ekstraksiyonlar tekrarlanabilir. PCR için negatif örnekler amplifikasyon gösteriyorsa, kaynağı belirlemek için sorun giderme yapılmalı ve ardından örnekler yeniden çalıştırılmalıdır. Düşük kontaminasyon seviyelerini hesaba katmak için, ekstraksiyon boşluklarının ve PCR negatiflerinin sıralanması önerilir, böylece kirleticiler hesaplama analizi sırasında gerekirse tanımlanabilir ve çıkarılabilir. Tersine, PCR amplifikasyonu da çeşitli nedenlerden dolayı başarısız olabilir. Çevresel örnekler için, PCR reaksiyonunun inhibisyonu genellikle Taq polimeraz23'emüdahale eden çeşitli maddelerden kaynaklanabilecek suçludur. İnhibisyondan şüphelenilirse, DNA özlerini seyreltmek için PCR sınıfı su (bkz. Malzeme Tablosu)kullanılabilir.
Bu protokolün birkaç önemli sınırlaması ve olası zorlukları vardır. Numune toplama hem su hem de tortu numuneleri için zor olabilir. Yeterli biyokütle elde etmek için ideal olarak 1 L akarsu suyunun bir filtreden itilmesi gerekir. Filtrenin gözeneklerinin mikropları yakalamak için küçük olması gerekir, ancak tortuyu da hapsedebilir. Son yağışlar nedeniyle suda çok fazla tortu varsa, filtre tıkanabilir ve tüm hacmin filtreden itilmesini zorlaştırır. Tortu toplama için, toplama sırasında tortu derinliğini tahmin etmek zor olabilir. Ayrıca, çakıl taşları ve kayalar daha düşük nükleik asit verimine yol açacağından ve mikrobiyal topluluğun doğru bir temsili olmayabileceğinden, toplanan tortunun ağırlıklı olarak toprak olduğundan emin olmak önemlidir. Son olarak, özellikle koruyucu kullanılmazsa, numunelerin toplamadan sonra buzda tutulması da hayati önem taşır.
Bu protokol hem metatranscriptomics hem de 16S laboratuvar protokollerini kapsasa da, bu iki yöntemin hem süreçte hem de sağladıkları veri türünde çok farklı olduğu vurgulanmalıdır. 16S rRNA geni, bakteriler ve arkealarda yüksek oranda korunan ve bir örnekteki bakteri topluluğunu karakterize etmek için yararlı olan yaygın olarak hedeflenen bir bölgedir. Hedefli ve spesifik bir yaklaşım olmasına rağmen, tür seviyesi çözünürlüğü genellikle ulaşılamaz ve yeni ayrılan türleri veya suşları karakterize etmek zordur. Aksine, metatranscriptomics, bir örnekte bulunan tüm aktif genleri ve mikropları yakalayan daha geniş bir yaklaşımdır. 16S sadece tanımlama için veri sağlarken, metatranscriptomics eksprese genler ve metabolik yollar gibi fonksiyonel veriler sağlayabilir. Her ikisi de değerlidir ve birleştirildiğinde, hangi bakterilerin mevcut olduğunu ve hangi genleri ifade ettiklerini ortaya çıkabilirler.
Bu makalede, fracking çalışması bağlamında hem 16S rRNA hem de metatranscriptomik analizler için alan toplama ve numune işleme yöntemleri açıklanmaktadır. Ayrıca, düşük biyokütle örneklerinden yüksek kaliteli DNA / RNA ve uzun süreli depolama için toplama yöntemlerini detaylandırıyor. Burada açıklanan yöntemler, mikrobiyal topluluklarının yapısını ve işlevini inceleyerek fracking'in yakındaki akışları nasıl etkilediğini öğrenme çabalarımızda örnek toplama ve işleme ile ilgili deneyimlerimizin doruk noktasıdır. Mikroplar rahatsızlıklara hızlı bir şekilde yanıt verir ve sonuç olarak hangi mikropların mevcut olduğu ve ifade ettikleri genler fracking'in ekosistemler üzerindeki etkileri hakkında bilgi sağlayabilir. Genel olarak, bu yöntemler, fracking'in bu önemli ekosistemleri nasıl etkilediğini anlamamızda paha biçilmez olabilir.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Yazarlar, bu yöntemlerin geliştirilmesine yol açan projelerin finansman kaynaklarını kabul etmek isterler: Howard Hughes Tıp Enstitüsü (http://www.hhmi.org) Precollege ve Lisans Bilimleri Eğitim Programı aracılığıyla, ayrıca Ulusal Bilim Vakfı (http://www.nsf.gov) tarafından NSF ödülleri DBI-1248096 ve CBET-1805549 aracılığıyla.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 200 Proof Ethanol | Thermo Fisher Scientific | A4094 | 400 mL need to be added to Buffer PE (see Qiagen QIAQuck Gel Extraction kit protocol) and 96 mL needs to be added to the DNA/RNA Wash Buffer (see ZymoBIOMICS DNA/RNA Miniprep kit protocol). Additional ethanol is needed for the ZymoBIOMICS DNA/RNA Miniprep and NEBNext® Ultra™ II RNA Library Prep with Sample Purification Beads kits. |
| Agarose | Thermo Fisher Scientific | BP1356-100 | 100 g per bottle. 0.6 g of agarose would be needed to make one 2% 30 mL gel. |
| Disinfecting Bleach | Walmart (Clorox) | No catalog number | Use a 10% bleach solution for cleaning the work area before and after lab procedures |
| DNA gel loading dye | Thermo Fisher Scientific | R0611 | Each user-made (i.e. non-e-gel) should include loading dye with all of the samples in the ratio of 1 µL dye to 5 µL sample |
| DNA ladder | MilliporeSigma | D3937-1VL | A ladder should be run on every gel/e-gel |
| DNA/RNA Shield (2x) | Zymo Research | R1200-125 | 3 mL per sediment sample (50 mL conical) and 2 mL per water sample (filter) |
| Ethidium bromide | Thermo Fisher Scientific | BP1302-10 | Used for staining user-made e-gels |
| Forward Primer | Integrated DNA Technologies (IDT) | 51-01-19-06 | 0.5 µL per PCR reaction |
| Isopropanol | MilliporeSigma | 563935-1L | Generally less than 2 mL per library. Volume needed varies by mass of excised gel fragment (see Qiagen QIAQuick Gel Extraction kit protocol). |
| PCR-grade water | MilliporeSigma | 3315932001 | 13 µL per PCR reaction (assuming 1 µL of sample DNA template is used) |
| Platinum Hot Start PCR Master Mix (2x) | Thermo Fisher Scientific | 13000012 | 10 µL per PCR reaction |
| Reverse Primer | Integrated DNA Technologies (IDT) | 51-01-19-07 | 0.5 µL per PCR reaction |
| TBE Buffer (Tris-borate-EDTA) | Thermo Fisher Scientific | B52 | 1 L of 10x TBE buffer (30 mL of 1x TBE buffer would be needed to make one 30 mL gel) |
| 1 L bottle | Thermo Fisher Scientific | 02-893-4E | One needed per stream (the same bottle can be used for multiple streams if it is sterilized between uses) |
| 1.5 mL Microcentrifuge tubes | MilliporeSigma | BR780400-450EA | 5 microcentrifuge tubes are needed per DNA extraction and an additional 3 are needed to purify RNA (see ZymoBIOMICS DNA/RNA Miniprep kit protocol) |
| 2% Agarose e-gel | Thermo Fisher Scientific | G401002 | Each gel can run 10 samples (so 9 with a PCR negative and 8 if the extraction negative is run on the same gel) |
| 50 mL Conicals | CellTreat | 229421 | 1 50 mL conical needed per sediment samples |
| 500 mL Beaker | MilliporeSigma | Z740580 | Only 1 needed (for flame sterilization) |
| Aluminum foil | Walmart (Reynolds KITCHEN) | No number | Aluminum foil can be folded and autoclaved. The part not exposed to the environment can then be used as a sterile, DNA and RNA free surface for processing filters (one folded piece per filter to avoid cross-contamination) |
| Autoclave | Gettinge | LSS 130 | Only one needed |
| Centrifuge | MilliporeSigma | EP5404000138-1EA | Only 1 needed |
| Cooler | ULINE | S-22567 | Just about any cooler can be used. This one is listed due to being made of foam, making it lighter and thus easier to take along for field sampling. |
| Disruptor Genie | Bio-Rad | 3591456 | Only one needed |
| Electrophoresis chamber | Bio-Rad | 1664000EDU | Only 1 needed |
| Electrophoresis power supply | Bio-Rad | 1645050 | Only 1 needed |
| Freezer (-20 C) | K2 SCIENTIFIC | K204SDF | One needed to store DNA extracts |
| Freezer (-80 C) | K2 SCIENTIFIC | K205ULT | One needed to store RNA extracts |
| Gloves | Thermo Fisher Scientific | 19-020-352 | The catalog number is for Medium gloves. |
| Heat block | MilliporeSigma | Z741333-1EA | Only one needed |
| Lab burner | Sterlitech | 177200-00 | Only one needed |
| Laminar Flow Hood | AirClean Systems | AC624LFUV | Only 1 needed |
| Library purification kit | Qiagen | 28704 | One kit has enough for 50 reactions |
| Magnet Plate | Alpaqua | A001219 | Only one needed |
| Microcentrifuge | Thermo Fisher Scientific | 75004061 | Only one needed |
| Micropipette (1000 µL volume) | Pipette.com | L-1000 | Only 1 needed |
| Micropipette (2 µL volume) | Pipette.com | L-2 | Only 1 needed |
| Micropipette (20 µL volume) | Pipette.com | L-20 | Only 1 needed |
| Micropipette (200 µL volume) | Pipette.com | L-200R | Only 1 needed |
| NEBNext Ultra II RNA Library Prep with Sample Purification Beads | New England BioLabs Inc. | E7775S | One kit has enough reagents for 24 samples. |
| Parafilm | MilliporeSigma | P7793-1EA | 2 1" x 1" squares are needed per filter |
| PCR Tubes | Thermo Fisher Scientific | AM12230 | One tube needed per reaction |
| Pipette tips (for 1000 µL volume) | Pipette.com | LF-1000 | Pack of 576 tips |
| Pipette tips (for 20 µL volume) | Pipette.com | LF-20 | Pack of 960 tips |
| Pipette tips (for 200 µL volume) | Pipette.com | LF-250 | Pack of 960 tips |
| PowerWulf ZXR1+ computer cluster | PSSC Labs | No number | This is just an example of a supercomputer powerful enough to perform metatranscriptomics analysis in a timely manner. Only one needed. |
| Qubit fluorometer starter kit | Thermo Fisher Scientific | Q33239 | Comes with a Qubit 4 fluorometer, enough reagent for 100 DNA assays, and 500 Qubit tubes |
| Scoopula | Thermo Fisher Scientific | 14-357Q | Only one needed |
| Sterile blades | AD Surgical | A600-P10-0 | One needed per filter |
| Sterivex-GP Pressure Filter Unit | MilliporeSigma | SVGP01050 | 1 filter needed per water sample |
| Thermocycler | Bio-Rad | 1861096 | Only one needed |
| Vise-grip | Irwin | 2078500 | Only one needed (for cracking open the filters) |
| Vortex-Genie 2 | MilliporeSigma | Z258415-1EA | Only 1 needed |
| WHIRL-PAK bags | ULINE | S-22729 | 1 needed per filter |
| ZymoBIOMICS DNA/RNA Miniprep kit | Zymo Research | R2002 | One kit has enough reagents for 50 samples. |
Referanslar
- The process of unconventional natural gas production. US EPA Available from: https://www.epa.gov/uog/process-unconventional-natural-gas-production (2013)
- Brittingham, M. C., Maloney, K. O., Farag, A. M., Harper, D. D., Bowen, Z. H. Ecological risks of shale oil and gas development to wildlife, aquatic resources, and their habitats. Environmental Science & Technology. 48 (19), 11034-11047 (2014).
- McBroom, M., Thomas, T., Zhang, Y. Soil erosion and surface water quality impacts of natural gas development in East Texas, USA. Water. 4 (4), 944-958 (2012).
- Maloney, K. O., Weller, D. E. Anthropogenic disturbance, and streams: land use and land-use change affect stream ecosystems via multiple pathways. Freshwater Biology. 56 (3), 611-626 (2011).
- Meyer, J. L., et al. The contribution of headwater streams to biodiversity in river networks1. JAWRA Journal of the American Water Resources Association. 43 (1), 86-103 (2007).
- Alexander, R. B., Boyer, E. W., Smith, R. A., Schwarz, G. E., Moore, R. B. The role of headwater streams in downstream water quality. Journal of the American Water Resources Association. 43 (1), 41-59 (2007).
- Ulrich, N., et al. Response of aquatic bacterial communities to hydraulic fracturing in Northwestern Pennsylvania: A five-year study. Scientific Reports. 8 (1), 5683 (2018).
- Chen See, J. R., et al. Bacterial biomarkers of Marcellus shale activity in Pennsylvania. Frontiers in Microbiology. 9, 1697 (2018).
- Rausch, P., et al. Comparative analysis of amplicon and metagenomic sequencing methods reveals key features in the evolution of animal metaorganisms. Microbiome. 7 (1), 133 (2019).
- Louca, S., Doebeli, M., Parfrey, L. W. Correcting for 16S rRNA gene copy numbers in microbiome surveys remains an unsolved problem. Microbiome. 6 (1), 41 (2018).
- Trexler, R., et al. Assessing impacts of unconventional natural gas extraction on microbial communities in headwater stream ecosystems in Northwestern Pennsylvania. Frontiers in Microbiology. 5, 522 (2014).
- Mumford, A. C., et al. Shale gas development has limited effects on stream biology and geochemistry in a gradient-based, multiparameter study in Pennsylvania. Proceedings of the National Academy of Sciences. 117 (7), 3670-3677 (2020).
- JoVE Core Biology DNA Isolation. Journal of Visualized Experiments Available from: https://www.jove.com/cn/science-education/10814/dna-isolation (2020)
- Oxford Gene Technology DNA Storage and Quality. OGT Available from: https://www.ogt.com/resources/literature/403_dna_storage_and_quality (2011)
- ThermoFisher SCIENTIFIC Technical Bulletin #159: Working with RNA. Thermoscientific Available from: https://www.thermofisher.com/us/en/home/references/ambion-tech-support/nuclease-enzymes/general-articles/working-with-rna.html (2020)
- QIAGEN AllPrep DNA/RNA Mini Kit. Qiagen Available from: https://www.qiagen.com/us/products/discovery-and-translational-research/dna-rna-purification/multianalyte-and-virus/allprep-dnarna-mini-kit/#orderinginformation (2020)
- ZymoBIOMICS DNA/RNA Miniprep Kit. Zymo Research Available from: https://www.zymoresearch.com/products/zymobiomics-dna-rna-miniprep-kit (2020)
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565 (2010).
- 16S Illumina amplicon protocol: Earth microbiome project. Earth microbiome project Available from: https://earthmicrobiome.org/protocols-and-standards/16s/ (2018)
- Gel Purification: Binding, washing and eluting a sample | Protocol. Journal of Visualized Experiments Available from: https://www.jove.com/v/5063/gel-purification (2020)
- New England Biolabs protocol for the use with NEBNext Poly(A) mRNA magnetic isolation module (E7490) and NEBNext Ultra II RNA library prep kit for Illumina (E7770, E7775). New England Biolabs Available from: https://www.neb.com/protocols/2017/03/04/protocol-for-use-with-purified-mrna-or-rrna-depleted-rna-and-nebnext-ultra-ii-rna-library-prep-ki (2020)
- Anderson, M. J. Permutational multivariate analysis of variance (PERMANOVA). Wiley StatsRef: Statistics Reference Online. , 1-15 (2017).
- Schrader, C., Schielke, A., Ellerbroek, L., Johne, R. PCR inhibitors - occurrence, properties and removal. Journal of Applied Microbiology. 113 (5), 1014-1026 (2012).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır