Method Article
Mikroalglerin Model İyileştirmeleri için Yüksek Verimli Metabolik Profil oluşturma
Bu Makalede
Özet
Bu protokol, yeşil bir mikroalga olan Chlamydomonas reinhardtii'nin metabolik gereksinimlerini tanımlamak ve mevcut bir metabolik ağ modelini iyileştirmek için bir fenotip mikroarray (PM) teknoloji platformunun kullanımını göstermektedir.
Özet
Metabolik modeller, bir organizmanın mevcut genom ek açıklamasına göre yeniden oluşturulur ve metabolik süreçleri sistem düzeyinde incelemek için tahmine dayalı araçlar sağlar. Genom ölçeğindeki metabolik modeller, deneysel olarak doğrulanmamış reaksiyonların yanı sıra boşluklar içerebilir. Yeni izole edilmiş mikroalgal türlerin yeniden yapılandırılmış modelleri, bu boşluklara bağlı zayıflıklara neden olacaktır, çünkü bu tür izolelerin metabolizması için genellikle seyrek biyokimyasal kanıtlar vardır. Fenotip mikroarray (PM) teknolojisi, çok çeşitli giriş metabolitlerine yanıt olarak hücresel metabolik aktiviteleri işlevsel olarak belirleyen etkili, yüksek verimli bir yöntemdir. Yüksek verimli fenotipik testlerin metabolik modelleme ile birleştirilmesi, genomik kanıtları desteklemek ve genişletmek için biyokimyasal kanıtlar sağlayarak mevcut metabolik ağ modellerinin hızla yeniden haline gelmesine veya optimize edilmesine izin verebilir. Bu çalışma, örnek olarak yeşil mikroalgal model türü Chlamydomonas reinhardtii kullanılarak mikroalglerin incelenmesi için PM testlerinin kullanımını gösterecektir. Bu çalışmada PM tarafından elde edilen 254'ün üzerinde reaksiyon için deneysel kanıtlar, genom ölçeğinde bir C. reinhardtii metabolik ağ modeli olan iRC1080'i yaklaşık yüzde 25 oranında genişletmek ve iyileştirmek için kullanılmıştır. Burada oluşturulan protokol, bilinen mikroalg mutantları ve yeni izolatlar da dahil olmak üzere diğer mikroalglerin metabolizmasının işlevsel olarak profillerinin oluşturulması için bir temel olarak kullanılabilir.
Giriş
Hedeflenen metabolitlerin gelişmiş ve istikrarlı üretimi için alg metabolizmasının optimize edilmesi, metabolik ağların sistem düzeyinde analizleri yoluyla karmaşık metabolik mühendislik stratejilerinin geliştirilmesini gerektirir. Metabolik ağ modelleri optimizasyon stratejilerinin hızlı gelişimi için rasyonel tasarımlara rehberlik edebilir1,2,3,4. Yaklaşık 160 mikroalgal tür sıralanmış olmasına rağmen5, bilgimize göre, sadece 44 alg metabolik model mevcuttur4,6,7. Genomik bilgilerin deneysel olarak doğrulanması için yüksek verimli metabolik fenotipik verilerin elde etmedeki zorluk nedeniyle, yüksek kaliteli ağ modellerinin yeniden inşası, alg genom diziliminin hızlı gelişiminin gerisinde kalmaktadır.
C. reinhardtii, alg bazlı çalışmalar için çekici bir model sistemidir. Bu tür fotoautotropik veya heterotropik olarak büyüyebilir ve temel ve uygulamalı araştırmalarda model organizma olarak yaygın olarak kullanılmıştır. Genom dizisi 20078'de yayınlandı , genom ölçeğindeki metabolik modeller daha sonra 9,10,11türleri için yeniden inşa edildi. C. reinhardtii (iRC1080) için genom ölçeği modeli Chang ve arkadaşları tarafından yeniden inşa edildi. 10 genomik ve literatür kanıtlarına dayanmaktadır (~250 kaynak gerektirir). 2.190 reaksiyonlu 1.706 metabolitvardır 10; ancak, modelin tamlığı o sırada yayınlanan mevcut deneysel kanıtların ötesinde doğrulanamadı.
Fenotip mikroarrays (PMs) teknolojisi, doku kültürü hücrelerinin yanı sıra heterotrofik mikroorganizmalar için metabolik profil oluşturma bilgileri sağlayabilen yüksek verimli bir platformdur. Özellikle, ilk olarak Chlamydomonas reinhardtii12 ve daha sonra kloroidium 13 ve Chlorella 14 türleri için bildirildiği gibi, mikroalglerdeki fenotip-genotip bilgi açığını gidermek için kullanılabilir. Binlerce metabolit, sinyal molekülü, ozmolit ve efektör moleküle verilen hücre yanıtlarını inceleyerek, PM tahlilleri fonksiyonel metabolik profilleme sağlayabilir ve işlev, metabolizma ve çevre duyarlılığı hakkında içgörüler sunabilir15,16,17. Özellikle, PM tahlilleri, her kuyuda bulunan farklı besin maddeleri, metabolitler veya ozmolitler ile 96 kuyulu mikro plakalarda hücrelerin metabolit kullanımını tespit eder. Ayrıca, antibiyotikler ve hormonlar gibi biyoaktif molekülleri de test etmek mümkündür. Tetrazolium bazlı bir redoks boyasının NADH azaltılması ile renk üretiminin yoğunluğuna göre belirlendiği gibi, substratların metabolik kullanımı hücre solunumu açısından değerlendirilir15,16,17. 96 kuyulu mikro plakalardaki deneyler fenotip mikroarray aleti (PMI) platformu ile zaman içinde otomatik olarak izlenebilir ve belirlenebilir. Yirmi 96 kuyulu mikro plaka, karbon, azot, kükürt ve fosfor kaynaklarının yanı sıra farklı ozmotik / iyon ve pH etkilerini kullanmak için hücresel fenotipleri incelemek için ortak set metabolitlerini temsil etmek üzere tasarlanmıştır. PM teknolojisi, mikroorganizmalar için mevcut genom ölçeğindeki metabolik modellerin15 , 16 , 17,18'ingüncellenmesi ve yükseltilmesi içinbaşarıylakullanılmıştır.
Burada gösterilen protokol ve veriler Chaiboonchoe ve ark.'ın daha önce yayınlanan çalışmalarına dayanmaktadır. 12 Sunulan çalışma, mikroalglerin metabolik fenotiplerini karakterize etmek ve C. reinhardtii'nin mevcut bir alg metabolik modelini genişletmek ve yeni metabolik modellerin yeniden inşasına rehberlik etmek için PM tahlil yönteminin kullanımını detaylandırmamaktadır.
Protokol
1. Fenotip Mikroarray Deneyleri
- ABD Minnesota Üniversitesi Chlamydomonas Kaynak Merkezi'nden C. reinhardtii strain CC-503'ü edinin (https://www.chlamycollection.org).
- Hücreleri taze Tris-Asetat-Fosfat (TAP) ortam19'da 400 μg/mL timentin son konsantrasyonlarda büyütün, 50 μg/mL ampisilin ve 400 mikromol foton/m 2 sn altında 100 μg/mL kanamycin (bakteri üremesini engellemek için),25°C'de, iki gün boyunca orta kütük fazı.
- Kültürü 22 °C'de 10 dakika boyunca 2.000 x g'da döndürün ve peletin rahatsız etmeden üst saltantayı atın.
- %0,1 tetrazolium menekşe boyası "D" içeren taze TAP ortamı hazırlayın.
NOT: Her plakada test edilen metabolit kategorisine bağlı olarak bazı besin maddelerini hariç tutmak için bu adımda TAP medyasını değiştirin (örneğin, azot kaynak plakaları için amonyum klorürü hariç tutun). - Peletin, hazırlanan taze TAP ortamlarında (adım 1.2'den) 1 x 106 hücre/mL'lik son konsantrasyona yeniden biriktirin.
- Kimyasal bileşik dizi tahlil plakaları (karbon kaynakları, azot kaynakları, fosfor ve kükürt kaynakları plakaları ve peptit azot kaynakları) kullanın.
- Test plakalarının her kuyusuna 100 μL hücre içeren bir ortam alquot aşılayın.
NOT: Tahlilleri çoğaltın. - Maya özü / pepton plakaları üzerinde hücreleri çizgi ve Smith ve diğerleri gibi gram boyama gerçekleştirmek. Bakteriyel kontaminasyonu izlemek için test öncesi ve sonrası 20.
- Kimyasal bileşik dizi test plakalarını mikro plaka okuyucu sistemine yerleştirin.
- Tüm plakaları 30 °C'de 7 güne kadar kuluçkaya yatırın ve mikro plaka okuyucu sistemini her 15 dakikada bir boya renk değişimini okuyacak şekilde programlayın.
NOT: Çoğu mikro plaka okuyucusu kuluçka sırasında sürekli ışık kaynağı sağlamadığı için, algler heterotrofik solunum yapabilmelidir.
2. Veri Analizi
- Ham kinetik verileri mikro plaka okuyucusundan CSV dosyaları olarak dışa aktarın, daha sonra R'deki Omnilog Fenotip Mikroarray (OPM) paketine giriş olarak kullanılacaktır.
- PM Kinetik veri dönüştürücü yazılımını kullanma; D5E veri dosyalarını yükleyin ve PM kinetik çözümleme yazılımında aşağıdaki komut satırlarını kullanarak OKA dosyalarına dönüştürün:
Yük | Alma (OKA dosyalarının klasörünü bulma) | Filtreleri Doldurma | | al Tüm Plakaları Ekle | Kapatmak.
| Dışa Aktar Okuma verilerini (Kinetik), biçim (CSV) (Tablo Başlığı)seçin ve plakaları seçin (her plaka (tek tek Dosyalar)) | Veri | verme Kurtarmak. - Fenotip Microarray (PM) veri analizini gerçekleştirmek için, R yazılım ortamında çalışan OPM yazılım paketi21,22'yi kullanın. Paket, öğretici ve başvuru belgeleri şu http://www.goeker.org/opm/. R için grafik kullanıcı arabirimi olan RStudio'da, aşağıdaki komutları kullanarak opm paketini ve bağımlılıklarını yükleyin:
kaynak (http://www.goeker.org/opm/install_opm.R)
kitaplık (opm) - Kinetik verilerin CSV dosyalarını içeren dizine gidin ve read_opm işlevini kullanarak verileri alın:
x <- read_opm(".", convert="grp", include=list ("csv:")) - Eğri parametresi tahminini kullanarak kinetik verileri toplar ve ayrıklaştırır.
For (i in 1 :length(x)) {
x[[i]] <_aggre(x[[i]], önyükleme = 0 L, çekirdek = 1 L, yöntem ="splines", seçenekler = set_spline_options("smooth.spline"))
x[[i]] <- do_disc(x[i]], kesme = YANLIŞ)
}
Meta verilerin #Collection
meta veriler <- collect_template("."))
metadata$Strain <- c("BLANK","CC- 503")
için (I in 1 :length(x)) {x[[i]] <- include_metadata(x[[i]], md = meta veriler, replace = TRUE)} - 96 kuyulu plakalar için solunum (veya büyüme) ölçümlerini (y ekseni) zamanın bir işlevi (x ekseni) olarak eşlemek için xy_plot işlevini kullanın.
yazdırma (xy_plot(x[[ 1 ]], ="Strain", theor.max = FALSE)) - Kinetik verilerin hızlı bir karşılaştırmalı genel görünümünü sağlamak için level_plot işlevini kullanarak verileri bir ısı haritası olarak görselleştirin.
level_plot(x, ana = list(), renkler = opm_opt("color.borders"), panel.headers = metadata$Strain, cex = NULL, strip.fmt = list(), striptext.fmt = list(), legend.sep =" ", space ="Lab", bias = 0.7 , num.colors = 200 L) - Ham kinetik eğrilerden önemli biyolojik bilgileri, eğri parametrelerini çıkarın ve gecikme fazını (φ), büyüme hızını (μ), maksimum hücre solunumunu (A) ve eğrinin altındaki alanı (AUC)21ekleyin. Pozitif metabolitleri tanımlamak için, her mikrowell plakasının boşluğuna ek olarak, boyanın ortamla abiyotik reaktivitesini temsil eden negatif kontrolün A değerlerini arka plan çıkarma değerleri olarak kullanın. Ayıklama işlevi A parametresini elde etmek için kullanılır.
opm_opt("curve.param")
param <- özü (x, as.labels = list("Strain")))
- PM Kinetik veri dönüştürücü yazılımını kullanma; D5E veri dosyalarını yükleyin ve PM kinetik çözümleme yazılımında aşağıdaki komut satırlarını kullanarak OKA dosyalarına dönüştürün:
3. Yeni Metabolitlerle İlişkili Reaksiyonların ve Genlerin Tanımlanması
- Kimyasal bileşik dizilerden bulunan metabolitleri kullanarak reaksiyonlar için Enzim Komisyonu numaralarını (VC' ler) tanımlamak için KEGG (Kyoto Genler ve Genomlar Ansiklopedisi) (http://www.genome.jp/kegg/) ve MetaSik (http://metacyc.org/) arayın23,2423,24.
- Tanımlanan EC numaralarını, Joint Genom Institute (JGI), Phytozome (http://www.phytozome.net) ve hakemli yayınlar 23,25 ,26,27gibi birden fazla kullanılabilir alg ek açıklama kaynağında arama temeli olarak kullanın.
- Bir sorgu belirli bir EC numarası için genetik kanıt döndürmezse, C. reinhardtii'yeen yakın türlerden başlayarak diğer organizmalardaki ilgili ilişkili proteinleri tanımlayın, ardından varsayılan ayarlara sahip NCBI PSI-BLAST sunucusunu kullanarak profil tabanlı bir arama yapın ve reaksiyon12ile ilişkili aday genleri tanımlamak için C. reinhardtii'de (nr) gereksiz olmayan proteinler (nr) kullanın.
- EMBL-EBI Pfam (http://pfam.xfam.org/search) veya InterPro (http://www.ebi.ac.uk/interpro/) protein etki alanı tahmin sunucuları aracılığıyla bu BLAST isabetlerini sorgulayarak, aranan EC numarasıyla ilgisi için PSI-BLAST isabetlerini E değerleriyle 0,05 < el ile küratörlüğünü el ile toplar. İkinci iki taramanın, protein için doğru enzimmatik aktivitenin tanımlanmasını sağlamak için kritik adımlar olduğunu unutmayın.
4. Model İyileştirme ve Değerlendirme
- En son COBRA Araç Kutusu v.3.0'ı kullanın28MATLAB'da29,30model iyileştirmesi için aşağıdaki adımları gerçekleştirmek için platform. COBRA Araç Kutusu aşağıdaki adımları izleyerek yüklenebilir: https://opencobra.github.io/cobratoolbox/stable/installation.html . Alternatif olarak, COBRA Toolbox'ın Python (COBRApy) gibi diğer açık kaynaklı programlama dillerinde de uygulandığını unutmayın31) ve şu adresinde mevcuttur: https://opencobra.github.io/cobrapy/ .
- COBRA Toolbox v.3.0'ı yükledikten sonra MATLAB'ı açın ve araç kutusunu başlatmak için aşağıdaki komutu yürütün:
initCobraToolbox; - Cobra Toolbox işlevlerini kullanarak ilişkili genleriyle tanımlanan reaksiyonları metabolik modele ekleyin, örneğin iRC1080, addReaction ve changeGeneAssociation. iRC1080 modelini içeren dizine gidin, http://bigg.ucsd.edu/models/iRC1080'den indirilin ve modeli yüklemek, yeniden adlandırmak ve yeni bir reaksiyon ve ilişkili geni eklemek için aşağıdaki komutları yürütün.
Yükle('iRC 1080 .mat')
modelNew = iRC 1080;
modelNew = addReaction(modelNew, 'D-ALA 2' , ...
{'d-ala[c]' , 'atp[c]' , ...
'D-aladata[c]' , 'adp[c]' , 'pi[c]' , ...
'h[c]' },[- 2 - 1 1 1 1 1 ],false);
modelNEW = changeGeneAssociation(modelNew, ...
'D-ALA 2','au.g 14655 _t 1' ); - Metabolit hücre içi olarak üretilmediği, ancak ortamdan alındığı bazı durumlarda, modele yeni metabolitler için taşıma reaksiyonları ekleyin. Bu taşıma reaksiyonları, bir metabolitin hücre dışı ortamdan sitozola pasif difüzyonunu temsil eder. Ek olarak, metabolit'i hücre dışı ortama girmek veya çıktı almak için addExchangeRxn işlevini kullanarak karşılık gelen bir yapay değişim reaksiyonu ekleyin.
modelNew = addReaction(modelNew, 'CYCPt' , ...
{'cycp[e]','cycp[c]' },[- 1 1 ],true)'
modelNew = addExchangeRxn(modelNew, 'cycp[e]' ,- 1000,1000); - Yeni sonuç modelinin davranışını test edin, örneğin, iBD1106, akı dengesi analizi (FBA) gerçekleştirerek optimizecbmodel işlevini kullanarak, nesnel işlev olarak biyokütlenin en üst düzeye çıkarılması için açık ve karanlık koşullar altında. Işık büyümesi için PRISM solar litho'nun ışık reaksiyonlarının alt ve üst sınırlarını 646,07 (maksimum oran) olarak ayarlayın. Karanlık büyüme için, tüm PRISM ışık reaksiyonlarının sınırlarını sıfıra ayarlayın. Karanlık ve açık koşullarda büyüme için daha önce10 olarak tanımlanan Biyokütle işlevini kullanın. FBA çözümü, reaksiyon akılarına (solution.v) ve daha düşük maliyete (solution.w) karşılık gelen iki vektörün yanı sıra metabolitlerin gölge fiyatlarına (solution.y) karşılık gelen bir vektörü çıkacaktır.
%Işık koşulu altında büyümeyi simüle:
modelNew = changeRxnBounds(modelNew,{ ...
% 'PRISM_solar_litho' , ...
'PRISM_solar_exo' , ...
'PRISM_incandescent_ 60 W' , ...
'PRISM_fluorescent_cool_ 215 W' , ...
'PRISM_metal_halide' , ...
'PRISM_high_pressure_sodium' , ...
'PRISM_growth_room' , ...
'PRISM_white_LED' , ...
'PRISM_red_LED_array_ 653 nm' , ...
'PRISM_red_LED_ 674 nm' , ...
'PRISM_fluorescent_warm_ 18 W' , ...
'PRISM_design_growth' , ...
}, 0, 'b' );
modelNew = changeObjective(modelNew, 'BIOMASS_Chlamy_mixo');
FBAsolutionNew = optimizeCbModel(modelNew, 'max'); - i BD1106 için elde edilen FBA çözümlerini iRC1080 için elde edilenlerle karşılaştırmak için iRC1080 için 4.1.4 adımlarınıyineleyin.
- Modelleri karşılaştırmak için kullanılabilecek bir dizi COBRA yöntemi vardır (örneğin, akı değişkenlik analizi, gen silme çalışmaları, sağlamlık analizleri, akı bölünmüş tahminler, FBA, örnekleme vb.). Ayrıntılı öğreticiler https://opencobra.github.io/cobratoolbox/stable/tutorials/index.html. Burada, iRC1080 modelinin rafine sürümü olan iBD1106 ile karşılaştırıldığı bir örnek, her modelde hesaba katılan metabolitlerin gölge fiyatlarını (biyokütle nesnel işlevinin sistem değişkenlerindeki değişikliklere duyarlılığı) elde ederek verilmiştir.
Metabolitler için gölge fiyatları elde edin:
shadowPrices = tablo(modelNew.mets, ...
modelNew.metNames, FBAsolutionNew.y);
- COBRA Toolbox v.3.0'ı yükledikten sonra MATLAB'ı açın ve araç kutusunu başlatmak için aşağıdaki komutu yürütün:
Sonuçlar
Model alga Chlamydomonas reinhardtii'nin Fenotip Mikroarray taraması
PM tahlilleri, alglerin çeşitli karbon, azot, kükürt ve fosfor kaynaklarını minimum bir ortamda kullanma yeteneğini test eder. Bu yöntem açıklamasında, karbon ve azot metabolizmasını tanımlamak için PM testlerinin nasıl kullanıldığını gösterdik. Karbon ve azot kullanımı kinetiği mikro plaka okuyucu ile ölçüldü. Veriler PMI yazılımı kullanılarak analiz edildi. Seçilen PM test plakalarının (PM01 ve PM03) özet kinetiği Şekil 1'degösterilmiştir. "Xy çizimleri", y ekseni ve x ekseninin sırasıyla ham ölçümlerin ve zamanın değerlerini temsil ettiği 96 kuyu plakalarının tahlilleri için çizilen zaman içinde solunum ölçümlerini görüntüler. Veriler, kinetik verilerin montajını karşılaştırmalı olarak analiz etmek için bir ısı haritası desenine dönüştürüldü.
PM verilerini kullanarak genom ölçeğindeki metabolik ağın rafine edilmesi boru hattı (Şekil 2), yüksek verimli PM testlerinin genomik aramalar tarafından sağlanan deneysel kanıtlarla entegrasyonunu göstermektedir.
PM01 - 04 ve PM10 plakalarından elde edilen PM verilerinin tekrarlanabilirliğini belirlemek için, iki bağımsız çoğaltma deneyinden elde edilen verileri birbirine karşı çizmek için doğrusal bir regresyon analiz edildi (Şekil 3). Şekil 3, verilerin çoğunluğunun 45° çizgisine düştükçe neredeyse benzer olduğunu, sadece birkaç aykırı bilginin mevcut olduğunu ve R2 belirleme katsayılarının 0,9 olduğunu göstermektedir. Algler için deneylerin tutarlılığı ve tekrarlanabilirliği bu arsa ile doğrulanır.
Yeni metabolitlerin tanımlanması
Pm tahlil yedi plakada 662 metabolit tanımladı; PM01-PM04 ve PM06-PM08, Gaz Kromatografisi Uçuş Süresi (GC-TOF) ise 77 metabolit32 (Şekil 4)tespit etmiştir. Bu iki seti iRC1080'de hesaplanan 1068 metabolitlerle karşılaştırırken, üç küme arasında yalnızca altı metabolit çakıştı ve 149 iRC1080 ve PM arasında çakıştı. Bu sonuç, metabolik profil oluşturma platformunun önemli bir yeni metabolik bilgi kaynağı olabileceğini göstermektedir.
Asetik asit, arka plan sinyalini çıkardıktan sonra PM01 plakasında destekleyici karbon olarak tespit edilen tek karbon kaynağıydı. Bu bulgu literatür33 ile tutarlıdır ve PM tahlillerinin özgüllüğünü göstermektedir. Başbakan tahlilleri, C. reinhardtii'nin büyüme için kullanabileceği yeni kükürt, fosfor ve azot kaynaklarını ortaya çıkardı. Kükürt metabolitleri sülfat, tiyosülfat, tetrathionat ve DL-Lipoamid'di. Fosfor kaynakları tiyofosfat, ditofosfat, D-3-fosfo-gliserik asit ve sisteamin-S-fosfattı. Azot kaynağı metabolitleri, daha az yaygın amino asitler de dahil olmak üzere L-amino ve D-amino asitlerdi; L-homoserine, L-pyroglutamic, metilamin, etlamin, etanolamin ve D,L-α-amino-butik ve 108 Di-peptitler ve beş Tri-peptit (Tablo 1). Yeni tanımlanan 128 metabolitin tümü KEGG ve MetaCyc'te ilişkili reaksiyonları, EC numaraları ve yolları için arandı.
Yeni 128 metabolit, 49 benzersiz EC numarasıyla ilişkilendirildi. Bunlardan 15 VC, dahil olmak üzere beş kaynak kullanılarak genomik kanıtlarına bağlandı; Fitozome Sürüm 10.0.234 JGI Sürüm4 35, AUGUSTUS 5.0 ve 5.210 Manichaikul ve ark. 36 ve KEGG13. Genomik kanıtı olmayan metabolitler, ilgili dizilerinin diğer organizmalarda bulunduğu Evrensel Protein Kaynağı web sitesine (UniProt, http://www.uniprot.org/)37,38 girildi. C. reinhardtii'deki homolog diziler, NCBI web sitesinden Konuma Özgü Yinelemeli BLAST (PSI-BLAST, https://blast.ncbi.nlm.nih.gov/Blast.cgi) çalıştırılarak yalnızca önemli hizalamalar üreten diziler (E-değeri <0.005) göz önünde bulundurularak tanımlanmıştır.
Model iyileştirmesi
Yeni 128 metabolitle ilişkili reaksiyonlar, kodlanmış genleriyle birlikte, iRC1080 modeline eklenerek ağı genişletti. Elde edilen model iBD1106, 2.444 reaksiyon, 1.959 metabolit ve 1.106 gen oluşturur (Tablo 2). Eklenen yeni 254 reaksiyon 20 amino asit oksidasyon reaksiyonlarıydı. Dört gen tarafından kodlanmış 108 di-peptit hidroliz reaksiyonları, beş tripeptid hidroliz reaksiyonları ve 120 taşıma reaksiyonları (Cre02.g096350.t1.3, au.g14655_t1, e_gwW.1.243.1, Cre12.g486350.t1.3).
Di-peptitlerin ve tri-peptitlerin hidrolizi için toplam 113 yeni reaksiyon eklendi. Di-peptitlerin ve tri-peptitlerin hidrolizi, biri di-peptitler (Cre02.g078650.t1.3) ve diğeri tri-peptitler (Cre16.g675350.t1.3) için olmak üzere iki genle ilişkilidir.
Fosfor kaynakları ile ilgili olarak, JLM_162926 genle ilişkili olarak sisteamin-S-fosfatın hidrolizinin sisteamin ve fosfat içine reaksiyonu eklenmiştir.
WoLF PSORT aracı39 (http://www.genscript.com/psort/wolf_psort.html) ve Ghamsari ve arkadaşları tarafından bildirilen sonuçlar. Yeni reaksiyonların gerçekleştiği hücresel bölmelerin spesifikasyonunu elde etmek için 35 uygulandı. WoLF PSORT, yeni reaksiyonlarla ilişkili protein dizilerini analiz ederek, reaksiyonlar için hücresel bölme olarak sitozol tahmin etti.
Oluşturulan metabolik model, biyokimyasal bilgiler eksik olduğunda boşluklar içerebilir. Bu gibi durumlarda, bir COBRA komutu olan gapFindkullanılır. Kök boşluklarını listeler ve yeni modelde yeni boşlukların tanıtılmasına izin verir. Metabolik bir modelde üretilemeyen metabolitler kök boşlukları40,41olarak adlandırılır. Kök boşluğunun analiz edildiğinde, hem iRC1080 hem de iBD1106 modellerinin aynı 91 boşluk içerdiği belirtilmiştir. Bu, yeni metabolitleri ve ilişkili reaksiyonlarını eklemenin herhangi bir ek kök boşluğu getirmediğini göstermektedir. Bu protokolde kullanılan fenotipleme yönteminin kök boşluklarını kapatmadığı unutulmamalıdır, çünkü orijinal kök boşluğu metabolitleri fenotipleme tahlillerinde ele alınmayan taşıma veya üretim mekanizmalarından yoksundur. IBD1106'nın metabolik davranışını açık ve karanlık koşullarda test etmek için akı dengesi analizi yapıldı; (asetat yok) ve (asetat ile), sırasıyla. Algoritma, nesnel bir işlev (biyokütle büyümesi) için biyokütle öncül reaksiyonlarını en üst düzeye çıkarır. Her metabolitin belirlenen nesnel işleve katılımını değerlendirmek için, tüm metabolitler için "gölge fiyatları" hesaplandı. Metabolit akı değişiklikleri ile ilgili nesnel işlevdeki değişiklik, bir metabolit30,42'nin gölge fiyatını tanımlar. Bir metabolitin "fazla" olup olmadığının veya nesnel işlevin "sınırlanıp sınırlanmadığının" göstergesi, örneğin biyokütle üretimi gibi gölge fiyat analizi ile belirlenebilir. Negatif veya pozitif gölge fiyat değerleri, ek olarak nesnel işlevi azaltacak veya artıracak metabolitleri ortaya koymaktadır. Gölge fiyatların sıfır değerleri, nesnel işlevi etkilemeyecek metabolitleri ortaya koymaktadır. Şekil 5'te iBD1106 ve iRC1080 arasındaki gölge fiyatların karşılaştırılması, çoğu metabolit için önemli bir değişikliğin gözlenmediğini göstermektedir; ancak, açık ve karanlık büyüme koşullarında sırasıyla 105 ve 70 olguda farklılıklar bulunur. Tablo 4 bu tür metabolitlerin örneklerini içerir.
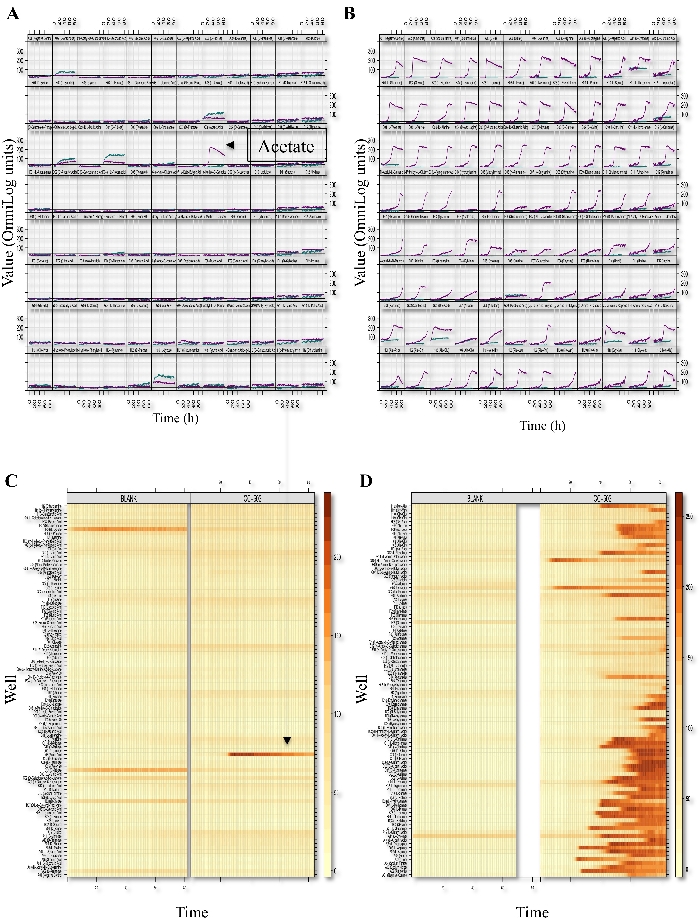
Şekil 1: C. reinhardtii'ninfenotipik mikroarray profillemi. PM01'in solunum XY çizimleri ve seviye çizimleri (Karbon kaynakları; A, C) ve PM03 (Azot kaynakları; B, D) test plakaları gösterilmiştir. Şekil, her hücrenin iyi bir plakayı ve dolayısıyla belirli bir metabolit veya büyüme ortamını temsil ettiği 8x12 dizisidir. Her hücrede veya iyi gösterimde eğriler, zamanın bir işlevi (x ekseni) olarak azaltma (y ekseni) ile boya dönüşümünü temsil eder. CC-503 ve boş kuyulardan gelen PM solunum eğrileri her hücrede gösterilir ve renkle gösterilir (deniz mavisi boş kuyuları, mor renk cc-503'ü temsil eder). Düzey çizimi, her solunum eğrisini zaman içinde ince yatay çizgi değiştirme rengi (veya değişmeden kalan) olarak temsil eder. Isı haritası renk değişiklikleri açık sarıdan (çok az solunum gerçekleşmedi) koyu turuncu veya kahverengiye (önemli solunum gerçekleşti) kadardır. C. reinhardtii (CC-503) tarafından kullanılan metabolitler ve boş plakalar gösterilir. Bu rakam Chaiboonchoe ve ark.'ın daha önce yayınlanan bir eserindendir. 12Bu rakamın daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
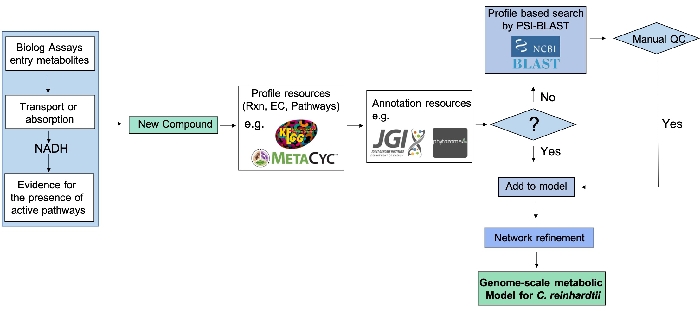
Şekil 2: PM verilerini kullanarak genom ölçeğinde metabolik ağ arıtma boru hattı. Yeni bir bileşik PM testinde pozitif test edildikten sonra, Enzim Komisyonu numarası (EC), reaksiyon ve yolu KEGG ve MetaCyc gibi mevcut veritabanlarından tanımlanır. Genomik kanıtlar daha sonra mevcut olduğunda genomik ve ek açıklama kaynaklarından çıkarılır ve genotip ile fenotip arasında bir bağlantı oluşturur. Doğrudan genomik kanıt kullanılamadığında, protein dizisi EC sayılarından tanımlanır ve genetik kanıtlar PSI-Blast ile tanımlanır. Yeniden oluşturulan metabolik ağ daha sonra yeni tanımlanan bileşiklere göre rafine edilir, ancak yalnızca ilgili veritabanlarını kullanarak protein etki alanlarını sorgulamayı gerektiren bir kalite kontrol adımından sonra. Bu rakam Chaiboonchoe ve ark. 12Bu rakamın daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
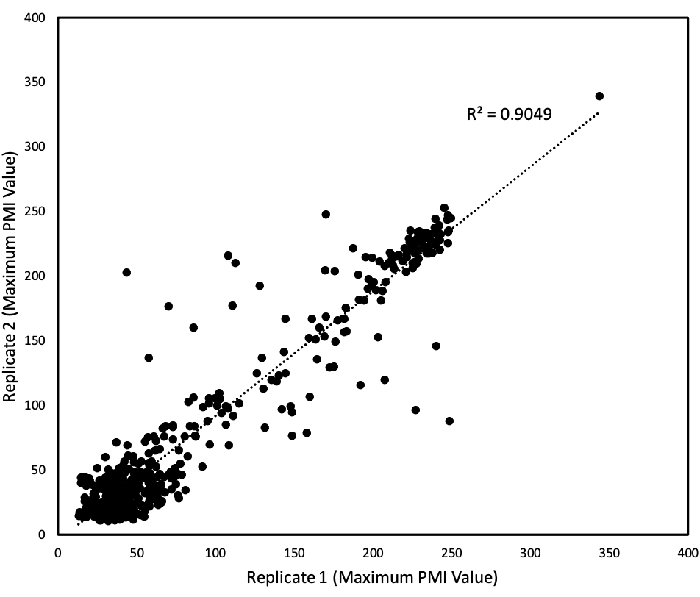
Şekil 3: PM testlerinin tekrarlanabilirliği. PMI değerleri 168 saatlik bir süre içinde toplandı ve maksimum PMI değerleri iki çoğaltma çalışması için çizildi. Her eksen, her çalışma için maksimum PMI değerlerini temsil eder (x ekseni bir çoğaltma çalışması ve y ekseni diğeri). Yeniden üretilen değerler her eksenden eşitlikçidir. Her nokta tek bir maksimum değeri temsil eder. Doğrusal regresyon bir elektronik tablo yazılımı tarafından gerçekleştirildi ve ortaya çıkan belirleme katsayısı (R2)gösterilmiştir. Bu rakam Chaiboonchoe ve ark. 12Bu rakamın daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 4: Metabolitlerin venn diyagramı. Venn diyagramı PM plakaları, iRC1080 metabolik modeli ve Gaz Kromatografi Uçuş Süresi (GC-TOF) deneyleri ile tanımlanan metabolitleri numaralandırır. Her daire, ilgili her çalışma yönteminde bulunan metabolitlerin toplam sayısını gösterir. Aynı zamanda, çakışan bölgeler bu yöntemler arasında paylaşılan metabolit sayısını temsil eder. iRC1080 metabolik modeli toplam 1.068 benzersiz metabolit içerir. GC-TOF toplam tanımladı 77metabolitler 32PM plakaları kullanılarak tanımlanan toplam 662 metabolit vardır. Bu rakam Chaiboonchoe ve ark.'ın daha önce yayınlanan çalışmalarındandır. 12Bu rakamın daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
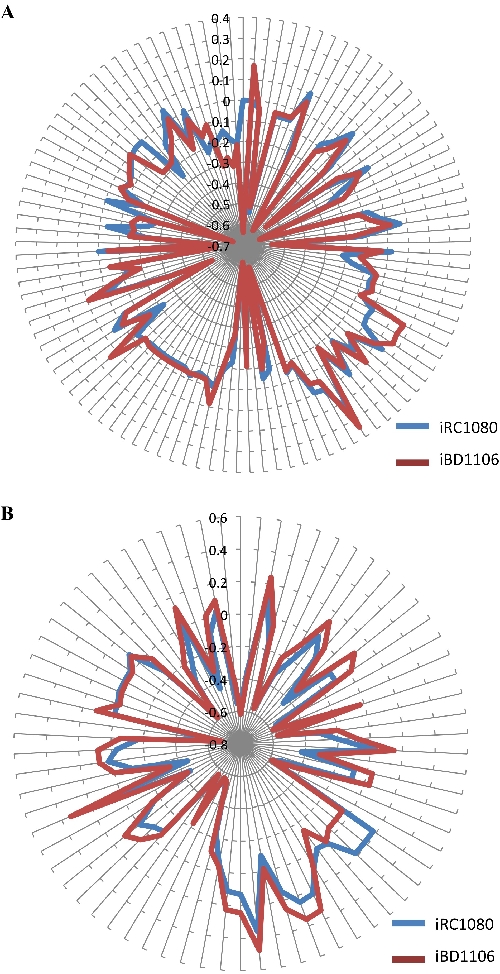
Şekil 5: Biyokütle maksimizasyonu için farklı koşullar altında iRC1080 ve iBD1106'daki metabolitlerin gölge fiyatları. "Radar çizimleri" üzerindeki her daire bir gölge fiyat değerine karşılık gelirken, bir çizimin merkezinden uzanan her çizgi bir metabolit gösterir. (A) Hafif bir büyüme koşulu altında iRC1080 ve iBD1106'nın gölge fiyatları ve metabolik davranışları; (B), karanlık büyüme durumu altında iRC1080 ve iBD1106'nın farklı metabolik davranışları. Bu rakam Chaiboonchoe ve ark.'ın daha önce yayınlanan çalışmalarındandır. 12Bu rakamın daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
| Biyoloji Kimyasal | EC* | Gen Ek Açıklaması | PSI-BLAST |
| Sisteamin-S-Fosfat | 3.1.3.1 | JLM_1629261,2,3,4 | |
| Tetrathionat | 1.8.2.2 | önemsiz E-değeri | |
| 1.8.5.2 | önemsiz E-değeri | ||
| D-Alanine | 1.4.1.1 | XP_001700222.1 | |
| 1.5.1.22 | başarısız el ile QC | ||
| 2.1.2.7 | önemsiz E-değeri | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 2.3.2.10 | önemsiz E-değeri | ||
| 2.3.2.14 | önemsiz E-değeri | ||
| 2.3.2.16 | önemsiz E-değeri | ||
| 2.3.2.17 | önemsiz E-değeri | ||
| 2.3.2.18 | önemsiz E-değeri | ||
| 2.6.1.21 | başarısız el ile QC | ||
| 3.4.13.22 | XP_001698572.1, XP_001693532.1, XP_001701890.1, XP_001700930.1 | ||
| 3.4.16.4 | Chlre2_kg.iskele_ 140000391,2,3 | ||
| 3.4.17.8 | başarısız el ile QC | ||
| 3.4.17.13 | önemsiz E-değeri | ||
| 3.4.17.14 | önemsiz E-değeri | ||
| 4.5.1.2 | önemsiz E-değeri | ||
| 6.1.1.13 | başarısız el ile QC | ||
| 6.1.2.1 | başarısız el ile QC | ||
| 6.3.2.4 | au.g14655_t11,2,3 | ||
| 6.3.2.10 | başarısız el ile QC | ||
| 6.3.2.16 | önemsiz E-değeri | ||
| 6.3.2.35 | önemsiz E-değeri | ||
| D-Kuşkonmaz | 1.4.5.1 | önemsiz E-değeri | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 3.1.1.96 | önemsiz E-değeri | ||
| 2.3.1.36 | önemsiz E-değeri | ||
| 1.4.99.1 | XP_001692123.1 | ||
| 3.5.1.77 | e_gwW.1.243.11,2 | ||
| 3.5.1.81 | önemsiz E-değeri | ||
| 5.1.1.10 | başarısız el ile QC | ||
| D-Aspartik Asit | 6.3.1.12 | önemsiz E-değeri | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| D-Glutamik Asit | 1.4.3.7 | önemsiz E-değeri | |
| 1.4.3.3 | önemsiz E-değeri | ||
| D-Lizin | 5.4.3.4 | önemsiz E-değeri | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| 6.3.2.37 | başarısız el ile QC | ||
| D-Serine | 2.7.11.8 | önemsiz E-değeri | |
| 2.7.11.17 | Cre12.g486350.t1.31,2,3,4 | ||
| 3.4.21.78 | başarısız el ile QC | ||
| 3.4.21.104 | başarısız el ile QC | ||
| 4.3.1.18 | g6244.t14 | başarısız el ile QC | |
| 6.3.2.35 | önemsiz E-değeri | ||
| 6.3.3.5 | önemsiz E-değeri | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| D-Valine | 1.21.3.1 | başarısız el ile QC | |
| 6.3.2.26 | başarısız el ile QC | ||
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| L-Pyroglutamic Asit | |||
| Tiyofosfat | |||
| Dithiophosphate | |||
| Etilaminin | 6.3.1.6 | ||
| D,L-a-Amino-Butik Asit | 2.1.1.49 | önemsiz E-değeri | |
| 1.4.3.3 | Cre02.g096350.t1.35 | ||
| Di-peptit | 3.4.13.18 | Cre02.g078650.t1.31 | |
| Tri-peptit | 3.4.11.4 | Cre16.g675350.t1.31 |
Tablo 1: Tanımlanan pozitif substrat kullanım metabolitlerinin listesi (C, P, S, N) iRC1080 metabolik modelinde mevcut değildir. *Gen tanımlanmazsa reaksiyona dahil edilemedi. 1 Fitozome sürüm 10.0.2 (http://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Creinhardtii). 2 JGI sürüm 4 35. 3 Augustus sürüm 510. 4 KEGG (http://www.genome.jp/kegg/kegg1.html). 5 JGI sürüm 3.136. Bu tablo Chaiboonchoe ve ark.'ın daha önce yayınlanmış çalışmalarındandır. 12
| Model | Reaksiyon | Metabolitleri | Gen |
| iRC1080 | 2,191 | 1,706 | 1,086 |
| iBD1106 | 2,445 | 1,959 | 1,106 |
Tablo 2: iRC1080 ve iBD1106'nıniçeriği. Bu tablo Chaiboonchoe ve ark.'ın daha önce yayınlanmış çalışmalarındandır. 12
| Reaksiyon kategorisi veya sınıfı | Tepki sayısı |
| Amino Asitler | 20 |
| Dipeptides | 108 |
| Tripeptides | 5 |
| Taşıma reaksiyoni | 120 |
Tablo 3: iBD1106'daki yeni tepkilerin özeti. Bu tablo Chaiboonchoe ve ark.'ın daha önce yayınlanmış çalışmalarındandır. 12
| Büyüme durumu | Metabolit | Ad | iRC1080 | iBD1106 | |
| 4r5au | 4-(1-D-Ribitylamino)-5-aminouracil | 0 | 0.168 | ||
| 5aprbu | 5-Amino-6-(5'-fosforibitylamino)urasil | -0.009 | 0.158 | ||
| Işık | pa1819Z18111Z | 1-(9Z)-oktasetnoyl,2-(11Z)-sekizgen-sn-gliserol3-fosfat | -0.009 | -0.65 | |
| Koyu | 4abut | 4-aminobutanoat | 0.18 | -0.05 | |
Tablo 4: iRC1080 ve iBD1106 için önemli gölge fiyatları örneği. Bu tablo Chaiboonchoe ve ark.'ın daha önce yayınlanmış çalışmalarındandır. 12
Tartışmalar
Yeşil mikroalganın metabolik fenotipingi, C. reinhardtii, burada yüksek verimli PM test plakaları ve değiştirilmemiş bir PMI kullanılarak tanımlanmıştır. Tahliller peptit azot kaynakları (PM06-08) ile birlikte toplam 190 karbon kaynağı (PM01 ve PM02), 95 azot kaynağı (PM03), 59 fosfor kaynağı ve 35 kükürt kaynağı (PM04) için kullanılmıştır. 148 besin için pozitif solunum gözlendi (C-kaynağı kullanımı için bir pozitif test, her S-kaynağı ve P-kaynağı kullanımı için dört pozitif test ve N-kaynak kullanımı için 139 pozitif tahlil). Ortamın substratları veya besin maddeleri (karbon, azot, fosfor veya kükürt) bileşeni, bu kaynakların her biri için test eden ilgili PM mikro plakalarına uygulandığında tanımlanan ortama eklenmemelidir.
Burada gösterilen yöntem, mevcut metabolik ağ modellerini genişletmek veya yeni modellerin yeniden yapılandırılmasını yönlendirmek için kullanılabilecek metabolik mikroalg fenotiplerini karakterize etmek için etkilidir. Ayrıca, çoğu mikroalgin beslenme gereksinimleri bilinmediğinden, bu platform bunları hızlı bir şekilde tanımlamak için kullanılabilir. Nelson ve diğerleri. 43, mikroalg Kloroidium sp. UTEX 3007'nin büyümesini destekleyen yeni bileşikleri tanımlamak için bu yöntemleri başarıyla uygulamış ve elde edilen bilgileri Chlamydomonas'ın aksine 40 farklı karbon kaynağı içeren tür giriş metabolitlerini tanımlamak için kullanmıştır.
Pm'nin mikroalglerin profilini çıkarmak için önemli bir sınırlaması, PMI'ın kuluçka odasında aydınlatma olmaması ve mikroalglerin heterotrofik metabolizmayı gerçekleştirebilmesi gerektiğidir. Işığın yokluğu, metabolik akıları hesaplamak için ışığı içeren modellerin yorumunu etkileyebilir. Koordinasyon fonksiyonlarına sahip gen çiftleri metabolik ağ merkezlerini oluşturmak için birlikte gelişti ve fotokintitik ve fotosetik olmayan ağ hub'ları arasındaki ayrımyapılabilir 44. Genel olarak, fotosetik ağ hub'ları (yani modeldeki yüksek bağlı düğümler) heterotrofik modellerin dışında kalır. Pratik amaçlar için, miksotrofik türlerde heterotrofik modelleme, ışık tarafından yönlendirdiği bilinen reaksiyonları atlamalı ve koşullar arasındaki enerji dengesi farklılıklarını hesaba katmalıdır. Bu nedenle, ışığa bağımlı ve ışıktan bağımsız metabolizma modelleme Chlamydomonas metabolikmodellemedestandart uygulamadır 6,45.
Trebouxiophytes gibi bazı yeşil mikroalglerin büyüme için çeşitli karbon moleküllerini özümsediği bilinmektedir ve bunun likenlerin üyeleri olarak uzun evrimsel tarihlerinden kaynaklandığı düşünülmektedir46. Chlamydomonas gibi klorofitler büyüme için asetat kullanabilirken, ticari olarak çok uzun zincirli çoklu doymamış yağ asitleri (VLC-PUFAs) üretme potansiyeli ile bilinen kahverengi deniz mikroalgası Tisochrysis lutea, asetat kullanamaz, ancak büyüme için gliserol kullanabilir47. 100 g l−1 kuru hücre ağırlığından fazla biyokütle konsantrasyonu, fed-batch modunda organik karbon kaynaklarının optimize edilmiş eklenmesiyle Chlorella ile elde edilmiştir48. Ayrıca, Chlorellavulgaris'e şeker eklenmesi CO2'ninelektrasyonunu yükseltebilir, böylece fotosetik büyüme sırasında katkı maddesi yararı sağlar49. Çoğu heterotrofik mikroalg de miksotropik olarak büyüyebilir, ancak Klorofit Krokloris zofingiensis'in şeker50ilavesi üzerine fotosentezi kapattığı gösterilmiştir.
Bacillariophyta bölümüne ait diatomlar, fitoplanktonların önemli bir grubudur. Diatomların çoğu sadece fotoautotropik olarak büyüyebilmesine rağmen, bazıları miktotropik veya heterotropik olarak yetiştirilebilir51. Örneğin, gliserol, Phaeodactylum tricornutum52 model türleri de dahil olmak üzere bazı diatomlarda CO2'ninyokluğunda ışıkta büyümeyi desteklediği bulunmuştur. Ayrıca, Nitzschia linearis gibi bazı bentik diatomlar karanlıkta karbonhidratlar üzerinde büyüyebilir53. Hücrelerin heterotrofik olarak büyümesini sağlamak için uygun organik karbon kaynaklarını destekleyerek PM testlerini diatomlara ve diğer alg gruplarına genişletmesi muhtemeldir ve bir mikstrofi stratejisi, minimum derecede gerekli bir ışık kaynağı sağlayan zorunlu ototrofi mikroalgleri için de potansiyel olarak kullanılabilir.
Verilerin tekrarlanabilirliğini değerlendirmek için, tüm plakalar için yinelenen tahlillerin yapılması şiddetle tavsiye edilir. Bir tahlil ancak negatif kontrolden ve ilgili boş kuyulardan çıktıktan sonra absorbans (PMI değeri) pozitifse pozitif olarak kabul edilebilir. Bu açıklama, test edilen bileşiğin varlığında, boyanın abiyotik reaksiyonunun ortamla bir yansımasıdır.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma için büyük destek, New York Üniversitesi Abu Dabi Araştırma Enstitüsü hibesi (73 71210 CGSB9) ve NYU Abu Dabi Fakültesi Araştırma Fonları (AD060) kapsamında Tamkeen tarafından finanse edilen NYUAD Genomik ve Sistem Biyolojisi Merkezi (CGSB) tarafından sağlandı. W.F. ayrıca Zhejiang Üniversitesi'nin Yüz Yetenek Programı tarafından desteklendi. Ashish Jaiswal'a videoyu kaydetmede yardım için teşekkür ederiz. Hong Cai'ye metabolik fenotip verilerini ürettiği için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Ampicillin | VWR | 97062-796 | |
| Biolog assay plates [ PM01-08] | Biolog, Hayward, CA, USA | ||
| Biolog Omnilog Instrument | Biolog, Hayward, CA, USA | ||
| Chlamydomonas reinhardtii strain CC-503 | Chlamydomonas Resource Center at the University of Minnesota, USA. | Regents of the University of Minnesota | |
| Kanamycin | VWR | 0408-EU-10G | |
| Tetrazolium Violet Dye “D” | Biolog, Hayward, CA, USA | ||
| Timentin | GlaxoSmithKline Australia Pty Ltd | 42010012-2 |
Referanslar
- Oberhardt, M. A., Palsson, B. &. #. 2. 1. 6. ;., Papin, J. A. J. M. Applications of genome-scale metabolic reconstructions. Molecular Systems Biology. 5 (1), 320 (2009).
- Schmidt, B. J., Lin-Schmidt, X., Chamberlin, A., Salehi-Ashtiani, K., Papin, J. A. Metabolic systems analysis to advance algal biotechnology. Biotechnology Journal. 5 (7), 660-670 (2010).
- Koskimaki, J. E., Blazier, A. S., Clarens, A. F., Papin, J. A. J. I. B. Computational models of algae metabolism for industrial applications. Industrial Biotechnology. 9 (4), 185-195 (2013).
- Koussa, J., Chaiboonchoe, A., Salehi-Ashtiani, K. J. B. Computational approaches for microalgal biofuel optimization: a review. BioMed Research. 2014, 649453 (2014).
- Nelson, D. R., et al. Large-scale genome sequencing reveals the driving forces of viruses in microalgal evolution. Cell Host & Microbe. 29 (2), 250-266 (2021).
- Shene, C., Asenjo, J. A., Chisti, Y. Metabolic modelling and simulation of the light and dark metabolism of Chlamydomonas reinhardtii. The Plant Journal. 96 (5), 1076-1088 (2018).
- Tibocha-Bonilla, J. D., Zuñiga, C., Godoy-Silva, R. D., Zengler, K. Advances in metabolic modeling of oleaginous microalgae. Biotechnology for Biofuels. 11 (1), 241 (2018).
- Merchant, S. S., et al. The Chlamydomonas genome reveals the evolution of key animal and plant functions. Science. 318 (5848), 245-250 (2007).
- May, P., Christian, J. -. O., Kempa, S., Walther, D. J. B. G. ChlamyCyc: an integrative systems biology database and web-portal for Chlamydomonas reinhardtii. BMC Genomics. 10 (1), 209 (2009).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), (2011).
- de Oliveira Dal'Molin, C. G., Quek, L. -. E., Palfreyman, R. W., Nielsen, L. K. AlgaGEM - a genome-scale metabolic reconstruction of algae based on the Chlamydomonas reinhardtii genome. BMC Genomics. 12 (5), (2011).
- Chaiboonchoe, A., et al. Microalgal metabolic network model refinement through high-throughput functional metabolic profiling. Frontiers in Bioengineering and Biotechnology. 2, 68 (2014).
- Kanehisa, M., et al. Data, information, knowledge and principle: back to metabolism in KEGG. Nucleic Acids Research. 42 (1), 199-205 (2014).
- Zuñiga, C., et al. Genome-scale metabolic model for the green alga Chlorella vulgaris UTEX 395 accurately predicts phenotypes under autotrophic, heterotrophic, and mixotrophic growth conditions. Plant Physiology. 172 (1), 589-602 (2016).
- Bochner, B. R. New technologies to assess genotype-phenotype relationships. Nature Reviews Genetics. 4 (4), 309-314 (2003).
- Bochner, B. R. Global phenotypic characterization of bacteria. FEMS Microbiology Reviews. 33 (1), 191-205 (2009).
- Bochner, B. R., Gadzinski, P., Panomitros, E. Phenotype microarrays for high-throughput phenotypic testing and assay of gene function. Genome Research. 11 (7), 1246-1255 (2001).
- Bartell, J. A., Yen, P., Varga, J. J., Goldberg, J. B., Papin, J. A. Comparative metabolic systems analysis of pathogenic Burkholderia. Journal of Bacteriology. 196 (2), 210-226 (2014).
- Gorman, D. S., Levine, R. J. P. o. t. N. A. o. S. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. PNAS. 54 (6), 1665-1669 (1965).
- Smith, A. C., Hussey, M. A. Gram stain protocols. American Society for Microbiology. , 1-9 (2005).
- Vaas, L. A. I., et al. opm: an R package for analysing OmniLog phenotype microarray data. Bioinformatics. 29 (14), 1823-1824 (2013).
- Vaas, L. A. I., Sikorski, J., Michael, V., Göker, M., Klenk, H. -. P. Visualization and Curve-Parameter Estimation Strategies for Efficient Exploration of Phenotype Microarray Kinetics. PLoS ONE. 7 (4), 34846 (2012).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes-a 2019 update. Nucleic Acids Research. 48 (1), 445-453 (2020).
- Kanehisa, M., Furumichi, M., Sato, Y., Ishiguro-Watanabe, M., Tanabe, M. KEGG: integrating viruses and cellular organisms. Nucleic Acids Research. , (2020).
- Lopez, D., Casero, D., Cokus, S. J., Merchant, S. S., Pellegrini, M. Algal Functional Annotation Tool: a web-based analysis suite to functionally interpret large gene lists using integrated annotation and expression data. BMC Bioinformatics. 12 (1), 282 (2011).
- Caspi, R., et al. The MetaCyc database of metabolic pathways and enzymes. Nucleic Acids Research. 46, 633-639 (2018).
- Sahoo, S., et al. dEMBF v2. 0: An Updated Database of Enzymes for Microalgal Biofuel Feedstock. Plant and Cell Physiology. 61 (5), 1019-1024 (2020).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 14 (3), 639-702 (2019).
- Heirendt, L., et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v. 3.0. Nature Protocols. 1, (2019).
- Orth, J. D., Thiele, I., Palsson, B. &. #. 2. 1. 6. ;. What is flux balance analysis. Nature Biotechnology. 28 (3), 245 (2010).
- Ebrahim, A., Lerman, J. A., Palsson, B. O., Hyduke, D. R. COBRApy: constraints-based reconstruction and analysis for python. BMC Systems Biology. 7 (1), 74 (2013).
- Bölling, C., Fiehn, O. Metabolite profiling of Chlamydomonas reinhardtii under nutrient deprivation. Plant Physiology. 139 (4), 1995-2005 (2005).
- Harris, E. H. . The Chlamydomonas sourcebook: introduction to Chlamydomonas and its laboratory use. 1, (2009).
- Goodstein, D. M., et al. Phytozome: a comparative platform for green plant genomics. Nucleic Acids Research. 40, 1178-1186 (2012).
- Ghamsari, L., et al. Genome-wide functional annotation and structural verification of metabolic ORFeome of Chlamydomonas reinhardtii. BMC Genomics. 12 (1), 4 (2011).
- Manichaikul, A., et al. Metabolic network analysis integrated with transcript verification for sequenced genomes. Nature Methods. 6 (8), 589-592 (2009).
- Apweiler, R., et al. UniProt: the Universal Protein knowledgebase. Nucleic Acids Research. 32, 115-119 (2004).
- Consortium, T. U. Activities at the universal protein resource (UniProt). Nucleic Acids Research. 42 (11), 7486-7486 (2014).
- Horton, P., et al. PSORT: protein localization predictor. Nucleic Acids Research. 35, 585-587 (2007).
- Becker, S. A., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox. Nature Protocols. 2 (3), 727-738 (2007).
- Schellenberger, J., et al. Quantitative prediction of cellular metabolism with constraint-based models: the COBRA Toolbox v2.0. Nature Protocols. 6 (9), 1290 (2011).
- Varma, A., Boesch, B. W., Palsson, B. O. Stoichiometric interpretation of Escherichia coli glucose catabolism under various oxygenation rates. Applied and Environmental Microbiology. 59 (8), 2465-2473 (1993).
- Nelson, D. R., et al. The genome and phenome of the green alga Chloroidium sp. UTEX 3007 reveal adaptive traits for desert acclimatization. eLife. , 25783 (2017).
- Chaiboonchoe, A., et al. Systems level analysis of the Chlamydomonas reinhardtii metabolic network reveals variability in evolutionary co-conservation. Molecular BioSystems. 12 (8), 2394-2407 (2016).
- Chang, R. L., et al. Metabolic network reconstruction of Chlamydomonas offers insight into light-driven algal metabolism. Molecular Systems Biology. 7 (1), 518 (2011).
- Rajendran, A., Hu, B. Mycoalgae biofilm: development of a novel platform technology using algae and fungal cultures. Biotechnology for Biofuels. 9 (1), 112 (2016).
- Hu, H., et al. Effect of cultivation mode on the production of docosahexaenoic acid by Tisochrysis lutea. AMB Express. 8 (1), 50 (2018).
- Bumbak, F., Cook, S., Zachleder, V., Hauser, S., Kovar, K. Best practices in heterotrophic high-cell-density microalgal processes: achievements, potential and possible limitations. Applied Microbiology and Biotechnology. 91 (1), 31 (2011).
- Fu, W., et al. Sugar-stimulated CO2 sequestration by the green microalga Chlorella vulgaris. Science of the Total Environment. 654, 275-283 (2019).
- Roth, M. S., et al. Regulation of oxygenic photosynthesis during trophic transitions in the green alga Chromochloris zofingiensis. The Plant Cell. , (2019).
- Villanova, V., et al. Investigating mixotrophic metabolism in the model diatom Phaeodactylum tricornutum. Philosophical Transactions of the Royal Society B: Biological Sciences. 372 (1728), 20160404 (2017).
- Cerón-García, M., et al. Mixotrophic growth of Phaeodactylum tricornutum on fructose and glycerol in fed-batch and semi-continuous modes. Bioresource Technology. 147, 569-576 (2013).
- Tuchman, N. C., Schollett, M. A., Rier, S. T., Geddes, P. . Advances in Algal Biology: A Commemoration of the Work of Rex Lowe. , 167-177 (2006).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır