Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Kesme Kuvvetleri ile Topluluk Kuvveti Spektroskopisi
Bu Makalede
Özet
Topluluk kuvvet spektroskopisi (EFS), biyofiziksel ve biyosensing alanlarında bir grup biyomoleküler yapının mekanik olarak açılması ve gerçek zamanlı olarak algılanması için sağlam bir tekniktir.
Özet
Floresan ve mekanokimyasal prensiplere dayanan tek moleküllü teknikler biyolojik algılamada üstün duyarlılık sağlar. Bununla birlikte, yüksek verim yeteneklerinin eksikliği nedeniyle, bu tekniklerin uygulanması biyofizikte sınırlıdır. Topluluk kuvvet spektroskopisi (EFS), bireysel moleküllerin mekanokimyasal çalışmalarını moleküler toplulukların çalışmalarına dönüştürerek büyük bir moleküler yapı kümesinin araştırılmasında yüksek verim göstermiştir. Bu protokolde, DNA ikincil yapıları (i-motifler), bir homojenizatör ucunun rotoru ile statoru arasındaki kesme akışında 77796 / s'ye kadar kesme hızlarında açılmıştır. Akış hızlarının ve moleküler boyutların i-motifin yaşadığı kesme kuvvetleri üzerindeki etkileri gösterilmiştir. EFS tekniği ayrıca DNA i-motifleri ve ligandlar arasındaki bağlanma afinitesini de ortaya çıkardı. Ayrıca, kesme kuvveti (yani, mekano-tıklama kimyası) ile harekete geçirilebilen bir tıklama kimyası reaksiyonu gösterdik. Bu sonuçlar, moleküler yapıların konformasyonunu kontrol etmek için kesme kuvveti kullanmanın etkinliğini ortaya koymaktadır.
Giriş
Tek moleküllü kuvvet spektroskopisi1'de (SMFS), bireysel moleküler yapıların mekanik özellikleri, atomik kuvvet mikroskobu, optik cımbız ve manyetik cımbız 2,3,4 gibi sofistike aletlerle incelenmiştir. Kuvvet üreten/algılayan kurulumlardaki moleküllerin aynı yönlülük gereksinimi veya manyetik cımbızdaki küçük görüş alanı ve minyatür santrifüj kuvvet mikroskobu (MCF)5,6,7,8 ile sınırlı olarak, SMFS kullanılarak aynı anda yalnızca sınırlı sayıda molekül araştırılabilir. SMFS'nin düşük verimi, büyük bir molekül kümesinin katılımını gerektiren moleküler tanıma alanındaki geniş uygulamasını önler.
Kesme akışı, büyük bir molekül kümesine kuvvet uygulamak için potansiyel bir çözüm sağlar9. Bir kanalın içindeki sıvı akışında, kanal yüzeyine ne kadar yakın olursa, akış hızı10 o kadar yavaş olur. Böyle bir akış hızı gradyanı, sınır yüzeyine paralel kesme gerilimine neden olur. Bir molekül bu kesme akışına yerleştirildiğinde, molekül kendisini yeniden yönlendirir, böylece kesme kuvveti uzun eksen11'e uygulandığında, uzun ekseni akış yönüyle hizalanır. Bu yeniden yönlendirmenin bir sonucu olarak, aynı tipteki tüm moleküllerin (sapların boyutu ve uzunluğu) aynı kesme kuvvetini yaşarken aynı yönde hizalanması beklenir.
Bu çalışma, DNA i-motifi tarafından örneklendiği gibi, büyük bir moleküler yapı kümesine kesme kuvveti uygulamak için böyle bir kesme akışını kullanmak için bir protokolü tanımlamaktadır. Bu protokolde, homojenizatör ucundaki rotor ve stator arasında bir kesme akışı oluşturulur. Bu çalışma, katlanmış DNA i-motif yapısının 9724-97245 s-1 kesme oranlarıyla açılabileceğini buldu. Ayrıca, L2H2-4OTD ligand ile i-motif arasında 36 μM'lik bir ayrışma sabiti bulundu. Bu değer, jel kayma testi 12 ile ölçülen 31 μM iletutarlıdır. Ayrıca, mevcut teknik, bir tıklama reaksiyonunu katalize etmek için şelatlı bakırı (I) açığa çıkarabilen i-motifi açmak için kullanılır. Bu protokol böylece, makul bir sürede (30 dakikadan daha kısa) düşük maliyetli aletlerle büyük bir dizi i-motif yapısının ortaya çıkmasına izin verir. Kesme kuvveti tekniğinin kuvvet spektroskopisinin verimini büyük ölçüde arttırdığı göz önüne alındığında, bu tekniğe topluluk kuvvet spektroskopisi (EFS) diyoruz. Bu protokol, bu kesme kuvvetine dayalı EFS'nin uygulanmasını kolaylaştırmak için deneysel kılavuzlar sağlamayı amaçlamaktadır.
Protokol
NOT: Bu protokolde kullanılan tüm tamponlar ve kimyasal reaktifler Tablo Malzemeleri'nde listelenmiştir.
1. Kesme kuvveti mikroskobunun hazırlanması
NOT: Kesme kuvveti mikroskobu, bir reaksiyon ünitesi (homojenizatör) ve bir algılama ünitesi (floresan mikroskobu) olmak üzere iki bölümden oluşur. Göz merceğinin büyütülmesi 10x'tir ve objektif lensin (hava) büyütmesi 4x'tir.
- Homojenizatörü ve mikroskobu bir montaj tablasına monte edin. Gözlük takın ve floresan mikroskobu açın ve ardından uygun bir dalga boyundaki uyarma ışık demetinin (burada 488 nm kullanıldı) homojenizatörün dispersiyon ucunun merkezinden geçtiğinden emin olmak için homojenizatörü ayarlayın.
- Enine kesitte 5 cm yüksekliğinde ve 1,5 cm2 x 1,5cm2 yüksekliğinde düz tabanlı bir reaksiyon odası hazırlayın. Arka planı azaltmak için, seçilen hazne malzemesinin cam gibi floresan olmadığından emin olun (bazı plastiklerde floresan vardır).
- İstenilen kesme kuvvetini sağlayabilen uygun bir homojenizatör dispersiyon ucu seçin.
NOT: Kesme hızı, aşağıdaki denkleme göre sabit bir dönme hızında rotor ve stator arasındaki mesafeye bağlıdır11 :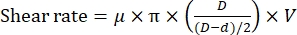
burada μ, 20 ° C'de suyun dinamik viskozitesidir; D, statorun iç çapıdır; d, rotorun dış çapıdır ve V, kesme hızıdır (rpm / s). - Reaksiyon odasını floresan mikroskobun numune aşamasına sıkıştırın ve ardından odayı homojenizatörün dispersiyon ucunu tutacak şekilde ayarlayın (Şekil 1). Dispersiyon ucunun, reaksiyon odasının alt yüzeyinin biraz üzerinde (~1 mm) olduğundan emin olun.
- Mikroskopun odağının dispersiyon ucunun yüzeyinde olduğundan emin olmak için homojenizatörün ve haznenin dikey konumunu birlikte ayarlayın. Ardından, algılama alanının (görüş alanı) rotor ve stator arasında ayarlandığından emin olmak için dağılım ucunun yatay konumunu ayarlayın (Şekil 1).
- Deneyde kullanılan floresan boyaya göre floresan kanallarını açın.
- Yüksek hızlı kesme deneyinden önce, dispersiyon ucunun reaksiyon odasına dokunmadan düzgün çalışabilmesini sağlamak için kesmeyi düşük kesme hızıyla (örneğin, 2.000 rpm) test etmek için deiyonize (DI) su kullanın.
2. Ligandlı ve ligandsız i-motiflerin açılması
- Hu ve ark.11'de açıklandığı gibi, DI suyunda sırasıyla iki ucunda bir boya ve bir söndürücü ile etiketlenmiş bir insan telomerik i-motif DNA'sı (Malzeme Tablosu) hazırlayın.
NOT: Dizi içeren i-motif: 5'-TAA CCC TAA CCC TAA CCC TAA CCC TAA. - DNA'yı pH 5.5 veya pH 7.4'te 30 mM MES tamponunda 5 μM'ye kadar seyreltin. DNA çözeltisine, Abraham Punnoose ve ark.13'e göre sentezlenen ligand L2H2-4OTD'yi 0-60 μM'lik bir konsantrasyon aralığında ekleyin.
- Floresan mikroskobu kullanarak deiyonize DI suyuyla doldurulan reaksiyon odasının arka plan floresan yoğunluğunu kesmeden kontrol edin ve en aza indirin. Arka plan floresansını en aza indirmenin kolay bir yolu, reaksiyon odasını DI suyuyla yıkamaktır. Arka plan floresan değerinin daha sonra veri analizlerinde çıkarılması gerekir.
NOT: Bu adımdan itibaren başıboş ışıktan kaçınılmalıdır. - Yazılımı kullanarak CCD kameranın parametrelerini ayarlayın. Önerilen parametreler aşağıdaki gibidir: pozlama süresi = 0,5 sn, CCD duyarlılığı = 1600 ve kayıt süresi = 20 dakika.
- Uzun bir pipet kullanarak, DNA çözeltisini boş ve temiz reaksiyon odasına ekleyin. Reaksiyon odasını kara bir kutu ile örtün. Ardından, verileri kaydetmek için CCD kamera açıkken 20 dakika boyunca 9.724 s−1 ila 97.245 s−1 (homojenizatörle ilişkili yazılım kullanılarak seçilmiştir) arasında değişen seçilmiş bir kesme hızında kesme gerçekleştirmek için homojenizatörü çalıştırın.
- Deneyden sonra, odayı çıkarın ve DI suyuyla yıkayın.
3. Kesme kuvveti ile çalıştırılan tıklama reaksiyonu
- DI suyunda i-motif DNA'sı hazırlayın. 10 μM CuCl ve 300 μM askorbik asit ile desteklenmiş 300 μM Tris tamponunda (pH 7.4) 10 μM i-motif DNA'sını 10 dakika boyunca inkübe edin (tüm konsantrasyonlar çözeltideki son konsantrasyonlardır).
NOT: CuCl, tıklama reaksiyonunun katalizörüdür. Askorbik asit, bakır (I) oksidasyonunu önleyecektir. - Çözeltiyi, 14.300 x g'lık bir santrifüj kuvvetinde bir ultrafiltrasyon cihazı ile ultra filtreleyin. Her filtrasyondan sonra çözeltiyi ~500 μL'ye kadar 300 μM askorbik asit ile desteklenmiş 30 mM Tris tamponu (pH 7.4) ile doldurun.
- Filtrelemeyi 3x tekrarlayın.
- Artık çözeltiyi toplayın ve 20 μM Calfluor 488 azid, 20 μM HPG ve 10 μM TBTA ile birlikte 300 μM askorbik asit ile desteklenmiş 30 mM Tris (pH 7.4) ekleyerek 300 μL'lik bir son hacim oluşturun. Reaktifler eklendikten sonra, çözeltiyi karanlık odaya taşıyın.
NOT: Bu adımdan sonra ışıktan kaçınılmalıdır. - Kesme deneylerinden önce mikroskop kullanarak DI suyuyla doldurulmuş reaksiyon odasının arka plan floresan yoğunluğunu kontrol edin ve en aza indirin. Arka plan floresansını en aza indirmenin kolay bir yolu, reaksiyon odasını DI suyuyla yıkamaktır.
- DNA çözeltisini uzun bir pipetle boş reaksiyon odasına ekleyin ve ardından homojenizatör kesmeyi CCD kamera açıkken 20 dakika boyunca 63.209 s−1 kesme hızında başlatın.
- Deneyden sonra, odayı çıkarın ve DI suyuyla yıkayın.
Sonuçlar
Şekil 1, EFS'deki topluluk moleküllerinin mekanik açılımını ve gerçek zamanlı algılanmasını özetlemektedir. Şekil 1B'de, i-motif DNA'nın floresan yoğunluğunun, pH 5.5 MES tamponunda 9.724 s-1 ila 97.245 s-1 arasında değişen kesme hızı ile arttığı gözlenmiştir. Bir kontrol olarak, aynı i-motif DNA'sı pH 7.4 MES tamponunda 63.209 s-1 oranında kesildiğinde floresan yoğunluğu artmadı...
Tartışmalar
Bu makalede açıklanan protokol, bir grup biyomoleküler yapının kesme kuvveti ile açılmasının gerçek zamanlı olarak araştırılmasına izin verir. Burada sunulan sonuçlar, DNA i-motif yapılarının kesme kuvveti ile açılabileceğinin altını çizmektedir. Ligand'a bağlı i-motifin açılması ve kesme kuvveti ile harekete geçirilmiş tıklama reaksiyonları, bu topluluk kuvveti spektroskopisi yöntemi için kavram kanıtı uygulamalarıydı.
Şekil 1'de
Açıklamalar
Yazarların çıkar çatışması yoktur.
Teşekkürler
Bu araştırma çalışması, Ulusal Bilim Vakfı [CBET-1904921] ve Ulusal Sağlık Enstitüleri [NIH R01CA236350] tarafından H. M. tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 3K MWCO Amicon | Millipore Sigma | ufc900324 | |
| Ascorbic acid | VWR | VWRC0143-100G | |
| Calfluor 488 azide | Click Chemistry Tools | 1369-1 | |
| CuCl | Thermo | ACRO270525000 | |
| Dispersion tip | Switzerland | PT-DA07/2EC-B101 | |
| DNA oligos | IDT | ||
| Dye | IDT | /5Cy5/ | |
| Fluorescence microscope | Janpan | Nikon TE2000-U | |
| Homogenizer | Switzerland | PT 3100D | |
| HPG | Santa Cruz Biotechnology | cs-295271 | |
| KCl | VWR | VWRC26760.295 | |
| MES | VWR | VWRCE169-500G | |
| Quencher | IDT | /3IAbRQSp/ | |
| TBTA | Tokyo Chemical Industry | T2993 | |
| Tris | VWR | VWRCE133-100G |
Referanslar
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: Optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences of the United States of America. 103 (16), 6190-6195 (2006).
- Grandbois, M., Beyer, M., Rief, M., Clausen-Schaumann, H., Gaub, H. E. How strong is a covalent bond. Science. 283 (5408), 1727-1730 (1999).
- Strick, T. R., Allemand, J. F., Bensimon, D., Croquette, V. Behavior of supercoiled DNA. Biophysical Journal. 74 (4), 2016-2028 (1998).
- Yang, D., Ward, A., Halvorsen, K., Wong, W. P. Multiplexed single-molecule force spectroscopy using a centrifuge. Nature Communications. 7, 11026 (2016).
- Su, H., et al. Light-responsive polymer particles as force clamps for the mechanical unfolding of target molecules. Nano Letters. 18 (4), 2630-2636 (2018).
- Kirkness, M. W. H., Forde, N. R. Single-molecule assay for proteolytic susceptibility: Force-induced collagen destabilization. Biophysical Journal. 114 (3), 570-576 (2018).
- Astumian, R. D. Thermodynamics and kinetics of molecular motors. Biophysical Journal. 98 (11), 2401-2409 (2010).
- Bekard, I. B., Asimakis, P., Bertolini, J., Dunstan, D. E. The effects of shear flow on protein structure and function. Biopolymers. 95 (11), 733-745 (2011).
- Chistiakov, D. A., Orekhov, A. N., Bobryshev, Y. V. Effects of shear stress on endothelial cells: go with the flow. Acta Physiologica. 219 (2), 382-408 (2017).
- Hu, C., Jonchhe, S., Pokhrel, P., Karna, D., Mao, H. Mechanical unfolding of ensemble biomolecular structures by shear force. Chemical Science. 12 (30), 10159-10164 (2021).
- Sedghi Masoud, S., et al. Analysis of interactions between telomeric i-motif DNA and a cyclic tetraoxazole compound. ChemBioChem. 19 (21), 2268-2272 (2018).
- Abraham Punnoose, J., et al. Adaptive and specific recognition of telomeric G-quadruplexes via polyvalency induced unstacking of binding units. Journal of the American Chemical Society. 139 (22), 7476-7484 (2017).
- Dhakal, S., et al. Coexistence of an ILPR i-motif and a partially folded structure with comparable mechanical stability revealed at the single-molecule level. Journal of the American Chemical Society. 132 (26), 8991-8997 (2010).
- Hu, C., Tahir, R., Mao, H. Single-molecule mechanochemical sensing. Accounts of Chemical Research. 55 (9), 1214-1225 (2022).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır