Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Protein Korunumunun Hızlı Değerlendirilmesi için Türler Arasında Tahmin Etmek İçin Dizi Hizalamasının Gösterilmesi Duyarlılık Aracı
Bu Makalede
Özet
Burada, ABD Çevre Koruma Ajansı Türler Arası Duyarlılığı Tahmin Etmek için Dizi Hizalama (SeqAPASS) aracının en son sürümünü kullanmak için bir protokol sunuyoruz. Bu protokol, protein korumasını hızlı bir şekilde analiz etmek ve türler arasında kimyasal duyarlılığın özelleştirilebilir ve kolayca yorumlanabilir tahminlerini sağlamak için çevrimiçi aracın uygulanmasını göstermektedir.
Özet
ABD Çevre Koruma Ajansı Türler Arası Duyarlılığı Tahmin Etmek için Dizi Hizalama (SeqAPASS) aracı, araştırmacıların ve düzenleyicilerin türler arasında toksisite bilgilerini tahmin etmelerini sağlayan hızlı, serbestçe kullanılabilir, çevrimiçi bir tarama uygulamasıdır. İnsan hücreleri, fareler, sıçanlar ve zebra balığı gibi model sistemlerdeki biyolojik hedefler için, çeşitli kimyasallar için toksisite verileri mevcuttur. Protein hedef korunumunun değerlendirilmesi yoluyla, bu araç, bu tür model sistemlerinden üretilen verileri, toksisite verilerinden yoksun binlerce başka türe tahmin etmek için kullanılabilir ve göreceli içsel kimyasal duyarlılık tahminleri verir. Aracın en son sürümleri (sürüm 2.0-6.1), yayın için verilerin hızlı sentezlenmesine, yorumlanmasına ve kullanılmasına ve ayrıca sunum kalitesinde grafiklere izin veren yeni özellikler içermektedir.
Bu özellikler arasında özelleştirilebilir veri görselleştirmeleri ve yorumlama kolaylığı için SeqAPASS verilerini özetlemek üzere tasarlanmış kapsamlı bir özet rapor bulunmaktadır. Bu makalede, kullanıcılara iş gönderme, çeşitli protein dizisi karşılaştırmaları düzeylerinde gezinme ve elde edilen verileri yorumlama ve görüntüleme konusunda rehberlik eden protokol açıklanmaktadır. SeqAPASS v2.0-6.0'ın yeni özellikleri vurgulanmıştır. Ayrıca, bu aracı kullanarak transtiretin ve opioid reseptör protein korunumuna odaklanan iki kullanım durumu tanımlanmıştır. Son olarak, SeqAPASS'ın güçlü yönleri ve sınırlamaları, alet için uygulanabilirlik alanını tanımlamak ve türler arası ekstrapolasyon için farklı uygulamaları vurgulamak için tartışılmaktadır.
Giriş
Geleneksel olarak, toksikoloji alanı, kimyasal güvenlik değerlendirmeleri için gerekli verileri sağlamak için tüm hayvan testlerinin kullanımına büyük ölçüde güvenmiştir. Bu tür yöntemler genellikle maliyetli ve kaynak yoğundur. Bununla birlikte, şu anda kullanılan çok sayıda kimyasal madde ve yeni kimyasalların geliştirilme hızından dolayı, küresel olarak kimyasal tarama için daha verimli yöntemlere ihtiyaç duyulmaktadır 1,2. Bu ihtiyaç ve bunun sonucunda hayvan testlerinden uzaklaşan paradigma kayması, yüksek verimli tarama testleri, yüksek verimli transkriptomikler, yeni nesil dizileme ve alternatif test stratejileri vaat eden hesaplamalı modelleme dahil olmak üzere birçok yeni yaklaşım yönteminin geliştirilmesine yol açmıştır 3,4.
Kimyasal maruziyetlerden potansiyel olarak etkilenen türlerin çeşitliliği genelinde kimyasal güvenliğin değerlendirilmesi, sadece geleneksel toksisite testleriyle değil, aynı zamanda yeni yaklaşım yöntemleriyle de kalıcı bir zorluk olmuştur. Karşılaştırmalı ve öngörücü toksikolojideki ilerlemeler, farklı türlerin göreceli duyarlılığını anlamak için çerçeveler sağlamıştır ve hesaplama yöntemlerindeki teknolojik gelişmeler bu yöntemlerin uygulanabilirliğini arttırmaya devam etmektedir. Son on yılda, türler arası ekstrapolasyon için öngörücü yaklaşımları desteklemek ve tipik model organizmaların 5,6,7,8'in ötesinde kimyasal güvenlik değerlendirmelerini geliştirmek için mevcut gen ve protein dizisi veritabanlarından ve belirli kimyasal moleküler hedeflerin bilgisinden yararlanan çeşitli stratejiler tartışılmıştır.
Bilimi eyleme geçirmek, öngörücü toksikolojideki bu temel çalışmalara dayanmak, kimyasal test çabalarına öncelik vermek ve karar vermeyi desteklemek için, ABD Çevre Koruma Ajansı Türler Arası Duyarlılığı Tahmin Etmek için Dizi Hizalama (SeqAPASS) aracı oluşturuldu. Bu araç,9. türlerin çeşitliliği boyunca kimyasal duyarlılığı tahmin etmek için sürekli genişleyen protein dizisi bilgilerinin genel depolarını kullanan halka açık ve serbestçe kullanılabilen web tabanlı bir uygulamadır. Bir türün belirli bir kimyasala karşı göreceli içsel duyarlılığının, o kimyasalın bilinen protein hedeflerinin korunumunu değerlendirerek belirlenebileceği ilkesine dayanarak, bu araç, bilinen hassasiyeti olan bir türün protein amino asit dizilerini, mevcut protein dizisi verileriyle tüm türlere karşı hızla karşılaştırır. Bu değerlendirme, (1) birincil amino asit dizisi, (2) fonksiyonel alan ve (3) kritik amino asit kalıntısı karşılaştırmaları dahil olmak üzere, her biri kimyasal-protein etkileşimi hakkında daha derinlemesine bilgi gerektiren ve duyarlılık tahmininde daha fazla taksonomik çözünürlük sağlayan üç analiz seviyesi ile tamamlanır. SeqAPAS'ın önemli bir gücü, kullanıcıların, ilgilendikleri kimyasal-protein veya protein-protein etkileşimi ile ilgili ne kadar bilgi mevcut olduğuna bağlı olarak hedef korumaya yönelik ek kanıt satırları ekleyerek değerlendirmelerini özelleştirebilmeleri ve rafine edebilmeleridir.
İlk versiyon 2016 yılında piyasaya sürüldü ve bu da kullanıcıların birincil amino asit dizilerini ve fonksiyonel etki alanlarını kimyasal duyarlılığı tahmin etmek için akıcı bir şekilde değerlendirmelerine izin verdi ve minimum veri görselleştirme yetenekleri içeriyordu (Tablo 1). Bireysel amino asit farklılıklarının, türlerin kimyasal duyarlılığını etkileyebilecek kimyasal-protein etkileşimlerinde türler arası farklılıkların önemli belirleyicileri olduğu gösterilmiştir10,11,12. Bu nedenle, doğrudan kimyasal etkileşim için önemli olan kritik amino asitleri dikkate almak için sonraki versiyonlar geliştirilmiştir13. Paydaş ve kullanıcı geri bildirimlerine yanıt veren bu araç, türler arası ekstrapolasyondaki zorlukları ele almak için hem araştırmacıların hem de düzenleyici toplulukların ihtiyaçlarını karşılamak üzere tasarlanmış ek yeni özelliklerle yıllık sürüm sürümlerine tabi tutulmuştur (Tablo 1). 2020'de SeqAPASS sürüm 5.0'ın piyasaya sürülmesi, veri görselleştirme ve veri sentezi seçeneklerini, harici bağlantıları, özet tablo ve rapor seçeneklerini ve grafik özelliklerini içeren kullanıcı merkezli özellikler ortaya çıkardı. Genel olarak, bu sürümün yeni nitelikleri ve yetenekleri veri sentezini, harici veritabanları arasında birlikte çalışabilirliği ve türler arası duyarlılık tahminleri için veri yorumlama kolaylığını geliştirdi.
Access restricted. Please log in or start a trial to view this content.
Protokol
1. Başlarken
NOT: Burada sunulan protokol, araç yardımcı programına ve temel özelliklere odaklanmıştır. Yöntemlerin, özelliklerin ve bileşenlerin ayrıntılı açıklamaları web sitesinde kapsamlı bir Kullanım Kılavuzunda bulunabilir (Tablo 1).
Tablo 1: SeqAPASS aracının evrimi. İlk dağıtımından itibaren SeqAPASS aracına eklenen özelliklerin ve güncelleştirmelerin listesi. Kısaltmalar: SeqAPASS = türler arası duyarlılığı tahmin etmek için dizi hizalaması; ECOTOX = ECOTOXicology bilgi bankası. Bu tabloyu indirmek için lütfen tıklayınız.
- Chrome'u kullanarak https://seqapass.epa.gov/seqapass'a gidin. Mevcut bir hesabı kullanmak için Oturum Aç'ı seçin veya kullanıcıların tamamladıkları işleri çalıştırmalarına, depolamalarına, erişmelerine ve özelleştirmelerine olanak tanıyan bir SeqAPASS hesabı oluşturmak için talimatları izleyin.
- Bir analiz yapmadan önce, mevcut literatürü veya önceden var olan verileri gözden geçirerek ilgilenilen bir proteini ve hedeflenen veya hassas bir türü tanımlayın (Şekil 1). SeqAPASS, sorgu proteinini tanımlamaya yardımcı olmak için dış kaynaklara bağlantılar içerdiğinden, ilgili kaynaklara erişmek için Bir Protein Hedefi Tanımla altındaki açılır düğmelere tıklayın.
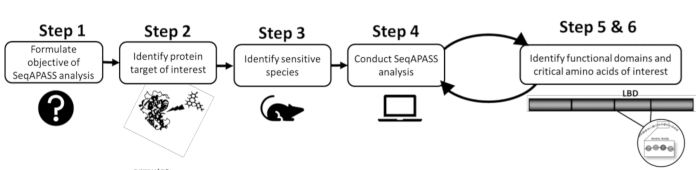
Şekil 1: SeqAPASS problem formülasyonu: Başarılı bir analiz için gerekli ön bilgilerin şematik diyagramı. Kısaltmalar: SeqAPASS = türler arası duyarlılığı tahmin etmek için dizi hizalaması; LBD = ligand bağlama etki alanı. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 2: Veritabanları arasında SeqAPASS birlikte çalışabilirliği. SeqAPASS'a entegre edilmiş harici araçların, veritabanlarının ve kaynakların şematik diyagramı. Kısaltmalar: SeqAPASS = türler arası duyarlılığı tahmin etmek için dizi hizalaması; AOP = advers sonuç yolu; NCBI = Ulusal Biyoteknoloji Bilgi Merkezi; ECOTOX = ECOTOXicology bilgi bankası. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Tablo 2: SeqAPASS aracına entegre bağlantılar, kaynaklar ve araçlar. SeqAPASS aracında kullanılan çeşitli veri kaynaklarının, bağlantıların ve kaynakların listesi. Kısaltma: SeqAPASS = türler arası duyarlılığı tahmin etmek için dizi hizalaması. Bu tabloyu indirmek için lütfen tıklayınız.
2. SeqAPASS sorgusu geliştirme ve çalıştırma: Düzey 1
NOT: Seviye 1 analizinde, bir sorgu proteininin tüm birincil amino asit dizisi, mevcut dizi bilgisine sahip tüm türlerin birincil amino asit dizileriyle karşılaştırılır. Bu araç, türler arasındaki amino asit dizilerini hızlı bir şekilde hizalamak ve karşılaştırmak için kamuya açık verileri araştırmak, toplamak ve derlemek için algoritmalar kullanır. Arka uç, Ulusal Biyoteknoloji Bilgi Merkezi (NCBI) veritabanlarından bilgi depolar ve stratejik olarak Protein Temel Yerel Hizalama Arama Aracı (BLASTp)54 ve Kısıtlama Tabanlı Çoklu Hizalama Aracı'nın (COBALT) bağımsız sürümlerini kullanır.
- Birincil Amino Asit Dizilerini Karşılaştır altında, Türlere Göre veya Katılıma Göre'yi tıklatın. İlgilenilen protein hedefini seçmek üzere türler listesinden yazmak veya seçmek için Türlere Göre seçimini kullanın.
- Protein katılımlarını (yani, NCBI protein kimliğini) doğrudan Katılıma Göre metin kutusuna katılım(lar)ı girerek gönderin.
- Sorguyu göndermek için İstek Çalıştırma'yı seçin. Gönderildikten sonra, tarayıcı penceresinin sağ üst köşesinde başarılı bir gönderim olduğunu belirten bir bildirim görünmesini bekleyin.
- Bu kullanıcı hesabı altında gerçekleştirilen tüm SeqAPASS çalıştırmalarının bir listesini görüntülemek ve tamamlanma yüzdesini kontrol etmek için sayfanın üst kısmındaki SeqAPASS Çalıştırma Durumu sekmesini seçin.
- Düzey 2 ve Düzey 3 çalıştırmalarının durumunu denetlemek için uygun radyo düğmesi seçiliyken Verileri Yenile'yi tıklatın.
- Söz konusu hesap altında tamamlanan tüm raporların listesine erişmek için sayfanın üst kısmındaki SeqAPASS Raporlarını Görüntüle sekmesini seçin.
- SeqAPASS Raporlarını Görüntüle sekmesinde, ilgilendiğiniz sorgu proteinini seçin. Düzey 1 Sorgu Protein Bilgileri sayfasını açmak ve sonuçları, veri özelleştirme seçeneklerini, görselleştirmeleri ve özet raporları görüntülemek için Seçili Raporu İste'ye tıklayın.
- Varsayılan olarak, verileri web tarayıcısında görüntülemek için Raporu Görüntüle'yi seçin. Alternatif olarak, ham verileri .zip dosyası olarak indirmek için Raporu Kaydet'i seçin.
NOT: Düzey 1 analizi için gereken süre, o andaki genel kullanıcı talebine, kuyruğa gönderilen işlerin sayısına ve gönderilen bir iş için var olan protein bilgilerinin miktarına bağlı olarak değişir (sürüm 5.1 için ortalama 23 dakika). Bir protein hedefi daha önce tamamlanmışsa, veriler gönderildikten sonra saniyeler içinde hazır olacaktır.
3. SeqAPASS sorgusu geliştirme ve çalıştırma: Düzey 2
NOT: Tüm protein dizisi doğrudan kimyasal bir etkileşime dahil olmadığından, Seviye 2 analizi, daha düşük taksonomik sıralarda (örneğin, sınıf, düzen, aile) duyarlılık tahminleri yapmak için yalnızca fonksiyonel alanın amino asit dizisini karşılaştırır.
- Düzey 1 Sorgu Protein Bilgileri sayfasından, Düzey 2 Sorgu menüsünü doldurmak için Düzey 2 üstbilgisinin yanındaki artı işaretini + işaretini tıklatın.
- İlgilenilen proteindeki (sorgu proteini) uygun etki alanlarını tanımlayın.
- Bir etki alanı tanımlanmamışsa, uygun etki alanı seçiminin tanımlanmasına yardımcı olabilecek NCBI Korunmuş Etki Alanları Veritabanı'na (CDD) (Tablo 1) tümleşik bağlantıyı tıklatın.
NOT: Genellikle, Düzey 2'de sorgu olarak yalnızca belirli isabet etki alanları seçilir.
- Bir etki alanı tanımlanmamışsa, uygun etki alanı seçiminin tanımlanmasına yardımcı olabilecek NCBI Korunmuş Etki Alanları Veritabanı'na (CDD) (Tablo 1) tümleşik bağlantıyı tıklatın.
- Sorgu proteininin işlevsel etki alanlarının listesini otomatik olarak doldurmak için Etki Alanı Seç kutusunu tıklatın.
- Açılır listeden etki alanı katılımlarını seçin ve Etki Alanı Çalıştırma İste düğmesini tıklatarak Düzey 2 sorgusunu başlatın. Gönderildikten sonra, gönderimin başarılı olduğunu belirten bir bildirimin görünmesini bekleyin.
- Düzey 2'yi yenile'yi tıklatın ve gönderimden sonraki saniyeler içinde kullanıma sunulacak olan Düzey 2 verilerini doldurmak için 3 çalışır.
- Düzey 2 Verilerini Görüntüle altında, açılır listeden tamamlanan etki alanı katılımlarını seçin ve sonuçları yeni bir sayfada açmak için Düzey 2 Verilerini Görüntüle düğmesini tıklatın.
4. Verilere erişme ve verileri anlama: SeqAPASS Seviye 1 ve Seviye 2
- Sonuçların raporunu görüntülemek için Sorgu Protein Bilgileri sayfasının en altına kaydırın - varsayılan olarak Düzey 1 ve 2 analizleriyle birlikte bir Birincil Rapor sağlanır. Tüm dizi isabetlerini ve hizalama metriklerini sağlayan daha ayrıntılı bir rapor görüntülemek için Tam rapor radyo düğmesini seçin. NCBI veritabanındaki şeffaf protein hizalaması ve taksonomi bilgilerine erişmek için her iki raporda da uygun erişim/kimlik/ada tıklayın.
- ECOTOX sütununu görüntülemek için sonuçlar tablosunun sağ tarafına kaydırın. Duyarlılık tahminleri olan türler için karşılık gelen toksisite verilerini hızla toplamak üzere ECOTOXicology bilgi bankası (ECOTOX) bağlantılarına tıklayın.
NOT: ECOTOX, sucul ve karasal bitkiler ve vahşi yaşam için tek kimyasal toksisite verileri sağlayan kapsamlı, halka açık bir bilgi tabanıdır. SeqAPASS v6.0, ilgili ECOTOX verileriyle kimyasal ve ilgili türlere göre daha hızlı bağlantı kurmak için bir ECOTOX widget'ı içerir. - Tabloyu e-tablo dosyası olarak kaydetmek için Tabloyu İndir'i tıklayın. Taksonomik gruba göre sıralanmış verileri sunan bir özet rapor tablosunu görüntülemek ve indirmek için Özet Raporu Görüntüle düğmesini tıklatın.
NOT: Veri özeti tabloları hem Birincil hem de Tam Raporlar için kullanılabilir ve belirli bir hedef için tahminlere genel bir bakış sağlar.
5. Veri ayarlarını manipüle etme: SeqAPASS Seviye 1 ve Seviye 2
NOT: Hem Seviye 1 hem de Seviye 2 analizlerinde, protein benzerliği ne kadar büyük olursa, bir kimyasalın proteinle sorgu türüne/proteine benzer şekilde etkileşime girme olasılığının o kadar yüksek olduğu ve bu moleküler hedefe sahip kimyasalların potansiyel etkilerine duyarlı hale geldiği varsayılmaktadır. Bu verilerin benzerliği nedeniyle, Düzey 1 ve 2 verilerini anlama adımları tek bir protokolde birlikte özetlenmiştir.
- Rapor ayarlarına erişmek ve bunları değiştirmek için Sorgu Protein Bilgileri'nin üst kısmındaki alt menülere bakın ve çoğu çözümlemede tüm rapor seçenekleri için varsayılan ayarları kullanın. Varsayılan ayarı değiştirmek için bilimsel gerekçe varsa, aşağıdaki isteğe bağlı adımları izleyin:
- (İSTEĞE BAĞLI) Duyarlılık kesme ayarlarını yeni bir sekmede görüntülemek ve ayarlamak için Duyarlılık Kesme'nin yanındaki + işaretini tıklatın. Açılır listeden yeni bir kesme değeri seçin veya kullanıcı tanımlı bir kesme değeri girin.
- (İSTEĞE BAĞLI) Varsayılan değerden başka bir şey isteniyorsa , E-Değer (tesadüfen gerçekleşmesi beklenen farklı hizalamaların sayısı) alanındaki sayıyı değiştirin.
NOT: E değeri kutudaki sayıdan büyük olan proteinler birincil rapordan elenecektir. - (İSTEĞE BAĞLI) Sonuç tablosundaki Filtrelenmiş Taksonomik Grup sütununda görüntülenecek taksonomik hiyerarşi düzeyini seçmek için Taksonomik Gruba Göre Sırala seçeneğini kullanın.
NOT: Taksonomik hiyerarşinin değiştirilmesi, kesimin üzerinde bulunan her filtrelenmiş gruptan türlere dayalı duyarlılık tahminini de değiştirecektir. - (İSTEĞE BAĞLI) Varsayılandan başka bir şey isteniyorsa, Ortak Etki Alanı (bir proteinin sonuçlara dahil edilmek üzere sorgu proteiniyle kaç ortak etki alanını paylaşması gerektiği) alanını değiştirin.
NOT: Varsayılan ayar 1 olduğundan, sorgu proteiniyle en az bir ortak etki alanını paylaşmayan tüm diziler dışlanır. - (İSTEĞE BAĞLI) Yalnızca Yüzde Benzerlik kesimden büyük veya ona eşitse ya da isabet ortolog adayı olarak tanımlanırsa Y'nin duyarlılık tahminlerini geri almak için Türler Boyunca Okunan Türler altında Hayır'ı seçin.
NOT: Bu ayar varsayılan olarak Evet olarak ayarlanır, yani tüm ortolog adayları, duyarlılık kesiminin üstünde listelenen tüm türler ve kesimin üstünde bir veya daha fazla tür bulunan aynı taksonomik gruptan kesimin altındaki tüm türler için Y'nin duyarlılık tahmini raporlanacaktır.
- Geçerli ayarları yakalayan bir dosyayı karşıdan yüklemek için Geçerli Rapor Ayarlarını İndir düğmesini tıklatın.
NOT: Seçilen belirli değerlendirme Düzeyi (1, 2 veya 3), raporda sunulan ayarları belirler.
6. Verileri görselleştirme: SeqAPASS Seviye 1 ve Seviye 2
- Görselleştirme'nin yanındaki artı işaretini + işaretini tıklatın ve kullanıcı tanımlı bilgileri ve etkileşimli bir sonuç grafiği seçme seçeneğini görüntüleyen ayrı bir sekme açmak için Verileri Görselleştir düğmesini tıklatın.
- Etkileşimli boxplot ve plot denetimlerini açmak ve boxplot görselleştirmesinin veri tablosundaki değişiklikleri yansıtacak ve yayın ve sunum kalitesinde grafikler sağlayacak şekilde etkin bir şekilde güncelleştirilmesine izin vermek için Boxplot'u tıklatın.
NOT: Varsayılan kutu grafiği, x ekseninde tür gruplarını ve y ekseninde yüzde benzerliğini görüntüler. Boxplots, duyarlılık kesimini (kesikli çizgi), sorgu türlerine kıyasla türler arasındaki yüzde benzerliği ve her bir taksonomik grup için ortalama ve medyan değerleri, 25. ve 75. yüzdelik dilimler ve çeyrekler arası aralıkla birlikte gösterir. Analizin amacına ve kullanıcının ihtiyaçlarına bağlı olarak, birçok boxplot özelliği aşağıdaki isteğe bağlı adımlarla değiştirilebilir.- (İSTEĞE BAĞLI) Görüntülenen taksonomik grupları özelleştirmek için, Denetimler bölümünün altındaki Taksonomik Gruplar kutusuna bakın. Adların üzerine gidip x'i seçerek veya Taksonomik Gruplar açılır menüsünü kullanarak grupları kaldırın.
- (İSTEĞE BAĞLI) İlgilenilen bir türü veya önceden tanımlanmış belirli grupları (örneğin, nesli tükenmekte olan veya tehdit altındaki türler) belirleyecek bir efsane eklemek için, en yüksek yüzde benzerliğine göre sıralanmış ilk üç türü listeleyen bir açılır kutuyu etkinleştirmek için imleci x eksenindeki taksonomik bir grup adının üzerine getirin. İlgili tür bilgilerini içeren bir açılır kutu oluşturmak için göstergedeki türlerin üzerine gelin. Türleri ve tahminleri listeleyen indirilebilir bir özet tablo oluşturmak için belirli bir taksonomik grubun kutusuna tıklayın.
- Bir dosya türü seçmek, genişlik/yükseklik çözünürlüğünü özelleştirmek ve görselleştirmeyi kaydetmek için Boxplot'u İndir'e tıklayın.
7. Bir SeqAPASS analizi geliştirmek ve yürütmek: Seviye 3
NOT: Bir Seviye 3 analizi, sorgu proteini içindeki kullanıcı tarafından tanımlanan amino asit kalıntılarını değerlendirir ve bu kalıntıların türler arasında korunmasını hızla karşılaştırır. Bu kalıntıların korunduğu türlerin, şablon türlere / proteine benzer şekilde bir kimyasalla etkileşime girme olasılığının daha yüksek olduğu varsayılmaktadır. Seviye 3 bireysel amino asitlere odaklandığından, bir analiz ancak kimyasal-protein veya protein-protein etkileşimi için kritik olan amino asit kalıntılarının ayrıntılı bilgisi mevcut olduğunda gerçekleştirilebilir.
- Düzey 3 Sorgu menüsünü doldurmak için Düzey 1 Sorgu Protein Bilgileri sayfasındaki Düzey 3 üstbilgisinin yanındaki artı işaretini + işaretini tıklatın.
- Mevcut literatürü sorgulamak için önceden tanımlanmış bir Boole dizesi oluşturan ve kullanıcıların Düzey 3 değerlendirmesinde kullanılacak kritik amino asitlerin tanımlanmasını desteklemek için uygun literatürü belirlemelerine yardımcı olan başvuru gezgini aracını açmak için Referans Gezgini'nin yanındaki artı işaretini + işaretini tıklatın (Tablo 2 ve Şekil 2).
- (İSTEĞE BAĞLI) Sorgu proteini otomatik olarak doldurulduktan sonra, ek proteinler eklemek için Protein Adı Ekle işlevini kullanın.
- Alakalı arama terimlerini içeren otomatik olarak oluşturulmuş bir arama dizesi içeren bir açılır pencereyi açmak için Google Akademik Oluştur Bağlantısını tıklayın.
- Arama dizesini kullanarak literatür veritabanını sorgulamak için Google Akademik'te Ara'yı tıklayın.
- Alternatif olarak, Panoya Kopyala'yı tıklatın ve Referans Gezgini'ndeki işlevleri kullanarak terimler ekleyerek veya kaldırarak arama dizesini özelleştirin.
8. Tanımlanmış literatürü kullanarak kritik amino asit kalıntılarını tanımlayın
- Kullanıcı tarafından seçilen türlerin hizalanacağı şablon sırasını Düzey 3 Sorgu menüsünde seçin.
NOT: Bu şablon dizisi genellikle kritik amino asitlerin tanımlandığı literatüre dayanarak seçilir ve Seviye 1 ve Seviye 2'de sorgulananlarla aynı veya farklı bir tür olabilir.- (İSTEĞE BAĞLI) Birincil/Tam Rapor tablolarında görünmeyen katılımları/dizileri karşılaştırmak için Ek Karşılaştırmalar kutusunu kullanın.
- Tamamlanan Düzey 3 çalışmasını tanımlamak için Düzey 3 Çalıştırma Adı girin metin kutusuna Düzey 3 çalıştırması için kullanıcı tanımlı bir ad girin. Her değerlendirme için benzersiz bir ad seçin.
- Taksonomik Grubu Seç alanında ilgilenilen taksonomik grubu seçin . Tabloyu o taksonomik gruba göre otomatik olarak filtrelemek için bir taksonomik grup seçin.
- Sonuç tablosunda, şablon dizisine hizalanacak herhangi bir türün yanındaki onay kutusunu manuel olarak tıklatın.
NOT: Uygun hizalamayı sağlamak için, şablonla her seferinde bir taksonomik grup karşılaştırılmalıdır. İlgilenilen türler için sadece benzer şekilde açıklamalı proteinleri seçin. Karşılaştırma için dizileri seçerken, belirli dizilere dikkat etmek önemlidir (örneğin, varsayımsal, DÜŞÜK KALİTE veya kısmi). Dahil edilmek için şeffaf bir gerekçe olmadığı sürece, eksik veya uygunsuz dizi bilgileri nedeniyle tahminleri çarpıtabilecekleri için bu dizileri hariç tutmak en iyisidir. - Tüm taksonomik ilgi gruplarını hizalamak için adımları yineleyin.
- Düzey 3 Çalıştırma Adı Seç menüsünü tamamlanan Düzey 3 işleriyle doldurmak ve verileri hemen bir Düzey 3 hizalamasından almak için tüm türler hizalandıktan sonra Düzey 2'yi yenile'yi tıklatın ve 3 çalışır.
- Birden çok taksonomik gruptaki hizalamaları birleştirmek için Düzey 3 Verilerini Birleştir'i tıklatın.
- Alternatif olarak, tek bir raporu görüntülemek için, Görüntülenecek Sorguyu Seç altında kullanıcı tanımlı adı seçin ve Düzey 3 Verilerini Görüntüle'yi tıklatın.
- Düzey 3 Raporlarını Birleştir menüsünde amino asit kalıntısı karşılaştırması için temel olarak kullanılacak Düzey 3 şablonunu seçin ve İleri'yi tıklatın.
- Düzey 3 işleri'nde, karşılaştırma için tamamlanan işleri seçin ve İleri'ye tıklayın. İsterseniz taksonomik grupları yeniden sıralamak için Sipariş Düzeyi 3 İşler işlevini kullanın. Birleşik taksonomik grupların hizalandığı bir Düzey 3 rapor sayfası oluşturmak için Düzey 3 Verilerini Görüntüle'yi tıklatın.
- Amino Asit Kalıntı Konumlarını Gir kutusuna virgülle ayrılmış amino asit konumlarını yazıp ardından Kalıntı Listesine Kopyala'yı seçerek şablon türleri için önceden tanımlanmış amino asit konumlarını seçin. Mekik kutusundan şablon dizisindeki kalıntıları doğrudan seçin.
- Sayfayı yenilemek ve Düzey 3 duyarlılık tahminlerini görüntülemek için Raporu Güncelleştir'i tıklatın.
NOT: Seviye 3, anahtar pozisyonlardaki farklılıkların protein etkileşimlerini etkileyip etkilemeyeceğini belirlemek için yan zincir fonksiyonel özelliklerinin (örneğin, alifatik, aromatik) ve moleküler boyutların (moleküler ağırlık farklılıkları >30 g / mol) temel tanımlayıcılarından türetilen basit bir kurallar kümesi kullanır13.
9. Seviye 3 SeqAPASS verilerini görselleştirme
NOT: Önceki Seviyelerde olduğu gibi, Birincil ve Tam raporlar kullanılabilir. Birincil Rapor, Düzey 1 ve 2'deki verilerle aynı verilere ek olarak, amino asit konumlarını, kısaltmaları ve şablon tahminiyle benzer bir evet/hayır (Y/N) duyarlılığını görüntüler. Benzer şekilde, Tam Rapor amino asit yan zincir sınıflandırması ve moleküler ağırlık hakkında bilgi içermektedir.
- Düzey 3 rapor sayfasında, sonuçların raporunu görüntülemek için en alta kaydırın. Tabloyu kaydetmek için raporun altındaki Tabloyu İndir'i tıklayın.
- Taksonomik gruba göre sıralanmış verileri sunan bir özet rapor tablosunu görüntülemek ve indirmek için Düzey 3 Özet Raporu Görüntüle'yi tıklatın. Kullanıcı tanımlı bilgileri ve sonuçları etkileşimli bir ısı haritası biçiminde görüntüleme seçeneğini görüntüleyen ayrı bir tarayıcı sekmesi açmak için Düzey 3 Rapor sayfasında Görselleştirme'nin yanındaki artı işaretini + işaretini tıklatın.
- Etkileşimli grafiği ve denetimleri açmak ve ısı haritası görselleştirmesinin veri tablosundaki değişiklikleri yansıtacak şekilde etkin bir şekilde güncellenmesine izin vermek için Görselleştirme Bilgileri sayfasında Isı Haritası'na tıklayın. Isı haritasını özelleştirmek için aşağıdaki isteğe bağlı adımları uygulayın.
- (İSTEĞE BAĞLI) Amino asit konumunu, tek harfli kısaltmayı ve amino asit benzerliğini görüntüleyen Basit Rapor veya seçilen her amino asit hakkında ayrıntılı bilgi görüntüleyen Tam Rapor arasında geçiş yapmak için Rapor Seçenekleri'ni seçin.
- (İSTEĞE BAĞLI) Türlerin Ortak Ad veya Bilimsel Ad ile görüntülenme şeklini değiştirmek için Rapor Seçenekleri'ni seçin.
NOT: Basit Raporda, amino asitler şablon amino aside Toplam Eşleşme (koyu mavi), Kısmi Eşleşme (açık mavi, yalnızca bir kriteri karşılayan ikameler) veya Eşleşme Değil (sarı, her iki kriteri de karşılamayan ikameler) olarak kategorize edilir. Tam Rapor , karşılaştırmaları Toplam Eşleşme (koyu mavi) veya Eşleşme Değil (sarı) olarak görüntüler. - (İSTEĞE BAĞLI) Ortolog Adayları, Tehdit Altındaki Türler, Nesli Tükenmekte Olan Türler veya Ortak Model Organizmalar gibi yararlı bilgileri vurgulamak için İsteğe Bağlı Seçimler'i seçin.
- (İSTEĞE BAĞLI) Sütun, gösterge ve metin ekleme veya kaldırma dahil olmak üzere ek özelleştirme seçeneklerini belirlemek için Isı Haritası Ayarları'nı seçin.
- Bir dosya türü seçmek ve görselleştirmeyi kaydetmek için Boxplot'u İndir'e tıklayın.
10. SeqAPASS Sonuçlarının Yorumlanması: Protein korunumu için kanıt çizgileri
NOT: Yorumlama kolaylığı için bu araç, verileri Düzeyler arasında tümleştirmek üzere tasarlanmış bir Karar Özet Raporu (DS Raporu) içerir. DS Raporu, kullanıcının seçtiği sonuçları (yani veri tablolarını ve/veya görselleştirmeleri) içerir ve aynı anda birden fazla tür için birden fazla Düzeyde duyarlılık tahminlerinin hızlı bir şekilde değerlendirilmesine olanak tanır.
- Sonuçlar veya veri görselleştirme sayfalarından DS Raporuna İtme Düzeyi # 'a tıklayın ve verilerin "iletilmesini" ve DS Report sekmesinin etkin olmasını bekleyin.
NOT: Sonuçlar veya herhangi bir değişiklik DS Raporu'na aktarılmamışsa, DS Raporu'na İtme Düzeyi # seçilene kadar etkin kalır. Bir ayar değiştirilmişse, değişiklikler rapora gönderilene kadar Yeni değişiklikleri göndermek için tıklatın metni görünür. Görselleştirmeler, değerlendirme sırasında herhangi bir zamanda DS Raporu'na gönderilebilir. - DS sayfasına erişmek için istediğiniz zaman DS Raporu sekmesini seçin.
NOT: Düzey 1'de hizalanmış tüm türler için, Nihai Karar Özet Raporu tablosu her analiz için önemli verileri ve duyarlılık tahminlerini içerir. DS tablosundaki bir tür Düzey 3 raporuna dahil edilmemişse ancak Düzey 1 ve/veya Düzey 2 işlerinde bulunmuşsa, tablodaki hücre Düzey 3 duyarlılık tahmini için uygulanamaz (NA) bir atama alır.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
SeqAPASS aracının uygulanmasını göstermek ve yeni özellikleri vurgulamak için, protein korunumunun türler arasında kimyasal duyarlılıkta farklılıklar olduğunu (insan transtiretini) ve hiçbir fark olmadığını (μ opioid reseptörü [MOR]) öngördüğü örnekleri temsil eden iki vaka çalışması tanımlanmıştır. Bu örneklerden ilki, olumsuz sonuç yolakları için uygulanabilirlik alanını tahmin etmek için protein dizisi / yapısal karşılaştırmaları ele alırken (AOP'ler, tanım için
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Toksikolojik açıdan ilgi çekici kimyasallara maruz kalabilecek canlı organizmaların genomik, fenotipik, fizyolojik ve davranışsal çeşitliliğini yakalamak için yeterli türün ampirik olarak test edilmesinin mümkün olmadığı yaygın olarak kabul edilmektedir. SeqAPAS'ın amacı, moleküler düzeyde karşılaştırmalar yoluyla test edilen organizmalardan yüzlerce veya binlerce başka türe kimyasal toksisite verilerinin / bilgisinin ekstrapolasyonuna yardımcı olmak ve bilgilendirmek için mevcut ve süre...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarların açıklayacağı bir çıkar çatışması yoktur.
Teşekkürler
Yazarlar, Dr. Daniel L. Villeneuve'e (U.S. EPA, Center for Computational Toxicology and Exposure) ve Dr. Jon A. Doering'e (Louisiana State University, Çevre Bilimleri Bölümü) makalenin daha önceki bir taslağı hakkında yorum yaptıkları için teşekkür etmektedir. Bu çalışma ABD Çevre Koruma Ajansı tarafından desteklenmiştir. Bu makalede ifade edilen görüşler yazarların görüşleridir ve ABD Çevre Koruma Ajansı'nın görüşlerini veya politikalarını yansıtmak zorunda değildir, ayrıca ticari adların veya ticari ürünlerin belirtilmesi federal hükümet tarafından onaylandığını göstermez.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Spreadsheet program | N/A | N/A | Any program that can be used to view and work with csv files (e.g. Microsoft Excel, OpenOffice Calc, Google Docs) can be used to access data export files. |
| Basic computing setup and internet access | N/A | N/A | SeqAPASS is a free, online tool that can be easily used via an internet connection. No software downloads are required. |
Referanslar
- Krewski, D., et al. Toxicity testing in the 21st century: a vision and a strategy. Journal of Toxicology and Environmental Health, Part B. 13 (2-4), 51-138 (2010).
- Wang, Z., Walker, G. W., Muir, D. C. G., Nagatani-Yoshida, K. Toward a global understanding of chemical pollution: A first comprehensive analysis of national and regional chemical inventories. Environmental Science & Technology. 54 (5), 2575-2584 (2020).
- Brooks, B. W., et al. Toxicology advances for 21st century chemical pollution. One Earth. 2 (4), 312-316 (2020).
- Kostal, J., Voutchkova-Kostal, A. Going all in: A strategic investment in in silico toxicology. Chemical Research in Toxicology. 33 (4), 880-888 (2020).
- Cheng, W., Doering, J. A., LaLone, C., Ng, C. Integrative computational approaches to inform relative bioaccumulation potential of per- and polyfluoroalkyl substances (PFAS) across species. Toxicology Sciences. 180 (2), 212-223 (2021).
- Kostich, M. S., Lazorchak, J. M. Risks to aquatic organisms posed by human pharmaceutical use. Science of the Total Environment. 389 (2-3), 329-339 (2008).
- Gunnarsson, L., Jauhiainen, A., Kristiansson, E., Nerman, O., Larsson, D. G. Evolutionary conservation of human drug targets in organisms used for environmental risk assessments. Environmental Science & Technology. 42 (15), 5807-5813 (2008).
- LaLone, C. A., et al. Evidence for cross species extrapolation of mammalian-based high-throughput screening assay results. Environmental Science & Technology. 52 (23), 13960-13971 (2018).
- LaLone, C. A., et al. Editor's highlight: Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS): A web-based tool for addressing the challenges of cross-species extrapolation of chemical toxicity. Toxicology Sciences. 153 (2), 228-245 (2016).
- Head, J. A., Hahn, M. E., Kennedy, S. W. Key amino acids in the aryl hydrocarbon receptor predict dioxin sensitivity in avian species. Environmental Science & Technology. 42 (19), 7535-7541 (2008).
- Bass, C., et al. Mutation of a nicotinic acetylcholine receptor β subunit is associated with resistance to neonicotinoid insecticides in the aphid Myzus persicae. BMC Neuroscience. 12, 51-51 (2011).
- Erdmanis, L., et al. Association of neonicotinoid insensitivity with a conserved residue in the loop d binding region of the tick nicotinic acetylcholine receptor. Biochemistry. 51 (23), 4627-4629 (2012).
- Doering, J. A., et al. et al. In silico site-directed mutagenesis informs species-specific predictions of chemical susceptibility derived from the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) tool. Toxicology Sciences. 166 (1), 131-145 (2018).
- Noyes, P. D., et al. Evaluating chemicals for thyroid disruption: Opportunities and challenges with in vitro testing and adverse outcome pathway approaches. Environmental Health Perspectives. 127 (9), 95001(2019).
- Park, G. Y., Jamerlan, A., Shim, K. H., An, S. S. A. Diagnostic and treatment approaches involving transthyretin in amyloidogenic diseases. Int J Mol Sci. 20 (12), 2982(2019).
- Rabah, S. A., Gowan, I. L., Pagnin, M., Osman, N., Richardson, S. J. Thyroid hormone distributor proteins during development in vertebrates. Front Endocrinol (Lausane). 10, 506(2019).
- Richardson, S. J. Cell and molecular biology of transthyretin and thyroid hormones. International Review of Cytology. 258, 137-193 (2007).
- Yamauchi, K., Ishihara, A. Transthyretin and Endocrine Disruptors. Recent Advances in Transthyretin Evolution, Structure and Biological Functions. Richardson, S. J., Cody, V. , Springer. Berlin Heidelberg, Germany. 159-171 (2009).
- Iakovleva, I., et al. Tetrabromobisphenol A is an efficient stabilizer of the transthyretin tetramer. PLoS One. 11 (4), 0153529(2016).
- Ishihara, A., Sawatsubashi, S., Yamauchi, K. Endocrine disrupting chemicals: Interference of thyroid hormone binding to transthyretins and to thyroid hormone receptors. Molecular and Cellular Endocrinology. 199 (1), 105-117 (2003).
- Kar, S., Sepúlveda, M. S., Roy, K., Leszczynski, J. Endocrine-disrupting activity of per- and polyfluoroalkyl substances: Exploring combined approaches of ligand and structure based modeling. Chemosphere. 184, 514-523 (2017).
- Morais-de-Sa, E., Pereira, P. J., Saraiva, M. J., Damas, A. M. The crystal structure of transthyretin in complex with diethylstilbestrol: A promising template for the design of amyloid inhibitors. Journal of Biological Chemistry. 279 (51), 53483-53490 (2004).
- Morgado, I., Campinho, M. A., Costa, R., Jacinto, R., Power, D. M. Disruption of the thyroid system by diethylstilbestrol and ioxynil in the sea bream (Sparus aurata). Aquatic Toxicology. 92 (4), 271-280 (2009).
- Yamauchi, K., Prapunpoj, P., Richardson, S. J. Effect of diethylstilbestrol on thyroid hormone binding to amphibian transthyretins. General and Comparative Endocrinology. 119 (3), 329-339 (2000).
- Zhang, J., et al. Structure-based virtual screening protocol for in silico identification of potential thyroid disrupting chemicals targeting transthyretin. Environmental Science & Technology. 50 (21), 11984-11993 (2016).
- Ren, X. M., et al. Binding interactions of perfluoroalkyl substances with thyroid hormone transport proteins and potential toxicological implications. Toxicology. 366-367, 32-42 (2016).
- Wilson, N., Mbabazi, K., Seth, P., Smith, H., Davis, N. L. Drug and opioid-involved overdose deaths - United States, 2017-2018. Morbidity and Mortality Weekly Report. 69 (11), 290-297 (2020).
- EPA. National Pollutant Discharge Elimination System (NPDES). United States Environmental Protection Agency. , Available from: https://www.epa.gov/npdes/npdes-resources (2018).
- Duvallet, C., Hayes, B. D., Erickson, T. B., Chai, P. R., Matus, M. Mapping community opioid exposure through wastewater-based epidemiology as a means to engage pharmacies in harm reduction efforts. Preventing Chronic Disease. 17, 200053(2020).
- Gushgari, A. J., Venkatesan, A. K., Chen, J., Steele, J. C., Halden, R. U. Long-term tracking of opioid consumption in two United States cities using wastewater-based epidemiology approach. Water Research. 161, 171-180 (2019).
- Lau, B., Bretaud, S., Huang, Y., Lin, E., Guo, S. Dissociation of food and opiate preference by a genetic mutation in zebrafish. Genes Brain Behave. 5 (7), 497-505 (2006).
- Bossé, G. D., Peterson, R. T. Development of an opioid self-administration assay to study drug seeking in zebrafish. Behavioural Brain Research. 335, 158-166 (2017).
- Mottaz, H., et al. Dose-dependent effects of morphine on lipopolysaccharide (LPS)-induced inflammation, and involvement of multixenobiotic resistance (MXR) transporters in LPS efflux in teleost fish. Environmental Pollution. 221, 105-115 (2017).
- Manglik, A., et al. Crystal structure of the µ-opioid receptor bound to a morphinan antagonist. Nature. 485 (7398), 321-326 (2012).
- Comer, S. D., Cahill, C. M. Fentanyl: Receptor pharmacology, abuse potential, and implications for treatment. Neuroscience & Biobehavioral Reviews. 106, 49-57 (2019).
- Podlewska, S., Bugno, R., Kudla, L., Bojarski, A. J., Przewlocki, R. Molecular modeling of µ opioid receptor ligands with various functional properties: PZM21, SR-17018, morphine, and fentanyl-simulated interaction patterns confronted with experimental data. Molecules. 25 (20), 4636(2020).
- Huang, W., et al. Structural insights into µ-opioid receptor activation. Nature. 524 (7565), 315-321 (2015).
- Lipiński, P. F. J., et al. Fentanyl family at the mu-opioid receptor: Uniform assessment of binding and computational analysis. Molecules. 24 (4), 740(2019).
- Boland, L. A., Angles, J. M. Feline permethrin toxicity: Retrospective study of 42 cases. Journal of Feline Medicine and Surgery. 12 (2), 61-71 (2010).
- Stevenson, B. J., Pignatelli, P., Nikou, D., Paine, M. J. Pinpointing P450s associated with pyrethroid metabolism in the dengue vector, Aedes aegypti: developing new tools to combat insecticide resistance. PLoS Neglected Tropical Diseases. 6 (3), 1595(2012).
- Ankley, G. T., Gray, L. E. Cross-species conservation of endocrine pathways: A critical analysis of tier 1 fish and rat screening assays with 12 model chemicals. Environmental Toxicology and Chemistry. 32 (5), 1084-1087 (2013).
- Meteyer, C. U., Rideout, B. A., Gilbert, M., Shivaprasad, H. L., Oaks, J. L. Pathology and proposed pathophysiology of diclofenac poisoning in free-living and experimentally exposed oriental white-backed vultures (Gyps bengalensis). Journal of Wildlife Diseases. 41 (4), 707-716 (2005).
- EPA. ECOTOX User Guide: ECOTOXicology Knowledgebase System. EPA, United States Environmental Protection Agency. , Available from: https://cfpub.epa.gov/ecotox/index.cfm (2021).
- ECOS Environmental Conservation Online System. U.S. Fish & Wildlife Service. , Available from: https://ecos.fws.gov/ecp/ (2021).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır