Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Lentivirüs Aracılı CRISPR / Cas9 Gen Düzenleme Kullanarak Nakavt Kas Hücre Hatlarının Geliştirilmesi
Bu Makalede
Özet
Protokol, kılavuz-RNA'ların tasarımından hücresel klonlamaya ve nakavt klonlarının karakterizasyonuna kadar CRISPR / Cas9 kullanarak nakavt miyoblastlarının nasıl üretileceğini açıklar.
Özet
Kümelenmiş düzenleyici aralıklı kısa palindromik tekrarların (CRISPR) / Cas 9'un önemli bir uygulaması, özellikle genetik tanı sırasında tanımlanan bir hastalıkla ilişkili yeni genlerin / proteinlerin işlevini incelemek için nakavt hücre hatlarının geliştirilmesidir. Bu tür hücre hatlarının gelişimi için, iki ana konunun çözülmesi gerekir: CRISPR araçlarının (Cas9 ve kılavuz RNA) seçilen hücrelere yüksek verimlilikle yerleştirilmesi ve Cas9 aktivitesinin seçilen genin spesifik olarak silinmesiyle sınırlandırılması. Burada açıklanan protokol, CRISPR araçlarının kas hücreleri gibi transfekte edilmesi zor hücrelere yerleştirilmesine adanmıştır. Bu protokol, halka açık plazmidlerle üretilen lentivirüslerin kullanımına dayanmaktadır ve tüm klonlama adımlarının ilgilenilen bir geni hedeflemek için tanımlandığı bilinmektedir. Cas9 aktivitesinin kontrolü, Cas9'u hedef alan bir kılavuz RNA'yı kodlayan bir lentivirüs ile hücrelerin transdüksiyonunun Cas9 ekspresyonunun aşamalı olarak ortadan kaldırılmasına izin verdiği KamiCas9 adı verilen daha önce tanımlanmış bir sistemin adaptasyonu kullanılarak gerçekleştirilmiştir. Bu protokol, kas hücre içi kalsiyum salınımında ve uyarım-kasılma eşleşmesinde yer alan bu önemli kalsiyum kanalının nakavtını doğrulamak için protein ve fonksiyonel düzeyde daha da karakterize edilen bir RYR1-nakavt insan kas hücresi hattının geliştirilmesine uygulanmıştır. Burada açıklanan prosedür, kas hücrelerindeki diğer genlere veya transfekte edilmesi zor diğer hücrelere kolayca uygulanabilir ve bu genleri insan hücrelerinde incelemek için değerli araçlar üretebilir.
Giriş
Gen diziliminin ilerlemesi ve belirli bir dokudaki bilinmeyen fonksiyonlara sahip genlerdeki mutasyonların tanımlanmasıyla, yeni bir hedef genin işlevini anlamak ve ilgili patofizyolojik mekanizmalara katılımını doğrulamak için ilgili hücresel modellerin geliştirilmesi önemli bir araç oluşturmaktadır. Ek olarak, bu modeller gelecekteki terapötik gelişmeler için büyük önem taşımaktadır 1,2 ve deneylerde hayvanların kullanımının azaltılmasına yönelik uluslararası önerilerle aynı doğrultuda nakavt hayvan modellerinin geliştirilmesine ilginç bir alternatif oluşturmaktadır. CRISPR / Cas9 kullanarak gen düzenleme, şu anda mevcut olan en güçlü araçlar arasındadır, bu da birçok nakavt / knock-in modelinin geliştirilmesine izin vermiştir ve CRISPR / Cas9 kullanarak hedeflenen gen doğrulaması, CRISPR / Cas93'ün en yaygın kullanılan uygulamaları arasındadır. Gen düzenlemenin başarısı, hedef hücre modelinde CRISPR araçlarını (kılavuz RNA'lar ve nükleaz Cas9) tanıtma yeteneğine dayanır; bu,kas hücreleri 4 gibi transfekt edilmesi zor birçok hücrede zor olabilir. Bu zorluk, birçok hücre tipini verimli bir şekilde dönüştürmek ve transgenini iletmek için büyük avantaja sahip olan virüsün, genellikle lentivirüsün kullanılmasıyla aşılabilir. Ancak en büyük dezavantajı, transgenin konakçı hücre genomuna entegrasyonudur, bu da entegrasyon bölgesinde lokalize genlerin potansiyel olarak değişmesine ve transgenin kalıcı ekspresyonuna yol açar, bu da nükleaz Cas9 durumunda zararlı sonuçlara neden olur5. Merienne ve meslektaşları6 tarafından, Cas9 geninin kendisini hedef alan ve Cas9 inaktivasyonuna yol açan bir kılavuz-RNA'nın hücrelerine girişten oluşan akıllı bir çözüm önerilmiştir. Bu stratejinin bir uyarlaması, burada, transfekte edilmesi zor hücrelerdeki hemen hemen her genin yok edilmesine izin veren kullanıcı dostu ve çok yönlü bir protokol olarak sunulmaktadır.
Burada sunulan protokolün amacı, ölümsüzleştirilmiş kas hücrelerine ilgi duyan bir genin inaktivasyonunu indüklemektir. Farklı ölümsüzleştirilmiş hücre türlerinde ilgilenilen herhangi bir geni yok etmek için kullanılabilir. Burada açıklanan protokol, kılavuz RNA'ları ve bunların lentiviral plazmidlere klonlanmasını tasarlamak, lentiviral vektörlerde CRISPR araçlarını üretmek, hücreleri farklı lentivirüslerle dönüştürmek ve homojen düzenlenmiş bir hücre hattı üretmek için hücreleri klonlamak için adımlar içerir.
Bu protokolü kullanarak, ölümsüzleştirilmiş insan iskelet kası hücreleri, hücre içi kalsiyum salınımı ve kas kasılmasında rol oynayan temel bir kalsiyum kanalı olan tip 1 ryanodin reseptörünün (RyR1) silinmesiyle geliştirilmiştir7. Genin nakavtı (KO), Western blot kullanılarak protein seviyesinde ve kalsiyum görüntüleme kullanılarak fonksiyonel düzeyde doğrulanmıştır.
Protokol
Kas biyopsileri, Avrupa tavsiyelerine ve Fransız mevzuatına uygun olarak Araştırma için Dokular Bankası'ndan (Myobank, AB ağı EuroBioBank'ın bir ortağı, Paris, Fransa) alınmıştır. Tüm bireylerden yazılı bilgilendirilmiş onam alınmıştır. Ölümsüzleştirilmiş miyoblastlar Dr. V. Mouly (Myology Institute, Paris, Fransa) tarafından nazikçe üretildi ve protokoller Myology Institute etik komitesi tarafından onaylandı (MESRI, n AC-2019-3502).
1. CRISPR kılavuz tasarımı
- Silinecek genin bölgesini tanımlayın. ensembl.org veya genome.ucsc.edu gibi genom tarayıcı araçlarını kullanarak genomik dizisini arayın ve silinecek bölgenin her iki tarafındaki kılavuz RNA'yı (gRNA) aramak için iki bölgenin kromozomik koordinatlarını belirleyin.
- Burada kullanılan gen için, RYR1 geninin FASTA dizisini ve ekzon 101'in dizisini aşağıdaki gibi elde edin. Toplulukta, insan genomunun en son sürümünde RYR1'i arayın, ilk girişi seçin ve protein kodlama dizisinin transkriptine tıklayın. Ardından, genin ekzonları listesine yönlendirmek için Exons'a tıklayın.
- Diziyi İndir'e tıklayın ve tüm genin tam fikir birliği dizisini indirmek için Yalnızca Genomik Dizi'yi seçin. Genin ekzon ve intronlarının listesini aşağı kaydırın ve hedeflenen olanları seçin.
- Gendeki karşılık gelen nükleotid dizisini bulun. İntronların nükleotid dizilerini, silinecek ekzonun hemen yukarı ve aşağı akışlarını seçin, bu da gRNA'ları aramak için kullanılacaktır.
- Burada kılavuz 1 ve kılavuz 2 olarak adlandırılan iki gRNA'yı, Crispor.tefor.net8 gibi çevrimiçi araçları kullanarak adım 1.1'de tanımlanan bölgelerde (silinecek bölgenin yukarı ve aşağı akış intronları) tasarlayın. Birkaç yüz baz çifti (bp) ile ayrılmış iki gRNA'yı seçin, her bir gRNA'nın dizisi, protospacer bitişik motifi (PAM) olmadan tam olarak 20 nükleotid uzunluğundadır. Hedef dışı sınırlamak için mevcut en iyi kılavuzları seçin. Kılavuz tasarımı örneği için Şekil 1'e bakın.
- İki kılavuz arasındaki dizinin silinmesi ve ilgilenilen genin yok edilmesiyle sonuçlanması beklenir, böylece iki kılavuzun konumunu, ilgilenilen gendeki temel bir diziyi veya temel bir ekzonu silecek şekilde seçer. Silinen sekansın / ekzonun, yalnızca genin alternatif bir transkriptinde bulunmadığından ve / veya proteinin önemli bir bölümünü kodladığından emin olun, böylece silinmesi fonksiyonel bir nakavtla sonuçlanacaktır.
NOT: Cas9 bölünme yerinin, protospacer bitişik motifinin (PAM) 3 bp yukarı yönünde meydana geleceği tahmin edilse de, daha büyük bir mesafede bölünme de meydana gelebilir, bu nedenle iyi bir çözüm, introndaki bölünme gibi bölünme bölgesinin kesin lokalizasyonuna bağlı değildir.
- İki kılavuz arasındaki dizinin silinmesi ve ilgilenilen genin yok edilmesiyle sonuçlanması beklenir, böylece iki kılavuzun konumunu, ilgilenilen gendeki temel bir diziyi veya temel bir ekzonu silecek şekilde seçer. Silinen sekansın / ekzonun, yalnızca genin alternatif bir transkriptinde bulunmadığından ve / veya proteinin önemli bir bölümünü kodladığından emin olun, böylece silinmesi fonksiyonel bir nakavtla sonuçlanacaktır.
- Aşağıdaki dizilere sahip olmak için PAM olmadan her gRNA için ters kompleman (RC) dizisini belirleyin: Kılavuz 1 ve Kılavuz 1-RC, Kılavuz 2 ve Kılavuz 2-RC.
- Plazmidlerin klonlanmasını gerçekleştirmek için Tablo 1'de sunulan primerleri sıralayın. Protokol boyunca, sterilH2O'da 10 nM konsantrasyonda primerler kullanın.
NOT: Bu primerlerdeki gRNA'lara eklenen diziler (kalın ve altı çizili), sırasıyla kılavuz, promotor ve trans-aktive edici Crispr RNA'dan (tracrRNA) önce ve sonra plazmidin dizisine karşılık gelir ve primerler ile plazmid arasında iyi bir örtüşme sağlamak için değiştirilmemelidir.
2. Plazmid klonlaması
NOT: Bu adımda, gRNA'lar lentivirüs üretimi için plazmid omurgasına yerleştirilecektir. İki gRNA'yı kodlayan bir kaset, ilk önce üst üste binen primerler kullanılarak ardışık polimeraz zincir reaksiyonları (PCR) tarafından üretilir. Yeni kaset daha sonra lentiviral omurga plazmidi #87919'a yerleştirilir.
- Aşağıdaki plazmidleri elde edin: plazmid #87919, bir lentiviral vektörde CRISPR kılavuz RNA'ları kodlayan ve bir lentiviral vektörde SpCas9 dizisi için kodlayan plazmid #87904.
- Kaset yapımı
NOT: Klonlama protokolü şu şekilde özetlenmiştir: Şekil 2.- 2 μL plazmid #87919, 2 μL primer_XmaIF, 2 μL primer_Guide1R, 25 μL polimeraz karışımı ve 19 μLH2O ile bir PCR (A) reaksiyonu çalıştırın. 60 °C'de 30 sn, 72 °C'de 1 dk 45 sn ve 72 °C'de 7 dk'lık son uzama. Adım 1.4'te tanımlanan primerlerin Tm'si 60 ° C'dir.
NOT: Program 1'deki uzama süresi oldukça uzun görünse de, bu uzama süresi doğru boyutta yeterli malzemenin üretilmesini sağlamak için seçilmiştir. Gerçekten de, plazmidde tekrarlanan diziler nedeniyle (RNA kılavuzlarından sonra tracrRNA dizisi plazmid #87919'da üç kez tekrarlanır) beklenen DNA'nın PCR amplifikasyonu zordur ve ardışık PCR'lerde ek küçük bantlar üretilir. Bu nedenle, farklı PCR ürünleri arasındaki rekabet nedeniyle, ya uzama süresi arttırılmıştır (en uzun olanı desteklemek ve sonunda yeterli saflaştırılmış malzemeye sahip olmak için) ya da uzun parça için bir dokunmatik PCR (program 2) kullanılmıştır (adım 2.2.6'da PCR (son) için açıklandığı gibi). - PCR ürünlerini Tris-Borate-EDTA tamponunda (TBE) %1'lik bir agaroz jeli üzerinde ayırın, 300 bp fragmanını tüketin ve saflaştırın. Üreticinin talimatını takiben, 20 μL'lik son hacimde elüsyon ile özel bir kit kullanarak saflaştırma gerçekleştirin. arıtılmış parçayı doğrudan kullanın veya adım 2.2.5 için -20 ° C'de saklayın.
- PCR programı 1'i kullanarak 2 μL plazmid #87919, 2 μL primer_Guide1F, 2 μL primer_Guide2R, 25 μL polimeraz karışımı ve 19 μLH2O ile bir PCR (B) reaksiyonu çalıştırın. PCR ürünlerini TBE'de% 1'lik bir agaroz jeli üzerinde ayırın, 400 bp fragmanını 20 μL elüsyon tamponunda tüketin ve saflaştırın. Saflaştırılmış parçayı doğrudan kullanın veya adım 2.2.5 için -20 ° C'de saklayın.
- PCR programı 1'i kullanarak 2 μL plazmid #87919, 2 μL primer_Guide2F, 2 μL primer_BlpIR, 25 μL polimeraz karışımı ve 19 μLH2O ile bir PCR (C) reaksiyonu çalıştırın. PCR ürünlerini TBE tamponunda% 1'lik bir agaroz jeli üzerinde ayırın. 600 bp parçayı 20 μL elüsyon tamponunda tüketin ve saflaştırın. Saflaştırılmış parçayı doğrudan kullanın veya adım 2.2.6 için -20 ° C'de saklayın.
NOT: Adım 2.2.1'in NOTUNDA açıklandığı gibi, plazmiddeki tekrarlanan bölgeler üzerinde astarın hibridizasyonu nedeniyle 900 bp'de başka bir parça görülebilir. Varsa, bu bant atılmalıdır. - PCR programı 1 ile 2 μL elüsyon PCR A (adım 2.2.2'den itibaren), 2 μL elüsyon PCR B (adım 2.2.3'ten itibaren), 2 μL primer_XmaIF, 2 μL primer_Guide2R, 25 μL polimeraz karışımı ve 19 μL H2O ile bir PCR (D) reaksiyonu çalıştırın. PCR ürünlerini TBE tamponunda% 1'lik bir agaroz jeli üzerinde ayırın; 700 bp parçayı 20 μL elüsyon tamponunda tüketin ve saflaştırın. Saflaştırılmış parçayı doğrudan kullanın veya adım 2.2.6 için -20 ° C'de saklayın.
NOT: Diğer bantlar, tekrarlanan bölgelerdeki primerlerin hibridizasyonu nedeniyle >1.000 bp, 400 bp ve 300 bp'de görülebilir ve atılmalıdır. - 4 μL elüsyon PCR C (adım 2.2.4'ten itibaren), 4 μL elüsyon PCR D (adım 2.2.5'ten itibaren), 4 μL primer_XmaIF, 4 μL primer_BlpIR, 50 μL polimeraz karışımı ve 34 μLH2O ile bir PCR (son) reaksiyonu çalıştırın. Kullanılan program aşağıdaki gibidir (PCR programı 2): 98 ° C'de 5 dakika boyunca ilk denatürasyon; altı döngü: 98 °C'de 30 sn, 66 °C'de 30 s (döngü başına 1 °C hibridizasyon sıcaklığında azalma), 72 °C'de 1 dk 45; 35 döngü: 98 °C'de 30 sn, 60 °C'de 30 sn, 72 °C'de 1 dk 45 ve 72 °C'de 5 dakikalık son uzama.
NOT: Bu son amplifikasyon için PCR programı, her kılavuzdan hemen sonra iki tekrarlanan tracrRNA dizisi içeren son amplifikasyonun büyüklüğü nedeniyle, öncekinden farklı olarak bir dokunmatik PCR'dir. - PCR ürünlerini TBE tamponunda% 1'lik bir agaroz jeli üzerinde ayırın; 20 μL elüsyon tamponunda yaklaşık 1.300 bp'de göç eden son kaseti tüketin ve saflaştırın. Salınan ürünü sayısallaştırın. Saflaştırılmış parçayı doğrudan kullanın veya adım 2.3.2.1 için -20 ° C'de saklayın. Bu, lentiviral plazmidin içine yerleştirilecek son üründür.
NOT: Diğer parçalar >1000 bp, 400 bp ve 300 bp'de görülebilir ve atılması gereken tamamlanmamış PCR fragmanlarına karşılık gelir.
- 2 μL plazmid #87919, 2 μL primer_XmaIF, 2 μL primer_Guide1R, 25 μL polimeraz karışımı ve 19 μLH2O ile bir PCR (A) reaksiyonu çalıştırın. 60 °C'de 30 sn, 72 °C'de 1 dk 45 sn ve 72 °C'de 7 dk'lık son uzama. Adım 1.4'te tanımlanan primerlerin Tm'si 60 ° C'dir.
- gRNA kasetinin lentiviral plazmid omurgasına yerleştirilmesi.
- Plazmidi #87919 numaralı plazmidin XmaI ve BlpI kısıtlama enzimleri ile çift sindirimi ile doğrusallaştırın.
- Reaksiyonu 15 μL önerilen tampon, 15 μL plazmid (1μg / μL), 7.5 μL BlpI enzimi (10 U / μL'de), 7.5 μL XmaI enzimi (10 U / μL'de) ve112.5μL H 2 O ile hazırlayın. Toplam miktarı% 1'lik bir agaroz jeli üzerine yükleyin, ~ 10 kb plazmidi uygun bir kit ile kesin ve saflaştırın. Elüsyon 20 μL tamponda gerçekleştirilir ve salınan ürün optik yoğunluk ölçümü kullanılarak ölçülür.
NOT: Büyük DNA fragmanı için Sun ve coll9 tarafından tanımlanan protokol gibi uygun bir saflaştırma protokolü kullanın.
- Reaksiyonu 15 μL önerilen tampon, 15 μL plazmid (1μg / μL), 7.5 μL BlpI enzimi (10 U / μL'de), 7.5 μL XmaI enzimi (10 U / μL'de) ve112.5μL H 2 O ile hazırlayın. Toplam miktarı% 1'lik bir agaroz jeli üzerine yükleyin, ~ 10 kb plazmidi uygun bir kit ile kesin ve saflaştırın. Elüsyon 20 μL tamponda gerçekleştirilir ve salınan ürün optik yoğunluk ölçümü kullanılarak ölçülür.
- gRNA kasetini ve plazmidi bağlayın. Reaksiyon karışımını adım 2.2.7'den gRNA kaseti ve adım 2.3.1.1'den doğrusallaştırılmış plazmid ile hazırlayın, 10 μL'lik bir son hacme sahip olmak için 2 μL enzim veH2O ekleyin. p_guides adı verilen son plazmidi üretmek için 50 ° C'de 15 dakika boyunca inkübe edin.
NOT: DNA kaset miktarı 50-100 ng arasında, plazmid miktarı ise 2:1 molar oranla 100-200 ng arasında olmalıdır.
- Plazmidi #87919 numaralı plazmidin XmaI ve BlpI kısıtlama enzimleri ile çift sindirimi ile doğrusallaştırın.
- Stbl3 (50 μL) veya XL10-Gold gibi kimyasal olarak yetkin E.coli'yi dönüştürmek için taze hazırlanmış plazmidin 2 μL'sini kullanın ve antibiyotiksiz 37 °C'de 1 saat büyümeden sonra 100 μg / mL ampisilin ile LB agar plakasına yayın. Gece boyunca 37 ° C'de inkübe edin. Birkaç koloni seçin ve üreticinin talimatlarını izleyerek ticari bir kit kullanarak mini bir hazırlık yapın.
- PCR programı 1'i kullanarak Primer_XmaI ve Primer_BlpR ile miniprep DNA üzerinde bir PCR amplifikasyonu gerçekleştirin (bkz. Şekil 2). PCR ürünlerini TBE tamponunda% 1'lik bir agaroz jeli üzerinde ayırın. Yaklaşık 1300 bp beklenen boyutta bir bant ile birkaç koloni (~ 5) seçin.
- gRNA kasetinin doğru yerleştirildiğini doğrulamak için Primer_XmaI veya Primer_BlpR kullanarak seçilen kolonilerin DNA dizilimini gerçekleştirin.
NOT: Dizi doğrulanmış kolonilerden biri çalışmada daha fazla kullanılmıştır ve plazmid p_guides olarak adlandırılır. - Adım 2.2'den (kaset yapımı) Primers-Killer F ve R ve Primers-mCherry F ve R ile tekrarlayın. Plazmid p_Killer olarak adlandırılır.
3. Lentivirüs üretimi
- Üreticinin talimatlarını izleyerek endotoksin içermeyen bir maxi-prep kiti kullanarak gerekli tüm plazmidlerin büyük bir kısmını üretin ve saflaştırın. Alikotları 2 μg / μL'de hazırlayın. -20 ° C'de saklayın
- Hücrelerin hazırlanması (1. Gün)
- % 10 fetal sığır serumu (FBS) ve% 1 penisilin / streptomisin ile desteklenmiş, Dulbecco'nun modifiye kartal ortamı (DMEM) yüksek glikozlu piruvatından oluşan 16 mL ortamda plaka başına 1 x 106 HEK293 hücre ile tohumlanmış 145 cm'lik 18 plaka hazırlayın. Hücreleri 37 ° C'de, 3 gün boyunca% 5'lik bir CO2 inkübatöründe yükseltin.
NOT: Lentivirüs üretimi, Biyogüvenlik Seviye 2 laboratuvarında, tek kullanımlık koruyucu giysi, koruyucu başlık ve eldivenler dahil olmak üzere uyarlanmış koruma donanımları kullanılarak dikkatli bir şekilde gerçekleştirilmelidir. Tüm deneyler, filtre uçlarına sahip laminer bir akış başlığı (BSLII güvenlik kabini) altında yapılmalıdır. Lentivirüsleri içeren tüm çözeltiler ve kullanılan tüm plastik/cam atıklar, %70 etanol veya başka bir virüs inaktive edici ile inaktive edilmelidir.
- % 10 fetal sığır serumu (FBS) ve% 1 penisilin / streptomisin ile desteklenmiş, Dulbecco'nun modifiye kartal ortamı (DMEM) yüksek glikozlu piruvatından oluşan 16 mL ortamda plaka başına 1 x 106 HEK293 hücre ile tohumlanmış 145 cm'lik 18 plaka hazırlayın. Hücreleri 37 ° C'de, 3 gün boyunca% 5'lik bir CO2 inkübatöründe yükseltin.
- Hücrelerin transfeksiyonu (4. Gün)
- Hücrelerin% 60 -% 65 birleşmesine ulaştıklarından emin olmak için hücrelerin birleşmesini kontrol edin.
- Her plaka için aşağıdakileri içeren transfeksiyon çözeltisini hazırlayın: İlgilenilen plazmidin 20.8 μg (p-kılavuzları veya p-Killer veya pCas9 #87904), zarfı kodlayan plazmidin 4.8 μg (VSV-G, #8454), lentiviral ambalaj için plazmid psPAX2'nin (#12260) 20.8 μg'ı, 136 μL kalsiyum fosfat veH2O ile 1.000 μL'lik son bir hacme ayarlayın.
NOT: Reaktiflerin optimum şekilde hazırlanmasını sağlamak için tüm plakalar için aynı anda bir karışım hazırlamayın; Aynı anda altı tabak için bir karışım hazırlayın, örneğin 18 tabak için, altı tabak için üç kez bir karışım hazırlayın. - Oda sıcaklığında (RT) en az 10 dakika inkübe edin ve hücrelere damla damla 2 mL çözelti ekleyin. Transfeksiyon reaktifini, plakanın geri, ileri, yukarı ve aşağı doğru hafifçe çalkalanması ile homojenize edin, 37 ° C'de, en az 5 saat boyunca% 5 CO2'de inkübe edin.
NOT: Bu noktadan itibaren ve lentivirüs üretiminin sonuna kadar, ikinci bir çift eldiven, koruma kılıfı ve tek kullanımlık bir plastron dahil olmak üzere ek koruma donanımı takın. - Transfeksiyondan beş saat sonra, ortamı plakalardan çıkarın ve transfeksiyon reaktiflerinden kurtulmak için PBS ile durulayın. 12 mL taze ortam ekleyin ve 37 ° C'de,% 5 CO2'de 48 saat inkübe edin.
- Viral parçacıkların toplanması (6. Gün)
- Ortamı tüm plakalardan toplayın ve bir araya getirin. Hücresel kalıntıları pelet etmek için 4 ° C'de 5 dakika boyunca 800 x g'de santrifüj. Süpernatantı 0,45 μm filtre kullanarak filtreleyin (birden fazla filtre gerekir).
- Sallanan bir kova rotorunda 4 °C'de 2 saat boyunca 68.300 x g'de santrifüj. Süpernatantı çıkarın ve tüpleri güvenlik kabininin altında 5-10 dakika boyunca bir kağıt üzerinde baş aşağı bırakarak mümkün olduğunca fazla sıvıyı uzaklaştırın ve ardından pelet başına 100 μL HEK proliferasyon ortamı ekleyin. 4 °C'de en az 2 saat sonra, yukarı ve aşağı pipetleyerek peletleri yeniden askıya alın. Tüm askıya alınmış peletleri bir havuza alın.
NOT: Peletler, alikotasyondan önce gece boyunca 4 ° C'de ortamda bırakılabilir. - Aliquot lentivirus 10 μL veya 25 μL numune boyutunda (kullanıma bağlı olarak) ve sıvı azot ile çıtçıt-dondurma. -80 °C'de saklayın. Çözülmüş bir aliquot'u dondurmayın.
- LV-kılavuzları, LV-Killer ve LV-Cas9 üretmek için 3.2 ila 3.4 arasındaki adımları ilgilendiğiniz diğer plazmidlerle tekrarlayın.
NOT: Alternatif olarak, lentivirüsler bir şirketten veya bir virüs tesisinden satın alınabilir.
4. Lentivirüs titrasyonu
NOT: Virüs titrasyonu HEK293 hücreleri üzerinde gerçekleştirilir. Titrasyon, sonraki adımlarda, ilgilenilen hücreler için hücre başına kesin sayıda lentivirüs (lentivirüs grubu ne olursa olsun) dahil etmek için önemlidir. Bir hücreyi verimli bir şekilde dönüştüren viral parçacıkların sayısına enfeksiyon çokluğu (MOI) denir: MOI 10, böylece hücre başına 10 viral parçacığın sokulmasına karşılık gelir. Donma/çözülme döngüsü lentivirüsün yaşayabilirliğini etkilediğinden, titrasyon dondurulmuş bir lentivirüs aliquot ile gerçekleştirilir ve sonraki her deney aynı havuzun yeni bir aliquot ile gerçekleştirilir. Burada bir titrasyon yöntemi açıklanmıştır, ancak başka yöntemler de kullanılabilir.
- 1. günde, altta cam kapaklı beş adet 35 mm'lik plakada ve kapaksız iki adet35 mm'lik plakada plaka başına 1 x 10 5 hücre tohumlayın.
- 2. günde, kapaksız iki plakadan, tripsinizasyondan sonra hücre sayısını toplayın ve sayın ve plaka başına ortalama hücre miktarını (N) belirleyin.
- Proliferasyon ortamında 1/10'da seyreltilmiş 100 μL lentivirüs hazırlayın. Beş kültürlü plakayı, 1 ila 50 μL seyreltilmiş virüs arasında farklı hacimlerde seyreltilmiş virüsle dönüştürün. Bu protokol için, beş plakayı dönüştürmek için aşağıdaki hacimleri kullanın: 1 μL, 5 μL, 10 μL, 20 μL, 50 μL. 37 ° C'de 48 saat boyunca,% 5 CO2'de inkübe edin.
- 4. günde, RT'deki örtülerin inkübasyonuyla% 4 paraformaldehit içinde 20 ila 30 dakika boyunca hücreleri sabitleyin. LV-Cas9 için, RT'de 10 dakika boyunca Fosfat Tampon Salin'de (PBS) %0.1 Triton X100 ile hücreleri geçirgenleştirin, RT'de 20 dakika boyunca PBS-%0.1 Triton X100-2% Keçi Serumu - %0.5 Sığır Serumu Albümini (BSA) ile doyurun ve RT'de 45 dakika boyunca birincil antikor anti-V5 (seyreltme 1/400) ile etiketleyin, ardından floresan sekonder antikor ile RT'de 30 dakika inkübasyon yapın. RT'de 10 dakika boyunca çekirdekleri Hoescht (PBS'de 10 μg/mL) ile etiketleyin.
- Kapak kapaklarını bir slayta monte edin ve 20x objektifle donatılmış bir floresan mikroskop kullanarak gözlemleyin. LV kılavuzları ve LV-Killer için, fiksasyondan sonra doğrudan çekirdek etiketlemesine ilerleyin ve kapak fişlerini monte edin. Her kapak kayması için, görüş alanındaki toplam çekirdek sayısını (toplam hücre sayısı) ve etiketlenen hücre sayısını (V5 veya mCherry ile) sayın ve her kapak fişi için etiketlenen hücrelerin oranını belirleyin.
- Dönüştürülen hücrelerin oranının en az% 10 olduğu ve% 50'yi geçmediği bir kapak kapağı seçin. Bu kapak kayması için iletim verimliliğini (X) belirleyin ve bu iletim verimliliğini elde etmek için kullanılan seyreltilmiş virüsün hacmini (V, μL cinsinden) not edin.
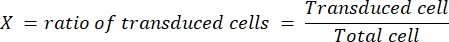
- Aşağıdaki formüle göre virüsün titresini (ip / mL olarak temsil edilen bulaşıcı parçacıklarda) belirleyin:

Seyreltme faktörü, adım 4.3'te gerçekleştirilen lentivirüsün seyreltilmesidir. HEK hücrelerinde belirlenen, rutin olarak LV-Cas9 için 1 x 10 9 ip/mL ve AG-kılavuz için1 x 10 10 ip/mL son titre elde ettik.
5. Miyoblastın transdüksiyonu
NOT: Ölümsüzleştirilmiş miyoblastlar, daha önce üretilen üç lentivirüs ile art arda dönüştürülür. %20 FBS, %2 penisilin/streptomisin, %2 Ultroser G ile takviye edilmiş ve 37 °C, %5 CO 2 ile takviye edilmiş Ham's F10'dan oluşan proliferasyon ortamında %50'nin altında bir yoğunlukta tutulurlar.
- Aşağıdaki formüle göre, seçilen sayıda hücreyi LV-Cas9 ve LV kılavuzları için MOI 10 ve LV-katil için MOI 20 ile tedavi etmek için gereken lentivirüs hacmini belirleyin:
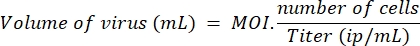
NOT: Paralel bir deneyde, transdüksiyon verimliliği bir kontrol lentivirüsü (lenti-GFP) kullanılarak miyoblastlar ve HEK üzerinde karşılaştırılmıştır ve bir miyoblastı bir HEK hücresine kıyasla verimli bir şekilde dönüştürmek için beş kat daha fazla lentivirüse ihtiyaç duyulduğunu belirledik. Böylece, HEK üzerinde ölçülen MOI 10, miyoblast başına iki viral partiküle karşılık gelir. Burada kullanılan MOI, HEK hücreleri üzerinde hesaplanır. - 1. günde, kuyu başına 100 μL proliferasyon ortamında 10.000 hücreli 96 kuyucuklu plakaları tohumlayın. 2. günde, adım 5.1'de hesaplanan uygun hacimdeki AG-kılavuzları ve LV-Cas9'u ekleyerek güvenlik kabini altındaki hücreleri dönüştürün. Hücreleri 7. güne kadar inkübatöre geri döndürün.
NOT: MOI'nin 25'ten yüksek kullanımı, özellikle LV-Cas9 için, yüksek konsantrasyonda lentivirüs konsantrasyonunda daha yüksek hücre ölümü nedeniyle düzenlenmiş hücrelerin sayısını artırmayabilir. - 7. günde, tripsinizasyon yapın ve hücreleri sayın. Hücreleri yeni bir plakada% 40 ila% 50'lik bir akıcılıkta tohumlayın ve hücreleri inkübatöre geri döndürün. Beş saat sonra, hücreleri LV-Killer ile 20'lik bir MOI'de (adım 5.1'de hesaplanan hacim) dönüştürün. Transfeksiyondan sonraki 5 ila 10 gün boyunca, en az iki pasaj boyunca hücreleri yükseltin ve her zaman% <50'lik düşük bir akıcılıkta tutun. Hücreler, büyümeleri normale döndüğünde (bölünme süresine göre tahmin edilen) bir sonraki adım olan hücresel klonlama için hazırdır.
NOT: Transdüksiyondan sonra proliferasyon biraz daha yavaş olabilir. Normal hücre büyümesi, bu deneysel prosedürden önce, hücrelerin bölünme süresinin tahmin edilmesiyle belirlenebilir.
6. Hücresel klonlama
NOT: Miyoblast transdüksiyonu zor olduğundan ve lentivirüs kullanırken bile% 100 verimliliğe asla ulaşmadığından, tamamen düzeltilmiş bir hücre hattı elde etmek için hücresel klonlama gereklidir. Bu sadece ölümsüzleştirilmiş hücrelerle veya birkaç hafta / ay boyunca kültürlenebilen ve büyütülebilen hücrelerle mümkündür.
- Hücreleri tripsinize edin ve sayın. Hücreleri proliferasyon ortamında 10 hücre / mL'de seyreltin ve hücreleri 100 μL / kuyu içi ortam içeren 96 delikli plakalarda 1 hücre / kuyucukta tohumlayın.
NOT: Tohumlanacak plakaların sayısı, beklenen gen düzenleme olasılığına bağlıdır, rutin olarak 2 ila 10 plaka kullanılır. - Hücreleri büyüme için izleyin ve miyoblastların akıcılığını% 50'nin altında tutarken, en az 35 mm'lik bir plakaya ulaşana kadar her bir kuyuyu daha büyük bir plakada kademeli olarak yükseltin. Bu adım, kullanılan hücrelere ve bir kuyuda izole edildikten sonra büyüme yeteneklerine bağlı olarak 2-6 hafta sürebilir.
7. Klon seçimi
NOT: Bu adım, büyüyen klonlardan hangilerinin uygun şekilde değiştirildiğini belirlemek için gerçekleştirilir.
- İlk kılavuzdan (Primer_BeforeGuide1F) önce bulunan bir astardan ve ikinci kılavuzdan (Primer_AfterGuide2R) sonra bulunan başka bir astardan oluşan bir astar seti tasarlayın, böylece varsayılan olarak değiştirilmiş diziyi çevreleyen bölgeyi güçlendirin. Burada kullanılan astarlar için Tablo 1'e bakın.
- Her klondan hücreleri toplayın, gelecekteki amplifikasyon için en az 300.000 hücre ayırın ve kalan hücreler üzerinde herhangi bir standart protokol kullanarak genomik DNA'yı çıkarın.
- Çok sayıda klon büyüyorsa, düzenlenmemiş olanları atmak için, DNA'yı bu havuzdan çıkarmak ve PCR ile test etmek için aynı tüpteki beş klondan hücreleri bir araya getirerek hızlı bir test yapın. Gerektiği kadar klonla tekrarlayın. Ardından, bireysel analiz yapmak için düzenlenmiş hücreleri içeren havuzları daha da ayırın.
- PCR ile düzenlemeyi aşağıdaki gibi kontrol edin. PCR reaksiyonunu 1 μL Primer_BeforeGuide1F, 1 μL Primer_AfterGuide2R, 12.5 μL polimeraz karışımı, 3 μL genomik DNA ve 7.5 μLH2O ile hazırlayın. Düzenlenmiş klonları tanımlamak için% 1'lik bir agaroz jeli üzerinde çalışın.
- Aşağıdaki gibi sıralayarak düzenlemeyi kontrol edin. Silme işlemini onaylamak ve düzenlemenin her klonda nasıl gerçekleştirildiğini belirlemek için seçilen klonların Sanger dizilimini gerçekleştirin. Yalnızca hedeflenen genin değiştirildiğinden ve gözlemlenen fizyolojik etkiden sorumlu olduğundan emin olmak için birden fazla düzenlenmiş klon tutun ve sonraki deneylerde kontrol klonu (CTRL) olarak kullanılacak düzenlenmemiş bir klon tutun.
- Seçili klonları genişletin. Her klonun akıcılığı yaklaşık% 50'ye ulaştığında, biyokimyasal ve fonksiyonel karakterizasyonları gerçekleştirmek için yeterli hücre üretilene kadar hücreleri tripsinize edin ve hücreleri daha büyük bir tabakta saklayın (genellikle klon başına 1 x 106'dan fazla) ve her klonun dondurulmuş alikotlarını gelecekte kullanmak üzere saklayın.
8. Düzenlenmiş klonların karakterizasyonu
NOT: Birkaç klon seçildikten ve DNA dizilimi ile doğrulandıktan sonra, hedeflenen genin silinmesi, Western blot kullanılarak protein seviyesinde ve bu gen için fonksiyonel bir hücresel tahlil mevcutsa fonksiyonel düzeyde doğrulanabilir. RYR1-KO durumunda, RyR1 bir kalsiyum kanalı olduğundan, fonksiyonel karakterizasyon, kültürlenmiş hücreler üzerinde kalsiyum görüntüleme kullanılarak gerçekleştirilmiştir.
- Düzenlenmiş klonlarda protein ekspresyonu
NOT: RyR1 sadece farklılaşmış miyotüpler10'da eksprese edilir. İfadesi, RyR1'in protein seviyesindeki delesyonunu ve Cas9 proteininin silinmesini doğrulamak için Western lekesi kullanılarak miyotüplerde değerlendirilmiştir.- Laminin ile kaplanmış 35 mm'lik bir plakada yaklaşık 1.76cm2'lik bir yüzeyde proliferasyon ortamında (yukarıda tarif edilen, adım 5) plaka 200.000 hücre (kalsiyum ile PBS'de 10 mg / mL'de 200 μL'lik bir laminin damlasına karşılık gelen yüzey). Hücreler 37 ° C'de 2-3 saat inkübe edildikten sonra,% 5 CO2 ile plakaya yapıştıktan sonra, kültür ortamını DMEM düşük glikoz +% 10 At serumu +% 1 penisilin / streptomisin'den oluşan bir farklılaşma ortamına kaydırın ve hücreleri 6 gün boyunca inkübatöre geri döndürün.
- 6 günlük farklılaşmadan sonra, proteaz inhibitörleri ile desteklenmiş 200 μL RIPA içeren hücreleri toplayın ve lize edin. Folin Lowry yöntemi11'i kullanarak protein konsantrasyonunu belirleyin.
- Laemmli denatürasyon tamponunda RT'de 30 dakika denatürasyondan sonra,% 5 -% 15 gradyan akrilamid jeli üzerine 15 μg protein yükleyin. Elektroforetik ayırmadan sonra, proteinleri 4 saat11 için 0.8 V'ta Immobilon P üzerine aktarın.
- %0,1 Aralık 20 ve %5 Yağsız kuru süt içeren PBS'de RT'de 30 dakika boyunca membranın doygunluğundan sonra, membranı RT'de 2 saat boyunca aynı tamponda seyreltilmiş birincil antikorlarla veya 4 °C'de bir gecede inkübe edin, membranı PBS-%0,1 Aralık 20 ile 5 dakika boyunca 5x yıkayın ve membranı RT'de 1 saat boyunca ikincil antikorlarla inkübe edin. Kullanılan birincil antikorlar şunlardır: Cas9'u tespit etmek için V5-etiketine karşı antikorlar (seyreltme: 1/5000), yükleme kontrolü olarak anti-GAPDH (seyreltme: 1/1000), anti-RyR1 antikoru 12,13 (seyreltme: 1/10.000), DHPR'nin alfa 1 alt birimine karşı antikor (seyreltme: 1/1000) ve miyozin ağır zincir MF20'ye karşı antikor (seyreltme: 1/1000).
- Membranı 5x PBS-0.1% Tween 20 ile 5 dakika boyunca yıkayın, fazla sıvıyı kurutun ve kemilüminesan substratı ekleyin. Kemilüminesan sinyali tespit etmek için substrat sağlayıcısı tarafından önerildiği gibi ilerleyin.
- Düzenlenmiş klonların fonksiyonel karakterizasyonu
NOT: RyR1'in işlevi, CTRL veya KO klonlarından üretilen diferansiye miyotüplerde kalsiyum görüntüleme kullanılarak değerlendirildi14.- Laminin ile kaplanmış 35 mm'lik tabakların merkezinde0,2 cm2'lik bir yüzey üzerinde 50.000 hücre plakası (kalsiyum içeren PBS'de 10 mg / mL'de 50 μL'lik bir laminin damlası ile kaplanmış yüzey) ve adım 8.1.1'de açıklandığı gibi 6 gün boyunca farklılaşmaya neden olur. Biyolojik bir üçlüye sahip olmak için her stimülasyon için üç plaka hazırlayın.
- Miyotüpleri 50 μL fluo 4-doğrudan ile yükleyin, farklılaşma ortamında 1:1 seyreltin ve 37 ° C'de 30 dakika boyunca inkübe edin. Hücreleri, 1 mg / mL'de glikoz ile desteklenmiş KREBS tamponu ile iki kez durulayın.
- Floresan varyasyonlarını ters çevrilmiş floresan mikroskop veya konfokal mikroskop ile 10x hedef kullanarak ölçün. Plakayı mikroskop aşamasına takın ve edinimi 90 s için saniyede 1 kare hızında başlatın.
- Kalan KREBS'yi çıkarın ve membran depolarizasyonu için 2 mL KCl (140 mM nihai konsantrasyon) veya RyR1 doğrudan stimülasyonu için 2 mL 4 CmC (500 μM nihai konsantrasyon) ekleyerek çerçeve 25'teki hücreleri uyarın. Kaydedilen alanda en az 10 miyotüp bulunduğundan emin olun.
- Özel bir yazılım kullanarak her miyotüpteki floresan varyasyonunu ölçün. Analiz için çanak başına en az 10 miyotüp (ideal olarak çanak başına 20-30 miyotüp) seçin, her miyotüpün uzun eksenine bir çizgi (veya bir İlgi Bölgesi (ROI)) çizin ve tüm çerçeveler için bu çizgi boyunca floresan F'yi toplayın.
- 1 ila 24 arasındaki çerçevelere karşılık gelen ilk floresan değeri olan F0'ı belirleyin. Floresan varyasyonunu (F-F0)/F0'ı 0'dan 90 saniyeye kadar zamanın bir fonksiyonu olarak çizin. Üç farklı kültürden en az 90 miyotüpten floresan varyasyonunu elde etmek için deneyi üç kez tekrarlayın. 90 miyotüp için tüm sonuçları toplayın ve her zaman diliminde (F-F0) / F0'ın ortalama ± SEM'ini hesaplayın. Her stimülasyon ve her klon için kalsiyum salınımının tepe genliğini ölçün.
Sonuçlar
Bu protokol, RyR1 proteinini kodlayan RYR1 genini yok etmek için RyR1'in daha önce16 olarak karakterize edildiği sağlıklı bir denek15'ten (insan miyoblastları için HM hücreleri olarak adlandırılır) ölümsüzleştirilmiş miyoblastlara uygulandı. RNA kılavuzlarının tasarımı, genin ekzon 101 ve intron 101'in bir kısmını kapsayan diziyi silmek için yapıldı. Ekzon 101'in bir kısmının silinmesinin okuma çerçevesinin bozulmasına neden olmas...
Tartışmalar
Patolojilerde yer alan bilinmeyen fonksiyona sahip genlerin karakterizasyonuna giden yolda önemli bir adım, bu genlerin işlevini incelemek için ilgili hücresel modellerin geliştirilmesidir. CRISPR / Cas9 kullanarak gen düzenlemenin kullanımı katlanarak büyüyen bir araştırma alanıdır ve burada sunulduğu gibi nakavt modellerinin geliştirilmesi en yaygın kullanılan uygulamaları arasındadır. Bu bağlamda, burada, ilgilenilen herhangi bir gende bir insan hücre hattı nakavtı geliştirmek için çok yö...
Açıklamalar
Yazarların açıklayacağı bir çıkar çatışması yoktur.
Teşekkürler
Bu çalışma, Association Française contre les myopathies (AFM-Téléthon) ve Auvergne-Rhône Alpes Région'dan (AURA) gelen hibelerle finanse edildi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
Referanslar
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır