Method Article
Kriyo-Elektron Tomogramlarının Derin Öğrenme Tabanlı Segmentasyonu
Bu Makalede
Özet
Bu, bir tomogramın bir kısmını eğitim girdisi olarak kullanarak kriyo-elektron tomogramlarının çok sınıflı segmentasyonu için çok dilimli bir U-Net'i eğitmek için kullanılan bir yöntemdir. Bu ağın diğer tomogramlara nasıl çıkarılacağını ve subtomogram ortalaması ve filament izleme gibi daha ileri analizler için segmentasyonların nasıl çıkarılacağını açıklıyoruz.
Özet
Kriyo-elektron tomografisi (kriyo-ET), araştırmacıların hücreleri yerli, hidratlanmış hallerinde şu anda mümkün olan en yüksek çözünürlükte görüntülemelerini sağlar. Bununla birlikte, tekniğin, ürettiği verileri analiz etmeyi zaman alıcı ve zor hale getiren birkaç sınırlaması vardır. Tek bir tomogramın elle bölümlendirilmesi saatlerden günlere kadar sürebilir, ancak mikroskop günde 50 veya daha fazla tomogramı kolayca üretebilir. Kriyo-ET için mevcut derin öğrenme segmentasyon programları mevcuttur, ancak bir seferde bir yapıyı segmentlere ayırmakla sınırlıdır. Burada, çok kesitli U-Net evrişimli sinir ağları, kriyo-tomogramlar içinde aynı anda birden fazla yapıyı otomatik olarak segmentlere ayırmak için eğitilir ve uygulanır. Uygun ön işleme ile, bu ağlar, her bir tomogram için ayrı ayrı ağları eğitmeye gerek kalmadan birçok tomograma sağlam bir şekilde çıkarılabilir. Bu iş akışı, çoğu durumda segmentasyon süresini 30 dakikanın altına düşürerek kriyo-elektron tomogramlarının analiz edilebileceği hızı önemli ölçüde artırır. Ayrıca, segmentasyonlar, hücresel bir bağlamda filament izlemenin doğruluğunu artırmak ve subtomogram ortalaması için koordinatları hızla çıkarmak için kullanılabilir.
Giriş
Son on yıldaki donanım ve yazılım gelişmeleri, kriyo-elektron mikroskobu (cryo-EM) için bir "çözünürlük devrimi" ile sonuçlanmıştır1,2. Daha iyi ve daha hızlı dedektörler3, veri toplamayı otomatikleştiren yazılım4,5 ve faz plakaları6 gibi sinyal artırıcı ilerlemelerle, büyük miktarlarda yüksek çözünürlüklü kriyo-EM verisi toplamak nispeten basittir.
Cryo-ET, doğal, hidratlanmış bir durumdahücresel ultrayapıya benzeri görülmemiş bir bakış açısı sunar 7,8,9,10. Birincil sınırlama numune kalınlığıdır, ancak tomografi11 için kalın hücresel ve doku örneklerinin inceltildiği odaklanmış iyon ışını (FIB) frezeleme gibi yöntemlerin benimsenmesiyle, kriyo-ET ile görüntülenebilecek ufuk sürekli genişlemektedir. En yeni mikroskoplar günde 50'den fazla tomogram üretebilmektedir ve bu oranın yalnızca hızlı veri toplama şemalarının geliştirilmesi nedeniyle artacağı tahmin edilmektedir12,13. Kriyo-ET tarafından üretilen büyük miktarda verinin analiz edilmesi, bu görüntüleme yöntemi için bir darboğaz olmaya devam etmektedir.
Tomografik bilginin nicel analizi, önce açıklamalı olmasını gerektirir. Geleneksel olarak, bu zaman alıcı olan bir uzman tarafından elle segmentasyon gerektirir; kriyo-tomogramda bulunan moleküler karmaşıklığa bağlı olarak, saatlerce veya günlerce özel dikkat gerektirebilir. Yapay sinir ağları bu soruna çekici bir çözümdür, çünkü segmentasyon çalışmasının büyük kısmını zamanın bir bölümünde yapmak için eğitilebilirler. Evrişimli sinir ağları (CNN'ler) özellikle bilgisayarlı görme görevleri için uygundur14 ve son zamanlarda kriyo-elektron tomogramlarının analizi için uyarlanmıştır15,16,17.
Geleneksel CNN'ler, biyolojik görüntü analizi görevleri için genellikle mümkün olmayan binlerce ek açıklamalı eğitim örneği gerektirir. Bu nedenle, U-Net mimarisi bu alanda18'de mükemmel olmuştur, çünkü ağı başarılı bir şekilde eğitmek için veri artırmaya dayanır ve büyük eğitim kümelerine bağımlılığı en aza indirir. Örneğin, bir U-Net mimarisi tek bir tomogramın yalnızca birkaç dilimi (dört veya beş dilim) ile eğitilebilir ve yeniden eğitilmeden diğer tomogramlara sağlam bir şekilde çıkarılabilir. Bu protokol, Dragonfly 2022.119 içindeki elektron kriyo-tomogramlarını segmentlere ayırmak için U-Net sinir ağı mimarilerini eğitmek için adım adım bir kılavuz sağlar.
Dragonfly, derin öğrenme modelleri tarafından 3D görüntü segmentasyonu ve analizi için kullanılan ticari olarak geliştirilmiş bir yazılımdır ve akademik kullanım için serbestçe kullanılabilir (bazı coğrafi kısıtlamalar geçerlidir). Uzman olmayan bir kişinin hem semantik segmentasyon hem de görüntü gürültüsünü gidermek için derin öğrenmenin güçlerinden tam olarak yararlanmasını sağlayan gelişmiş bir grafik arayüze sahiptir. Bu protokol, yapay sinir ağlarını eğitmek için Dragonfly içindeki kriyo-elektron tomogramlarının nasıl önceden işleneceğini ve ek açıklama ekleneceğini gösterir; bu da daha sonra büyük veri kümelerini hızlı bir şekilde segmentlere ayırmak için çıkarılabilir. Ayrıca, filament izleme ve alt tomogram ortalaması için koordinat ekstraksiyonu gibi daha ileri analizler için segmentlere ayrılmış verilerin nasıl kullanılacağını tartışır ve kısaca gösterir.
Protokol
NOT: Dragonfly 2022.1, yüksek performanslı bir iş istasyonu gerektirir. Sistem önerileri, bu protokol için kullanılan iş istasyonunun donanımıyla birlikte Malzeme Tablosuna dahil edilmiştir. Bu protokolde kullanılan tüm tomogramlar, 3,3 ila 13,2 ang/pix piksel boyutundan 4 kat bağlanır. Temsili sonuçlarda kullanılan örnekler, bu kurumun etik standartlarına uygun hayvan bakım yönergelerini izleyen bir şirketten ( Malzeme Tablosuna bakınız) elde edilmiştir. Bu protokolde kullanılan tomogram ve eğitim girdisi olarak oluşturulan çoklu yatırım getirisi, Ek Dosya 1'e (https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct bulunabilir) paketlenmiş bir veri kümesi olarak dahil edilmiştir, böylece kullanıcı istediği takdirde aynı verilerle birlikte takip edebilir. Dragonfly ayrıca, kullanıcıların eğitilmiş ağları paylaşabilecekleri Infinite Toolbox adlı açık erişimli bir veritabanına da ev sahipliği yapıyor.
1. Kurulum
- Varsayılan çalışma alanını değiştirme:
- Çalışma alanını bu protokolde kullanılanı yansıtacak şekilde değiştirmek için Ana panelin sol tarafında, Sahnenin Görünüm Özellikleri bölümüne gidin ve Göstergeleri Göster'in seçimini kaldırın. Mizanpaj bölümüne ilerleyin ve Tek sahne ile Dört Eşit Görünüm görünümünü seçin.
- Varsayılan birimi güncellemek için Dosya | Tercihler. Açılan pencerede, Varsayılan birimi milimetreden nanometreye değiştirin.
- Yararlı varsayılan tuş bağlantıları:
- Artı işaretlerini 2B görünümlerde görüntülemek ve 3B görünümde 3B ses yüksekliği döndürmeye izin vermek için Esc tuşuna basın. 2B görünümlerde artı işaretlerini gizlemek ve 3B görünümde 2B çeviri ve 3B birim çevirisine izin vermek için X düğmesine basın.
- Diğer 2B görünümlerde görüntüleme düzleminin açısını değiştirmek üzere tıklatılıp sürüklenebilen küçük okları görmek için imleci artı işaretlerinin üzerine getirin.
- Her iki görünümde de yakınlaştırma durumuna girmek için Z tuşuna basın, böylece kullanıcıların yakınlaştırmak ve uzaklaştırmak için herhangi bir yeri tıklatıp sürüklemelerine olanak tanır.
- Dört Görünüm sahnesindeki bir görünüme çift tıklayarak yalnızca o görünüme odaklanın; dört görünümün tümüne dönmek için tekrar çift tıklayın.
- Kolay içe aktarma için Özellikler sekmesindeki her şeyi ORS nesnesi olarak dışa aktararak ilerlemeyi düzenli aralıklarla kaydedin. Listedeki tüm nesneleri seçin ve Dışa Aktar'a sağ tıklayın | ORS'nin itiraz ettiği gibi. Dosyayı adlandırın ve kaydedin. Alternatif olarak, Dosya | Oturumu Kaydet. Yazılımdaki otomatik kaydetme özelliğini kullanmak için, Dosya | Tercihler | Otomatik kaydetme.
2. Görüntü içe aktarma
- Görüntü içe aktarma için Dosya | Görüntü dosyalarını içe aktarın. Ekle'yi tıklayın, görüntü dosyasına gidin ve Aç'ı tıklayın | Sonraki | Bitirin.
NOT: Yazılım .rec dosyalarını tanımıyor. Tüm tomogramlar .mrc sonekine sahip olmalıdır. Sağlanan verileri kullanıyorsanız, bunun yerine Dosya | Nesneleri içe aktarın. Training.ORSObject dosyasına gidin ve Aç'ı ve ardından Tamam'ı tıklayın.
3. Ön işleme (Şekil 1.1)
- Özel bir yoğunluk ölçeği oluşturun (veri kümeleri genelinde görüntü yoğunluklarını kalibre etmek için kullanılır). Yardımcı Programlar'a gidin | Boyut Birimi Yöneticisi. Sol altta, yeni bir Boyut Birimi oluşturmak için + işaretine tıklayın.
- İlgilendiğiniz tüm tomogramlarda bulunan yüksek yoğunluklu (parlak) ve düşük yoğunluklu (karanlık) bir özellik seçin. Birime bir ad ve bir kısaltma verin (örneğin, bu ölçek için referans boncuklarını 0,0 Standart yoğunluğa ve arka planı 100,0 olarak ayarlayın). Özel boyut birimini kaydedin.
NOT: Özel yoğunluk ölçeği, farklı zamanlarda veya farklı ekipmanlarda toplanmasına rağmen tüm verilerin aynı yoğunluk ölçeğinde olmasını sağlamak için oluşturulan ve verilere uygulanan rasgele bir ölçektir. Sinyalin içine düştüğü aralığı en iyi temsil eden açık ve koyu özellikleri seçin. Verilerde herhangi bir referans yoksa, bölümlere ayrılacak en karanlık özelliği seçin (örneğin, proteinin en karanlık bölgesi). - Görüntüleri özel yoğunluk ölçeğine göre kalibre etmek için, ekranın sağ tarafındaki Özellikler sütununda veri kümesine sağ tıklayın ve Yoğunluk Ölçeğini Kalibre Et'i seçin. Ekranın sol tarafındaki Ana sekmesinde, Prob bölümüne ilerleyin. Dairesel prob aletini uygun bir çapa sahip kullanarak, tomogramın arka plan bölgesinde birkaç yere tıklayın ve ortalama sayıyı Ham Yoğunluk sütununa kaydedin; Referans işaretleyicileri için bu işlemi tekrarlayın, ardından Kalibre Et'i tıklayın. Gerekirse, Ana sekmenin Pencere Tesviye bölümündeki Alan aracıyla yapıları yeniden görünür hale getirmek için kontrastı ayarlayın.
- Görüntü filtreleme:
NOT: Görüntü filtreleme gürültüyü azaltabilir ve sinyali artırabilir. Bu protokol, bu veriler için en iyi şekilde çalıştıkları için yazılımda yerleşik olarak bulunan üç filtre kullanır, ancak kullanılabilir birçok filtre vardır. İlgilenilen veriler için bir görüntü filtreleme protokolüne karar verildikten sonra, segmentasyondan önce tüm tomogramlara tam olarak aynı protokolün uygulanması gerekecektir.- Sol taraftaki ana sekmede, Görüntü İşleme Paneline doğru aşağı kaydırın. Gelişmiş'i tıklatın ve yeni bir pencerenin açılmasını bekleyin. Özellikler panelinden filtrelenecek veri kümesini seçin ve veri kümesinin solundaki göz simgesini tıklatarak görünür hale getirin.
- İşlemler panelinden açılır menüyü kullanarak ilk işlem için Histogram Eşitleme'yi (Kontrast bölümünün altında) seçin. İşlem Ekle'yi seçin | Gauss dili (Yumuşatma bölümünün altında). Çekirdek boyutunu 3B olarak değiştirin.
- Üçüncü bir işlem ekleyin; tıklayın, sonra Netliği Kaldır'ı seçin ( Keskinleştirme bölümünün altında). Bunun için çıktıyı bırakın. Tüm dilimlere uygulayın ve filtrelemenin çalışmasına izin verin, ardından ana arayüze dönmek için Görüntü İşleme penceresini kapatın.
4. Eğitim verileri oluşturma (Şekil 1.2)
- Filtrelenmemiş veri kümesini gizleyerek önce Veri Özellikleri panelinde solundaki göz simgesini tıklatarak eğitim alanını tanımlayın. Ardından, yeni filtrelenen veri kümesini gösterin (otomatik olarak DataSet-HistEq-Gauss-Unsharp olarak adlandırılır). Filtrelenmiş veri kümesini kullanarak, tomogramın ilgilenilen tüm özellikleri içeren bir alt bölgesini tanımlayın.
- İlgilenilen bölgenin etrafında bir kutu oluşturmak için, sol tarafta, ana sekmede, Şekiller kategorisine gidin ve Kutu Oluştur'u seçin. Dört Görünüm panelindeyken, tüm boyutlarda yalnızca ilgilenilen bölgeyi içine almak için kutunun kenarlarını yönlendirmeye/sürüklemeye yardımcı olması için farklı 2B düzlemleri kullanın. Veri listesinde, Kutu bölgesini seçin ve göz simgesinin yanındaki gri kareyi tıklatarak daha kolay görüntülemek için kenarlığın rengini değiştirin.
NOT: 2D U-Net için en küçük yama boyutu 32 x 32 pikseldir; 400 x 400 x 50 piksel, başlamak için makul bir kutu boyutudur. - Çoklu yatırım getirisi oluşturmak için sol tarafta Segmentasyon sekmesini seçin | Yeni'yi işaretleyin ve Çoklu Yatırım Getirisi Olarak Oluştur'u işaretleyin. Sınıf sayısının, ilgilenilen özelliklerin sayısına + bir arka plan sınıfına karşılık geldiğinden emin olun. Çoklu yatırım getirisi Eğitim Verilerini adlandırın ve Tamam'a tıklamadan önce geometrinin veri kümesine karşılık geldiğinden emin olun.
- Eğitim verilerini segmentlere ayırma
- Kutulu bölgenin sınırları içine girene kadar veriler arasında gezinin. Sağdaki özellikler menüsünden Çoklu Yatırım Getirisi'ni seçin. Çoklu yatırım getirisindeki ilk boş sınıf adını çift tıklatarak adlandırın.
- 2D fırça ile boyayın. Soldaki segmentasyon sekmesinde, 2B araçlara gidin ve dairesel bir fırça seçin. Ardından, açılır menüden Adaptive Gaussian veya Local OTSU'yu seçin. Boyama yapmak için sol ctrl tuşunu basılı tutun ve tıklayın. Silmek için sol üst karakteri basılı tutun ve tıklayın.
NOT: Fırça, o anda seçili olan sınıfın rengini yansıtır. - Çoklu yatırım getirisindeki her nesne sınıfı için önceki adımı yineleyin. Kutulu bölgedeki tüm yapıların tamamen bölümlere ayrıldığından veya ağ tarafından arka plan olarak kabul edileceğinden emin olun.
- Tüm yapılar etiketlendiğinde, Multi-ROI'de Background sınıfına sağ tıklayın ve Tüm Etiketlenmemiş Vokselleri Sınıfa Ekle'yi seçin.
- Maske adlı yeni bir tek sınıf yatırım getirisi oluşturun. Geometrinin filtrelenmiş veri kümesine ayarlandığından emin olun ve ardından uygula'ya tıklayın. Sağdaki özellikler sekmesinde, Kutuya sağ tıklayın ve YG'ye Ekle'yi seçin. Maske YG'sine ekleyin.
- Maskeyi kullanarak eğitim verilerini kırpmak için Özellikler sekmesinde, Ctrl tuşunu basılı tutup her birine tıklayarak hem Eğitim Verileri çoklu yatırım getirisini hem de Maske YG'sini seçin. Ardından, Boole işlemleri etiketli bölümdeki veri özellikleri listesinin altındaki Kesişim'i tıklatın. Yeni veri kümesini Kırpılmış Eğitim Girişi olarak adlandırın ve Tamam'a tıklamadan önce geometrinin filtrelenmiş veri kümesine karşılık geldiğinden emin olun.
5. Yinelemeli eğitim için segmentasyon sihirbazını kullanma (Şekil 1.3)
- Önce Özellikler sekmesinde filtrelenmiş veri kümesini sağ tıklatıp ardından Segmentasyon Sihirbazı seçeneğini belirleyerek eğitim verilerini segmentasyon sihirbazına aktarın. Yeni bir pencere açıldığında, sağ taraftaki giriş sekmesini arayın. Çoklu Yatırım Getirisinden Çerçeveleri İçe Aktar'a tıklayın ve Kırpılmış Eğitim Girişi'ni seçin.
- (İsteğe bağlı) Eğitim ilerlemesini gerçek zamanlı olarak izlemek için bir Görsel Geri Bildirim Çerçevesi oluşturun.
- Segmentlere ayrılmamış verilerden bir çerçeve seçin ve yeni bir çerçeve olarak eklemek için + işaretini tıklayın. Çerçevenin sağındaki karışık etiketi çift tıklatın ve İzleme olarak değiştirin.
- Yeni bir Sinir Ağı Modeli oluşturmak için, Modeller sekmesinde sağ tarafta, yeni bir model oluşturmak için + düğmesine tıklayın. Listeden U-Net'i seçin ve ardından giriş ölçümlendirmesi için 2,5B ve 5 dilimleri seçip Oluştur'a tıklayın.
- Ağı eğitmek için, SegWiz penceresinin sağ alt köşesindeki Eğit'e tıklayın.
NOT: Eğitim, ilerlemeyi kaybetmeden erken durdurulabilir. - Yeni kareleri segmentlere ayırmak üzere eğitilmiş ağı kullanmak için U-Net eğitimi tamamlandığında yeni bir çerçeve oluşturun ve Tahmin Et'i (sağ altta) tıklayın. Ardından, segmentasyonu gerçek kareye aktarmak için tahmin edilen karenin sağ üst köşesindeki Yukarı okunu tıklayın.
- Tahmini düzeltmek için, Ctrl tuşunu basılı tutarak iki sınıfı tıklatarak birinin parçalanmış piksellerini diğerine değiştirin. Her iki sınıfı da seçin ve yalnızca her iki sınıfa ait pikselleri boyamak için fırçayla boyayın. Segmentasyonu en az beş yeni çerçevede düzeltin.
NOT: Her iki sınıf da seçiliyken fırça aracıyla boyama, normalde olduğu gibi shift tuşunu basılı tutarak silmek yerine, birinci sınıfın piksellerini ikinci sınıfa dönüştüreceği anlamına gelir. Ctrl tuşunu basılı tutarak tıklamak tersini gerçekleştirir. - Yinelemeli eğitim için, Eğit düğmesine tekrar tıklayın ve ağın 30-40 dönem daha daha fazla eğitim almasına izin verin, bu noktada eğitimi durdurun ve başka bir eğitim turu için 4.5 ve 4.6 adımlarını tekrarlayın.
NOT: Bu şekilde, bir model tek bir veri kümesi kullanılarak yinelemeli olarak eğitilebilir ve geliştirilebilir. - Ağı yayımlamak için, performansından memnun kaldığınızda, Segmentasyon Sihirbazı'ndan çıkın. Hangi modellerin yayınlanacağını (kaydedileceğini) soran otomatik olarak açılan iletişim kutusunda, başarılı ağı seçin, adlandırın ve ardından ağı segmentasyon sihirbazı dışında kullanılabilir hale getirmek için yayınlayın .
6. Ağı uygulama (Şekil 1.4)
- Önce eğitim tomogramına uygulamak için Özellikler panelinde filtrelenmiş veri kümesini seçin. Soldaki Segmentasyon panelinde, AI ile segmentasyon bölümüne ilerleyin. Doğru veri kümesinin seçildiğinden emin olun, açılır menüde yeni yayınlanan modeli seçin, ardından Segmentlere Ayır | Tüm Dilimler. Alternatif olarak, segmentasyonun tek dilimli önizlemesini görüntülemek için Önizleme'yi seçin.
- Bir çıkarım veri kümesine uygulamak için yeni tomogramı içeri aktarın. Adım 3'e göre ön işlem (Şekil 1.1). Segmentasyon panelinde, AI ile segment bölümüne gidin. Yeni filtrelenen tomogramın seçilen veri kümesi olduğundan emin olma, önceden eğitilmiş modeli seçin ve Segmentlere Ayır | Tüm Dilimler.
7. Segmentasyon manipülasyonu ve temizliği
- Önce gürültüyü ve ilgi özelliğini bölümlere ayıran sınıflardan birini seçerek gürültüyü hızla temizleyin. Sağ tıklayın | Process Adaları | Voxel Count tarafından kaldır | Bir voksel boyutu seçin. Küçük başlayın (~ 200) ve gürültünün çoğunu gidermek için sayıyı kademeli olarak artırın.
- Segmentasyon düzeltmesi için, yalnızca bu sınıflara ait pikselleri boyamak üzere iki sınıfı Ctrl tuşunu basılı tutarak tıklatın. İkinci sınıfın piksellerini birinci sınıfa değiştirmek için segmentasyon araçlarıyla Ctrl tuşunu basılı tutarak tıklayın + sürükleyin ve tam tersini gerçekleştirmek için Shift tuşunu basılı tutarak tıklayın + sürükleyin. Yanlış etiketlenmiş pikselleri hızlı bir şekilde düzeltmek için bunu yapmaya devam edin.
- Bağlı bileşenleri ayırın.
- Bir sınıf seçin. Çoklu Yatırım Getirisi'nde bir sınıfa sağ tıklayın | Aynı sınıfın başka bir bileşenine bağlı olmayan her bileşen için yeni bir sınıf oluşturmak üzere Ayrı Bağlı Bileşenler. Sınıfları kolayca birleştirmek için Multi-ROI'in altındaki düğmeleri kullanın.
- Yatırım getirisini İkili/TIFF olarak dışa aktarın.
- Multi-ROI'de bir sınıf seçin, ardından sağ tıklayın ve Sınıfı ROI olarak Ayıklayın. Yukarıdaki özellikler panelinde yeni YG'yi seçin, sağ tıklayın | İhracat | İkili olarak YG (tüm görüntüleri tek bir dosyaya dışa aktarma seçeneğinin seçili olduğundan emin olun).
NOT: Kullanıcılar IMOD programı tif2mrc 20'yi kullanarak tiff'ten mrc formatına kolaycadönüştürebilirler. Bu, filament izleme için kullanışlıdır.
- Multi-ROI'de bir sınıf seçin, ardından sağ tıklayın ve Sınıfı ROI olarak Ayıklayın. Yukarıdaki özellikler panelinde yeni YG'yi seçin, sağ tıklayın | İhracat | İkili olarak YG (tüm görüntüleri tek bir dosyaya dışa aktarma seçeneğinin seçili olduğundan emin olun).
8. ROI'den alt tomogram ortalaması için koordinatlar oluşturma
- Bir sınıf ayıklayın.
- Ortalama almak için kullanılacak sınıfa sağ tıklayın | Extract Class as ROI. Sağ tıklama sınıfı YG | Bağlı Bileşenler | Yeni Çoklu Yatırım Getirisi (26 bağlı).
- Koordinatlar oluşturun.
- Yeni Çoklu Yatırım Getirisi'ne sağ tıklayın | Skaler Jeneratör. Veri Kümesiyle Temel Ölçümler'i genişletin | X, Y ve Z kütlelerinin ağırlıklı merkezini kontrol edin. Veri kümesini ve işlemi seçin. Çoklu Yatırım Getirisi | Skaler değerleri dışa aktarın. Tüm skaler yuvaları seç'i işaretleyin, ardından çoklu yatırım getirisindeki her sınıf için merkezcil dünya koordinatlarını CSV dosyası olarak oluşturmak üzere Tamam'ı işaretleyin.
NOT: Parçacıklar birbirine yakınsa ve segmentasyonlar temas ediyorsa, bileşenleri çoklu yatırım getirisine ayırmak için bir havza dönüşümü gerçekleştirmek gerekebilir.
- Yeni Çoklu Yatırım Getirisi'ne sağ tıklayın | Skaler Jeneratör. Veri Kümesiyle Temel Ölçümler'i genişletin | X, Y ve Z kütlelerinin ağırlıklı merkezini kontrol edin. Veri kümesini ve işlemi seçin. Çoklu Yatırım Getirisi | Skaler değerleri dışa aktarın. Tüm skaler yuvaları seç'i işaretleyin, ardından çoklu yatırım getirisindeki her sınıf için merkezcil dünya koordinatlarını CSV dosyası olarak oluşturmak üzere Tamam'ı işaretleyin.
9. Havza dönüşümü
- Ortalama almak için kullanılacak Çoklu Yatırım Getirisi'nde sınıfa sağ tıklayarak sınıfı ayıklayın | Extract Class as ROI. Buna ROI Watershed Mask adını verin.
- (İsteğe bağlı) Delikleri kapatın.
- Parçalanmış parçacıkların delikleri veya açıklıkları varsa, bunları havza için kapatın. Veri Özellikleri'nde YG'yi tıklatın. Segmentasyon sekmesinde (solda), Morfolojik İşlemler'e gidin ve deliksiz katı segmentasyonlar elde etmek için gerekli olan Dilate, Erode ve Close kombinasyonunu kullanın.
- YG'ye tıklayarak yatırım getirisini tersine çevirme | Seçili nesneyi kopyalayın (veri özelliklerinin altında). Kopyalanan YG'yi seçin ve Segmentasyon sekmesinde sol tarafta Ters Çevir'i tıklayın.
- Ters çevrilmiş YG'ye sağ tıklayarak bir mesafe haritası oluşturun | Eşleme Oluştur | Mesafe Haritası. Daha sonra kullanmak için, mesafe haritasının bir kopyasını oluşturun ve ters çevirin (sağ tıklayın | Değiştir ve Dönüştür | Değerleri Tersine Çevir | Uygula). Bu ters çevrilmiş haritayı Manzara olarak adlandırın.
- Tohum noktaları oluşturun.
- Yatırım getirisini gizleyin ve Mesafe Haritası'nı görüntüleyin. Segmentasyon sekmesinde, Aralık Tanımla'yı tıklayın ve her noktanın ortasındaki yalnızca birkaç piksel vurgulanana ve hiçbiri başka bir noktaya bağlanmayana kadar aralığı azaltın. Aralık bölümünün en altında, Yeni'ye Ekle'yi tıklayın. Bu yeni ROI Seedpoints adını verin.
- Havza dönüşümü gerçekleştirin.
- Seedpoints YG'sine sağ tıklayın | Bağlı Bileşenler | Yeni Çoklu Yatırım Getirisi (26 bağlı). Yeni oluşturulan Çoklu Yatırım Getirisi'ne sağ tıklayın | Havza Dönüşümü. Manzara adlı mesafe haritasını seçin ve Tamam'ı tıklatın; Watershed Mask adlı ROI'yi seçin ve her bir tohum noktasından bir havza dönüşümü hesaplamak ve bireysel parçacıkları çoklu ROI'de ayrı sınıflara ayırmak için Tamam'ı tıklayın. Adım 8.2'deki gibi koordinatlar oluşturun.
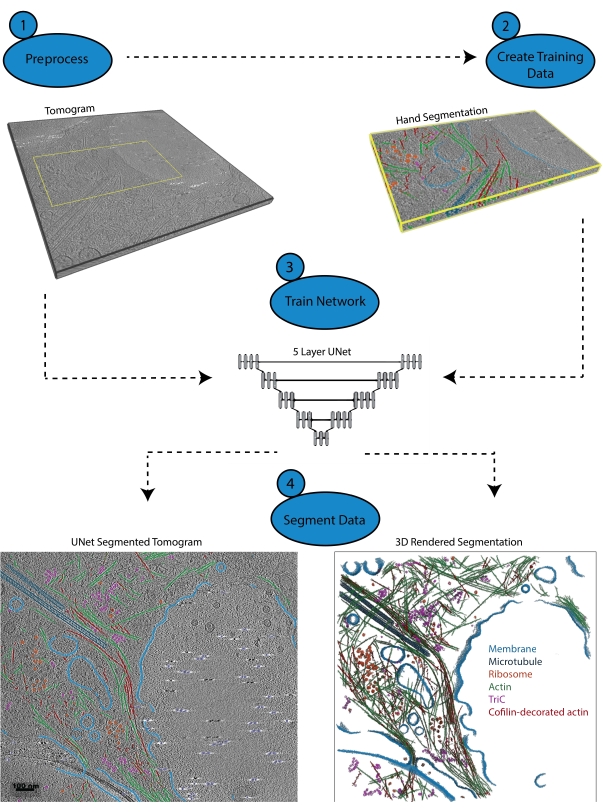
Şekil 1: İş akışı. 1) Yoğunluk ölçeğini kalibre ederek ve veri kümesini filtreleyerek eğitim tomogramını önceden işleyin. 2) Bir tomogramın küçük bir kısmını, kullanıcının tanımlamak istediği tüm uygun etiketlerle elle segmentlere ayırarak eğitim verilerini oluşturun. 3) Giriş olarak filtrelenmiş tomogramı ve eğitim çıkışı olarak el segmentasyonunu kullanarak, segmentasyon sihirbazında beş katmanlı, çok dilimli bir U-Net eğitilir. 4) Eğitilen ağ, açıklama eklemek için tam tomograma uygulanabilir ve her bölümlenmiş sınıftan bir 3D oluşturma oluşturulabilir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Sonuçlar
Protokolü takiben, beş parçalı bir U-Net, beş sınıfı tanımlamak için tek bir tomogram (Şekil 2A) üzerinde eğitildi: Membran, Mikrotübüller, Aktin, Referans belirteçleri ve Arka Plan. Ağ yinelemeli olarak toplam üç kez eğitildi ve daha sonra tomogramı tamamen segmentlere ayırmak ve açıklama eklemek için tomograma uygulandı (Şekil 2B, C). En az temizleme işlemi 7.1 ve 7.2 numaralı adımlar kullanılarak gerçekleştirilmiştir. Sonraki üç ilgi çekici tomogram (Şekil 2D, G, J) ön işleme için yazılıma yüklendi. Görüntü içe aktarmadan önce, tomogramlardan biri (Şekil 2J), farklı bir mikroskopta biraz farklı bir büyütmede toplandığı için 17.22 ş/ px'ten 13.3 ş/ px'e piksel boyutu ayarlaması gerektiriyordu. IMOD program squeezevol aşağıdaki komutla yeniden boyutlandırmak için kullanılmıştır:
'squeezevol -f 0.772 inputfile.mrc outputfile.mrc'
Bu komutta -f , piksel boyutunun değiştirileceği faktörü ifade eder (bu durumda: 13.3/17.22). İçe aktarma işleminden sonra, üç çıkarım hedefinin tümü 3.2 ve 3.3 numaralı adımlara göre önceden işlendi ve ardından beş dilimli U-Net uygulandı. Minimum temizlik tekrar yapıldı. Son segmentasyonlar Şekil 2'de gösterilmiştir.
Her tomogramdan mikrotübül segmentasyonları ikili (adım 7.4) TIF dosyaları olarak dışa aktarıldı, MRC'ye (IMOD tif2mrc programı) dönüştürüldü ve daha sonra silindir korelasyonu ve filament izleme için kullanıldı. Filamentlerin ikili segmentasyonları, tomogramlar üzerinde izlemekten çok daha sağlam filament izleme ile sonuçlanır. Filament izlemeden koordinat haritaları (Şekil 3), mikrotübül oryantasyonunu belirlemek için en yakın komşu ölçümleri (filament paketleme) ve tek filamentler boyunca sarmal alt tomogram ortalaması gibi daha ileri analizler için kullanılacaktır.
Başarısız veya yetersiz eğitilmiş ağların belirlenmesi kolaydır. Başarısız bir ağ herhangi bir yapıyı segmentlere ayıramazken, yetersiz eğitilmiş bir ağ tipik olarak bazı yapıları doğru bir şekilde bölümlere ayırır ve önemli sayıda yanlış pozitif ve yanlış negatife sahip olur. Bu ağlar, performanslarını artırmak için düzeltilebilir ve yinelemeli olarak eğitilebilir. Segmentasyon sihirbazı, eğitildikten sonra modelin zar benzerlik katsayısını (SegWiz'de skor olarak adlandırılır) otomatik olarak hesaplar. Bu istatistik, eğitim verileri ile U-Net segmentasyonu arasındaki benzerliğin bir tahminini verir. Dragonfly 2022.1 ayrıca, arayüzün üst kısmındaki Yapay Zeka sekmesinden erişilebilen bir modelin performansını değerlendirmek için yerleşik bir araca sahiptir (kullanım için belgelere bakın).
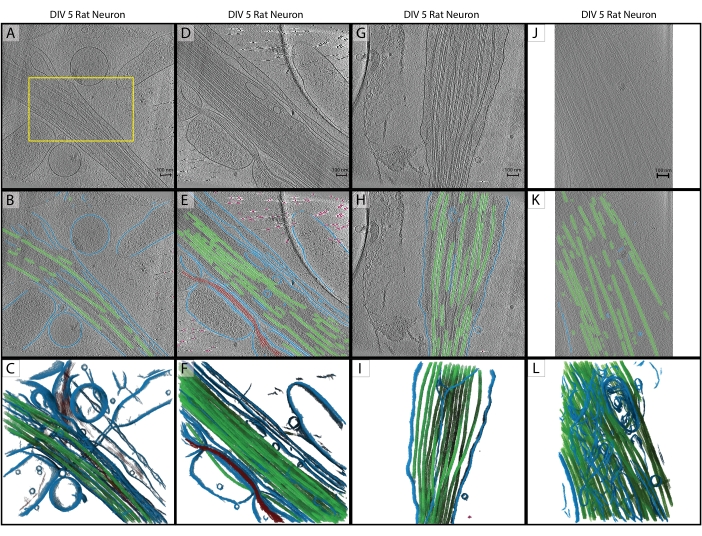
Şekil 2: Çıkarım. (A-C) 2019 yılında bir Titan Krios üzerinde toplanan bir DIV 5 hipokampal sıçan nöronunun orijinal eğitim tomogramı. Bu, IMOD'de CTF düzeltmesi ile geriye dönük bir rekonstrüksiyondur. (A) Sarı kutu, eğitim girişi için el segmentasyonunun yapıldığı bölgeyi temsil eder. (B) Eğitim tamamlandıktan sonra U-Net'ten 2D segmentasyon. (C) Membran (mavi), mikrotübüller (yeşil) ve aktin (kırmızı) gösteren parçalanmış bölgelerin 3D görüntüsü. (D-F) DIV 5 hipokampal sıçan nöronu, eğitim tomogramı ile aynı seanstan. (E) Ek eğitim ve hızlı temizlik olmadan U-Net'ten 2D segmentasyon. Membran (mavi), mikrotübüller (yeşil), aktin (kırmızı), referanslar (pembe). (F) Bölümlere ayrılmış bölgelerin 3B olarak oluşturulması. (G-I) 2019 oturumundan DIV 5 hipokampal sıçan nöronu. (H) Hızlı temizleme ve (I) 3B oluşturma ile U-Net'ten 2B segmentasyon. (J-L) DIV 5 hipokampal sıçan nöronu, 2021 yılında farklı bir Titan Krios üzerinde farklı bir büyütmede toplandı. Piksel boyutu, eğitim tomogramına uyacak şekilde IMOD programı squeezevol ile değiştirildi. (K) Hızlı temizleme ile U-Net'ten 2B segmentasyon, uygun ön işleme ve (L) segmentasyonun 3B oluşturulması ile veri kümeleri arasında sağlam çıkarım gösterir. Ölçek çubukları = 100 nm. Kısaltmalar: DIV = gün in vitro; CTF = kontrast transfer fonksiyonu. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
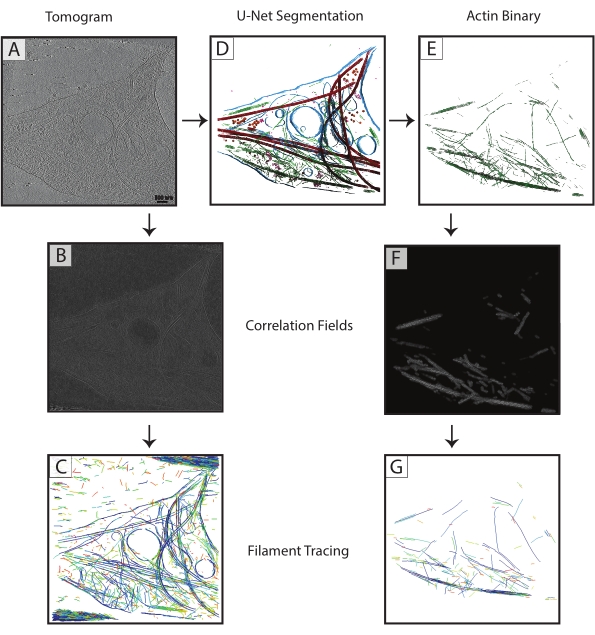
Şekil 3: Filament izleme iyileşmesi . (A) Titan Krios üzerinde toplanan bir DIV 4 sıçan hipokampal nöronunun Tomogramı. (B) Aktin filamentleri üzerindeki silindir korelasyonundan elde edilen korelasyon haritası. (C) Parametreleri tanımlamak için korelasyon haritasındaki aktin filamentlerinin yoğunluklarını kullanarak aktinin filament takibi. İzleme, membranı ve mikrotübülleri ve gürültüyü yakalarken, sadece aktini izlemeye çalışır. (D) Tomogramın U-Net segmentasyonu. Mavi renkte vurgulanan membran, kırmızı renkte mikrotübüller, turuncu renkte ribozomlar, morda triC ve yeşil renkte aktin. (E) Filament izleme için ikili maske olarak ekstrakte edilen aktin segmentasyonu. (F) Silindir korelasyonundan (B)'den aynı parametrelerle oluşturulan korelasyon haritası. (G) Tomogramdan sadece aktin filamentlerinin filament takibi önemli ölçüde iyileştirildi. Kısaltma: DIV = gün in vitro. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Ek Dosya 1: Bu protokolde kullanılan tomogram ve eğitim girdisi olarak oluşturulan çoklu yatırım getirisi, paketlenmiş bir veri kümesi (Training.ORSObject) olarak dahil edilir. Bkz. https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct.
Tartışmalar
Bu protokol, tek bir tomogramdan çok sınıflı bir U-Net'i eğitmek için Dragonfly 2022.1 yazılımını kullanmak için bir prosedür ve bu ağın aynı veri kümesinden olması gerekmeyen diğer tomogramlara nasıl çıkarılacağını ortaya koymaktadır. Eğitim nispeten hızlıdır (tamamen eğitilen ağa ve kullanılan donanıma bağlı olarak, dönem başına 3-5 dakika kadar hızlı veya birkaç saat kadar yavaş olabilir) ve öğrenmesini geliştirmek için bir ağı yeniden eğitmek sezgiseldir. Ön işleme adımları her tomogram için gerçekleştirildiği sürece, çıkarım tipik olarak sağlamdır.
Tutarlı ön işleme, derin öğrenme çıkarımı için en kritik adımdır. Yazılımda birçok görüntüleme filtresi vardır ve kullanıcı belirli veri kümeleri için hangi filtrelerin en iyi şekilde çalıştığını belirlemek için denemeler yapabilir; eğitim tomogramında kullanılan filtreleme ne olursa olsun, çıkarım tomogramlarına aynı şekilde uygulanması gerektiğini unutmayın. Ağa doğru ve yeterli eğitim bilgisi sağlamak için de özen gösterilmelidir. Eğitim dilimleri içinde bölümlere ayrılmış tüm özelliklerin mümkün olduğunca dikkatli ve hassas bir şekilde bölümlere ayrılması hayati önem taşımaktadır.
Görüntü segmentasyonu, sofistike bir ticari sınıf kullanıcı arayüzü ile kolaylaştırılmıştır. El segmentasyonu için gerekli tüm araçları sağlar ve voksellerin eğitim ve yeniden eğitimden önce herhangi bir sınıftan diğerine kolayca yeniden atanmasını sağlar. Kullanıcının vokselleri tomogramın tüm bağlamında elle segmentlere ayırmasına izin verilir ve bunlara birden fazla görünüm ve ses seviyesini serbestçe döndürme yeteneği verilir. Ek olarak, yazılım, daha iyi performans gösterme eğiliminde olan16 ve birden fazla tek sınıflı ağ ile segmentasyondan daha hızlı olan çok sınıflı ağları kullanma yeteneği sağlar.
Tabii ki, bir sinir ağının yeteneklerinde sınırlamalar vardır. Cryo-ET verileri, doğası gereği, açısal örneklemede çok gürültülü ve sınırlıdır, bu da aynı nesnelerde yöne özgü bozulmalara yol açar21. Eğitim, yapıları doğru bir şekilde elle segmentlere ayırmak için bir uzmana dayanır ve başarılı bir ağ yalnızca verilen eğitim verileri kadar iyidir (veya kötüdür). Sinyali artırmak için görüntü filtreleme eğitmen için yararlıdır, ancak belirli bir yapının tüm piksellerini doğru bir şekilde tanımlamanın zor olduğu birçok durum vardır. Bu nedenle, eğitim segmentasyonunu oluştururken büyük özen gösterilmesi önemlidir, böylece ağ eğitim sırasında öğrenilebilecek en iyi bilgiye sahip olur.
Bu iş akışı her kullanıcının tercihine göre kolayca değiştirilebilir. Tüm tomogramların tam olarak aynı şekilde önceden işlenmesi gerekli olsa da, protokolde kullanılan filtrelerin tam olarak kullanılması gerekli değildir. Yazılımın çok sayıda görüntü filtreleme seçeneği vardır ve birçok tomogramı kapsayan büyük bir segmentasyon projesine başlamadan önce bunları kullanıcının özel verileri için optimize etmeniz önerilir. Kullanılabilecek epeyce ağ mimarisi de vardır: Çok dilimli bir U-Net'in bu laboratuvardan gelen veriler için en iyi şekilde çalıştığı bulunmuştur, ancak başka bir kullanıcı başka bir mimarinin (3D U-Net veya Sensor 3D gibi) daha iyi çalıştığını görebilir. Segmentasyon sihirbazı, aynı eğitim verilerini kullanarak birden çok ağın performansını karşılaştırmak için kullanışlı bir arabirim sağlar.
Burada sunulanlar gibi araçlar, tam tomogramların elle bölümlendirilmesini geçmişin bir görevi haline getirecektir. Sağlam bir şekilde çıkarılabilir iyi eğitilmiş sinir ağlarıyla, tomografik verilerin mikroskopun toplayabildiği kadar hızlı bir şekilde yeniden yapılandırıldığı, işlendiği ve tamamen bölümlere ayrıldığı bir iş akışı oluşturmak tamamen mümkündür.
Açıklamalar
Bu protokol için açık erişim lisansı Object Research Systems tarafından ödendi.
Teşekkürler
Bu çalışma, Penn State College of Medicine ve Biyokimya ve Moleküler Biyoloji Bölümü ile Tütün Uzlaşma Fonu (TSF) hibesi 4100079742-EXT tarafından desteklenmiştir. Bu projede kullanılan CryoEM ve CryoET Core (RRID: SCR_021178) hizmetleri ve araçları, kısmen, Pennsylvania Eyalet Üniversitesi Tıp Fakültesi tarafından, Araştırma ve Lisansüstü Öğrenciler Dekan Yardımcısı Ofisi ve Pennsylvania Sağlık Bakanlığı aracılığıyla Tütün Yerleşim Fonları (CURE) kullanılarak finanse edilmiştir. İçerik yalnızca yazarların sorumluluğundadır ve mutlaka Üniversite veya Tıp Fakültesi'nin resmi görüşlerini temsil etmemektedir. Pennsylvania Sağlık Bakanlığı, herhangi bir analiz, yorum veya sonuç için özellikle sorumluluk kabul etmez.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Dragonfly 2022.1 | Object Research Systems | https://www.theobjects.com/dragonfly/index.html | |
| E18 Rat Dissociated Hippocampus | Transnetyx Tissue | KTSDEDHP | https://tissue.transnetyx.com/faqs |
| IMOD | University of Colorado | https://bio3d.colorado.edu/imod/ | |
| Intel® Xeon® Gold 6124 CPU 3.2GHz | Intel | https://www.intel.com/content/www/us/en/products/sku/120493/intel-xeon-gold-6134-processor-24-75m-cache-3-20-ghz/specifications.html | |
| NVIDIA Quadro P4000 | NVIDIA | https://www.nvidia.com/content/dam/en-zz/Solutions/design-visualization/productspage/quadro/quadro-desktop/quadro-pascal-p4000-data-sheet-a4-nvidia-704358-r2-web.pdf | |
| Windows 10 Enterprise 2016 | Microsoft | https://www.microsoft.com/en-us/evalcenter/evaluate-windows-10-enterprise | |
| Workstation Minimum Requirements | https://theobjects.com/dragonfly/system-requirements.html |
Referanslar
- Bai, X. -C., Mcmullan, G., Scheres, S. H. W. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- de Oliveira, T. M., van Beek, L., Shilliday, F., Debreczeni, J., Phillips, C. Cryo-EM: The resolution revolution and drug discovery. SLAS Discovery. 26 (1), 17-31 (2021).
- Danev, R., Yanagisawa, H., Kikkawa, M. Cryo-EM performance testing of hardware and data acquisition strategies. Microscopy. 70 (6), 487-497 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Tomography 5 and Tomo Live Software User-friendly batch acquisition for and on-the-fly reconstruction for cryo-electron tomography Datasheet. , Available from: https://assets.thermofisher.com/TFS-Assets/MSD/Datasheets/tomography-5-software-ds0362.pdf (2022).
- Danev, R., Baumeister, W. Expanding the boundaries of cryo-EM with phase plates. Current Opinion in Structural Biology. 46, 87-94 (2017).
- Hylton, R. K., Swulius, M. T. Challenges and triumphs in cryo-electron tomography. iScience. 24 (9), (2021).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Oikonomou, C. M., Jensen, G. J. Cellular electron cryotomography: Toward structural biology in situ. Annual Review of Biochemistry. 86, 873-896 (2017).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-the cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Lam, V., Villa, E. Practical approaches for Cryo-FIB milling and applications for cellular cryo-electron tomography. Methods in Molecular Biology. 2215, 49-82 (2021).
- Chreifi, G., Chen, S., Metskas, L. A., Kaplan, M., Jensen, G. J. Rapid tilt-series acquisition for electron cryotomography. Journal of Structural Biology. 205 (2), 163-169 (2019).
- Eisenstein, F., Danev, R., Pilhofer, M. Improved applicability and robustness of fast cryo-electron tomography data acquisition. Journal of Structural Biology. 208 (2), 107-114 (2019).
- Esteva, A., et al. Deep learning-enabled medical computer vision. npj Digital Medicine. 4 (1), (2021).
- Liu, Y. -T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nature Methods. 14 (10), 983-985 (2017).
- Ronneberger, O., Fischer, P., Brox, T. U-net: Convolutional networks for biomedical image segmentation. Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 9351, 234-241 (2015).
- Dragonfly 2021.3 (Computer Software). , Available from: http://www.theobjects.com/dragonfly (2021).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iancu, C. V., et al. A "flip-flop" rotation stage for routine dual-axis electron cryotomography. Journal of Structural Biology. 151 (3), 288-297 (2005).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır