Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
TACI: 3D Kalsiyum Görüntüleme Analizi için Bir ImageJ Eklentisi
Bu Makalede
Özet
TrackMate Kalsiyum Görüntüleme Analizi (TACI), z eksenindeki hareketi inceleyen ve bir hücrenin yoğunluğunu karşılık gelen zaman noktasında temsil etmek için her z-yığınının maksimum değerini tanımlayan 3D kalsiyum görüntüleme analizi için açık kaynaklı bir ImageJ eklentisidir. Yanal (x / y) yönde üst üste binen nöronları, ancak farklı z-düzlemlerinde ayırabilir.
Özet
Sinirbilimdeki araştırmalar, veri kümelerinden kapsamlı bilgiler çıkarmak için karmaşık görüntüleme ve hesaplama araçlarını kullanacak şekilde gelişmiştir. Kalsiyum görüntüleme, güvenilir sonuçlar elde etmek için sofistike yazılımlar gerektiren yaygın olarak kullanılan bir tekniktir, ancak birçok laboratuvar, protokolleri modern standartları karşılayacak şekilde güncellerken hesaplama yöntemlerini benimsemekte zorlanmaktadır. Programlama bilgisi eksikliği ve yazılım için ödeme duvarları nedeniyle zorluklar ortaya çıkmaktadır. Ek olarak, ilgilenilen hücreler kalsiyum görüntüleme sırasında her yönde hareketler gösterir. Hareketi lateral (x/y) yönde düzeltmek için birçok yaklaşım geliştirilmiştir.
Bu makalede, 3D kalsiyum görüntülemede z eksenindeki hareketi incelemek için yeni bir ImageJ eklentisi olan TrackMate Analysis of Calcium Imaging (TACI) kullanılarak gerçekleştirilen bir iş akışı açıklanmaktadır. Bu yazılım, bir nöronun göründüğü tüm z-pozisyonlarından maksimum floresan değerini tanımlar ve nöronun yoğunluğunu karşılık gelen t-pozisyonunda temsil etmek için kullanır. Bu nedenle, bu araç yanal (x / y) yönde üst üste binen, ancak farklı z-düzlemlerinde görünen nöronları ayırabilir. Bir ImageJ eklentisi olarak TACI, 3D kalsiyum görüntüleme analizi için kullanıcı dostu, açık kaynaklı bir hesaplama aracıdır. Bu iş akışını, sıcaklık dalgalanması sırasında her yöne hareketleri gösteren sinek larva ısıya duyarlı nöronları ve sinek beyninden elde edilen bir 3D kalsiyum görüntüleme veri kümesini kullanarak doğruladık.
Giriş
Hücre içi kalsiyum seviyesi, nöronal uyarılabilirliğin kesin bir belirtecidir. Kalsiyum görüntüleme, nöronal aktiviteyi anlamak için hücre içi kalsiyumdaki değişiklikleri ölçer1. Nörobilimdeki çalışmalar, genetik yaklaşımlar yoluyla belirli nöron kümelerinde noninvaziv olarak ifade edilebilen GCaMP 2,3 gibi genetik olarak kodlanmış kalsiyum göstergeleri (GECI'ler) de dahil olmak üzere hücre içi kalsiyum konsantrasyonunu ölçmek için tekniklerin geliştirilmesi nedeniyle bu yöntemi giderek daha fazla kullanmıştır. Lazerlerin ve mikroskop bileşenlerinin düşük maliyetleri de kalsiyum görüntüleme kullanımını arttırmıştır4. Daha da önemlisi, kalsiyum görüntüleme, serbestçe hareket eden hayvanlarda aynı anda tek nöronların yanı sıra büyük nöron popülasyonlarının kaydedilmesine ve incelenmesine izin verir5.
Bununla birlikte, kalsiyum görüntüleme verilerinin analizi zordur, çünkü (1) zaman içinde bireysel hücrelerin floresansındaki değişiklikleri izlemeyi içerir, (2) floresan sinyali nöronal yanıtlarla aralıklı olarak kaybolur veya yeniden ortaya çıkar ve (3) nöronlar her yönde, özellikle bir odak düzleminin içinde ve dışında hareket edebilir veya birden fazla düzlemde görünebilir4, 6. Manuel analiz zaman alıcıdır ve kayıtların uzunluğu ve nöronların sayısı arttıkça pratik hale gelir. Kalsiyum görüntüleme analiz sürecini hızlandırmak için çeşitli yazılım programları geliştirilmiştir. Önceden, yazılım sınırlı bir deneysel bağlamda tasarlandı ve diğer laboratuvarların onu benimsemesini zorlaştırdı. Yazılım paylaşımı için modern standartları karşılamaya yönelik son çabalar, farklı gruplardaki kalsiyum görüntüleme verilerini tutarlı bir şekilde analiz edebilen çeşitli araçların geliştirilmesine yol açmıştır 7,8,9,10,11,12,13,14,15,16,17,18,19 . Bununla birlikte, bu araçların çoğu programlama bilgisi gerektirir ve / veya ticari yazılıma bağlıdır. Programlama bilgisi ve yazılım ödeme duvarlarının eksikliği, araştırmacıları bu yöntemleri benimsemekten caydırmaktadır. Dahası, bu araçların birçoğu x / y hareketini düzeltmeye odaklanır, ancak z eksenindeki hareketin de açıkça teşhis edilmesi ve düzeltilmesi gerekir6. Z-sürüklenmesi sergileyen ve çoklu z-düzlemlerinde görünen nöronlara odaklanan 3D kalsiyum görüntülemeyi analiz etmek için hesaplamalı bir araca ihtiyaç vardır. İdeal olarak, bu araç açık kaynaklı yazılım kullanmalı ve diğer laboratuvarların kolayca benimsemesine izin vermek için programlama bilgisi gerektirmemelidir.
Burada, 3D kalsiyum görüntüleme verilerini analiz etmek için yeni bir ImageJ eklentisi olan TACI'yi geliştirdik. İlk olarak, yazılım gerekirse yeniden adlandırır ve 3D kalsiyum görüntüleme verilerini z-konumlarına göre düzenler. İlgilenilen hücreler her z-pozisyonunda izlenir ve floresan yoğunlukları TrackMate veya diğer hesaplama araçları tarafından çıkarılır. TACI daha sonra z eksenindeki hareketi incelemek için uygulanır. Bir z-yığınının maksimum değerini tanımlar ve karşılık gelen zaman noktasında bir hücrenin yoğunluğunu temsil etmek için kullanır. Bu iş akışı, 3D kalsiyum görüntülemeyi her yöne hareket ederek ve / veya yanal (x / y) yönde üst üste binen ancak farklı z konumlarında görünen nöronlarla analiz etmek için uygundur. Bu iş akışını doğrulamak için, sinek larva ısıl duyarlı nöronlarından ve beyindeki mantar nöronlarından 3D kalsiyum görüntüleme veri kümeleri kullanılmıştır. Not olarak, TACI açık kaynaklı bir ImageJ eklentisidir ve herhangi bir programlama bilgisi gerektirmez.
Protokol
1. Kalsiyum görüntüleme
- Sinek larvaları hazırlığı
NOT: Sinekler ve larvalar 25 °C'de 12 saat:12 saat ışık:karanlık döngü altında tutulur.- Sinekleri CO2 ile uyuşturun. Her sinek şişesine 20-45 erkek ve 20-45 dişi ayırın ve CO 2 maruziyetinden kurtulmak için onlara en az24 saat ila 48 saat verin.
NOT: CO2'ye sinek maruziyeti mümkün olan en kısa süre sürmelidir. - Larva yaşını senkronize etmek için, sineklerin üzerine maya granülleri içeren yeni şişelere dokunun ve yumurta bırakmaları için 4-8 saat bekleyin. Sinekleri yeni şişelere çevirerek çıkarın.
- Larvaları 72 saatte 10 mL% 20 w / v sakkaroz çözeltisi kullanarak toplayın.
- Sinekleri CO2 ile uyuşturun. Her sinek şişesine 20-45 erkek ve 20-45 dişi ayırın ve CO 2 maruziyetinden kurtulmak için onlara en az24 saat ila 48 saat verin.
- Mikroskop ve sıcaklık kontrol kurulumu
- Aşağıdaki ayarları kullanarak görüntülemeyi bir konfokal mikroskopta ( Malzeme Tablosuna bakınız) ve bir sahne eki ile z eksenli bir piezo sahnesinde gerçekleştirin: lazer, Argon; tarama modu, çerçeve; çerçeve boyutu, 512 x 512; hız, maksimum; kanallar / bit derinliği, 1/8 Bit; yakınlaştırma, 1.5; z-yığını, dilim = 15, Keep = dilim; odak cihazları ve stratejisi, Kesin odak; odak, Kesin odaklanma.
- Bir termoelektrik soğutucu oluşturmak için ısı transfer bileşiği ile bir Peltier soğutma modülünü bir ısı emiciye takın. Peltier, 2 A güç kaynağı ile güçlendirilmiştir.
- Sıcaklığı kaydetmek için bir veri toplama cihazına bir termokupl mikroprobu takın.
- Kalsiyum görüntüleme
- Larvaları 1x fosfat tamponlu salin (PBS) içinde 3x durulayın.
- Bir cam kızağın ortasına 75 μL 1x PBS pipet.
- 1x PBS'ye bir veya iki larva koyun ve termokupl mikroprobunu larvaların yanına yerleştirin.
NOT: Larva ve mikroprob arasındaki mesafe ~ 5 mm olmalıdır. Larvalar, mikroprob kendilerine çok yakın konumlandırılmışsa hareket edebilir. Büyük bir mesafe yanlış sıcaklık okumalarına neden olabilir. - Larva ve termokupl mikroprobunu bir cam kapak kayışı ile örtün. Kapak kapağını oje ile kapatın.
- Slaytı mikroskop aşamasına yerleştirin, 25x hedefi kullanarak odağı bulun ve termoelektrik soğutucuyu slaytın üzerine yerleştirin.
NOT: Peltier, larvaların sıcaklık uyaranlarını iletmek için bulunduğu slayta doğrudan yerleştirilir. - Konfokal yazılımda, ilgilenilen floresan hücrelere odaklanın. Aşırı doygunluğu önlemek için lazer gücünü ayarlayın. Z-stack ayarında ilk ve son dilim konumlarını ayarlayın.
NOT: Fotobeyazlatmayı önlemek için kayıttan önce mümkün olan en düşük lazer gücünü kullanın. - Z-stack taramasını ve sıcaklık kaydını aynı anda başlatın.
- Peltier yüzey sıcaklığını değiştirmek için Peltier'e güç veren güç kaynağını kontrol edin. Sıcaklığı azaltmak için güç kaynağını açın ve sıcaklığı artırmak için kapatın.
- Z-stack taramasını ve sıcaklık kaydını durdurun.
2. 3D kalsiyum görüntüleme verilerinin analizi
- Kalsiyum görüntüleme verilerini TIFF dosyalarına aktarın ve bunları içindeki TIFF dosyalarının temel adıyla aynı ada sahip bir klasöre kaydedin.
Not: dosya adı virgül içermemelidir.- Kalsiyum görüntüleme verilerini ZEN'den (siyah baskı) dışa aktarmak için aşağıdaki parametreleri kullanın: dosya türü, TIFF; sıkıştırma, LZW; kanallar, 2; Z-pozisyonu, hepsi; zaman, hepsi; faz, 1; bölge, dolu.
- Kurma
- TACI-Calcium_Imaging.jar'i Github'dan indirin (https://github.com/niflylab/TACI_CalciumImagingPlugin/releases).
- Menü çubuğunda Eklentiler'e tıklayarak ve ardından açılır menüde Yükle'ye tıklayarak eklentiyi FIJI'ye yükleyin (Eklentiler | Yükle). Ardından FIJI'yi yeniden başlatın.
NOT: Eklentileri KULLANMAYIN | Eklentiyi yükleyin. - Eklentiler'e tıklayarak ve ardından TACI-Kalsiyum Görüntüleme'yi seçerek eklentiyi çalıştırın (Eklentiler | TACI-Kalsiyum Görüntüleme).
- TIFF dosya adlarını gerekli yapıya dönüştürmek için RENAME işlevini kullanın.
NOT: Araç, varsayılan yapı olarak filename_h#t#z#c#.tif kullanır (h# ve c# isteğe bağlıdır; #: pozitif bir tamsayı). Görüntü dosya adları varsayılan yapıda değilse, RENAME işlevi yürütülmelidir.- Klasörlere Gözat'ı tıklatarak TIFF görüntülerinin yeniden adlandırılması gereken klasörü seçin.
- Parametre bilgilerini doldurun. Dosya Adı, Faz, Maksimum T-Konumu, Maksimum Z-Konumu ve Kanal dahil olmak üzere beş parametre listelenir. Her parametrenin üç değeri vardır: Önceki Metin, Parametre Değeri ve Sıra.
NOT: Bilgiler büyük/küçük harfe duyarlıdır. Görüntü dosya adları parametreden önce bir tümcecik içeriyorsa, tümceciği karşılık gelen parametrenin Önceki Metni olarak doldurun. Görüntü dosya adları tüm parametrelerden sonra bir tümcecik içeriyorsa, ifadeyi Gönderi Metni olarak doldurun.- Dosya Adı, Maksimum T-Konumu ve Maksimum Z-Konumu girdiğinizden ve Maksimum T-Konumu ve Maksimum Z-Konumu parametre değerlerinin tüm basamakları içerdiğinden emin olun.
- Dosya Adı'nın klasör adı kullanılarak otomatik olarak doldurulmasını bekleyin.
- TIFF dosya adları faz ve kanalı içermiyorsa, karşılık gelen parametre değerini boş bırakın ve Sırayla Na'yı seçin.
- Aynı ada sahip bir klasör oluşturmak için Yeniden Adlandır'ı tıklatın ve _r. Klasörde, TIFF dosya adlarının ORGANIZE işleviyle uyumlu olacak şekilde yeniden yapılandırıldığını gözlemleyin.
NOT: _r TIFF görüntülerinin dosya adlarına eklenmiştir.
- Aynı z konumundan TIFF görüntülerini tek bir klasöre kaydetmek için ORGANIZE işlevini kullanın.
NOT: Klasör adı, içindeki TIFF dosyalarının temel adıyla aynı ada sahip olmalı ve virgül içermemelidir.- Klasörlere Gözat'ı tıklatarak TIFF görüntülerinin düzenlenmesi gereken klasörü seçin.
- CSV dosyası parametresi (param.csv) varsa, parametre değerlerinin otomatik olarak doldurulmasını bekleyin.
Not: param.csv dosyası gerekli bir biçime sahiptir. Dosya adı, aşama, position_t, position_z, kanal ve is_gray dahil olmak üzere parametreler, satır 1'de A sütunundan başlayarak soldan sağa doğru doldurulmalıdır. - CSV dosyası parametresi (param.csv) yoksa, parametre değerlerini el ile doldurun. Faz ve Kanal parametre değerlerinin harf içerdiğinden emin olun, T Konumu ve Z Konumu parametre değerleri t ve z konumlarının en büyük sayıları olmalıdır. Görüntü dosya adları fazı veya kanalı içermiyorsa, Na girin.
- Gerektiğinde gri tonlamalı TIFF görüntüleri oluşturun. Görüntüleri gri tonlamak için Görüntüler gri mi? kutusunun işaretini kaldırın.
- Aynı ada ve _gray_stacks sahip bir klasör oluşturmak ve klasörde aynı ada ve _# (#: z-konumları) değerine sahip klasörler oluşturmak için Düzenle'yi tıklatın. TIFF dosyalarının z-konumlarına göre karşılık gelen klasörlere ayrıldığını ve parametrelerin ve değerlerinin bulunabileceği param.csv adlı bir dosyanın oluşturulduğunu gözlemleyin.
- Her z-konumundan ilgili hücrelerin floresan yoğunluklarını çıkarmak için FIJI'de TrackMate kullanın.
NOT: Bu adım diğer görüntüleme yazılımları tarafından da gerçekleştirilebilir.- FIJI tarafından bir z-position klasöründe TIFF görüntülerini açın.
- Eklentiler'e tıklayarak TrackMate'i çalıştırın | Takip | TrackMate öğesine gidin ve gerekirse aşağıdaki parametreleri ayarlayın.
- DoG veya LoG dedektörleri kullanın.
- Blob çapını, eşiğini ve medyan filtresini değiştirin. Blob çapını, hücrelerin çapına benzer olacak şekilde ayarlayın. Hücreler oval ise, blob çapını alt eksene benzer olacak şekilde ayarlayın.
NOT: Eşiğin artırılması, sinyal yoğunluklarını etkilemeden arka plan gürültüsünün sinyal olarak alınmasını önlemeye yardımcı olur (Ek Şekil 1). Bununla birlikte, eşikteki bir artış gerçek sinyalleri kaçırabilir (Ek Şekil 1). Sinyaller güçlüyse, Tuz ve Biber gürültüsünü azaltmak için medyan filtreyi kullanın. - Filtreleri, alakasız sinyallerin tümünü olmasa da bazılarını kaldıracak şekilde ayarlayın. X ve Y filtreleri uzamsal filtrelerdir. Gerçek sinyallerden uzak olan alakasız sinyalleri kaldırmak için X ve Y filtrelerini kullanın.
NOT: Bir görüntüde filtreler ayarlandığında, gerçek sinyallerin kaldırılmadığından emin olmak için diğer tüm görüntüleri kontrol etmek çok önemlidir. - Bağlantı maksimum mesafesini, boşluk kapatma maksimum mesafesini ve boşluk kapatma maksimum kare boşluğunu ayarlayın. Bağlantı maksimum mesafesini ve boşluk kapatma maksimum mesafesini , özellikle numuneler zaman içinde önemli ölçüde hareket ettiğinde, parça sayısını azaltmaya yardımcı olmak için blob çapının 3x-5 katı olacak şekilde ayarlayın. Boşluk kapanan maksimum kare boşluğunu , yığındaki görüntü sayısına ayarlayın.
- İlgilenilen bölgelerin (YG) floresan yoğunluklarını bir CSV dosyasına aktarın. Eski bir TrackMate sürümü kullanılıyorsa, Bir eylem seçin penceresinde Tüm noktaların istatistiklerini dışa aktar'ı seçin. TrackMate sürümü 7.6.1 veya daha yüksekse, Görüntü seçenekleri penceresinde Noktalar'ı seçin. Etkileşimli dosyaları CSV dosyalarına dışa aktarın veya kaydedin.
NOT: Her iki dosya da görüntü penceresiyle etkileşimlidir; Bir YG'yi vurgulamak, görüntü penceresinde karşılık gelen YG'yi görüntüler. Bu dosyalar, ilgili zaman noktalarında (MEAN_INTENSITY) ilgili hücrelerin ortalama yoğunluklarını (MEAN_INTENSITY_CH1 veya POSITION_T) içerir. Aynı TRACK_IDs, farklı zaman noktalarında aynı yatırım getirilerini temsil etmelidir. Ancak, bu her zaman doğru değildir ve gerektiğinde manuel olarak düzeltilmesi gerekebilir. TrackMate bazı zaman noktalarında yatırım getirilerini tanımazsa, bu zaman noktaları görüntülenmez.
- Arka plan floresan yoğunluğunu ayıklayın ve Background_list.csv dosyasını oluşturun.
NOT: Bu çalışmada, her z-pozisyonu için arka plan yoğunluğu, floresan sinyalleri içermeyen ve farklı zaman noktalarından gelen üç ila beş komşu aynı boyutlu blobun ortalama değeri kullanılarak tahmin edilmiştir.- Background_list.csv dosyası gerekli bir biçime sahiptir: her sütun, Nöron 0'dan başlayarak bir nöronun bilgilerini içerir. 1. satırdaki Nöron 0 gibi nöron numaralarını doldurun. Ardından, analiz edilen her z-pozisyonu için arka plan yoğunluğunu sağlayın - Nöron 0 için beş z-pozisyonu analiz edilirse, Nöron 0'ın altındaki beş arka plan yoğunluğu değerini doldurun.
Not: Background_list.csv dosyası TACI EXTRACT işlevi için gereklidir. Arka plan ihmal edilebilir ise, her nöronun sıfır arka plan yoğunluğuna sahip Background_list.csv sağlanmalıdır.
- Background_list.csv dosyası gerekli bir biçime sahiptir: her sütun, Nöron 0'dan başlayarak bir nöronun bilgilerini içerir. 1. satırdaki Nöron 0 gibi nöron numaralarını doldurun. Ardından, analiz edilen her z-pozisyonu için arka plan yoğunluğunu sağlayın - Nöron 0 için beş z-pozisyonu analiz edilirse, Nöron 0'ın altındaki beş arka plan yoğunluğu değerini doldurun.
- Her t-pozisyonunda maksimum floresan yoğunluklarını tanımlamak ve denklem (1)'de gösterildiği gibi ΔF/F0'ı hesaplamak için EXTRACT işlevini kullanın.
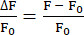 (1)
(1)- Bir klasör oluşturun ve Neuron 0'dan başlayarak nöron numarasını kullanarak adlandırın. İlgili nöronun floresan bilgilerini içeren CSV dosyalarını klasöre kaydedin. Her CSV dosyası bir z konumunun bilgilerini içerdiğinden, CSV dosyalarının sayısı analiz edilen z konumu sayısına eşittir. CSV dosyalarını Mean_Intensity#.csv olarak adlandırın (# z konumunu temsil eder) ve her CSV dosyası en az iki sütun içerir: POSITION_T ve MEAN_INTENSITY veya MEAN_INTENSITY_CH1.
NOT: İlgilendiğiniz her hücre için bir klasör oluşturun. - Adım 2.7.1'de oluşturulan Background_list.csv dosya ve klasörleri tek bir klasöre kaydedin. Dosyalara Gözat'a tıklayarak bu klasörü seçin.
NOT: Background_list.csv dosyasındaki arka plan değerlerinin sayısı, her nöron için Mean_Intensity#.csv dosyalarının sayısıyla eşleşmelidir. - Arka Plan Dosyası otomatik olarak doldurulur. T Pozisyonu Sayısı için en fazla sayıda t-pozisyonunu doldurun.
- Her nöron için CSV dosyaları ve grafikleri içeren bir sonuç klasörü oluşturmak için Ayıkla'yı tıklatın. CSV dosyaları, her t konumunda maksimum floresan yoğunluğu ve ΔF/F0 hakkında bilgi içerir. Grafikler, t-konumları üzerinden ΔF/F0'ın çizgi grafikleridir.
NOT: Bu çalışmada ΔF/F0 denklemi (1) kullanılarak hesaplanmıştır. Her z-pozisyonunun ilk değeri F0 olarak kullanılmıştır. Bu F0 uygun değilse20, eklenti python_files klasöründeki her nöron için ham veri içeren dosyalar sağlar.
- Bir klasör oluşturun ve Neuron 0'dan başlayarak nöron numarasını kullanarak adlandırın. İlgili nöronun floresan bilgilerini içeren CSV dosyalarını klasöre kaydedin. Her CSV dosyası bir z konumunun bilgilerini içerdiğinden, CSV dosyalarının sayısı analiz edilen z konumu sayısına eşittir. CSV dosyalarını Mean_Intensity#.csv olarak adlandırın (# z konumunu temsil eder) ve her CSV dosyası en az iki sütun içerir: POSITION_T ve MEAN_INTENSITY veya MEAN_INTENSITY_CH1.
- Her nöronun ΔF /F 0'ının ortalamasını almak, SEM'i hesaplamak ve ortalama ΔF/F0'ı t-konumları üzerinden çizmek için MERGE işlevini kullanın.
- Dosyalara Gözat'a tıklayarak EXTRACT işlevi tarafından oluşturulan sonuçlar klasörünü seçin.
- T Pozisyonu Sayısını, en fazla sayıda t-pozisyonu ile doldurun.
- merged_data dosyası ve merged_data.csv grafiği de dahil olmak üzere bir Average_dF_F0.png klasörü oluşturmak için Birleştir'i tıklatın. CSV dosyası, her t-konumundaki ortalama ve SEM ΔF/F0 bilgilerini içerir. Grafik, t-pozisyonları üzerinden ortalama ΔF/F0'ın çizgi grafiğidir.
Sonuçlar
3D kalsiyum görüntüleme analizi iş akışı
Bu çalışmada, yeni bir ImageJ eklentisi olan TACI'yi geliştirdik ve z-sürüklenmesini izlemek ve çoklu z-pozisyonlarında görünen bireysel hücrelerin tepkilerini belirleyen 3D kalsiyum görüntülemeyi analiz etmek için bir iş akışı tanımladık (Şekil 1). Bu aracın dört işlevi vardır: RENAME, ORGANIZE, EXTRACT ve MERGE. İlk olarak, gör?...
Tartışmalar
Bu çalışma yeni bir ImageJ eklentisi olan TACI'yi geliştirdi ve 3D kalsiyum görüntülemeyi analiz eden bir iş akışını tanımladı. Şu anda mevcut olan birçok araç, x / y hareketini düzeltmeye odaklanmaktadır, ancak z eksenindeki hareketin de açıkça teşhis edilmesi veya düzeltilmesi gerekir6. Canlı bir organizmada görüntü elde etme sırasında, organizma hareketsiz hale getirildiğinde bile z ekseni üzerindeki hareket kaçınılmazdır ve sıcaklık değişimi gibi bazı u...
Açıklamalar
Yazarların açıklayacağı bir çıkar çatışması yoktur.
Teşekkürler
Kalsiyum görüntüleme verilerini toplamak için Fralin Görüntüleme Merkezi'ndeki bir Zeiss LSM 880 kullanıldı. Dr. Michelle L Olsen ve Yuhang Pan'a IMARIS yazılımındaki yardımları için teşekkür ederiz. Dr. Lenwood S. Heath'e el yazması hakkındaki yapıcı yorumları için ve Steven Giavasis'e GitHub README dosyasındaki yorumları için teşekkür ederiz. Bu çalışma NIH R21MH122987 (https://www.nimh.nih.gov/index.shtml) ve NIH R01GM140130 (https://www.nigms.nih.gov/) tarafından L.N.'ye desteklenmiştir. Fon verenlerin çalışma tasarımı, veri toplama ve analizi, yayınlama kararı veya makalenin hazırlanmasında hiçbir rolü yoktu.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Blunt Fill Needel | BD | 303129 | |
| Calcium chloride dihydrate | Fisher Scientific | 10035-04-8 | Fly food ingredient |
| Carbon dioxide | Airgas | UN1013 | Size 200 High Pressure Steel Cylinder |
| CO2 bubbler kit | Genesee | 59-180 | |
| Confocal microscope LSM880 | Zeiss | 4109002107876000 | An inverted Axio Observer Z1, equipped with 5 lasers, 2 standard PMT detectors, 32-channel GaAsP dectectors, an Airyscan detector, and Definite Focus.2. |
| DAQami software | Measurement Computing | ||
| Dextrose | Genesee | 62-113 | Fly food ingredient |
| Drosophila Agar | Genesee | 66-111 | Fly food ingredient |
| Ethanol | Decon Labs, Inc. | 64-17-5 | Fly food ingredient |
| Fly line: Ir21a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: Ir21a-Gal80 | Dr. Lina Ni lab | ||
| Fly line: Ir68a-Gal4 | Dr. Aravinthan DT Samuel lab | A kind gift | |
| Fly line: Ir93a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: UAS-GCaMP6 | Bloomington Drosophila Stock Center | 42750 | |
| Flypad | Genesee | 59-114 | |
| General purpose forged brass regulator | Gentec | G152 | |
| Gibco PBS pH 7.4 (1x) | Thermo Fisher Scientific | 10010-031 | |
| Green Drosophila tubing | Genesee | 59-124 | |
| Heat transfer compound | MG Chemicals | 860-60G | |
| Heatsink | Digi-Key Electronics | ATS2193-ND | Resize to 12.9 x 5.5 cm |
| Illuminator | AmScope | LED-6W | |
| Inactive Dry Yeast | Genesee | 62-108 | Fly food ingredient |
| Incubator | Pervical | DR-41VL | Light: dark cycle: 12h:12h; temperature: 25 °C; humidity: 40-50% RH. |
| Methyl-4-hydroxybenzoate | Thermo Scientific | 126965000 | Fly food ingrediete |
| Micro cover glass | VWR | 48382-126 | 22 x 40 mm |
| Microscope slides | Fisher Scientific | 12-544-2 | 25 x 75 x 1.0 mm |
| Nail polish | Kleancolor | ||
| Narrow Drosophila vials | Genesee | 32-113RL | |
| Objective | Zeiss | 420852-9871-000 | LD LCI Plan-Apochromat 25x/0.8 Imm Corr DIC M27 |
| Peltier cooling module | TE Technology | TE-127-1.0-0.8 | 30 x 30 mm |
| Plugs | Genesee | 49-102 | |
| Power Supply | Circuit Specialists | CSI1802X | 10 volt DC 2.0 amp linear bench power supply |
| Princeton Artist Brush Nepture | Princeton Artist Brush Co. | Series 4750, size 2 | |
| Sodium potassium L-tartrate tetrahydrate | Thermo Scientific | 033241-36 | Fly food ingredient |
| Stage insert | Wienecke and Sinske | 432339-9030-000 | |
| Stereo Microscope | Olympus | SZ61 | Any stereo microscope works |
| T-Fitting | Genesee | 59-123 | |
| Thermocouple data acquisition device | Measurement Computing | USB-2001-TC | Single channel |
| Thermocouple microprobe | Physitemp | IT-24P | |
| Yellow Cornmeal | Genesee | 62-101 | Fly food ingredient |
| Z-axis piezo stage | Wienecke and Sinske | 432339-9000-000 |
Referanslar
- Grienberger, C., Konnerth, A. Imaging calcium in neurons. Neuron. 73 (5), 862-885 (2012).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nature Biotechnology. 19 (2), 137-141 (2001).
- Zhang, Y., et al. jGCaMP8 fast genetically encoded calcium indicators. Janelia Research Campus. , (2020).
- Robbins, M., Christensen, C. N., Kaminski, C. F., Zlatic, M. Calcium imaging analysis - How far have we come. F1000Research. 10, 258 (2021).
- Oh, J., Lee, C., Kaang, B. K. Imaging and analysis of genetically encoded calcium indicators linking neural circuits and behaviors. The Korean Journal of Physiology & Pharmacology. 23 (4), 237-249 (2019).
- Stringer, C., Pachitariu, M. Computational processing of neural recordings from calcium imaging data. Current Opinion in Neurobiology. 55, 22-31 (2019).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Nguyen, J. P., Linder, A. N., Plummer, G. S., Shaevitz, J. W., Leifer, A. M. Automatically tracking neurons in a moving and deforming brain. PLoS Computational Biology. 13 (5), 1005517 (2017).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. EMC2: A versatile algorithm for robust tracking of calcium dynamics from individual neurons in behaving animals. bioRxiv. , (2021).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, 38173 (2019).
- Delestro, F., et al. In vivo large-scale analysis of Drosophila neuronal calcium traces by automated tracking of single somata. Scientific Reports. 10, 7153 (2020).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25 (2020).
- Eglen, S. J., et al. Toward standard practices for sharing computer code and programs in neuroscience. Nature Neuroscience. 20 (6), 770-773 (2017).
- Pachitariu, M., et al. Suite2p: Beyond 10,000 neurons with standard two-photon microscopy. bioRxiv. , (2017).
- Corder, G., et al. An amygdalar neural ensemble that encodes the unpleasantness of pain. Science. 363 (6424), 276-281 (2019).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. Tracking calcium dynamics from individual neurons in behaving animals. PLoS Computational Biology. 17 (10), 1009432 (2021).
- Kolar, K., Dondorp, D., Zwiggelaar, J. C., Høyer, J., Chatzigeorgiou, M. Mesmerize is a dynamically adaptable user-friendly analysis platform for 2D and 3D calcium imaging data. Nature Communications. 12, 6569 (2021).
- Moein, M., et al. CaSiAn: A Calcium Signaling Analyzer tool. Bioinformatics. 34 (17), 3052-3054 (2018).
- Zhou, P., et al. Efficient and accurate extraction of in vivo calcium signals from microendoscopic video data. Elife. 7, 28728 (2018).
- Neugornet, A., O'Donovan, B., Ortinski, P. I. Comparative effects of event detection methods on the analysis and interpretation of Ca(2+) imaging data. Frontiers in Neuroscience. 15, 620869 (2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Fazeli, E., et al. Automated cell tracking using StarDist and TrackMate. F1000Research. 9, 1279 (2020).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Ni, L., et al. The ionotropic receptors IR21a and IR25a mediate cool sensing in Drosophila. Elife. 5, 13254 (2016).
- Omelchenko, A. A., et al. Cool and warm ionotropic receptors control multiple thermotaxes in Drosophila larvae. Frontiers in Molecular Neuroscience. , (2022).
- Sanchez-Alcaniz, J. A., et al. An expression atlas of variant ionotropic glutamate receptors identifies a molecular basis of carbonation sensing. Nature Communications. 9 (1), 4252 (2018).
- Hernandez-Nunez, L., et al. Synchronous and opponent thermosensors use flexible cross-inhibition to orchestrate thermal homeostasis. Science Advances. 7 (35), (2021).
- Klein, M., et al. Sensory determinants of behavioral dynamics in Drosophila thermotaxis. Proceedings of the National Academy of Sciences of the United States of America. 112 (2), 220-229 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır