Nesiller Arası Epigenetik Kalıtımın Analizi C. elegans Floresan Raportör ve Kromatin İmmünopresipitasyon (ChIP) Kullanılarak
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bu protokol, C. elegans'ta RNAi kaynaklı susturma ve ilişkili kromatin modifikasyonlarının epigenetik kalıtımını incelemek için bir RNA girişimini ve ChIP testini açıklar.
Özet
Nesiller arası epigenetik kalıtım (TEI), kodlamayan RNA'lar ve kromatin modifikasyonları gibi faktörler aracılığıyla genom dizisini değiştirmeden germ hattı boyunca bilgi aktarımına izin verir. Nematod Caenorhabditis elegans'ta RNA interferansı (RNAi) kalıtımı olgusu, bu model organizmanın kısa yaşam döngüsünden, kendi kendine yayılmasından ve şeffaflığından yararlanan TEI'yi araştırmak için etkili bir modeldir. RNAi kalıtımında, hayvanların RNAi'ye maruz kalması, ilk tetikleyicinin yokluğunda birden fazla nesil boyunca devam eden hedef lokusta gen susturulmasına ve değişmiş kromatin imzalarına yol açar. Bu protokol, germ hattı ile eksprese edilen bir nükleer yeşil floresan protein (GFP) raportörü kullanılarak C. elegans'ta RNAi kalıtımının analizini açıklar. Muhabir susturma, GFP'yi hedefleyen çift sarmallı RNA eksprese eden bakterilerin hayvanlarla beslenmesiyle başlatılır. Her nesilde, senkronize gelişimi sürdürmek için hayvanlar geçirilir ve raportör gen susturma mikroskopi ile belirlenir. Seçilmiş nesillerde, GFP raportör lokusunda histon modifikasyon zenginleşmesini ölçmek için kromatin immünopresipitasyon (ChIP)-kantitatif polimeraz zincir reaksiyonu (qPCR) için popülasyonlar toplanır ve işlenir. RNAi kalıtımını incelemek için bu protokol, küçük RNA ve kromatin yolaklarındaki TEI faktörlerini daha fazla araştırmak için kolayca değiştirilebilir ve diğer analizlerle birleştirilebilir.
Giriş
Epigenetik kalıtım, gen düzenleyici bilgilerin nesiller boyunca aktarılmasına izin verir ve bu nedenle ebeveynlerin çevresinin veya deneyimlerinin soylarını etkilemesine izin verebilir. C. elegans'ta, eksojen çift sarmallı RNA (dsRNA) tarafından başlatılan germ hattı gen susturması, orijinal tetikleyiciyemaruz kalmayan döllerde birden fazla nesil için kalıtsal olabilir 1,2,3,4. RNA interferansı (RNAi) kalıtımı olarak adlandırılan bu süreç, C. elegans'ta piRNA tarafından başlatılan çok kuşaklı susturma2,5, paramutasyon/RNAe (RNA kaynaklı epigenetik susturma)6,7,8 ve çok kopyalı dizi tarafından başlatılan susturma9 dahil olmak üzere, küçük RNA ve kromatin düzenleme makineleri için örtüşen ancak farklı gereksinimleri olan birkaç ilgili epigenetik susturma fenomeninden biridir . C. elegans eksojen RNAi'de dsRNA, hedef mRNA'larını tanımak için birincil Argonaute proteinleri ile bir kompleks içinde hareket eden küçük enterferans yapan RNA'lara (siRNA'lar) işlenir. Bu hedefleme, hem sitoplazmik hem de nükleer susturma yolları yoluyla hedef mRNA'yı susturmak için ikincil Argonotlar ile ilişkili ikincil siRNA'ların amplifikasyonuna yol açar. Germ hattı eksprese edilen RNAi hedefleri için, nükleer ikincil Argonaute HRDE-1 ve ek nükleer RNAi faktörleri, yeni ortaya çıkan transkriptleri hedef alır, bu da transkripsiyonel baskılama ve H3K9me310 dahil olmak üzere baskılayıcı kromatin işaretlerini biriktirmek için histon metiltransferazların işe alınmasına neden olur. Histon H3K9me3, RNAi kalıtımı11,12 ile germ hattı ile eksprese edilen yeşil floresan protein (GFP) transgenlerinin kalıtsal susturulmasının kurulmasını destekler.
Bu protokolün amacı, RNAi kalıtımı için bir raportör olarak germ hattında bir GFP-histon füzyon proteinini eksprese eden bir transgen kullanmak ve kromatin immünopresipitasyon ve kantitatif polimeraz zincir reaksiyonu (ChIP-qPCR) kullanarak raportör transgen lokusunda histon modifikasyonlarındaki değişiklikleri test etmektir. Bu protokol, raportör susturmayı başlatmak için standartlaştırılmış bir plaka bazlı RNAi besleme yaklaşımını açıklar. Ayrıca, alkalin hipoklorit tedavisi ('ağartma') ile gravid yetişkinlerden utero embriyoları izole ederek hayvanların nesiller arasında geçirilmesi için ayrıntılı bir zaman çizelgesi sağlar. Floresan mikroskobu ile popülasyonun bir alt kümesinde GFP susturma sıklığının izlenmesi ve histon H3K9me3 ChIP-qPCR için yöntemler ve temsili veriler de açıklanmaktadır. Raportör tabanlı RNAi kalıtım deneyleri, epigenetik düzenlemede genetik ve çevresel faktörlerin rollerini işlevsel olarak incelemek için oldukça izlenebilir bir sistem sağlar 13,14 ve bu tür raportörleri kullanan genetik taramalar, hem 2,3,15 için gerekli olan genleri hem de16,17 nesiller arası epigenetik kalıtım süresini negatif olarak düzenleyen genleri tanımlamıştır.
Protokol
NOT: Şekil 1'de bir tahlil zaman çizelgesi verilmiştir.
1. RNAi nematod büyüme ortamı (RNAi-NGM) plakalarının hazırlanması
- GFP veya kontrol RNAi vektörlerini içeren E. coli HT115 (DE3), gliserol stoklarından 100 μg / mL ampisilin ile desteklenmiş Luria-Bertani (LB) agar plakalarına çizin. Bakterileri gece boyunca 37 °C'de inkübe edin.
- Ertesi gün, RNAi-NGM plakaları hazırlayın (% 1.7 [a / h] agar,% 0.3 [a / h] NaCl,% 0.25 [a / h] pepton, 1 mM CaCl2, 5 μg / mL kolesterol, 25 mM potasyum fosfat tamponu [pH 6.0], 1 mMMgS4, 25 μg / mL karbenisilin ve 5 mM izopropil β-d-1-tiyogalaktopiranosid [IPTG]) standart protokolegöre 18.
- Testteki her bir suş için, GFP RNAi ile tohumlanmış en az altı RNAi-NGM plakası (üç biyolojik kopyanın her biri için iki plaka) ve kontrol RNAi (bir biyolojik kopya) ile tohumlanmış iki RNAi-NGM plakası hazırlayın.
- Işıktan korumak için plakaları folyo ile gevşek bir şekilde örtün ve gece boyunca oda sıcaklığında kurumaya bırakın.
- RNAi-NGM plakasının dökülmesiyle aynı gün, 4 mL sıvı RNAi bakteri kültürü hazırlayın.
- Steril koşullar altında, kültür tüplerine 10 μg/mL tetrasiklin ve 100 μg/mL ampisilin ile takviye edilmiş 4 mL LB suyu alikot. LB agar plakalarından her tüpe tek koloniler seçin ve 37 °C'de yaklaşık 16 saat çalkalayarak gece boyunca inkübe edin.
- RNAi bakterilerini RNAi-NGM plakalarına tohumlayın.
- Gece boyunca büyüdükten sonra, LB suyu ile 1:5 bakteri seyreltmesi kullanarak kültürlerin optik yoğunluğunu (OD600) ölçün.
- 10 μg/mL tetrasiklin ve 100 μg/mL ampisilin ile takviye edilmiş LB suyu kullanarak RNAi bakterilerini göreceli OD600 /2'ye seyreltin.
- Steril koşullar altında, her bir RNAi-NGM plakasına 150 μL kontrol veya GFP RNAi bakterisi ekleyin.
- Plakaları folyo ile örtün ve kullanmadan önce bakterilerin oda sıcaklığında en az 24 saat üremesine izin verin.
NOT: Kullanılmayan RNAi-NGM plakaları, karanlıkta kapalı bir kapta 4 °C'de 1 haftaya kadar ters çevrilmiş olarak saklanabilir.
2. RNAi kalıtım testinin başlatılması: P0 nesli için embriyoların ağartılması ve kaplanması
NOT: RNAi Kalıtım testine başlamadan önce, GFP raportörü mjIs134 [mex-5p::gfp-h2b::tbb-2 3'UTR]7 içeren solucanlar, en az iki nesil boyunca 21 °C'de aç bırakılmadan muhafaza edilmelidir.
- RNAi kalıtım testinin başlamasından 4 gün (96 saat) önce (Şekil 1), OP50-1 bakterileri ile tohumlanmış her 35 mm'lik standart NGM plakasına üç veya dört gravid yetişkin seçin.
NOT: Gerinim başına üç NGM plakası yeterlidir. Doğurganlığı azalmış suşlar için, plaka başına dörtten fazla hayvan gerekebilir. - 4 gün sonra, yetişkin popülasyonun embriyoları tabağa bırakmaya başladığından emin olun. Triton X-100 (TX-100) (22 mM KH 2 PO 4, 42 mM Na2HPO 4, 86 mM NaCl, 1mMMgSO 4,% 0.01 (h / h) TX-100) ile desteklenmiş 800 μL M9 tamponu kullanarak plakalardaki solucanları 1.5 mL'lik tüplere yıkayın. Yıkamayı tekrarlayın ve aynı tüpe koyun.
- Embriyoları aşağıdaki gibi ağartarak popülasyondan izole edin.
- 1.5:1 hacim oranında ağartıcı (%6 sodyum hipoklorit) ve 10 N NaOH içeren bir ağartma solüsyonu hazırlayın. Her numune için 250 μL kullanılabilecek şekilde yeterli miktarda hazırlayın.
- 1.5 mL'lik tüpleri solucanlarla birlikte 1.000 x g'da 1.5-2 dakika santrifüjleyin.
- Solucan 'peletini' bozmadan 100 μL bırakarak süpernatanı aspire edin. Alternatif olarak, solucanların aspirasyondan önce yerleşmesine izin vermek için tüpler yaklaşık 2 dakika bırakılabilir.
- 750 μL'lik bir nihai hacim elde etmek için her tüpe 650 μL ddH2Oekleyin.
NOT: Aşağıdaki adımlar zamana duyarlıdır. - Her tüpe 250 μL ağartma solüsyonu ekleyin ve bir zamanlayıcı başlatın.
- Her 1-2 dakikada bir, tüpleri iyice girdaplayın. 5 dakika sonra, yetişkinlerin bozulmasını izlemek için stereoskopun altındaki solucanları kontrol edin.
- Solucanlar tamamen çözülene ve sadece embriyolar kalana kadar girdap yapmaya devam edin. Tüpleri hemen 1,5 dakika boyunca 1.000 x g'da santrifüjleyin.
NOT: Beyazlatma genellikle 6-7 dakika içinde tamamlanır, ancak salınan embriyoları gözlemlemek için yeterli bir büyütmede (40x) bir stereoskop altında izlenmelidir. Solucanları uzun süre ağartıcıda bırakmayın, çünkü bu embriyoları tehlikeye atacaktır. - Santrifüjlemeden sonra, süpernatanı aspire edin ve yaklaşık 50-100 μL bırakın. Yıkamak için TX-100 ile 1 mL M9 tamponu ekleyin ve vorteksleme ile karıştırın. 1.000 x g'da 1.5-2 dakika tekrar santrifüjleyin ve en az iki kez daha yıkayın.
- Süpernatanı son yıkamadan aspire edin ve tüpte yaklaşık 100 μL bırakın. Girdap yaparak karıştırın. Her tüpten 2 μL'yi etiketli bir cam slayt üzerine iki kez pipetleyin. Mikrolitre başına embriyo konsantrasyonunu tahmin etmek için bir çetele sayacı kullanarak embriyoları sayın.
NOT: Birden fazla buzlu kuyucuğa sahip slaytlar, birden fazla suşun kopyalarını saymak için burada kullanılabilir. Sayma slaytları yıkanabilir ve tekrar kullanılabilir.
- Yarı senkronize bir popülasyon artışı başlatmak için, her bir RNAi-NGM plakasına 250 embriyo içeren bir hacmi karıştırın ve pipetleyin. Her kopya için iki plaka kullanın. Sıvının emilmesine izin verin.
- RNAi ile tedavi edilen P0 nesli için, 21 ° C'de 4 gün (96 saat) inkübe edin (embriyolardan yetişkinliğe kadar). Zamanlamalar genotipe göre değişebilir.
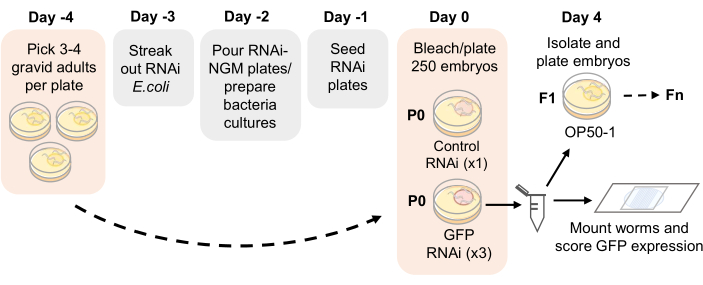
Şekil 1: RNAi kalıtım testi şeması. RNAi-NGM plaka hazırlığı ve RNAi kalıtım testi kurulumu için önerilen zaman çizelgesi. Gravid erginleri -4. günde OP50-1 ile tohumlanmış NGM plakalarına toplanır. 4 gün sonra ergin döller ağartılır ve embriyolar RNAi-NGM plakalarına kaplanır. P0 nesli, 21 °C'de 4 gün boyunca RNAi'ye maruz bırakılır. Solucanlar yetişkinliğe ulaştığında, kopyalar ağartma ile geçirilir ve her nesilde germ hattı GFP ekspresyonu için puanlanır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
3. Germ hattı GFP ekspresyonu için her neslin aktarılması ve puanlanması
NOT: Puanlamayı kolaylaştırmak için, solucanları hizalamak için doğrusal çıkıntılara sahip agaroz pedleri oluşturmak için bir vinil plak kullanın. Bu yöntem Rivera Gomez ve Schvarzstein19'dan uyarlanmıştır.
- Bir mikrodalgada ısıtarak küçük bir şişede agarozu ddH2O içinde çözerek% 1 (a / h) agaroz hazırlayın. Bir karıştırma çubuğu ekleyin ve folyo ile örtün. Yeniden eritilecekse, agarozu karıştırırken bir ocak gözünde 200 °C'de ısıtın. Eridikten sonra ısıyı 80 °C'ye düşürün.
- TX-100 ile 800 μL M9 tamponu kullanarak 1,5 mL'lik bir tüpe NGM plakalarındaki solucanları yıkayın. Tüm gravid yetişkinleri çıkarmak için plakaları iki kez yıkayın. Hayvanların verimli bir şekilde toplandığını doğrulamak için stereoskopun altındaki plakaları kontrol edin.
- Tüpleri 1.000 x g'da 1.5-2 dakika santrifüjleyin veya solucanların 2 dakika oturmasına izin verin. Süpernatanı aspire edin ve solucan 'peletini' bozmadan 100 μL bırakın.
- Solucanları monte etmek için agaroz pedleri aşağıdaki gibi hazırlayın.
- Bir cam Pasteur pipeti kullanarak (dar ucu kırılmış olarak) vinil plağın üzerine (daha önce19) erimiş %1 (a/h) agaroz damlatın.
- Bir cam slaytı, kayıttaki çizgiler yatay veya dikey olacak şekilde yönlendirin ve slaytı hızlı bir şekilde agaroz damlasının üzerine yerleştirin. Cam sürgüyü kayıttan çıkarmadan önce yaklaşık 30 saniye bekleyin.
NOT: Birden fazla genotip puanlanıyorsa, bir slaytta daha büyük bir agaroz pedi yapmak ve bir bıçak kullanarak ikiye kesmek faydalı olabilir.
- Solucanları agaroz pedlerine monte edin.
- Stereoskop altında, agaroz pedine yaklaşık 5 μL 5 mM levamisol ekleyin. Levamizol seyreltmesi her hafta taze yapılmalıdır.
- 5-10 μL solucanı karıştırmak ve agaroz pedine aktarmak için solucan tüplerini hafifçe vurun. Solucanların sayısını, çoğaltılan solucan başına yaklaşık 40 solucan olacak şekilde eklenirken gözle tahmin edin.
- Daha kolay puanlama için bir kirpik penası kullanarak solucanları sıralar halinde hizalayın. Bir cam lamel ekleyin.
NOT: Hayvanların bu şekilde monte edildiği kaydıraklar kurumadan önce birkaç saat dayanabilir.
- Slaytları puanlamadan önce ağartma protokolünü kullanarak embriyoları kalan hayvanlardan izole edin (bkz. adım 2.3). OP50-120 ile tohumlanmış 35 mm NGM plakalarına 250 embriyo yerleştirin. Sıvı emildikten sonra plakaları ters çevirin ve 21 °C inkübatöre geri koyun.
- GFP filtre setine sahip bir floresan stereoskop kullanın. Bir çetele sayacı kullanarak her çoğaltma için slaytlardaki GFP pozitif ve GFP negatif solucanların sayısını sayın ve kaydedin.
NOT: GFP pozitif solucanlar, germ hatlarında ve uteroda embriyolarında nükleer GFP ekspresyonuna sahip olacaktır. GFP negatif solucanların germ hatları floresan olmaz, ancak GFP daha sonraki nesillerde tekrar açılmaya başladığında, floresan loş olabilir. Gözlenen herhangi bir otofloresansı hesaba katmak için ek transgenik olmayan solucan suşlarının monte edilmesi yararlı olabilir. - Popülasyonu, yukarıda tarif edildiği gibi, 21 ° C'de yaklaşık 4 günde bir (96 saat) ağartma yoluyla geçiş. Alternatif olarak, kolaylık sağlamak için 3 günlük/4 günlük alternatif bir programda geçiş yapılabilir. 72 saat sonra daha düşük bir embriyo verimi olabileceğinden, dönüşümlü olarak daha kısa geçen nesiller için fazladan ~ 50 embriyo yerleştirin.
NOT: P0 neslinin geçiş süresini, kaplamadan 4 gün sonra embriyolarda tutarlı tutun.
4. ChIP için hayvan toplama
NOT: Hayvan sayısı ve zamanlama suşa, gelişim aşamasına, epitopa ve immünopresipitasyon (IP) hedeflerinin sayısına bağlıdır. Aşağıdaki örnekte, üç IP için hayvanları toplayın: H3K9me3, histon H3 ve IgG kontrolü. ChIP protokolü Askjaer ve ark.21'den uyarlanmıştır.
- ChIP numune toplamadan önce, RNAi kalıtım testinin önceki neslini ek üç NGM plakası (kopya başına en az dört plakaya) genişletin. 21 °C'de 4 gün büyütün.
- Embriyoları izole etmek için gravid yetişkinleri ağartın (bkz. adım 2.3).
- Her suş için, yaklaşık 3.500 embriyoyu 14 plaka üzerine (plaka başına 250) yerleştirin ve genç yetişkin aşamasına kadar 21 ° C'de 3 gün boyunca büyütün.
- Hayvanları% 0.01 (h / h) TX-100 (PBS / TX) içeren fosfat tamponlu salin (PBS) ile 1.5 mL'lik tüplere yıkayın. 2 dakika boyunca 1.000 x g'da santrifüjleyin. Hayvanları bir suştan bir tüpe aspire edin ve toplayın ve en az 1 mL PBS / TX ile üç kez daha yıkayın.
- 1.000 μL'ye aspire edin, karıştırmak için ters çevirin ve 3 μL'deki hayvan sayısını üç kez sayın (bkz. adım 2.3.9).
- Hayvanların konsantrasyonunu hesaplayın ve formaldehit çapraz bağlama için 3.000 hayvana karşılık gelen bir hacmi yeni bir tüpe aktarın.
- Toplam hacmin solucan pelet hacminin en az 10 katı olduğundan emin olun.
5. Formaldehit çapraz bağlama
DİKKAT: Buhara maruz kalmayı önlemek için çeker ocakta formaldehit ile çalışın.
- Solucanları içeren tüpe formaldehit (% 1.8 nihai konsantrasyon) ekleyin. Oda sıcaklığında 6 dakika döndürün.
- Hemen sıvı nitrojen içinde dondurun. Bu adımda deneye ara verilebilir ve numuneler -80 °C'de saklanabilir.
- Çapraz bağlı numuneyi oda sıcaklığındaki bir su banyosunda 3 dakika çözdürün ve oda sıcaklığında 16 dakika döndürün.
- 125 mM'lik bir son konsantrasyona 1.25 M glisin ekleyin ve 5 dakika döndürün.
NOT: Manyetik boncuk elüsyonuna kadar, numuneleri buz üzerinde veya 4 °C'de tutun ve buz gibi soğuk tamponlar kullanın. - Numuneyi 3 dakika boyunca 1.000 x g'da santrifüjleyin. Her seferinde 1 mL PBS/TX ile üç kez yıkayın. 1 mL resüspansiyon tamponu (150 mM NaCl, 50 mM HEPES-KOH [pH 7.5], 1 mM etilendiamintetraasetik asit [EDTA], %0.01 TX-100, proteaz inhibitörü [5 mL başına bir tablet]) ile iki kez yıkayın.
- Son yıkamadan sonra, toplam hacmin pelet hacminin en az üç katı ve minimum ~100 μL olmasını sağlamak için yeterli tampon bırakın.
6. Sonikasyon
NOT: Sonikasyon parametreleri, sonikatör ve hayvan aşamasının tipine ve modeline bağlıdır. Numune hacmi ve konsantrasyonu, açma/kapama aralıkları, döngü sayısı ve güç ayarı gibi parametrelerin ampirik olarak optimize edilmesi gerekir. Örneğin, bir sonikasyon süresi boyunca, bir protein tahlili kullanarak solucan lizizini izleyin ve konsantrasyonun bir platoya ne zaman ulaştığını belirleyin. Ek olarak, genomik DNA'nın ortalama kesme boyutunun yaklaşık 200-1,000 bp olduğu zaman, %1.5'lik bir agaroz/tris-asetat-EDTA (TAE) jeli üzerinde çapraz bağlanmanın tersine çevrilmesinin ardından saflaştırılan DNA'nın elektroforezi ile izleyin.
- Önceki adımdaki numunelerin hacmini ölçün. Karıştırın ve 90-120 μL'yi bir polistiren sonikasyon tüpüne ayırın.
- 2x deterjan (150 mM NaCl, 50 mM HEPES-KOH [pH 7.5], 1 mM EDTA, %0.2 sodyum deoksikolat, %0.7 sarkosil) içeren eşit hacimde yeniden süspansiyon tamponu ekleyin.
- 4 ° C'de 7 dakika (20 s açık / 40 s kapalı) boyunca% 50 güçte bir su banyosu sonikatöründe sonikat. Pipetleme ile yavaşça karıştırın. Sonikasyonu 7 dakika daha tekrarlayın.
- Sonikasyonlu lizatı 1.5 mL'lik bir tüpe aktarın. Deterjan içermeyen 0,5 hacim yeniden süspansiyon tamponu ekleyin (150 mM NaCl, 50 mM HEPES-KOH [pH 7,5], 1 mM EDTA) (örneğin, 200 μL numuneye 100 μL tampon ekleyin).
- 4 °C'de 15 dakika boyunca 13.000 x g'da santrifüjleyin. Lizat süpernatanı tutun ve yeni bir tüpe aktarın.
- İsteğe bağlı olarak, her bir lizatın konsantrasyonlarını belirlemek için bir protein tahlili yapın. Bu adım, büyük numune miktarları veya tahliller arası karşılaştırmalar için yararlı olabilir. Bununla birlikte, yukarıda açıklandığı gibi hayvan sayısının standartlaştırılması, burada açıklanan numune miktarları için iyi sonuç verir, çünkü qPCR ile belirlenen kromatin/DNA verimi ile daha iyi bir korelasyon vardır.
7. İmmünopresipitasyon
NOT: Antikor ve manyetik boncuk miktarını lizatın hacmine ve konsantrasyonuna göre ölçeklendirin.
- Lizat süpernatanı dört parçaya bölün: her IP için üç eşit hacim ve Giriş olarak bir IP'nin hacminin %10'u (örneğin, 100 μL IP #1, 100 μL IP #2, 100 μL IP #3, 10 μL Giriş). IP lizatını 200 μL'lik bir PCR tüpüne aktarın ve Giriş lizatını -20 °C'de 1.5 mL'lik bir tüpte saklayın.
- Uygun IP örneğine 0.5 μg anti-H3K9me3 antikoru, anti-histon H3 antikoru veya IgG ekleyin. Rotasyon ile gece boyunca 4 °C'de inkübe edin.
- Ertesi gün, IP başına 9 μL protein G kaplı manyetik boncukları tek bir 1.5 mL'lik tüpe ayırın. Boncukları iki kez 1 mL FA-150 (150 mM NaCl, 50 mM HEPES-KOH [pH 7.5], 1 mM EDTA, %1 TX-100, %0.1 sodyum deoksikolat) ile yıkayın.
- FA-150 tamponundaki manyetik boncukları, yukarıdaki adım 7.3'te stoktan alınan orijinal hacme yeniden süspanse edin. Her IP'ye 7,5 μL ekleyin. Rotasyonla 2 saat boyunca 4 ° C'de inkübe edin.
8. Yıkama ve elüsyon
NOT: Manyetik boncukların kurumamasını sağlamak için, önceki yıkamayı aspire ettikten sonra her bir yıkama veya elüsyon tamponunu hızlı bir şekilde ekleyin.
- Boncukları yıkamak için her seferinde aşağıdaki tampon çözeltilerinden 0.2-1 mL kullanın. Yıkamaları aspire etmek için boncukları manyetik bir stand üzerinde toplayın. Her yıkamayı 4 °C'de 5 dakika döndürerek inkübe edin. Aşağıdaki sırayı takip edin.
- FA-150 ile iki kez yıkayın.
- FA-1M ile bir kez yıkayın (1 M NaCl ile FA-150).
- FA-0.5M (0.5 M NaCl ile FA-150) ile bir kez yıkayın. Yeni bir PCR tüpüne aktarın.
- TE-LiCl (250 mM LiCl, 10 mM Tris-Cl [pH 8.0], 1 mM EDTA, %1 IGEPAL CA-630, %1 sodyum deoksikolat) ile bir kez yıkayın.
- TE + (50 mM NaCl, 10 mM Tris-Cl [pH 8.0], 1 mM EDTA,% 0.005 IGEPAL CA-630) ile iki kez yıkayın.
NOT: Aksi belirtilmedikçe oda sıcaklığında numune işlemeye devam edin.
- Son yıkama tamponunu aspire edin. Manyetik boncukları 50 μL ChIP elüsyon tamponu (200 mM NaCl, 10 mM Tris-Cl [pH 8.0], 1 mM EDTA,% 1 sodyum dodesil sülfat [SDS]) ile yeniden süspanse edin ve 1.5 mL'lik bir tüpe aktarın.
- 65 °C'de 15 dakika boyunca bir termomikserde 1,000 rpm'de dakikada 5 saniye karıştırarak elute edin.
- Boncukları manyetik bir stand üzerinde toplayın ve süpernatanı yeni bir tüpe aktarın.
- Elüsyonu başka bir 50 μL ChIP elüsyon tamponu ile tekrarlayın. Süpernatantları toplam 100 μL için bir araya getirin. Aşağıda ayrıntılı olarak açıklandığı gibi çapraz bağlanmayı ve DNA elüsyonunu tersine çevirmeye devam edin.
9. Ters çapraz bağlama ve DNA elüsyonu
- Giriş lizat örneğini çözün. ChIP elüsyon tamponu ile 100 μL'ye kadar doldurun.
- Her IP ve Giriş örneğine 16,5 μg RNase A ekleyin. 37 °C'de 1 saat inkübe edin.
- 40 μg Proteinaz K ekleyin ve 55 °C'de 2 saat inkübe edin. Daha sonra gece boyunca 65 °C'de inkübe edin.
- Numuneleri oda sıcaklığına soğutun ve DNA'yı bir spin-kolon kiti ile saflaştırın.
10. qPCR reaksiyonu kurulumu ve çalıştırması
NOT: Primer, reaksiyon kurulumu ve termosiklatör parametreleri, kullanımdaki qPCR reaksiyon karışımı için üreticinin tavsiyelerine uyacak şekilde değiştirilmelidir.
- GFP RNAi raportörünü ve H3K9me3 pozitif ve negatif zenginleştirme kontrol bölgelerini hedefleyen primer setleri hazırlayın. Astar erime sıcaklığı 60 °C'dir. Astar dizileri için Malzeme Tablosuna bakın.
- Giriş DNA'sı için dört adet dörtlü seri seyreltme yapın (örneğin, 1:5, 1:20, 1:80, 1:320).
NOT: IP DNA, daha fazla reaksiyona izin vermek için doğrudan veya seyreltilmiş (örneğin, 1:2 veya 1:3) kullanılabilir. qPCR performansı etkilenebileceğinden, seyreltmenin uygunluğu ampirik olarak belirlenmelidir. - Bir PCR plakasında bir primer setine karşılık gelen tüm reaksiyonları düzenleyin. Her Giriş DNA seyreltmesi ve her IP DNA örneği için teknik yinelenen reaksiyonlar ayarlayın. Her 10 μL reaksiyon, 1.5 μL Giriş veya IP DNA (veya kontrol olarak elüsyon tamponu), qPCR ana karışımı (1x nihai konsantrasyon), ileri primer ve ters primer (her primer için 400 nM nihai konsantrasyon) içerir.
- Gerçek zamanlı bir termocycler'da aşağıdaki programı çalıştırın: ilk denatürasyon: 4 dakika boyunca 95 °C; amplifikasyon ve floresan algılama: 10 sn boyunca 95 °C'lik 40 döngü ve 30 sn boyunca 60 °C'de bir plaka okunduğunda; son uzatma: 5 dakika boyunca 60 °C; erime eğrisi: 0,5 °C'lik artışlarla 60 °C'den 90 °C'ye, adım başına 5 sn.
11. Amplifikasyon verimliliğinin belirlenmesi ve ürün özgüllüğünün doğrulanması
- Her bir Giriş DNA dilüsyon seti için, x ekseninde [log10(1/seyreltme)] ve y ekseninde [Input Cq] ile dört veri noktasını çizin. En uygun çizginin eğimini belirleyin.
- Amplifikasyon verimliliğini hesaplayın. İdeal astar setleri sürekli olarak %95-%100 verimlilik göstermelidir.
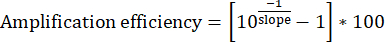
- Tüm reaksiyonların keskin bir erime eğrisi tepe noktasına sahip olduğunu ve aynı primer setine sahip reaksiyonların aynı erime sıcaklığına sahip olduğunu kontrol edin. Birden fazla tepe noktası veya farklı erime sıcaklıkları, spesifik olmayan amplifikasyonu gösterebilir.
- İsteğe bağlı olarak, ürün bandı boyutunu doğrulamak için oda sıcaklığında standart %2 agaroz/TAE jeli üzerinde reaksiyonlar çalıştırın.
12. Giriş yüzdesinin hesaplanması
- Giriş seyreltme faktörünü hesaplayın. IP lizat hacminin %10'u Girdi olarak kaydedildiğinden, Girdi DNA'sının 1:5 seyreltilmesi için seyreltme faktörü 50'dir.
- IP seyreltme faktörünü belirleyin. IP DNA'sı qPCR'den önce seyreltilmemişse, seyreltme faktörü 1'dir.
- Seyreltme faktörleri için ayarlanmış IP ve Giriş arasındaki Cq farkını hesaplayın.
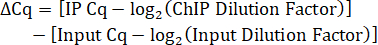
- Giriş yüzdesini hesaplayın.
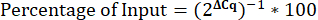
Sonuçlar
Germ hattı ile eksprese edilen GFP-histon H2B [mex-5p::gfp-h2b::tbb-2 3'UTR]7 raportörünü taşıyan hayvanlar (Şekil 2A) beslenerek GFP RNAi'ye veya kontrol RNAi'ye maruz bırakıldı ve protokolde ve Şekil 1'de açıklandığı gibi geçti. Germ hattındaki nükleer GFP sinyali, her nesildeki popülasyonun bir örneği için bir floresan diseksiyon mikroskobu kullanılarak manuel olarak puanlandı. Transgenin susturulması, GFP RNAi ile tedavi edilen skorlanmış P0 ve F1 hayvanlarında tamamen nüfuz ediciydi (Şekil 2B). F2 neslinde, GFP susturma kalıtımı sergileyen popülasyonun oranı yaklaşık% 50 idi. F5 kuşağı tarafından, popülasyonun çoğunluğu susturma kalıtımı göstermedi ve F10 kuşağı tarafından, tüm hayvanlar GFP'yi ifade ettiği için kalıtım tespit edilmedi.
RNAi ile indüklenen susturmaya karşılık gelen histon H3K9me3 zenginleşmesindeki değişikliği belirlemek için, GFP RNAi veya kontrol RNAi tedavisini takiben F1 nesil hayvanlarda ChIP-qPCR gerçekleştirildi. Beklendiği gibi, GFP RNAi ile tedavi edilen popülasyon, kontrol RNAi ile tedavi edilen hayvanlara kıyasla GFP hedefinde ve 1.3 kb aşağı akış bölgesinde daha yüksek histon H3K9me3 seviyeleri sergiledi (Şekil 2C). Histon H3K9me3 ChIP'nin özgüllüğü, bu işarette zenginleştirildiği bilinen, ancak yakındaki bir negatif kontrol lokusunda (hrp-2) olmayan bir pozitif kontrol lokusunda (clec-18) zenginleştirme ile desteklenir. Beklendiği gibi tüm qPCR lokuslarında histon H3 zenginleşmesi ve IgG kontrol immünopresipitasyonlarında arka plana yakın zenginleşme de tespit edildi. Raportördeki ChIP zenginleştirmesi clec-18 pozitif kontrol lokusuna normalleştirildiğinde, GFP RNAi üzerinde daha yüksek histon H3K9me3 zenginleşmesi gösterilirken, histon H3 zenginleştirmesi kontrol ve GFP RNAi tedavileri arasında benzerdir (Şekil 2D). GFP RNAi'nin clec-18 lokusundaki histon H3K9me3 veya toplam histon H3 doluluğunu etkilemesi beklenmediğinden, bu normalizasyon, GFP RNAi ve kontrol RNAi numuneleri arasındaki ChIP verimliliğindeki farklılıklar gibi teknik varyasyonlara karşı hafifletir. RNAi tedavileri arasında histon H3K9me3 ve histon H3 seviyelerinin kat değişimi, GFP RNAi kaynaklı susturma üzerine histon doluluğundan bağımsız olarak GFP muhabirine özgü histon H3K9me3 zenginleşmesini gösterir (Şekil 2E).
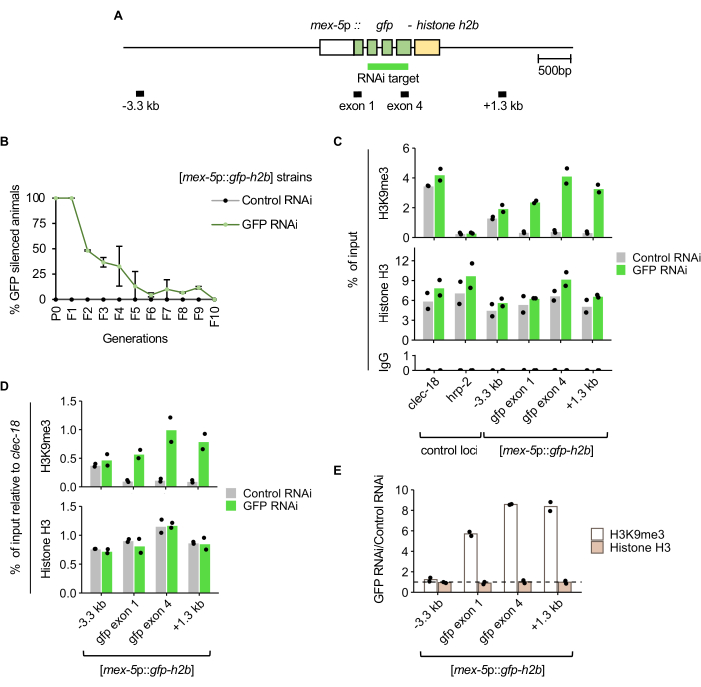
Şekil 2: GFP RNAi kaynaklı susturma, RNAi hedefinde yüksek H3K9me3 zenginleşmesine karşılık gelir. (A) qPCR amplikon bölgeleri etiketli germ hattı ile eksprese edilen GFP RNAi raportörü mjIs134[mex-5p::gfp-h2b::tbb-2 3'UTR] diyagramı. (B) GFP ekspresyonu, 21 ° C'de RNAi tedavilerinden sonra nesiller boyunca puanlandı. Hata çubukları, iki biyolojik kopyadan standart sapmayı temsil eder. (C) İki biyolojik kopyadan F1 genç yetişkinlerde H3K9me3, histon H3 ve IgG kontrolünün ChIP-qPCR'si. clec-18 ve hrp-2, sırasıyla H3K9me3 zenginleştirmesi için pozitif ve negatif kontrol lokuslarıdır. (D) GFP RNAi raporlayıcısında H3K9me3 ve histon H3 zenginleşmesi, clec-18 pozitif kontrol lokusuna normalize edildi. (E) CLEC-18'e normalizasyon ile GFP RNAi ve kontrol RNAi ile tedavi edilen hayvanlar arasında H3K9me3 ve histon H3 zenginleşmesinde değişiklik. Noktalı çizgi, 1'lik bir katlama değişimini temsil eder. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Bu protokolde, dsRNA, C. elegans18'de standart bir yöntem haline gelen besleme yoluyla tanıtılır. RNAi kalıtım deneyleri için besleme yaklaşımı, büyük bir P0 popülasyonu 2,11,12,22,23,24,25 elde etmek için kolay bir yöntem sağlar. Bununla birlikte, RNAi maruziyetinin zamanlaması ve süresi, transgen susturmanın etkinliğini etkiler26 ve RNAi bakterilerinin konsantrasyonu, kalıtsal RNAi susturmanınkalıcılığını etkiler 1. Bu nedenle, standartlaştırılmış RNAi bakterileri ve solucan büyümesi, tutarlı bir GFP susturma seviyesi ve kalıtım süresi elde etmek için önemlidir. Burada embriyolar RNAi plakaları üzerine kaplanır, böylece P0 hayvanları yumurtadan RNAi bakterilerine maruz kalır. Alternatif yaklaşımlar, senkronize L1 24,27 veyaL4 25 evre hayvanlarını RNAi-NGM plakalarına kaplamıştır. Ek olarak, açlık ve diğer stresler RNAi kalıtımının13 sürdürülmesini etkilediğinden, gıda arzının aşırı kalabalıklaşmasını ve tükenmesini önlemek için plakalar izlenmelidir. Beslenme yoluyla RNAi'yi başlatmaya bir alternatif olarak, öncü C. elegans RNAi kalıtım çalışmalarından bazıları, dsRNA konsantrasyonunun daha fazla kontrolünü sağlayangonad enjeksiyonu yoluyla RNAi'yi indükledi 1,28.
Bu protokolde, her nesil, daha önce tarif edildiği gibialkalin hipoklorit muamelesi ile bir popülasyon olarak kabul edilir 16,22,23,24. Hipoklorit arıtımı, F1 neslinin ebeveyn ortamından22 RNAi bakterileri ile kontamine olmamasını sağlar ve olası istenmeyen popülasyon darboğazını önler. Bununla birlikte, toplu geçiş de bir sınırlama olabilir, çünkü bireysel hayvanlar farklı kalıtım modellerinesahip olabilir 1,29. Her nesli oluşturmak için alternatif bir yöntem, tek tek hayvanlarıseçmektir 1,2,9,11,25. Bu yaklaşım, soylar içindeki fenotiplerin izlenmesine ve genetik çaprazlamaların dahil edilmesine izin verir. Soy analizi, düşük penetranslı fenotipler için de avantajlı olabilir12.
Floresan protein ekspresyonu, RNAi kaynaklı susturma 2,3,4,12,14,15,16,25,30'un güçlü ve kullanışlı bir okumasıdır. Bu protokolde açıklandığı gibi, GFP ifadesi manuel olarak AÇIK veya KAPALI 11,12,15,16,25 olarak puanlanabilir. Manuel puanlama,nitel yoğunluk seviyeleri 3,4,14 atanarak daha da iyileştirilebilir. Alternatif olarak, mikroskopi görüntülerinin 11,12,14,25,27 otomatik yoğunluk puanlaması veya canlı hayvanların akış sitometrisi floresan ölçümü2 kantitatif ve yüksek verimli bir okuma sağlayabilir. Bununla birlikte, raportör ifadesi germ hattı ile sınırlı olduğundan, otomatik yaklaşımların bir uyarısı, özellikle çalışma anormal germ hattı gelişimine sahip mutantları içeriyorsa, susturulmuş GFP ekspresyonu olan hayvanlar ile germ hattı olmayan hayvanlar arasında ayrım yapmaları gerektiğidir. GFP floresansına alternatif olarak, RNAi hedef pre-mRNA ve mRNA seviyeleri 2,16,23,24'ü ölçmek için ters transkripsiyon (RT)-qPCR kullanılabilir. Bu yaklaşım, RNA'yı hedef alan susturmanın daha doğrudan bir okumasını sağlar ve özellikle susturmanın görünür bir fenotip üretmediği diğer RNAi hedefleri için yararlıdır. Yapay GFP raportörlerinin kullanılmasının bir sınırlaması, eksojen ve endojen dizilerin nesiller arası RNAi11'de diferansiyel olarak düzenlenmesidir. Bu nedenle, sıcaklığa duyarlı embriyonik öldürücü alel oma-1(zu405)1,11,16,25 gibi endojen hedeflerle yapılan çalışmalar, floresan transgen raportörlerine tamamlayıcı bir yaklaşım olarak düşünülmelidir.
ChIP'nin RNAi kalıtım testleri bağlamında analizi, tedaviler ve replikasyonlar arasında karşılaştırma yapılmasını gerektirir. İlk olarak, numuneler arasındaki başlangıç malzemesindeki farklılıkları hesaba katmak için, ChIP sinyali, 'Giriş yüzdesi' ile aynı lokusta Giriş sinyaline normalleştirilir. Bir histon H3 ChIP'nin paralel olarak işlenmesi, herhangi bir histon modifikasyon değişikliğinin nükleozom yoğunluğundaki değişikliklere karşılık gelip gelmediğini belirlemeye yardımcı olacaktır. Ek olarak, ChIP çok adımlı bir süreç olduğundan, verimlilik numuneler arasında değişebilir. Uygun pozitif ve negatif kontrol lokuslarının seçimi, numuneler ve deneyler arasındaki sinyal-gürültü oranını değerlendirmek ve karşılaştırmak için yararlıdır. Ek olarak, numuneler arasındaki karşılaştırmaları kolaylaştırmak için, RNAi hedefindeki ChIP DNA qPCR eşik döngüsü değerleri genelliklebir kontrol odağı 3,11,15,23,30,31'e normalleştirilir. RNAi tedavisinin etkilerini değerlendirmek için, tedavi RNAi'sindeki ChIP sinyalinin kontrol veya RNAi olmayan koşullara oranı da karşılaştırılır. Mevcut yaklaşımın bir sınırlaması, ChIP'nin bütün hayvanlarla gerçekleştirilmesidir, RNAi tedavisine verilen yanıt ise germ hattına özgü olabilir. Bu uyarının üstesinden gelmek için bir yaklaşım, izole germ hattı çekirdeklerini kullanarak ChIP gerçekleştirmektir. ChIP'yi optimize etmek için ek teknik hususlar da başka bir yerde kapsamlı bir şekilde tartışılmıştır32,33.
Genel olarak, bu RNAi kalıtımı ve ChIP protokolü, nesiller arası epigenetik düzenlemeyi daha fazla keşfetmek için diğer tekniklerle entegre edilebilecek ayrıntılı ve uyarlanması kolay bir temel sağlar. Örneğin, kromatin manzarasının hem RNAi hedefine yakın hem de genom çapında bir ölçekte daha ayrıntılı bir görünümü için ChIP DNA'dan (ChIP-seq) yüksek verimli dizileme kütüphaneleri oluşturulabilir.
Açıklamalar
Tüm yazarlar, açıklanacak herhangi bir çelişkileri olmadığını onaylar.
Teşekkürler
Araçları geliştiren ve paylaşan ve bu makalede çalışmalarına atıfta bulunulan C. elegans topluluğundaki laboratuvarlara teşekkür etmek istiyoruz. Bazı suşlar, NIH Araştırma Altyapısı Programları Ofisi (P40 OD010440) tarafından finanse edilen CGC tarafından sağlanmıştır. Bu çalışma, Kanada Sağlık Araştırmaları Enstitüleri (CIHR) Projesi tarafından A.L.S.'ye (PJT-175245) hibe ile desteklenmiştir. CL, Kanada Doğa Bilimleri ve Mühendislik Araştırma Konseyi (NSERC) lisansüstü bursu (PGS-D) tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Agarose | Bioshop | AGA002 | |
| Ampicillin | Bioshop | AMP201 | Make a 100 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Anti-H3K9me3 Rabbit Polyclonal Antibody | Abcam | ab8898 | Concentration is batch-dependent (0.9 - 1 mg/mL). |
| Anti-Histone H3 Rabbit Polyclonal Antibody | Abcam | ab1791 | Concentration is batch-dependent (0.7 - 1 mg/mL). |
| Bleach (6% Sodium hypochlorite) | Lavo | 02358107 | |
| C. elegans strain with GFP RNAi Reporter | NA | SX1263 | Sapetschnig et al. 2015 (ref. 7). A gift from E. Miska lab, University of Cambridge. |
| Carbenicillin | BioShop | CAR544 | Make a 25 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Dynabeads Protein G Magnetic Beads | Invitrogen | 10003D | |
| E. coli strain HT115(DE3) | Caenorhabditis Genetics Center (CGC) | HT115(DE3) | |
| E. coli strain OP50-1 | Caenorhabditis Genetics Center (CGC) | OP50-1 | |
| EDTA (0.5 M, pH 8.0) | Invitrogen | 15575020 | |
| Fluorescence Stereoscope | Zeiss | Axio Zoom.V16 | |
| Formaldehyde (37%) | Sigma | F8775 | |
| Glycine | Sigma | 50046 | Make a 1.25 M solution and store at 4 °C. |
| HEPES-KOH (1 M, pH 7.5) | Teknova | H1035 | |
| Hydrophobic Printed Slides, 10 wells | VWR | 100488-904 | |
| IGEPAL CA-630 (Octylphenol ethoxylate) | BioShop | NON999 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
| IPTG (Isopropyl-β-D-thiogalactoside) | BioShop | IPT001 | Make a 0.2 g/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725122 | |
| LB Agar Plates supplemented with 100 µg/mL Ampicillin | NA | NA | Standard lab recipe. |
| Levamisole (Tetramisole hydrochloride) | Sigma | L9756 | Make a 200 mM solution in ultrapure water. Store at -20 °C. |
| LiCl (8 M) | Sigma | L7026 | |
| M9 Buffer | NA | NA | 22 mM KH2PO4, 42 mM Na2HPO4, 86 mM NaCl, 1 mM MgSO4. |
| Magnetic Separator (1.5 mL tubes) | Applied Biosystems | A13346 | |
| Magnetic Separator (0.2 mL tubes) | Permagen | MSR812 | |
| Microscope Cover Glass | Fisher Scientific | 12541B | |
| Microscope Slide | Technologist Choice | LAB-037 | |
| NaCl (5 M) | Promega | V4221 | For ChIP buffers. |
| NaOH | Sigma | S5881 | Make a 10 M solution and store at room temperature. |
| NGM Plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 μg/mL cholesterol, 25 mM Potassium phosphate pH 6.0, 1 mM MgSO4, 50 µg/mL streptomycin. |
| Normal Rabbit IgG (1 mg/mL) | Cell Signaling Technology | 2729 | |
| Petri Dishes (35 mm x 10 mm) | Sarstedt | 82.1135.500 | |
| Phosphate Buffered Saline (10X) | Fisher BioReagents | BP3991 | |
| Plasmid - Control RNAi | Addgene | L4440 (Plasmid #1654) | |
| Plasmid - GFP-targetting RNAi | Addgene | L4417 (Plasmid #1649) | Note, alternative L4440-derived plasmids targeting GFP can be used. |
| Primer pair [-3.3 kb upstream of gfp] | Integrated DNA Technologies | NA | F: AAACCAAAGGACGAGAGATTCA, R: GGCTCGATCAAGTAAAATTTCG |
| Primer pair [+1.3 kb downstream of gfp] | Integrated DNA Technologies | NA | F: TCGACCAGTTCTAAAGTCACCG, R: ACGTGCGGGATCATTTCTTACT |
| Primer pair [clec-18] | Integrated DNA Technologies | NA | F: TGCTCCATGACCTCAACAACA, R: AGTACAGTTCACCGATCCAGA |
| Primer pair [gfp exon 1] | Integrated DNA Technologies | NA | F: CTGGAGTTGTCCCAATTCTTGT, R: GGGTAAGTTTTCCGTATGTTGC |
| Primer pair [gfp exon 4] | Integrated DNA Technologies | NA | F: GATGGCCCTGTCCTTTTACCA, R: ATGCCATGTGTAATCCCAGCA |
| Primer pair [hrp-2] | Integrated DNA Technologies | NA | F: CGTCAACAGGGAGCAGCTG, R: CCTCCGAACTTTCTCTGTCCA |
| Protease Inhibitor Cocktail Tablet | Roche | 11836170001 | |
| Proteinase K | Bioline | BIO-37084 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Real-Time PCR Detection System | Bio-Rad | CFX96 | |
| RNAi-NGM plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 µg/mL cholesterol, 25 mM Potassium phosphate buffer pH 6.0, 1 mM MgSO4, 25 µg/mL carbenicillin and 5 mM IPTG. |
| RNase A | Sigma | R4642 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma | L5777 | Make a 10% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| SDS | Sigma | 74255 | Make a 10% (w/v) solution and store at room temperature. |
| Sodium deoxycholate | Sigma | 30970 | Make a 5% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| Sonication Tube | Evergreen | 214-3721-010 | |
| Sonication Tube Cap | Evergreen | 300-2911-020 | |
| Sonicator | Qsonica | Q800R3-110 | |
| Streptomycin sulfate | Bioshop | STP101 | Make a 50 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| TAE buffer (1X) | NA | NA | 40 mM Tris, 20 mM acetate, 1 mM EDTA |
| Tally counter clicker | Uline | H-7350 | |
| Tetracycline | Bioshop | TET701 | Make a 5 mg/mL solution in ethanol and store at -20 °C. |
| Thermomixer | Eppendorf | 05-400-205 | |
| Tris-HCl (1 M, pH 8.0) | Invitrogen | 15568025 | |
| Triton X-100 | Sigma | T8787 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
Referanslar
- Alcazar, R. M., Lin, R., Fire, A. Z. Transmission dynamics of heritable silencing induced by double-stranded RNA in Caenorhabditis elegans. Genetics. 180 (3), 1275-1288 (2008).
- Ashe, A., et al. piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell. 150 (1), 88-99 (2012).
- Buckley, B. A., et al. A nuclear Argonaute promotes multigenerational epigenetic inheritance and germline immortality. Nature. 489 (7416), 447-451 (2012).
- Vastenhouw, N. L., et al. Long-term gene silencing by RNAi. Nature. 442 (7105), 882 (2006).
- Lee, H. -. C., et al. C. elegans piRNAs mediate the genome-wide surveillance of germline transcripts. Cell. 150 (1), 78-87 (2012).
- Luteijn, M. J., et al. Extremely stable Piwi-induced gene silencing in Caenorhabditis elegans. The EMBO Journal. 31 (16), 3422-3430 (2012).
- Sapetschnig, A., Sarkies, P., Lehrbach, N. J., Miska, E. A. Tertiary siRNAs mediate paramutation in C. elegans. PLoS Genetics. 11 (3), e1005078 (2015).
- Shirayama, M., et al. piRNAs initiate an epigenetic memory of nonself RNA in the C. elegans germline. Cell. 150 (1), 65-77 (2012).
- Minkina, O., Hunter, C. P. Stable heritable germline silencing directs somatic silencing at an endogenous locus. Molecular Cell. 65 (4), 659-670 (2017).
- Seroussi, U., et al. Mechanisms of epigenetic regulation by C. elegans nuclear RNA interference pathways. Seminars in Cell & Developmental Biology. 127, 142-154 (2022).
- Lev, I., Gingold, H., Rechavi, O. H3K9me3 is required for inheritance of small RNAs that target a unique subset of newly evolved genes. eLife. 8, e40448 (2019).
- Woodhouse, R. M., et al. Chromatin modifiers SET-25 and SET-32 are required for establishment but not long-term maintenance of transgenerational epigenetic inheritance. Cell Reports. 25 (8), 2259-2272 (2018).
- Houri-Zeevi, L., Teichman, G., Gingold, H., Rechavi, O. Stress resets ancestral heritable small RNA responses. eLife. 10, e65797 (2021).
- Houri-Ze'evi, L., et al. A tunable mechanism determines the duration of the transgenerational small RNA inheritance in C. elegans. Cell. 165 (1), 88-99 (2016).
- Spracklin, G., et al. The RNAi inheritance machinery of Caenorhabditis elegans. Genetics. 206 (3), 1403-1416 (2017).
- Perales, R., et al. Transgenerational epigenetic inheritance is negatively regulated by the HERI-1 chromodomain protein. Genetics. 210 (4), 1287-1299 (2018).
- Shukla, A., Perales, R., Kennedy, S. piRNAs coordinate poly(UG) tailing to prevent aberrant and perpetual gene silencing. Current Biology. 31 (20), 4473-4485 (2021).
- Ahringer, J. Reverse GeneticsWormBook: the Online Review of C. elegans Biology. WormBook. , (2006).
- Rivera Gomez, K., Schvarzstein, M. Immobilization of nematodes for live imaging using an agarose pad produced with a Vinyl Record. microPublication Biology. 2018, (2018).
- Stiernagle, T. Maintenance of C. elegans. WormBook: The Online Review of C. Elegans Biology. , 1-11 (2006).
- Askjaer, P., Ercan, S., Meister, P. Modern techniques for the analysis of chromatin and nuclear organization in C. elegans. WormBook: the Online Review of C. elegans Biology. , 1-35 (2014).
- Gu, S. G., et al. Amplification of siRNA in Caenorhabditis elegans generates a transgenerational sequence-targeted histone H3 lysine 9 methylation footprint. Nature Genetics. 44 (2), 157-164 (2012).
- Kalinava, N., Ni, J. Z., Peterman, K., Chen, E., Gu, S. G. Decoupling the downstream effects of germline nuclear RNAi reveals that H3K9me3 is dispensable for heritable RNAi and the maintenance of endogenous siRNA-mediated transcriptional silencing in Caenorhabditis elegans. Epigenetics & Chromatin. 10, 6 (2017).
- Kalinava, N., et al. elegans heterochromatin factor SET-32 plays an essential role in transgenerational establishment of nuclear RNAi-mediated epigenetic silencing. Cell Reports. 25 (8), 2273-2284 (2018).
- Lev, I., et al. MET-2-dependent H3K9 methylation suppresses transgenerational small RNA inheritance. Current Biology. 27 (8), 1138-1147 (2017).
- Kamath, R. S., Martinez-Campos, M., Zipperlen, P., Fraser, A. G., Ahringer, J. Effectiveness of specific RNA-mediated interference through ingested double-stranded RNA in Caenorhabditis elegans. Genome Biology. 2 (1), (2001).
- Xu, F., et al. A cytoplasmic Argonaute protein promotes the inheritance of RNAi. Cell Reports. 23 (8), 2482-2494 (2018).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287 (5462), 2494-2497 (2000).
- Houri-Zeevi, L., Korem Kohanim, Y., Antonova, O., Rechavi, O. Three rules explain transgenerational small RNA inheritance in C. elegans. Cell. 182 (5), 1186-1197 (2020).
- Burton, N. O., Burkhart, K. B., Kennedy, S. Nuclear RNAi maintains heritable gene silencing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences. 108 (49), 19683-19688 (2011).
- Mao, H., et al. The Nrde pathway mediates small-RNA-directed histone H3 lysine 27 trimethylation in Caenorhabditis elegans. Current Biology. 25 (18), 2398-2403 (2015).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Mukhopadhyay, A., Deplancke, B., Walhout, A. J. M., Tissenbaum, H. A. Chromatin immunoprecipitation (ChIP) coupled to detection by quantitative real-time PCR to study transcription factor binding to DNA in Caenorhabditis elegans. Nature Protocols. 3 (4), 698-709 (2008).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır