Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
İnsan Kök Hücre Kaynaklı Orta Beyin Dopaminerjik Nöronlarının Fenotipik Profillemesi
Bu Makalede
Özet
Bu protokol, insan orta beyin dopaminerjik nöronlarının hücre kültürünü, ardından immünolojik boyamayı ve genetik veya kimyasal modülasyonlara bağlı fenotipik varyasyonların tanımlanmasına izin veren elde edilen mikroskobik yüksek içerikli görüntülerden nöronal fenotipik profillerin oluşturulmasını tanımlar.
Özet
Parkinson hastalığı (PH), orta beyin dopaminerjik (mDA) nöron kaybına neden olan bir dizi hücre biyolojik süreciyle bağlantılıdır. Mevcut birçok in vitro PD hücresel modeli karmaşıklıktan yoksundur ve çoklu fenotipleri hesaba katmaz. İnsan kaynaklı pluripotent kök hücre (iPSC) kaynaklı mDA nöronlarındaki fenotipik profilleme, PD ile ilgili bir hücre tipinde bir dizi nöronal fenotipi paralel olarak aynı anda ölçerek bu eksiklikleri giderebilir. Burada, ticari olarak temin edilebilen insan mDA nöronlarından fenotipik profiller elde etmek ve analiz etmek için bir protokol açıklıyoruz. Nükleer, α-sinüklein, Tirozin hidroksilaz (TH) ve Mikrotübül ile ilişkili protein 2 (MAP2) ile ilgili fenotipleri görselleştirmek için nörona özgü bir floresan boyama paneli kullanılır. Açıklanan fenotipik profilleme protokolü, 384 oyuklu plakalar, otomatik sıvı işleme ve yüksek verimli mikroskopi kullandığından ölçeklenebilir. Protokolün faydası, Lösin açısından zengin tekrar kinaz 2 (LRRK2) geninde PD'ye bağlı G2019S mutasyonunu taşıyan sağlıklı donör mDA nöronları ve mDA nöronları kullanılarak örneklendirilmiştir. Her iki hücre hattı da LRRK2 kinaz inhibitörü PFE-360 ile tedavi edildi ve fenotipik değişiklikler ölçüldü. Ek olarak, çok boyutlu fenotipik profillerin kümeleme veya makine öğrenimine dayalı denetimli sınıflandırma yöntemleri kullanılarak nasıl analiz edilebileceğini gösteriyoruz. Açıklanan protokol, özellikle nöronal hastalık modellemesi üzerinde çalışan veya insan nöronlarındaki kimyasal bileşik etkilerini inceleyen araştırmacıların ilgisini çekecektir.
Giriş
Parkinson hastalığında (PH) çeşitli hücre biyolojik süreçleri bozulur. Örneğin, mitokondriyal disfonksiyon, oksidatif stres, protein yıkım kusurları, veziküler kaçakçılığın bozulması ve endolizozomal fonksiyon, orta beyin dopaminerjik (mDA) nöron kaybı ile ilişkilendirilmiştir ve PD1'de yaygın olarak gözlenir. Bu nedenle, PH, birbiriyle etkileşime girebilen ve birbirini kötüleştirebilen çoklu hastalık mekanizmalarını içeriyor gibi görünmektedir. Bu mekanik etkileşimi araştırmanın yararlı bir yolu, kapsamlı bir fenotipik parmak izinin veya orta beyin dopaminerjik (mDA) nöronlarının profilinin oluşturulmasıdır.
Fenotipik profilleme, ölçülebilir özelliklerin bir koleksiyonuna dayalı olarak bir örneklem profilinin oluşturulmasını içeren bir yaklaşımdır ve ikincisi, bu profile dayalı olarak bir örneklem hakkında tahminlerde bulunmayı içerir 2,3. Profil oluşturmanın amacı, bazıları daha önce bir hastalık veya tedavi ile ilişkilendirilmemiş olabilecek çok çeşitli özellikleri yakalamaktır3. Sonuç olarak, profil oluşturma beklenmedik biyolojik süreçleri ortaya çıkarabilir. Fenotipik profilleme tipik olarak floresan lekeli hücrelere dayanır ve fenotipik profiller oluşturmak için Hücre Boyama gibi standartlaştırılmış testler geliştirilmiştir4. Son zamanlarda, fenotipik profilleme, örneğin, küçük moleküllerin karakterizasyonu veya yalnızca hasta kaynaklı fibroblastlaradayalı PD alt tiplerinin doğru tahmini için uygulanmıştır 5,6. Bu ilerlemelere rağmen, fenotipik profilleme, LRRK2 G2019S gibi PD'ye bağlı mutasyonları eksprese eden insan kaynaklı pluripotent kök hücre (iPSC) türevi mDA nöronları gibi mitotik sonrası farklılaşmış hücrelere nadiren uygulanmıştır. iPSC'den türetilen modellerin önemli zorlukları arasında, farklılaşma grupları veya genotipler arasında ince veya değişken patolojik özelliklerin varlığı ve izole PD fenotiplerinin hastalığın tüm karmaşıklığını yakalayamaması yer alır. Ayrıca, iPSC nöronal modelleri fizyolojik olarak ilgili olsa da, teknik karmaşıklık ile ilgili endişeler nedeniyle PD ilaç keşif süreçlerinde nadiren kullanılmaktadır 7,8.
Daha önce, hem genetik hem de kimyasal bileşik kaynaklı fenotipik değişikliklere duyarlı olan insan mDA nöronlarında PD ile ilişkili çoklu patofizyolojik fenotipleri ölçmek için sağlam bir metodoloji geliştirmiştik9. Bu makale, mDA nöronlarından fenotipik profiller oluşturmak için bu metodolojinin daha da optimize edilmiş bir versiyonunu ayrıntılı olarak açıklamaktadır (Şekil 1). Bu protokolün, yüksek kaliteli mDA nöronlarının kullanımı ve teknik tekrarlanabilirlik gibi daha önce açıklanan fenotipik profilleme yaklaşımlarına göre çeşitli avantajları vardır. İlk kez, bu protokol, kimyasal bozulmalardan sonra fizyolojik olarak ilgili post-mitotik mDA nöronlarında fenotipik profillemeyi yüksek düzeyde ölçeklenebilir bir şekilde mümkün kılar. Tamamen farklılaşmış ve dondurularak korunmuş mDA nöronları ticari olarak temin edilebilir ve partiden partiye farklılaşma değişkenliğini önemli ölçüde azaltır. İkinci olarak, teknik değişkenlik, iyi tanımlanmış bir deneysel tasarım (yani, kültür süresi veya kenar kuyularından kaçınma), otomatik sıvı işleme ve otomatik mikroskopi kullanılarak daha da azaltılabilir. Ek olarak, denetimsiz kümeleme veya denetimli sınıflandırma yaklaşımları kullanılarak fenotipik profil analizinin ilk adımları burada özetlenmiştir ve fenotipik profilleme verilerinin nasıl analiz edilebileceğini göstermektedir. Bu protokol, genetik veya kimyasal bozulmaların neden olduğu mDA nöronlarının fenotipik değişiklikleriyle ilgilenen araştırmacılar için, özellikle yüksek düzeyde ölçeklenebilir bir çalışma kurulumu gerektiğinde, örneğin tarama kampanyaları sırasında veya daha az sayıda bileşiğin etkilerinin incelenmesi gerektiğinde, örneğin toksik etkileri belirlemek için kullanılacaktır. Özetle, insan nöronlarının fenotipik profillemesinin uygulanmasının, hastalıkla ilgili karmaşık fenotipleri incelemek ve ilaç adaylarının hücresel etkilerini karakterize etmek için değerli bir teknik olduğu tahmin edilmektedir.
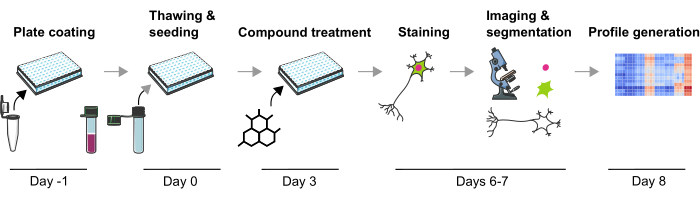
Şekil 1: İnsan iPSC'den türetilen mDA nöronlarından görüntü tabanlı fenotipik profiller oluşturmak için deneysel protokolün şematik tasviri. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protokol
1. Nöron tohumlaması için besiyeri ve plakaların hazırlanması (1. Gün)
- Plakaları Gün-1'de nöron tohumlamasına hazırlamak için, kullanmadan hemen önce Laminin'i oda sıcaklığına (RT) ısıtın. Laminin stok çözeltisini (0.1 mg / mL) soğuk PBS + / + (Ca 2 + ve Mg2 + ile) 1/10 oranında seyrelterek Laminin çözeltisini hazırlayın.
NOT: Tüm reaktifler Malzeme Tablosunda listelenmiştir. Çözeltilerin ve tamponların bileşimleri Tablo 1-4'te açıklanmıştır. - Daha sonra, önceden kaplanmış 384 oyuklu bir Poli-D-Lizin (PDL) plakasının her bir oyuğuna 25 μL Laminin çözeltisi ekleyin ve gece boyunca 4 ° C'de inkübe edin. Kaplanmış plakalar 4 °C'de bir haftaya kadar saklanabilir.
NOT: Protokol burada bir haftaya kadar duraklatılabilir. Plastik film kullanarak plakaları kapatın. - Komple Bakım Ortamı hazırlayın ve bir aya kadar 4 °C'de saklayın (Tablo 1).
2. Nöronların çözülmesi (Gün 0)
- 0. Günde nöronları çözmek için, bir su banyosunu 37 °C'ye ısıtın ve Komple Bakım Ortamını ışıktan korunarak RT'ye dengeleyin.
- Ticari olarak elde edilen donmuş nöronları içeren şişeyi ( Malzeme Tablosuna bakınız) sıvı nitrojen tankından çıkarın ve kuru buz üzerine yerleştirin. Ardından şişeyi 2 dakika boyunca su banyosuna koyun. Sıvı tamamen çözüldüğünde, şişeyi% 70 etanol ile dezenfekte edin.
- Çözülmüş nöronları bir P1000 pipeti (yaklaşık 370 μL) ile aspire edin ve yukarı ve aşağı pipetleme olmadan 50 mL'lik bir santrifüj tüpüne aktarın. Ardından, şişeyi 630 μL Tam Bakım Ortamı ile durulayın, 50 mL'lik tüpte damla damla (45° açı) dağıtın. Dağıtırken yavaşça çalkalayın.
NOT: Hücrelerin ozmotik yırtılmasını önlemek için ortamı yavaşça, damla damla dağıtmak çok önemlidir. 45°'lik açı ve hafif çalkalama, pipetleme sırasında yüksek lokal ozmotik basıncı en aza indirir. - Benzer şekilde, bir P1000 pipeti ile 50 mL santrifüj tüpüne 1 mL Tam Bakım Ortamı ekleyin. Ardından, yavaşça 2 mL Tam Bakım Ortamı ekleyin. Dağıtırken dikkatlice çalkalayın.
- Hücreleri sayın. 10 μL Tripan Blue ve 10 μL hücre süspansiyonlu bir mikrotüp hazırlayın ve bir sayma odası lamına (10 μL) ekleyin. Otomatik bir hücre sayacı kullanarak (Malzeme Tablosuna bakın) veya manuel olarak sayım yapın.
- Saydıktan sonra, nöronları içeren 50 mL'lik tüpü RT'de 5 dakika boyunca 400 x g'de santrifüjleyin ve süpernatanı çıkarın. Bir P1000 pipet ve 1 mL Tam Bakım Ortamı kullanarak peleti dikkatlice yeniden süspanse edin. Ardından istenen konsantrasyona ulaşmak için gerekli hacmi ekleyin (300.000 hücre / mL, ayrıca bir sonraki adıma bakın).
3. Hazırlanan plakalarda nöronların tohumlanması (Gün 0)
- 0. Günde hazırlanan plakalardaki nöronları tohumlamak için, kaplanmış plakaları buzdolabından çıkarın, hücre kültürü kaputunun altına yerleştirin ve yaklaşık 30 dakika boyunca RT'ye dengelenmelerini sağlayın.
- Tohumlamadan hemen önce, otomatik bir sıvı işleyici veya 16 kanallı bir pipet ile 15 μL'lik kaplama solüsyonunu aspire edin. Kaplamanın zarar görmesini önlemek için oyuk başına yaklaşık 10 μL bırakın.
- Daha sonra, 16 kanallı bir pipet ile oyuk başına 300.000 hücre / mL (adım 2.6'da hazırlanan) içeren 50 μL hücre çözeltisi dağıtın, bu da oyuk başına 15.000 tohumlanmış nöron ve 60 μL'lik bir nihai hacim ile sonuçlanır.
- 384 oyuklu bir plakada, fenotipik profilleri etkileyebilecek olası kenar etkilerini en aza indirmek için sütun 1, 2, 23, 24 ve A, B, O ve P satırlarını kullanmaktan kaçının. Kullanılmayan boş kuyuları 80 μL PBS ile doldurun. Plakaları 37 °C ve %5 CO2'de inkübe edin.
4. Orta değişim veya bileşik tedavi (3. Gün)
- Kuyu sayısına bağlı olarak, RT'de uygun hacimde Komple Bakım Ortamını önceden ısıtın. Işıktan koruyun.
- Orta düzeyde bir değişiklik gerekiyorsa, adım 4.4'e geçin. Bileşik işlem isteniyorsa, bileşik stok konsantrasyonunu ve kullanılan çözücüyü (su, DMSO, metanol vb.) doğrulayın.
- Test edilecek tüm istenen konsantrasyonlar için 1.5x konsantre bileşik çözelti hazırlayın. Bileşik dilüsyonları Tam Bakım Ortamı kullanarak hazırlayın.
NOT: Nötr bir kontrol olarak, ilgili çözücüyü test edilen bileşikle aynı konsantrasyonda kullanın. Farklı konsantrasyonlarda çoklu veya bilinmeyen bileşikler kullanılıyorsa, fenotipik profil üzerindeki çözücü etkilerini ölçmek için ayrı bir doz-yanıt deneyi yapılması tavsiye edilir. - Otomatik pipetleme sistemi kullanılıyorsa, 384 oyuklu bir saklama plakasına 60 μL 1,5x bileşik solüsyon ekleyin. Alternatif olarak, 16 kanallı bir pipet kullanın. Orta düzeyde bir değişiklik gerekiyorsa, bunun yerine 60 μL Tam Bakım Ortamı ekleyin.
- Otomatik pipetleme sistemini kullanarak, 20 μL/kuyucuk tutmak için nöron içeren plakadan oyuk başına 40 μL ortamı aspire edin ve atın. Ardından, istenen nihai konsantrasyonu elde etmek için her bir oyuğa 384 oyuklu saklama plakasından 40 μL/kuyucuk 1.5x bileşik çözelti ekleyin.
NOT: Tam ortam değişiklikleri yapmamak, ancak nöronal halıya veya kaplamaya zarar vermemek için her zaman kuyuda artık ortam bırakmak çok önemlidir. - Nöronal kültürün bu protokolde tarif edilen 6 günden fazla yapılması durumunda, ortamı 2-3 günde bir değiştirin.
5. Nöron fiksasyonu ve boyanması (6-7. günler)
- Nöronları 6. ve 7. günlerde sabitlemek ve boyamak için, tüm dağıtım ve yıkama adımları için otomatik bir sıvı işleyici kullanın. Alternatif olarak, 16 kanallı bir pipet kullanın.
- Triton X-100'ü 1x PBS'de seyrelterek %10'luk bir Triton X-100 çözeltisi hazırlayın. Çözelti homojen olana kadar girdap. 4 °C'de saklayın.
- Nöronları sabitlemek için, %4'lük bir nihai konsantrasyonla sonuçlanan 20 μL/kuyu %16 PFA dağıtın. Plakayı RT'de 30 dakika inkübe edin ve 1x PBS ile üç kez yıkayın. Son yıkamadan sonra 20 μL/kuyu PBS bırakın.
DİKKAT: PFA, oral, dermal ve solunum toksisitesine neden olduğu bilinen tehlikeli bir madde olarak kabul edilmektedir. Aynı zamanda gözler için bir tehdit oluşturur ve genetik mutasyonlara ve kansere yol açabilir. PFA'nın uygun şekilde kullanılması, uygun havalandırmanın sağlanmasına ek olarak göz ve yüz koruması gibi uygun kişisel koruyucu ekipmanların kullanılmasını gerektirir. PFA'nın çevreye salınmasını önlemek önemlidir. - Geçirgenlik ve blokaj için 2x blokaj solüsyonu hazırlayın (Tablo 2).
- 20 μL / kuyucuk 2x bloke edici çözelti (1x nihai konsantrasyon) ekleyin, RT'de 1 saat inkübe edin ve PBS ile bir kez yıkayın. Yıkadıktan sonra 20 μL/kuyu PBS tutun.
- Primer antikor (bakınız Malzeme Tablosu) boyama için, 2x primer boyama tamponu hazırlayın (Tablo 3).
- 20 μL/kuyucuk 2x birincil boyama tamponu (1x nihai konsantrasyon) ekleyin ve gece boyunca 4 °C'de inkübe edin. Ertesi sabah 7. günde, PBS ile üç kez yıkayın. Yıkadıktan sonra 20 μL/kuyu PBS bırakın.
- İkincil antikor (bkz . Malzeme Tablosu) boyama için, 2x ikincil boyama tamponu hazırlayın (Tablo 4).
- 20 μL / kuyucuk 2x ikincil boyama tamponu (1x nihai konsantrasyon) ekleyin, RT'de ışıktan uzakta 2 saat inkübe edin ve PBS ile üç kez yıkayın. Son yıkamadan sonra 100 μL PBS / kuyu bırakın.
- Buharlaşmayı en aza indirmek için plakaya alüminyum sızdırmazlık ekleyin. Alternatif olarak, plakayı plastik film ve alüminyum folyo kullanarak örtün. Görüntü almaya devam edin veya plakayı 4 °C'de saklayın.
NOT: Protokol burada bir haftaya kadar duraklatılabilir. Arka planda floresan gözlenirse, engelleme süresini artırmayı düşünün. Boyama yetersizse, birincil antikor konsantrasyonunu veya inkübasyon süresini artırmayı deneyin.
- Buharlaşmayı en aza indirmek için plakaya alüminyum sızdırmazlık ekleyin. Alternatif olarak, plakayı plastik film ve alüminyum folyo kullanarak örtün. Görüntü almaya devam edin veya plakayı 4 °C'de saklayın.
6. Floresan lekeli nöronların görüntülenmesi (7. Gün)
- 7. Günde kaplanmış, kültürlenmiş ve boyanmış nöronların görüntülerini elde edin. İdeal olarak, otomatik bir konfokal floresan mikroskobu kullanın (bkz. Alternatif olarak, görüntüleri manuel olarak alın.
- Sırasıyla 405 nm, 488 nm, 561 nm ve 647 nm lazerler kullanarak Hoechst, TH, α-sinüklein ve MAP2 kanallarını elde edin (Şekil 2).
NOT: Fenotipik profilleme için yeterli miktarda ayrıntılı veri oluşturmak için 40x'lik bir objektif kullanın ve 2 μm ile ayrılmış 3 Z diliminden oluşan Z yığınlarını kullanarak 16 alan/kuyu elde edin. Nöronal halıda pipetlemeye bağlı hasarlar varsa, bu alanları görüntülemekten kaçınmaya çalışın. - Mikroskoba ve kameraya bağlı olarak, floresan yoğunluklarının optimum dinamik aralığını elde etmek için dört floresan kanalının her biri için pozlama sürelerini ve uyarma yoğunluklarını ayrı ayrı ayarlayın.
NOT: Görüntüleme yazılımı, ideal pozlama süresini belirlemek için genellikle bir histogram sağlar. Histogram düşük sinyal aralığında çok fazla sola kaydırılırsa, pozlama süresi çok kısadır veya uyarma yoğunluğu çok düşüktür. Sağdaki maksimum sinyal seviyesinde keskin bir uçurum varsa, sinyal değeri doymuştur. Bu durumda, uyarma yoğunluğunu azaltın veya maruz kalma süresini kısaltın. - Görüntüleri .tif gibi kayıpsız ve açık bir biçimde saklayın.
7. Görüntü işleme (Gün 8)
- Kantitatif fenotipik profillerin oluşturulması için görüntü segmentasyonu ve fenotipik özellik çıkarımı gereklidir. Görüntü segmentasyonu ve özellik çıkarma için PhenoLink yazılımını kullanın (Malzeme Tablosu). PhenoLink'i yükleme talimatları GitHub deposunda (https://github.com/Ksilink/PhenoLink) bulunabilir.
NOT: Görüntü segmentasyonu, bir görüntüdeki farklı nesneleri veya bölgeleri tanımlamak ve ayırmak için kullanılırken, fenotipik özellik çıkarımı, bu bölgelerden ilgili bilgileri analiz etmek ve çıkarmak için kullanılır. Çok kanallı floresan görüntülerden nicel bilgileri çıkarmak için CellProfiler 10, ImageJ/FIJI 11, Napari12 veya Knime Analytics Platform13 gibi çeşitli alternatif yazılım çözümleri mevcuttur. - Aydınlatma düzeltmeli ham görüntülerde görüntü segmentasyonu gerçekleştirin. Arka plan sinyalinin minimum olması ve istenen segmentli sinyalin ham görüntüdeki sinyale karşılık gelmesi için plaka başına ilgili floresan kanal yoğunluğu eşiklerini ampirik olarak belirleyin. Aynı gün işlenen ve boyanan plakalar tipik olarak segmentasyon için karşılaştırılabilir kanal yoğunluğu eşikleri gerektirir.
- Canlıları ölü hücrelerden ayırmak için çekirdek boyutunu ve yoğunluğunu tanımlayın. 40x görüntü kullanırken, diğer tüm varsayılan parametreleri koruyun ve yazılımı çalıştırın. Kuyu başına yüz yirmi altı nicel görüntü özelliği hesaplanacaktır (Ek Tablo 1).
- Fenotipik profiller oluşturmak ve farklı hücre hatlarından veya tedavi koşullarından fenotipik profilleri karşılaştırmak için elde edilen tablo nicel verileri kullanın. Her satır biyolojik bir duruma (kuyuya) karşılık gelir ve her sütun belirlenmiş bir fenotipik özelliğe karşılık gelir.
NOT: Kullanımını göstermek için veri analizi boru hattıyla birlikte örnek bir çıktı dosyası sunuyoruz (bkz. Ek olarak, Şekil 3 , fenotipik bir profilin bileşimini göstermektedir.
8. Fenotipik profil oluşturma ve görselleştirme (8. Gün)
- Bilgisayarınızda Python ve Jupyter yüklü değilse, Anaconda Dağıtımını kurun ve Jupyter yazılımını açın. Sağlanan Jupyter not defterini ve sağlanan diğer tüm dosyaları indirin ve bunları aynı dizine kaydedin (bkz. Jupyter yazılımını kullanarak Jupyter not defteri dosyasını açın.
NOT: Anaconda, Python gibi programlama dilleri için ücretsiz ve açık kaynaklı bir platformdur. Bu platform, fenotipik profiller (https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling) oluşturmak ve analiz etmek için sağlanan Jupyter not defterini yürütebilen Python yorumlayıcısı Jupyter ile birlikte gelir. - Jupyter yazılımını kullanarak Jupyter not defterini hücre hücre yürütün. Sağlanan örnek veriler .fth ve dosya .txt gereksinimlerinin Jupyter not defteri ile aynı dizinde bulunması gerekir. Her Jupyter not defteri hücresine, işlevselliğini açıklamak için açıklama eklenir.
NOT: Jupyter notebook'u veri yükleme ve ölçeklendirme ile başlayarak doğru sırayla kullanmak ve sonuna kadar hücre hücre ilerlemek çok önemlidir. Tüm veriler ve grafik çıktısı, Jupyter not defteri kaynak dizininde yeni oluşturulan bir klasörde depolanır. Şekil 4'te iş akışı ve iş akışı çıktısı gösterilmektedir.
Sonuçlar
mDA nöronlarında fenotipik profilleme, hücresel biyolojinin birçok yönünü ve deneysel modülasyon sırasındaki değişikliklerini ölçmenin etkili bir yoludur. Bu metodolojiyi örneklemek için, bu çalışmada kriyoprezervasyonlu LRRK2 G2019S ve sağlıklı donör mDA nöronları kullanılmıştır. Bu nöronlar yaklaşık 37 gündür farklılaşmıştır, post-mitotik ve eksprese nöronal belirteçlerdir (TUBB3 ve MAP2) ve FOXA2 ile kombinasyon halinde tirozin hidroksilaz (TH) dahil olmak üzere dopaminerjik n...
Tartışmalar
Fenotipik profilleme, floresan boyama, mikroskopi ve görüntü analizi uygulayarak hücrelerdeki çok sayıda fenotipi ölçmek için kullanılan bir tekniktir3. Fenotipik profiller, hücresel biyolojide tek bir okuma kullanıldığında fark edilmeyebilecek karmaşık değişiklikleri anlamak için hücre hatları veya diğer deneysel koşullar arasında elde edilebilir ve karşılaştırılabilir. Burada, PD hücresel biyolojisini modellemek için sıklıkla kullanılan bir hücre tipi olan ins...
Açıklamalar
Tüm yazarlar Ksilink tarafından istihdam edilmektedir.
Teşekkürler
Yazarlar, sunulan protokolün tasarımına yol açan değerli yardımları ve tartışmaları için Ksilink'teki tüm meslektaşlarına teşekkür eder.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse - Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit - Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Referanslar
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson's disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson's disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson's Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson's Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson's disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson's disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson's disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson's disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır