Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
CRISPR-Cas9 Adeno-İlişkili Virüs (AAV) ve 2 Hücreli Embriyo Elektroporasyonu Kullanılarak Sıçan Embriyolarının Genom Düzenlemesi
Bu Makalede
Özet
Bu protokol, genetiği değiştirilmiş sıçan modelleri üretmek için optimize edilmiş bir yaklaşım sunar. Adeno-ilişkili virüs (AAV), bir DNA onarım şablonu sunmak için kullanılır ve elektroporasyon, 2 hücreli embriyoda genom düzenleme işlemini tamamlamak için CRISPR-Cas9 reaktiflerini iletmek için kullanılır.
Özet
Genom düzenleme teknolojisi, sıçanlar da dahil olmak üzere genetiği değiştirilmiş hayvanlar üretmek için yaygın olarak kullanılmaktadır. DNA onarım şablonlarının ve CRISPR-Cas reaktiflerinin sitoplazmik veya pronükleer enjeksiyonu, embriyolara en yaygın dağıtım yöntemidir. Bununla birlikte, bu tür bir mikromanipülasyon, özel ekipmana erişim gerektirir, zahmetlidir ve belirli bir düzeyde teknik beceri gerektirir. Ayrıca, mikroenjeksiyon teknikleri genellikle embriyo üzerindeki mekanik stres nedeniyle daha düşük embriyo sağkalımına neden olur. Bu protokolde, mikroenjeksiyona gerek kalmadan CRISPR-Cas9 genom düzenlemesi ile birlikte çalışacak büyük DNA onarım şablonları sunmak için optimize edilmiş bir yöntem geliştirdik. Bu protokol, 2 hücreli embriyoları modifiye etmek için elektroporasyon yoluyla CRISPR-Cas9 ribonükleoproteininin (RNP) verilmesi ile birlikte tek sarmallı DNA donör şablonlarının AAV aracılı DNA iletimini birleştirir. Bu yeni stratejiyi kullanarak, %42 ile %90 arasında verimlilikle 1.2 ila 3.0 kb boyutlarında DNA dizilerinin eklenmesini taşıyan hedefli knock-in sıçan modellerini başarıyla ürettik.
Giriş
CRISPR tabanlı genom düzenleme araçlarının geliştirilmesi, yeni ve daha karmaşık genetiği değiştirilmiş sıçan modellerini verimli bir şekilde üretme yeteneğimizi hızlandırdı. Tek kılavuzlu RNA, Cas9 nükleaz ile birlikte, genom içindeki ilgilenilen DNA dizilerini hedefleyen ve çift sarmallı DNA kırılmalarına neden olan Ribonükleoprotein (RNP) kompleksleri oluşturmak üzere birleştirilir. Hücresel DNA onarım mekanizmaları hataya açık olduğundan, onarım işlemi sırasında hedef genin işlevini bozabilecek eklemeler ve silmeler (INDEL'ler) ortaya çıkar. Genom düzenleme reaktifleri ile birlikte istenen bir tasarlanmış DNA dizisinin (onarım şablonu) birlikte verilmesi durumunda, onarım şablonunun çift sarmallı DNA kırılmasını içeren bölgeye eklenmesi, homolojiye yönelik onarım (HDR) adı verilen bir işlemle gerçekleşir. Bu, hedeflenen DNA eklemeleri/ikameleri (knock-in'ler) ile hayvan modelleri oluşturmak için etkili bir stratejidir. Bir sınırlama, knock-in dizilerinin genellikle büyük boyutlu olmasıdır, bunun gen düzenleme verimliliğini azalttığı ve böylece istenen modelin oluşturulmasını daha zor hale getirdiği gösterilmiştir1. Zincirleme verimliliği artırmaya yönelik stratejiler, hem çift sarmallı DNA (dsDNA) hem de tek sarmallı DNA (ssDNA) onarım şablonlarının doğrusallaştırılmasını ve DNA onarım şablonlarının kimyasal modifikasyonunuiçermektedir 2,3,4. Ek olarak, HDR uyarıcı bileşiklerle birlikte pronükleer mikroenjeksiyon, mikroenjeksiyon ile birlikte elektrik darbelerinin uygulanması ve 2 hücreli embriyolara zamanlanmış mikroenjeksiyon denenmiştir 5,6,7. Bu yaklaşımların bazılarının başarısına rağmen, 1.0 kb'den daha büyük DNA dizilerinin dahil edilmesi teknik olarak zor olmaya devam etmektedir.
Reaktifleri kültürlenmiş hücre hatlarına sokmak için yaygın bir yöntem olan elektroporasyon, CRISPR-Cas9 bileşenlerini embriyolara vermek için mikroenjeksiyona bir alternatif sunar. İlk olarak sıçan embriyolarında8 gösterilen embriyo elektroporasyonu, o zamandan beri farelerde 9,10,11,12,13, domuzlarda14,15 ve diğer hayvan model organizmalarında 16,17,18 bir dağıtım yöntemi olarak başarıyla kullanılmıştır.. CRISPR-Cas9 reaktifleri içeren ortamda süspanse edilen embriyolar, iki elektrot arasında bir küvete veya bir cam slayt üzerine yerleştirilir ve doğrudan elektrik akımı darbelerine maruz bırakılır. Bu, CRISPR-Cas9 bileşenlerinin embriyolara girdiği zona pellucida ve embriyo plazma zarında geçici açıklıklar oluşturur. Tipik olarak, geçici açıklıkları oluşturmak için orta seviyeli elektriksel "gözenek" darbeleri kullanılır, ardından negatif yüklü genom düzenleme bileşenlerinin hareketini kolaylaştıran daha düşük seviyeli elektriksel "transfer darbeleri" gelir. Embriyo elektroporasyonu verimlidir, yüksek verime sahiptir ve gerçekleştirilmesi kolaydır. Bununla birlikte, embriyo elektroporasyonunun küçük (<200 bp) ssDNA onarım şablonlarının tanıtımı için oldukça başarılı olduğu gösterilmiş olsa da, daha büyük (>1.0 kb) onarım şablonlarının başarılı elektroporasyonuna dair çok az rapor vardır13,19. Bu boyut kısıtlaması, büyük yerleştirmeler gerektiren knock-in hayvan modelleri oluşturmak için embriyo elektroporasyonunun önemli bir sınırlamasını temsil eder.
Gen terapisi bağlamında, adeno-ilişkili virüsler (AAV'ler), hem bölünen hem de bölünmeyen hücrelerin in vivo enfektivitesi, patojenite eksikliği ve nadir genomik entegrasyon nedeniyle genetik materyali iletmek için uzun süredir araç olarak kullanılmaktadır20,21. Son zamanlarda, daha fazla çalışma, DNA onarım şablonlarını ve CRISPR reaktiflerini tanıtmak için AAV'leri CRISPR-Cas9 teknolojisiyle birleştirdi 22,23,24. Bu yaklaşım, mikroenjeksiyon tekniklerine ihtiyaç duymadan daha büyük DNA onarım şablonlarının sunulmasına izin verir.
HDR yolu, hücre döngüsünün geç S ve G2 fazlarında daha aktiftir25,26. İn vitro olarak yapılan çalışmalarda, CRISPR-Cas9 RNP'lerinin ve DNA onarım şablonlarının G2 ile senkronize hücrelere verilmesi veya Cas9 proteininin varlığının bir Cas9-Geminin füzyon proteini2 kullanılarak geç S ve G2 fazlarına kısıtlanmasıyla zincirleme verimlilikte önemli artışlar elde edilmiştir. Ayrıca, 2 hücreli aşama embriyosunun genişletilmiş G2 fazı sırasında meydana gelen büyük bir zigotik genom aktivasyonu (ZGA) olayı vardır ve bu, açık bir kromatin durumu ile ilişkilidir. Bunun, CRISPR-Cas9 RNP'lerine ve onarım şablonlarına genomik DNA'ya daha fazla erişilebilirlik sağladığı tahmin edilmektedir.
Amacımız, embriyo gelişiminin 2 hücreli aşamasında CRISPR-Cas9 RNP'leri tanıtmak için AAV yaklaşımını embriyo elektroporasyonu ile birleştirerek tüm bu gözlemleri geliştirmekti. Bu strateji, DNA eklemelerinin hedeflenen genetik mühendisliği için etkili bir yöntem oluşturmak için AAV'nin daha büyük DNA onarım şablonu dağıtım kapasitesinden, elektroporasyonun teknik kolaylığından ve embriyo gelişimi sırasında genomik erişilebilirlik için daha uygun 2 hücreli zaman noktasından yararlanır. Bu protokolde vurgulandığı gibi, optimize edilmiş yöntemimiz, mikroenjeksiyon tekniklerine ihtiyaç duymadan 1.2 ila 3.0 kb boyutunda DNA dizilerinin eklenmesini taşıyan hedefli knock-in sıçan modellerinin üretilmesine izin verir.
Protokol
Tüm deneysel prosedürler Missouri Üniversitesi Kurumsal Hayvan Bakımı ve Kullanımı Komitesi (ACUC protokolü #25580) tarafından onaylandı ve Laboratuvar Hayvanlarının Kullanımı ve Bakımı Kılavuzu'nda belirtilen yönergelere göre gerçekleştirildi.
1. AAV aracılı DNA onarım şablonu teslimi
- DNA yapısını, iki homoloji kolu arasında istenen knock-in dizisini içerecek şekilde tasarlayın. Tipik homoloji kol uzunlukları 300-500 bp uzunluğundadır. Uygun bir plazmit omurgasına klonlanarak ve rekombinant ssAAV1 veya ssAAV6'ya (serotip 1 veya 6) paketlenerek tüm DNA onarım şablonunun (homoloji kolları + istenen knock-in dizisi) ters çevrilmiş terminal tekrarları (ITR'ler) ile çevrili olduğundan emin olun.
NOT: DNA onarım şablonunda sgRNA hedef dizisi mevcutsa, bu diziyi bozmak için sessiz mutasyonlar tasarlamak ve böylece doğru hedeflenmiş alelin CRISPR-Cas9 aracılı düzenlemesini önlemek önemlidir.
DİKKAT: Rekombinant AAV viral vektörleri entegre değildir ve BSL-1 olarak sınıflandırılır. Virüse maruz kalmış embriyoların pipetlenmesi ve işlenmesi için standart aseptik teknikler kullanılır. - 35 mm'lik Petri kabında mineral yağ altında 30 μL KSOM-R ortamına27,28 3 × 107 genom kopyası (GC)/μL'lik bir nihai konsantrasyona kadar tasarlanmış ssAAV1 veya ssAAV6 (DNA onarım şablonuyla paketlenmiş) ekleyin.
- Görünür pronükleuslara sahip zigotları, tasarlanmış ssAAV içeren KSOM-R ortamına yerleştirin.
NOT: Bu protokolü kullanarak, AAV serotip 1 ve 6, verimli DNA onarım şablonu iletimi için kolayca geçebildiğinden, zona pellucida'yı inceltmeye gerek yoktur. - Yeterli viral transdüksiyona izin vermek için% 5 CO2 ve maksimum nem ile 37 ° C'de 18-20 saat inkübe edin.
2. Elektroporasyon hazırlığı
- Ertesi gün, steril bir RNaz içermeyen tüpte Opti-MEM ortamında seyreltilmiş 100 ng/μL sgRNA ve 100 ng/μL Cas9 proteini içeren bir elektroporasyon karışımı yapın. Yavaşça karıştırın.
- RNP oluşumuna izin vermek için oda sıcaklığında 10 dakika inkübe edin.
- İnkübasyon süresi boyunca, elektroporatörü açın ve uçları 1,5 mm boşluklu bir cam kayar elektroda bağlayın.
- Elektroporasyon parametrelerini Tablo 1'de açıklandığı gibi ayarlayın.
3. 2 hücreli embriyo elektroporasyonu
- Çözeltiye hava kabarcıkları girmemesine dikkat ederek, cam slayt üzerindeki iki elektrot arasına 5 μL CRISPR elektroporasyon karışımını nazikçe pipetleyin.
- 2 hücreli aşama embriyosunu AAV çözeltisinden çıkarın. 2 hücreli aşamadaki embriyoları, elektrotların kenarlarına dokunmalarına izin vermeden karışımda sıralayın. Bu lizisi önlemek için yapılır.
- Empedansı ölçmek için Ohm düğmesine basın. Empedansın 0,100 ile 0,300 kΩ arasında olduğundan emin olmak için kontrol edin. Reaksiyon hacmi empedansı değiştirecektir (empedansı azaltmak için hacim ekleyin ve empedansı artırmak için hacmi çıkarın).
- Başlat'a basın. İşlemin tamamlanması birkaç saniye sürer.
- Elektroporasyondan hemen sonra embriyoları çıkarın ve 30 μL'lik taze KSOM-R besiyerinde 3 kez yıkayın.
- Embriyoları, mineral yağ altında 500 μL damla KSOM-R ortamında in vitro kültür için% 5 CO2 ve maksimum nem ile 37 ° C'lik inkübatöre geri yerleştirin. Alternatif olarak, canlı yavrular üretmek için embriyoları cerrahi olarak 1.5 günlük bir koitum sonrası (dpc) yalancı gebe dişi sıçana aktarın.
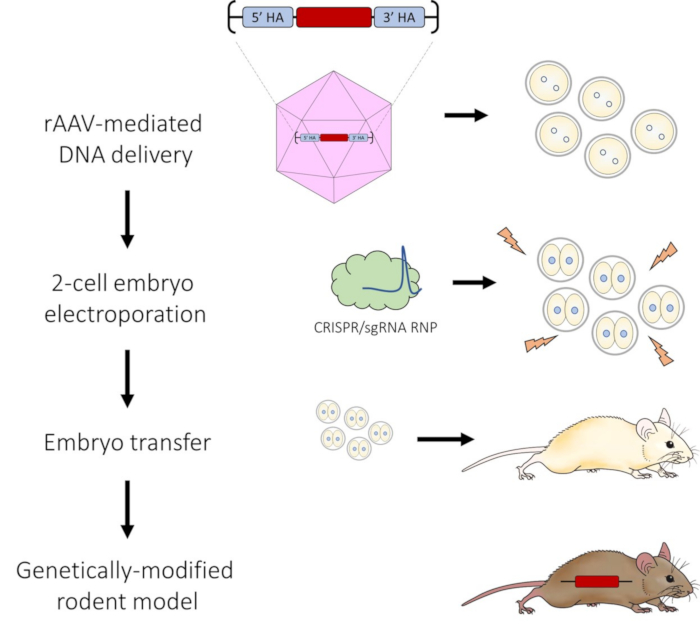
Şekil 1: CRISPR-Cas9 genom düzenlemesi için AAV aracılı DNA iletimi ve 2 hücreli embriyo elektroporasyon boru hattının şeması. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Sonuçlar
Protokolü takiben, 2 hücreli embriyolar CRISPR-Cas9 RNP'ler ile elektroporasyona tabi tutulduktan sonra yüksek verimli HDR'ye izin veren DNA onarım şablonunun etkili AAV aracılı iletimi vardır. Videoda gösterildiği gibi, başarılı bir elektroporasyon işlemi, her elektrotta kabarcıkların oluşmasına (Şekil 2C) ve empedansın 0.100 ile 0.300 kΩ aralığında kalmasına neden olur. Elektroporasyondan sonra, embriyoların zona pellucida içindek...
Tartışmalar
CRISPR-Cas genom düzenleme sistemi, çeşitli hayvan türlerinde hem basit hem de karmaşık, özelleştirilmiş genetik modifikasyonların verimli bir şekilde üretilmesine izin vererek genetik mühendisliği alanında devrim yarattı. Genom düzenleme ile ilgili tekniklerde sık sık yapılan iyileştirmeler ve iyileştirmeler, çok yönlülüğünü daha da artırmaktadır. Burada, CRISPR-Cas9 reaktiflerinin 2 hücreli kemirgen embriyolarına 2 hücreli embriyo elektroporasyonu ile birlikte ssAAV aracılı DNA ileti...
Açıklamalar
Yazarların beyan edecek herhangi bir çıkar çatışması yoktur.
Teşekkürler
Yazarlar, videografi ve video düzenleme konusundaki yardımları için Nolan Davis'e teşekkür eder.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Leads with alligator clips for electrodes | NepaGene | C117 | |
| Mineral oil | MilliporeSigma | M5310 | |
| NepaGene21 Super Electroporator | NepaGene | NEPA21 | |
| Platinum plate electrodes on slide glass | NepaGene | CUY501P1-1.5 | |
| PURedit Cas9 Protein | MilliporeSigma | PECAS9 | |
| sgRNA (chemically-modified) | Synthego | N/A | |
| ssAAV1 or ssAAV6 packaged DNA repair template | Vectorbuilder | N/A |
Referanslar
- Lau, C. H., Tin, C., Suh, Y. CRISPR-based strategies for targeted transgene knock-in and gene correction. Fac Rev. 9, 20 (2020).
- Gutschner, T., Haemmerle, M., Genovese, G., Draetta, G. F., Chin, L. Post-translational Regulation of Cas9 during G1 Enhances Homology-Directed Repair. Cell Rep. 14 (6), 1555-1566 (2016).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35 (2017).
- Miura, H., Quadros, R. M., Gurumurthy, C. B., Ohtsuka, M. Easi-CRISPR for creating knock-in and conditional knockout mouse models using long ssDNA donors. Nat Protoc. 13 (1), 195-215 (2018).
- Gu, B., Posfai, E., Gertsenstein, M., Rossant, J. Efficient generation of large-fragment knock-in mouse models using 2-cell (2C)-homologous recombination (HR)-CRISPR. Curr Protoc Mouse Biol. 10 (1), e67 (2020).
- Gu, B., Posfai, E., Rossant, J. Efficient generation of targeted large insertions by microinjection into two-cell-stage mouse embryos. Nat Biotechnol. 36 (7), 632-637 (2018).
- Kurihara, T., et al. DNA repair protein RAD51 enhances the CRISPR/Cas9-mediated knock-in efficiency in brain neurons. Biochem Biophys Res Commun. 524 (3), 621-628 (2020).
- Kaneko, T., Sakuma, T., Yamamoto, T., Mashimo, T. Simple knockout by electroporation of engineered endonucleases into intact rat embryos. Sci Rep. 4, 6382 (2014).
- Chen, S., Lee, B., Lee, A. Y., Modzelewski, A. J., He, L. Highly efficient mouse genome editing by CRISPR ribonucleoprotein electroporation of zygotes. J Biol Chem. 291 (28), 14457-14467 (2016).
- Hashimoto, M., Yamashita, Y., Takemoto, T. Electroporation of Cas9 protein/sgRNA into early pronuclear zygotes generates non-mosaic mutants in the mouse. Dev Biol. 418 (1), 1-9 (2016).
- Wang, W., et al. Delivery of Cas9 protein into mouse zygotes through a series of electroporation dramatically increases the efficiency of model creation. J Genet Genomics. 43 (5), 319-327 (2016).
- Troder, S. E., et al. An optimized electroporation approach for efficient CRISPR/Cas9 genome editing in murine zygotes. PLoS One. 13 (5), e0196891 (2018).
- Teixeira, M., et al. Electroporation of mice zygotes with dual guide RNA/Cas9 complexes for simple and efficient cloning-free genome editing. Sci Rep. 8 (1), 474 (2018).
- Tanihara, F., et al. Efficient generation of GGTA1-deficient pigs by electroporation of the CRISPR/Cas9 system into in vitro-fertilized zygotes. BMC Biotechnol. 20 (1), 40 (2020).
- Tanihara, F., et al. Generation of CD163-edited pig via electroporation of the CRISPR/Cas9 system into porcine in vitro-fertilized zygotes. Anim Biotechnol. 32 (2), 147-154 (2021).
- Camargo, L. S. A., Owen, J. R., Van Eenennaam, A. L., Ross, P. J. Efficient one-step knockout by electroporation of ribonucleoproteins into zona-intact bovine embryos. Front Genet. 11, 570069 (2020).
- Lin, J. C., Van Eenennaam, A. L. Electroporation-mediated genome editing of livestock zygotes. Front Genet. 12, 648482 (2021).
- Betters, E., Charney, R. M., Garcia-Castro, M. I. Electroporation and in vitro culture of early rabbit embryos. Data Brief. 21, 316-320 (2018).
- Miyasaka, Y., et al. CLICK: one-step generation of conditional knockout mice. BMC Genomics. 19 (1), 318 (2018).
- Epstein, B. E., Schaffer, D. V. Combining engineered nucleases with adeno-associated viral vectors for therapeutic gene editing. Adv Exp Med Biol. 1016, 29-42 (2017).
- Gaj, T., Epstein, B. E., Schaffer, D. V. Genome engineering using adeno-associated virus: basic and clinical research applications. Mol Ther. 24 (3), 458-464 (2016).
- Chen, S., et al. CRISPR-READI: Efficient generation of knockin mice by CRISPR RNP electroporation and AAV donor infection. Cell Rep. 27 (13), 3780-3789 (2019).
- Mizuno, N., et al. Intra-embryo gene cassette knockin by CRISPR/CAS9-mediated genome editing with adeno-associated viral vector. iScience. 9, 286-297 (2018).
- Davis, D. J., et al. Efficient DNA knock-in using AAV-mediated delivery with 2-cell embryo CRISPR-Cas9 electroporation. Front Genome Ed. 5, 1256451 (2023).
- Smirnikhina, S. A., Zaynitdinova, M. I., Sergeeva, V. A., Lavrov, A. V. Improving homology-directed repair in genome editing experiments by influencing the cell cycle. Int J Mol Sci. 23 (11), (2022).
- Takata, M., et al. Homologous recombination and non-homologous end-joining pathways of DNA double-strand break repair have overlapping roles in the maintenance of chromosomal integrity in vertebrate cells. EMBO J. 17 (18), 5497-5508 (1998).
- Men, H., Stone, B. J., Bryda, E. C. Media optimization to promote rat embryonic development to the blastocyst stage in vitro. Theriogenology. 151, 81-85 (2020).
- Men, H., Amos-Landgraf, J. M., Bryda, E. C., Franklin, C. L. KSOM-R supports both mouse and rat preimplantation embryo development in vitro. Theriogenology. 198, 69-74 (2023).
- Romeo, C., et al. AAV diffuses across zona pellucida for effortless gene delivery to fertilized eggs. Biochem Biophys Res Commun. 526 (1), 85-90 (2020).
- Fulka, J., Moor, R. M., Fulka, J. Electrofusion of mammalian oocytes and embryonic cells. Methods Mol Biol. 48, 309-316 (1995).
- Rems, L., et al. Cell electrofusion using nanosecond electric pulses. Sci Rep. 3, 3382 (2013).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır