Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Drosophila Larva Beyin ve Hücre Hattından Poli A Kuyruk Uzunluğunun Ölçülmesi
Bu Makalede
Özet
Protokol, diğer türlerden dokulara veya hücre tiplerine kolayca uyarlanabilen, Drosophila sinir sisteminden ilgilenilen genin poli(A) uzunluğunu ölçmek için verimli ve güvenilir bir yöntemi açıklar.
Özet
Poliadenilasyon, mRNA moleküllerinin 3' ucuna poli(A) kuyrukları ekleyen çok önemli bir transkripsiyon sonrası modifikasyondur. Poli(A) kuyruğunun uzunluğu, hücresel süreçler tarafından sıkı bir şekilde düzenlenir. mRNA poliadenilasyonunun düzensizliği, anormal gen ekspresyonu ve kanser, nörolojik bozukluklar ve gelişimsel anormallikler dahil olmak üzere çeşitli hastalıklarla ilişkilendirilmiştir. Bu nedenle, poliadenilasyonun dinamiklerini anlamak, mRNA işleme ve transkripsiyon sonrası gen regülasyonunun karmaşıklıklarını çözmek için hayati önem taşır.
Bu makale, Drosophila larva beyinlerinden ve Drosophila Schneider S2 hücrelerinden izole edilen RNA örneklerinde poli(A) kuyruk uzunluklarını ölçmek için bir yöntem sunmaktadır. Maya poli(A) polimeraz kullanılarak mRNA'nın 3' ucuna G/I kalıntılarının enzimatik eklenmesini içeren guanozin/inosin (G/I) kuyruk yaklaşımını kullandık. Bu modifikasyon, RNA'nın 3' ucunu enzimatik bozulmadan korur. Korunan tam uzunluktaki poli(A) kuyruklar daha sonra evrensel bir antisens astar kullanılarak ters kopyalanır. Daha sonra, PCR amplifikasyonu, ters transkripsiyon için kullanılan evrensel bir dizi oligo ile birlikte ilgilenilen geni hedefleyen gene özgü bir oligo kullanılarak gerçekleştirilir.
Bu, ilgilenilen genin poli(A) kuyruklarını kapsayan PCR ürünleri üretir. Poliadenilasyon tek tip bir modifikasyon olmadığından ve farklı uzunluklarda kuyruklarla sonuçlandığından, PCR ürünleri bir dizi boyut gösterir ve bu da agaroz jel üzerinde bir leke desenine yol açar. Son olarak, PCR ürünleri yüksek çözünürlüklü kapiler jel elektroforezine tabi tutulur, ardından poli(A) PCR ürünlerinin ve gene özgü PCR ürününün boyutları kullanılarak miktar tayini yapılır. Bu teknik, poli(A) kuyruk uzunluklarını analiz etmek için basit ve güvenilir bir araç sunarak, mRNA regülasyonunu yöneten karmaşık mekanizmalar hakkında daha derin bilgiler edinmemizi sağlar.
Giriş
Çoğu ökaryotik mRNA, kanonik poli(A) polimerazlar tarafından şablonlanmamış adenosinlerin eklenmesiyle çekirdekteki 3' terminallerinde transkripsiyon sonrası poliadenillenir. Bozulmamış bir poli(A) kuyruğu, mRNA'nın yaşam döngüsü boyunca çok önemlidir, çünkü mRNA nükleer ihracatı1 için gereklidir, translasyon verimliliğiniartırmak için poli(A) bağlayıcı proteinlerle etkileşimi kolaylaştırır 2 ve bozunmayakarşı direnç kazandırır 3. Bazı durumlarda, poli(A) kuyruğu, kanonik olmayan poli(A) polimerazlar4 tarafından kolaylaştırılan sitoplazmada da uzamaya uğrayabilir. Sitoplazmada, poli (A) kuyruk uzunluğu dinamik olarak değişir ve mRNA molekülünün ömrünü etkiler. Kuyruk uzunluğunu 5,6,7 modüle etmek için çok sayıda polimeraz ve deadenilaz bilinmektedir. Örneğin, poli(A) kuyruklarının kısalması translasyonel baskılama ile ilişkilidir, oysa poli(A) kuyruklarının uzatılması translasyonu artırır 8,9.
Biriken genomik çalışmalar, ökaryotik biyolojinin çeşitli yönlerinde poli(A) kuyruk uzunluğunun temel önemini göstermiştir. Bu, germ hücresi gelişimi, erken embriyonik gelişim, öğrenme ve hafıza için nöronal sinaptik plastisite ve inflamatuar yanıttaki rolleri içerir10. Poli(A) kuyruk uzunluklarını ölçmek için geliştirilmiş çok sayıda yöntem ve tahlil vardır. Örneğin, RNase H/oligo(dT) testi, poli(A) kuyruk uzunluğu 11,12'yi incelemek için oligo(dT) varlığında veya yokluğunda RNase H'den yararlanır. Poli(A) kuyruğunu incelemek için diğer yöntemler arasında, cDNA uçlarının hızlı amplifikasyonu poli(A) testi (RACE-PAT)12,13 ve ligaz aracılı poli(A) testi (LM-PAT)14 gibi 3' uçların PCR amplifikasyonu yer alır. PAT tahlilinin diğer modifikasyonları arasında ePAT15 ve sPAT16 bulunur. Enzimatik G-tailing17,18 veya 3' ucunun G/I-tailing'i, PAT testinin diğer varyasyonlarıdır. Bu tekniklerin daha fazla modifikasyonu, yüksek çözünürlüklü poli(A) testi (Hire-PAT)19 olarak adlandırılan yüksek çözünürlüklü analiz için kılcal jel elektroforezi ile birlikte floresan etiketli primerlerin kullanımını içerir. Bu PCR güdümlü tahliller, hızlı ve yüksek hassasiyetli poli(A) uzunluk kantitasyonuna izin verir.
Yeni nesil dizilemenin geliştirilmesiyle birlikte, PAL-seq20 ve TAIL-seq21 gibi yüksek verimli bir dizileme yöntemi, transkriptom çapında bir ölçekte poliadenilasyon analizlerine izin verir. Bununla birlikte, bu yöntemler yalnızca 36-51 nükleotidin kısa dizileme okumalarını sağlar. Bu nedenle, FLAM-Seq22 , tam uzunlukta mRNA'nın küresel kuyruk uzunluğu profillemesi için geliştirilmiştir ve uzun okumalar sağlar. Nanopor teknolojisi23 , poli(A) kuyruk uzunluğu tahminleri için PCR'den bağımsız, doğrudan RNA veya doğrudan cDNA dizilimi sağlar. Ancak, bu yüksek verimli yöntemler sınırsız değildir. Büyük miktarlarda başlangıç malzemesi gerektirirler, pahalıdırlar ve zaman alıcıdırlar. Ayrıca, nadir transkriptleri analiz etmek, yüksek verimli yöntemlerle son derece zor olabilir ve düşük verimli PCR tabanlı yöntemler, pilot deneyler ve diğer yöntemlerin doğrulanması için az sayıda transkriptin analiz edilmesi gerektiğinde hala bir avantaj sağlar.
Yakın zamanda, Dscam1 mRNA'larının Drosophila'da kısa poli(A) kuyrukları içerdiğini gösterdik, bu da sitoplazmik poli(A)-bağlayıcı proteinin Dscam1 3'UTR üzerinde G/I kuyruk yöntemi24 kullanılarak kanonik olmayan bir şekilde bağlanmasını gerektirir. Burada, Drosophila sinir sistemi ve Drosophila S2 hücrelerinden mRNA'ların poli(A) uzunluğunun doku hazırlığı ve ölçülmesi için kolaylaştırılmış bir prosedür sunuyoruz.
Access restricted. Please log in or start a trial to view this content.
Protokol
1. Drosophila larvalarının yetiştirilmesi ve seçilmesi
- Nemlendirilmiş bir inkübatörde 25 ° C'de standart sinek yemi ortamında sinek suşunu (w1118, vahşi tip) koruyun / kültürleyin.
- Yumurtlamadan 72 saat sonra 10 dolaşan 3. instar larvasını seçin.
- Larvaları 35 mm'lik boş bir Petri kabına koyun ve larvaları forseps kullanarak musluk suyu içeren yeni kaba aktararak nazikçe yıkayın. Kalan yiyecekleri çıkarmak için bunu 2 kez yapın.
2. Drosophila larvalarından beyin izolasyonu (Şekil 1)
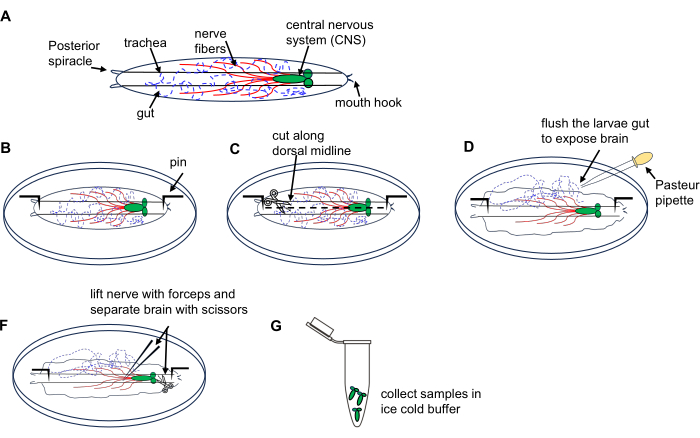
Şekil 1: Drosophila larva beyninin 3. instar gezinme evresinden diseksiyonu. (A) Drosophila larvalarının şematik çizimleri. (B-G) Larva diseksiyonu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- Buz gibi soğuk PBS içeren bir diseksiyon kabına 10 larva yerleştirin.
- Larva dorsal tarafını yukarı yerleştirin (uzunluğu boyunca uzanan trakeal tüplerle tanımlanır) ve her bir ucunu tabağın dibine sabitleyin, ardından arka uçta vücut duvarında küçük bir kesi yapın.
- Mikrodiseksiyon makası kullanarak vücut duvarını dorsal orta hat boyunca ön uca doğru kesin.
- Beyni açığa çıkarmak için tabakta PBS'li bir Pasteur pipeti 3x kullanarak larvaların içini kısaca yıkayın.
- Forseps kullanarak beyni bulun ve kaldırın ve mikrodiseksiyon makası kullanarak dikkatlice izole edin.
- Diseke beyinleri buz üzerinde buz gibi soğuk PBS ile doldurulmuş 1.5 mL'lik bir mikrosantrifüj tüpüne aktarın; Tüm larva beyinlerini toplayın. Bölüm 4'te açıklandığı gibi bir RNA mikro hazırlık kiti kullanarak RNA ekstraksiyonuna devam edin.
NOT: Doku hasarını ve RNA bozulmasını önlemek için 15 dakika içinde 10 larva diseksiyonu yapın.
3. Drosophila S2 Schneider hücreleri
- Drosophila Schneider'in ortamındaki Drosophila S2 hücrelerini, nemlendirilmiş bir inkübatörde 25 ° C'de% 10 fetal sığır serumu (FBS) ile desteklenmiş olarak, minimum% 90 canlılık ile 8 × 106 ila 10 ×10 6 hücre / mL yoğunluğa kadar büyütün.
- 50 mL'lik steril konik bir tüpte, 25 ° C'ye önceden ısıtılmış %10 FBS ile desteklenmiş Schneider'in Drosophila ortamı ile hücreleri 2,5 × 106 hücre / mL'ye seyreltin.
- 8 mL hücre süspansiyonunu (20 × 106 hücre) 100 mm'lik bir kültür plakasına aktarın ve 12 mL'ye (1. gün) getirmek için 4 mL ortam ekleyin.
- Kültürlenmiş hücreleri nemlendirilmiş bir inkübatörde 25 ° C'de inkübe edin.
NOT: Hücreler 12-16 saat sonra (2. gün) plakaya gevşek bir şekilde yapışır. - Hücreleri uygun DNA plazmitleri ile transfekte edin24.
- Nemlendirilmiş bir inkübatörde 48 saat inkübe edin.
- İnkübasyondan sonra, hafif pipetleme ile 5 mL buz gibi PBS ekleyerek hücreleri toplayın (4. gün).
- Hücreleri 15 mL'lik bir tüpe aktarın.
- 4 ° C'de 5 dakika boyunca 1.000 × g'da santrifüj ederek hücreleri peletleyin.
- Hücreleri buz gibi PBS ile nazikçe pipetleyerek 2 kez durulayın ve hücreleri 4 °C'de 5 dakika boyunca 1.000 × g'da santrifüjleyerek toplayın.
- Bir RNA miniprep kiti kullanarak RNA ekstraksiyonu gerçekleştirin.
NOT: Steril bir laminer akış başlığı içinde aşağıdaki adımları gerçekleştirin.
4. Drosophila larvaları beyninden ve S2 hücrelerinden toplam RNA ekstraksiyonu
- Larva beyni: PBS'yi kısa santrifüjleme ile çıkarın (5.000 × g'da 8 sn kısa sıkma).
- 600 μL RNA lizis tamponu ekleyin ve plastik bir tokmak ile 10x homojenize edin. Tam lizizi sağlamak için tüpü stereo mikroskop altında görsel olarak inceleyin.
- Doku kalıntılarını gidermek için 1.000 × g'da 4 °C'de 5 dakika santrifüjleyin. Temizlenmiş süpernatanı nükleaz içermeyen bir mikrosantrifüj tüpüne aktarın.
- Üreticinin talimatlarına göre bir RNA mikro hazırlık kiti kullanarak RNA'yı izole edin.
NOT: Örneklerde az miktarda RNA bulunması nedeniyle larva beyin örnekleri için bir RNA mikro hazırlık kitinin kullanılması önemlidir. - S2 hücreleri: PBS'yi çıkarın ve RNA'yı üreticinin talimatlarına göre izole edin.
- Spektrofotometri ve agaroz jel elektroforezi ile RNA verimini ve kalitesini ölçün.
- Ekstrakte edilen RNA'nın optik yoğunluğunu sırasıyla A260 nm ve A280 nm'de ölçerek ekstrakte edilen RNA'nın saflığını ve miktarını belirleyin. Aşağı akış uygulamaları için A260 nm/A280 nm oranının ≥2.0 ve RNA konsantrasyonunun >350 ng/μL olduğundan emin olun.
NOT: 10 Drosophila larva beyninden tipik bir RNA verimi ~500-800 ng/μL veya 5 μL'de 2.5-4 μg'dir. S2 hücreleri için verim ~2-3 μg/μL'dir (15 μL'de 15-30 μg). İzole RNA, uzun süreli depolama için -80 °C'de saklanabilir.
- Ekstrakte edilen RNA'nın optik yoğunluğunu sırasıyla A260 nm ve A280 nm'de ölçerek ekstrakte edilen RNA'nın saflığını ve miktarını belirleyin. Aşağı akış uygulamaları için A260 nm/A280 nm oranının ≥2.0 ve RNA konsantrasyonunun >350 ng/μL olduğundan emin olun.
5. RNA jelinin hazırlanması ve elektroforez
- % 1.5 Denatüre RNA jeli (100 mL)
NOT: Formaldehit, ciltle teması ve buharların solunması yoluyla toksiktir; Kimyasal bir davlumbazda tutun.- Üç agaroz tableti (1.5 g) 82 mL MOPS tamponunda (Ek Dosya 1) tabletler ince parçacıklar oluşturmak üzere tamamen parçalanana kadar çözün.
- Agaroz bulamacını, çözelti berraklaşana ve tüm parçacıklar tamamen çözülene kadar bir mikrodalgada ısıtın.
- Çözeltiyi ~60 °C'ye soğutun.
- 18 mL %37 formaldehit ekleyin, ardından hafifçe döndürerek karıştırın. Çözeltiyi döküm tepsisine dökün ve çeker ocakta katılaşmasına izin verin.
- RNA numunesi hazırlama ve elektroforez
- RNA örneğini 200 ng'ye (5 μL'de) seyreltin ve 5 μL 2x RNA yükleme boyası ekleyin.
- Numuneleri kuru bir banyoda 5 dakika boyunca 70 °C'de ısıtın.
- İlk şeride 2 μL RNA merdiveni ve bitişik şeride 10 μL numune yükleyin.
- 5 x 6 cm'lik bir jel için 60 dakika boyunca 100 V'ta MOPS tamponunda elektroforez gerçekleştirin.
NOT: Amplikon boyutlarına bağlı olarak elektroforez koşullarını ayarlayın. - Jeli bir UV aydınlatıcı üzerinde görselleştirin.
NOT: Tek bir bant ~600 nükleotid boyutunun varlığı, bozulmamış RNA preparatını gösterir (bkz. Şekil 2A).
6. Poli (A) kuyruk uzunluğu ölçümü
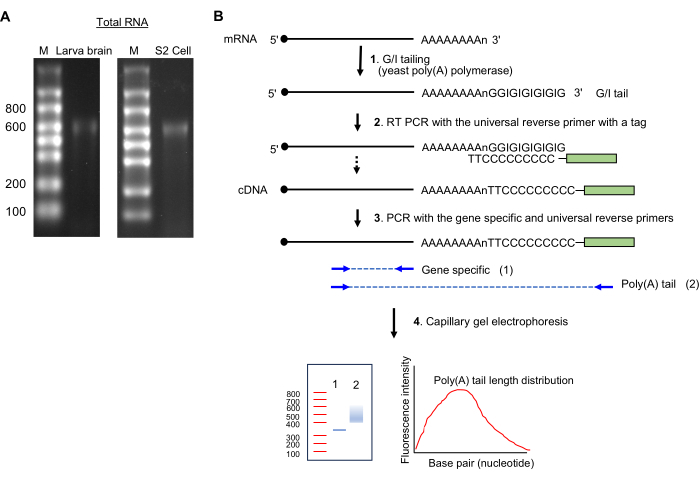
Şekil 2: RNA numunesi hazırlama ve poli(A)-kuyruk testi. (A) RNA jel görüntüleri, %1.5'lik bir formaldehit agaroz jeli üzerinde Drosophila larva beyninden (solda) ve S2 hücrelerinden (sağda) toplam RNA'yı gösterir. Tek sarmallı RNA merdiven boyutları, M şeridindeki nükleotidlerde gösterilir. rRNA'dan gelen ~600 nt'de büyük bir RNA bantlamasına dikkat edin. (B) Poli(A)-kuyruk tahlilinin şemaları. Kısaltma: G/I = guanozin/inosin. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- GI kuyruğu (Şekil 2B)
- Reaktifleri buz üzerinde tutarak aşağıdaki karışımı (20 μL) hazırlayın: 14 μL'ye kadar toplam RNA örneği (1 μg), 4 μL 5x Kuyruk tampon karışımı ve 2 μL 10x Kuyruk enzim karışımı.
- Bir termocycler'da 37 °C'de 60 dakika inkübe edin.
- 1.5 μL kuyruk durdurma çözeltisi ekleyin; 2 dakika buz üzerinde tutun.
NOT: Ters transkripsiyona devam edin veya GI kuyruklu RNA örneklerini ters transkripsiyona geçmeye hazır olana kadar -80 °C'de saklayın.
- Ters transkripsiyon ve PCR amplifikasyonu
- Karışımı hazırlayarak ve Ek Dosya 1'de açıklanan koşullar altında inkübe ederek cDNA'yı sentezleyin.
- cDNA örneklerini seyreltin ve Ek Dosya 1'de belirtildiği gibi DNA'yı çoğaltmak için PCR gerçekleştirin.
7. Agaroz jel elektroforezi ile PCR ürün analizi
- Kalite kontrol için 45 dakika boyunca 100 V'ta elektroforez ile %2.5'lik bir agaroz jel üzerinde adım 6.2.2'deki PCR ürünlerinin küçük bir kısmını (2-5 μL) analiz edin.
- Jel ekstrakte edilen PCR bantlarını sıralayarak gene özgü ve kuyruğa özgü reaksiyonlar için PCR'nin özgüllüğünü doğrulayın.
8. Kılcal elektroforez
- Yüksek hassasiyetli bir DNA kitine sahip biyoanalizörü kullanarak gen spesifik ve poli(A) spesifik PCR'den 1 μL PCR ürünleri (0,5-5 ng/μL) üzerinde yüksek çözünürlüklü jel elektroforezi gerçekleştirin. Başarılı bir koşuyu gösteren iyi çözülmüş zirveleri arayın.
9. Veri analizi: poli(A) kuyruk uzunluğu ölçümü (Şekil 3)
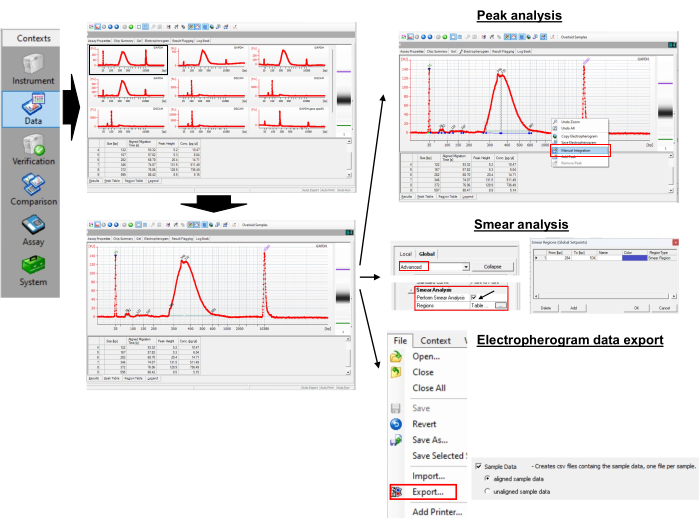
Şekil 3: Poli(A) kuyruk uzunluğu ve tepe değeri ölçümü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- Verilere erişme
- Verilere erişmek için yazılımda xad dosyasını açın.
- Ağaç görünümü panelinde örnek adını veya merdiveni seçin.
NOT: Bu, seçilen numuneler için sonucu elektroferogramlar veya jel benzeri görüntüler olarak gösterecektir. Alt işaretleyici 35 baz çifti (bp) ve üst işaretleyici 10.380 bp, merdiven verilerini (50-7.000 bp) numune kuyularından gelen verilerle hizalamak için kullanılan dahili standartlardır. - Ayrıntıları görüntülemek için elektroferogramları ve jel benzeri görüntüleri yakınlaştırın ve uzaklaştırın.
- Tepe ve smear analizinin yapılması
- En yüksek boyutları elde etmek için, seçilen bir numunenin elektroferogramını açın.
- Elektroferograma sağ tıklayın ve yatay çizgiyi sürükleyerek tepe noktalarını manuel olarak seçmek için manuel entegrasyonu seçin.
- Tepe tablosundaki tepe değerlerine dikkat edin. En büyük Tepe Yüksekliğine sahip zirveyi belirleyin. Bu, tek bir numune için poli(A) kuyruk uzunluğunun bir tepe noktasıdır. Gösterilen örnekte 346 bp'dir.
- Üst menüdeki Ayar Noktalarını Göster/Gizle simgesini etkinleştirin ve sağ tarafta yeni bir panelin açılmasını bekleyin.
- Gelişmiş'i seçin, Smear Analizi Gerçekleştir'i bulmak için aşağı kaydırın ve onay kutusunu seçin. Bu, Bölge tablosunu elektroferogram sekmesine ekleyecektir.
- Bir örnekten bir elektroferogram seçin ve Kimden [bp] ve Kime [bp] menüsünü gösteren Bölge tablosuna gidin. Başlangıç ve bitişi [bp] ayarlamak için elektroferograma sağ tıklayın ve Bölge Ekle'yi eklemek için Bölge'yi seçin.
- Bölge tablosundaki herhangi bir hücreye sağ tıklayın ve özel bölgelerin ayarlanabileceği küçük yeni bir pencere açmak için Bölgeleri Değiştir'i seçin.
NOT: Örneğin, GAPDH için 300 bp ile 550 bp arasında bir bölge kullandık. Gene özgü GAPDH PCR, 265 bp'de bir pik verdi. Üniversal primer (Tablo 1), G/I-kuyruklu RNA'lara tavlama yoluyla poli(A) PCR'nin uzunluğunu 35 bp uzatır. Böylece, GAPDH RNA'sındaki ilk adenin nükleotidi 300 bp'de (265 + 35) başlar. Maksimum poli(A) kuyruk uzunluğunu keyfi olarak 250 (300 + 250 = 550) ile sınırladık. Bölge tablosundan, program bölge içindeki ortalama boyutu 387 bp olarak döndürür. - İlgilenilen mRNA'daki poli(A) kuyruk uzunluğunu hesaplamak için denklem (1)'i kullanın:
Poli(A) kuyruk uzunluğu = (A - B - 35) (1)
A, elektroferogramdan elde edilen poli(A)-spesifik PCR ürününün ortalama bp'sidir (yani, GAPDH için 387 bp), B, elektroferogramdan elde edilen gene özgü PCR ürününün pik bp'sidir (yani, GAPDH için 265 bp) ve "35", evrensel ters primer etiketinin uzunluğudur.
NOT: Yukarıdaki hesaplamadan, GAPDH'nin ortalama poli(A) kuyruk uzunluğu 387 - 265 - 35= 87 bp'dir.
10. Poli(A) kuyruk uzunluğu dağılımının görselleştirilmesi
- Verileri Dosya | İhracat | Örnek verileri almak için Örnek Veriler.
NOT: Dışa aktarılan csv dosyası, X ekseninde bp yerine çalışma zamanını görüntüler. - Bir numunenin elektroferogramına gidin ve Bölge Tablosundaki bölge tablosunu otomatik olarak bp'den çalışma zamanına dönüştürmek için Elektroferogram sekmesindeki Boyutları Göster'i işaretleyin. Örnekte, 70,39 sn ila 86,28 sn, 300 bp ila 550 bp'ye karşılık gelir.
- csv dosyasını açın ve bir grafik oluşturmak için 70,39 sn ile 86,28 sn arasındaki çalışma süresi değerlerini seçin. bp boyutlarını grafikteki X eksenine göre görselleştirmek için, bp boyutlarına sahip elektroferogramı bir görüntü dosyası olarak dışa aktarın ve elektronik tabloda oluşturulan grafiğin üzerine yerleştirin. Bu, poli(A) kuyruk dağılımındaki bp boyutlarıyla uygun şekilde eşleşecektir.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Burada, Drosophila larva beyinlerinden Dscam1 ve GAPDH'nin poli(A) kuyruk uzunluğunu analiz ettik (Şekil 4). İzole edilen RNA'lar, kalite kontrol için bir agaroz jel üzerinde görselleştirildi. Yaklaşık 600 nükleotid boyutunda tek bir RNA bandı, bozulmamış RNA preparatını gösterir (Şekil 2A). RNA'lar, bir Agilent 2100 biyoanalizörü kullanılarak G/I kuyruğuna ve yüksek çözünürlüklü kapiler elektroforeze tabi t...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Bu protokolde, Drosophila larva beynini dolaşan 3. instar aşamasından diseksiyon tekniğini ve Drosophila S2 hücrelerinden örnek hazırlamayı açıklıyoruz. mRNA'ların kararsız doğası nedeniyle, numune toplama ekstra dikkat gerektirir. Larva beyin diseksiyonu için izolasyon sırasında beyinler zarar görmemeli ve uzun süre solüsyon içinde tutulmamalıdır. Bir diseksiyon turu için diseksiyon süresini 8-10 dakika olarak tutmak esastır. Diseksiyon solüsyonunun RNAaz inhibit?...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarların ifşa edecek herhangi bir çıkar çatışması yoktur.
Teşekkürler
Bu çalışma, Ulusal Nörolojik Bozukluklar ve İnme Enstitüsü tarafından JK'ye R01NS116463 ve Ulusal Sağlık Enstitüleri Hibe P20GM103650 tarafından desteklenen ve bu çalışmada bildirilen araştırmalar için kullanılan Nevada Üniversitesi, Reno'daki Hücresel ve Moleküler Görüntüleme Çekirdeği tesisi tarafından desteklenmiştir.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
Referanslar
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435(2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622(2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57(2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294(2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır