Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Pluripotent Kök Hücre Farklılaşmasını İzlemek için Canlı Hücre Görüntü Tabanlı Makine Öğrenimi Stratejisi
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Mevcut pluripotent kök hücre (PSC)-fonksiyonel hücre farklılaşma sistemleri şu anda ciddi hattan hatta ve partiden partiye değişkenlik sorunları tarafından engellenmektedir. Burada, kardiyak farklılaşmayı ana örnek olarak kullanarak, görüntü tabanlı makine öğrenimine dayalı PSC farklılaşma sürecini akıllıca izlemek ve modüle etmek için bir protokol sunuyoruz.
Özet
Pluripotent kök hücre (PSC) teknolojileri, ilaç keşfi, hastalık modellemesi ve rejeneratif tıpta yaygın olarak kullanılmaktadır. Bununla birlikte, mevcut PSC'den fonksiyona hücre farklılaştırma sistemleri, ciddi hattan hatta ve partiden partiye değişkenlik sorunları tarafından engellenmektedir. Bu nedenle hücre farklılaşmasının gerçek zamanlı olarak hassas kontrolü önemlidir. Bu protokolde, parlak alan görüntü tabanlı makine öğrenimi kullanarak hücre farklılaşmasındaki değişkenliğin üstesinden gelen, invaziv olmayan ve akıllı bir strateji açıklıyoruz. PSC'den kardiyomiyosite farklılaşmayı örnek olarak alan bu metodoloji, ilk PSC durumunun kontrolü, farklılaşma koşullarına erken değerlendirme ve müdahale ve yanlış farklılaşmış hücre kontaminasyonunun ortadan kaldırılması için ayrıntılı bilgi sağlar ve birlikte PSC'lerden fonksiyonel hücrelere sürekli olarak yüksek kaliteli farklılaşmayı gerçekleştirir. Prensip olarak, bu strateji, hücre üretimini desteklemek ve hücre kaderi dönüşümü sırasındaki mekanizmaları daha iyi anlamamızı sağlamak için çok adımlı diğer hücre farklılaşması veya yeniden programlama sistemlerine genişletilebilir.
Giriş
Pluripotent kök hücreler (PSC'ler), in vitro olarak birçok hücre tipine farklılaşma konusunda olağanüstü bir yeteneğe sahiptir. Bu farklılaşmış fonksiyonel hücreler, tümü araştırma veya klinik uygulamalar için değerli olan hücre tedavisi, hastalık modellemesi ve ilaç geliştirme için kullanılabilir 1,2,3. Örneğin, PSC'leri kardiyomiyositlere (CM'ler) ayırmak için çeşitli yöntemler geliştirilmiştir4,5,6,7. Bu CM'ler, ilaçların kardiyotoksisite testi, kalp hastalığının modellenmesi ve hücre nakliiçin uygulanabilir 8,9,10,11.
Bununla birlikte, PSC'den terminal farklılaşmış hücrelere dönüşüm aşamalı bir süreçtir ve farklılaşma işlemi sırasında çoklu bozulmalar hücreleri farklı hücre kaderlerine götürebilir. PSC hatlarının farklı genetik geçmişleri ve epigenetik işaretleri, belirli bir soya farklılaşma potansiyelini etkiler 12,13,14,15; PSC pasajlarının sayısı ve birikmiş gen mutasyonları da PSC heterojenliğinin kaynaklarıdır; Farklı deneyciler tarafından kullanılan deneysel işlemlerdeki farklılıklar da tamamen farklı farklılaşma sonuçlarına yol açabilir 16,17,18,19,20. Bu nedenle, şu anda PSC'den türetilmiş hücre üretimindeki ana sorunlardan biri, hücre hatları ve partileri arasındaki kararsızlıktır 21,22,23,24,25. PSC farklılaşmasındaki kararsızlık genellikle önemli ölçüde zaman ve emek kaynakları tüketen çok sayıda tekrarlanan deneye yol açar. Bu sorunu ele almak için, hücre hatları ve partileri arasındaki değişkenliği en aza indiren ve böylece farklılaşmanın kararlılığını ve sağlamlığını artıran bir strateji geliştirmek çok önemlidir.
Son zamanlarda, yüksek çözünürlüklü mikroskopi ve makine öğrenimindeki (ML) gelişmeler, hücre biyolojisinde makine öğrenimi tabanlı kantitatif görüntü analizinin uygulanmasını kolaylaştırarak, hücre görüntüleme özelliklerinde değerli bilgilerin kullanılmasını mümkün kılmıştır 26,27,28,29,30,31,32,33,34. Önceki çalışmamızda, PSC farklılaşmasının kararlılığını ve verimliliğini artırmak için PSC farklılaşma durumunu gerçek zamanlı olarak izlemek ve müdahale etmek için canlı hücre görüntü tabanlı bir makine öğrenimi stratejisi önerdik (Şekil 1)35. PSC'den kardiyomiyosit farklılaşmasını örnek alarak, rastgele orman modelleri kullanarak ilk PSC durumunu değerlendirdik, lojistik regresyon modellerini kullanarak optimal farklılaşma durumunu tahmin ettik ve derin öğrenme tabanlı Grad-CAM36 ve pix2pix37 kullanarak başarıyla farklılaşmış hücreleri tanıdık. Makine öğrenimi modelleri, alan, çevre, dışbükeylik, katılık, parlaklık, hareket hızı ve derin evrişimli sinir ağları tarafından çıkarılan diğer örtük özellikler dahil olmak üzere bir dizi parlak alan morfolojik özelliğinden hücre soylarını tanımlamayı öğrendi. Bu yerleşik makine öğrenimi modellerinden elde edilen çıkarımlara dayanarak, ilk PSC durumunun kontrolünü, farklılaşma koşullarında erken değerlendirme ve müdahaleyi ve yanlış farklılaşmış hücre kontaminasyonunun ortadan kaldırılmasını gerçekleştirdik ve birlikte kardiyak farklılaşma sürecinin kapsamlı ve doğru bir modülasyonunu sağladık. Burada stratejiyi geliştirmek için adım adım bir protokol sunuyoruz.
Protokol
1. Hücre farklılaşması ve karakterizasyonu
- Kültür reaktiflerinin ve kültür plakalarının hazırlanması
- 48 mL bazal ortama 2 mL takviye ve% 0.2 Penisilin-Streptomisin ekleyerek PSC kültür ortamını hazırlayın. Eki alın ve -20 °C'de saklayın. Bu ortamı 4 °C'de 4 haftaya kadar saklayın.
- 500 mL bazal ortama 1 mL takviye ve% 0.2 Penisilin-Streptomisin ekleyerek PSC hazırlama ortamını hazırlayın. Kullanırken, ortamı bir kerelik kullanım için önceden ısıtın ve ortamı 4 °C'de 3 haftaya kadar saklayın.
- RPMI 1640'a 1x B27 takviyesi eksi insülin ve% 1 Penisilin-Streptomisin ekleyerek CM farklılaşma ortamını hazırlayın. RPMI 1640'a 1x B27 takviyesi ve% 1 Penisilin-Streptomisin ekleyerek CM bakım ortamını hazırlayın. Bu iki tür besiyerini 4 °C'de 2 hafta saklayın.
- Matrigel'i buza batırın ve gece boyunca 4 °C'de tutun. Matrigel'in tamamen çözüldüğünden emin olun; daha sonra, daha sonra kullanmak üzere -80 ° C'de saklanan 1 mL alikotlara alikot yapın ve Matrigel çalışma çözeltisini hazırlamak için 4 ° C'de 49 mL DMEM / F12'ye 1 mL Matrigel ekleyin. 6 oyuklu bir kültür plakasının oyuğu başına 850 μL bu Matrigel çalışma solüsyonunu ekleyin ve plakayı 37 °C'de en az 30 dakika inkübe edin. CM farklılaşmasında kullanılan 96 oyuklu kültür plakası için, oyuk başına 35 μL Matrigel çalışma çözeltisi ekleyin.
NOT: Matrigel'i her zaman buz üzerinde tutun; pipet uçları ve tüpleri 4 °C'de önceden soğutulmalıdır. Matrigel çalışma solüsyonu 4 ° C'de 2 hafta saklanabilir. Matrigel çalışma solüsyonunu pipetlerken kabarcıklardan kaçınma konusunda dikkatli olun.
- PSC bakımı ve geçişi
- PSC'leri 6 oyuklu kültür plakalarında tutun. Hücre yoğunluğunu mikroskop altında onaylayın ve birleşme ~%70'e ulaştığında geçişe hazırlanın (Şekil 2A).
- Hücreleri pas geçmek üzere sindirmek için EDTA kullanın. PSC kültür ortamını, PBS'yi ve EDTA'yı 37 ° C'lik bir su banyosunda ısıtın ve ortama 27632 μM'lik bir nihai konsantrasyona kadar Y5 ekleyin.
- PSC'leri 1 mL PBS ile yıkayın, oyuk başına 1 mL EDTA ekleyin ve plakayı 37 ° C'lik bir inkübatörde 3 dakika inkübe edin.
- Hücreler kültür plakasından ayrılmadan önce EDTA'yı aspire edin, ardından hücreleri yeniden süspanse etmek için kuyu 3-4x'in tabanını nazikçe durulamak için 1 mL PSC kültür ortamını kullanın.
NOT: Bu, santrifüjleme adımını atlamamıza ve geçiş işlemlerinde hücre hasarını azaltmamıza olanak tanır. - DMEM / F12'yi 6 oyuklu bir kültür plakasının Matrigel kaplı bir kuyusundan aspire edin ve oyuk başına 2 mL taze PSC kültür ortamı ekleyin (Y27632 eklendi). Hücre süspansiyonunu 1:6 ila 1:12 bölme oranıyla kuyucuklara ekleyin. Plakayı %5 CO2 içeren 37 °C inkübatöre yerleştirin.
- PSC kültür ortamını (Y27632 olmadan) geçtikten 12 ila 24 saat sonra değiştirin; Ardından, ortamı değiştirin ve hücre durumunu ve birleşmesini her gün kontrol edin.
- PSC'lerin Kardiyak Farklılaşması
- CM farklılaşması için, PSC'leri PSC hazırlama ortamında (Y27632 eklendi) 96 oyuklu bir kültür plakasına tohumlayın. CM farklılaşması için, PSC'leri PSC hazırlama ortamında (Y27632 eklendi) 96 oyuklu bir kültür plakasına tohumlamak için yukarıdaki adımların aynısını izleyin (adım 1.2.2-1.2.6).
NOT: Bu aşamada, EDTA tedavi süresi, PSC'leri yeterince ayırmak için uygun şekilde uzatılabilir (5 dakika veya daha fazla), böylece klonlar geçişten sonra daha küçük olabilir. Çalışmamız, aynı alan içinde daha küçük kolonilerin farklılaşmaya elverişli olduğunu göstermiştir. - Aşama I: PSC'ler %80-90 birleşmeye ulaştığında, ortamı 2-20 μM CHIR99021 (CHIR) ile CM farklılaşma ortamına geçirin (Şekil 2B). Ortam değiştirmenin zaman noktası 0. gün olarak adlandırılır. 24-48 saat CHIR işleminden sonra, ortamı taze CM farklılaşma ortamına değiştirin.
NOT: CHIR, mezoderm indüksiyonu için bir GSK3beta inhibitörüdür. CM farklılaşması için optimal CHIR dozu, farklı hücre hatları ve partileri ile dalgalanır. Örneğin, optimal CHIR dozu iPS-B1 için 8-10 μM, 48 saattir. Makine öğrenimi modelini eğitmek üzere çeşitli parlak alan görüntüleri toplamak için, burada farklı farklılaşma verimlilikleri için her bir deney grubu için CHIR konsantrasyonlarını titre ettik. - Aşama II: 72 saatte (3. gün), ortamı 5 μM IWR1 ile desteklenmiş CM farklılaşma ortamı ile değiştirin ve 48 saat (5. gün) için kültür. Ortamı CM farklılaşma ortamı ile 1-2 gün (6-7 güne) değiştirin. Bu noktada, PSC'ler kardiyak progenitör hücrelere (CPC) farklılaşır.
- Aşama III: Ortamı CM bakım ortamıyla değiştirin ve ortamı her 3 günde bir değiştirin. 10. günde veya 12. günde, daha fazla analiz için CM'yi hasat edin.
- CM farklılaşması için, PSC'leri PSC hazırlama ortamında (Y27632 eklendi) 96 oyuklu bir kültür plakasına tohumlayın. CM farklılaşması için, PSC'leri PSC hazırlama ortamında (Y27632 eklendi) 96 oyuklu bir kültür plakasına tohumlamak için yukarıdaki adımların aynısını izleyin (adım 1.2.2-1.2.6).
- CM'nin immünofloresan boyaması
- 10. veya 12. günde, hücreleri PBS ile yıkayın ve oda sıcaklığında 15 dakika boyunca PBS'de% 4 paraformaldehit ile sabitleyin. CM'leri 2x'i PBS ile yıkayın ve plakayı 4 °C'de ~1 haftaya kadar saklayın.
NOT: Paraformaldehit, keskin bir kokuya sahip toksik bir reaktiftir. Kimyasal bir başlıkta çalıştırın ve gözleri ve elleri koruyun. - Boyama zamanında, hücreleri oda sıcaklığında 15 dakika boyunca geçirgenleştirici solüsyon (PBS'de% 0.1 Triton X-100) ile muamele edin; daha sonra, hücreleri oda sıcaklığında 30 dakika boyunca bloke edici çözelti (PBS'de% 3 Eşek Serumu içeren% 0.1 Triton X-100) inkübe edin.
- CM'leri tanımlamak için numuneyi gece boyunca 4 ° C'de bloke edici çözelti içinde seyreltilmiş Kardiyak Troponin T (cTnT) birincil antikoru (1:300) ile inkübe edin.
- Birincil antikoru toplayın ve hücreleri PBS 3x ile yıkayın. Numuneyi, karanlık bir ortamda 37 ° C'de 1 saat boyunca% 1 sığır serum albümini ile PBS'de ikincil antikorlarla inkübe edin.
NOT: Toplanan primer antikor 1 hafta içinde 2-3 kez tekrar kullanılabilir.
- Birincil antikoru toplayın ve hücreleri PBS 3x ile yıkayın. Numuneyi, karanlık bir ortamda 37 ° C'de 1 saat boyunca% 1 sığır serum albümini ile PBS'de ikincil antikorlarla inkübe edin.
- İkincil antikoru hücrelerden çıkarın ve hücreleri 3x PBS ile yıkayın. Çekirdekleri oda sıcaklığında 5 dakika boyamak için Hoechst 33342 (PBS'de 1:1.000) kullanın. Hücreyi PBS kullanarak 3x durulayın ve kurumasını önlemek için oyuk başına 100 μL PBS ekleyin. Plakayı 4 °C'de karanlık bir yerde saklayın ve 1 hafta içinde floresan görüntüler elde edin.
- 10. veya 12. günde, hücreleri PBS ile yıkayın ve oda sıcaklığında 15 dakika boyunca PBS'de% 4 paraformaldehit ile sabitleyin. CM'leri 2x'i PBS ile yıkayın ve plakayı 4 °C'de ~1 haftaya kadar saklayın.
2. Farklılaşma süreci boyunca görüntü akışı elde etme
- Görüntülerin toplanması için deneysel tasarımın kurulması
- CM farklılaşmasında farklı aşamaların parlak alan görüntülerini ve farklılaşma sonucunun cTnT immünofloresan görüntülerini toplamak için canlı hücre kültürünü ve görüntülemeyi destekleyen otomatik bir mikroskop kullanın (Şekil 2A-E). Deney programını tasarlamak ve mikroskobu kontrol etmek için mikroskobu destekleyen yazılımı kullanın.
- Yazılımı açın ve yeni bir deneysel tasarım oluşturun. Görüntüleme için 5x objektif ve 2x tüp lens seçin. Kutucuklar ve Z yığını seçeneklerini kontrol edin.
- Kanallar menüsünde parlak alan görüntüleme için TL Parlak alan kanalı ekleyin. İmmünofloresan görüntüleme için AF488 ve H3342 kanalları ekleyin. Harici dijital CMOS kamera ile görüntüleme için Imaging Setup (Görüntüleme Ayarları) menüsündeki ışık yolunu değiştirin.
- Edinme Modu menüsünü açın ve sinyal-gürültü oranını artırmak için 2 x 2 gruplamayı ayarlayın.
- Z yığını menüsünü açın ve Orta modeli seçin. Tarama sırasında dilim sayısını ve aralıkları ayarlayın. Analizde odak içi görüntüler için 3-6 μm aralıklarla 3-5 dilim seçin.
- Navigasyon ve Döşemeler penceresinde, döşeme bölgelerini Taşıyıcıya göre ayarlayın ve bir kuyu için 25 döşeme (5 sütun x 5 satır) ayarlayın. Kutucuklar menüsünde, Örnek Taşıyıcı'da Multiwell 96 şablonunu seçin ve bitişik bölgelerde %5-% 15 arasında bir çakışma ayarlayın.
- Sürekli görüntüleme gerekiyorsa, Time serisi seçeneğini işaretleyin ve Zaman serisi menüsünde süreyi ve aralığı ayarlayın. Aşama I'de görüntü akışını toplamak için, aralıksız 12 saatlik bir süre ayarlayın.
- Parlak alan ve immünofloresan görüntülerin elde edilmesi
- Görüntü almak için önce hücre kültürü plakasını numune tepsisine koyun ve numuneyi mikroskobun içine yükleyin. Deneysel tasarıma uyması için Multiwell 96 şablonunu seçin. Numune canlı hücrelerden oluşuyorsa, kültür için uygun koşulu (37 °C, %5 CO2) korumak için ısıtma sistemini ve CO2 pompasını açın.
- Önceden ayarlanmış deneysel projeyi açın ve yeni bir proje olarak kaydedin. Fayanslar menüsünü açın ve plakanın konumunu manuel olarak kalibre edin. Navigasyon ve Döşemeler penceresinde, gereken kuyuları seçin ve bu kuyucuklar için döşeme bölgeleri oluşturmak için Oluştur'a tıklayın.
- Kutucuklar menüsünde Döşeme Bölgelerini Doğrula'ya tıklayın ve tüm kuyuları doğrulamak için Otomatik Odaklama'yı çalıştırın. Ardından, fare tekerleğini kaydırarak parlak alanın altındaki her bir kuyucuğun odağını manuel olarak düzeltin.
- Gerekli kanalları işaretleyin ve Kanallar menüsünde maruz kalma sürelerini ayarlayın. Önerilen maruz kalma süreleri Parlak için 1.625 ms, H3342 için 100 ms ve AF550 için 488 ms'dir. Tüm kanalların görüntülerini kontrol etmek için tek bir çırpıda oynatın.
- Son olarak, Denemeyi başlat düğmesine tıklayın ve otomatik görüntülemeyi bekleyin. Tipik olarak, 96 oyuklu bir kültür plakasının tamamının (96 kuyu, üç katman, parlak alan altındaki her kuyucuk için 25 karo) taranmasını bitirmek ~ 1,2 saat sürer. İşleme çerçevesinde, Görüntü Dışa Aktar'ı seçin, sıkıştırılmamış TIFF formatının veya PNG formatının dosya türünü seçin ve uygulayın. Daha fazla analiz için kuyunun ortasındaki 9 döşemenin (3 x 3) resimlerini kullanın (Şekil 2A-E).
3. Farklılaşma sürecinin her aşamasında imaj tabanlı makine öğrenimi stratejisinin oluşturulması
- Yazılım hazırlığı
- pytorch (1.9.0), numpy, scipy, scikit-learn, pandas, visdom, scikit-image, opencv-python ve matplotlib paketleriyle Python ortamını yükleyin.
- Jupyter Notebook'u yükleyin.
- MATLAB'ı yükleyin.
- Kodu ve örnek veri kümelerini https://github.com/zhaoyanglab/ML-for-PSC-differentiation'dan indirin.
NOT: Makine öğreniminin performansı, veri kümelerinin kalitesine ve modellerin ve hiper parametrelerin seçimine bağlıdır. Örnek uygulamanın tüm koşullar için en uygun olacağı garanti edilmez. Makine öğrenimi mühendisleri kodu özelleştirmeli ve modeli ve hiper parametreleri kendi özel ihtiyaçlarına göre uyarlamalıdır.
- PSC aşamasında makine öğrenimi stratejisi: İlk PSC koloni durumlarının kontrolü
Veri seti: 0 saatte (CHIR işleminden önce) parlak alan görüntü çiftleri ve son farklılaşma verimliliği
Model: rastgele orman- 0 saatte (CHIR tedavisinden önce) parlak alan görüntülerinden ve son cTnT floresan görüntülerinden oluşan bir veri seti hazırlayın. 0 saatlik parlak alan görüntülerinin morfolojik profillerinin çeşitliliğini artırmak için, veri kümesinin, geçişten sonraki bir zaman aralığında farklılaşma başlatılmış olarak, farklı hücre hatlarından PSC kolonilerini içerdiğinden emin olun. Veri setindeki kuyuları aşama I'de en uygun CHIR koşullarıyla tedavi edin.
NOT: Kolonilerin optimum CHIR koşullarıyla muamele edilmesini sağlamak için, aynı parti içindeki kuyucuklar titre edilmiş CHIR dozları ile muamele edilebilir. Veri setine yalnızca yüksek verimli CHIR koşullarına sahip kuyular dahil edilir. - cTnT floresan görüntülerinden hesaplanan Farklılaşma Verimliliği İndeksi ile her bir kuyucuğun farklılaşma verimliliğini ölçün, bu
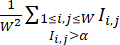 (burada
(burada 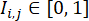 W × W floresan görüntüsündeki piksel konumundaki (i, j) floresan yoğunluğunu temsil eder ve toplam, yoğunluğu bir eşiğin üzerinde olan tüm piksel konumları üzerinden alınır α, genellikle 0,5 olarak ayarlanır). Her hücre hattında, bu hücre hattındaki tüm kuyucuklar için maksimum Farklılaşma Verimlilik İndeksini hesaplayın ve Farklılaşma Verimlilik İndekslerini maksimum değere göre normalleştirin.
W × W floresan görüntüsündeki piksel konumundaki (i, j) floresan yoğunluğunu temsil eder ve toplam, yoğunluğu bir eşiğin üzerinde olan tüm piksel konumları üzerinden alınır α, genellikle 0,5 olarak ayarlanır). Her hücre hattında, bu hücre hattındaki tüm kuyucuklar için maksimum Farklılaşma Verimlilik İndeksini hesaplayın ve Farklılaşma Verimlilik İndekslerini maksimum değere göre normalleştirin.
NOT: Her bir hücre hattının farklılaşma potansiyeli doğası gereği farklı olduğundan, böyle bir normalizasyon, farklılaşma sonucuna katkıda bulunan çeşitli hücre hatları arasında paylaşılan koloni morfolojik özelliklerine daha fazla vurgu yapacaktır. Normalizasyon gerçekten de farklı hücre hatlarından PSC kolonilerini karşılaştırılabilir hale getirir ve hücre hatlarının neden olduğu yanlılığı azaltır. - Koloni şeklinin özelliklerini ortaya çıkaran yüksek boyutlu özelliklerle 0 saatlik parlak alan görüntülerinin morfolojik profillerini ölçün. Parlak alan görüntülerinde koloni bölgelerinin ana hatlarını çizmek için yerel entropiyi hesaplayın ( yarıçapı r ile); Yerel entropisi bir eşik değerinden daha büyük olan pikseller σ hücrelerle kaplanarak tanımlanır. r = 10 piksel ve σ = 3 parametre ayarını kullanın. Çalışma dizinini "./colony_control" olarak değiştirin; parlak alan görüntülerini, hücre bölgelerinin ikili görüntülerini ve cTnT floresan görüntülerini "colony_control/ image_data/CD00-*/[brightfield|cell_region|ctnt]/S*.png" konumuna kaydedin; ve diğerlerinin yanı sıra koloni alanı, çevre, katılık, dışbükeylik, dairesellik ve merkez kontur mesafeleri ile ilgili özellikleri hesaplamak için Jupyter Notebook Compute_features".ipynb" komutunu çalıştırın.
- Veri kümesini, genel verilerin %50 ila %80'ini oluşturacak şekilde rastgele bir eğitim kümesine ve bir test kümesine bölün. Son bloğu çalıştırın: Veri kümesini bölmek için Jupyter Notebook'ta veri kümesini bölün (Şekil 3A).
- 0 saatlik parlak alan görüntü özelliklerinden farklılaşma verimliliğini tahmin etmek için eğitim kümesinde rastgele bir orman regresyon modeli eğitin. Modeli eğitmek ve test etmek için Jupyter Notebook "Machine_learning.ipynb" komutunu çalıştırın.
- Test kümesinde eğitilen rastgele orman modelini değerlendirin. Pearson korelasyon katsayısını kullanarak tahmin edilen farklılaşma verimliliğinin gerçek olanla yüksek oranda ilişkili olduğunu onaylayın (Şekil 3B).
NOT: Test seti eğitim aşamasında yer almadığından, test seti üzerinde tahmin edilen ve gerçek farklılaşma verimliliği arasında yüksek bir tutarlılık, modelin görünmeyen numuneler üzerinde iyi bir genelleme yapabileceğini gösterir. - Eğitilen rastgele orman modelinden özellik önem derecesi elde edin ve en önemli 8 özelliği seçin. Özellik aralığını bölmelere bölerek ve her bölmedeki ortalama farklılaştırma verimliliğini hesaplayarak nihai farklılaştırma verimliliği ile ilişkilerini görselleştirin. Görselleştirmeyi gerçekleştirmek için Jupyter Notebook "Feature_importance.ipynb" komutunu çalıştırın (Şekil 3C).
NOT: Görselleştirme, PSC koloni şeklindeki değişikliklerin farklılaşmaya nasıl katkıda bulunduğunu anlamaya yardımcı olabilir. - PSC koloni durumlarını gerçek zamanlı olarak izlemek için eğitilen modeli yeni toplu işlemlerde uygulayın. Parlak alan görüntüsünü her saat yakalayın, görüntüyü eğitilen modele geçirin ve tahmin edilen farklılaşma verimliliğini elde edin. Tahmin edilen verimlilik bir eşiği aştığında (örneğin, %50), farklılaşma sürecini başlatın. Tahmin edilen verimlilik eşiğe ulaşamazsa, bu örnekleri atın.
NOT: PSC aşamasında, PSC kolonilerinin farklılaşma potansiyeli önce yükselecek ve daha sonra azalacaktır. Gerçek zamanlı izleme, deneycilerin farklılaşmayı başlatmak için en elverişli zaman noktasını belirlemelerini sağlar.
- 0 saatte (CHIR tedavisinden önce) parlak alan görüntülerinden ve son cTnT floresan görüntülerinden oluşan bir veri seti hazırlayın. 0 saatlik parlak alan görüntülerinin morfolojik profillerinin çeşitliliğini artırmak için, veri kümesinin, geçişten sonraki bir zaman aralığında farklılaşma başlatılmış olarak, farklı hücre hatlarından PSC kolonilerini içerdiğinden emin olun. Veri setindeki kuyuları aşama I'de en uygun CHIR koşullarıyla tedavi edin.
- Aşama I'de makine öğrenimi stratejisi: CHIR dozlarının erken değerlendirilmesi ve ayarlanması
Veri seti: 0-12 saatte parlak alan görüntü akışı çiftleri ve CHIR konsantrasyon etiketleri (düşük/optimal/yüksek)
Model: lojistik regresyon- Görüntülerin her saat 0 ile 12 saat arasında elde edildiği, bütünü iyi parlak alan görüntü akışlarından oluşan bir veri kümesi hazırlayın. Her bir oyuğun CHIR dozlarını (yani, CHIR konsantrasyonları ve süresinin kombinasyonları) kaydedin. Çeşitliliği artırmak için veri setinin titre edilmiş CHIR dozlarına sahip farklı partiler ve kuyucuklar içerdiğinden emin olun (örneğin, konsantrasyon = 2, 4, 6, 8, 10 ve 12 μM; süre = 24, 36 ve 48 saat). Çalışma dizinini "./stage_I" olarak değiştirin; görüntüleri "./data/image/CD01-*/S*/T*.png" olarak kaydedin, burada CD01-* (CD01-1, CD01-2, CD01-3, CD01-4) parti adıdır, S* (S1, S2, ..., S96) kuyu indeksidir ve T*.png (T1.png, T2.png, ..., T10.png) görüntü akışının farklı zaman adımlarında kuyunun önceden işlenmiş parlak alan görüntüsüdür.
- Her partide, cTnT+ hücrelerinin sayısının floresan görüntüsündeki toplam Hoechst+ hücre sayısına bölünmesiyle hesaplanan her bir kuyucuk için cTnT+ hücrelerinin yüzdesini hesaplayın. Aynı CHIR doz koşuluna sahip paralel kuyucuklar üzerindeki cTnT + hücrelerinin yüzdesinin ortalaması. Her kuyucuk için CHIR doz durumunu ve cTnT + hücrelerinin yüzdesini "./data/image/CD01-*/CD01-?_exp_config.csv" bir csv dosyasına kaydedin.
- Her partide, aşağıdaki kriterlere göre her CHIR süresi altında "düşük", "optimal" ve "yüksek" CHIR konsantrasyon aralığını belirleyin. Bir eşiğin üzerinde (örneğin, %20) ortalama cTnT + hücre yüzdesine sahip CHIR konsantrasyonları "optimal" olarak tanımlanır. Optimal aralığın ötesindeki CHIR konsantrasyonları "düşük" veya "yüksek" olarak tanımlanır. Her CHIR süresi altında, kuyucukları karşılık gelen CHIR konsantrasyonlarına göre "düşük", "optimal" ve "yüksek" olarak etiketleyin.
NOT: Ampirik olarak, CHIR konsantrasyonları ile bir parti içindeki yüksek verimli kuyuların süresi arasında negatif bir korelasyon vardır: daha yüksek CHIR süresi için optimal konsantrasyonlar azalır. Başka bir deyişle, CHIR konsantrasyonlarının optimalliği CHIR süresine bağlıdır ve bu nedenle, kuyuların etiketleri farklı CHIR süreleri arasında farklılık gösterir. - Her partide, her CHIR süresi altında, optimumdan sapmasını ölçmek için her konsantrasyon c için ΔCHIR konsantrasyonunu hesaplayın. Optimum CHIR konsantrasyon aralığını [c1, c2] ile belirtin ve ardından ΔCHIR konsantrasyonu (c) = c - c1 (c < c1 ise), 0 (c 1 ≤ c ≤ c2 ise) veya c - c2 (c > c2 ise).
- Veri kümesindeki görüntü akışlarının özelliklerini ayıklayarak, I. aşama sırasında hücrelerin morfolojik değişikliklerini yansıtmalarını sağlayın. Yerel entropi, alan, çevreler, parlaklık, optik akış ve fraktal boyut ile ilgili görüntü özelliklerini hesaplamak için şunu çalıştırın:
cd ./data
python compute_features.py
CD..
NOT: Morfolojik değişiklikleri vurgulamak için, bazı özelliklerin değerleri (örn. alan, çevreler ve optik akış) görüntü akışlarındaki ilk temel değerleriyle normalleştirilebilir. - Veri kümesini rastgele bir eğitim kümesine ve bir test kümesine bölün. Veri kümesini bölmek ve veri kümelerini Veri Çerçeveleri olarak düzenlemek için Jupyter Notebook "./data/prepare_dataset.ipynb" komutunu çalıştırın (Şekil 3D).
- Her CHIR süresi için, eğitim setindeki görüntü akışı özelliklerinden CHIR konsantrasyon etiketini (düşük/optimal/yüksek) tahmin etmek için bir lojistik regresyon modeli eğitin. Doğruluk, kesinlik, geri çağırma, F1 puanı ve eğrinin altındaki alan (AUC) kullanarak test setinde eğitilen lojistik regresyon modelinin sınıflandırma performansını değerlendirin. Eğitim ve test için Jupyter Notebook "machine_learning.ipynb" çalıştırın (Şekil 3E).
- CHIR doz değerlendirmesinde model performansını değerlendirin. Test setinde, aynı CHIR konsantrasyonuna sahip paralel kuyuların tahmin edilen etiketlerini, Sapma
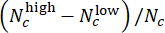 Skorları (-1 ile 1 arasında değişen) kullanarak birleştirin, burada Nc, c konsantrasyonuna sahip kuyuların sayısını gösterir ve
Skorları (-1 ile 1 arasında değişen) kullanarak birleştirin, burada Nc, c konsantrasyonuna sahip kuyuların sayısını gösterir ve 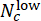 aralarındaki
aralarındaki 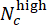 kuyular sırasıyla "düşük" ve "yüksek" olarak tahmin edilir. Tahmin edilen Sapma Skorlarının, Pearson korelasyon katsayısını kullanarak her bir CHIR dozu için gerçek ΔCHIR konsantrasyonu ile yüksek oranda ilişkili olduğunu onaylayın.
kuyular sırasıyla "düşük" ve "yüksek" olarak tahmin edilir. Tahmin edilen Sapma Skorlarının, Pearson korelasyon katsayısını kullanarak her bir CHIR dozu için gerçek ΔCHIR konsantrasyonu ile yüksek oranda ilişkili olduğunu onaylayın. - Modelin genelleştirme performanslarını test etmek için toplu işler arası doğrulama gerçekleştirin. Her turda, test için bir parti bırakırken diğerlerini eğitim için kullanın. Pearson korelasyon katsayısını kullanarak her test partisindeki her CHIR dozu için tahmin edilen Sapma Skorlarını ve gerçek ΔCHIR konsantrasyonunu karşılaştırın. Toplu iş doğrulamasını uygulamak için Jupyter Notebook "cross_batch_validation.ipynb" komutunu çalıştırın (Şekil 3F).
NOT: Seçilen bir CHIR süresi h için, h süresi altında eğitilen lojistik regresyon modelinden konsantrasyona sahip tüm kuyular için tahmin, CHIR konsantrasyonu c'nin optimalliğini yansıtan bir Sapma Puanına dönüştürülecektir. 0'a yakın bir Sapma Skoru, konsantrasyon c'nin h süresi altında optimal olacağının tahmin edildiğini gösterirken, negatif veya pozitif bir Sapma Skoru, CHIR dozlarının daha sonra ayarlanması için bir sinyal olarak hizmet edebilecek bir düşük doz veya aşırı doz durumunu gösterir. Tahmin erken (12 saat) yapıldığından, aşama I'in geri kalanında nihai CHIR süresinin ve CHIR konsantrasyonunun hala ayarlanabileceğini unutmayın. - Yeni partilerdeki CHIR dozlarını değerlendirmek için eğitimli lojistik regresyon modellerini uygulayın. Belirli bir CHIR konsantrasyonu için, konsantrasyonlu paralel kuyular için 0-12 saatlik görüntü akışı özelliklerini hesaplayın, özellikleri farklı CHIR süreleri altında eğitilen lojistik regresyon modellerine iletin ve buna göre her kuyu için tahmin edilen etiketleri elde edin. Modellerin tahminine bağlı olarak, farklı CHIR süreleri altında her bir CHIR konsantrasyonunu değerlendirmek için Sapma Puanlarını hesaplayın.
- Model tabanlı CHIR doz değerlendirmesi ile, CHIR sürelerini veya konsantrasyonlarını 48 saatten önce optimuma ayarlayarak her bir optimal olmayan CHIR konsantrasyonunun altındaki kuyuları kurtarın.
- Ayarlama süresi: Optimum CHIR süresi ile ilgili model tahminine göre, bu kuyucuklardaki CHIR içeren ortamın CM farklılaşma ortamına geçirildiği zaman noktasını ayarlayın. En uygun zaman noktası kaçırılırsa, bu örnekleri atın.
- Konsantrasyonun ayarlanması: önceden ayarlanmış CHIR süresi altında, mevcut konsantrasyon "düşük" olarak tahmin ediliyorsa, CHIR konsantrasyonunu artırmak için ortama doğrudan yüksek bir CHIR konsantrasyonu ekleyin. Gerektiğinde CHIR'i DMSO'da (50 mM) PBS ile seyreltin, uygun miktarda sıvı ekleyin ve plakayı hafifçe sallayın. Mevcut konsantrasyonun "yüksek" olduğu tahmin ediliyorsa, belirli bir hacimde CHIR içeren ortamı aspire edin ve aynı hacimde CM farklılaşma ortamı ile destekleyin.
NOT: Farklılaşmanın ilk 48 saatinde, hücreler hücre büyümesini ve farklılaşmasını iyileştirmek için faktörler salgılar, bu nedenle ortamı tamamen değiştirmeyin. Ek olarak, farklılaşmada orta hacmi sabit tutmak gerekir. Bu nedenle, CHIR konsantrasyonunu ayarlarken eklenen sıvı hacminin mümkün olduğunca küçük olduğundan emin olun.
- Aşama II'de makine öğrenimi stratejisi: CM tarafından taahhüt edilen TBM'lerin tanınması
Veri seti: 6. günde parlak alan görüntüleri çiftleri ve CM tarafından taahhüt edilen TBM'lerin manuel olarak açıklamalı maskeleri
Model: Zayıf denetimli öğrenme için Grad-CAM36 ile ResNeSt38- 6. günde parlak alan görüntülerinden oluşan bir veri seti hazırlayın. Görüntü akışlarındaki cTnT+ hücrelerini 12. günden 6. güne kadar izleyerek parlak alan görüntülerinde CM tarafından taahhüt edilen TBM'lere manuel olarak açıklama ekleyin.
- CM tarafından taahhüt edilen TBM'lerin parlak alan görüntülerini ve karşılık gelen manuel ek açıklamalarını (maskeler olarak adlandırılır), her bir yama tam kuyu görüntü boyutunun 1/5,5'i boyutunda olacak şekilde yamalar halinde kırpın. Bitişik yamaların eğitim setinde %50 ve test setinde %75 örtüşme paylaştığı yerlerde yamaları çakışan bir şekilde ayıklayın. %≥30 CM taahhütlü TBM'lere sahip yamaları "pozitif" olarak etiketleyin. CM tarafından taahhüt edilen TBM'leri olmayan yamaları "negatif" olarak etiketleyin. Çalışma dizinini "stage_II" olarak değiştirin. Görüntüleri kırpmak için "./Crop_and_Reconstruct/mask_crop.m" komutunu çalıştırın. Parlak alan yamalarına etiketler (0: negatif; 1: pozitif) atamak için "./Crop_and_Reconstruct/classification.m" komutunu çalıştırın (Şekil 3G).
- Etiketli görüntü yamalarını rastgele bir eğitim setine ve bir test setine bölün.
- Bu yamaları sınıflandırmayı öğrenmek için derin bir evrişimli sinir ağı olan ResNeSt38'i eğitin. Modelin yüksek bir sınıflandırma doğruluğu gösterdiğini doğrulamak için test setinde eğitilen ResNeSt'i değerlendirin. Eğitim ve test için sırasıyla "train.py" ve "test.py" çalıştırın (Şekil 3H).
- Isı haritalarıyla temsil edilen ResNeSt çıkarımına en çok katkıda bulunan bölgeleri vurgulamak için Grad-CAM36'yı kullanın. Görüntülenen TBM (IR-CPC) bölgeleri olarak adlandırılan, tahmin edilen CM taahhütlü TBM bölgelerini elde etmek için ısı haritalarını ikili hale getirin (örneğin, maksimum %10 × bir eşik kullanarak).
- Doğruluk, F1 puanı, kesinlik, geri çağırma, özgüllük ve birleşim üzerinden kesişim (IoU) kullanarak IR-CPC bölgelerini test setinde manuel olarak açıklamalı maskelerle karşılaştırın. Pearson korelasyon katsayısını kullanarak IR-CPC bölgelerinin oranını Farklılaşma Verimlilik İndeksleri (nihai cTnT floresan görüntülerinden hesaplanan) ile karşılaştırın. Performans değerlendirmesi için "./Evaluation/index_calculate.m" ve "./Evaluation/Pearson_correlation.m" komutunu çalıştırın (Şekil 3I).
- Aşama II'de yeni partiler için TBM'leri tanımak için eğitilen modeli uygulayın. Tam kuyu parlak alan görüntülerini yamalar halinde kırpın, yamaları eğitimli ResNeSt'e geçirin ve yama düzeyinde IR-CPC bölgelerini elde edin, bunlar daha sonra tüm kuyu IR-CPC bölgelerini elde etmek için birleştirilir. Nihai farklılaşma verimliliğinin bir göstergesi olarak IR-CPC bölgelerinin fraksiyonunu hesaplayın. Hücre kontaminasyonunu azaltmak için IR-CPC'leri saflaştırın (aşağıda açıklanmıştır).
NOT: CPC tanıma modeli, nihai farklılaşma verimliliğinin erken bir değerlendirmesini yapabildiğinden, farklılaşma sürecine elverişli olan küçük moleküllerin taranması için yüksek verimli bir okuma görevi de görebilir, bu da farklılaşma protokolünü daha da optimize edebilir35.
- Görüntülenen ve tanınan TBM'lerin bölge seçici saflaştırılması
NOT: Fotoaktivasyon deneyleri, motorlu bir kademe ile kolaylaştırılmış ters çevrilmiş bir floresan mikroskobuna dayanmaktadır.- Eğitilen model tarafından tahmin edilen IR-CPC bölgelerine göre, herhangi bir IR-CPC üretemeyen kuyuları atın. Bu CM taahhütlü TBM'leri saflaştırmak için, sitotoksik olmayan fotoaktive edilebilir bir prob olan Çift Aktive Edilebilir Hücre İzleyici 1 (DACT-1) kullanarak CPColmayan 39'u bölge seçici olarak etiketleyin.
- DACT-1'i (10 mM) DMSO'da çözün ve -20 °C'de saklayın. 6. gün hücrelerini CM farklılaşma ortamında 1 μM DACT-1 ile 37 ° C'de 30 dakika inkübe edin.
- Canlı hücreleri mikroskop altında inceleyin ve ilgilenilen bölgeyi (ROI) MATLAB'da çokgen olarak seçin. Çokgen içinde 20 μm aralıklarla paralel çizgiler çizin ve kesişme noktalarının koordinatlarını hesaplayın.
- Motorlu aşamanın hareketli izini iz çizgilerine göre ayarlayın, ardından aşama 0,12 mm/s'de hareket eder, böylece sahneye monte edilen 405nm lazer çizgisi (20 μm çapında), TBM olmayanları kısıtlı bir şekilde fotoaktif hale getirmek için ROI'yi küresel olarak tarayabilir. Işınlamadan sonra, 561 nm lazer çizgisi kullanarak DACT-1 etiketli hücreleri tespit edin.
- 37 ° C'de 5-7 dakika boyunca% 0.05 Tripsin-EDTA (PBS ile seyreltilmiş) kullanarak hücreleri ayırın ve plakayı 2 dakika boyunca bir inkübatörde hafifçe sallayın. Hücreleri 40 μm'lik bir hücre süzgecinden süzün ve 500 × g'da 3 dakika santrifüjleyin. Hücreleri %0,5 BSA'da yeniden süspanse edin ve sıralanana kadar tüpü buz üzerinde tutun. Ayrıca, sıralama için negatif kontrol olarak ışınlanmamış bir numune hazırlayın.
- Akış sitometrisi sıralama sistemini başlatın ve sıralayıcı yazılımını kurun. 561 nm dalga boylu lazerleri seçin ve DACT1+ sıralaması için 610/20 bant geçiren bir filtre kullanın. Her örnek için, tek hücreyi seçmek için ileri saçılma (FSC) ve yan saçılma (SSC) olmak üzere iki değişkenli bir çizimde hücre popülasyonunu daire içine almak için çokgen bir geçit aracı kullanın. Ardından, arka plan floresansını doğrulamak için negatif kontrolü çalıştırın, bölge seçici olarak ışınlanmış numuneyi çalıştırın ve DACT1+ popülasyonu için kapıyı çizin.
- % 10 FBS ve 5 μM Y27632 ile CM bakım ortamında sıralanmış hücreleri toplayın ve yeniden askıya alın ve sıralanan hücreleri Matrigel kaplı 96 oyuklu bir plakada tohumlayın.
- Ertesi gün, ortamı CM bakım ortamı olarak değiştirin ve yüksek saflıkta CM'lerin hasat edilmesini bekleyin.
- Aşama III'te makine öğrenimi stratejisi: CM'lerin tanınması
Veri seti: 12. gündeki parlak alan görüntüleri çiftleri ve son cTnT floresan görüntüleri
Model: pix2pix37- CM'lerin parlak alan görüntülerinden ve karşılık gelen cTnT floresan görüntülerinden oluşan bir veri kümesi hazırlayın. Çeşitliliği artırmak için, veri setinin çeşitli farklılaşma verimliliklerine ve hücre hatlarına sahip kuyuları içerdiğinden emin olun.
- Veri kümesini rastgele bir eğitim kümesine ve bir test kümesine bölün. Çalışma dizinini "stage_III" olarak değiştirin. Parlak alan görüntülerini ve karşılık gelen cTnT floresan görüntülerini sırasıyla "./pix2pix/datasets/(CM|CM_new_cell_lines)/A/(train|test)/*.png" ve ".../B/(train|test)/*.png" konumuna kaydedin. Aşağıdaki komutu çalıştırın:
cd pix2pix/veri kümeleri
python combine_A_and_B.py --fold_A ./CM/A --fold_B ./CM/B --fold_AB ./CM/ --no_multiprocessing
CD.. - pix2pix model37'yi eğitim setinde eğitin. Aşağıdaki komutu çalıştırın (Şekil 3J):
python train.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 256 --lr 2e-4 --n_epochs 1000 --n_epochs_decay 0 --norm örneği --netD n_layers --n_layers_D 1 --batch_size 16 --direction AtoB --save_epoch_freq 100 --dataset_mode hizalı --use_resize_conv --tohum 1234 - Eğitilen pix2pix modelini test setine uygulayın. Aşağıdaki komutu çalıştırın:
python test.py --dataroot ./datasets/CM --name brightfield2fluorescence --model pix2pix --direction AtoB --input_nc 1 --output_nc 1 --load_size 1536 --crop_size 1536 --use_resize_conv --eval --num_test 1000 - Model tarafından tahmin edilen floresan görüntülerini gerçek (yani deneysel olarak elde edilen) görüntülerle karşılaştırın. Piksel düzeyinde, Pearson korelasyon katsayısını kullanarak tahmin edilen yoğunlukları ve gerçek yoğunlukları karşılaştırın. Ek olarak, görüntü düzeyinde, Pearson korelasyon katsayısını kullanarak tahmin edilen Farklılaşma Verimlilik İndekslerini gerçek olanlarla karşılaştırın. Bu karşılaştırmalar için "stage_III/evaluation/pixel_correlation.m" ve "image_correlation.ipynb" çalıştırın (Şekil 3K,L).
- Aşama III'te yeni partilerdeki CM'leri tanımak için eğitilen modeli uygulayın. CM'lerin yeni parlak alan görüntülerini eğitimli pix2pix modeline aktarın ve floresan görüntülerinin ve farklılaşma verimliliğinin in-silico bir tahminini elde edin.
NOT: Hücre kontaminasyonunu azaltmak için, model tarafından tanınan CM'leri saflaştırmak için DACT-1 tabanlı fotoaktif hücre sınıflandırması da geçerlidir.
Sonuçlar
Parlak alan görüntüleme ve makine öğrenimine dayalı olarak, genel farklılaşma süreci akıllı bir şekilde izlenebilir ve optimize edilebilir. PSC aşamasında, farklılaşmayı başlatmak için en uygun veya uygun zaman noktasını belirlemek için ilk PSC kolonilerinin morfolojik özelliklerine göre nihai farklılaşma verimliliğini tahmin edebilen bir makine öğrenimi modeli geliştirdik (Şekil 4A,B). Rastgele orman modeli ta...
Tartışmalar
Burada, mevcut PSC uygulaması ve çevirisindeki en büyük sorunlardan biri olan hücre farklılaşmasındaki değişkenliğin üstesinden gelmek için ayrıntılı bir protokol tanımladık. Canlı hücreli parlak alan görüntüleme ve makine öğreniminden yararlanarak, hücre hatları ve partiler arasında tutarlı bir şekilde yüksek verimlilik elde etmek için PSC farklılaşmasını yinelemeli olarak optimize ettik. Bununla birlikte, yukarıdaki farklılaşma sürecinde, protoko...
Açıklamalar
Yang Zhao, Jue Zhang, Xiaochun Yang, Yao Wang ve Daichao Chen, bu belgede (202210525166.X) bildirilen PSC farklılaşma stratejisi için bir patent başvurusunda bulunuyorlar.
Teşekkürler
Bu stratejinin temelini attıkları için Qiushi Sun, Yao Wang, Yu Xia, Jinyu Yang, Chang Lin, Zimu Cen, Dongdong Liang, Rong Wei, Ze Xu, Guangyin Xi, Gang Xue, Can Ye, Li-Peng Wang, Peng Zou, Shi-Qiang Wang, Pablo Rivera-Fuentes, Salome Püntener, Zhixing Chen, Yi Liu ve Jue Zhang'a teşekkür ederiz. Bu çalışma, Çin Ulusal Anahtar Ar-Ge Programı (2018YFA0800504, 2019YFA0110000) ve Çin İnsanlı Uzay Programı (HYZHXM01020) Uzay Tıbbi Deney Projesi tarafından Yang Zhao'ya desteklenmiştir. Şekil 1 , BioRender.com ile oluşturulmuştur.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | Diluted digests were used for CPC and CM digestion |
| 4% Paraformaldehyde in PBS | KeyGEN BioTECH | KGIHC016 | |
| 6-well Cell Culture Plate | NEST | 703001 | |
| 96-well Cell Culture Plate | NEST | 701001 | |
| B27 Supplement | Gibco | 17504044 | |
| B27 Supplement Minus Insulin | Gibco | A1895601 | |
| Bovine serum albumin (BSA) | GPC BIOTECH | AA904-100G | |
| Celldiscoverer 7 | Zeiss | Instruments used to take bright-field images throughout differentiation and final cTnT images | |
| CHIR99021 | Selleck | S1263 | |
| DMEM/F12 | Gibco | 12634010 | |
| Donkey anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Thermo | A-21202 | Secondary Antibody |
| FACSAria III | BD Biosciences | Flow cytometry sorter | |
| Fetal Bovine Serum (FBS) | VISTECH | SE100-B | |
| Hoechst 33342 | YEASEN | 40732ES03 | |
| Human Pluripotent Stem Cell Chemical-defined Medium | Cauliscell Inc | 400105 | Basal medium of PSC preparation medium |
| iPS-18 | TaKaRa | Y00300 | |
| iPS-B1 | Cellapy | CA4025106 | |
| iPS-F | Nuwacell | RC01001-B | |
| iPS-M | Nuwacell | RC01001-A | |
| IWR1-1-endo | Selleck | S7086 | IWR1 |
| Jupyter Notebook | N/A | Version 6.4.0 | https://jupyter.org/ |
| MATLAB | MathWorks | Version R2020a | Software for scientific computation and image annotation |
| Matrigel Matrix | Corning | 354230 | Matrigel |
| Mouse monoclonal IgG1 anti-cTnT | Thermo | MA5-12960 | cTnT primary antibody |
| Normal Donkey Serum | Jackson | 017-000-121 | |
| ORCA-Flash 4.0 V3 digital CMOS camera | Hamamatsu | C13440-20CU | The digital camera assembled on Celldiscoverer7 |
| PBS | NEB | 21-040-CVR | |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Pluripotency Growth Mater 1 basal medium | Cellapy | CA1007500-1 | Basal medium of PSC culture medium |
| Pluripotency Growth Mater 1 supplement | Cellapy | CA1007500-2 | Supplement of PSC culture medium |
| Prism | Graphpad | Version 8/9 | Statistical software for statistical analysis and plotting |
| Python | N/A | version 3.6 | Python 3 environment for scientific computation, with packages pytorch (1.9.0), numpy, scipy, pandas, visdom, scikit-learn, scikit-image, opencv-python, and matplotlib software for scientific computation and image annotation. |
| RPMI 1640 | Gibco | 11875176 | |
| Supplement hPSC-CDM (500x) | Cauliscell Inc | 00015 | Supplement of PSC preparation medium |
| TiE | Nikon | An inverted fluorescence microscope (with modification) for region-selevtive purification | |
| Triton X-100 | Amresco | 9002-93-1 | |
| Versene Solution | Thermo | 15040066 | EDTA solution for PSC digestion |
| Y27632 | Selleck | S6390 | |
| Zen | Zeiss | Version 3.1 | A supporting software of Celldiscoverer7 for image acquisition, processing and analysis |
Referanslar
- Yoshida, Y., Yamanaka, S. Induced pluripotent stem cells 10 years later: for cardiac applications. Circ Res. 120 (12), 1958-1968 (2017).
- Shi, Y., Inoue, H., Wu, J. C., Yamanaka, S. Induced pluripotent stem cell technology: a decade of progress. Nat Rev Drug Discov. 16 (2), 115-130 (2017).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Burridge, P. W., et al. Chemically defined generation of human cardiomyocytes. Nat Methods. 11 (8), 855-860 (2014).
- Mordwinkin, N. M., Burridge, P. W., Wu, J. C. A review of human pluripotent stem cell-derived cardiomyocytes for high-throughput drug discovery, cardiotoxicity screening, and publication standards. J Cardiovasc Transl Res. 6 (1), 22-30 (2013).
- Vanden Berg, C. W., Elliott, D. A., Braam, S. R., Mummery, C. L., Davis, R. P. Differentiation of human pluripotent stem cells to cardiomyocytes under defined conditions. Methods Mol Biol. 1353, 163-180 (2016).
- Burridge, P. W., Holmström, A., Wu, J. C. Chemically defined culture and cardiomyocyte differentiation of human pluripotent stem cells. Curr Protoc Hum Genet. 87, 1-15 (2015).
- Li, J., et al. Human pluripotent stem cell-derived cardiac tissue-like constructs for repairing the infarcted myocardium. Stem Cell Rep. 9 (5), 1546-1559 (2017).
- Smith, A. S. T., Macadangdang, J., Leung, W., Laflamme, M. A., Kim, D. -. H. Human iPSC-derived cardiomyocytes and tissue engineering strategies for disease modeling and drug screening. Biotechnol Adv. 35 (1), 77-94 (2017).
- Sharma, A., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes to assess drug cardiotoxicity. Nat Protoc. 13 (12), 3018-3041 (2018).
- Mehta, A., et al. Identification of a targeted and testable antiarrhythmic therapy for long-QT syndrome type 2 using a patient-specific cellular model. Eur Heart J. 39 (16), 1446-1455 (2018).
- Cahan, P., Daley, G. Q. Origins and implications of pluripotent stem cell variability and heterogeneity. Nat Rev Mol Cell Biol. 14 (6), 357-368 (2013).
- Kim, K., et al. Epigenetic memory in induced pluripotent stem cells. Nature. 467 (7313), 285-290 (2010).
- Rouhani, F., et al. Genetic background drives transcriptional variation in human induced pluripotent stem cells. PLoS Genet. 10 (6), e1004432 (2014).
- Kim, K., et al. Donor cell type can influence the epigenome and differentiation potential of human induced pluripotent stem cells. Nat Biotechnol. 29 (12), 1117-1119 (2011).
- Garitaonandia, I., et al. Increased risk of genetic and epigenetic instability in human embryonic stem cells associated with specific culture conditions. PLoS One. 10 (2), e0118307 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Matoba, N., Yamashita, T., Takayama, K., Sakurai, F., Mizuguchi, H. Optimal human iPS cell culture method for efficient hepatic differentiation. Differentiation. 104, 13-21 (2018).
- Volpato, V., et al. Reproducibility of molecular phenotypes after long-term differentiation to human iPSC-derived neurons: A multi-site omics study. Stem Cell Rep. 11 (4), 897-911 (2018).
- Anderson, N. C., et al. Balancing serendipity and reproducibility: Pluripotent stem cells as experimental systems for intellectual and developmental disorders. Stem Cell Rep. 16 (6), 1446-1457 (2021).
- Sepac, A., et al. Comparison of cardiomyogenic potential among human ESC and iPSC lines. Cell Transplant. 21 (11), 2523-2530 (2012).
- Laco, F., et al. Unraveling the inconsistencies of cardiac differentiation efficiency induced by the GSK3β inhibitor CHIR99021 in human pluripotent stem cells. Stem Cell Rep. 10 (6), 1851-1866 (2018).
- Volpato, V., Webber, C. Addressing variability in iPSC-derived models of human disease: guidelines to promote reproducibility. Dis Model Mech. 13 (1), 042317 (2020).
- Hu, B. -. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Huo, J., et al. Evaluation of batch variations in induced pluripotent stem cell-derived human cardiomyocytes from 2 major suppliers. Toxicol Sci. 156 (1), 25-38 (2017).
- Rostam, H. M., Reynolds, P. M., Alexander, M. R., Gadegaard, N., Ghaemmaghami, A. M. Image based machine learning for identification of macrophage subsets. Sci Rep. 7 (1), 3521 (2017).
- Christiansen, E. M., et al. In silico labeling: Predicting fluorescent labels in unlabeled images. Cell. 173 (3), 792-803 (2018).
- Buggenthin, F., et al. Prospective identification of hematopoietic lineage choice by deep learning. Nat Methods. 14 (4), 403-406 (2017).
- Smith, Z. D., Nachman, I., Regev, A., Meissner, A. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event. Nat Biotechnol. 28 (5), 521-526 (2010).
- Fan, K., Zhang, S., Zhang, Y., Lu, J., Holcombe, M., Zhang, X. A machine learning assisted, label-free, non-invasive approach for somatic reprogramming in induced pluripotent stem cell colony formation detection and prediction. Sci Rep. 7 (1), 13496 (2017).
- Kusumoto, D., et al. Automated deep learning-based system to identify endothelial cells derived from induced pluripotent stem cells. Stem Cell Rep. 10 (6), 1687-1695 (2018).
- Zhu, Y., et al. Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun. 12 (1), 2614 (2021).
- Qian, T., et al. Label-free imaging for quality control of cardiomyocyte differentiation. Nat Commun. 12 (1), 4580 (2021).
- Ounkomol, C., Seshamani, S., Maleckar, M. M., Collman, F., Johnson, G. R. Label-free prediction of three-dimensional fluorescence images from transmitted-light microscopy. Nat Methods. 15 (11), 917-920 (2018).
- Yang, X., et al. A live-cell image-based machine learning strategy for reducing variability in PSC differentiation systems. Cell Discovery. 9 (1), 53 (2023).
- Selvaraju, R. R., et al. Grad-CAM: Visual explanations from deep networks via gradient-based localization. Proc IEEE Int Conf Computer Vision (ICCV). , 618-626 (2017).
- Isola, P., Zhu, J. -. Y., Zhou, T., Efros, A. A. Image-to-image translation with conditional adversarial networks. Proc IEEE Conf Computer Vision Pattern Recognition (CVPR). , 5967-5976 (2017).
- Zhang, H., et al. ResNeSt: Split-attention networks. Proc IEEE/CVF Conf Computer Vision Pattern Recognition Workshops (CVPRW). , 2735-2745 (2022).
- Halabi, E. A., et al. Dual-activatable cell tracker for controlled and prolonged single-cell labeling). ACS Chem. Biol. 15 (6), 1613-1620 (2020).
- Hannan, N. R. F., Segeritz, C. -. P., Touboul, T., Vallier, L. Production of hepatocyte-like cells from human pluripotent stem cells. Nat Protoc. 8 (2), 430-437 (2013).
- Zhang, H., Shen, M., Wu, J. C. Generation of quiescent cardiac fibroblasts derived from human induced pluripotent stem cells. Methods Mol Biol. 2454, 109-115 (2022).
- Harding, M. J., McGraw, H. F., Nechiporuk, A. The roles and regulation of multicellular rosette structures during morphogenesis. Development. 141 (13), 2549-2558 (2014).
- Najm, F. J., et al. Rapid and robust generation of functional oligodendrocyte progenitor cells from epiblast stem cells. Nat Methods. 8 (11), 957-962 (2011).
- Hogrebe, N. J., Maxwell, K. G., Augsornworawat, P., Millman, J. R. Generation of insulin-producing pancreatic β cells from multiple human stem cell lines. Nat Protoc. 16 (9), 4109-4143 (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır