Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Çok Hücreli Sferoidlerin Üretimi ve Çok Parametreli Canlı Hücre Floresan Ömür Boyu Görüntüleme Mikroskobu (FLIM)
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Burada, takip çok parametreli canlı hücre mikroskobu gerçekleştirmek için farklı çok hücreli sferoid oluşturma yöntemlerini açıklıyoruz. Floresan ömür boyu görüntüleme mikroskobu (FLIM), hücresel otofloresan, boyama boyaları ve nanopartiküller kullanılarak, canlı üç boyutlu (3D) kanser ve kök hücre türevli sferoidlerde hücre metabolizması, hipoksi ve hücre ölümünün analizi için yaklaşım gösterilmiştir.
Özet
Çok hücreli tümör sferoidleri, tümör mikroçevresini yeniden üretmek, ilaç tedavilerini test etmek ve optimize etmek ve biyo ve nanosensörleri 3D bağlamda kullanmak için popüler bir 3D doku mikroagregat modelidir. Üretim kolaylığı, öngörülebilir boyutları, büyümeleri ve gözlemlenen besin ve metabolit gradyanları, 3B niş benzeri hücre mikro ortamını özetlemek için önemlidir. Bununla birlikte, sferoid heterojenliği ve üretim yöntemlerinin değişkenliği, genel hücre metabolizmasını, canlılığını ve ilaç yanıtını etkileyebilir. Bu, kök ve kanser hücresi biyolojisinde in vitro 3D doku modellerinde olduğu gibi boyut, değişkenlik, biyofabrikasyon ihtiyaçları ve kullanım gereksinimleri göz önüne alındığında en uygun metodolojiyi seçmeyi zorlaştırır. Özellikle, sferoid üretimi, optik metabolik görüntüleme, floresan ömür boyu görüntüleme mikroskobu (FLIM), nanosensörlerle küresel hipoksinin izlenmesi veya canlılık gibi kantitatif canlı mikroskoplarla uyumluluklarını etkileyebilir. Burada, canlı geniş alan, konfokal ve iki fotonlu mikroskoplarla uyumluluklarını vurgulayan bir dizi geleneksel sferoid oluşum protokolü sunulmaktadır. Çoğullanmış otofloresan FLIM ile ve çeşitli kanser türleri ve kök hücre sferoidleri kullanılarak analiz hattına yönelik takip görüntülemesi de sunulmaktadır.
Giriş
Çok hücreli sferoidler, hücrelerin kendi kendine toplanmasıyla elde edilen ve küresel bir şekil sergileyen bir grup 3B doku modelini temsil eder. Hücre-hücre ve hücre-matris etkileşimini in vitro olarak taklit etmek ve çok sayıda kanser ve kök hücre türevi yapı içinde 3B bir bağlamı yeniden üretmek için yaygın olarak kullanılırlar. Hücre bağlanmasını azaltmak ve agregasyonu teşvik etmek için çeşitli teknikler kullanılır. Bunlar, yüzey gerilimine1 dayanan asılı-damla yöntemini; ultra düşük bağlantı plakaları, mikro kalıplar ve mikro kuyular gibi hücre bağlanma itme yöntemleri 2,3; akustik dalga tabanlı yaklaşım4; akış kaynaklı toplama yöntemleri (eğirme şişeleri, biyoreaktör ve mikroakışkan cihazlar)5; manyetik parçacık destekli oluşum6 ve toplamayı teşvik eden sentetik ve ECM bazlı matrislerin ve iskelelerin kullanımı 7,8,9.
Kanser araştırmaları, geliştirilmesi ve yeni ilaç tedavilerinin doğrulanmasında, sferoidler, besinlerin, atık ürünlerin ve O2'nin mekansal difüzyonla sınırlı gradyanlarını özetleme yetenekleri nedeniyle çekici bir modeldir ve genellikle katı tümörler için tipik olan nekrotik bir çekirdek oluşumuna yol açar10,11. Bu daha güvenilir ve sofistike in vitro modeller, hayvan araştırmalarının 3R ilkesine (değiştirme, azaltma ve iyileştirme) göre hayvan modellerinin (Gıda ve İlaç İdaresi [FDA] Modernizasyon Yasası 2.012) kapsamlı kullanımına duyulan ihtiyacı sorgulamaktadır. Kansere ek olarak, sferoidler uygulamalarını kök hücre araştırmalarında bulurlar. Örneğin, pluripotent kök hücreler, indüklenmiş pluripotent kök hücrelerin (iPSC'ler) nöral öncü hücreler13 veya yumurtalık granüloza hücreleri13,14 gibi doğrudan hastalardan elde edilmesi zor olan özel hücre tiplerine farklılaşması için kullanılabilen embriyoid cisimler (EB) oluşturma kapasitesine sahiptir. Ayrıca, bir EB oluşumu genellikle nöral15, retinal16, kardiyak17, karaciğer18, mide19 ve bağırsak organoidleri20 gibi daha karmaşık organoid modellerin geliştirilmesinde ilk adımdır. Deneyler için uygun bir sferoid oluşturma yöntemi seçerken boyut, tekrarlanabilirlik, verim ve aşağı akış uygulamaları gibi faktörler göz önünde bulundurulmalıdır.
3B kültürün artan karmaşıklığı, 2B kültüre kıyasla daha yüksek değişkenliğe yol açabilir. Besin bileşimi21, ortam buharlaşması22, viskozite23, pH kontrolü24, sferoid oluşum yöntemi ve hatta kültürdeki süre 25,26 gibi faktörler, değişen morfoloji, boyut, canlılık ve farklı kemorezistanslı sferoidlerin elde edilmesine neden olabilir27,28. Son araştırmalar, küresel oksijen gradyanlarının her zaman statik olmadığını ve küresel heterojenliği etkileyen oluşum yöntemi, küresel boyut ve hücre dışı viskoziteden etkilendiğini göstermiştir29. Sferoidlerde tekrarlanabilirliği ve veri erişilebilirliğini iyileştirmek için, MISpheroID bilgi tabanıgeliştirilmiştir 26 ve tekrarlanabilir bir sonuç için minimum bilgi olarak hücre hattını, kültür ortamını, oluşum yöntemini ve sferoid boyutunu belirler. Bu nedenle, kuyu boyutu (maksimum sferoid boyutunun bir tahmini verilen), kullanılan sarf malzemeleri, hazırlama süresi ve sferoidleri mikroskopi kaplarına taşımadan izleme olasılığı dahil olmak üzere çoklu yüksek verimli (SphericalPlate 5D, laboratuvar yapımı mikro kalıplar ve Mikrodoku kalıpları) ve düşük bağlanma yöntemleri (yani, Biofloat ve Lipidure kaplı 96 kuyulu plakalar, hem iskelesiz hem de iskele tabanlı) arasında ayrıntılı bir karşılaştırma yapılmıştır (Şekil 1 ve Tablo 1). İkincisi, uzun vadeli çalışmalara olanak tanırken, yüksek verimli yöntemlerle üretilen sferoidler genellikle son nokta deneyleriyle sonuçlanır. 5DspheriPlate'in ızgaraları dışındaki tüm yöntemler istenmeyen otofloresan getirmez, bu nedenle mikroskopide doğrudan kullanımlarını sağlar.
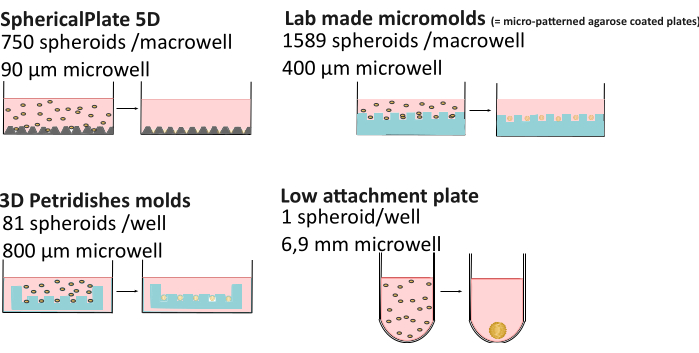
Şekil 1: Sferoid oluşum yöntemleri açıklanmıştır. Plakaya patentli mikro kuyuları entegre eden SphericalPlate 5D gibi yüksek verimli yöntemler, laboratuvarda üretilen mikro kalıplar ve MicroTissue kalıpları, agarozda (mavi) birden fazla mikro kuyu yapmak için damgalar kullanır. Lipidure (Amsbio) ve Biofloat (Sarstedt) gibi düşük bağlantılı plakalar, hücre yüzeyi yapışmasını engelleyen ve hücrenin kendi kendine toplanmasını destekleyen yapışmayan bir kaplama kullanır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| 5D Küresel Plaka | Kendi ürettiği mikro kalıplar | Mikro doku | Düşük bağlanma yöntemleri | |
| Sferoid/kuyu sayısı | 750 | 1589 | 81 | 1 |
| Çap kuyusu | 90 μm | 400 μm | 800 μm | 1 Milimetre |
| Kültür hacmi | 1 mL | 5 mL | 1 mL | 200 μL |
| Diğer sarf malzemeleri | / | 7 mL% 3 agaroz | 500 μL% 2 agaroz | / * |
| Hazırlanma zamanı | 10 dk | 2 saat + 3 gün medya adaptasyonu | 0,5 saat + 15 dk medya adaptasyonu | 10–30 dk + 1 saat kurutma |
| Izleme | Evet | Hayır** | Evet | Evet |
| Otofloresan | Evet | Hayır | Hayır | Hayır |
| Yeniden | Hayır | Evet | Evet | Hayır** |
| Masraf | €€ | € | €€€€ | €€€€: Kaplama ve Matrigel |
| €€: Ticari 96 kuyulu plaka | ||||
| *Bazı hücre hatları, kompakt sferoidler oluşturmak için ECM (yani %2-%5 Matrigel) ilavesine ihtiyaç duyar. | ||||
| **Kaplama tükenene kadar tekrar kullanılabilir. Bununla birlikte, her plaka az miktarda ortam tüketir ve zamanla toz birikebilir. Filtre sterilizasyonuna düzenli olarak ihtiyaç duyulur. | ||||
Tablo 1: Çoklu sferoid oluşturma yöntemlerinin karşılaştırılması29. "İzleme": bir mikroskopi çanağına aktarmaya gerek kalmadan sferoidi izleme yeteneği. €: 0-50€, €€: 50-150€ , €€€: 150-500€ , €€€€: >500€
Floresan mikroskobu, hücre ölümü, canlılık, proliferasyon, metabolizma, viskozite ve hatta mekanik özellikler dahil olmak üzere sferoidler içindeki temel biyolojik yönlerin doğrudan izlenmesini sağlar30. Floresan ömür boyu görüntüleme mikroskobu (FLIM), (mikro) ortamlarında 31,32,33,34 floresan prob etkileşimlerini incelemek için ek bir nicel boyut sağlar ve farklı emisyon ömürlerine göre örtüşen emisyon spektrumlarının çözülmesine izin verir 35,36 ve içsel hücresel otofloresansa dayalı hücre metabolizmasının araştırılması. Bu nedenle, nikotinamid adenin dinükleotid fosfat (NAD (P) H), flavin mononükleotid (FMN), flavin adenin dinükleotid (FAD), protoporfirin IX ve diğerleri gibi yaygın hücresel otofloresan bileşikler bir ve iki foton FLIM ile ölçülebilir ve glikoz katabolizması, oksidatif fosforilasyon (OxPhos) ve hücre redoks durumuna genel bir bakış sağlar. NAD(P)H serbest sitoplazmik veya proteine bağlı mitokondriyal formlardabulunur 37,38. Benzer şekilde, FAD'ın oksitlenmiş hali, serbest formun daha uzun bir ömrüne sahip floresandır. NAD(P)H ve FAD mikroskopileri genellikle numune fotohasarını önlemeyi amaçlayan iki foton uyarılmış FLIM'i içerir39. Sıklıkla, 'optik metabolik görüntüleme' FLIM, sferoid veya organoid metabolizması, oksijenasyon, proliferasyon ve hücre canlılığının daha eksiksiz bir resmini sağlamak için boya bazlı problar, genetik olarak kodlanmış biyosensörler, fosforesans ömür boyu görüntüleme mikroskobu (PLIM) ve oransal yoğunluğa dayalı ölçümlerin kullanımıyla birleştirilebilir 29,30,31. Ek olarak, FLIM, bir ilacın hedef alanı 33,40,41 ile bağlanmasını araştırmak için alıcı ile yakın temas halindeyken donör floroforun ömür boyu değişimini ölçmek için Förster rezonans enerji transferi (FRET) yöntemi ile de birleştirilebilir.
Elde edilen FLIM görüntüleri, kullanım ömrünü piksel piksel hesaplamak için tipik olarak analiz edilir. Şu anda, floresan ömrünü elde etmek için kullanılan en az 3 yaygın strateji vardır: yarı kantitatif 'hızlı FLIM'42 (bazen 'tau sense'43,44 olarak anılır), bir, iki veya üç üstel uydurma kullanarak bozunma eğrisi uydurma ve fazör dönüşümü ve fazör grafiği analizi ile 'uydurmasız' yaklaşım. Satıcıya bağlı olarak, ölçülen FLIM verilerini işlemek için sağlanan (LAS X, Symphotime, SPCImage, vb.) veya açık kaynaklı yazılım (örneğin, FLIMfit45, FLIMJ46 veya diğerleri47) kullanılabilir. Tipik olarak, satıcı tarafından sağlanan yazılım, ön veri analizi için yararlıdır, açık kaynaklı çözümler, örneğin fazör grafikleri ve 3D görselleştirme kullanarak daha doğru çalışmalar sağlayabilir.
Sferoidleri incelemek için bir yöntem olarak FLIM'in kullanışlılığına ve çekiciliğine rağmen, çok az deneysel protokol mevcuttur ve FLIM'i içeren başarılı canlı multiparametrik mikroskopi deneyleri için en uygun oluşum yönteminin seçiminde genel bir bilgi eksikliği vardır. Burada, yaygın olarak kullanılan sferoid oluşum protokollerinin ayrıntılı bir karşılaştırması, yakın zamanda doğrulanmış ve karakterize edilmiş uzak kırmızı ve yakın kızılötesi (NIR) oksijen algılayıcı nanosensör (MMIR1) ile morfolojilerine, canlılıklarına ve oksijenasyonlarına dayalı olarak sunulmaktadır. Katyonik nanoparçacık, referans O2 duyarsız aza-BODIPY (uyarma 650 nm, emisyon 675 nm) ve NIR O2 duyarlı metaloporfirin, PtTPTBPF (uyarma 620 nm, emisyon 760 nm) olmak üzere iki raportör boya ile emprenye edilir. MMIR1, hücresel toksisite oluşturmadan ve kararlı sinyallere, uzun süreli izlemeye ve çoğullamaya izin vermeden geleneksel bir floresan mikroskobu (oransal analiz kullanılarak) veya fosforesans ömür mikroskobu (PLIM) üzerinde oksijen gradyanlarının gerçek zamanlı analizini sağlar25,29. Boyalar veya nanosensörler ile boyama ihtiyacına, sferoid verimine veya hücre tipine bağlı olarak en uygun oluşum protokolü seçilebilir. Sferoidlerin canlılığı ve oksijenasyonu ile ilgili çalışmalar, kanser ve kök hücre kaynaklı sferoidler ile ilgili olduğundan, sunulan protokoller ayrıca bu modellerle NAD(P)H-FLIM ve FAD-FLIM örneklerini ve beklenen tipik sonuçlarını da içermektedir. Sunulan görüntüleme ve analiz boru hatları, en popüler zamanla ilişkili tek foton sayma tabanlı FLIM mikroskopi platformlarını hedeflemektedir.
Protokol
1. Çok hücreli sferoidlerin oluşumu
- Hücre kültürü
NOT: Hücre kültürleri, Amerikan Tipi Kültür Koleksiyonu (ATCC), Lonza, Sigma-Aldrich veya diğer satıcılardan toplanabilir. ATCC, tercih edilen büyüme ortamı, alt kültürleme prosedürleri, biyogüvenlik seviyesi, büyüme oranı ve STR profilleri dahil olmak üzere gerekli tüm kullanım bilgilerini sağlar. Burada, McCoy'un 5A ortamında (VWR, 392-0420) %10 FBS ve 1 mM Sodyum Piruvat ile desteklenmiş 500 hücre / sferoid insan kolon kanseri hücre hattı HCT116 kullanılır. Günlük olarak izlenen uzun süreli deneyler için ortama 10 mM HEPES, pH 7.2 eklenebilir.- % 70 -% 90 birleşmeye ulaşmak için hücre kültürünü büyütün.
- Hücreleri önceden ısıtılmış (37 °C) steril PBS (25 cm2 için 5 mL veya 75 cm2 şişe başına 10 mL) ile durulayın.
- % 0.05 tripsin - 1 mM EDTA çözeltisi (25 cm² için 0.5 mL veya 75 cm2 şişe başına 1 mL) ekleyin ve% 5 CO2,% 95 nemde 37 ° C'de 5-10 dakika inkübe edin hücre ayrılmasına ulaşmak için.
NOT: Transmisyon ışığı (parlak alan) mikroskobu altında kontrol hücresi ayrılması. Hücrelerin ayrışma enzim çözeltisine aşırı maruz kalması, canlılıklarını etkileyebilir. - % 10 FBS (1 mL ayrışma çözeltisi başına en az 5 mL ortam) içeren fazla hücre kültürü ortamı ekleyerek tripsini nötralize edin.
NOT: Düşük FBS veya FBS içermeyen kültür ortamında kültürlenen hücreler için, tripsin ile muamele edilen hücre kültürüne 0.5 mL %100 FBS ilavesi ve ardından hücreleri orijinal kültür ortamlarına aktarmak için santrifüjleme ile tripsin nötralizasyonu yapılabilir. - Ortamda tek hücreli süspansiyon elde etmek için pipetleme yoluyla hücre agregalarını ayırın.
NOT: Üstte 1000 μL pipet ucu bulunan serolojik bir pipetle pipetleme, büyük hacimli süspansiyon hücre kültüründe tek hücreli süspansiyon oluşumunu büyük ölçüde iyileştirir. - Hücre süspansiyonunun 1 mL'si başına hücre sayısını saymak için bir sayma odası (Neubauer tarafından yönetilen hemositometre veya alternatifleri) kullanın.
- Mililitre başına istenen hücre sayısını elde etmek için hücre süspansiyonunu seyreltin.
- O2'nin oransal analizi için hücre süspansiyonuna 10 μg / mL'lik bir nihai konsantrasyonda konsantre O2 probu (nanopartiküller) çözeltisi ekleyin.
NOT: Homojen hücre süspansiyonunu sağlamak için (prob ile), sferoid oluşumundan önce birkaç kez yeniden süspanse edin. O2 probu gerekli değilse, bu adımı atlayın ve sferoidlerin oluşumuna devam edin. iPSC'ler işlenirken değiştirilmiş bir protokol uygulanmalıdır. Kısaca, iPSC'ler Geltrex kaplı plakalar üzerinde koloniler halinde büyütülür ve satıcı tarafından sağlanan protokol48'de açıklandığı gibi ReLeSR kullanılarak geçirilir. Küresel oluşum gününde, koloniler büyük, kompakt olmalı ve belirgin sınırlara sahip çok katmanlı merkezler sergilemelidir. Hücreleri önceden ısıtılmış steril PBS ile durulayın. 1 mL yumuşak hücre ayrışma reaktifi (GCDR) ekleyin ve 37 ° C'de 8-10 dakika inkübe edin. Hücreleri kuyudan nazikçe ayırmak ve tek hücreli bir süspansiyon elde etmek için 1000 μL'lik bir uç kullanın. Tek hücreli süspansiyonu steril 50 mL'lik bir konik tüpe aktarın ve GCDR'yi nötralize etmek için 4 mL önceden ısıtılmış DMEM-F12 ortamı ekleyin. İyiliği 1 mL DMEM-F12 ile yıkayın ve hücre süspansiyonunun geri kalanına aktarın. 300 x g'da 5 dakika santrifüjleyin. Daha fazla deney için 1 mL uygun ortamda yeniden süspanse edin. Bu yazıda anlatılan deneyler için mTeSR + 10 μM Kaya İnhibitörü kullanılmıştır. Mililitre başına istenen hücre sayısını elde etmek için hücre süspansiyonunu sayın ve seyreltin.
- Sferoid oluşum yöntemleri
- 3D Petri kabı mikro kalıpları
NOT: Bu yüksek verimli yöntem, 800 μm çap ve 800 μm derinliğe sahip 9 x 9 mikro kalıp dizisinde aynı anda çok sayıda sferoid (81 sferoid) oluşturmak için kullanılır.- 3D Petri kaplarını dH 2O'da dökmek için mikrokalıpları durulayın ve otoklavlanabilir bir kaba koyun.
- 2 g elektroforez dereceli agaroz tozu ölçün ve 200 mL'lik kuru bir otoklav güvenli cam şişeye koyun.
NOT: Şişenin ve agaroz tozunun sıvı veya nem olmadan kuru olduğundan emin olun. - 3D Petri kaplarını ve şişeleri agaroz tozu ile kuru bir döngüde 30 dakika boyunca dökmek için otoklav mikro kalıpları.
- 100 mL ultra saf suya 0,9 g NaCl ekleyerek %0,9 w/v'lik bir tuzlu su çözeltisi hazırlayın ve otoklavlama ile sterilize edin.
NOT: NaCl üretici tarafından tavsiye edilir. Agaroz stabilitesini arttırır. - Sterilize edilmiş agaroz tozuna steril salin ekleyerek agaroz çözeltisini hazırlayın. Basınç oluşumunu önlemek için gevşek bir şekilde takılan kapağı vidalayın. Agaroz tozunu karıştırmak için şişeyi döndürün.
- Agaroz tozunu bir mikrodalga fırın kullanarak kaynatın ve çözün. Mikrodalgayı sık sık durdurun (~ her 10 saniyede bir). Şişeyi döndürün ve agaroz eriyene kadar tekrarlayın.
DİKKAT: Agaroz çözeltisi sıcaktır ve dikkatli kullanım gerektirir. Eritme işleminden hemen sonra çalkalama, çözeltinin kaptan dışarı çıkmasına neden olabilir. Kazaları önlemek için, kapasitenin %50'sinden fazla olmayan yeterince büyük kaplar kullanın ve uygun kişisel koruma (eldiven, fırın eldiveni, göz koruması ve laboratuvar önlüğü) kullanın. - Çözünmüş agaroz çözeltisini 60-70 °C'ye kadar soğumaya bırakın. Aseptik teknikler ve koşullar kullanılarak, erimiş agarozu mikro kalıba pipetleyin (12 seri için 500 μL veya 24 seri için 330 μL).
NOT: Agarozu karıştırırken veya pipetlerken kabarcık oluşumunu önleyin. Agaroz katılaşmadan önce pipetleme veya hafifçe sıyırma yoluyla mikro kalıbın küçük parçalarında sıkışmış küçük kabarcıkları çıkarın. - Agarozun yaklaşık 2-3 dakika katılaşmasına izin verin. Daha sonra, 3D Petri kabını çıkarmak için mikro kalıbı dikkatlice esnetin ve 12 oyuklu bir doku kültürü plakasına aktarın.
NOT: Mikro kalıbın aşırı esnemesi, agaroz kalıbı içinde çatlaklar oluşmasına neden olabilir. - 3D Petri kabını dengelemek için 2,5 mL/kuyucuk hücre kültürü ortamı ekleyin. 15 dakika veya daha uzun süre kuluçkaya yatırın. Kültür ortamını çıkarın ve taze ortamla değiştirin. 3D Petri kabını bir kültür ortamı ile dengelemek için bir kez daha tekrarlayın.
NOT: Protokol, hücre tohumlanana kadar burada kesilebilir. Uzun süreli depolama için (4 ° C'de 2 haftaya kadar), orta yerine PBS çözeltisi kullanın. - Kültür ortamını (veya PBS'yi) çevreleyen 3D Petri kabını tamamen çıkarın ve doku kültürü plakasını eğerek 3D Petri kabının içindeki ortamı doğru bir şekilde çıkarın.
- 40.500 hücre içeren 190 μL hücre süspansiyonunu hücre tohumlama odasına damla damla dikkatlice tohumlayın (bkz. adım 1.1).
NOT: Agaroz damgalarındaki mikro kuyucukların sayısı, damga başına üretilen sferoid sayısını belirler. Bu durumda, bu agaroz küfü 81 mikrokuyu (81 x 500 hücre/sferoid) içerir. Bir makrokuyuya eklenen süspansiyondaki hücre konsantrasyonunun değişimi, sferoid başına hücre sayısının değiştirilmesine izin verir, böylece sferoidin boyutunu kontrol eder. - Hücrelerin 3D Petri dsh'nin özelliklerine yerleşmesi için ~ 10 dakika bekleyin. Ardından, 3D Petri kabının dışına 2 mL orta ekleyin.
- Doku kültürü plakasını hücre kültürü inkübatörüne yerleştirin ve 3D Petri Kabını çevreleyen ortamı gerektiği gibi değiştirin.
- Düşük bağlantı plakası
NOT: Bu yöntem, oyuk başına tek bir sferoid oluşturmak için kullanılır. Kaplamalı plakalar (Lipidure veya Biofloat) ticari olarak temin edilebilir (1.2.2.1-1.2.2.4 adımlarını atlayın). Alternatif olarak, kaplama ayrı olarak satın alınabilir ve işlenmemiş çok kuyulu plakaları kaplamak için kullanılabilir. Bu kuyucuklarda daha hızlı buharlaşma nedeniyle 96 oyuklu plakaların kenarlarındaki kuyuların steril su veya PBS ile doldurulması ve böylece sferoidler için kuyu sayısının 60 ile sınırlandırılması önerilir. Daha az sferoide ihtiyaç duyulması durumunda, çevredeki boş kuyuları su veya PBS ile doldurun. Küresel oluşumu engelledikleri için küçük parçacıkların kuyulara girmesini önlemek için sıvı işleme için tozsuz uçlar kullanın.- 0.25 g polimer tozunu bir cam şişe kabında 50 mL etanol içinde çözerek %0.5 w/v'lik bir kaplama çözeltisi hazırlayın. Kaplamayı filtreyle sterilize edin.
NOT: Filtre sterilizasyonu ve sonraki tüm adımlar, laminer akış altında steril koşullarda gerçekleştirilmelidir. - 96 U-tabanlı bir kültür plakasının her bir oyuğuna 200 μL kaplama çözeltisi ekleyin.
- 1 dakika inkübe edin ve fazla kaplamayı çıkarın.
NOT: Kaplama çözeltisi birden çok kez kullanılabilir. Oda sıcaklığında (RT) bir cam kapta saklayın. Plastik kaplar tavsiye edilmez, çünkü plastik kısmen çözülebilir ve çözeltinin bir parçası haline gelebilir. Toz varsa, filtre 0,22 μm polietersülfon (PES) veya naylon şırınga filtreleri ile sterilize edin. - 96 oyuklu plakayı yaklaşık 1 saat kurumaya bırakın.
NOT: Hücrelerin hücre dışı bir matrise ihtiyacı varsa, adım 1.2.3'e geçin. Kaplamalı plakalar alüminyuma sarıldığında 1 aya kadar RT'de saklanabilir. Daha az miktarda hücre/kuyu tohumlarken, plakayı 300 g'da 5 dakika santrifüjlemek hücrelerin aşağı çekilmesine yardımcı olabilir.
- 0.25 g polimer tozunu bir cam şişe kabında 50 mL etanol içinde çözerek %0.5 w/v'lik bir kaplama çözeltisi hazırlayın. Kaplamayı filtreyle sterilize edin.
- Hücre dışı matriks destekli oluşum protokolü
NOT: Bazı hücre hatları kendi başlarına yeterli hücre dışı matris (ECM) üretmezler ve kompakt sferoidler 49,50,51 oluşturmak için Matrigel, Cultrex veya Geltrex gibi ECM'nin eklenmesine ihtiyaç duyarlar. Meme kanseri MDA-MB-231, insan dermal papillası, prostat kanseri hücreleri ve diğerleri gibi hücre tipleri için, ECM'nin eklenmesini gerektiren aşağıdaki modifikasyonlar52 ile adım 1.2.2'yi kullanmak mümkündür. Tozun küresel oluşumuna müdahale etmesini önlemek için tercihen sıvı kullanımı için tozsuz uçlar kullanın. Adım 1 ve 4-7, bir biyolojik güvenlik kabini (sınıf II) altında gerçekleştirilmelidir.- Yüzey işlemi için 1.2.2.1-1.2.2.4 (alçak bağlantı plakaları) adımlarıyla devam edin.
- 96 oyuklu plakayı buzdolabında 4 °C'de önceden soğutun.
- Santrifüjü 96 oyuklu plaka için doğru adaptörle hazırlayın ve 4 °C'ye kadar soğutun.
- Önceden soğutulmuş (4 °C) hücre kültürü ortamında %5'lik bir bazal membran matrisi (BMM) çözeltisi hazırlayın.
NOT: BMM, RT'de hızlı bir şekilde çapraz bağlanır. Kullanım sırasında stokları ve çözeltileri buz üzerinde tutun. - Hücre süspansiyonunu hazırlayın (bkz. adım 1.1).
- Her kuyucuğa 50 μL BMM çözeltisi ekleyin.
- BMM çözeltisinin (25.000 hücre / kuyucuk) üzerine her bir oyuğa yavaşça 50 μL hücre süspansiyonu ekleyin.
NOT: Bu hacmi kuyuya patlatmayın, aksi takdirde hücreler kuyuların kenarlarına yayılır ve dipte toplanmaz. Hücre yoğunluğu buna göre ayarlanarak oyuk başına daha az sayıda hücre elde edilebilir. Tüm hücreler tüm tohumlama yoğunluklarında sferoidler oluşturmaz; Optimizasyon, hücre tipi ve istenen boyut açısından gerçekleştirilmelidir. Sağlanan hacimlerle, BMM'nin nihai konsantrasyonu% 2.5'tir. Farklı bir konsantrasyona ihtiyaç duyulursa, stok çözeltisi daha düşük/daha yüksek bir konsantrasyonda hazırlanmalıdır. - 96 oyuklu plakayı 300 x g ve 4 °C'de 5 dakika santrifüjleyin.
NOT: Bu adım olmadan, hücreler kuyunun dibinde düzgün bir şekilde toplanmaz ve çok sayıda küçük agreganın oluşmasına neden olur. Bu aşamada çapraz bağlanmayı önlemek için santrifüjün soğutulması gerekir. - Plakayı hücre kültürü inkübatörüne yerleştirin. Agregalar tohumlamadan sonraki 4. günde olgun kabul edilir.
NOT: Küresel oluşumu ile ilgili ek protokoller için Ek Dosya 1'e bakın.
- 3D Petri kabı mikro kalıpları
2. Sferoidlerin canlı mikroskobu
- Canlı görüntüleme analizi için sferoidlerin hazırlanması
NOT: Deney tasarımına bağlı olarak (örneğin, uzun süreli izleme veya son nokta analizi, mikroskop kurulumu veya ölçülen floresanın spektral özellikleri) veya küresel üretim yönteminin mikroskopi ile uyumsuzluğuna bağlı olarak (örneğin, numune kalınlığı, malzemenin otofloresansı, görüntüleme sırasında sferoidlerin yüzdürülmesi) sferoidlerin üretildikleri plakada doğrudan izlenmesi, mümkün olmayabilir. Protokol, çoğu ters çevrilmiş geniş alan ve konfokal mikroskoplar için uygun olan görüntüleme için sferoidlerin hazırlanmasını açıklar.- Görüntüleme ortamını hazırlayın ve önceden ısıtın (37 °C): HEPES-Na, pH 7.2 (10 mM), sodyum piruvat (1 mM), L-glutamin (2 mM) ve glikoz (5 mM) ile desteklenmiş DMEM, fenol kırmızısı içermez.
NOT: Görüntüleme sırasında CO2 kontrolü sağlanmışsa, sodyum bikarbonat tek başına veya HEPES-Na ile kombinasyon halinde kullanılabilir24. Bazı hücre kültürü tipleri HEPES'in varlığını tolere edemez. Deneysel tasarıma bağlı olarak, piruvat, glutamin ve glikoz içeriği değiştirilebilir. - Kaplanmış (güçlü bir küresel yapışma için) veya kaplamasız (düşük küresel yapışma) kaplamalı (kalınlık # 1.5) steril mikroskopi kapları (ticari olarak temin edilebilir veya laboratuvar yapımı) hazırlayın.
NOT: Kaplama ihtiyacı ve türü, hücre tipine, sferoidlerin yapışma özelliklerine ve 3B'den 2B kültür arayüzüne hücre göçünün hızına bağlıdır. Kaplama, 3D organizasyonun kaybını kolaylaştırabileceği, sferoidlerdeki mikro gradyanların şeklini değiştirebileceği ve sonuç olarak hücre davranışını değiştirebileceği için bunun dikkate alınması önemlidir. Bazı deneyler için (örneğin, ilaç stimülasyonuna yanıtı analiz eden görüntüleme), yüzeye güçlü bir küresel yapışma gereklidir ve jelatin, BMM, kollajen, kollajen / poli-D-lizin veya poli-D-lizin ile bir kaplama tercih edilir. - O2 probu ile boyanmış sferoidleri mikro desenli agaroz veya 96 oyuklu plakanın mikro oyuklarından nazikçe yıkayın ve hala yüzen sferoidleri 2 mL'lik bir şişeye aktarın.
NOT: Yüksek verimli yöntemden tüm sferoidlerin toplanmasını sağlamak için, tüm sferoid süspansiyonları tek bir şişede birleştirerek kalıbı ek hacimde kültür ortamı ile 1-3 kez durulayın. Düşük bir bağlantı plakasındaki sferoidler için, deney için az sayıda sferoid yeterliyse, sferoidleri tek tek oyuklardan bir şişeye veya doğrudan bir mikroskopi kabına toplayın. Büyük sferoidleri aktarırken, pipetleme sırasında hasar görmemesi için pipet ucunun ucunu kesin. - Sferoidlerin şişenin dibine yerleşmesine ve görünür bir topak oluşturmasına izin vermek için şişeyi 5 dakikaya kadar dikey konumda bırakın.
- Ortamı tüpten çıkarın, sferoidleri rahatsız etmeyin ve yeterli miktarda taze kültürde nazikçe yeniden süspanse edin.
NOT: Sferoid kullanımının rahatlığı için, sferoidlerin görüntüleme ortamına aktarılması bu aşamada küçük partiler halinde de yapılabilir; 2.1.7 ve 2.1.8 adımlarına bakın. Farklı deney gruplarından gelen sferoidler, ortam bileşimi ve ortam ön koşullandırma süresi metabolizmalarını etkileyebileceğinden, eşit şekilde ele alınmalıdır. - Sferoidler yüzerken, mikroskopi çanağının oyuğu başına eşit hacimde küresel süspansiyon aktarın.
- Mikroskopi kabının/kuyusunun yüzeyine yapışmalarını sağlamak için sferoidleri bir CO 2 inkübatörde 37 ° C'de1-2 saat inkübe edin. Görüntüleme için adım 2.1.9 ile devam edin. Sferoidlerin ek problarla boyanması için adım 2.1.8 ile devam edin.
NOT: 3B sferoidden 2B yüzey arayüzüne hücre göçünün hızı, zamanın bir fonksiyonudur. 3D organizasyon kaybını önlemek için, inkübasyon süresi hücre tipine, yüzey kaplama tipine ve deneyin tasarımına göre optimize edilmelidir. Örneğin, HCT116, küresel boyuta bağlı olarak, kollajen IV / poli-D-lizin kaplı yüzeye uygun bir küresel bağlantı için en az 2 saate ihtiyaç duyarken, hDPSC bağlantısı ve 2D arayüze geçiş son derece hızlıdır ve 1-2 saat içinde 3D organizasyonun kaybına yol açar. Aşırı yayılma nedeniyle '2D sferoidlerin' görüntülenmesini önlemek için, azaltılmış inkübasyon süresi ile kaplanmamış cam yüzeyler kullanılır. - Bilinen bir küresel süspansiyon hacmine önerilen veya ampirik olarak optimize edilmiş konsantrasyonlarda floresan prob(lar)ı ekleyin. Görüntülemeden önce 37 ° C CO2 inkübatöründe 1 saat inkübe edin.
NOT: Canlı / ölü tahlil için, standart nihai konsantrasyon 1 μg / mL'de propidyum iyodür ve Calcein Green-kullanın. Probidyum iyodür boyamanın iPSC embriyoid cisimleri üzerindeki toksik etkisini önlemek için, nihai propidyum iyodür konsantrasyonu 0.5 μg/mL idi. Büyük küresel boyut nedeniyle probun difüzyonu verimli değilse, prob yükleme süresi uzayabilir. Yükleme süresi her zaman sferoidin yüzeye bağlanması için gereken toplam inkübasyon süresinin bir parçası olarak düşünülmelidir. Eğer sferoid tutunması için daha uzun bir süreye ihtiyaç duyuluyorsa boyama işlemi bu sürenin sonunda düzenlenmelidir. Daha uzun kuluçka süresinin 3D organizasyon kaybına yol açabileceğini unutmayın. - Hücre kültürü ortamını veya floresan probları içeren ortamı çıkarın ve gerekli miktarda görüntüleme ortamı ile değiştirin. Küresel görüntüleme yolunda ortam floresan arka planı olmadığından emin olmak için, ortam değiştirme (yıkama) adımını 5 defaya kadar tekrarlayın.
NOT: Ortam değişimi sırasında sferoidin çıkarılmasını önlemek için, ortamın mikroskopi kaplarının kenarlarından 200 μL'lik bir pipetle dikkatlice aspire edilmesi ve ortamın eklenmesinin mikroskopi kabında duvardan veya yana doğru yapılması önerilir. - Hemen görüntüleme protokolünün 2.2.1 adımına geçin.
NOT: Görüntülemeye hazırlık ile gerçek görüntüleme edinimi arasında çok uzun bir ara, hücre metabolizmasını (örneğin, değişen ortam bileşimi yoluyla), canlılığı (son nokta analizi için kullanılan bazı floresan problar, uzun bir inkübasyon süresinden sonra hücre ölümünü uyarabilen toksik etkilere sahiptir) etkileyebilir ve ayrıca 3D organizasyon kaybına yol açabilir. Birden fazla sferoid grubunun veya deneysel koşulların karşılaştırılması gerekiyorsa, tedavi, ön koşullandırma ve görüntüleme prosedürlerinin zamanlamasını analiz edilen gruplar arasında mümkün olduğunca eşit tutmak için deneysel tasarım buna göre geliştirilmelidir.
- Görüntüleme ortamını hazırlayın ve önceden ısıtın (37 °C): HEPES-Na, pH 7.2 (10 mM), sodyum piruvat (1 mM), L-glutamin (2 mM) ve glikoz (5 mM) ile desteklenmiş DMEM, fenol kırmızısı içermez.
- Görüntü elde etme
NOT: Protokol, Stellaris 8 Falcon (Leica) konfokal mikroskobu ve Leica Application Suite X (LAS X) yazılım sürümü 4.7 kullanılarak canlı sferoidlerin multiparametrik görüntülemesini açıklar. Bununla birlikte, alternatif mikroskopi platformlarında böyle bir analiz yapmak için sadece küçük değişiklikler gerekecektir.- Görüntülemeden 30-60 dakika önce sıcaklık klima kontrol ünitesini açın. Gerekli havalandırma hızını ve sıcaklığı (35-37 °C) ayarlayın.
NOT: Sıcaklık kontrolüne ek olarak, görüntüleme sırasında gaz konsantrasyonunun (örn. CO2 veya O2) kontrol edilmesi gerekiyorsa, görüntülemeden önce gerekli koşullara ulaşmak için ilgili cihazlar da önceden başlatılmalıdır. - Mikroskobu ve bağlı cihazları açın (yani, WLL lazer, bilgisayar, suya daldırma hedefi için su pompası ve diğer çalışan elektronik bloklar). Tam mikroskop kurulumuyla birlikte verilen mikroskop kontrol yazılımını (örneğin, LAS X Makine Modu veya Çevresel Kontrollü Makine Modu) başlatın ve aşama kalibrasyonunu başlatın.
- Yazılımda gerekli hedefi seçin ve gerekirse daldırma sıvısını uygulayın.
NOT: Canlı mikroskopi için, yeterli ('uzun') çalışma mesafesine sahip su veya gliserol daldırma hedeflerinin kullanılması önerilir, ör., HC Fluotar L 25x/0.95 W VIS IR (2.4 mm çalışma mesafesi), HC PL Apo 40x/1.25 GLYC (0.35 mm çalışma mesafesi) veya hava hedefleri için en az NA = 0.4 veya daha yüksek. Büyütme ve çalışma mesafesi seçimi, görüntülenen numunenin doğasına ve boyutuna ve ölçülen floresan sinyallerine bağlıdır (parlaklık, kuantum verimi, boyama verimliliği, bkz. örneğin, boyalar ve nanopartiküller üzerine tartışma53). Büyük nesneler (sferoidler veya organoidler, >500 μm boyutunda), 'biyo-reaktör' veya mikroakışkan çipler, uzun çalışma mesafesi hedefleri ve daha düşük büyütme gerektirirken, tek tek hücrelerin veya hücre organellerinin analizi, genellikle 'mozaik' görüntüleme yoluyla elde edilen yüksek büyütme gerektirir. - Sahnede sferoidlerle mikroskopi çanağını kurun. Odağı ayarlayın ve bir nesne/ilgi alanı (ROI) bulun.
NOT: Küçük, zayıf floresan, zayıf kontrastlı veya nadir nesneler bulunması gerekiyorsa ve odağı bulmak zorsa, mikroskopi çanağının duvarlarına önceden odaklanma yapılması ve kuyunun köşelerinden birinden başlayarak ilgilenilen nesne için yüzeyin serpantin ile 'taranması' önerilir. - Proje Aç penceresini seçin ve ilgili simgeye tıklayın, Yeni Proje Oluştur. Araştırma projesi dosyasına standart bir ad verin (örneğin, "YY-AA-DD+ açıklaması" ile başlayarak). Görüntüleme sırasında, üretilen tüm görüntüler otomatik olarak oluşturulan proje .lif dosyasına kaydedilecektir.
- Edinme penceresini açın. Ölçülen floresanın bilinen spektral özelliklerine (uyarma/absorbans ve emisyon spektrumları) dayalı olarak beyaz ışık lazer (WLL) uyarma dalga boyunu ve gerekli hibrit veya rezonans tarama dedektörleri (HyD S, HyD X veya HyD R tipi) aralığını ayarlayın. Tarama için Çizgi veya Çerçeve türlerini seçin.
NOT: Piyasada bulunan floresan boyaların çoğu için, spektral özellikler LAS X Dye Assistant paketinde bulunabilir (veya eklenebilir). Prob spektral özellikleriyle uyumlu ve FLIM durumunda foton sayımı ile uyumlu (yani HyD X veya HyD R) uygun spektrum hassasiyet aralığına sahip dedektörü seçin. Multiparametrik görüntüleme için WLL'yi birden fazla uyarma pozisyonuna ayarlayın (örneğin, FAD/Flavins'in ve oransal MMIR1 O2 probunun iki floresan kanalının eşzamanlı görüntülenmesi için - referans ve hassas, uyarma/emisyon ayarları 460 nm/510-590 nm HyD X1 ve 614 nm/631-690 HyD X3 ve 724-800 nm HyD R olabilir. Dedektörler farklı spektral hassasiyete sahip olabileceğinden, emisyonu toplamak için uygun dedektörü atamak önemlidir54. - (FLIM için isteğe bağlı) Edinme penceresinde, foton sayımı (bozunma toplama) ile birlikte görüntüleme gerçekleştirmek için FLIM modunu seçin. Hemen, FLIM verilerinde gezinmek ve analiz etmek için ek bir 'LAS X yazılımında FLIM modülü' açılacaktır.
- (FLIM için isteğe bağlı) Beklenen florofor ortalama ömrüne göre WLL darbe tekrarlama oranını seçin.
NOT: Lazer darbesinin frekansı, tam floresan bozunmasını toplamak için ayarlanmalıdır. Bu, mikroskoba takılan bir Darbe Seçici özelliği kullanılarak yapılabilir. Floresan bozunmasının lazer darbesi ile üst üste binmesi, tahmini floresan ömrünün kısalmasına yol açacaktır. Beklenen ortalama floresan ömründen 4-5 kat daha uzun darbe aralıklarına sahip olmanız önerilir (örneğin, 5 ns'ye kadar olan ömürler için 25 ns/40 MHz). Birçok darbeli lazer, sabit bir 80 MHz tekrarlama oranına sahiptir (yalnızca 2-3 ns'ye kadar bir aralık için idealdir). Bu, deney için doğru floroforları seçmek için önemlidir. - HIZLI CANLI modunu kullanarak önizleme görüntülemesini başlatın ve görüntüleme nesnesinin ince odağını ilgilenilen bir bölüme ayarlayın.
DİKKAT: Lazer güvenlik kurallarına kesinlikle uyun. Her zaman lazer güvenlik kurallarını göz önünde bulundurun ve iletim ışığını açmadan, göz merceğine veya örneğe bakmadan önce görüntülemenin durmasını bekleyin.
NOT: HIZLI CANLI modunda, 600 Hz'lik yüksek hızlı tarama (çift yönlü X tarama modu kullanılıyorsa maksimum kare hızı 4,43/sn'ye karşılık gelir), 256 x 256 piksel çözünürlük, floresanı foto ağartmadan korumak için görüntüye otomatik olarak uygulanır. İğne deliğini açın (örn. 3-4 AU'ya kadar) ve/veya floresan sinyali nesneye odaklanamayacak kadar zayıfsa lazer yoğunluğunu artırın. Eksik çürüme toplamadan kaçının. - (FLIM için isteğe bağlı) Bir FLIM modülü penceresinde (Canlı mod) görüntüleme sırasında görünen bir piksel yoğunluğu histogramına bakarak, ~1 foton/lazer darbe limitini (kırmızı çizgi) elde etmek için uygun lazer yoğunluğunu/iğne deliği boyutunu ve çözünürlüğünü ayarlayın. Yığılma etkisi riskini dışlamak için 1'den önemli ölçüde daha yükseğe çıkmaktan kaçının. Gerekirse, bir bozunma penceresinde tam bir bozunma koleksiyonuna sahip olmak için WLL darbe tekrarlama oranını ayarlayın (eksik bozunma toplamasını önlemek için, bkz. adım 2.2.8).
NOT: Foton sayısı (yoğunluk), montaj analizi veya fazör çizim bulutu için güvenilir bir bozunmayı yeniden oluşturmak için yeterli değilse, birkaç tarama tekrarı uygulayın (çerçeveler veya çizgiler veya tarama süresini ayarlayın), lazer yoğunluğunu artırın ve/veya çözünürlüğü feda edin (taranan ROI boyutu). Lazer tekrarlama oranının azaltılmasının, güvenilir bir bozunma rekonstrüksiyonu için daha fazla foton toplanmasını gerektirdiğini ve görüntüleme parametrelerinin ek bir düzeltmesinin gerekebileceğini unutmayın. Yoğun ışığın ve uzun sürekli aydınlatmanın hücre canlılığı ve metabolizması üzerindeki potansiyel etkisinin farkında olun55. Canlılık ve metabolizma üzerindeki olumsuz etki, uyarma ışığının yoğunluğuna, süresine ve dalga boyuna ve ayrıca görüntüleme yöntemine (örneğin, tek fotonlu konfokal ve çoklu fotonlu görüntüleme) bağlı olarak her bir durumda farklı olabilir. Görüntüleme parametrelerini buna göre ayarlayın ve gerekirse pilot deneylerde Calcein Green-veya propidyum iyodür yoğunluğu ile hücre canlılığını/ölümünü kontrol edin56. Mümkün olduğunda, canlı mikroskopi sırasında yeterli bir floresan sinyaline ulaşmak için floresan probu boyama protokolünün daha ileri optimizasyonları düşünülmelidir. - (3D z-stack için isteğe bağlı) Fast Live modundayken koordinatları, tarama yönünü ayarlayın ve bunları Z yığını penceresinde Başlangıç ve Bitiş (XYZ Tarama Modu) olarak nitelendirin. Z adım boyutunu veya adım sayısını seçin.
NOT: Yazılım, kullanılan çözünürlük ve tarama parametrelerine bağlı olarak 'optimum' adım sayısını otomatik olarak hesaplarken, canlı 3D rekonstrüksiyon normalde hızlı alım elde etmek için daha az sayıda adım gerektirebilir, örneğin, 1-2 μm adım boyutu, 50-100 μm yığın boyutu, çift yönlü tarama, toplam tarama süresinin 2-3 dakikasını gerektirir. Hücre altı organellerin, hücrelerin ve 3D hücre modelinin de ölçümler sırasında hareket edebileceğini unutmayın. Ek olarak, ışık penetrasyon derinliği ve saçılma sınırları nedeniyle, konfokal FLIM üzerinde tipik olarak sadece 50-100 μm tarama derinliği elde edilebilir. - Gerekli tüm ayarlar uygulandığında görüntülemeye başlayın.
- Resme uygun bir ad verin.
- İletim ışığı modunda bir sonraki görüntüleme nesnesini arayın ve görüntüleme prosedürünü önceden optimize edilmiş görüntüleme ayarlarıyla tekrarlayın (adım 2.2.8-2.2.12).
NOT: Yoğunluğa dayalı karşılaştırma veya yoğunluk oranı analizi için (örneğin, MMIR O2 probu tabanlı oksijenasyon analizi), analiz edilen tüm nesneler için her zaman aynı görüntüleme ayarlarını koruyun (büyütme ve objektif lens tipi, lazer yoğunluğu, güç ve darbe frekansı, uyarma dalga boyu, dedektör aralığı, iğne deliği, tarama hızı, piksel bekleme süresi ve çözünürlüğü). Bununla birlikte, floresan ömrü floresan yoğunluğuna bağlı olmadığından ve güvenilir hesaplama için uygun sayıda fotonun toplanmasını gerektirdiğinden, FLIM görüntüleme parametreleri, toplanan foton sayılarını farklı tedaviler veya deneysel koşullar arasında karşılaştırılabilir tutmak için deney boyunca yeniden ayarlanabilir. Bu nedenle, hem yoğunluğa dayalı hem de floresan ömrüne dayalı analizin gerekli olduğu multiparametrik analiz için, karşılaştırılan deney gruplarındaki tüm nesneler için optimize edilmiş evrensel görüntüleme ayarları uygulanmalıdır. "Yalnızca FLIM" karşılaştırması için, LAS X yazılımı bireysel görüntü ölçümü için IRF'nin hesaplanmasını sağladığından, biraz farklı görüntüleme ayarlarıyla elde edilen görüntüleri karşılaştırmak mümkündür42. Bununla birlikte, LAS X dışındaki FLIM uydurma analizi için (örneğin, FLIMfit45), görüntüleme yazılımından dışa aktarılamayacağından, cihaz yanıt fonksiyonu (IRF) her farklı görüntüleme koşulu için ölçülmelidir. Bu nedenle, deneysel tasarımın ve iş yükünün basitliği için, veri kümesindeki tüm görüntüler için aynı görüntüleme ayarlarının uygulanması önerilir. Daha sonra, karşılık gelen IRF ölçümleri, ölçülen spektral kanalın57,58,59 emisyon özellikleri, altın nanopartikül lüminesansı 60 veya çoklu foton FLIM61 için ikinci harmonik üretim sinyali ile söndürülmüş veya hızlı floresan ömürlü floroforların (ps aralığı dahilinde) kullanılmasıyla yapılabilir. LAS X yazılımında, önceden optimize edilmiş görüntüleme parametreleri, ilgilenilen dosyaya sağ tıklanarak ve Görüntü Ayarlarını Uygula seçilerek yeni bir proje için yüklenebilir. - Görüntüleme oturumu sonlandırıldığında, görüntüleme projesini kaydedin. Görüntüleme seansını sonlandırmak için, numuneyi mikroskopi aşamasından çıkarın ve görüntüleme tesisinde uygulanan standart prosedüre göre objektifi daldırma sıvısından (kullanılıyorsa) temizleyin. Projeyi ve yazılımı kapatın. Mikroskobu, lazerleri ve bağlı tüm cihazları kapatın.
- Görüntüleme veri analizine devam edin (adım 2.3).
- Görüntülemeden 30-60 dakika önce sıcaklık klima kontrol ünitesini açın. Gerekli havalandırma hızını ve sıcaklığı (35-37 °C) ayarlayın.
- LAS X FLIM modülü ve FIJI ile NAD(P)H/FAD-FLIM fazör görüntü işleme
NOT: Protokol, NAD(P)H ve FAD/Flavins otofloresan FLIM örneklerinde frekans alanı verileri için görüntülenen sferoidlerin floresan ömrü analizini açıklar. NAD(P)H otofloresan ölçümü, kısa ve daha uzun NAD(P)H otofloresan ömrü bileşenlerinin sırasıyla glikoliz veya oksidatif fosforilasyon (OxPhos) ile ilişkili olduğu metabolik analiz için altın standart haline geldi. Bu, metabolik yörünge boyunca bir fazör grafiği üzerinde, ölçülen serbest NAD (P) H veya proteine bağlı NAD (P) H standartlarına doğru kayma ile analiz edilebilir31,62. Metabolik kaymanın yörüngesini analiz etmek ve deney grupları arasındaki bir çizim üzerinde fazör bulutlarının konumunu karşılaştırmak için (aşağıdaki adım 2.3.6'ya bakınız), fazör bulutunun geometrik merkezinin (centroid) hesaplanmasına dayalı basitleştirilmiş bir fazör koordinatları analizi uygulandı29. Açıklanan protokol, FIJI'de merkez koordinatlarının hesaplanmasını ve koordinatlar kullanılarak bir fazör grafiği üzerindeki iki nokta arasındaki mesafenin ölçülmesini gösterir (örneğin, küresel NAD(P)H otofloresan fazör bulutunun bir merkezinden "serbest NAD(P)H" teorik noktasına olan mesafe). Benzer şekilde, FAD ve diğer otofloresan sinyalleri analiz edilebilir. Bir veri kümesi 1 ile .lif (LAS X yazılımı gereklidir) veya .ptu Bu prosedürü öğrenmek için dosya biçimleri sağlanmıştır (Ek Dosya 2, Ek Dosya 3, Ek Dosya 4, Ek Dosya 5, Ek Dosya 6, Ek Dosya 7, Ek Dosya 8, Ek Dosya 9, ve Ek Dosya 10).- FLIM modülünü LAS X'te açın, Projeyi Aç'ı seçin ve otofloresan NAD(P)H/FAD analizi için küresel görüntü dosyasını (.lif) yükleyin.
NOT: FLIM modülündeki olası hatalar ve ara veri kaybı nedeniyle, NAD(P)H/FAD analizi için orijinal küresel görüntü dosyasının (.lif) bir kopyasını kullanın ve ham dosyayı değiştirmeden tutun. - Tek bir görüntü seçin ve FLIM analiz arayüzüne gidin. Fazör grafiğine erişmek ve fazör analiz modunu etkinleştirmek için Fasor'a tıklayın. Gürültüyü en aza indirmek ve tüm fazör analizleri için veri görünürlüğünü artırmak için filtre (Medyan veya Dalgacık) uygulayın ve Eşiği ayarlayın. Harmonikleri seçin. NAD (P) H verilerine dayalı metabolik değişimin analizi için 2.3.3-2.3.5 adımlarıyla ilerleyin. Fazör grafiklerinin genel karşılaştırması için adım 2.3.6'dan devam edin.
NOT: Analiz ayarlarını (filtre türü, harmonikler, eşik, gruplama ve fazör ROI'leri) karşılaştırılan bir veri kümesindeki tüm görüntülere eşit olarak uygulayın. - (NAD(P)H analizi için isteğe bağlı) Veri kümesiyle ilgili herhangi bir görüntüyü seçin ve standart bir fazör çizim alanının evrensel bir dairesi üzerinde 0,45 ns'nin konumunu doğru bir şekilde bulmak için İki Bileşen için Çizim Oranı İmleci seçeneğini kullanın. Bu konum, normalde mono-üstel bozunmaya62 yakın olan saf homojen bir serbest NAD(P)h çözeltisinin ortalama floresan ömrüne atanacaktır.
NOT: Serbest-NADH ve serbest-NAD(P)H, 0.3 ns ve 0.7 ns63 olmak üzere iki kısa ömürlü bileşen ile su çözeltisinde benzer spektral özelliklere ve benzer floresan ömrü değerlerine sahiptir. Bu nedenle, fazör tabanlı analizin basitliği ve ömür boyu bileşenler arasındaki küçük bir fark nedeniyle, floresan bozunmalarının mono-üstel olduğu kabul edilir, bu da fazör bulutunun evrensel bir daire üzerinde tahsis edilmesine izin verir. Bir serbest NAD(P)H formunun referans ortalama ömrü de benzer bir analiz için bir fazör uzayda ölçülebilir ve çizilebilir. Referans ömrü literatür62'ye göre seçilmiştir; Diğer kaynaklarda, çözeltideki serbest NAD(P)H'nin biraz farklı değerinin bulunabileceğini unutmayın (0.4 ns64). - (NAD(P)H analizi için isteğe bağlı) Fazör grafiğini, ayrılmış serbest NAD(P)H ömrüne sahip olarak dışa aktarın (bkz. adım 2.3.3) grafiğe sağ tıklayıp Verileri Dışa Aktar'ı seçerek. Fazör grafiğini .tiff formatında bir dosya olarak belirlenmiş bir depolama klasörüne aktarın.
NOT: LAS X FLIM modülünden dışa aktarılan fazör çizim görüntüsünün orijinal piksel boyutu her zaman 1024 x 600 pikseldir. Veri dışa aktarma ve ön analiz için başka bir yazılım kullanılıyorsa, tüm fazör çizim görüntülerinin aynı boyut ve çözünürlükte dışa aktarıldığından emin olun. - Sferoid ile ilgili fazör bulutunu dışa aktarmak için, görüntüdeki küresel yatırım getirisini seçmek için LAS X FLIM modülündeki İmleç Çiz aracını kullanın. Oluşturulan fazör grafiğini adım 2.3.4'te belirtildiği gibi dışa aktarın.
NOT: Fazör uzayının karşılık gelen g ve s (x ve y'ye benzer) koordinatları, ömürlerine göre seçilen ROI'nin her pikseline atanacak ve bir frekans alanı veri seti 64,65'e dönüştürülecektir. Benzer tf (faz ömrü) ve tm (modülasyon ömrü) değerlerine sahip piksel kümesi, renk kodlamasının (koyu maviden kırmızıya kadar bir aralıkla) ömür boyu değerlerin bolluğunu yansıtacağı bir çizim üzerinde bir bulut deseni (fazör bulutu) oluşturacaktır. Bulutun evrensel bir daire üzerindeki veya içindeki konumu ile, tek veya çok üstel bozunmalar ayırt edilebilir. Bazı ölçüm ortamları, yoğunluk eşiği araçlarıyla basitçe çıkarılamayan bir fazör grafiği üzerinde karşılık gelen bir bulutun ortaya çıkmasına neden olan güçlü (otomatik) floresan sergiler. Bu model, merkez koordinatlarının hesaplanmasını etkileyecektir ve dışa aktarılan fazör grafiğinden çıkarılmalıdır. Küresel ROI ile çalışmak, ilgili olmayan piksellerin daha fazla fazör analizinden hariç tutulmasına izin verir. - Bir veri kümesindeki tüm küresel ROI'ler için fazör grafiği dışa aktarma prosedürünü tekrarlayın (bkz. adım 2.3.4 ve 2.3.6). Ek olarak, daha fazla karşılaştırmalı analiz için tam veri setini garanti etmek ve dışa aktarılan tüm görüntülerin aynı piksel boyutuna sahip olduğundan emin olmak için dışa aktarılan .tiff görüntü kümesini kontrol edin (adım 2.3.4'teki NOT'a bakın).
NOT: Protokolün bu aşamasında, görüntü seti, serbest NAD(P)H konumuna (literatüre veya ampirik olarak elde edilen verilere dayanarak) ve tüm küresel ROI ROI'sine (veya spesifik analiz için gerekirse alternatif ROI modellerine) sahip fazör grafiklerini içermelidir. Bu adımdan sonra, FIJI'de ve daha sonra bir elektronik tabloda daha fazla analiz yapılacaktır. Analiz aracı penceresini kullanarak, FIJI'de Ölçeği Ayarlayın seçeneğini belirleyin ve tüm fazör çizim görüntülerinin aynı birim türüyle, örneğin yalnızca piksel cinsinden kalibre edildiğinden emin olun. Değilse, seçilen için Ölçeği Ayarla penceresinde birim uzunluğu ayarlayın (örneğin, piksel tabanlı ölçek için, Piksel cinsinden Mesafe alanına 1 koyun ve uzunluk birimini Piksel olarak ayarlayın). Daha fazla karşılaştırma için, dışa aktarılan tüm verileri aynı birim türünü kullanarak ölçün. - (NAD(P)H analizi için isteğe bağlı) Karşılık gelen dışa aktarılan fazör çizim görüntüsünde serbest NAD(P)H ortalama floresan ömrünün piksel noktası konumunu belirleyin (bkz. adım 2.3.3): Fazör görüntüsünü FIJI ile açın, evrensel yarım daire ile İki Bileşen için Oran İmleci çizgisi arasındaki kesişimi bir piksel çözünürlüğüyle net bir şekilde görselleştirmek için görüntüyü büyütün; Kesişimi seçmek için Dikdörtgen ROI aracını kullanın.
NOT: Bir sonraki adımda koordinatlarının doğru bir şekilde belirlenmesi için dikdörtgen seçimin kesişme noktasının etrafında küçük bir alan olduğundan emin olun (adım 2.3.9). - (NAD(P)H analizi için isteğe bağlı) Analiz aracını açın, Ölçümleri Ayarla penceresini seçin ve ölçüm parametresi olarak Centroid'i seçin. Serbest NAD(P)H referans noktasının merkez koordinatlarını belirlemek için Analiz aracı penceresinde Ölç'e tıklayın. Bu koordinatları bir e-tabloya aktarın.
NOT: Free-NAD(P)H koordinatları, bir veri setinde bu noktadan küresel fazör bulutu konumuna olan mesafeleri karşılaştırmak için referans noktası olarak kullanılacaktır (NAD(P)H FLIM otofloresan analizinde glikoliz ve OxPhos arasındaki metabolik kaymayı karakterize etmenin yolu) - FIJI'yi kullanarak, küresel fazör bulutunun görüntüsünü açın. Görüntü penceresi aracını açın, Ayarla'yı seçin ve araç çubuğunda Renk Eşiği'ni seçin. Tercih edilen Eşikleme Yöntemini seçin (örneğin, Otsu) ve en bol piksel koordinatlarına sahip bir fazör bulutu parçasının seçimi için parametreleri daraltmak için Ton Değeri ve Parlaklık Değerini ayarlayın. Küme alanını tanımlamak için Seç'e tıklayın.
NOT: Analiz edilmesi gereken tüm fazör çizim görüntüleri için aynı eşik parametrelerini koruyun. Sunulan NAD(P)H-FLIM verileri için, Ton Değeri 9 ve Parlaklık Değeri 160 olarak ayarlanmış Otsu eşikleme yöntemi seçildi ve tüm fazör görüntülerine uygulandı. Alternatif olarak, seçilen alan, daha fazla analiz için bir fazör ROI kitaplığı oluşturmak için ROI Manager'a kopyalanabilir (Düzenle > Seçimi > Yöneticiye Ekle yolunu izleyin). - Seçimi korurken, adım 2.3.9'da açıklanan prosedürü izleyerek seçilen alanın merkez koordinatlarını hesaplayın. Bu koordinatları elektronik tablo dosyasına aktarın.
- Elektronik tabloda bir veri kümesi oluşturmak üzere tüm ROI fazör görüntülerinin merkez koordinatlarını belirlemek için 2.3.9 ve 2.3.10 adımlarını tekrarlayın.
NOT: ROI Manager ROI kitaplığını kullanmak, ROI analizini basitleştirmeye ve düzenlemeye yardımcı olur (bkz. adım 2.3.10) - (NAD(P)H analizi için isteğe bağlı) Farklı karşılaştırma gruplarından dışa aktarılan referans free-NAD(P)H ve sferoid ROI koordinatlarını içeren elektronik tabloyu açın. Belirlenen koordinatları ve aşağıdaki denklemi kullanarak her bir Spheroid fazör centroid ile referans serbest NAD(P)H konumu arasındaki mesafeyi hesaplayın:
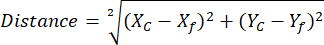
Burada, Xc ve Yc merkez koordinatlarıdır, Xf ve Yf referans koordinatlarıdır.
NOT: Referans ömrüne doğru kaymanın belirlenmesi için centroid parametresinin uygulanması, yalnızca bir veri setindeki tüm centroidlerin referans noktasına doğru aynı doğrusal yörüngede bulunması durumunda uygundur. Bunu kontrol etmek için, veri setindeki tüm merkez noktaları, aynı koordinat alanındaki referans noktası ile birlikte çizilmeli ve doğrusal trend hizalaması gerçekleştirilmelidir. Tüm noktalardan çizilen doğrusal eğilim çizgisinin R2 katsayısı 1'e yakınsa (örneğin, R2 0.8-0.99 ise), mesafe analizinin uygun olduğu varsayılır. - Karşılaştırma için tüm verileri uygun şekilde düzenleyin ve ilgili herhangi bir yazılımı (örneğin, Origin, MatLab) kullanarak istatistiksel analiz yapın. Veri seti özelliklerine (dağılım normalliği, istatistiksel birimlerin sayısı vb.) göre uygun istatistiksel testi seçin.
NOT: NAD(P)H analizi için, deneysel koşullara bağlı olarak metabolik kaymayı karakterize etmek için mesafe değerlerini karşılaştırın. Deney grupları arasındaki herhangi bir fazör grafiğinin karşılaştırılması için, ROI fazör bulutu merkez koordinatlarının karşılaştırmasını gerçekleştirin.
- FLIM modülünü LAS X'te açın, Projeyi Aç'ı seçin ve otofloresan NAD(P)H/FAD analizi için küresel görüntü dosyasını (.lif) yükleyin.
Sonuçlar
Uygun sferoid oluşum yönteminin seçilmesi
Seçilen sferoid oluşturma yöntemi, sferoidlerin boyutunu, şeklini, hücre yoğunluğunu, canlılığını ve ilaç duyarlılığını büyük ölçüde etkileyebilir (Şekil 2). Daha önce, çoklu yüksek verimli (SphericalPlate 5D, laboratuvar yapımı mikro kalıplar ve MicroTissue kalıpları) ve 'orta verimli' düşük bağlanma (Biofloat ve Lipidür kaplı 96 oyuklu plakalar) yöntemlerinin etkileri, sferoidlerin canl?...
Tartışmalar
Çok hücreli sferoidler, tümör ve kök hücre niş mikro çevreleri, ilaç keşfi ve biyofabrikasyon için 'doku yapı taşlarının' geliştirilmesi çalışmalarında tercih edilen bir yöntem haline gelmektedir. Sferoidlerin heterojen iç mimarisi, besin gradyanları ve oksijenasyon, nispeten basitleştirilmiş ve erişilebilir bir ortamda in vivo doku ve tümörlerinkini taklit edebilir. Daha fazla metodolojik şeffaflığa 26,28 ve standardizasyona71
Açıklamalar
Açıklanacak bir şey yok.
Teşekkürler
Bu çalışma, Ghent Üniversitesi'nin Özel Araştırma Fonu (BOF) hibeleri ile desteklenmiştir (BOF/STA/202009/003; BOF/IOP/2022/058), Flanders Araştırma Vakfı (FWO, I001922N) ve Avrupa Birliği, fliMAGIN3D-DN Ufuk Avrupa-MSCA-DN No. 101073507.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | Also available from Sigma |
| 10 mL serological pipets | VWR | 612-3700 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 12 well cell-culture plates, sterile | Greiner bio-one | 665-180 | Similar products are also available from Sarstedt, Corning and other companies. |
| 12 Well Chamber slide, removable | Ibidi | 81201 | Also available from Grace Bio-Labs, ThermoFisher Scientific and others |
| 15 mL centrifuge tubes | Nerbe plus | 02-502-3001 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 3D Petri Dish micromolds | Microtissue | Z764000-6EA | |
| 6 well cell-culture plates, sterile | Greiner bio-one | 657160 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 70% ethanol | ChemLab | CL02.0537.5000 | |
| Biofloat | Sarstedt | 83.3925.400 | Commercial available coated 96-well plate for spheroid formation |
| Calcein Green-AM | Tebubio | AS-89201 | Apply in dilution 1:1000 |
| CellSens Dimension software | Olympus | version 3 | |
| Collagen from human placenta, type IV | Sigma | C5533 | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Confocal FLIM Microscope | Leica Microsystems | N/A | Stellaris 8 Falcon inverted microscope with white-light laser, HyD X detectors, climate / T control chamber (OkoLab), 25x/0.95 W objective |
| D(+)-Glucose | Merck | 8342 | Prepare 1 M stock solution, 1:100 for preparation of imaging medium (final concentration 10 mM) |
| Dulbecco's modified Eagle's medium (DMEM), phenol red-, glucose-, pyruvate- and glutamine-free | Sigma-Aldrich | D5030-10X1L | For preparation of imaging medium |
| Fetal Bovine Serum (FBS) | Gibco | 10270-098 | Also available from Sigma. Needs to be heat-inactivated before use. |
| HEPES (1M) | Gibco | 15630-080 | Dilution 1/100 for preparation of imaging medium (final concentration 10 mM) |
| Human colon cancer cells HCT116 | ATCC | ||
| ImageJ | NIH | version 1.54f | |
| Leica Application Suite X (LAS X) | Leica Microsystems | version 4.6.1.27508 | |
| L-glutamine | Gibco | 25030 | Also available from Sigma. Apply in dilution 1:100. |
| Lipidure-CM5206 | Amsbio | AMS.52000034GB1G | |
| McCoy's 5A, need addition of 1 mM Sodium Pyruvate and 10 mM HEPES | VWR | 392-0420 | Standard growth medium for HCT116 cells |
| micro-patterned 3D-printed PDMS stamps | N/A | N/A | Provided by the Centre for Microsystems Technology, Professor Dr. Jan Vanfleteren, Ghent University |
| NaCl | Chemlab | CL00.1429.100 | |
| Neubauer couting chamber | Fisher Scientific | 15980396 | |
| O2 probes: MMIR1 | N/A | N/A | Full characterization, validation and some applications can be found at: https://www.biorxiv.org/content/10.1101/2023.12.11.571110 v1 |
| PBS | Fisher scientific | Gibco18912014 | Dissolve PBS tablet in 500 mL of distilled water. |
| Pen Strep :Penicillin (10,000 U/mL) / streptomycin (10,000 μg/mL) 100x solution | Gibco | 15140-122 | Also available from Sigma. Apply in dilution 1:100. |
| Poly-D-lysine | Sigma | P6407-5mg | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Propidium Iodide | Sigma-Aldrich | 25535-16-4 | Cell death staining, use 1 µg/mL at 1h incubation |
| PVDF syringe filter 0.22 µm | Novolab | A35149 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Sodium pyruvate (100 mM) | Gibco | 11360-070 | Dilution 1/100 for preparation of imaging medium (final concentration 1mM) |
| SphericalPlate 5D 24-well | Kugelmeiers | SP5D-24W | |
| sterile petridish | Greiner bio-one | 633181 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (25 cm² ) | VWR | 734-2311 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (75 cm²) | VWR | 734-2313 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| U-bottom 96-well plate | VWR | 10062-900 | Similar products are also available from Sarstedt, Corning, Greiner Bio-one and other companies |
| Ultrapure Agarose | Invitrogen (Life Technologies) | 16500-500 | Other types of Agarose such as Agarose low melting point (A-9414, Sigma), Agarose for routine use (A-9539, Sigma) |
| Widefield fluorescence inverted microscope | Olympus | N/A | Inverted fluorescence microscope IX81, with motorised Z-axis control, CoolLED pE4000 (16 channels, 365-770 nm), ORCA-Flash4.0LT (Hamamatsu) cMOS camera, glass warming plate Okolab, CellSens Dimension v.3 software and air objectives 4x/0.13 UPlanFLN and 40x/0.6 LUCPlanFLN. (Optional, for high-resolution imaging) 60x/1.0 LUMPLFLN water |
Referanslar
- Foty, R. A simple hanging drop cell culture protocol for generation of 3d spheroids. J Vis Exp. (51), e2720 (2011).
- Moskovits, N., et al. Establishing 3-dimensional spheroids from patient-derived tumor samples and evaluating their sensitivity to drugs. J Vis Exp. (190), e64564 (2022).
- Griner, L. M., et al. Generation of high-throughput three-dimensional tumor spheroids for drug screening. J Vis Exp. (139), e57476 (2018).
- Qian, Y., Wei, X., Chen, K., Xu, M. Three-dimensional acoustic assembly device for mass manufacturing of cell spheroids. J Vis Exp. (200), e66078 (2023).
- He, H., et al. Dynamic formation of cellular aggregates of chondrocytes and mesenchymal stem cells in spinner flask. Cell Prolif. 52 (4), e12587 (2019).
- Perez, J. E., Nagle, I., Wilhelm, C. Magnetic molding of tumor spheroids: Emerging model for cancer screening. Biofabrication. 13 (1), 015018 (2020).
- Kingsley, D. M., et al. Laser-based 3d bioprinting for spatial and size control of tumor spheroids and embryoid bodies. Acta Biomater. 95, 357-370 (2019).
- Guillaume, O., et al. Hybrid spheroid microscaffolds as modular tissue units to build macro-tissue assemblies for tissue engineering. Acta Biomater. 165, 72-85 (2023).
- Danilevicius, P., et al. laser-made 3D microscaffolds for tissue spheroid encagement. Biointerphases. 10 (2), 021011 (2015).
- Jamieson, L. E., Harrison, D. J., Campbell, C. Chemical analysis of multicellular tumour spheroids. Analyst. 140 (12), 3910-3920 (2015).
- Dmitriev, R., Borisov, S., Jenkins, J., Papkovsky, D. Multiparametric imaging of tumor spheroids with ultra-bright and tunable nanoparticle O2 probes. SPIE BiOS. 9328, (2015).
- Zushin, P. -. J. H., Mukherjee, S., Wu, J. C. FDA Modernization Act 2.0: transitioning beyond animal models with human cells, organoids, and AI/ML-based approaches. J Clin Invest. 133 (21), e175824 (2023).
- Kim, D. -. S., et al. Robust enhancement of neural differentiation from human es and ips cells regardless of their innate difference in differentiation propensity. Stem Cell Rev Rep. 6 (2), 270-281 (2010).
- Hart, D., Gutiérrez, D. R., Biason-Lauber, A. Generation of a human ovarian granulosa cell model from induced pluripotent stem cells. bioRxiv. , 2022 (2022).
- Chiaradia, I., et al. Tissue morphology influences the temporal program of human brain organoid development. Cell Stem Cell. 30 (10), 1351-1367 (2023).
- Wagstaff, E. L., Ten Asbroek, A. L., Ten Brink, J. B., Jansonius, N. M., Bergen, A. A. An alternative approach to produce versatile retinal organoids with accelerated ganglion cell development. Sci Rep. 11 (1), 1101 (2021).
- Thavandiran, N., et al. Design and formulation of functional pluripotent stem cell-derived cardiac microtissues. Proc Natl Acad Sci U S A. 110 (49), E4698-E4707 (2013).
- Harrison, S. P., et al. Scalable production of tissue-like vascularized liver organoids from human pscs. Exp Mol Med. 55 (9), 2005-2024 (2023).
- Noguchi, T. -. A. K., Kurisaki, A. Formation of stomach tissue by organoid culture using mouse embryonic stem cells. Methods Mol Biol. 2017, 217-228 (2017).
- Takahashi, J., et al. Suspension culture in a rotating bioreactor for efficient generation of human intestinal organoids. Cell Reports Methods. 2 (11), 100337 (2022).
- Lagziel, S., Gottlieb, E., Shlomi, T. Mind your media. Nat Metab. 2 (12), 1369-1372 (2020).
- Das, V., Fürst, T., Gurská, S., Džubák, P., Hajdúch, M. Reproducibility of uniform spheroid formation in 384-well plates: The effect of medium evaporation. J Biomol Screen. 21 (9), 923-930 (2016).
- Bera, K., et al. Extracellular fluid viscosity enhances cell migration and cancer dissemination. Nature. 611 (7935), 365-373 (2022).
- Michl, J., Park, K. C., Swietach, P. Evidence-based guidelines for controlling ph in mammalian live-cell culture systems. Commun Biol. 2, 144 (2019).
- Okkelman, I. A., Vercruysse, C., Kondrashina, A. V., Borisov, S. M., Dmitriev, R. I. Affordable oxygen microscopy-assisted biofabrication of multicellular spheroids. J Vis Exp. (182), e63403 (2022).
- Peirsman, A., et al. Mispheroid: A knowledgebase and transparency tool for minimum information in spheroid identity. Nat Methods. 18 (11), 1294-1303 (2021).
- Raghavan, S., et al. Comparative analysis of tumor spheroid generation techniques for differential in vitro drug toxicity. Oncotarget. 7 (13), 16948 (2016).
- Froehlich, K., et al. Generation of multicellular breast cancer tumor spheroids: Comparison of different protocols. J Mammary Gland Biol Neoplasia. 21 (3-4), 89-98 (2016).
- Debruyne, A. C., et al. Live microscopy of multicellular spheroids with the multimodal near-infrared nanoparticles reveals differences in oxygenation gradients. ACS Nano. 18 (19), 12168-12186 (2024).
- Debruyne, A. C., Okkelman, I. A., Dmitriev, R. I. Balance between the cell viability and death in 3D. Semin Cell Dev Biol. 144, 55-66 (2023).
- Barroso, M., Monaghan, M. G., Niesner, R., Dmitriev, R. I. Probing organoid metabolism using fluorescence lifetime imaging microscopy (flim): The next frontier of drug discovery and disease understanding. Adv Drug Deliv Rev. 201, 115081 (2023).
- Becker, W. Fluorescence lifetime imaging-techniques and applications. J Microsc. 247 (2), 119-136 (2012).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. J Cell Sci. 134 (9), 1-17 (2021).
- Sarder, P., Maji, D., Achilefu, S. Molecular probes for fluorescence lifetime imaging. Bioconjug Chem. 26 (6), 963-974 (2015).
- Alfonso-Garcia, A., et al. Mesoscopic fluorescence lifetime imaging: Fundamental principles, clinical applications and future directions. J Biophotonics. 14 (6), e202000472 (2021).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: Fundamentals and advances in instrumentation, analysis, and applications. J Biomed Opt. 25 (7), 1-43 (2020).
- Yellen, G. Fueling thought: Management of glycolysis and oxidative phosphorylation in neuronal metabolism. J Cell Biol. 217 (7), 2235-2246 (2018).
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomark Med. 4 (2), 241-263 (2010).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of nad (p) h and fad. Antioxid Redox Signal. 30 (6), 875-889 (2019).
- Verma, A., et al. Fluorescence lifetime imaging for quantification of targeted drug delivery in varying tumor microenvironments. bioRxiv. , 2024 (2024).
- Smith, J. T., et al. In vivo quantitative fret small animal imaging: Intensity versus lifetime-based fret. Biophys Rep. 3 (2), 100110 (2023).
- Alvarez, L. A., et al. Application Note: SP8 Falcon: A novel concept in fluorescence lifetime imaging enabling video-rate confocal flim. Nat Methods. , (2019).
- Roberti, M. J., et al. TauSense: A fluorescence lifetime-based tool set for everyday imaging. Nat. Methods. , (2020).
- Auer, J. M. T., Murphy, L. C., Xiao, D., Li, D. U., Wheeler, A. P. Non-fitting flim-fret facilitates analysis of protein interactions in live zebrafish embryos. J Microsc. 291 (1), 43-56 (2023).
- Warren, S. C., et al. Rapid global fitting of large fluorescence lifetime imaging microscopy datasets. PLoS One. 8 (8), e70687 (2013).
- Gao, D., et al. Flimj: An open-source imagej toolkit for fluorescence lifetime image data analysis. PloS One. 15 (12), e0238327 (2020).
- Tullis, I. D. C., Ameer-Beg, S. M., Barber, P. R., Rankov, V., Vojnovic, B. Mapping femtosecond pulse front distortion and group velocity dispersion in multiphoton microscopy. Proc. SPIE 6089, Multiphoton Microscopy in the Biomedical Sciences VI. , 60890 (2006).
- Zhou, Y., et al. One-step derivation of functional mesenchymal stem cells from human pluripotent stem cells. Bio Protoc. 8 (22), e3080 (2018).
- Benton, G., Arnaoutova, I., George, J., Kleinman, H. K., Koblinski, J. Matrigel: From discovery and ECM mimicry to assays and models for cancer research. Adv Drug Deliv Rev. 79-80, 3-18 (2014).
- Badea, M. A., et al. Influence of matrigel on single-and multiple-spheroid cultures in breast cancer research. SLAS Discov. 24 (5), 563-578 (2019).
- Lang, S., Sharrard, R., Stark, M., Villette, J., Maitland, N. Prostate epithelial cell lines form spheroids with evidence of glandular differentiation in three-dimensional matrigel cultures. Br J Cancer. 85 (4), 590-599 (2001).
- Barra, J., et al. DMT1-dependent endosome-mitochondria interactions regulate mitochondrial iron translocation and metastatic outgrowth. Oncogene. 43 (9), 650-667 (2024).
- Dmitriev, R. I., Papkovsky, D. B. Intracellular probes for imaging oxygen concentration: How good are they. Methods Appl Fluoresc. 3 (3), 034001 (2015).
- Schweikhard, V., et al. Application Note: The power HyD family of detectors for confocal microscopy. Nat Methods. , (2020).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon flim microscopy. Sci Rep. 12 (1), 11938 (2022).
- Bush, P. G., Wokosin, D. L., Hall, A. C. Two-versus one photon excitation laser scanning microscopy: Critical importance of excitation wavelength. Front Biosci. 12, 2646-2657 (2007).
- Liu, M., et al. Instrument response standard in time-resolved fluorescence spectroscopy at visible wavelength: Quenched fluorescein sodium. Appl Spectrosc. 68 (5), 577-583 (2014).
- Szabelski, M., et al. Collisional quenching of erythrosine b as a potential reference dye for impulse response function evaluation. Appl Spectrosc. 63 (3), 363-368 (2009).
- Chib, R., et al. Standard reference for instrument response function in fluorescence lifetime measurements in visible and near infrared. Meas Sci Technol. 27 (2), 027001 (2015).
- Talbot, C. B., et al. Application of ultrafast gold luminescence to measuring the instrument response function for multispectral multiphoton fluorescence lifetime imaging. Opt Express. 19 (15), 13848-13861 (2011).
- Recording the instrument response function of a multiphoton flim system. Becker & Hickl Available from: https://www.becker-hickl.com/literature/application-notes/recording-the-instrument-response-function-of-a-multiphoton-flim-system/ (2007)
- Leben, R., Köhler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free nad (p) h fluorescence lifetime imaging. Int J Mol Sci. 20 (22), 5565 (2019).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using nadh and nadph autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Gottlieb, D., Asadipour, B., Kostina, P., Ung, T. P. L., Stringari, C. FLUTE: A python gui for interactive phasor analysis of flim data. Biol Imaging. 3, e21 (2023).
- Malacrida, L., Ranjit, S., Jameson, D. M., Gratton, E. The phasor plot: A universal circle to advance fluorescence lifetime analysis and interpretation. Annu Rev Biophys. 50, 575-593 (2021).
- Okkelman, I., Vandenberghe, W., Dmitriev, R. Role of preconditioning with oxygen and glucose deprivation in promoting differentiation of dental pulp stem cells in 3D culture. Mol Biol Cell. 34 (2), 1212 (2022).
- Park, J. H., et al. The effect of bmp-mimetic peptide tethering bioinks on the differentiation of dental pulp stem cells (DPSCs) in 3D bioprinted dental constructs. Biofabrication. 12 (3), 035029 (2020).
- Pașca, S. P. The rise of three-dimensional human brain cultures. Nature. 553 (7689), 437-445 (2018).
- Dmitriev, R. I., Zhdanov, A. V., Nolan, Y. M., Papkovsky, D. B. Imaging of neurosphere oxygenation with phosphorescent probes. Biomaterials. 34 (37), 9307-9317 (2013).
- Bryanskaya, E. O., et al. High levels of FAD autofluorescence indicate pathology preceding cell death. Biochim Biophys Acta Gen Subj. 1868 (1), 130520 (2024).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 152 (2021).
- Okkelman, I. A., Neto, N., Papkovsky, D. B., Monaghan, M. G., Dmitriev, R. I. A deeper understanding of intestinal organoid metabolism revealed by combining fluorescence lifetime imaging microscopy (flim) and extracellular flux analyses. Redox Biol. 30, 101420 (2020).
- Gstraunthaler, G., Seppi, T., Pfaller, W. Impact of culture conditions, culture media volumes, and glucose content on metabolic properties of renal epithelial cell cultures: Are renal cells in tissue culture hypoxic. Cell Physiol Biochem. 9 (3), 150-172 (1999).
- Glickman, R. D. Phototoxicity to the retina: Mechanisms of damage. Int J Toxicol. 21 (6), 473-490 (2002).
- Golub, A. S., Pittman, R. N. Monitoring Parameters of Oxygen Transport to Cells in the Microcirculation. Quenched-Phosphorescence Detection of Molecular Oxygen: Applications in Life Sciences. , (2018).
- Nishigaki, T., Wood, C. D., Shiba, K., Baba, S. A., Darszon, A. Stroboscopic illumination using light-emitting diodes reduces phototoxicity in fluorescence cell imaging. Biotechniques. 41 (2), 191-197 (2006).
- Penjweini, R., Loew, H. G., Hamblin, M. R., Kratky, K. W. Long-term monitoring of live cell proliferation in presence of pvp-hypericin: A new strategy using ms pulses of led and the fluorescent dye cfse. J Microsc. 245 (1), 100-108 (2012).
- Carrasco Kind, M., et al. flimview: A software framework to handle, visualize and analyze flim data. F1000Research. 9, 574 (2020).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. Pam: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophys J. 114 (7), 1518-1528 (2018).
- Chen, S. -. J., Sinsuebphon, N., Barroso, M., Intes, X., Michalet, X. . Optical Molecular Probes, Imaging and Drug Delivery. , (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır