Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Tardigrad Hypsibius exemplaris'te qRT-PCR ile Bağıl Transkript Seviyelerinin Karşılaştırılması için Tek Hayvanlı, Tek Tüplü RNA Ekstraksiyonu
Bu Makalede
Özet
Bu çalışma, tardigrad Hypsibius exemplaris'te gen ekspresyonunu analiz etmek için hızlı bir RNA ekstraksiyonu ve transkript seviyesi karşılaştırma yöntemi sunmaktadır.Fiziksel lizis kullanan bu yüksek verimli yöntem, başlangıç materyali olarak tek bir tardigrad gerektirir ve kantitatif ters transkripsiyon polimeraz zincir reaksiyonu (qRT-PCR) için sağlam cDNA üretimi ile sonuçlanır.
Özet
Tardigrad Hypsibius exemplaris , çevresel aşırılıklarda hayatta kalma yeteneği ile ünlü, gelişmekte olan bir model organizmadır. Bu tür ekstremotoleransın moleküler mekanizmalarını ve genetik temelini araştırmak için birçok çalışma, büyük kohortlardan bireysel hayvanlara kadar değişen popülasyonlarda gerçekleştirilebilen RNA dizilimine (RNA-dizilimi) dayanır. Ters transkripsiyon polimeraz zincir reaksiyonu (RT-PCR) ve RNA interferansı (RNAi) daha sonra sırasıyla RNA-seq bulgularını doğrulamak ve aday genler için genetik gereksinimleri değerlendirmek için kullanılır. Bu tür çalışmalar, RNA ekstraksiyonu ve kantitatif RT-PCR (qRT-PCR) ile göreceli transkript seviyelerinin ölçümü için verimli, doğru ve uygun fiyatlı bir yöntem gerektirir. Bu çalışma, RNA'yı yalnızca tek tek tardigradlardan güvenilir bir şekilde izole etmekle kalmayıp aynı zamanda her ekstraksiyon için gereken süreyi ve maliyeti azaltan verimli bir tek tardigrad, tek tüplü RNA ekstraksiyon yöntemi (STST) sunar. Bu RNA ekstraksiyon yöntemi, kantitatif PCR (qRT-PCR) ile çoklu transkriptleri amplifiye etmek ve tespit etmek için kullanılabilecek miktarlarda cDNA verir. Yöntem, iki ısı şoku regüle proteini, Isı-Şok Proteini 70 β2 (HSP70 β2) ve Isı-Şoku Proteini 90α'yı (HSP90α) kodlayan genlerin ekspresyonundaki dinamik değişikliklerin analiz edilmesiyle doğrulanmıştır ve qRT-PCR kullanarak ısıya maruz kalan bireylerde nispi ekspresyon seviyelerini değerlendirmeyi mümkün kılar. STST, mevcut toplu ve tek tardigrad RNA ekstraksiyon yöntemlerini etkili bir şekilde tamamlar ve bireysel tardigrad transkripsiyonel seviyelerin qRT-PCR ile hızlı ve uygun maliyetli bir şekilde incelenmesine izin verir.
Giriş
Tardigradlar, diğer yaşam biçimlerinin çoğu için ölümcül olan aşırı koşullarda hayatta kalma yetenekleriyle tanınan küçük, çok hücreli hayvanlardır1. Örneğin, bu hayvanlar, insanlar için öldürücü olaniyonlaştırıcı radyasyon dozunun yaklaşık 1000 katı hayatta kalabilir 2,3,4,5,6,7,8,9,10, neredeyse tamamen kuruma 11,12,13,14,15, ilave edilmediğinde donma kriyoprotektanlar 16,17,18 ve kurumuş hallerinde uzay boşluğu bile 19,20. Aşırı ortamlarda hayatta kalma konusundaki benzersiz kapasiteleri nedeniyle, bu hayvanlar karmaşık, çok hücreli organizmalarda aşırı toleransı anlamak için temel modeller haline gelmiştir 1,21,22,23.
Transgenez ve germ hattı gen modifikasyonu da dahil olmak üzere bu olağanüstü hayvanların stabil genetik manipülasyonu, yakın zamana kadar zor kalmıştır24,25. Bu nedenle, ekstremotoleransın moleküler mekanizmalarını ortaya çıkarmak için yapılan deneylerin çoğu, RNA dizilimi yoluyla transkripsiyonel profilleme yoluyla gerçekleştirilir. Radyasyon 8,9,26,27,28, ısı stresi 29, donma stresi 12 ve kuruma 27,30,31,32,33 gibi çeşitli aşırı koşullar altında tardigradlar için birçok değerli ve bilgilendirici RNA dizileme veri seti mevcuttur. Bu çalışmalardan bazıları, ekstremotolerans konusundaki moleküler anlayışımızı aydınlatmak için toplu RNA ekstraksiyonu ve saflaştırma yöntemlerini kullanmıştır. Bununla birlikte, birçok hayvandan RNA transkriptlerinin toplu olarak çıkarılması, bireyler arasındaki gen ekspresyonundaki varyasyonun analizini önler, böylece daha rafine veri setlerinin potansiyel zenginliğini kaçırır. Daha da önemlisi, bu çalışmalar genellikle hem çevresel stres faktörlerinden kurtulan hem de hayatta kalmayan hayvanları içeren heterojen hayvan popülasyonlarını analiz eder. Bu nedenle, bu çalışmalar, birden fazla ve potansiyel olarak önemli ölçüde farklı yanıt durumlarından gelen ifade verilerinin ortalamasının alınmasıyla karıştırılır. Bu sorunu ele almak için, Arakawa ve ark., 201634, bir RNA ekstraksiyon kiti ve ardından girdi olarak tek 34,35,36 veya birden fazla30,37,38 hayvan kullanarak doğrusal bir PCR amplifikasyon adımı uygulayan zarif bir düşük girdili RNA-seq boru hattı geliştirdi. Bu çalışmalar, tardigrad ekstremotolerans22'yi anlamamız için temel teşkil etmiştir. İlginç bir şekilde, bu protokol aynı zamanda başlangıç materyali24 olarak yedi hayvan kullanılarak qRT-PCR'ye de uygulanmıştır.
Çoğu model organizmada, RNA-seq yoluyla potansiyel hedefleri belirledikten sonra, RNA-seq tarafından tanımlanan transkripsiyonel değişiklikleri doğrulamak ve aday genlerin ekspresyon zaman seyrini yüksek çözünürlüklü bir şekilde değerlendirmek için qRT-PCR gerçekleştirilir. Tanımlanan genlerin işlevini test etmek için, bu tür çalışmaları genellikle moleküler hedeflerin39,40 RNAi aracılı yıkımı ve ekstremotolerant kapasitenin12,41 analizi takip eder. Her bir RNAi yıkımının etkinliği tipik olarak transkript bolluğundaki azalmayı doğrudan izleyerek qRT-PCR ile doğrulanır. Bununla birlikte, RNAi, tardigradlarda emek yoğun bir süreçtir, çünkü her dsRNA'nın bireylere manuel mikroenjeksiyon yoluyla verilmesi gerekir39,40. Bu stratejinin düşük verimli doğası nedeniyle, tek hayvanlardan qRT-PCR için uyarlanmış hızlı, düşük maliyetli bir RNA ekstraksiyon yöntemi, tardigrad araştırmaları için oldukça değerli olacaktır. Tek tardigradlardan RNA'yı çıkarmak için önceki yöntemler geliştirilmiş olsa da, bu protokoller ekstraksiyonlarını qRT-PCR ile birleştirmemiştir, bunun yerine optik yoğunluğa dayalı yöntemleredayanmaktadır 12,40,41. Bu zorluklardan motive olarak, tek H. exemplaris'ten qRT-PCR için kullanılabilecek miktar ve kalite olarak RNA'yı güvenilir bir şekilde veren bir protokol geliştirmeye çalıştık.
Caenorhabditis elegans42 için geliştirilen tek hayvanlı bir RNA ekstraksiyon protokolünden uyarlanan STST, H. exemplaris için optimize edilmiştir. Ekstraksiyon yöntemi, kütikülü fiziksel olarak bozan, RNA ekstraksiyonuna ve ardından cDNA sentezine izin veren altı hızlı donma-çözülme adımından oluşur. STST yöntemi, Boothby, 2018 43 tarafından açıklandığı gibi toplu RNA ekstraksiyon yöntemlerine kıyasla ekstraksiyon süresini 24 kattan fazla ve Arakawa ve diğerleri, 201634 tarafından tarif edildiği gibi tek tardigrad RNA ekstraksiyon kitlerine kıyasla %30 azaltır. Ayrıca, RNA ekstraksiyon kiti preparatlarına kıyasla numune-deneyci etkileşimlerinin sayısı 5'ten sadece 1'e düşürülür, böylece eksojen ribonükleazlar tarafından kontaminasyon riski azaltılır. Yüksek oranda eksprese edilen genleri sorgularken, STST yöntemi, tek tardigrad başına 25 kantitatif RT-PCR reaksiyonu için yeterli cDNA üretir ve reaksiyon başına toplam 25 μL cDNA hacminin sadece 1 μL'sini gerektirir. Bununla birlikte, daha düşük bolluk transkriptleri için şablon konsantrasyonlarının ampirik olarak belirlenmesi gerekir.
Gen ekspresyonundaki dinamik değişiklikleri analiz etmek için STST yönteminin etkinliği, 20 dakika boyunca 35 ° C'de kısa süreli ısı şokuna yanıt olarak ısı şoku proteini-90α (HSP90α) ve ısı şoku proteini 70β2 (HSP70β2) kodlayan genlerin diferansiyel ekspresyonunun araştırılmasıyla değerlendirildi. Çoğu ökaryotik organizmada hem HSP70β2 hem de HSP90α, kısa süreli ısı şokuna maruz kalmanın (20 dakika) ardından hızla yukarı regüle edilir42. H. exemplaris'teki analiz, tek ısıl işlem görmüş tardigradlardan ekstrakte edilen hem HSP70β2 hem de HSP90α kodlayan RNA'ların, kısa süreli ısıya maruz kalmayı takiben ekspresyonda istatistiksel olarak anlamlı artışlar gösterdiğini ortaya koydu. Bu bulgular, STST protokolünün zaman içinde bireysel hayvanlarda gen ekspresyonundaki dinamik değişiklikleri analiz etmek için kullanılabileceğini göstermektedir.
STST ekstraksiyon yöntemi, hızlı ve ucuz RNA ekstraksiyonunu ve ardından transkript seviyelerinin qRT-PCR ile karşılaştırılmasını kolaylaştırarak RNA-seq gibi mevcut deneysel yöntemleri tamamlamalıdır. Bu yöntem aynı zamanda, manuel olarak enjekte edilen bireylerde RNAi'nin verimliliğini ve penetransını tek başına optik yoğunluktan daha kantitatif olarak değerlendirmek için değerli olacaktır. Son olarak, benzer kütiküler yapıları ve fiziksel özellikleri nedeniyle, bu yöntemin diğer tardigrad türlerinde gen ekspresyonunu analiz etmek için de etkili olması muhtemeldir44.
Access restricted. Please log in or start a trial to view this content.
Protokol
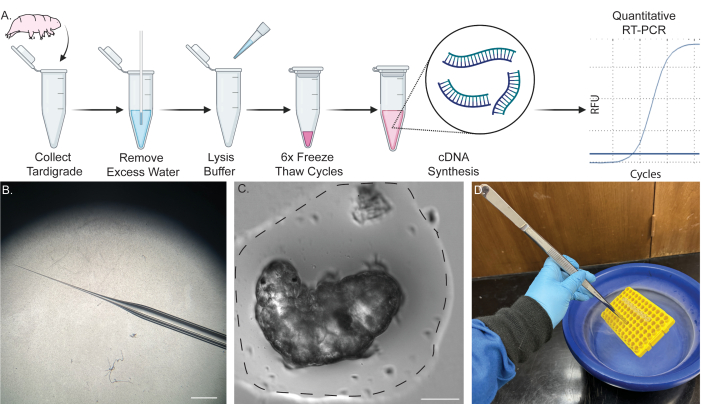
Şekil 1: Tek bir tardigraddan RNA ekstraksiyonu için tek tüplü boru hattı. (A) Altı donma-çözülme döngüsü ve müteakip cDNA sentezi dahil olmak üzere tek bir tardigraddan RNA ekstraksiyonu protokolünü gösteren şema. Örnekler daha sonra RT-PCR ve qRT-PCR için kullanılabilir. (B) Suyun uzaklaştırılması için kullanılan mikropipet konikliğinin görüntüsü. Ölçek çubuğu: 2 mm. (C) Küçük bir su hacmindeki bir tardigradın parlak alan görüntüsü (noktalı çizgi). Başarılı bir ekstraksiyon için çoğu suyun gösterilen ölçüde uzaklaştırılması gerekir ve lizis tamponunun seyreltilmesini önler. Ölçek çubuğu: 50 μm. (D) Numuneleri güvenli bir şekilde hızlı bir şekilde dondurmak-çözmek için uzun forseps kullanarak numunelerin sıvı nitrojene daldırılmasını gösteren resim. İçeriğin bir kısmı BioRender'da oluşturuldu. Kirk, M. (2022) BioRender.com/d93s511 Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
NOT: Şekil 1A, prosedürün bir şemasını göstermektedir. Ayrıntılı tardigrad ve alg kültürleme prosedürleri için daha önce yayınlanmışraporlara bakın 45,46,47.
1. Kaynak suyunun sterilizasyonu
- 5 galonluk bir su sürahisinden 2 L kaynak suyunu (Ayrıntılar için Malzeme Tablosuna bakın) 2 L'lik otoklava dayanıklı bir cam şişeye dökün.
- Kapağı otoklava dayanıklı şişeye yerleştirin ve az miktarda otoklav bandı ile kapatın. Şişeyi sıkmayın; Kapağı üstüne yerleştirin.
- Kaynak suyunu, kurutma adımı olmadan ıslak bir döngüde 50 dakika boyunca otoklavlayın.
- Suyun oda sıcaklığına (RT) gelmesine izin verin ve RT'de saklamadan önce kapağı sıkıca kapatın.
2. Cam mikropipet çekme (pipet çektirme ile)
- Bir cam mikropipeti (OD: 1 mm, ID: 0.58 mm, Uzunluk: 10 cm) bir mikropipet çekiciye sabitleyin. Pipet şeklini değiştireceği ve filamente zarar vereceği için ısıtma filamenti ile temastan kaçının.
- Her filament ve pipet çektirmesi için pipetin çekilme miktarını ampirik olarak belirleyin. Bununla birlikte, optimizasyon için bir başlangıç noktası olarak hizmet etmek için 78 °C ve 182,2 g çekme ağırlığına sahip tek bir çekme adımı kullanın.
- Cam mikropipeti keskin uçlu iki cam mikropipete ayırmak için filamentin ısınmasına ve yerçekimine izin verin (Şekil 1B).
- Bu çekilmiş cam mikropipetleri, yerinde tutmak ve keskin uçların kırılmasını önlemek için balmumu veya kil içeren 100 mm'lik kapalı bir Petri kabında saklayın.
3. Cam mikropipet çekme (pipet çektirme olmadan)
- Düşük ayarda bir Bunsen brülörü veya başka bir kontrollü alev kaynağını yakın.
- Her iki elinizde bir ucu olan bir cam mikropipet alın.
- Cam erimeye başlayana kadar cam mikropipetin ortasını alevin üzerinde tutun. Ardından, iki ucu hızla ayırın. Bu, çok hassas iki keskin uç oluşturacaktır.
- Ucu bir çift steril ince forseps ile hafifçe kırın.
- Bu çekilmiş cam mikropipetleri, yerinde tutmak ve keskin uçların kırılmasını önlemek için balmumu veya kil içeren 100 mm'lik kapalı bir Petri kabında saklayın.
4. RNA ekstraksiyonu
- Kriyo güvenli bir kapta 0,5 L sıvı nitrojen elde edin.
DİKKAT: Sıvı nitrojen kriyojeniktir ve cilde veya gözlere maruz kaldığında yanıklara neden olabilir. Tutarken koruyucu giysi, su sıçratan gözlük, nitril eldiven, kriyo eldiven, laboratuvar önlüğü ve kapalı burunlu ayakkabılar kullanın. Sıvıyı taşımadan önce kabın sıvı nitrojen için güvenli olduğundan emin olun. Bu adım için etanol-kuru buz banyosu kullanmak da mümkün olabilir. - cDNA sentezi ana karışımı yapın: 1 μL rastgele heksamer primer, 2 μL DNaz, 4 μL 5x RT Tamponu, 1 μL Enzim Karışımı, 1 μL H2Ove 1 μL 10 mM dNTP içeren 10 μL'lik bir çözelti. Bu çözeltiyi buz üzerinde saklayın.
- Tardigrad lizis tamponu hazırlayın (5 mM Tris (pH = 8)% 0.5 (h / v) Deterjan 1,% 0.5 (h / v) Deterjan 2, steril nükleaz içermeyen Suda% 0.25 mM EDTA).
NOT: Bu solüsyon 6 ay boyunca tezgah üstünde saklanabilir. Bununla birlikte, steriliteyi koruyun ve potansiyel RNAse kirletici kaynaklardan kaçının. - Ekstraksiyonlar için yeterli lizis tamponu (2 μL / tardigrad).
- Tardigrad lizis tampon çözeltisine RNAse inhibitörü 4 U / μL'lik bir nihai konsantrasyona ekleyin.
- Çözeltiyi buz üzerinde saklamadan önce 5 saniye boyunca 2000 x g hızında bir tezgah üstü santrifüjde çözeltiyi RT'de vorteksleyin ve döndürün.
- Steril filtre uçlu bir P1000 pipet kullanarak deney için gerektiği kadar tardigradı kültürden çıkarın ve bunları 35 mm'lik steril bir Petri kabına yerleştirin.
NOT: Herhangi bir sayıda tardigrad bu şekilde işlenebilir. Genellikle, ekstraksiyon için koşul başına üç tardigrad işlenir. - Tardigradları 1 mL otoklavlanmış steril kaynak suyu ve steril filtre uçlu P1000 pipet kullanarak üç kez yıkayın. Bunları yavaşça yukarı ve aşağı pipetlemek, alg kirleticilerinin giderilmesine yardımcı olur.
- 25x ila 50x büyütmede bir diseksiyon mikroskobu kullanarak, steril filtre uçlu bir P10 pipeti kullanarak bu yıkanmış kültürden tek bir tardigradı yeni bir steril 35 mm Petri kabına aktarın.
- Tek tardigradı 100 μL steril nükleaz içermeyen suda yıkamak için steril filtre uçlu bir P200 pipet kullanın.
NOT: Bu yıkama adımı, ribonükleazlar da dahil olmak üzere kirleticileri daha fazla uzaklaştırmak için kullanılır. - Yıkanmış tardigradı, steril filtre uçlu bir P10 pipeti kullanarak 1-2 μL steril nükleaz içermeyen su içinde temiz, steril bir PCR tüpünün dibine aktarın ve tardigradın ucun kenarına yapışmadığından emin olun.
- Tardigradı 25x büyütmede diseksiyon mikroskobu altında görselleştirin.
- Suyun suyun uzaklaştırılmasını kolaylaştırmak için, çekilen cam mikropipetin ucunu tüpün dışına hafifçe kırın. Deliğin suyu çekecek kadar büyük olduğundan, ancak tardigradı çekmeyecek kadar büyük olduğundan emin olun.
- Çekilmiş bir cam mikropipetin kılcal hareketini kullanarak, hayvan yaklaşık iki tardigrad uzunluğu çapında küçük bir su kabarcığı ile çevrili olana kadar suyu çıkarın.
- Su seviyesinin uygun olduğundan ve tardigradın sulu kaldığından emin olmak için diseksiyon kapsamı aracılığıyla su giderme işlemini izleyin.
NOT: Şekil 1C , ne kadar suyun çıkarılması gerektiğine dair bir örnek sunar. Bu kritik bir adımdır. Kurumasını önlemek için tardigradı küçük bir su kabarcığı çevreleyecektir, ancak lizis tamponunun seyreltilmesini önlemek için mümkün olduğunca fazla su çıkarılmalıdır. Kalan su seviyelerinin bir örneği için lütfen Şekil 1C'ye bakın. - Suyu çıkardıktan hemen sonra, tüpün dibine 2 μL tardigrad lizis tamponu ekleyin, kısaca girdap yapın ve tüpü bir masa üstü santrifüj üzerinde 2000 x g'da 5 saniye boyunca RT'de santrifüjleyin.
- Tardigradları içeren numuneleri hemen bir PCR tüp rafına yerleştirin ve raf tarafından sıkıca tutulduğundan emin olun.
- Bir çift uzun kaba forseps kullanarak rafı kavrayın ve numuneleri içeren rafı tamamen donana kadar sıvı nitrojene hafifçe batırın (Şekil 1D).
- Rafı sıvı nitrojenden çıkarın ve hemen buzun üzerine yerleştirin. Numunenin çözülmesine izin verin (~ 45 s ila 1 dakika sürer). Numuneyi her 15 saniyede bir buzdan çıkararak ve gözle görülür şekilde inceleyerek izleyin. Örnek gözle görülür şekilde şeffaf hale geldikten sonra bir sonraki adıma geçin.
- 4.18-4.19 adımlarını beş kez daha tekrarlayın. Maksimum lizis ve ekstraksiyon için toplam altı donma-çözülme döngüsü gereklidir (Şekil 2A,B).
- Donma-çözülme tamamlandığında, numuneleri buzun üzerine yerleştirin ve hemen bir sonraki adıma geçin. Numuneleri bu noktada depolama için dondurmayın, çünkü bu, cDNA hazırlığı için mevcut RNA'yı azaltacaktır.
5. cDNA sentezi
- Tardigrad lizat içeren PCR tüpüne 2 μL cDNA sentezi ana karışımı ekleyin. Tüpü kısa bir süre hafifçe vurun ve numuneleri buz üzerinde değiştirmeden önce bir masa üstü santrifüj ile 5 saniye boyunca 2000 x g'da RT'de döndürün.
- Numuneleri bir termodöngüleyiciye yerleştirin ve 25 ° C'de 10 dakika boyunca tavlama primerlerine, ters transkripsiyon gerçekleştirmek için 55 ° C'de 30 dakika boyunca inkübe edin ve son olarak enzimleri 85 ° C'de 5 dakika boyunca ısıyla inaktive edin.
- İnkübasyondan sonra, tüpü hemen buzun üzerine yerleştirin ve 21 μL steril nükleaz içermeyen su ekleyerek numuneyi toplam 25 μL hacme seyreltin. Düşük kopya sayılı transkriptler için, bu seyreltme adımını ampirik olarak belirlendiği şekilde değiştirin.
6. qPCR (Kısmi Çözünürlük)
- Daha büyük miktarlarda tardigraddan hazırlanan toplam RNA'yı kullanarak astar setinin tavlama sıcaklığını belirleyin, örneğin, Boothby, 201843'te sunulan toplu ekstraksiyon yöntemi.
- qRT-PCR'yi çalıştırmadan önce optimum tavlama sıcaklığını belirlemek için bir PCR sıcaklık gradyanı çalıştırın (bu protokolde kullanılan tüm PCR ayarları için Tablo 1 ve Tablo 2'ye bakın).
- Bir tüp indikatör boya süper karışımını buz üzerinde çözdürün ve ışıktan izole edin. Buz üzerine 96 oyuklu bir qPCR plakası yerleştirin ve istenen kuyucuk sayısına 5 μL süper karışım, 2 μL su, her bir primerden 1 μL (10 μM) ve 1 μL cDNA ürünü yerleştirin.
- PCR plakasını plaka contası ile kapatın ve astar seti için uygun bir tavlama sıcaklığı kullanarak qRT-PCR'yi çalıştırın (bu belgede kullanılan tüm qRT-PCR ayarları için Tablo 3'e bakın).
7. Nicelik belirleme ve sonuçların yorumlanması
- Sonuçları kantitatif olarak, ifadesinin empoze edilen koşullar boyunca sabit olması beklenen bir veya daha fazla kontrol temizlik geni ile karşılaştırın. Bu çalışma için aktin geni kullanıldı.
- Her kuyucuk için Ct değerlerini veya döngü eşiğini elde edin ve kontrol temizlik gen reaksiyonlarının Ct değerleriyle karşılaştırın. Aşağıdaki denklemi kullanarak gen ifadesindeki kıvrım değişimini hesaplayın:
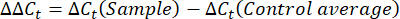
NOT: Fold gen ekspresyonu her transkript ve tardigrad için 2- (ΔΔCt)48 olarak çizilir. - Ct değerinden transkript numarasının kabaca bir tahminini elde etmek için aşağıdaki denklemi kullanın:
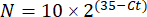
N, transkript sayısıdır ve 2, varsayılan PCR verimliliği veya PCR48 döngüsü başına floresanstaki kat artışıdır.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Tek tardigrad RNA ekstraksiyonunun geliştirilmesi ve optimizasyonu
Tardigradlarda RNA ekstraksiyonu için Ly ve ark., 201542 protokolünü uyarlayan STST sistemi, preparatın miktarını ve kalitesini en üst düzeye çıkarmak için optimize edilmiştir (Şekil 1A). RT-PCR, aktin transkriptleri için gerçekleştirildi ve ekzon 1 ve 2'yi kapsayan 527 bp'lik bir bölgeyi amplifiye ederek transkript verimini ölçt...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Bu çalışma, tek tardigrad qRT-PCR için RNA'nın ekstraksiyonu için etkili bir yöntem sunmaktadır. STST metodolojisini mevcut tek bir tardigrad RNA ekstraksiyon kiti ile doğrudan karşılaştırmak, STST RNA ekstraksiyonunun >200 kat daha yüksek miktarlarda aktin RNA transkripti sağladığını, maliyeti numune başına bir doların altına düşürdüğünü ve ekstraksiyon için gereken süreyi %30 oranında azalttığını ortaya koydu. STST'yi ilgili bir biyolojik soruya uyg...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarlar ifşa etmek için herhangi bir çıkar çatışması beyan etmezler.
Teşekkürler
NIH Ruth Kirschstein Bursu # 5F32AG081056-02 ve Dr. Molly J. Kirk'ü destekleyen Errett Fisher Doktora Sonrası Bursu, Chaoming Xu'yu destekleyen Crowe Ailesi Bursu ve California Üniversitesi, Santa Barbara Akademik Senato Bursu ve bu araştırma çabalarını destekleyen NIH hibeleri #R01GM143771 ve #2R01HD081266. Yazarlar ayrıca, Kaliforniya Üniversitesi, Santa Barbara ve Kaliforniya Üniversitesi, Rektörlük Ofisi tarafından desteklenen Kaliforniya Nanosistemler Enstitüsü bünyesindeki Biyolojik Nanoyapılar Laboratuvarı'nın kullanımını da kabul etmektedir.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
Referanslar
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890(2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884(2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333(2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621(2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330(2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495(2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245(2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, Ł The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983(2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522(2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120(2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298(2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405(2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143(2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266(2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663(2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168(2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063(2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24(2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888(2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- Goldstein, B. Hypsibius dujardini. collection notes and culture protocol from Bob McNuff. , At http://tardigrades.bio.unc.edu/protocols/CollectionCulture.pdf (2007).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508(2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424(2009).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır