Method Article
Maya Yüzey Ekranı ile Protein Mühendisliği
Bu Makalede
Özet
Bu protokol, ilgilenilen bir antijene bağlanan protein varyantlarını zenginleştirmek için maya yüzeyi teşhir seçimi kampanyalarının yürütülmesi için temel adımları açıklar.
Özet
Protein mühendisliği, belirli bir proteinin mevcut fonksiyonlarının iyileştirilmesini veya yeni fonksiyonların üretilmesini sağlar. Protein mühendisliği alanında en yaygın kullanılan ve çok yönlü araçlardan biri, maya yüzeyinde rastgele bir protein havuzunun eksprese edildiği maya yüzey ekranıdır. Fenotip (örneğin, maya ile görüntülenen proteinin ilgilenilen antijene bağlanması) ve genotipin (protein varyantını kodlayan plazmit) bağlantısı, bu kütüphanenin istenen özellikler için seçilmesini ve ardından zenginleştirilmiş varyantların dizilenmesini sağlar. Manyetik boncuk seçimi ile akış sitometrik sıralamayı birleştirerek, bir hedef antijene daha iyi bağlanmış protein varyantları seçilebilir ve zenginleştirilebilir. Özellikle, afinite olgunlaşmasına ek olarak, bir hedefe bağlanma, herhangi bir ilk bağlanma afinitesi olmadan da elde edilebilir. Burada, bir maya yüzeyi teşhir seçimi kampanyasının tüm önemli kısımlarını kapsayan ve tipik maya yüzeyi teşhir sonuçlarına örnekler veren adım adım bir protokol sunuyoruz. Maya yüzey görüntülemesinin, akış sitometrisine erişimi olan herhangi bir moleküler biyoloji laboratuvarında kurulabilecek, geniş çapta uygulanabilir ve sağlam bir yöntem olduğunu gösteriyoruz.
Giriş
Maya yüzey gösterimi, protein mühendisliği alanındaki en önemli teknolojilerden biridir. Geliştirilmiş afinite veya stabilite gibi istenen özelliklere sahip protein varyantlarının seçilmesini sağlar. İlk olarak 1997'detanıtılan 1, faj ekranı 2,3, ribozom ekranı4 ve memeli hücresi ekranı 5,6,7'nin yanı sıra en yaygın kullanılan görüntüleme teknolojilerinden biridir. İlgilenilen protein (POI), proteinleri tutturmak için kaynaştırılarak maya hücrelerinin yüzeyinde görüntülenir. Bir dizi farklı çapa proteini mevcuttur ve en yaygın olarak POI, maya aglütininin çiftleşme proteini Aga2p 1,8'in C-terminaline kaynaştırılır. Ek olarak, POI tipik olarak, floresan etiketli antikorlar ve akış sitometrisi kullanılarak ekran seviyesinin tespit edilmesini sağlayan bir hemaglutinin etiketi (HA-etiketi) ve c-myc etiketi gibi iki etiketle çevrilidir (Şekil 1A). Tipik maya seçimi kampanyaları, manyetik boncuk seçimleri ve akış sitometrik sıralamasının bir kombinasyonunu içerir. Boncuk seçimleri, büyük hücre sayılarının işlenmesini ve hedef antijene bağlanan protein varyantlarının zenginleşmesini sağlar, çünkü antijen yüklü boncuklarla çok değerlikli etkileşimler avidite etkilerine yol açar ve bu nedenle düşük afiniteli varyantların kaybını önler (Şekil 1B). Akış sitometrik analizi ve seçimi, görüntülenen POI varyantlarının etiketli antijene bağlanmasını görselleştirme avantajı sunar. Sonuç olarak, bağlayıcı popülasyonlar sıralanabilir ve yetiştirilebilir, bu da birkaç sıralama turu boyunca istenen özelliklere sahip protein varyantlarının zenginleşmesine yol açar. Ayrıca, çeşitliliği ve dolayısıyla proteinin afinitesine ve/veya stabilitesine katkıda bulunan ek mutasyonlar bulma olasılığını daha da artırmak için ek rastgele mutajenez turları gerçekleştirilebilir.
Maya yüzey gösterimi, (a) oksidatif protein katlanmasının yanı sıra ökaryotik post-translasyonel modifikasyonları (N-glikosilasyon gibi) sağlayan ökaryotik ekspresyon mekanizması, (b) proteini çevreleyen iki peptit etiketinin tespiti nedeniyle ekspresyon normalizasyonu, (c) akış sitometrisi ile seçim ilerlemesinin görsel olarak incelenmesi (örneğin, bağlanma hücrelerinin yüzdesi ve bağlanma yoğunluğu) ve (d) tek tek protein mutantlarını analiz etme olasılığı gibi belirli avantajlar sunar. maya (örneğin, termostabilitenin yanı sıra afinitenin analiz edilmesi), zahmetli protein ekspresyonu ve saflaştırılmasına zaman kazandıran bir alternatif sunar9. Aslında, maya yüzeyinde görüntülenen proteinlerin hem afiniteleri (KDdeğerleri) hem de stabiliteleri (T50 değerleri), biyofiziksel yöntemler ve çözünür proteinler 9,10,11,12 kullanılarak elde edilen verilerle iyi korelasyonlar göstermiştir. Maya yüzey gösterimi, antikor fragmanları 13,14,15,16, 10. tip III fibronektin alanı 17,18, rcSso7d 19,20 veya knottinler21 gibi çeşitli proteinlerin mühendisliği için kullanılmıştır.. Benzer şekilde, randomize pozisyonları ve amino asit kodon kullanımını değiştirerek maya kütüphanesi tasarımlarını optimize etmek için kapsamlı araştırmalar yapılmıştır 17,22,23. Maya yüzey gösteriminin stabilite mühendisliği 14,15,24,25, afinite 18,26,27, enzimatik aktivite 28,29,30,31 ve protein ekspresyonu32 için başarılı olduğu kanıtlanmıştır. Ek olarak, küçük bir molekülün varlığında veya yokluğunda koşullu bağlanma gibi daha karmaşık uygulamalar, maya yüzey ekranı20 kullanılarak gerçekleştirildi.
Bu protokolde, küçük molekül A112020'nin varlığında antijen insan retinol bağlayıcı protein 4'e (hRBP4) karşı seçilen G4 kütüphanesi (10. tip III fibronektin alanı, Fn3'e dayalı) örneği ile maya yüzeyi gösterimli bir seçim kampanyası için tüm temel adımları açıklıyoruz. Bu seçim, moleküler bir anahtar olarak kullanılabilecek küçük bir moleküle bağlı olan bir protein-protein etkileşimi elde etmek için gerçekleştirildi. Dikkat çekici bir şekilde, maya yüzey gösterimi ile alternatif yaklaşımlar mümkün olsa da, tipik maya seçimleri genellikle daha önce herhangi bir bağlanma afinitesi olmadan bir hedef antijene bağlanmayı hedefler. Bir maya kütüphanesinin yetiştirilmesini, boncuk seçimlerini, akış sitometrik sıralamasını ve hataya açık PCR (epPCR) ile afinite olgunlaşmasını içeren bir maya seçimi kampanyasının tüm adımlarını kapsıyoruz. Bu nedenle, bu protokol önceki maya yüzeyi görüntüleme protokollerini33,34 tamamlar ve herhangi bir maya kütüphanesi ve tercih edilen hedef antijen ile maya yüzeyi görüntüleme seçimleri (Şekil 1) için bir temel olarak kullanılabilir.
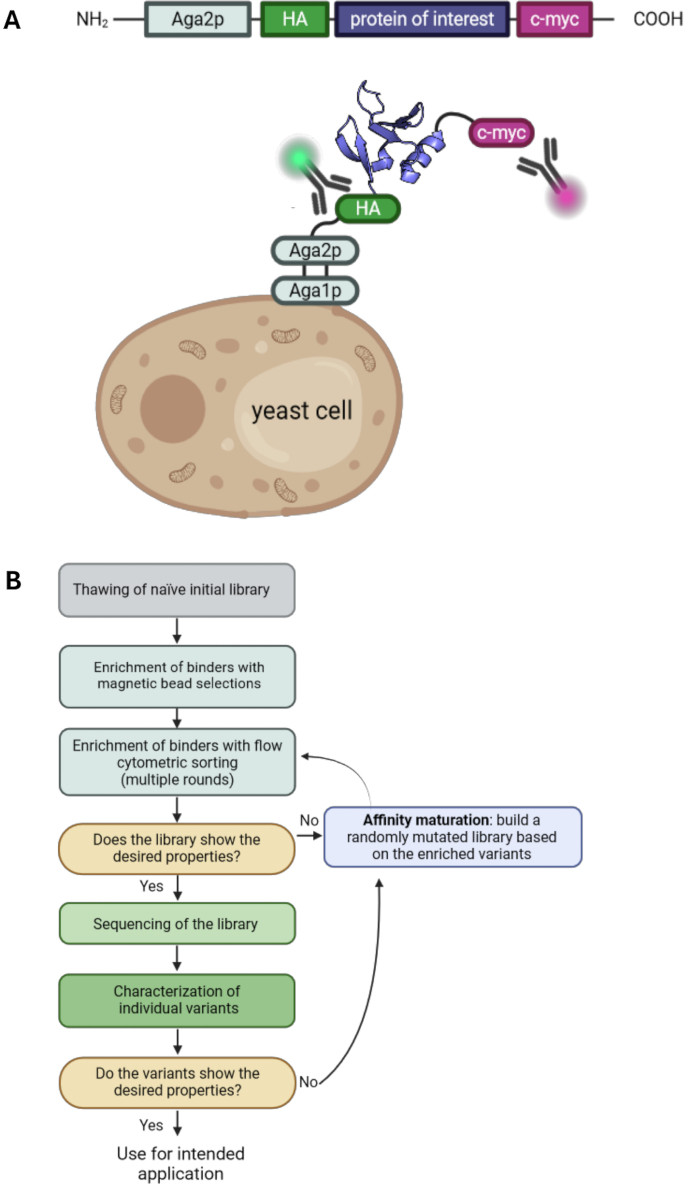
Şekil 1: Maya yüzeyi görüntüleme prensibi ve maya yüzeyi görüntüleme seçimleri için tipik bir iş akışı. (A) POI, bir maya yüzeyi görüntüleme vektörüne klonlanır ve tipik olarak bir N-terminali HA- ve bir C-terminali c-myc-etiketi ile çevrilidir. Yapı, yüzeyde sergilenmek üzere maya çiftleşme proteini Aga2p ile kaynaştırılır. Gösterilen protein, PDB ID: 6QBA20'den tasarlanmış bağlayıcı "RS3" dür. (B) Boncuk seçimleri ve akış sitometrik sıralaması ile protein varyantlarının istenen özelliklerle zenginleştirilmesini ve ayrıca afinite olgunlaşması için epPCR'yi birleştiren maya yüzeyi görüntüleme seçimi kampanyaları için tipik bir iş akışını gösteren akış şeması. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protokol
1. Maya kütüphanelerinin çözülmesi ve yetiştirilmesi
- Dondurulmuş maya kütüphanesinin yeterli miktarda alikotunu RT'de çözün ve hemen SD-CAA'da (bakınız Tablo 1) yaklaşık 1'lik bir OD600'e seyreltin (bu, 1 x 107 hücre / mL'ye karşılık gelir).
NOT: Kitaplığın çeşitliliğini en az 10 kat kapsadığınızdan emin olun. Bu nedenle, kitaplık boyutu 2,5 x 108 klonu oluşturuyorsa (bu, bu örnekte kullanılan G4 kitaplığının çeşitliliğidir), 1'in OD600'üne ulaşmak için 250 mL SD-CAA'da en az 2,5 x 109 hücre aşılayın. Bu protokol aynı zamanda sıvı kültürlerden de başlatılabilir (bir kütüphane ile taze dönüştürülmüş maya hücreleri). - Canlı hücrelerin sayısını değerlendirin.
- Bu maya süspansiyonundan (OD600 /1'de) 100 μL'lik bir alikot alın ve mikrosantrifüj tüplerinde 900 μL SD-CAA ortamı ile 1:10'luk bir seyreltme serisi gerçekleştirin.
- SD-CAA plakalarında 10-5'e kadar seyreltmeler hazırlayın ve 100 μL seyreltme 10-3, 10-4 ve 10-5'i plakalayın (Tablo 1). Plakaları 30 °C'de 2-3 gün inkübe edin. Çözüldükten sonra canlı hücre sayısının kütüphane çeşitliliğini en az 10 kat aştığından emin olun.
- Bu arada, kalan hücre süspansiyonu ile devam edin ve G4 kütüphanesini içeren maya kültürünü, çalkalarken (180 rpm) 30 ° C'de gece boyunca cam şişelerde yetiştirin. Gece boyunca inkübasyondan sonra, bölüm 2 (Maya yüzeyinde protein ekspresyonunun indüksiyonu) ile devam edin.
- 2-3 günlük inkübasyondan sonra, SD-CAA plakalarındaki kolonileri görsel inceleme ile sayın. Çözüldükten sonra canlı hücrelerin nihai sayısını hesaplarken, seyreltme faktörünü ve kaplamanın (yani 100 μL) orijinal kültür hacmine (yani 250 mL) oranını göz önünde bulundurun.
Canlı hücreler/mL = plakadaki koloni sayısı × seyreltme faktörü
Canlı hücreler (toplam) = (canlı hücreler/mL) × kültür hacmi
| Orta/tampon | Parça | Konsantrasyon [g/L] | Yorumlar/Açıklama | |||
| SD-CAA (SD-CAA) | D-glikoz | 20 | Tüm ortam bileşenlerini 1000 mL ddH2O ve steril filtratta tek kullanımlık 0,22 μm steril filtrelerle çözün. | |||
| Maya azot bazı | 6.7 | |||||
| Casmino asitleri | 5 | |||||
| Sitrik asit monohidrat | 7.4 | |||||
| Tri-sodyum sitrat dihidrat | 10.83 | |||||
| SG-CAA | D-galaktoz | 20 | Tüm ortam bileşenlerini 1000 mL ddH2O ve steril filtratta tek kullanımlık 0,22 μm steril filtrelerle çözün. | |||
| D-glikoz | 2 | |||||
| Maya azot bazı | 6.7 | |||||
| Kaamino asitler | 5 | |||||
| Di-sodyum hidrojen fosfat heptahidrat | 10.2 | |||||
| Sodyum dihidrojen fosfat monohidrat | 8.56 | |||||
| SD-CAA plakaları | Sorbitol | 182 | Sorbitol, di-sodyum hidrojen fosfat heptahidrat, sodyum dihidrojen fosfat monohidrat ve agar-agar'ı 900 mLddH2Ove otoklavda çözün. Kalan bileşenleri 100 mL ddH2O'da çözün ve steril bir şekilde süzün ve otoklavlanmış ortam ılık olduğunda ekleyin. | |||
| Di-sodyum hidrojen fosfat heptahidrat | 10.2 | |||||
| Sodyum dihidrojen fosfat monohidrat | 7.44 | |||||
| Ağar-agar | 15 | |||||
| D-glikoz | 20 | |||||
| Maya azot bazı | 6.7 | |||||
| Kaamino asitler | 5 | |||||
| YPD (YPD) | Pepton | 20 | 10x D-glikoz stoğu (200 g/L) hazırlayın ve tek kullanımlık 0,22 μm steril filtrelerle steril süzüntü hazırlayın. Pepton ve maya ekstraktını 900 mL ddH2O ve otoklavda çözün. Ilık olduğunda 100 mL 10x D-glikoz ekleyin. | |||
| Maya özü | 10 | |||||
| D-glikoz | 20 | |||||
| YPD plakaları | Pepton | 20 | 10x D-glikoz stoğu (200 g/L) hazırlayın ve tek kullanımlık 0,22 μm steril filtrelerle steril süzüntü hazırlayın. Pepton, maya özütü ve agar-agar'ı 900 mL ddH2O ve otoklavda çözün. Ilık olduğunda 100 mL 10x D-glikoz ekleyin. | |||
| Maya özü | 10 | |||||
| D-glikoz | 20 | |||||
| Ağar-agar | 15 | |||||
| PBSA (Radyo ve Orman | BSA | 1 | BSA'yı PBS'de çözün ve tek kullanımlık 0,22 μm steril filtrelerle steril süzün. | |||
Tablo 1: Ortam ve tampon bileşimi.
2. Maya yüzeyinde protein ekspresyonunun indüksiyonu
- Gece boyunca inkübasyondan sonra, maya kültürleri tipik olarak 2-20 arasında bir OD600'e ulaşır. Maya kültürünün yoğunluğunu belirlemek için OD600'ü bir fotometre ile ölçün.
NOT: OD ölçümleri yalnızca 0.1-1 aralığında doğrusaldır. Bu nedenle, OD 600'ü belirlemek için SD-CAA'da seyreltmeler (tipik olarak 1:20 ve 1:50) gerçekleştirin.- SD-CAA'daki hücreleri 1 OD600'e seyreltin ve çeşitliliğin 10 kat kaplandığından emin olun (yani, G4 kütüphanesinin seyreltilmesi için 2,5 x 109 hücre kullanılır). Hücrelerin üstel bir büyüme aşamasına ulaşmasını sağlamak için çalkalarken 30 °C'de inkübasyona devam edin.
- Gece boyunca kültürden maya kütüphanelerinin (% 15 gliserol içeren SD-CAA'da) gliserol stoklarını (çeşitliliği 50x kapsayan) hazırlayın. Bu amaçla, gerekli sayıda maya hücresini (2000 x g, 3 dk, 4 °C) santrifüjleyin, süpernatanı atın ve hücre peletini %15 gliserol ile desteklenmiş 1 mL SD-CAA ortamında yeniden süspanse edin. Gliserol stoklarını hemen -80 °C'de dondurun.
NOT: Maya kültürleri dondurulmadan önce durağan bir faza büyütülürse, maya hücrelerinin canlılığı artar. Daha sonraki seçim turlarında, kütüphanelerin çeşitliliği daha düşük olduğunda, tipik olarak 700 μL maya kültürü kullanmak ve bunu% 50 gliserol ile desteklenmiş 300 μL SD-CAA ile karıştırmak yeterlidir. Burada, maya kültürleri tipik olarak durağan fazda OD600 değerlerini 15 olarak gösterir, ancak gerçek değer kullanılan fotometreye bağlı olabilir. Böylece, durağan faz tekrarlanan OD600 ölçümleri ile belirlenebilir. OD600 değeri 2 saatlik bir inkübasyon süresi boyunca artmazsa, durağan bir faza ulaşılır.
- Maya kültürünün seyreltilmesinden 4-6 saat sonra 5 dakika, 2000 x g'da 10x çeşitliliği kapsayacak şekilde uygun bir hücre hacmini santrifüjleyin (tipik OD600 değerleri 4-6 arasındadır) ve süpernatanı atın.
- Hücre peletini SG-CAA'da yeniden süspanse edin (bkz. Tablo 1) OD600 /1 elde etmek için ve taze bir cam şişeye aktarın.
NOT: Galaktoz içeren bir ortamda yeniden süspansiyon, galaktozla indüklenebilen promotor tarafından yüzeydeki proteinin ekspresyonunu indükler. - POI'nin yüzey ekspresyonunu indüklemek için maya kültürlerini gece boyunca 20 °C'de inkübe ederken 180 rpm'de çalkalayın. Gece boyunca indüksiyonu takiben, maya kültürleri tipik olarak 2-5 arasında OD600 değerlerine ulaşacaktır; indüksiyon sıcaklığı 37 °C'ye yükseltilirse, OD600 değerlerinin 4-10 olmasını bekleyin.
NOT: Sıcaklığın 20°C'ye düşürülmesi hücre büyümesini yavaşlatır ve tipik olarak daha iyi görüntü seviyeleri sağlar. Bununla birlikte, 37 ° C'de indüksiyon, stabilize mutantlara doğru seçim basıncını artırmak için yararlı olabilir35.
3. Maya kütüphanelerinin ilk boncuk seçim turu (pozitif seçim)
NOT: Standart bir boncuk seçim prosedürü 6 adımdan oluşur (Tablo 2).
| Gün | Adım | |
| 0 | Gecelik kültür | |
| 1 | Maya hücrelerinin yüzeyinde protein ekspresyonunun indüksiyonu | |
| 2 | 1 pozitif seçim ile ilk boncuk seçimi | |
| 3 | Boncukların çıkarılması, pasajlanması, maya hücrelerinin yüzeyinde protein ekspresyonunun indüklenmesi ve kütüphanenin dondurulması | |
| 4 | 3 negatif ve 1 pozitif seçim ile ikinci boncuk seçimi | |
| 5 | Boncukların çıkarılması ve kütüphanenin dondurulması | |
Tablo 2: Bir maya kütüphanesinin boncuk seçimlerinin yürütülmesi için tipik zaman çizelgesi.
- Boncukların hazırlanması
- İlk boncuk seçimi için boncukları hazırlayın (Malzeme Tablosuna bakın): Her tüp için, 10 μL biyotin bağlayıcı manyetik boncukları (4 ×10 5 boncuk/μL) 990 μL PBSA (Tablo 1) içinde yeniden süspanse edin yıkamak için, tüpü kapak açıkken 2 dakika boyunca manyetik bir rafa yerleştirin. Süpernatanı dikkatlice çıkarın. Yıkama adımını 1 mL PBSA ile tekrarlayın.
NOT: Aynı biyotinile antijene sahip birden fazla boncuk tüpüne ihtiyaç duyulursa, tek tip bir antijen-boncuk çözeltisi elde etmek için bu adımda tek bir tüpte hazırlanabilirler. Bu durumda, bir sonraki adımda antijen miktarının buna göre artırılması gerekir. - Boncukları, 1.5 mL'lik bir mikrosantrifüj tüpünde 6.7-33 pmol biyotinile antijen (burada: 0.125 mg / mL konsantrasyonda 6.86 μL hRBP4) ile toplam 1 mL PBSA hacminde yeniden süspanse edin.
NOT: 6.7 pmol antijen yeterli iken, antijen sınırlayıcı değilse 33 pmol kullanılmalıdır. Seçim, bu durumda olduğu gibi küçük moleküllü bir ilaca veya liganda bağlı bir antijen için yapılırsa, bu ilaç/ligand seçim sırasında mevcut olmalı ve bu adımda eklenmelidir (burada: 5 μM A1120). - Hafifçe çalkalama veya döndürme ile 4 ° C'de 2 saat inkübe edin.
NOT: Bu adım sırasında hücreler hazırlanabilir. - İnkübasyondan sonra ve hücrelere eklenmeden önce, tüpü kapak açıkken 2 dakika boyunca manyetik bir rafa yerleştirin. Süpernatanı çıkarın ve antijen yüklü boncukları yukarıda tarif edildiği gibi 1 mL PBSA ile yıkayın. Yıkama adımını tekrarlayın.
NOT: Bir ilaca veya liganda bağlı bir antijen seçilirse, yıkama tamponu bu ilacı veya ligandı fazla miktarda içermelidir (burada: 5 μM A1120). - Antijen yüklü boncukları 50 μL PBSA içinde yeniden süspanse edin (varsa ilaç veya ligandın varlığında).
NOT: Aynı antijen yüklü boncuklar birden fazla tüp için hazırlanmışsa, hacmi buna göre ayarlayın (G4 kütüphanesi için, buradaki son hacim iki tüp için 100 μL veren boncuklardır).
- İlk boncuk seçimi için boncukları hazırlayın (Malzeme Tablosuna bakın): Her tüp için, 10 μL biyotin bağlayıcı manyetik boncukları (4 ×10 5 boncuk/μL) 990 μL PBSA (Tablo 1) içinde yeniden süspanse edin yıkamak için, tüpü kapak açıkken 2 dakika boyunca manyetik bir rafa yerleştirin. Süpernatanı dikkatlice çıkarın. Yıkama adımını 1 mL PBSA ile tekrarlayın.
- Maya hücrelerinin hazırlanması
- Yukarıda tarif edildiği gibi indüklenmiş maya kültürünün OD600'ünü ölçün.
- Kütüphanenin 10 katı çeşitliliği (2000 x g, 10 dk, 4 °C) kaplayacak kadar yeterli hücreyi peletleyin. G4 kitaplığı için, santrifüjleme için 2,5 x 109 hücre kullanın. Süpernatanı çıkarın ve hücreleri 10 mL PBSA ile yıkayın.
- Tekrar santrifüjleyin (2000 x g, 10 dk, 4 °C) ve hücreleri uygun PBSA hacminde (burada: 2 mL) 1.25 x 109 hücre / mL konsantrasyona yeniden süspanse edin ve bir mikrosantrifüj tüpüne aktarın.
NOT: PBSA'nın hacmi hücre sayısına bağlıdır. - Santrifüjleyin (2000 x g, 3 dk, 4 °C) ve 950 μL PBSA'da yeniden süspanse edin.
- Maya hücrelerinin antijen yüklü boncuklarla inkübasyonu
- 950 μL hücre süspansiyonuna 50 μL antijen yüklü boncuk ekleyin ve çalkalama veya döndürme sırasında 4 ° C'de 2 saat inkübe edin.
- İnkübasyondan sonra, hücre-antijen-boncuk çözeltisini kapağı açık olarak manyetik bir rafa yerleştirin. Tüm maya kültürünü manyetik seçime tabi tutmak için sıvıyı kapaktan tüpe pipetleyin. Bağlanmamış hücreleri dikkatlice atmadan önce 2 dakika bekleyin. Hemen 1 mL PBSA ile yıkayın.
NOT: Seçimler bir ilaç/ligand varlığında yapılırsa - bu örnekte A1120 - bunun PBSA'da bulunması gerekir. - PBSA ile yıkama adımından sonra, kurumasını önlemek için hücreleri 1 mL SD-CAA içinde hızla yeniden süspanse edin.
- 200 mL SD-CAA ekleyin ve yukarıda tarif edildiği gibi seri seyreltmeler gerçekleştirin. SD-CAA plakalarına 100 μL koyun ve yeni maksimum çeşitliliği belirlemek için 30 °C'de 2-3 gün inkübe edin. Burada, ilk boncuk seçiminden sonra G4 kütüphanesinin çeşitliliği 1.25 x106 hücredir.
NOT: Orijinal kitaplık bu seçim turunda 10 kat fazla örneklendiğinden, çoğu klonun seçimden sonra zenginleştirilmiş havuzda birden çok kez bulunacağını varsaymak mantıklıdır. Sonuç olarak, gerçek çeşitlilik daha düşük olacaktır ve bu sayı maksimum değer olarak kabul edilmelidir. Genellikle, bu ilk seçim turunda çeşitlilik en az 100 kat azaltılır. Kalan kültürü (200 mL) 30 ° C'de çalkalayarak (180 rpm) en az 16 saat inkübe edin. Protokol, hücreleri 4 ° C'ye yerleştirerek gece boyunca inkübasyondan sonra duraklatılabilir.
4. Boncukların çıkarılması ve bir sonraki boncuk seçim turundan önce ekim yapılması
- OD600'ü yukarıda açıklandığı gibi ölçün. Çeşitliliği en az 10 kat (2000 x g, 3 dk, RT) kaplamak için yeterli hücreleri peletleyin. Süpernatanı atın ve hücreleri 1 mL SD-CAA'da yeniden süspanse edin.
- Bir mikrosantrifüj tüpüne aktarın ve kapak açıkken 2 dakika boyunca manyetik bir rafa yerleştirin. Bağlanmamış hücreleri yeni bir mikrosantrifüj tüpüne aktarın ve ayırma işlemini tekrarlayın.
- Bağlanmamış hücreleri 100 mL SD-CAA içinde seyreltin ve 3-4 OD600'e ulaşılana kadar 180 rpm'de çalkalayarak 30 ° C'de inkübe edin.
NOT: Boncukların çıkarılmasından önce OD600 4'ten düşükse, hücreler inkübasyon olmadan doğrudan sonra indüklenebilir. - 3-4'lük bir OD600'e ulaşıldığında, çeşitliliği en az 10x (2000 x g, 3 dk, RT) kapsayacak şekilde yeterli sayıda hücreyi santrifüjleyin ve protein ekspresyonunun indüksiyonu için SG-CAA'da 1'in OD600'üne yeniden süspanse edin ve gece boyunca 180 rpm'de çalkalarken 20 ° C'de inkübe edin.
- Kalan SD-CAA maya kültürü durağan büyüme fazına ulaştığında (OD600 15'in üzerinde), çeşitliliği 50x (2000 x g, 3 dk, RT) kaplamak için yeterli hücreyi peletleyin. Hücreleri santrifüjleme (2000 x g, 3 dakika) ile dondurun, ardından hücre peletinin %15 gliserol içeren SD-CAA'da yeniden süspansiyonu ve -80 °C'de saklanması.
5. 3 negatif ve 1 pozitif seçim ile ikinci boncuk seçim turu
- Hücreleri ve antijen boncuklarını yukarıda tarif edildiği gibi hazırlayın. Negatif seçimler için çıplak boncuklardan oluşan bir çözelti (antijen eklemeden) hazırlayın.
NOT: 3 negatif seçim yapıldığından, boncuklar her üç tur için de birlikte hazırlanabilir. - Yıkadıktan sonra, antijen boncuklarını 50 μL PBSA'da yeniden süspanse edin ve çıplak boncukları 150 μL PBSA'da yeniden süspanse edin (eğer üç negatif seçim için de hazırlanmışlarsa).
- İlk negatif seçim için, PBSA'daki 950 μL yıkanmış hücreye 50 μL yıkanmış çıplak boncuk ekleyin ve 4 ° C'de 1,5 saat inkübe edin.
- İnkübasyondan sonra, çıplak boncuk hücreli süspansiyonları içeren tüpleri, kapağı açık olacak şekilde manyetik bir rafa yerleştirin. Kapaktaki sıvıyı tüpe pipetleyin ve 2 dakika bekleyin. Bağlanmamış hücreleri yeni bir mikrosantrifüj tüpüne aktarın ve 50 μL yıkanmış çıplak boncuk ekleyin.
- İki tur daha negatif seçim için prosedürü tekrarlayın. Bu arada, pozitif seçim için antijen yüklü boncuklar hazırlayın.
- 3 negatif seçim turundan sonra, hücrelere 50 μL antijen yüklü boncuk çözeltisi ekleyin ve 4 ° C'de 2 saat inkübe edin.
- Antijen yüklü boncukları içeren hücreleri, kapağı açık olacak şekilde manyetik bir rafa yerleştirin ve kapaktaki herhangi bir sıvıyı tüpe pipetleyin. İlişkisiz hücreleri atmadan önce 2 dakika bekleyin.
- Kalan tüm adımları ilk antijen boncuğu seçimi için açıklandığı gibi gerçekleştirin. İlk pozitif seçimden (adım 3.3.2) tek fark, yıkama adımlarının sayısıdır: PBSA ile bir kez yıkamak yerine, seçimin sıkılığını artırmak için boncuk hücresi süspansiyonunu PBSA ile iki kez yıkayın.
NOT: Genellikle, çeşitlilik tekrar 10x-100x oranında azaltılır. Bu ikinci boncuk seçiminden sonra G4 kütüphanesinin maksimum çeşitliliği 3.45 x 105 hücreydi. Yukarıda belirtildiği gibi, kütüphanenin aşırı örneklenmesi nedeniyle gerçek çeşitliliğin daha düşük olması beklenmektedir.
6. Akış sitometrik sıralama ile kütüphane seçimi
- SG-CAA'da yüzey ekspresyonunun gece boyunca indüksiyonundan sonra, yukarıda tarif edildiği gibi OD600'ü ölçün. Çeşitliliğin 10 katını kaplamak için yeterli hücreleri pelet haline getirin (2000 x g, 5 dk, RT). Süpernatanı atın.
- Peleti PBSA'da yeniden süspanse edin ve mikrosantrifüj tüplerine aktarın. Her tüp için boyama için 3 x 107 hücre kullanın. Çeşitliliğe bağlı olarak gerektiği kadar tüp hazırlayın. Antijen olmadan boyama için bir kontrol tüpü hazırlayın.
NOT: Örneğin, 30 x 106 hücrelerin sıralanması gerekiyorsa, görünür bir hücre peleti elde etmek ve sonraki adımlarda hücre kaybını önlemek için 300 x 106 hücreyi pelet haline getirin. 5 mL PBSA'da yeniden süspanse edin ve her biri 500 μL'lik 3 ila 4 alikotu mikrosantrifüj tüplerine aktarın. Boyama işlemi sırasında hücrelerin bir kısmı kaybedileceğinden, sıralanması gereken sayıdan yaklaşık 3 kat daha fazla hücre hazırlayın. Seçim bir ilaç / ligand varlığında gerçekleştirilirse, sadece antijen ile ancak ilaç / ligand olmadan ek bir kontrol hazırlayın. - Tekrar santrifüjleyin (2000 x g, 5 dk, RT), peleti antijen içeren 200 μL PBSA (5 μM A1120 ile hRBP4) içinde yeniden süspanse edin ve 4 ° C'de 1 saat inkübe edin.
NOT: Bu noktadan sonraki tüm adımlar buz üzerinde ve soğuk reaktiflerle gerçekleştirilmelidir. Antikor veya antijen tükenmesini önlemek için, tüm reaktifler, tüpteki maya tarafından görüntülenen proteinlerin toplam sayısına kıyasla stokiyometrik fazlalıkta eklenmelidir. Aksi takdirde, antijenlerin ve/veya antikorların sınırlı mevcudiyeti nedeniyle maya ile görüntülenen varyantların bağlanmasını sınırlama riski olabilir. Genellikle, hesaplama için maya hücresi başına 5 x 104 molekül kullanılır ve en az 10 kat stokiyometrik fazla antijen ve antikor eklenmesi önerilir. Antijen çok sınırlıysa, fazlalık 5 kata kadar azaltılabilir. Ligand tükenmesini önlemek için hesaplamalar için örnekler de dahil olmak üzere daha ayrıntılı bir açıklama, önceki bir protokol9'da verilmiştir. - İnkübasyondan sonra, hücreleri santrifüjleyin (2000 x g, 5 dk, 4 °C) ve 1 mL PBSA ile yıkayın (seçimler sırasında bir ilaç/ligand varsa - bu durumda A1120 olarak - bunun yıkama tamponunda bulunması gerekir).
- Hücreleri, bağlı antijenin görüntülenmesi ve tespiti için antikorları içeren 100 μL soğuk PBSA'da yeniden süspanse edin ve 4 ° C'de 30 dakika inkübe edin.
- Tam uzunlukta görüntülenen POI'yi tespit etmek için anti-c-myc antikoru (son seyreltme 1:100) kullanın. Görüntülenen proteinlerin toplam miktarını tespit etmek için anti-HA antikoru (son seyreltme 1:50) kullanın. Sırasıyla 6x HIS etiketi veya biyotin içeren çözünür antijene bağlanan maya hücrelerini seçmek için Penta-His antikoru (son seyreltme 1:200) veya streptavidin (son seyreltme 1:200) kullanın.
- İkincil reaktife bağlanan protein varyantlarının zenginleşmesini önlemek için sıralama turları arasında algılama reaktifleri arasında geçiş yapın.
NOT: Farklı florofor kombinasyonları kullanılabilir, ancak Alexa Fluor 488 ve Alexa Fluor 647, akış sitometrisinde kompanzasyon gerektirmedikleri için iyi seçeneklerdir. Antikor tükenmesini önlemek için, tüpte maya ile görüntülenen proteinlerin toplam sayısına kıyasla stokiyometrik fazlalık eklenmelidir. Genellikle, hesaplamalar maya hücresi başına 5 x 104 moleküle dayanır. Daha ayrıntılı bir açıklama önceki protokollerdeverilmiştir 9.
- İnkübasyondan sonra, hücreleri santrifüjleyin (2000 x g, 5 dk, 4 °C) ve pelete 1 mL PBSA (hRBP4 ile seçilen G4 kütüphanesi 5 μM A1120 için ilaç/ligand içeren) ekleyin ve tekrar santrifüjleyin. Süpernatantın çoğunu çıkarın ve peletin kurumasını önlemek için sadece 20-30 μL tutun.
- Peleti soğuk PBSA'da sadece sıralamadan hemen önce yeniden süspanse edin ve istenen özelliklere sahip Fn3 moleküllerini gösteren maya hücreleri için sıralama yapın (yani, A1120 varlığında hRBP4'e bağlanma).
NOT: Geçit stratejisi Şekil 2A'da gösterilmiştir. - Hücreleri doğrudan SD-CAA ortamına sıralayın, sıralamadan sonra daha fazla SD-CAA ortamı ekleyin ve 180 rpm'de çalkalayarak 30 ° C'de inkübe edin.
NOT: Ayırmadan sonra hacmin önemli ölçüde artması durumunda, fazla kılıf sıvısını çıkarmak için bir santrifüjleme adımı (2000 x g, 5 dk, RT) gerçekleştirilmeli ve hücreler taze bir SD-CAA ortamında yeniden süspanse edilmelidir. Yeni tüpe sıralanan hücre sayısını yazın, çünkü bu, bir sonraki sıralama turu için yeni maksimum çeşitlilik olur. Yukarıda belirtildiği gibi, kütüphanenin gerçek çeşitliliği, kütüphanenin aşırı örneklenmesi nedeniyle daha düşük olacaktır. - Maya kütüphanesinde bağlayıcı protein varyantlarının zenginleşmesini görmek için akış sitometrik sıralamalarını birkaç tur boyunca tekrarlayın (Şekil 2B).
NOT: Analitik akış sitometrik deneyinde kitaplıkları yan yana karşılaştırmak faydalı olabilir. Daha sonraki seçim turlarında, antijen konsantrasyonunun düşürülmesi, daha düşük ve daha yüksek afiniteli bağlayıcılar arasında ayrım yapmada ve düşük konsantrasyonlarda hedefe daha yüksek bir bağlanma sinyali gösteren protein varyantları için spesifik olarak sıralamada faydalı olabilir (Şekil 2C). Daha sonraki seçim turlarında, artan termostabilite için sıralama yapmak da mümkündür. Bu amaçla, Bölüm 7'de açıklandığı gibi kütüphanenin epPCR ile rastgele mutajenezi gerçekleştirildi. İndüksiyon, OD600'ün belirlenmesi ve hücrelerin yıkanması ile diğer akış sitometrik sıralama turlarına benzer hücreler hazırlayın. Maya hücrelerini PBSA'ya 10 dakika boyunca buz üzerine yerleştirin, ardından yüksek bir sıcaklıkta ısı şoku için bir termosiklerde 10 dakikalık bir inkübasyon yapın. Daha sonra, 10 dakika boyunca tekrar buzun üzerine koyun ve boyama protokolüne devam edin. Stabilize edilmiş varyantlar, termal denatürasyona direnecek ve bu nedenle, ısı inkübasyonundan sonra sürekli bağlanma gösterecektir. Wildtype proteinin ilk termostabilitesine bağlı olarak ısı şoku için farklı sıcaklıklar test edilebilir. Tipik olarak, 40-80 °C arasında değişen sıcaklıklar uygundur. Bununla birlikte, 48 ° C'nin üzerinde, maya hücrelerinin canlılığı büyük ölçüde azalacaktır. Bu nedenle, adım 7.1'de tartışıldığı gibi, ısı şoklu maya kütüphanesini sıraladıktan sonra plazmitleri çıkarmak gerekir. Bu durumda, EBY100 hücrelerinin bir sonraki sıralama turu36'ya geçmeden önce zenginleştirilmiş varyantlarla taze bir şekilde dönüştürülmesi gerekir. Termostabiliteyi arttırmak için daha ayrıntılı bir protokol başka bir yerde bulunabilir37. Görüntülenen proteinin denatürasyonu geri dönüşümlü ise, bu yöntem işe yaramayacaktır. Alternatif olarak, sıralamadan bir gün önce 20 °C yerine 37 °C'de protein ekspresyonunu indükleyin ve hala verimli tam uzunlukta görüntü gösteren protein varyantları için sıralayın.
7. Rastgele mutasyonları tanıtmak için epPCR ile afinite olgunlaşması
NOT: epPCR kullanılarak afinite olgunlaşması, ilk akış sitometrik sıralama turundan önce veya akış sitometrik sıralama turları arasında gerçekleştirilebilir. A1120 varlığında hRBP4 ile G4 kütüphanesinin seçimi için, ilk akış sitometrik sıralama turundan önce afinite olgunlaşması gerçekleştirildi. Bu aynı zamanda boncuk seçiminden sonraki kütüphane boyutuna ve akış sitometrisi ile tespit edilebilen bağlanma sinyaline de bağlıdır. Özellikle, boncuk seçimlerinden sonraki afinitelerin akış sitometrik deneylerinde bir sinyal elde etmek için yeterli olmadığı durumlarda (çünkü antijen yıkama adımları sırasında hızlı bir şekilde ayrışır), epPCR, daha sonra akış sitometrisi yoluyla tespit edilebilen ve seçilebilen gelişmiş varyantlar üretebilir.
- Aşağıdaki değişikliklerle ticari plazmid miniprep II kitini ( Malzeme Tablosuna bakınız) kullanarak zenginleştirilmiş maya kitaplığından bir maya miniprep DNA ekstraksiyonu gerçekleştirin:
- Gece boyunca kültürün hücre yoğunluğunu ölçün, sabahları 0.2 OD600'e seyreltin ve çalkalama ile 30 ° C'de inkübe edin.
- OD600 0.6'ya ulaştığında, hücreleri 2 dakika boyunca 600 x g'da santrifüjleyerek 3 mL kültür hasat edin.
- Pelete 200 μL Çözelti 1 ekleyin. 6 μL Zimolaz ekleyin. Pelet hafifçe vurarak veya hafif girdaplama ile yeniden süspanse edin ve 37 ° C'de 3 saat inkübe edin.
- 200 μL Çözelti 2 ekleyin ve hafifçe karıştırın. 400 μL Çözelti 3 ekleyin ve hafifçe karıştırın.
- 3 dakika boyunca maksimum hızda santrifüjleyin.
- Süpernatanı spin-I kolonuna aktarın, 30 saniye boyunca maksimum hızda santrifüjleyin ve akışı atın.
- 550 μL etanol içeren Yıkama Tamponu ile yıkayın ve maksimum hızda 2 dakika santrifüjleyin.
- Kolonu temiz bir mikrosantrifüj tüpüne aktarın ve kalan yıkama tamponunu çıkarmak için maksimum hızda 2 dakika santrifüjleyin.
- Kolonu yeni bir mikrosantrifüj tüpüne aktarın, 10 μL nükleaz içermeyen su ekleyin ve DNA'yı elüte etmek için maksimum hızda 1 dakika döndürün.
- EpPCR şablonunu yükseltmek için bir ilk PCR gerçekleştirin.
- Maya yüzeyi görüntüleme vektöründeki ekin dışında tavlanan primerleri kullanarak DNA'yı PCR ile amplifiye edin.
NOT: Maya mini hazırlığı sırasında ilgilenilen plazmid ile birlikte çok sayıda genomik maya DNA'sı ayrıştırıldığından, epPCR için bir şablon olarak kullanılacak temiz bir DNA fragmanı elde etmek için bu adım önerilir. - pCTCON2 için fwd 5'- GGCTCTGGTGGAGGCGGTAGCGGAGGCGGAG primerlerini kullanın
GGTCGGCTAGC ve rev 5'- CTATTACAAGTCC
Tablo 3'te açıklanan reaksiyon bileşenleri ve Tablo 4'te gösterilen döngü koşulları ile TCTTCAGAAATAAGCTTTTGTTCGGATCC. - PCR'yi bir PCR ve DNA temizleme kiti ile saflaştırın. 20 μL nükleaz içermeyen H2O'da elute edin.
- Maya yüzeyi görüntüleme vektöründeki ekin dışında tavlanan primerleri kullanarak DNA'yı PCR ile amplifiye edin.
- Rastgele mutasyonları tanıtmak için epPCR gerçekleştirin.
- Belirtilen bileşenler (Tablo 5) ve koşullar (Tablo 6) ile nükleotid analogları ile bir epPCR gerçekleştirin.
- Tüm epPCR ürününü 120 V'ta 45 dakika boyunca hazırlayıcı% 1 agaroz jel üzerinde çalıştırın. Bir işaretleyici ekleyin ve epPCR ürünü ile merdiven arasında bir şerit bırakın. Ekin beklenen boyutuna karşılık gelen istenen bandı çıkarın, bir jel ekstraksiyon kiti kullanarak saflaştırın ve DNA konsantrasyonunu ölçün.
- Randomize DNA'yı PCR ile çoğaltın.
- DNA'nın 50 ng'sini bir Q5 polimeraz ile çoğaltın. Belirtilen koşullarla (Tablo 2) her elektroporasyon için 100 x 7 μL PCR reaksiyonu ayarlayın (Tablo 8). İlk PCR için kullanılanla aynı primerleri kullanın.
NOT: Mayaya elektroporasyon için büyük miktarlarda DNA gereklidir. - 5 μL PCR ürününü %1'lik bir agaroz jel üzerinde analiz edin, 120 V ile 45 dakika çalıştırın.
- DNA'nın 50 ng'sini bir Q5 polimeraz ile çoğaltın. Belirtilen koşullarla (Tablo 2) her elektroporasyon için 100 x 7 μL PCR reaksiyonu ayarlayın (Tablo 8). İlk PCR için kullanılanla aynı primerleri kullanın.
- Amplifiye edilmiş DNA'yı etanol çökeltme ile saflaştırın.
- Birlikte 2 x 100 μL havuzlayın. 20 μL (hacmin% 10'u) steril filtrelenmiş 3 M sodyum asetat, pH 5.2 ekleyin.
NOT: Bu tampon önceden hazırlanabilir ve RT'de birkaç ay saklanabilir. - 440 μL %100 etanol (veya en az 2 x hacim) ekleyin ve RT'de 2 dakika inkübe edin.
- RT'de 5 dakika boyunca 20.000 x g'da santrifüjleyin. Süpernatanı çıkarın, 500 μL% 70 etanol ekleyin ve kısaca karıştırın.
- RT'de 5 dakika boyunca 20.000 x g'da santrifüjleyin. Süpernatanı çıkarın, 500 μL% 100 etanol ekleyin ve kısaca karıştırın.
- RT'de 5 dakika boyunca 20.000 x g'da santrifüjleyin. Süpernatanı çıkarın ve tüm etanol buharlaşana kadar DNA peletini laminer bir akış başlığı altında kurumaya bırakın.
NOT: Bu işlemi hızlandırmak için 48 °C'de bir ısı bloğu kullanın. DNA peleti her zaman görünür olmayabilir. - Kurutulmuş DNA peletini 10 μL nükleaz içermeyen H2O içinde çözün.
NOT: DNA mümkün olduğunca az hacimde çözülmelidir. DNA konsantrasyonunu ölçmeyin.
- Birlikte 2 x 100 μL havuzlayın. 20 μL (hacmin% 10'u) steril filtrelenmiş 3 M sodyum asetat, pH 5.2 ekleyin.
| Hacim [μL] | Son konsantrasyon | |
| 5x Q5 geliştirici | 10 | 1 katı |
| 5x Q5 tamponu | 10 | 1 katı |
| Astar fwd 10 μM | 2.5 | 0,5 μM |
| Astar devir 10 μM | 2.5 | 0,5 μM |
| dNTP'ler 10 mM | 1 | 200 μM |
| Q5 polimeraz | 0.5 | 20 U/mL |
| Maya miniprep'inden DNA | 10 | |
| Nükleaz içermeyen H2O | 13.5 |
Tablo 3: İzole maya miniprepinden POI genlerinin amplifikasyonu için 1. adım PCR koşulları.
| Adım | Sıcaklık | Saat |
| İlk denatürasyon | 98 °C | 30 Saniye |
| 25 döngü | 98 °C | 10 saniye |
| 72 °C | 30 Saniye | |
| 72 °C | 30 Saniye | |
| Son uzatma | 72 °C | 2 dk |
| Tutmak | 4 °C |
Tablo 4: İzole maya miniprepinden POI genlerinin amplifikasyonu için 1. adım PCR için döngü koşulları.
| Hacim [μL] | Son konsantrasyon | |
| Nükleaz içermeyen H2O | 50'ye kadar | |
| 10x Thermopol tamponu | 5 | 1 katı |
| Primer_fwd (10 μM) | 2.5 | 0,5 μM |
| Primer_rev (10 μM) | 2.5 | 0,5 μM |
| dNTP'ler (10 mM) | 1 | 200 μM |
| 8-okso-dGTP (100 μM) | 1 | 2 μM |
| dPTP (100 μM) | 1 | 2 μM |
| 1. PCR'den PCR ürünü | XX | 50 ng |
| Taq DNA polimeraz | 0.5 | 0,05 U/μL |
Tablo 5: POI DNA'nın 1. basamak PCR ile amplifikasyonundan sonra gerçekleştirilen epPCR için koşullar.
| Adım | Sıcaklık | Saat |
| İlk denatürasyon | 94 °C | 30 Saniye |
| 15 döngü | 94 °C | 45 Saniye |
| 60 °C | 30 Saniye | |
| 72 °C | 1 dk | |
| Son uzatma | 72 °C | 10 dk |
| Tutmak | 4 °C |
Tablo 6: epPCR için bisiklet koşulları.
| Hacim [μL] | Son konsantrasyon | |
| 5x Q5 geliştirici | 20 | 1 katı |
| 5x Q5 tamponu | 20 | 1 katı |
| Astar fwd 10 μM | 5 | 0,5 μM |
| Astar devir 10 μM | 5 | 0,5 μM |
| dNTP'ler 10 mM | 1 | 200 μM |
| Q5 polimeraz | 1 | 20 U/mL |
| 50 ng DNA | XX | |
| ddH20 | 100'e kadar |
Tablo 7: EBY2 hücrelerinin elektroporasyonundan önce epPCR ürününün amplifikasyonu için 100. adım PCR koşulları.
| Adım | Sıcaklık | Saat |
| İlk denatürasyon | 98 °C | 30 Saniye |
| 25 döngü | 98 °C | 10 saniye |
| 72 °C | 30 Saniye | |
| 72 °C | 30 Saniye | |
| Son uzatma | 72 °C | 2 dk |
| Tutmak | 4 °C |
Tablo 8: epPCR ürününün amplifikasyonu için 2. adım PCR için döngü koşulları.
8. Elektroporasyon için maya görüntüleme vektörünün doğrusallaştırılması
- pCTCON2 maya yüzeyi görüntüleme vektörünü doğrusallaştırın (Addgene plazmidi #41843).
- Büyük ölçekli bir özet gerçekleştirin ve bu sindirilmiş doğrusallaştırılmış vektörü diğer tüm elektroporasyonlar için kullanın.
- İlk doğrusallaştırma için 2 x 200 μg pCTCON2 plazmit DNA'sını Sal I ile 37 ° C'de 24 saat boyunca sindirin (Tablo 9).
- Ek parçayı çıkarmak için 37 ° C'de 24 saat boyunca NheI ve BamHI ile sindirin (Tablo 10).
- Son olarak, tüpe her enzimden 5 μL ekleyin (Sal I, NheI, BamHI) ve 37 ° C'de 24 saat boyunca tekrar inkübe edin.
- Sindirilmiş vektörü hazırlayıcı bir agaroz jeli üzerinde çalıştırın. Bantları çıkarın ve bir jel ekstraksiyon kiti ile saflaştırın. Vektörü 20 μL'de ısıtın, DNA konsantrasyonunu ölçün ve kabaca 2 μg/μL'ye ayarlayın.
NOT: Plazmidin uzun süre sindirilmesi, ekin yeterli bölünmesini sağlar ve elektroporasyondan sonra vektörden daha az arka plan ile sonuçlanır.
| DNA | 200 μg |
| 10x CutSmartTampon | 50 μL |
| Sal I-HF (NEB) | 30 μL (60 U) |
| H2O | 500 μL'ye kadar |
Tablo 9: Maya yüzeyinin büyük ölçekli sindiriminin ilk adımı için koşullar pCTCON2 vektörünü gösterir.
| pCTCON2 (Sindirilen Sal) | 500 μL |
| 10x CutSmartTampon | 37,5 μL |
| NheI-HF (NEB) | 15 μL (30 U) |
| BamHI-HF (NEB) | 15 μL (30 U) |
| H2O | 875 μL'ye kadar |
Tablo 10: Maya yüzeyinin büyük ölçekli sindiriminin ikinci aşaması için koşullar pCTCON2 vektörünü gösterir.
9. EBY100'ün randomize DNA ve doğrusallaştırılmış vektör ile elektroporasyonu
- EBY100 maya hücrelerini (ATCC, Malzeme Tablosuna bakınız) elektroporasyondan 3 gün önce bir gliserol stoğundan vurarak hazırlayın. Dönüştürülmemiş EBY100 hücreleri maya yüzeyi görüntüleme vektörünü içermediğinden, tam ortam YPD plakaları kullanın (Tablo 1). 30 °C'de inkübe edin.
- Elektroporasyondan bir gün önce, tek bir EBY100 kolonisini 30 mL taze hazırlanmış YPD ortamına aşılayın (Tablo 1). 180 rpm'de çalkalarken gece boyunca 30 °C'de inkübe edin.
NOT: YPD ortamı 1 aydan eski olmamalıdır. - Ertesi sabah, EBY100 gece kültürünü 100 mL YPD ortamında 0.2'lik bir OD600'e seyreltin ve 180 rpm'de çalkalarken 30 ° C'de inkübe edin.
NOT: 2 elektroporasyon için 50 mL'lik bir kültür hacmi yeterlidir. Tipik olarak, 100 mL, kontroller için yeterli hücreye sahip olacak şekilde hazırlanır (sadece hücreler ve hücreler artı vektör). - 1.3-1.5'lik bir OD600'e ulaşıldığında, hücreleri 50 mL'lik tüplerde 2000 x g'da 3 dakika boyunca pelet haline getirin ve süpernatanı çıkarın.
- Peletin 25 mL 100 mM steril filtrelenmiş lityum asetat içinde yeniden süspanse edilmesi ( Malzeme Tablosuna bakınız). Taze hazırlanmış steril filtreli DTT'yi (1 M) (Malzeme Tablosuna bakınız) 10 mM'lik bir nihai konsantrasyona ekleyin.
NOT: Lityum asetat hacmi, orijinal YPD hacminin yarısı kadar olmalıdır. Gerekirse ölçeği küçültün veya artırın. Lityum asetat (100 mM) önceden hazırlanabilir ve RT'de birkaç ay saklanabilir, ancak 1 M DTT stoğu kullanımdan hemen önce hazırlanmalı ve buz üzerinde tutulmalıdır. - Hücreleri 10 dakika çalkalarken 30 ° C'de inkübe edin.
- 3 dakika boyunca 2000 x g'da santrifüjleyin, süpernatanı atın ve hücreleri buzun üzerine yerleştirin.
NOT: Aşağıdaki tüm adımlar buz üzerinde ve soğutulmuş reaktifler, küvetler ve mikrosantrifüj tüpleri ile gerçekleştirilmelidir. - Peleti 25 mL soğuk steril H2O. Santrifüjde 2000 x g'da 3 dakika boyunca tekrar süspanse edin.
NOT: Kullanılan H2O hacmi, orijinal YPD hacminin yarısı kadar olmalıdır. - 350 μL steril H2O içinde tekrar süspanse edin.
NOT: Yeniden süspansiyondan sonraki toplam hacim 500 μL olmalıdır. Gerekirse daha fazla H2O ekleyin. - DNA kütüphanesini paralel olarak hazırlayın: Etanol ile saflaştırılmış ekleri içeren mikrosantrifüj tüplerini buz üzerine yerleştirin ve 4 μg doğrusallaştırılmış pCTCON2 vektörü ekleyin.
- 250 μL hücreyi DNA'ya ekleyin ve keserek hafifçe karıştırın. Önceden soğutulmuş 2 mm'lik bir elektroporasyon küvetine aktarın ve elektroporasyona kadar buz üzerinde tutun.
NOT: Elektroporasyonun başarısını belirlemek için kontroller ekleyin (yalnızca DNA'sı olmayan hücreler ve yalnızca eklemesiz doğrusallaştırılmış vektöre sahip hücreler). - Aşağıdaki koşullara sahip 2 mm elektroporasyon küvetleri ( Malzeme Tablosuna bakın) kullanarak elektropoze edin: kare dalga protokolü, tek darbe, 500 V, 15 ms darbe süresi.
NOT: Tipik sarkmalar %3 - %9'dur. Diğer elektroporatörler de kullanılabilir, ancak belirtilen koşullar bu çalışmada kullanılan cihaz için optimize edilmiştir. - Hücreleri 1 mL önceden ısıtılmış YPD ile kurtarın, önceden ısıtılmış bir mikrosantrifüj tüpüne aktarın ve 30 ° C'de 1 saat boyunca sallamadan inkübe edin.
NOT: Geri kazanım tüplerinin uygun sıcaklıkta kalmasını sağlamak için ısıtma bloğunu elektroporatörün yanına yerleştirin. - SD-CAA plakaları üzerinde 100 μL 10-3, 10-4, 10-5 ve 10-6 hücre seyreltmeleri (bkz. Tablo 1) ve 30 ° C'de dönüştürücülerin büyümesine izin verir.
NOT: Teorik kütüphane çeşitliliği, burada 1 mL olan orijinal kültür hacmine dönüştürücü sayısının geri hesaplanmasıyla elde edilir. Tipik olarak, bu dönüşüm protokolü yaklaşık 107-10 8 dönüştürülmüş hücre verir. - Kalan hücreleri 3 dakika boyunca 2000 x g'da peletleyin, 200 mL SD-CAA'da yeniden süspanse edin ve gece boyunca 180 rpm'de çalkalayarak 30 ° C'de inkübe edin. EpPCR randomize kütüphanesini içeren transforme hücreler ya SD-CAA'da geçilebilir, hem de ertesi gün SG-CAA'da yüzey ekspresyonu için indüklenebilir veya daha fazla kullanıma kadar 4 ° C'de saklanabilir. Çeşitliliği belirledikten sonra epPCR sonrası kütüphanenin gliserol stoklarını dondurun. Ertesi gün, hücrelerin bir kısmı, bir plazmit elde edilmeden transformasyon prosedüründe taşınan dönüştürülmemiş hücreler olmaya devam edeceğinden, bu etkiyi telafi etmek için ilk geçiş adımında kütüphane çeşitliliğinin en az 20 katının geçirilmesi önerilir.
10. Birkaç seçim turundan sonra maya kütüphanelerinin sıralanması
- Yukarıda tarif edildiği gibi bir maya mini hazırlığı yaparak maya DNA'sını çıkarın.
- Ticari E. coli 10-beta elektro-yetkin hücreleri (bkz . Malzeme Tablosu) izole edilmiş DNA ile elektrostatik hale getirin.
- Elektroporasyon küvetlerini (1 mm, Malzeme Tablosuna bakın) ve mikrosantrifüj tüplerini buzun üzerine yerleştirin. Elektroporasyondan sonra hücreleri geri kazanmak için 37 ° C'de 950 μL büyüme ortamı ile mikrosantrifüj tüplerini önceden ısıtın.
- Hücreleri buz üzerinde çözdürün ve tüpü hafifçe vurarak karıştırın. Hücrelerin 50 μL'sini soğutulmuş bir mikrosantrifüj tüpüne aktarın. 1 μL maya miniprep DNA'sı ekleyin.
- Hücre-DNA karışımını dikkatlice soğutulmuş bir elektroporasyon küvetine aktarın.
NOT: Kabarcıklardan kaçının ve hücrelerin küvetin dibinde biriktiğinden emin olun. - Aşağıdaki koşulları kullanarak bir elektroporasyon cihazı ile elektropoze edin: 2.0 kV; 200 Ω; 25 μF olarak adlandırılır.
NOT: Tipik zaman sabitleri 4.8-5.1 ms'dir. Diğer elektroporatörler de kullanılabilir, ancak belirtilen koşullar bu çalışmada kullanılan cihaz için optimize edilmiştir. - Küvetlere hemen 950 μL 37 °C önceden ısıtılmış büyüme ortamı ( Malzeme Tablosuna bakınız) ekleyin, hafifçe karıştırın ve ılık mikrosantrifüj tüplerine geri aktarın.
NOT: Geri kazanım tüplerinin uygun sıcaklıkta kalmasını sağlamak için ısıtma bloğunu elektroporatörün yanına yerleştirin. - 37 °C ve 300 rpm'de 40 dakika inkübe edin. Plaka 10 μL, 100 μL ve hücrelerin geri kalanı (santrifüjlemeden sonra) önceden ısıtılmış seçici bir LB plakasına (bakınız Tablo 1). Plakaları gece boyunca 37 °C'de inkübe edin.
NOT: pCTCON2 vektörü için, antibiyotik seçimi için ampisilin kullanılır. - LB agar ve gerekli antibiyotik (burada ampisilin kullanılır) içeren bir kopya plaka hazırlayın: 96 oyuklu bir plakanın her bir oyuğuna 100 μg/mL ampisilin ile 200 μL LB agar pipetleyin ve katılaşmasına izin verin.
- Oyuk başına 100 μg/mL ampisilin içeren 50 μL LB içeren 96 kuyulu bir sıralama plakası hazırlayın.
- Plakalardan tek bir koloni seçin ve önce kopya plakasını ve sıralama plakasının aynı kuyusunu aşılayın.
NOT: Kopya plakası, sıvı bir kültürün aşılanması ve bir mini hazırlık yapılması yoluyla istenen plazmitlerin kolayca elde edilebilmesini sağlar. - Kopya plakasını gece boyunca 37 °C'de inkübe edin ve ardından 4 °C'de saklayın.
- Sıralama plakasını plastik veya alüminyum folyo ile kaplanmış olarak sallarken (180 rpm) 37 °C'de 3-4 saat inkübe edin.
- Sıralama plakasını kapatın ve 500 μL 10 μM pCTCON2 sıralama astarı (5'-CGTTTGTCAGTAATTGCGGTTCTC) ile birlikte dizileme için gönderin.
- Dizilerin analizi için, elde edilen DNA dizilerini, örneğin EMBOSS Transeq38 gibi uygun bir seçim programı kullanarak amino asit dizilerine çevirin. Ardından, varyant dizilerini birbirine ve ebeveyn vahşi tip proteinine hizalamak için Çoklu Dizi Hizalama (MUSCLE)39 veya benzer bir hizalama aracı kullanın.
NOT: Farklı seçim turlarının kütüphanelerinin dizilimini gerçekleştirerek (örneğin, daha önceki bir seçim turundan sonra ve ayrıca yüksek afiniteli varyantları zenginleştirmek için daha düşük antijen konsantrasyonlarına sahip ek seçim turlarından sonra), farklı varyantları tanımlamak mümkündür. Bu, büyük ölçüde sıralama turları sırasında uygulanan kapıların sıkılığına ve dolayısıyla kütüphanelerin çeşitliliğine bağlıdır.
Sonuçlar
G4 kütüphanesi, küçük moleküllü ilaç A1120'ye bağlı antijen hRBP4'e karşı seçildi. Akış sitometrik sıralama için kütüphanelerin boyanması, Yöntem 6'da açıklandığı gibi gerçekleştirildi ve uygulanan geçit stratejisi Şekil 2A'da gösterildi. Bir ilk geçit, hücre morfolojisine dayalı olarak tüm hücreleri içeriyordu ve ikinci kapı (FSC-Genişlik histogramı), tek hücreleri seçmek ve hücre agregalarını çıkarmak için uygulanan sıkı bir geçit stratejisi gösterdi. Üçüncü ve son kapı, protein varyantlarının (x ekseni) antijen bağlanmasına (y ekseni) karşı görüntüsünü gösterdi. Hem görüntüleme hem de bağlanma sinyalleri gösteren maya hücreleri sıralandı. Daha da önemlisi, sıralama kapısı, bağlanma alanlarını yüksek bağlanma sinyali ve dolayısıyla yüksek afinite ile zenginleştirmek için katı bir şekilde ayarlandı. Bu sıkı seçim, seçim kampanyası boyunca hedef antijene spesifik olarak bağlanan maya hücrelerinin görüntülenmesinde bir zenginleştirme sağladı (Şekil 2B). Daha sonraki akış sitometrik sıralama turlarında, antijen konsantrasyonu 10 kat azaltıldı (100 nM'den 10 nM'ye). Bu nedenle, genel bağlanma sinyali azaldı ve sadece yüksek afiniteye sahip bağlayıcılar hala tespit edilebilir ve sıralanabilirdi (Şekil 2C).
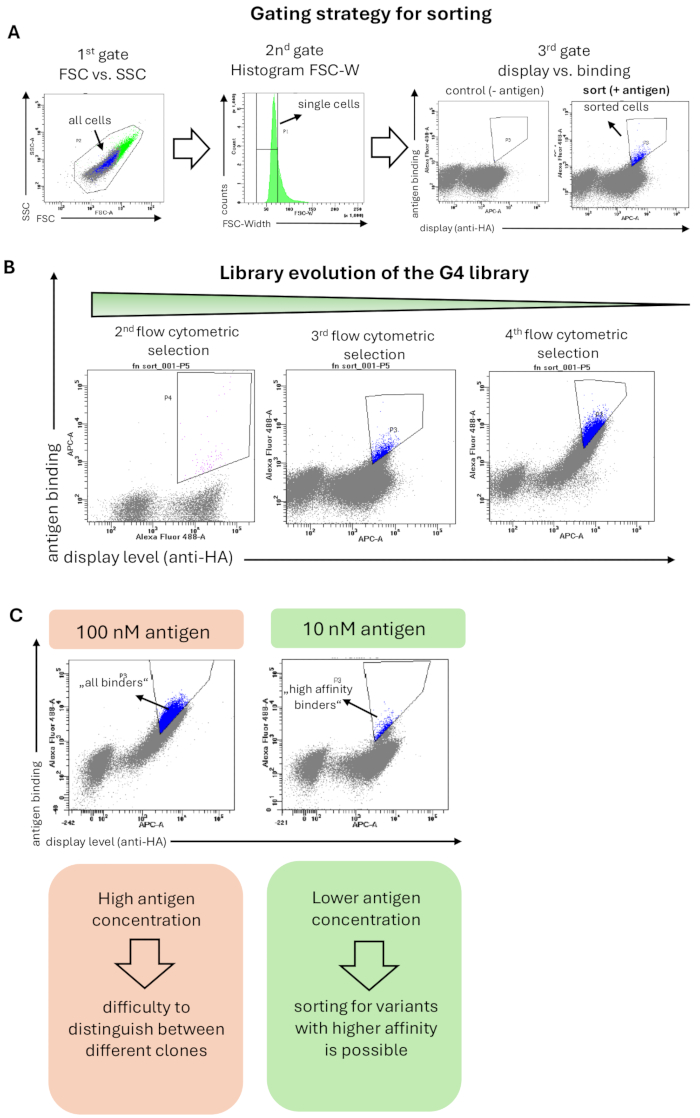
Şekil 2: Antijene (A1120 varlığında hRBP4) bağlanmak için Fn3 tabanlı G4 kütüphanesinin bir maya yüzeyi ekran seçiminden elde edilen temsili sonuçlar. (A) Maya kütüphanelerinin sınıflandırılması için genel geçit stratejisi. İlk kapı (FSC ve SSC), tüm maya hücrelerini seçmek ve saçılma olaylarını hariç tutmaktır; ikinci kapı (FSC-W'nin histogramı) hücre agregalarını uzaklaştırmayı ve yalnızca tek maya hücrelerini seçmeyi amaçlar. Üçüncü kapı, antijene bağlanmaya karşı yüzey görüntüleme seviyesini (HA- veya c-myc-tag'in tespiti) çizer (burada anti-His antikoru tarafından tespit edilen 5 μM A1120 varlığında hRBP4). Kütüphane ayrıca antijen bağlanmasının beklenmediği durumlarda sadece ikincil antikorlarla (antijensiz) boyandı. Sıralanan hücreler mavi renkle vurgulanır. (B) G4 kütüphanesinin 3 tur akış sitometrik sıralama boyunca evrimi. Bağlanma popülasyonunun zenginleşmesi her seçim turunda gözlemlenebilir. (C) Daha düşük antijen konsantrasyonlarının kullanılması, hedef antijene karşı daha yüksek afiniteye sahip protein varyantlarının seçilmesini sağlar. Antijen konsantrasyonunun (burada hRBP4) 10 kat azaltılması üzerine, daha yüksek (sıralanmış hücreler, mavi) veya daha düşük afiniteye sahip klonların varlığını gösteren farklı köşegenler ortaya çıkar. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Maya yüzey gösterimi, protein mühendisliğinde kullanılan temel yöntemlerden biri olarak gelişmiştir. Yaygın olarak afinite 1,18,40,41, ekspresyon/stabilite 24,27,42,43 ve aktivite28,44 mühendisliği için kullanılmasına rağmen, epitop haritalama45,46 veya maya hücrelerinin yüzeyindeki bireysel mutantların karakterizasyonu gibi daha ileri kullanımlar 9 da mümkündür. Bu protokolde, manyetik boncuklarla seçim ve akış sitometrik sıralama ile maya kütüphanesinin afinite olgunlaşması için epPCR ile çeşitlendirilmesi de dahil olmak üzere bir maya yüzeyi teşhir seçimi kampanyası başlatmak için temel adımları sunuyoruz.
Konvansiyonel maya yüzey teşhir seçimleri için temel bir gereklilik, yeterli kalitede çözünür proteinin mevcudiyetidir. Yüksek saflıkta ve tanımlanmış bir oligomerizasyon durumuna sahip iyi katlanmış bir hedef proteinle başlamak (yani, monomerik protein sadece monomer olarak bulunmalıdır), hedef antijene yüksek afiniteye sahip bir protein varyantı için seçim yapmak için en yüksek başarı oranını sağlar. İfade edilmesi zor hedef proteinler için bir alternatif, bu sınırlamayı aşmak için makul bir strateji sunan hücre bazlı seçimlerdir47. Bununla birlikte, maya yüzeyi ekranı, zahmetli ve zaman yoğun klonlama, çözünür bir formatta ekspresyon ve protein saflaştırma işlemine gerek kalmadan elde edilen protein varyantlarını doğrudan maya yüzeyinde karakterize etme imkanı gibi birçok avantaj sunar. Varyantların hem afinitesi hem de stabilitesi doğrudan maya yüzeyi9 üzerinde analiz edilebilir.
Bu protokolde, protein varyantlarının G4 kütüphanesinin, daha spesifik olarak insan fibronektininin 10. tip III alanının, küçük molekül A1120'nin varlığında antijen hRBP4'e bağlanmak için nasıl seçildiğini gösteriyoruz. Boncuk seçimleri ve akış sitometrik sıralamasının kombinasyonu, seçim turları boyunca hedef antijene artan bir bağlanma gösteren varyantların zenginleşmesini sağladı (Şekil 2B). Daha düşük konsantrasyonlarda antijen kullanılmasının, yüksek afiniteli protein varyantlarının seçimini mümkün kıldığını gösterdik (Şekil 2C). Tipik olarak, maya ekran seçimleri ile elde edilebilecek afiniteler nanomolar ve hatta pikomolar aralık18'dedir. Nihai afiniteler, hedef antijene, seçim turlarının sayısına ve afinite olgunlaşmasına, kullanılan bağlama iskelesine ve uygulanan geçit stratejisine bağlıdır. Bireysel protein varyantlarının karakterizasyonu bu protokolde ele alınmamıştır, ancak önceki çalışmamızda ayrıntılı olarak açıklanmıştır9. Maya gösterimi başlangıçta scFvs 1,40 gibi antikor fragmanlarının mühendisliği için kullanılmış olsa da, yöntem antikor bazlı olmayan proteinler için de yaygın olarak kullanılmaktadır10.
Özetlemek gerekirse, maya yüzey ekranı, hemen hemen her hedef proteine bağlanma ve/veya artan stabilite gibi yeni veya geliştirilmiş özelliklere sahip protein varyantlarının üretilmesini sağlayan güçlü bir protein mühendisliği aracıdır.
Açıklamalar
M.W.T., Miltenyi Biotec'ten fon aldı. Tüm yazarlar, maya yüzey ekranı kullanılarak geliştirilen teknolojiler ve mühendislik proteinleri için patent başvurularında mucitlerdir.
Teşekkürler
Bu çalışma, Avusturya Bilim Fonu (FWF Projesi W1224 - Proteinlerin Biyomoleküler Teknolojisi Doktora Programı - BioToP ve FWF Projesi ESP 465-B), Avusturya Federal Dijital ve Ekonomik İşler Bakanlığı, Avusturya Ulusal Araştırma, Teknoloji ve Geliştirme Vakfı tarafından Christian Doppler Araştırma Derneği'ne (Yeni Nesil CAR T Hücreleri için Christian Doppler Laboratuvarı) desteklenmiştir. ve St. Anna Çocuk Kanser Araştırma Enstitüsü'ne (Viyana, Avusturya) özel bağışlarla. E.S., Avusturya Bilimler Akademisi'nin St. Anna Çocuk Kanseri Araştırma Enstitüsü'nün DOC Bursu sahibidir. Figürler BioRender.com ile oluşturulmuştur.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
Referanslar
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır