CMG Helikaz ile DNA Çözmenin Tek Moleküllü Gerçek Zamanlı Görselleştirilmesi
Bu Makalede
Özet
Bu protokol, CMG sarmal ile çözülen DNA'nın canlı görselleştirilmesi için tek moleküllü bir testin gerçekleştirilmesini gösterir. (1) bir DNA substratının hazırlanmasını, (2) floresan etiketli Drosophila melanogaster CMG helikazının saflaştırılmasını, (3) toplam iç yansıma floresan (TIRF) mikroskobu için bir mikroakışkan akış hücresinin hazırlanmasını ve (4) tek moleküllü DNA çözme tahlilini açıklar.
Özet
Sadık genom duplikasyonu, bölünen hücrelerin genetik stabilitesini korumak için gereklidir. DNA replikasyonu, S fazı sırasında, replizom adı verilen dinamik bir protein kompleksi tarafından gerçekleştirilir. Yanıtın kalbinde, DNA çift sarmalının iki zincirini DNA polimerazların her bir zinciri kopyalayabilmesi için ayıran CDC45-MCM2-7-GINS (CMG) sarmal bulunur. Genom çoğaltması sırasında, yanıtlayıcılar çok sayıda engel ve zorluğun üstesinden gelmelidir. Bunların her biri genom stabilitesini tehdit eder, çünkü DNA'nın tam ve doğru bir şekilde kopyalanmaması mutasyonlara, hastalıklara veya hücre ölümüne yol açabilir. Bu nedenle, CMG'nin hem normal replikasyon hem de replikasyon stresi sırasında replizomda nasıl çalıştığını anlamak büyük ilgi çekicidir. Burada, yüzeye bağlı gerilmiş DNA moleküllerinin bireysel CMG kompleksleri tarafından gerçek zamanlı olarak görselleştirilmesine izin veren rekombinant saflaştırılmış proteinleri kullanan bir toplam iç yansıma floresan (TIRF) mikroskobu testini açıklıyoruz. Bu tahlil, tek molekül düzeyinde CMG davranışını araştırmak için güçlü bir platform sağlar ve helikaz dinamiklerinin reaksiyon koşulları üzerinde gerçek zamanlı kontrol ile doğrudan gözlemlenmesine olanak tanır.
Giriş
DNA replikasyonu, bir hücrenin mutasyonları, hastalıkları veya ölümü önlemek için genomunu doğru bir şekilde kopyalaması gerektiğinden sıkı bir şekilde düzenlenir. Ökaryotik DNA replikasyonu, ebeveyn DNA'sını çözen ve yeni DNA'yı sentezlemek için bir şablon olarak tek sarmallı DNA'yı (ssDNA) kullanan replizom kompleksi tarafından gerçekleştirilir. G1 fazında, MCM2-7'nin katalitik olarak aktif olmayan çift heksamerleri, replikasyon kökenleri1'de çift sarmallı DNA'ya (dsDNA) yüklenir. S fazında, MCM2-7 kompleksleri, CDC45 ve GINS2'nin bağlanmasıyla 11 alt birimli CMG kompleksleri (CDC45, MCM2-7, GINS) oluşturmak üzere aktive edilir. Her CMG, DNA'nın zıt yönlerde çözülmesini başlatır ve replizomanın kendisini3 etrafında düzenlediği çekirdek birimi oluşturur.
Yirmi yıl önce, CMG helikaz ilk olarak DNA replikasyonu4 için gerekli olan 11 alt birimli bir kompleks olarak tanımlandı. O zamandan beri, CMG anlayışımız, yükleme ve aktivasyon 5,6'dan DNA çözme ve sonlandırma7'ye kadar önemli ölçüde ilerlemiştir. Geleneksel biyokimyasal ve yapısal biyoloji teknikleri bu keşiflerin çoğu için kritik öneme sahiptir; bununla birlikte, bu yöntemler genellikle CMG'nin daha dinamik yönlerini inceleme yeteneklerinde sınırlıydı. Tek moleküllü yöntemler, aktivitelerini her seferinde bir molekül ölçmek veya görselleştirmek için tek tek biyomoleküllerin fiziksel manipülasyonunu kullanır. Bu, diğer teknikler tarafından sıklıkla gözden kaçan veya tespit edilemeyen proteinlerin gerçek zamanlı dinamikleri hakkında fikir vermek için kullanılabilir 8,9.
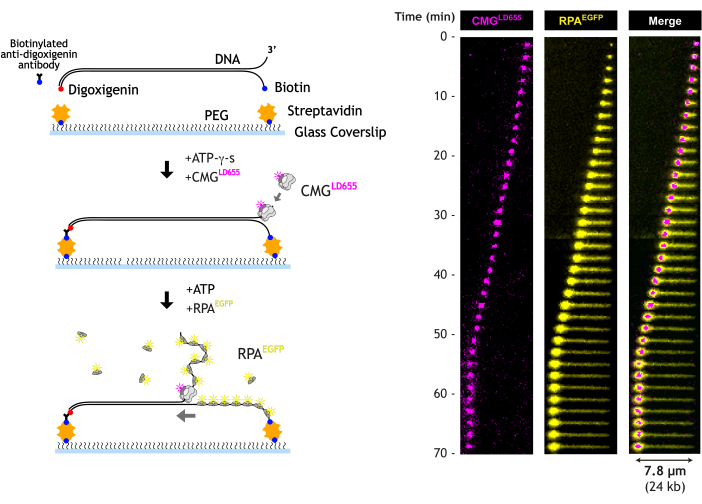
Burada, CMG helikaz ile DNA çözülmesini gerçek zamanlı olarak görselleştirmek için bir toplam iç yansıma floresan (TIRF) mikroskobu testini açıklıyoruz. Saflaştırılmış, floresan olarak etiketlenmiş CMG, önceden yapılmış bir DNA çatal yapısı içeren uzun DNA'nın serbest 3 'ucuna yüklenir. Doğrusal DNA, DNA'nın her iki ucunu sırayla yüzeye bağlayarak bir mikroakışkan akış hücresinde bir biotin-PEG lamel üzerinde gerilir. Bu yaklaşım, veri analizi sırasında hesaba katılması gereken varyasyonu önemli ölçüde azaltan daha düzgün DNA bağlantısına izin verir. ATP-γ-s varlığında, CMG, çatalın 3' ucundaki tek sarmallı DNA'ya yüklenir. ATP-γ-s, CMG'nin DNA'ya bağlanmasına izin veren ancak çözülmesine izin vermeyen, yavaş hidrolize olabilen bir ATP analoğudur. Daha sonra ATP'nin saflaştırılmış, floresan etiketli RPA ile birlikte eklenmesi, CMG'yi aktive eder ve kapsamlı DNA çözülmesini başlatır. Görsel olarak, CMG DNA boyunca yer değiştirir ve arkasında büyüyen bir RPA'ya bağlı ssDNA yolu bırakır. Bağlanmamış DNA ucu, CMG ile hareket eder ve RPA bağlanmasının neden olduğu sıkıştırma nedeniyle "sıkı bir top" oluşturur. Akış hücresi tasarımı, tamponun çözme sırasında herhangi bir noktada değiştirilmesine izin vererek, her deney sırasında ve üzerinde büyük kontrol sağlar.
Bu protokol, birbirinden bağımsız olarak gerçekleştirilebilen dört yönteme ayrılmıştır. Bölüm 1, tek moleküllü tahliller için 20 kb'lık doğrusal çatallı bir DNA substratının hazırlanmasını açıklar. Bölüm 2, Drosophila melanogaster CMG'nin (DmCMG) saflaştırılmasını ve floresan etiketlemesini özetlemektedir. DmCMG'nin ifadesi ile ilgili temel bilgiler notlar bölümünde yer almaktadır. Bölüm 3, bir TIRF mikroskobu üzerinde kullanılabilecek bir mikroakışkan akış hücresinin hazırlanmasını kapsar. Bölüm 4, tek moleküllü DNA çözme testinin nasıl gerçekleştirileceğini açıklar.
Protokol
1. Tek moleküllü tahlillerde kullanılan 20 kb'lık lineer çatallı DNA'nın hazırlanması (Şekil 1)
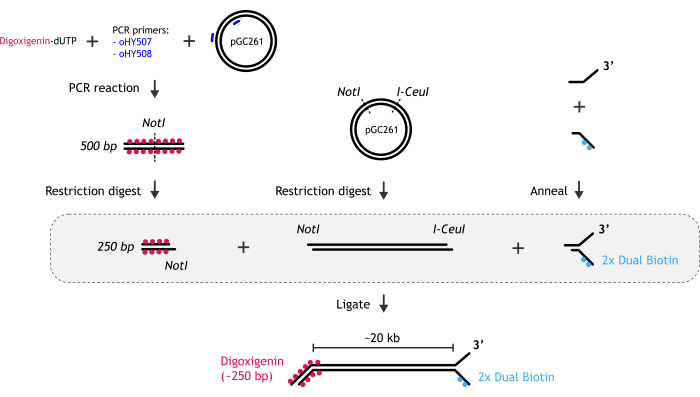
Şekil 1: DNA substrat hazırlığının grafiksel gösterimi. (A) Biyotinile DNA çatal ucu, kısmen tamamlayıcı iki oligonükleotidin tavlanmasıyla oluşturulur: biyotinillenmiş ve biyotinillenmemiş olan. (B) Ana dsDNA fragmanı (~ 20 kb), her iki uçta farklı çıkıntılara sahip doğrusal bir DNA oluşturmak için pGC261 plazmidinin iki enzimle kısıtlama sindirimi ile üretilir. (C) Digoksijenenin dubleks DNA ucu, digoksijenenin-dUTP varlığında gerçekleştirilen bir PCR reaksiyonu ve ardından kısıtlama sindirimi ile elde edilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- Biyotinile DNA çatal ucunun oluşturulması.
- Çatal DNA'sını tavlayın.
- İki oligonükleotidi (oHY502 ve oHYbio85) 1x STE tamponunda (100 mM NaCl, 10 mM Tris, pH 8.0; 1 mM etilendiamintetraasetik asit [EDTA]) karıştırın. Son oligonükleotid konsantrasyonu 10 μM'dir ve son reaksiyon hacmi 100 μL'dir.
- Reaksiyonu bir ısı bloğu üzerinde 80 ° C'de 5 dakika inkübe edin. Isı bloğunu kapatın ve reaksiyonun kademeli olarak soğumasını bekleyin, sıcaklık 30 °C'nin altına düşene kadar ısı bloğunda bırakın.
- Tris-Borat-EDTA (TBE)-Poliakrilamid jel elektroforezi (PAGE) jeli üzerindeki tavlama verimliliğini doğrulayın.
- % 8 akrilamid - TBE jeli hazırlayın (4 jel için: 32.8 mL H2O; 4.8 mL 10x TBE tamponu (1 M Tris, 1 M borik asit, 20 mM EDTA), 9.6 mL %40 Akrilamid / Bis Çözeltisi, 37.5: 1; 0.8 mL %10 Amonyum persülfat [APS], 40 μL Tetrametiletilendiamin [TEMED]).
DİKKAT: TBE, akrilamid, APS, TEMED toksik/zararlıdır. - RT'de 1 saat boyunca% 8 akrilamid - TBE jeli üzerinde% 8 akrilamid - TBE jeli üzerinde 6x yükleme boyası ile seyreltilmiş tek tek oligonükleotidlerin yanında küçük bir hacim (1 μL) tavlanmış oligonükleotid çalıştırın.
- Jeli, uygun bir nükleik asit boyası ile desteklenmiş 1x TBE'de 30 dakika boyunca bir tahterevalli sallanan çalkalayıcıda bekletin, ardından jeli bir jel görüntüleme sistemi kullanarak tarayın.
- % 8 akrilamid - TBE jeli hazırlayın (4 jel için: 32.8 mL H2O; 4.8 mL 10x TBE tamponu (1 M Tris, 1 M borik asit, 20 mM EDTA), 9.6 mL %40 Akrilamid / Bis Çözeltisi, 37.5: 1; 0.8 mL %10 Amonyum persülfat [APS], 40 μL Tetrametiletilendiamin [TEMED]).
- Jel eksizyonu için TBE-PAGE jel üzerinde tavlanmış DNA'yı çözün.
- Oligonükleotidler uygun şekilde tavlanırsa, numunenin geri kalanını BSA (~ 100 μL tavlanmış numune, 25 μL 6x yükleme boyası, 2 μL 33 mg / mL BSA) varlığında çözün. Daha büyük hacmi jel üzerine yüklemek için, birkaç kuyucuğu steril bir neşter ile birleştirin.
- Jeli, uygun nükleik asit boyası ile desteklenmiş 1x TBE'de 30 dakika boyunca bir tahterevalli sallanan çalkalayıcı üzerinde bırakın.
- Jeli bir cam tepsiye aktarın ve DNA'yı mavi ışık altında karanlık bir odada görselleştirin. Mavi ışık kullanırken uygun bir koruyucu ekran veya koruyucu gözlük kullanın.
- İstenilen bandı steril bir neşter ile kesin. Fazla jeli kesin.
- Tavlanmış çatalı elektroelüsyon ile saflaştırın.
- Jel parçasını yerleştirecek kadar uzun bir diyaliz tüpü parçası kesin, 1x TBE'de ıslatın ve bir ucunu kapatın.
- BSA'yı 500 μL 1x TBE'de 0,3 mg / mL'ye seyreltin ve tüm hacmi tüpe pipetleyin.
- Jel parçasını içeri kaydırmak için metal spatulayı kullanın. Elektroelüsyon sırasında DNA'nın göç etmesi için tampon alanı sağlamak için jel parçasını borunun bir tarafına hareket ettirin.
- Fazla tamponu sıkın ve borunun ikinci ucunu klipsleyin.
NOT: Kalan hacim, nihai hacmi ve konsantrasyonu belirleyecektir. - Diyaliz tüpünü 1x TBE tamponundaki agaroz elektroforez tankına yerleştirin ve tüpün tamamen suya daldırıldığından emin olun.
NOT: DNA'nın jelden diyaliz torbasına geçebilmesi için jel parçasının negatif elektroda en yakın tarafa yerleştirildiğinden emin olun. - Elektroelüsyonu 1-2 saat boyunca 80 V'ta çalıştırın.
- Elektronik DNA'yı 10 mM Tris tamponuna (pH 8) diyalize edin.
- Elektroelüsyondan sonra diyaliz torbasını tanktan çıkarın ve bir ucunu doku ile kurulayın. Borunun orta kısmına, jelin ve numunenin bulunduğu doku ile dokunmaktan kaçının.
- Klipsi kurutulmuş uçtan çıkarın ve DNA'yı tampona karıştırmak için numuneyi borunun içinde yukarı ve aşağı nazikçe pipetleyin.
- Temiz bir spatulayı 1x TBE tamponuna batırın ve jel parçasını borudan çıkarın.
NOT: Jel parçasını çıkarırken mümkün olduğunca az tampon çıkarın. - Diyaliz torbasının açık ucunu tekrar kapatın ve içerideki havayı alın.
- Diyaliz torbasını 1,5 L 10 mM Tris tamponu (pH 8), 20 mM NaCl ve 2 mM MgCl2 ile doldurulmuş 2 L'lik bir behere yerleştirin. Boruyu tampona tamamen daldırırken boruyu beherin kenarına yapıştırmak için klipsleri parafilm ile sarın.
NOT: Tuz, kısa oligo dubleksleri stabilize eder. - Tamponu hafifçe karıştırmak için manyetik bir çubuk kullanın ve numuneyi oda sıcaklığında (RT) en az 3-4 saat veya gece boyunca 4 °C'de diyalize edin.
- Diyalizden sonra, diyaliz torbasının bir ucunu bir mendille kurulayın (DNA'yı seyreltmemek için) ve bu ucun klipsini açın.
- Numuneyi borunun içinde yukarı ve aşağı nazikçe pipetleyin ve 1.5 mL'lik temiz bir tüpe aktarın.
- Bir mikrohacim spektrofotometresi kullanarak DNA'nın konsantrasyonunu ölçün.
- Çatal DNA'sını tavlayın.
- 20 kb'lık parça oluşturma
- NotI/I-CeuI enzimlerini kullanarak pGC261 plazmid18'in kısıtlama sindirimini gerçekleştirin.
- 8 μL NotI-HF (20.000 U/mL), 8 μL I-CeuI (5.000 U/mL) ve pGC261 plazmitini (200 μL'lik bir reaksiyonda nihai plazmit konsantrasyonu ~ 40 ng / μL) restriksiyon enzimleri üreticileri tarafından önerilen bir tamponda nazikçe karıştırın.
- Reaksiyonu gece boyunca 37 °C'de inkübe edin.
- Jel eksizyonu için agaroz jel üzerinde sindirilmiş DNA'yı çözün.
- İSTEĞE BAĞLI: Tüm reaksiyonu yüklemeden önce sindirim verimliliğini test etmek için önce %0.6'lık bir agaroz jel üzerinde küçük bir miktar reaksiyon çalıştırın.
- 1.2.2.3-1.2.2.4 adımlarını izleyerek% 0.6 agaroz jeli hazırlayın.
- 0.48 g agaroz tozunu 80 mL 1x TBE tamponu ile karıştırın. Çözeltiyi kaynayana kadar mikrodalgada pişirin. Agarozun tamamen eridiğinden emin olmak için döndürün. Birkaç dakika soğumaya bırakın, ardından belirtilen tanka dökün.
- Numunenin daha büyük hacmini barındıracak şekilde kuyuları birleştirmek için, önceden tarağın birden fazla kuyusuna bir parça bant yapıştırın.
- Jel katılaştıktan sonra tarağı nazikçe çıkarın ve tankı 1x TBE tamponu ile doldurun. Numuneyi DNA yükleme boyası ile nazikçe karıştırın ve jeli 1 saat boyunca 120 V'ta çalıştırın.
- Jeli, lekelenene kadar bir tahterevalli sallanan çalkalayıcı üzerinde uygun bir nükleik asit boyası ile desteklenmiş 1x TBE içinde bırakın.
- Lekeli jeli bir cam tepsiye aktarın ve DNA'yı mavi ışık altında karanlık bir odada görselleştirin.
- İstenilen bandı steril bir neşter ile kesin. Fazla jeli kesin.
- İsteğe bağlı: İstenen parçayı adım 1.1.4 ve 1.1.5'te açıklandığı gibi elektroelüsyon ile saflaştırın. Diyaliz tamponundaki tuzu burada atlayın, çünkü bu sadece kısa oligo dupleksi stabilize etmek için gereklidir.
- NotI/I-CeuI enzimlerini kullanarak pGC261 plazmid18'in kısıtlama sindirimini gerçekleştirin.
- Digoksigenin dubleks DNA ucunun üretilmesi
- Şablon olarak pGC261 DNA'yı kullanarak digoksigenin-dUTP (dig-dUTP) ile PCR reaksiyonu gerçekleştirin.
- Karışım: 400 μL su, 8 μL pGC261 (0.8 ng/μL), 3.5 μL primer oHY507 (100 μM), 3.5 μL primer oHY508 (100 μM), 8 μL dig-dUTP (1 mM), 400 μL 2x PCR karışımı (DNA polimeraz (20 μL), 10 mM dNTP'ler (40 μL), 5x yüksek kaliteli (HF) tampon (400 μL) ve su (540 μL).
- Bir termal döngüleyicide aşağıdaki reaksiyonu çalıştırın:
98 °C - 1 dk
30x: 98 °C - 20 sn; 65 °C - 20 sn; 72 °C - 30 sn;
72 °C - 10 dk
4 °C - Basılı tutun
- Digoxigenin etiketli PCR ürününü sindirin ve saflaştırın
- Ticari bir DNA saflaştırma kiti kullanarak PCR ürününü saflaştırın.
- 10 μL NotI-HF'yi (20.000 U/mL) enzim tedarikçisi tarafından önerilen bir tamponda PCR ürünü ile karıştırın (~200 μL'lik bir reaksiyonda ~50 ng/μL'lik nihai DNA konsantrasyonu). Reaksiyonu gece boyunca 37 °C'de inkübe edin.
- Ticari bir DNA saflaştırma kiti kullanarak sindirilmiş DNA'yı saflaştırın.
- Şablon olarak pGC261 DNA'yı kullanarak digoksigenin-dUTP (dig-dUTP) ile PCR reaksiyonu gerçekleştirin.
- DNA substratını yapmak için bileşenleri birleştirin.
- Biyotinile çatal ucunu, 20 kb'lık DNA fragmanını ve digoksigenin etiketli Not-I ile muamele edilmiş PCR fragmanını 5 μL T4 DNA ligaz (400.000 U/mL) ile enzim tedarikçisi tarafından 200 μL'lik bir reaksiyonda nazikçe karıştırın. Bununla birlikte, eklenen 20 kb'lık DNA fragmanına (1-5 μg) dayanarak, hem biyotinile çatal ucunun hem de PCR fragmanının yaklaşık 100 kat molar fazlalığını ekleyin.
- Reaksiyonu PCR tüplerine (her biri 50 μL) alın ve gece boyunca bir termal döngüleyicide 16 °C'de inkübe edin.
- Bağlanmış numuneyi %0.6'lık bir agaroz jel üzerinde çözün ve bağlanmış DNA'yı adım 1.2.2 ve 1.2.3'te açıklandığı gibi elektroelüsyon ile saflaştırın.
- Flaş donmuş DNA'yı -80 °C'de saklayın.
2. Drosophila melanogaster CMG'nin saflaştırılması (Şekil 2)

Şekil 2: Drosophila melanogaster CMG'nin 4 L Hi Five hücresinden saflaştırılması. Proteinler, MOPS tamponu varlığında 200 V altında% 4 -% 12 Bis-Tris poliakrilamid jel üzerinde çözüldü. Numune, saflaştırmanın her aşamasında gösterilir (hücre lizatı - 2 μL, FLAG elüsyon - 10 μL, ilk iyon değişim kolonundan sonra - 10 μL ve etiketlemeden sonra ve ikinci iyon değişim kolonundan sonra - 1 μL. (A) Coomassie boyama, (10 μL) floresan etiketlemeden önce ve sonra (1 μL) CMG kompleksinin 11 alt biriminin tümünün varlığını doğrular. (B) MCM3 alt biriminin etiketleme verimliliği, uzun geçiren kırmızı (LPR) filtre kullanan bir floresan görüntü analizörü ile Cy5 taraması yapılarak doğrulanmıştır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
NOT: Floresan olarak etiketlenmiş Drosophila melanogaster CMG'yi hazırlamak için, MCM3 alt biriminde (pFastBac1 vektöründe) 10 N-terminal FLAG Tag'in akış aşağısına bir TEV bölünme bölgesi (ENLYFQG) ve ardından dört Gly kalıntısı eklendi. Kompleksi ifade etmek için bakulovirüs ekspresyon sistemi kullanıldı. İlk transfeksiyon için, Sf21 hücreleri her CMG alt birimi (P1 virüs aşaması) için ayrı ayrı kullanıldı. Virüsleri daha da çoğaltmak için Sf9 hücreleri kullanıldı (P2 virüs aşaması). Daha sonra, Sf9 hücre kültürleri (her CMG alt birimi için 100 mL; 0.5 x 106 hücre / mL),% 10 fetal buzağı serumu (P3 virüs aşaması) ile desteklenmiş 0.5 mL P2 virüsü ile enfekte edildi. Tüm CMG kompleksini 4 L Hi Five hücrelerinde (1 x 106 hücre / mL) eksprese etmek için, alt birimlerin her biri için 200 mL P3 virüsü kullanıldı. CMG kompleksini ifade eden Hi Five hücrelerinin hasat edilmesinden sonra, hücre peleti sıvı nitrojen içinde hızlı dondurulabilir ve -80 ° C'de saklanabilir. Tüm saflaştırmayı buz üzerinde veya 4 °C'de gerçekleştirin. Tamponlar, indirgeyici ajanların (DTT veya 2-Merkaptoetanol) ve proteaz inhibitörlerinin (DİKKAT) kullanımdan hemen önce eklenmesi şartıyla önceden hazırlanabilir. Tüm tamponların önceden soğutulduğundan, filtrelendiğinden ve gazının alındığından emin olun.
- Aşağıdaki arabellekleri hazırlayın.
- 25 mM 4- (2-hidroksietil) -1-piperazineetansülfonik asit (HEPES) pH 7.5,% 0.02 Tween-20,% 10 gliserol, 15 mM KCl, 2 mM MgCl2, 2 mM 2-merkaptoetanol, 1 mM EDTA ve 1 mM etilen glikol-bis (β-aminoetil eter) -N, N, N', N', N'-tetraasetik asit (EGTA).
DİKKAT: 2-merkaptoetanol, DTT ve EDTA toksik/zararlıdır - ANTI-FLAG M2 afinite jeli hazırlamak için Tris tamponlu salin (TBS; 0.1 M Glycine HCl, pH 3.5) tamponu hazırlayın.
- 25 mM HEPES pH 7.5, %0.02 Tween-20, %10 gliserol, 100 mM KCl, 1 mM DTT, 1 mM EDTA ve 1 mM EGTA'yı karıştırarak Tampon A-100'ü hazırlayın.
- 25 mM HEPES pH 7.5, 50 mM sodyum asetat, 10 mM magnezyum asetat, %10 gliserol ve 1 mM DTT ekleyerek diyaliz tamponu hazırlayın.
- 50 mM Tris-HCl pH 7.5 ve 150 mM NaCl'yi karıştırarak TBS tamponunu hazırlayın.
- 25 mM 4- (2-hidroksietil) -1-piperazineetansülfonik asit (HEPES) pH 7.5,% 0.02 Tween-20,% 10 gliserol, 15 mM KCl, 2 mM MgCl2, 2 mM 2-merkaptoetanol, 1 mM EDTA ve 1 mM etilen glikol-bis (β-aminoetil eter) -N, N, N', N', N'-tetraasetik asit (EGTA).
- Proteaz inhibitörü kokteyl tabletleri ile desteklenmiş 200 mL soğuk resüspansiyon tamponu A'da 4 L Hi Five hücre kültüründen peleti yeniden süspanse edin. Tüm pelet tamamen yeniden süspanse olana kadar tüpleri ters çevirerek karıştırın.
- Yeniden süspanse edilmiş hücreleri önceden soğutulmuş 40 mL Dounce homojenizatöre aktarın. Mümkün olduğu kadar çok hücreyi geri kazanmak için, hücre peletinin dondurulması için kullanılan tüpleri aynı tip tamponla yıkayın ve Dounce homojenizatöre ekleyin. Homojenizatördeki toplam hacmin önerilen sınırı aşmadığından emin olun. Hücreleri buz üzerinde 60-70 vuruşla parçalayın.
NOT: Pistonun borunun dibine ulaştığından emin olun, ancak homojenizatör kırılabileceğinden çok fazla basınç uygulamayın. Pistonu sıvı seviyesinin üzerine çıkarmamaya dikkat edin, çünkü bu kabarcıklara neden olur. - Numunenin tamamı homojen hale gelene kadar önceki adımı tekrarlayın ve önceden soğutulmuş beherde birleştirin. Numunenin son hacmini değerlendirin. Damla damla, hafifçe karıştırarak 100 mM'lik son konsantrasyona filtrelenmiş KCl ekleyin.
- Santrifüjü önceden 4 °C'ye soğutun. Numuneyi santrifüj tüplerine dökün ve üretici tarafından önerilen hacme ulaşıldığından emin olun. Tüpleri bir ölçekte dengeleyin.
NOT: Çok küçük hacimli tüpler santrifüjleme sırasında kırılabilir. - Parçalanmış hücreleri 23.500 x g'da 4 ° C'de 15-30 dakika santrifüjleyin. Bu esnada ANTI-FLAG M2 afinite jelini bir sonraki adıma uygun olarak hazırlayınız.
- ANTI-FLAG M2 afinite jeli (%50 süspansiyon) içeren şişeyi nazikçe karıştırın. Bir P-100 pipet ucunun ucunu kesin (ucu genişletmek için) ve 4 mL süspansiyonu (2 mL boncuk) hemen 20 mL'lik bir poli-hazırlık kromatografi kolonuna aktarmak için kullanın. Uçta sıkışmış boncukları kurtarmak için, ucu TBS ile yıkayın ve sütuna ekleyin. Tüm boncukların kolonun içine yerleşmesine izin verin, ancak kurutmamaya dikkat edin.
- Reçineyi 3x boncuk hacmi (toplam 6 mL) 0.1 M Glisin HCl pH 3.5 ile yıkayın. Daha sonra, boncukları 3-5 boncuk hacmi TBS (6-10 mL) ile yıkayın. Ardından, boncukları Tampon A-100 ile 3 kez (6 mL) yıkayın.
NOT: Boncukları 0.1 M Glisin HCl pH 3.5 çözeltisinde 20 dakikadan daha uzun süre bırakmayın. Akış basıncını ve yıkama hızını artırmak için kolonu bir kapakla örtün. Reçinenin hiçbir noktada kurumasına izin vermeyin. - Reçineyi hazırladıktan sonra, reçinenin üzerinde 2 mL Tampon A-100 bırakın ve kolonun çıkışını kapatın.
- Santrifüjlemeden sonra, süpernatanı önceden soğutulmuş 50 mL'lik tüplere yavaşça dökün. Peleti bozmamaya dikkat edin. Daha sonra SDS-PAGE elektroforezi ile doğrulamak için küçük bir alikot (~ 10 μL) alın.
- 50 mL'lik tüplerin her birine eşit miktarda ANTI-FLAG M2 boncuk (önceki adıma uygun olarak hazırlanmış) ekleyin. Kolondaki tüm boncukları, birkaç mililitre ek Tampon A-100 ile yeniden süspanse ederek ve bu yeniden süspansiyonu tüplere aktararak kurtarmaya çalışın.
- Numuneyi (süpernatant) ANTI-FLAG M2 reçine ile 4 °C'de 2,5 saat döndürerek inkübe edin.
- İnkübasyondan sonra, numuneyi 4 ° C'de 5 dakika boyunca 1.000 x g'da döndürün. Bir pipet kullanarak, süpernatantın çoğunu çıkarın. Kesilmiş bir uç kullanarak, boncukları kalan süpernatantın birkaç mililitresinde yeniden süspanse edin ve bunları iki adet 15 mL'lik tüpe aktarın. 50 mL'lik tüplerin duvarlarına yapışan boncukları kurtarmak için birkaç mililitre Tampon A-100 ekleyin ve bunu 15 mL'lik tüplere de aktarın.
- 15 mL'lik tüpleri 1,000 x g'da 4 ° C'de 5 dakika döndürün. Süpernatanı çıkarın.
- Her tüpe ~ 14 mL Tampon A-100 ekleyerek, ardından 4 ° C'de kısa, yumuşak dönüş ve ardından 4 ° C'de 5 dakika boyunca 1000 x g'da sıkarak boncukları yıkayın. Yıkamaları iki kez tekrarlayın. Daha sonra SDS-PAGE elektroforezi ile doğrulamak için her yıkamadan (~ 10 μL) küçük alikotlar alın.
- Bu adım (2.16), önceki adımlara (2.13-2.15) alternatif bir prosedürdür.
- ANTI-FLAG M2 reçinesi ile inkübasyondan sonra, numuneyi iki adet 20 mL'lik poli-prep kromatografi kolonuna dökün.
- Bu yöntemi seçerseniz, boncukların kolonun dibine yerleşmesine ve bağlanmamış numunenin yerçekimi akışıyla kolondan geçmesine izin verin. Daha sonra, Tampon A-100'ü doğrudan kolona ekleyin ve numuneyi yerçekimi akışı yoluyla yıkamasına izin verin.
- Boncukları (Tampon A-100 içinde yeniden süspanse edilmiş) iki adet 10 mL'lik poli-hazırlık kromatografi kolonuna aktarın.
- Tampon A-100'ün reçineden geçmesine izin verin, tampon seviyesi reçinenin tepesine ulaştığında çıkışı kapatın ve 200 μg / mL (DYKDDDDK) peptit (FLAG elüsyon tamponu) ile desteklenmiş Tampon A-100 ekleyerek proteini boncuklardan ayırın.
- İlk elüsyon için, 3 mL FLAG elüsyon tamponu ekleyin ve kapalı sütunu RT'de 15 dakika boyunca yavaşça döndürün. İlk elüsyon fraksiyonunu toplamak için çıkışı açın.
- Çıkışı kapatın ve her sütuna 2 mL FLAG elüsyon tamponu ekleyin. 10 dakika daha döndürün ve ikinci elüsyon fraksiyonunu toplayın.
- Tüm ayrıştırılmış fraksiyonları bir araya getirin (~ 10 mL) ve 4 ° C'de tutun. Daha sonra SDS-PAGE elektroforezi ile doğrulamak için ayrıştırılmış fraksiyonun (~ 10 μL) küçük bir alikotunu alın.
- 0.22 μm şırınga filtreleri kullanarak ayrıştırılan numuneyi filtreleyin.
- Önceden, üreticinin protokolüne uygun olarak bir protein saflaştırma sistemine bağlı CaptoHiRes Q (5/50) sütununu (yüksek çözünürlüklü iyon değişim kromatografisi sütunu) hazırlayın. Ardından, sütunu Tampon A-100 ile dengeleyin ve filtrelenmiş numuneyi dengelenmiş sütuna yükleyin.
- Sütunu 20 sütun hacmi (CV) Tampon A-100 ile yıkayın (kolonun hacmi ~ 1 mL olduğundan toplam ~ 20 mL).
- Proteini elüte etmek için önceden iki tampon hazırlayın: Tampon A-100 ile aynı bileşime sahip, ancak 100 mM KCl yerine 550 mM KCl ile Tampon A-100 ve Tampon B. 100 mM'den 550 mM KCl'ye artan tuz konsantrasyonu ile 20 CV (~ 20 mL) için gradyan elüsyonu ayarlayın. Bir fraksiyon toplayıcıda 0.3-0.5 mL elüsiyon toplayın.
NOT: CMG, Tampon B'nin yaklaşık% 70 -% 75 konsantrasyonunda elüte olmalıdır. SDS-PAGE elektroforezi için yıkama ve ayrıştırılmış fraksiyonların küçük alikotlarını alın. - Seçilen fraksiyonlarda CMG'nin mevcut olduğunu doğrulamak için SDS-PAGE elektroforezi gerçekleştirin. Bu arada, kolonu ve arıtma sistemini üreticinin protokolüne göre temizleyin.
- 1 mL numune başına 50 μL TEV proteaz (1 mg/mL) karıştırarak seçilen fraksiyonları gece boyunca TEV proteaz ile inkübe edin.
- Bir diyaliz tüpünü önceden ıslatın ve zarın hasar görmediğinden emin olun. CMG/TEV karışımını diyaliz tüpüne ekleyin ve 1,5 L önceden soğutulmuş Tampon A-100 ve içindeki manyetik çubuk ile 2 L'lik behere yerleştirin. Gece boyunca 4°C'de hafifçe karıştırarak diyalize edin.
- Numuneyi 5 mM CaCl2 varlığında 50 μM LD655 işaretli peptit ve 10 μg/mL Sortase enzimi ile karıştırın. Reaksiyonu 4 ° C'de inkübe edin, 30 dakika döndürün ve tüpün ışıktan kaplandığından emin olun. SDS-PAGE elektroforezi için etiketlemeden önce ve sonra küçük bir alikot alın.
- Peptit çökelebileceğinden, saflaştırma sistemine yüklemeden önce etiketli numuneyi 0,22 μm santrifüj filtreleri kullanarak filtreleyin.
- Yüksek çözünürlüklü iyon değişim kromatografisi kolonunu daha önce açıklandığı gibi önceden hazırlayın (adım 2.20). Bu sefer numuneyi ışıktan korumak için kolonu ve fraksiyon toplayıcı sistemini alüminyum folyo ile kaplayın.
- Serbest peptidi çıkarmak için, filtrelenmiş numuneyi yüksek çözünürlüklü iyon değişim kromatografisi kolonuna yükleyin. Saflaştırmayı daha önce anlatıldığı gibi gerçekleştirin (adım 2.21-2.22). Etiketli CMG'nin daha öncekine benzer bir Tampon-B konsantrasyonunda ortaya çıktığından emin olun.
- Seçilen fraksiyonların kalitesini doğrulamak için SDS-PAGE elektroforezi gerçekleştirin. Floresansı görüntülemek için, yüklemeden önce numuneleri kaynatmayın ve elektroforez tankının ışıktan korunduğundan emin olun. Önce bir jel görüntüleme sistemi kullanarak protein floresansını görselleştirin, ardından jeli Coomassie boyası ile boyayın ve tüm proteinleri görselleştirmek için tekrar görüntüleyin.
- Seçilen fraksiyonları gece boyunca 4 ° C'de, 1,5 L diyaliz tamponuna karşı diyaliz edin. Gerekirse, numuneyi konsantre edin.
- Proteini sıvı nitrojen içinde dondurun ve daha fazla kullanılana kadar -80 ° C'de saklayın.
3. Akış hücresinin hazırlanması (Şekil 3)
- Fırında pişirme adımını atlayarak daha önce açıklanan protokol11'i izleyerek biotin-PEG lamellerini hazırlayın. Biotin-PEG lameller RT'de vakum altında en az 1 ay stabildir. Akış hücresinin hazırlanması için biotin-PEG lamelini (24 mm x 60 mm) ikiye bölün (yaklaşık 24 mm x 30 mm).
NOT: Lamel ile akış kanallarının olacağı orta kısmına dokunmamaya dikkat ediniz. - Bir cam slaytı yaklaşık 2,4 cm x 1 cm'lik parçalar halinde aşındırarak ve oturtarak küçük cam parçaları hazırlayın.
- Biri diğerinden biraz daha geniş olan 1.4 mm elmas kaplı bir matkap ucu kullanarak 0.8 mm aralıklarla iki delik açın (0.043" = giriş, 0.048" = çıkış). Boru yerine oturana kadar deliği genişletmek için aynı matkap ucunu kullanarak giriş/çıkış borusunun bir parçasını yerleştirmeye çalışarak deliklerin doğru boyutta olduğunu test edin. Borunun kolayca düşmeyecek kadar sıkı oturduğundan emin olun.
NOT: Ölü hacmi en aza indirmek için daha dar giriş borusu kullanılır. - Cam parçalarıyla aynı şekilde çift taraflı bant kesin.
- Slaytı bant üzerinde hizalayın ve bant üzerindeki konumlarını işaretlemek için her delikten bir iğne saplayın. Bir tıraş bıçağı kullanarak her iki deliği de kapsayan bir kanal kesin.
NOT: Kanalı çok uzun süre kesmeyin, çünkü her deliğin uzak tarafındaki fazla alan, sıvı aktığında kuru kalabilir ve akış sırasında hava kabarcıkları ile ilgili sorunlara yol açabilir. Keserken, uzun kenarları tek bir temiz kesimde kesmeye çalışın, çünkü birden fazla kesimden kaynaklanan yıpranmış kenarlar akış kanalının kalitesini etkileyebilir. Benzer bir amaçla, akış kanalının kenarlarını kesmeyin, çünkü kullanılabilir parçaya yapılan kesikler sıvı akışını etkileyebilir ve hatta monte edilmiş akış hücresinde sızıntı yapabilir. - Cam parçasını kuruyana kadar aseton ve doku ile temizleyin, ardından temiz bir yüzeye yerleştirin. Yapışkan bandın bir tarafını soyun ve her iki delik de kanalın içinde tamamen olacak şekilde cam parçasının üzerine yapıştırın.
- Yüzeye sert ancak orta derecede bir basınçla bastırarak kabarcıkları ortadan kaldırmak için bir p1000 pipet ucu kullanarak bandı cam parçaya kapatın. Mühürlemek için tüm yüzeyin üzerinden geçin.
- Bandın ikinci kenarını her iki cam parçasından soyun ve yapışkan tarafı yukarı bakacak şekilde temiz bir yüzeye yerleştirin. Slaytları birbirine yakın yerleştirin, ancak lamele takılacakları konuma dokunmayın (epoksinin her bir cam parçasının etrafında tam bir sızdırmazlık oluşturması için aralarında yeterli boşluk bırakın).
- Yarım biotin-PEG lamelini plastik cımbız kullanarak, sadece kenarından tutarak alın (akış kanalını oluşturacak alana dokunmayın) ve PEG ile işlevselleştirilmiş tarafı yapıştırıcının üzerine indirin. Yerine sabitlemek için parmağınızla bastırarak sabitleyin, ardından hava kabarcıklarını çıkarmak için yüzeyi bir pipet ucuyla orta derecede sıkıca ovalayarak contayı tamamlayın (bu kırılabileceğinden akış kanalının alanında değil), ardından ters çevirin.
NOT: Lameli dikkatli tutun. - Her akış hücresi için, her tip polietilen borudan (PE20 ve PE60) ~ 10 cm kesin. Ölü hacmi azaltmak için daha dar boruyu akış odasına giriş olarak kullanın.
- Sürgüdeki deliklere elle boru yerleştirin. Deliklerin çapı doğruysa, borunun yerleştirildikten sonra delikte kendi kendine durduğundan emin olun. Borunun yerleştirilmesini biraz daha kolaylaştırmak için boru ucunu hafif bir açıyla <45° kesin ve borunun lamele karşı bir sızdırmazlık oluşturmasını önleyin, bu da uç düzse yapabilir (bu, akışı engelleyebilir).
NOT: Borunun cam parçaya doğru şekilde oturması, akış hücresi yapısını daha güvenilir hale getirir ve akış hücresine kabarcıkların girmesini önlemeye yardımcı olur. - Epoksi bileşenlerini iyice karıştırın, ardından boruyu kapatmak ve her cam parçasının etrafında bir sızdırmazlık oluşturmak için epoksiyi delmek için bir p200 ucu kullanın. Lamel turunun kenarına kadar gidecek kadar epoksi ekleyin, çünkü bu aynı zamanda hassas lameli de güçlendirecektir. Bununla birlikte, lamelin alt tarafına epoksi girmediğinden emin olun, çünkü bu, sahnenin üzerinde düz durmasını engelleyebilir.
NOT: Gerekirse, az miktarda fazla epoksi, bir tıraş bıçağı kullanılarak alt taraftan kazınabilir. - Epoksinin tamamen kürlenmesi için en az 30-60 dakika bekletin.
NOT: Akış hücresi hemen kullanılmayacaksa, tamamen sertleştikten sonra RT'de vakum altında saklayın. - Akış hücresini kullandıktan sonra, cam parçalarını geri kazanın ve süresiz olarak yeniden kullanın. Epoksi ve çift taraflı yapıştırıcıyı yumuşatmak için hortumu dışarı çekin ve akış hücresini en az 24 saat boyunca aseton içeren bir sürgülü kavanoza yerleştirin, böylece cam parçalarının kolayca çıkarılmasını sağlayın. Başka bir akış hücresi yapmak için tekrar kullanmadan önce, cam parçalarını sünger ve sabunlu suyla ovarak temizleyin, kurulayın ve aseton ve kağıt mendil ile ovalayın.

Şekil 3: Akış hücresi hazırlığının grafiksel gösterimi. (A) Cam parçasının boyutuna uyacak şekilde çift taraflı bant kesin. Sürgüyü bandın üstüne hizalayın ve her deliğin konumunu bir iğne ile işaretleyin. Bir tıraş bıçağı kullanarak, bir kanal oluşturmak için her tutuşun etrafını kesin. (B) Bandın bir tarafını soyun ve bandı cam parçasının üzerine yapıştırın. Her iki deliğin de kanalın içinde olduğundan emin olun. Bandın ikinci ucunu soyun ve biotin-PEG lamelini üstüne yapıştırın. (C) Polietilen boruyu her deliğe yerleştirin ve boruyu epoksi ile yerine kapatın, her bir cam parçasını da lamel ile kapatın. (D) Her iki kanalı da kullandıktan sonra, boruyu dışarı çekin ve akış hücresini asetonla doldurulmuş bir boyama kavanozuna yerleştirin. Yaklaşık 24 saat sonra, epoksi ve bant yumuşamış olacak ve akış hücresinin katmanları soyulabilir. Cam parçaları geri kazanılabilir ve bir sonraki akış hücresini yapmak için süresiz olarak yeniden kullanılmak üzere asetonda saklanabilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
4. CMG aracılı DNA çözülmesini görselleştirmek için tek moleküllü TIRF testi
- Aşağıdaki arabellekleri hazırlayın.
- Bloke edici tampon hazırlayın (20 mM Tris, pH 8.0; 50 mM NaCl; 2 mM EDTA; 0.2 mg / mL BSA). Tamponu 0,22 μm'lik bir şırınga filtresi kullanarak filtreleyin ve 4 °C'de saklayın.
- 10x Reaksiyon tamponu 1 (250 mM HEPES pH 7.5; 50 mM NaCl; 100 mM Magnezyum Asetat) hazırlayın. Tamponu 0,22 μm'lik bir şırınga filtresi kullanarak filtreleyin ve 4 °C'de saklayın.
- 10x Reaksiyon tamponu 2 hazırlayın (250 mM Tris, pH 7.5; 100 mM Magnezyum Asetat; 1.25 M Potasyum glutamat; 1 mM EDTA; %0.025 Ara-20). Tamponu 0,22 μm'lik bir şırınga filtresi kullanarak filtreleyin ve 4 °C'de saklayın.
- Açık tüpleri bir desikatöre yerleştirerek ve 15 dakika vakum altında bırakarak yaklaşık 2,5 mL bloke edici tampon ve 5 mL ultra saf suyun gazını alın.
NOT: Gaz giderme sırasında kabarcıklar sıvının sıçramasına neden olabileceğinden tüpleri ağzına kadar doldurmayın. - Akış hücresini bir mikroskop tablasına yerleştirin ve her iki ucunda yapışkan bir macun kullanarak yerine sabitleyin. Akış hücresinin edinim sırasında daha sonra hareket etmesini önlemek için iyi bir şekilde emniyete almak önemlidir.
- Akış hücresi çıkış hortumunu, diğer ucunda bir iğne ile şırıngaya bağlı boru bulunan şırınga pompasına bağlayın. Akış hücresini bağlamak için bu iğneyi çıkış borusuna sokun.
- Objektif ısıtıcısını 30 °C'ye getirin.
- Bir tüpteki 1-2 mL gazı alınmış suyu, giriş borusunun yerleştirilebileceği ve borunun dibine ulaşabileceği şekilde akış hücresinin yakınında ayrı bir yapışkan macun parçasına sabitleyin.
- Kanaldan su akıtın, ardından giriş borusunun yakınında sıkışan kabarcıkları çıkarmak için daha hızlı akış kullanın ve gerekirse daha fazla su ekleyin. Aksi takdirde, büyük kabarcıklar deneylerde daha sonra yerinden çıkabilir ve kanaldan geçebilir. Akışın tamamen durması için birkaç dakika bekletin, çünkü hızlı akış durdurulduktan sonra şırınga içindeki basınç stabilize olduğu için sıvının bir süre daha fazla çekilmesine neden olabilir.
NOT: Bir baloncuğa temas eden herhangi bir DNA, yeniden sulandırılsa bile kullanılamaz hale gelir. - 20 μL'lik 1 mg / mL streptavidin alikotuna 100 μL gazı alınmış bloke edici tampon ekleyin. Açık tüpü yapışkan macuna takın ve giriş tüpünü sudan streptavidin'e aktarın. 2 dakika boyunca 40 μL / dak hızında akış yapın (toplam 80 μL) ve 5 dakika inkübe edin.
- Fazla streptavidini bloke edici tamponla yıkayın (100 μL için 50 μL / dak).
- Biyotinile edilmiş DNA'da akış, 25 nM SYTOX portakal ile bloke edici tamponda seyreltilir. DNA'nın yüzeye gerçek zamanlı olarak bağlanmasını izlemek için 532 nm lazerle canlı görüntü kullanan görüntü.
- Yüzeydeki DNA'nın yaklaşık yoğunluğu elde edildiğinde, serbest DNA'yı yıkamak için yine 25 nM SYTOX (100 μL için 50 μL/dak) ile bloke edici tamponda akıtın.
- 25 nM SYTOX turuncu (300 μL için 100 μL / dak) içeren bloke edici tamponda seyreltilmiş biyotinile anti-digoksigenin antikorunda (~ 10 μg / mL) akış.
NOT: SYTOX turuncu, DNA'nın baz çiftleri arasında interkalasyon yaparak kontur uzunluğunu uzatır. Bu, DNA'nın uçtan uca uzunluğunun SYTOX turuncusunda arttığı, ikinci bağın yüzeye birinciden daha fazla yapışmasına izin verdiği ve SYTOX turuncusu daha sonra yıkandığında daha iyi gerilmiş DNA verdiği anlamına gelir. Her DNA ucunda farklı bir parça kullanılması, DNA'nın lamel yüzeyine daha düzgün bir şekilde bağlanmasına izin verir (Şekil 4). Her iki uç da biyotinile edildiğinde, aynı adımda bağlanırlar ve bu da bağlanma pozisyonlarında önemli değişikliklere neden olur. Bir uçta digoksigenin kullanmak, ikinci ucu bağlamadan önce DNA kapsamı üzerinde daha fazla kontrol sağlar. Bu aynı zamanda DNA kapsamının inkübasyon ile arttırılmasına da izin verir. - Biyotinile edilmiş anti-digoksigenin antikorunu ve SYTOX turuncusunu bloke edici tamponla (100 μL için 50 μL / dak) yıkayın.
- 120 μL 'ATP-g-s karışımı' oluşturun (1x Reaksiyon tamponu 1, 0.75 mg / mL BSA, 1.25 mg / mL kazein, 8 mM DTT, 0.33 mM ATP-g-s), yeni bir tüpe 30 μL ekleyin, ardından orijinal tüpten akış hücresine 50 μL akış hücresine (50 μL için 50 μL / dak). Bu, çıkış borusundan geri akış veya difüzyon yoluyla tampon karıştırmanın etkisini en aza indirmeye yardımcı olur.
- 30 μL ATP-g-s karışımında ~ 100 nM finale saflaştırılmış CMG ekleyin, ardından 20 μL için 20 μL / dk'da akıtın. 15 dakika inkübe edin.
- 120 μL 'ATP/RPA karışımı' (1x Reaksiyon tamponu 1, 0.75 mg/mL BSA, 8 mM DTT, 3.3 mM ATP, 20 nM EGFP-hRPA) oluşturun ve 80 μL için 40 μL/dk'da akış hücresine akış.
- Hemen görüntü almaya başlayın. Her 30 saniyede bir her kare için 6 x 6 görüş alanı (veya deney için uygun bir kare hızı) elde edin. Daha düşük kare hızları, lazer maruziyetini ve florofor foto ağartmayı azaltabilir. EGFP-hRPA'yı %1 güçte 488 nm lazerle görselleştirin. CMG etiketliyse, örneğin LD655, CMG'yi 640 nm lazerle (maksimum güç = 30 mW) %10 güçte görselleştirin. SYTOX turuncu lekeli DNA'yı %0,5 güçte 532 nm lazerle görselleştirin. Her lazeri 50-100 ms pozlama ile kullanın.
NOT: Akış hücresinin giriş borusu bir sıvı tüpünden diğerine her aktarıldığında, sıvıdan çıkarmadan önce borunun ucunu tüpün altına birkaç kez vurun. Bu, aktarım sırasında baloncukların içeri girmesini önlemeye yardımcı olur. Giriş borusunun ucunun sıvıdan uzak tutulma süresini en aza indirin ve ucu yukarı doğru tutmayın veya ucu önemli ölçüde kaldırmayın, çünkü bu, sıvının kanala doğru akmasına ve havanın uca çekilmesine neden olur. Giriş borusunu aktarırken, boruyu epoksiden çekerek akış hücresine sabitleyebileceğinden, boruya gerilim uygulamamaya çalışın. DNA gevşetmesini görüntülemek için, çift sarmallı DNA'yı görselleştirmek için SYTOX turuncu varlığında, ATP/RPA karışımı için 10x Reaksiyon tamponu 1'i 10x Reaksiyon tamponu 2 ile değiştirin. SYTOX turuncu bu koşullarda DNA'ya daha iyi bağlanır. Her deneyin sonunda şırınga pompasını tamamen boşaltın. Ara sıra suyla doldurarak ve birkaç kez boşaltarak veya söküp sabunlu suyla yıkayarak yıkayın.

Şekil 4: DNA'nın yüzeye bağlanması. (A) DNA substratlarını her iki uçta biyotin ile bağlarken, iki bağ arasındaki mesafe, uçların yüzeye nasıl temas ettiğine bağlı olarak değişebilir (i). Bir uçta digoksigenin kullanılarak, her bir ucun bağlanması, daha tutarlı bağ mesafeleri ve daha düzgün bir şekilde gerilmiş DNA için zamansal olarak ayrılabilir (ii). (B) Her iki uçtan bağlanmış (digoksigenin etiketli) ve floresan interkalasyonlu nükleik asit boyası ile boyanmış DNA'yı gösteren örnek görüş alanı. Her iki ucundan bağlı olan DNA bir çizgi olarak görünürken, sadece bir uçtan bağlı olan DNA lekeler olarak görünür. İdeal olarak, DNA, diğer DNA'larla örtüşmeden mümkün olduğunca yoğun bir şekilde bağlanmalıdır. Görüntü 512 x 512 pikseldir (piksel boyutu = 154,6 nm). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Temsili Sonuçlar
CMG DNA'yı çözdüğünde, karakteristik bir RPA yolu zamanla büyüyecektir (Şekil 5). Çözülmemiş DNA'nın 5' ucu yüzeye bağlanır; bu nedenle, tether ve çatal arasında doğrusal bir RPA sinyali uzantısı olarak görülür. 3' uç bağlı değildir ve bu nedenle çatalla birlikte hareket eder ve kompakt bir EGFP-RPA sinyali olarak gözlemlenir. Sıkıştırılmış çözülmemiş translokasyon ipliğinin konumu, yaklaşık olarak 640 nm'lik bir lazerle görselleştirilen LD655-CMG ile birlikte hareket eden replikasyon çatalının konumuna karşılık gelir.
DNA substratına verilen hasarı en aza indirmek önemlidir, çünkü tek sarmallı DNA çentikleri gibi hasarlar, gözlemlenebilir çözülme olaylarının sayısını azaltır ve toplanabilecek veri miktarını sınırlar (Şekil 6).
Şekil 5: Tek moleküllü DNA çözme deneyi. DNA substratı bir lamel yüzeyine bağlanır. LD655 ile işaretlenmiş saflaştırılmış CMG, ATP-G-S'de 15 dakika boyunca DNA ile inkübe edilir. ATP ve saflaştırılmış EGFP etiketli RPA eklenir ve CMG ile kapsamlı DNA çözülmesi başlatılır. Bir karikatür şeması (solda) ve temsili verilerin kymografı (sağda) gösterilmektedir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
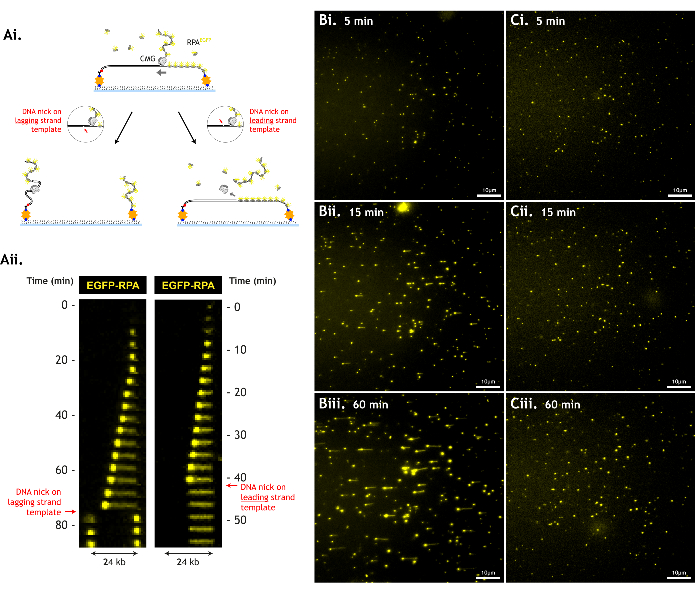
Şekil 6: DNA hasarı, tahlil verimini azaltır. (A) CMG, DNA omurgasındaki bir kırılmayı (DNA çentik) geçerek DNA'yı çözemez. Önde gelen iplik şablonundaki bir çentik, CMG'nin DNA'dan kaymasına neden olur ve hem CMG hem de önde gelen iplik şablonu kaybolur. Gecikmeli iplik şablonundaki bir çentik, gecikmeli iplik şablonunun DNA'nın geri kalanından ayrılmasına neden olur ve her DNA parçası kendi bağlarına geri çekilir. Bu, (i) karikatür şemaları ve (ii) bu olayların kymografları (ii) ile gösterilmiştir. Tek bir görüş alanında (B) minimum hasarlı bir DNA substratına karşı (C) (i) 5 dk, (ii) 15 dk ve (iii) 60 dk'da daha hasarlı bir DNA substratı ile temsili veriler. Daha fazla hasar görmüş DNA substratı, benzer çözme aktivitesi seviyelerine rağmen (5 dakikada büyüyen RPA noktalarının benzer yoğunluğu, benzer CMG yükleme/çözme verimliliğini gösterir) CMG'ler daha erken çentiklerle karşılaştığından, uzun çözülme yolları oluşturmaz. Görüş alanı 512 x 512 pikseldir (piksel boyutu = 154,6 nm). %1 lazer gücü (488 nm) görüntüleme EGFP-RPA. 10 μm'yi gösteren ölçek çubuğu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Bu tahlil, bireysel CMG'lerin gerçek zamanlı dinamiklerini hem ayrı ayrı hem de istenen ek faktörler bağlamında gözlemlemek ve araştırmak için bir platform sağlar. Bununla birlikte, birçok tek moleküllü floresan tekniğinde olduğu gibi, üstesinden gelmek için optimizasyon gerektirebilecek bazı yaygın zorluklar vardır. Bunlar genellikle floroforların uzun süreler boyunca görüntülenmesi (fotoağartma, parlaklık), DNA substrat hazırlığı (DNA hasarı), akış hücresi yüzeyinin kalitesi (arka plan gürültüsü, spesifik olmayan etkileşimler) veya saflaştırılmış protein preparatının kalitesi (nükleaz kontaminasyonu, etiketleme verimliliği) ile ilgilidir.
Her florofor fotostabilite ve parlaklık açısından farklılık gösterir, bu nedenle uygun bir molekül seçmek önemlidir. RPA gibi floresan etiketli oligomerik proteinleri görüntülerken, birçok florofor yakın mesafede uyarılarak görünür bir sinyal üreteceğinden daha düşük bir lazer gücü kullanılabilir. Tek floroforların görüntülenmesi için, örneğin, tek bir alt birim üzerinde etiketlenmiş CMG, floroforu net bir şekilde gözlemlemek için daha yüksek bir lazer gücüne ihtiyaç vardır. Florofor ömrü, lazer maruziyetini en aza indirerek, örneğin görüntülerin çekilme sıklığını azaltarak uzatılabilir. Ek olarak, bir floroforun uyarılması, fotoağartmaya katkıda bulunabilecek reaktif oksijen türleri (ROS) üretir. Görüntüleme tamponuna bir oksijen süpürme sistemi dahil etmek, ROS'u ortadan kaldırarak floroforların ömrünü uzatabilir. Bununla birlikte, bazı oksijen temizleme sistemleri pH12'yi etkileyebilir.
DNA substratının hazırlanması ile ilgili olarak, çentikler veya tek sarmallı boşluklar gibi DNA hasarını en aza indirmek çok önemlidir. Aşırı hasar, DNA'nın kapsamlı bir şekilde çözülmesini önleyerek ne kadar veri toplanabileceğini sınırlar. Hasar, mekanik kesme, nükleaz kontaminasyonunun bir sonucu olarak aşırı ısınma veya görüntüleme sırasında oluşan ROS'tan kaynaklanabilir. DNA örneğini dikkatli bir şekilde kullanarak, pipetleme için geniş delikli uçlar kullanarak, yavaş pipetleyerek ve numuneyi hafifçe vurmaktan kaçınarak kesme en aza indirilebilir. ROS'un etkisi, lazer maruziyetini azaltarak veya görüntüleme tamponuna bir oksijen süpürme sistemi dahil ederek en aza indirilebilir. DNA substratının hazırlanmasından sonra, bir çözme reaksiyonu gerçekleştirmeden önce hasarı onarmak için ticari DNA onarım kitlerini kullanmak mümkündür.
DNA çözmenin verimliliği ayrıca CMG'nin saflığına ve aktivitesine de bağlıdır. Optimizasyonun gerekli olduğu yerleri belirlemek için her saflaştırma adımından sonra SDS-PAGE elektroforezi ile numune saflığını değerlendirmek iyi bir uygulamadır. Son adımdan sonra çok fazla kirletici gözlenirse, CaptoHiRes Q (5/50) sütunundan elüsyon için kullanılan tuz gradyan hacimlerinin değiştirilmesine yardımcı olabilir. Lamel yüzeyinde istenmeyen bir arka plan oluşturabileceğinden, protein etiketlemesi için kullanılan fazla floresan peptidin çıkarılması da son derece önemlidir. DNA substratını bozabileceğinden, nükleaz kontaminasyonundan kaçınmak da önemlidir. Bir deneyden sonra, kalan DNA'yı SYTOX turuncu ile boyamak, DNA'nın önemli ölçüde bozulup bozulmadığını kontrol etmenin iyi bir yolu olabilir. Bir deney boyunca belirli bir düzeyde DNA hasarı kaçınılmazdır, ancak önemli hasar genellikle sorunlu nükleaz kontaminasyonunu gösterir.
Test aynı zamanda doğal olarak, kırınım sınırlı noktaların çözünürlüğü ile sınırlıdır ve floresan proteinlerin ayrı olarak ayırt edilmeleri için yüzlerce baz çifti uzakta (daha fazla değilse) olmasını gerektirir. Bu, CMG ilerlemesinin ve etkileşimlerinin gözlemlenebileceği ayrıntıyı sınırlar.
Her analiz için gözlemlediğimiz çözülme olaylarının sayısı değişir. Başarılı bir deney için, 512x512 piksel (piksel boyutu = 154,6 nm) görüş alanı başına yeterli uzunlukta en az birkaç RPA yolu görmeyi bekliyoruz. Aynı deneyde birden fazla görüş alanı görüntülenebilir ve gerektiğinde daha fazla veri toplanmasına olanak tanır. Yolların yararlı olması için aynı uzunlukta olması veya DNA'nın sonuna ulaşması gerekmez. Örneğin, her deney için ortalama bağ mesafesi, CMG eklenmeden önce SYTOX ile boyanmış DNA'nın uzunluğu ölçülerek belirlenebilir. Bu, mesafeyi 'μm kat edilen'den 'kb çözülmemiş'e dönüştürerek herhangi bir RPA yolu için (çatalı gözle görülür şekilde hareket ettirmek için yeterli DNA çözüldüğü sürece) ne kadar DNA'nın çözüldüğünü tahmin etmek için kullanılabilir.
CMG, çeşitli DNA substratları üzerinde çözme aktivitesi sergiler, ancak CMG10'un ayak izini barındırmak için en az 30 nt'lik bir poliT flebi üzerinde serbest bir 3' DNA ucu sağlamak önemlidir. Çatalda birden fazla biyotin parçasının bulunması, sağlam yüzey bağlaması sağlar. DNA substratının geri kalanı, farklı DNA dizileri, uzunlukları ve kimyasal modifikasyonları içerecek şekilde çok çeşitli şekillerde yeniden tasarlanabilir. DNA'nın konformasyonu, farklı konsantrasyonlarda magnezyum asetat kullanılarak değiştirilebilir. Daha yüksek konsantrasyonlarda (≥10 mM) magnezyum asetat için, RPA kaplı ssDNA filamenti sıkıştırılır ve bu da çözülme sırasında RPA bağlanması ile öğretilen DNA'nın çekilmesine yol açar. Bu, DNA'nın aşırı hareket etmesini önlediği, CMG'nin pozisyonunun ve çözülme ilerlemesinin daha doğru bir şekilde ölçülmesine izin verdiği için yararlı olabilir. Düşük konsantrasyonlarda (~ 3 mM) magnezyum asetat , RPA-ssDNA baştan sona rahat kalır.
Açıklanan tek moleküllü tahlil, DNA replikasyonunun daha ileri yönlerini araştırmak için üzerine inşa edilebilen ve değiştirilebilen bir platformu temsil eder. DNA replikasyonu sırasında CMG, replisome ve bileşenlerinin etrafında toplandığı bir çekirdek görevi görür. Bu nedenle, CMG dinamikleri üzerindeki etkilerini incelemek için TIMELESS, TIPIN ve CLASPIN gibi aksesuar faktörler de dahil olmak üzere bu teste ek saflaştırılmış proteinler eklenebilir. Bu proteinlerin replikasyon çatallarının13 hızını etkilediği gösterilmiştir, ancak CMG çözülme hızını nasıl etkiledikleri açık değildir. Bu nedenle, bu testi kullanarak farklı replizom proteinlerinin CMG'yi nasıl etkilediğini araştırmak ilginç olacaktır. DNA polimerazların eklenmesi, daha önce maya proteinleri14 ile tarif edildiği gibi, tek başına DNA çözülmesinin ötesinde DNA replikasyonu hakkında daha iyi bir fikir verebilir. Ayrıca, modifiye edilmiş CMG'nin saflaştırılması, belirli mutasyonların veya translasyon sonrası modifikasyonların helikaz aktivitesini nasıl etkilediğinin daha iyi anlaşılmasını sağlayabilir15,16. Ek olarak, farklı DNA substratlarının tasarlanması, replikasyon stresini taklit eden çeşitli koşullar altında CMG ile DNA çözülmesininincelenmesine izin verebilir 17. Bu modifikasyonlar, DNA engellerini 9,18, zincirler arası çapraz bağları 19,20,2 1 ve DNA ipliklerindeki22 süreksizlikleri içerir.
Açıklamalar
Yazarların rekabet eden finansal çıkarları veya diğer çıkar çatışmaları yoktur.
Teşekkürler
pGC261 plazmidini sağladığı için Gheorghe Chistol'e ve peptit sentezi ve etiketlemesi için Francis Crick Enstitüsü Kimyasal Biyoloji Tesisi'ne teşekkür ederiz. Bu çalışma, Cancer Research UK, UK Medical Research Council ve The Wellcome Trust'tan (CC2133) çekirdek fon alan Francis Crick Enstitüsü tarafından finanse edilmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
Referanslar
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819 (2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159 (2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713 (2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331 (2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır