Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
SV40 Tabanlı İnsan Epizomu İçinde Yapıya Eğilimli DNA Tekrarları Yoluyla Replikasyonun Elektroforetik Analizi
Bu Makalede
Özet
Burada, 2 boyutlu jel elektroforezi kullanarak patojenik, yapıya eğilimli tekrarlar yoluyla replikasyon ilerlemesini analiz etme prosedürünü özetliyoruz.
Özet
İki boyutlu nötr/nötr jel elektroforezi (2DGE), doğal engeller yoluyla DNA replikasyonunu analiz etmek için bir kıyaslama tekniği olarak ortaya çıktı. Bu protokol, insan hücrelerinde simian virüsü 40 (SV40) tabanlı epizom içinde yapıya eğilimli, genişletilebilir DNA tekrarları yoluyla replikasyon çatalı ilerlemesinin nasıl analiz edileceğini açıklar. Kısaca, insan hücrelerine plazmit transfeksiyonu üzerine, replikasyon ara ürünleri, modifiye edilmiş Hirt protokolü ile izole edilir ve replike edilmemiş DNA'yı çıkarmak için DpnI kısıtlama enzimi ile muamele edilir. Ara ürünler daha sonra uygun kısıtlama enzimleri tarafından sindirilir ve ilgilenilen tekrarı 3-5 kb uzunluğundaki bir DNA fragmanının orijin-distal yarısına yerleştirir. Replikasyon ara ürünleri, önce boyuta ve sonra şekle göre iki dikey boyuta ayrılır. Southern blot hibridizasyonunu takiben, bu yaklaşım araştırmacıların, replikasyon Y-yayının azalan yarısında çeşitli yapı oluşturan tekrarlarda çatal durmasını gözlemlemelerine olanak tanır. Ayrıca, durak alanının bu şekilde konumlandırılması, çatalın tersine çevrilmesi, yakınsak bir çatalın ortaya çıkması ve yeniden kombinasyonel çatalın yeniden başlatılması gibi tekrar aracılı çatal durdurmanın çeşitli sonuçlarının görselleştirilmesine olanak tanır.
Giriş
Kısa tandem tekrarları (STR) küçük, tipik olarak 2-9 baz çifti (bp), insan genomunun yaklaşık %3'ünü oluşturan tekrarlayan DNA dizileridir1. STR, gen regülasyonunda önemli bir rol oynar2; bununla birlikte, tekrarlayan bileşimleri, onları kanonik olmayan DNA ikincil yapı oluşumuna ve ardından genetik kararsızlığa eğilimli hale getirir 3,4. Solak sarmallardan saç tokalarına/haç formlarına, üç ve dört sarmallı sarmallara kadar, bu alternatif DNA yapıları, replizom için içsel zorluklara neden olur. İkincil yapı oluşumu için doğal bir ön koşul, DNA replikasyonu için bir ön koşul olan DNA'nın çözülmesidir. Bu, genomun işleyişi için benzersiz bir bilmece sunar, çünkü bu yapıların çoğu replikasyon sırasında oluşabilir, replisome ilerlemesini engeller ve nihayetinde replikasyon çatalının durmasına 5,6,7 veya ciddi vakalarda çatal çökmesine ve DNA kırılmasına 8,9 neden olabilir. Hem durdurulan çatalların yeniden başlatılmasının hem de DNA onarım yollarının, tekrarlanan genişlemeler10,11 ve karmaşık genom yeniden düzenlemeleri (CGR)12,13 gibi tekrarlanan kararsızlığa yol açtığı gösterilmiştir. Bu olaylar, Frajil X sendromu, Huntington hastalığı, Friedreich ataksisi ve diğerleri14,15 ve Emmanuel sendromu16 gibi CGR hastalıkları dahil olmak üzere tekrar genişleme bozuklukları olarak bilinen yaklaşık 60 insan hastalığının gelişmesine neden olabilir. Bu nedenle, tekrarlanan kararsızlıktan kaynaklanan insan hastalığının mekanizmalarını daha iyi anlamak için, bu tekrarlar yoluyla replikasyon çatalı ilerlemesinin ayrıntılarını incelemek zorunludur.
1980'lerin ortalarında, Brewer ve Fangman'ın Saccharomyces cerevisiae'de replikasyon başlangıcının otonom replikasyon dizisinde (genellikle ARS olarak bilinir) meydana geldiğine dair doğrudan kanıt sağlamaya çalıştıkları17 replikasyon ilerlemesini incelemek için bir teknik ortaya çıktı. Bunu yaparken, 2 boyutlu Nötr / Nötr jel elektroforezi (2DGE) 18 olarak bilinen Bell ve Byers'tan daha önceki bir yöntemi uyarlayarak, agarozdaki maya replikasyon ara ürünlerinin yapılarını ayırdılar. Bu teknik, doğrusal olmayan DNA'nın agaroz jel içinde aynı kütlenin doğrusal eşdeğerinden farklı şekilde hareket ettiği gerçeğini kullandı. Daha spesifik olarak, 2DGE'de, izole edilmiş DNA, belirli bir ilgi bölgesinde kapsamlı bir replikasyon haritası oluşturmak için önce öncelikle boyuta ve daha sonra öncelikle şekle göre iki dikey boyutta ayrılır. Orijinal makalelerinde, Brewer ve Fangman bunu, kopyalanmamış DNA'yı kopyalanmış muadillerine bağlayan "basit Y" yapılarından veya replikasyon çatallarından oluşan bir yay olarak gösterdiler. Ayrıca, gözlemlenen diğer ara ürünleri sırasıyla replikasyon kökenlerini ve yakınsak çatalları temsil eden "kabarcıklar" ve "çift Y'ler" olarak tanımlarlar.
2DGE, belirli bir zamanda DNA replikasyon ara ürünlerinin nispi popülasyonlarını incelemek için kullanılabilir. Bu nedenle, bir ara ürün popülasyonu diğerinden daha yaygınsa, bu görselleştirme üzerine belirgin olacaktır. Bu, 2DGE'yi yapı oluşturan tekrarlar gibi zorlu diziler aracılığıyla replikasyon ilerlemesini incelemek için özellikle kullanışlı bir araç haline getirir. Örneğin, analiz edilen bölge, replikasyon çatalının durmasını indükleyebilen bir dizi içeriyorsa, bu, yay üzerinde bir çıkıntı olarak ortaya çıkacaktır (Şekil 1A), bu da o lokusta bir replikasyon çatalı birikimini gösterir. Bu, maya 19,20,21'de hem saç tokası oluşturan tekrar dizilerinin hem de insan hücrelerinde (22,23,24) tripleks oluşturan tekrarların replikasyonu ile görülebilir. Durdurmaya ek olarak, 2DGE, rekombinant ara ürünler25 durumunda olduğu gibi, replikasyon sırasında oluşan standart basit Y'lere uymayan DNA yapılarını gözlemlemek için kullanılabilir. Bu ara ürünler daha ağır ve daha dallı X şeklinde bir yapıya sahiptir ve bu nedenle hem birinci hem de ikinci boyutlarda standart çoğaltma çatallarından daha yavaş hareket eder. Replikasyon çatalının tersine çevrilmesi20,24,26 ile ilgili olarak da benzer sonuçlar gözlemlenebilir. Güçlü replikasyon stresine yanıt olarak, ökaryotik hücrelerin durmuş çatalları kurtarmak için replikasyon çatalının tersine çevrilmesini kullandığı gösterilmiştir. Bu ters çatallar, durmuş çatallara benzer moleküler ağırlığa sahiptir; yine de tavuk ayağı yapıları, Y şeklindeki tamamlayıcılarına göre ikinci boyutta daha yavaş elektroforetik hareketliliğe neden olur ve bu da yaydan yukarı ve dışarı bir uzantı ile sonuçlanır.
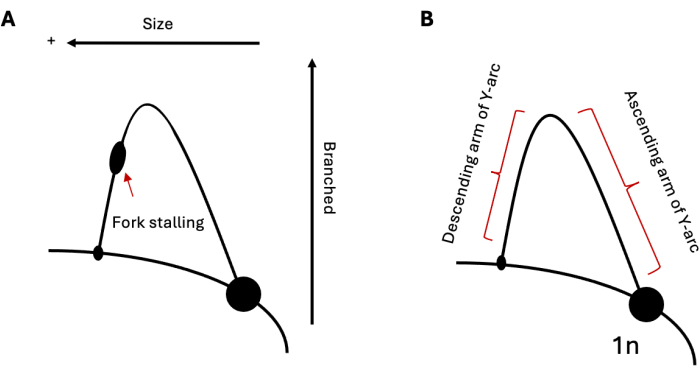
Şekil 1: DNA replikasyonunun 2D Jel elektroforez analizi. (A) Çatal durmasını indükleyebilen yapı oluşturan bir tekrar yoluyla replikasyonu gösteren tipik bir 2DGE'nin şeması. Ara boyut ve yapı, elektroforetik hareketliliği etkileyecektir. (B) Sırasıyla artan ve alçalan kolları etiketlenmiş Y arkı örneğin. Kısaltma: 2DGE = İki boyutlu nötr/nötr jel elektroforezi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Doğal olarak, 2DGE'nin en önemli yönlerinden biri, replikasyon ara ürünlerinin kalitesi ve miktarı ile ilgilidir. Bununla birlikte, memeli hücrelerinde endojen lokuslar yoluyla replikasyonun 2DGE analizinin çözünürlüğü, 6 × 109 bp diploid insan genomu içindeki tek bir kopya hedef dizisi için yetersizdir, ancak bu, ağır şekilde çoğaltılmış DHFR lokus27 veya ribozomal RNA28 gibi çok kopyalı genler için yapılmıştır. SV40 tabanlı replikasyon, ökaryotik hücrelerde replikasyonu incelemek için verimli ve iyi karakterize edilmiş bir araçtır29. Enfeksiyon üzerine nükleozomlara parsellenen viral genomu çoğaltmak için konakçı replisome mekanizmasının çoğunu kullanan güvenilir bir ökaryotik replikasyon modeli sağlar30,31. Memeli replisome'un iki dikkate değer istisnası, konakçı CMG kompleksi yerine T-antijeninin (Tag) replikatif DNA sarmalı olarak hizmet etmesi ve DNA polimeraz deltasının hem önde gelen hem de gecikmeli DNA zincirlerinisentezlemesidir 32. Bu sistemden, orijinal olarak Massimo Lopes laboratuvarında22 oluşturulan bir plazmit içinde bir SV40 replikasyon kaynağından aşağı akışta patojenik yapı oluşturan tekrarlar yerleştirerek yararlandık. Daha da önemlisi, bu plazmit aynı zamanda Tag'ın kendisini kodlayan geni de içerir, böylece çeşitli kültürlenmiş insan hücrelerine transfeksiyon üzerine yapısal ve son derece güçlü replikasyonu ile sonuçlanır. Bu özellik, insan hücrelerinde patojenik tekrarların replikasyonu sırasında ve buna yanıt olarak oluşan ara ürünlerin 2DGE analizi için ideal olan büyük miktarda ürüne yol açar. Burada, 2 boyutlu jel elektroforezi kullanarak SV40 tabanlı insan epizomu içinde yapı oluşturan tekrarların replikasyonunu görselleştirmek için ayrıntılı bir yöntemi açıklıyoruz.
Protokol
NOT: Memeli hücrelerinde ana hatlarıyla belirtilen 2DGE analizimiz için tasarlanan plazmit, yapıya eğilimli tekrarların birkaç kb yukarısında bir SV40 replikasyon kaynağı içermelidir (Şekil 2). Tekrarların plazmide klonlanması gereken orijine göre hangi oryantasyonu seçerken, önde gelen ve gecikmeli sentez akılda tutulmalıdır.

Şekil 2: 2DGE analizi için tekrar içeren plazmitin sindirimi. Yapıya eğilimli yinelemeler, sağa hareket eden çoğaltma çatalından birkaç kb aşağı akışta gösterilir. Benzersiz kesiciler 1 ve 2 ile sindirim, sindirilen parçanın yarı noktasının ötesindeki dizi göz önüne alındığında, tekrar dizisini Y-yayının alçalan koluna yerleştirecektir. Kısaltma: 2DGE = İki boyutlu nötr/nötr jel elektroforezi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
1. Memeli hücrelerine plazmid transfeksiyonu
- Transfeksiyondan önce 600.000 HEK293T hücreyi 10 cm'lik bir doku kültürü plakasına tohumlayın. Hücrelerin gece boyunca 37 °C'de iyileşmesine izin verin.
NOT: Bu deney için birçok hücre hattı kullanılabilir, ancak en iyi sonuçlar için SV40 Etiketini içeren hücreler önerilir. DİKKAT: HEK293T hücreleri BSL-2 olarak kabul edilir ve tüm kültür çalışmaları uygun aseptik teknik ve uygun KKD kullanılarak bir biyogüvenlik kabininde yapılmalıdır. - Hücreler %60 birleşmeye ulaştığında, üreticinin protokolüne uygun transfeksiyon reaktifleri kullanarak 8 μg tekrar içeren plazmit DNA'yı tohumlanmış hücrelere aktarın.
- Bu zaman noktasında ara ürünleri izole etmiyorsanız, eski ortamı aspire edin ve 24 saat sonra 10 mL taze ortamla değiştirin.
- Transfeksiyondan 24-48 saat sonra hücreleri toplamaya başlayın.
- Ortamı aspire edin ve 10 mL fosfat tamponlu salin (PBS) ile dikkatlice yıkayın. 0.5 mL tripsin kullanarak hücreleri ayırın ve toplayın ve 4 dakika boyunca 340 × g'da döndürün.
- Süpernatanı aspire edin ve hücre peletlerini PBS ile yıkayın. 340 × g'da 4 dakika boyunca tekrar döndürün ve süpernatanı aspire edin.
NOT: Deney burada hücre peletlerinin -80 °C'de dondurulmasıyla duraklatılabilir. Transfeksiyondan 48 saat sonra en iyi çözünürlüğü izole eden replikasyon ara ürünlerini bulduk; Bununla birlikte, 24 saat uygulanabilir sonuçlar vermiştir.
2. Replikasyon ara ürünlerinin izolasyonu
- Hücreleri 50 mL konik tüplerde 1.5 mL modifiye Hirt lizis tamponu [10 mM tris-HCl (pH 7.5), 10 mM etilendiamintetraasetik asit (EDTA)] içinde yeniden süspanse edin ve hücre lizizine başlayın.
- Nükleazları uzaklaştırmak için% 0.6'lık bir nihai konsantrasyona (yaklaşık 650 μL stok% 2 SDS) sodyum dodesil sülfat (SDS) ve 100 μg / mL'lik bir nihai konsantrasyona (yaklaşık 10 μL stok, 20 mg / mL proteinaz K) proteinaz K ekleyin.
- Homojen olana kadar pipetleyerek nazikçe karıştırın ve karışımı 37 °C'de en az 90 dakika inkübe edin.
- NaCl konsantrasyonunu 1 M'ye (yaklaşık 540 μL stok, 5 M NaCl) yükseltin ve homojen olana kadar hafifçe karıştırın. Hücre kalıntılarının, RNA'nın ve proteinin tuzlanarak çökelmesine izin vermek için gece boyunca (18-24 saat) 4 ° C'de inkübe edin.
NOT: Karışım oldukça viskoz olacaktır, bu nedenle iyi karıştırırken dikkatli olun ve sabırlı olun. - Ertesi gün, DNA'yı hücre kalıntısı, RNA ve proteinden ayırın.
- Karışımı 29.500 × g'da 4 °C'de 45 dakika santrifüjleyin.
- DNA içeren süpernatanı aktarın, bir hacim fenol:kloroform:izoamil alkol 25:24:1 (h/v) ekleyin ve homojen olana kadar kısaca karıştırın.
DİKKAT: Fenol: kloroform: izoamil alkol tehlikeli bir maddedir ve kimyasal davlumbazda uygun KKD ile kullanılmalıdır. - Oda sıcaklığında 5 dakika boyunca 15.000 × g'da tekrar santrifüjleyin. Sulu tabakayı yeni bir konik tüpe aktarın.
- İzole edilmiş DNA'yı çökeltin ve yıkayın.
- Bir hacim saf izopropanol ekleyin ve oda sıcaklığında en az 5 dakika inkübe edin. DNA'yı 15.000 × g'da 4 ° C'de 30 dakika boyunca döndürün.
- Süpernatanı boşaltın ve fazla tuzu çıkarmak için peleti soğuk %70 etanol ile yıkayın.
- 4 ° C'de 30 dakika boyunca 15.000 × g'da bir kez daha döndürün, havayla kurutun ve peleti Tris-EDTA (TE) tamponunda (10 mM Tris-HCl, 1 mM EDTA) nazikçe yeniden süspanse edin.
NOT: Deney burada duraklatılabilir ve numuneler -20 °C'de dondurulabilir; bununla birlikte, DNA replikasyon ara ürünlerinin kalitesini azaltabileceğinden, donma/çözülme döngüsünden kaçınılmalıdır.
3. Numune hazırlama ve 2 boyutlu jel elektroforezi
- Plazmit replikasyonunun izole edilmiş ara ürünlerini sindirin.
- Plazmid DNA'yı sindirmek için numuneye 100 birim uygun kısıtlama enzimi ekleyin, spesifik olarak tekrar içeren diziyi doğrusal parçanın orijin-distal yarısına yerleştirin (Şekil 2). Ek olarak, metillenmiş DNA'yı kesmek için DpnI ekleyin, böylece kültürlenmiş insan hücrelerinde tam olarak kopyalanmamış herhangi bir plazmit DNA'yı çıkarın.
NOT: En iyi sonuçlar için, kısıtlama enzimleri, yapıya eğilimli diziyi Y-yayının inen koluna yerleştiren 3-5 kb'lık bir parça veren benzersiz kesiciler olmalıdır. - Tam plazmit sindirimine izin vermek için numuneleri 37 ° C'de 6-10 saat inkübe edin.
- DNA'yı 2.5 hacim soğuk saf etanol ile çökeltin ve gece boyunca -20 ° C'de inkübe edin veya bir hacim izopropanol ekleyin ve oda sıcaklığında 5 dakika inkübe edin.
- Sindirilmiş ve çökeltilmiş numuneleri 15.000 × g'da 4 °C'de 30 dakika santrifüjleyin.
- Süpernatanı boşaltın ve numuneyi soğuk% 70 etanol ile yıkayın. 15.000 × g'da 4 °C'de 30 dakika boyunca tekrar döndürün.
- Süpernatanı boşaltın, 10 dakika havayla kurutun ve numuneleri 15 μL TE tamponunda yeniden süspanse edin.
- Plazmid DNA'yı sindirmek için numuneye 100 birim uygun kısıtlama enzimi ekleyin, spesifik olarak tekrar içeren diziyi doğrusal parçanın orijin-distal yarısına yerleştirin (Şekil 2). Ek olarak, metillenmiş DNA'yı kesmek için DpnI ekleyin, böylece kültürlenmiş insan hücrelerinde tam olarak kopyalanmamış herhangi bir plazmit DNA'yı çıkarın.
- Birinci boyut agaroz jelini 1x Tris-borat-EDTA (TBE) (89 mM tris bazı, 89 mM borik asit, 2 mM EDTA) içinde% 0.4-0.5'te hazırlayın. Çözeltinin en az 1 saat katılaşmasına izin verin.
- Numuneleri ilk boyuta yüklemeye başlayın.
- Merdiveni jelin en sol kenarına göre ilk 3 cm'ye yerleştirin. Ardından, hazırlanan numunelerin tamamını, her çift arasında 3 cm olacak şekilde yükleyin.
- Ara ürünleri boyutlarına göre ayırmak için jeli 19-24 saat boyunca 0,85 V / cm'de 1x TBE'de çalıştırın. DNA'ya zarar verebilecek numuneleri ışıktan korumak için haznenin kapalı olduğundan emin olun.
- Ertesi gün, jeli tampondan çıkarın ve bir cetvel kullanarak sindirilmiş doğrusal parçanın yerini tahmin edin.
- Merdiveni içeren jelin ilk 3 cm'sini eksize edin ve jel segmentini 10-15 dakika boyunca 0.3 μg / mL etidyum bromür içeren 1x TBE'de boyayın. Bir jel dokümantasyon sistemi kullanarak merdiveni görselleştirin.
- Tahmini konuma 1,3 cm ekleyin ve a değerini elde edin. Ardından, a değerinden 7,5 cm çıkarın ve b değerini elde edin. Cetveli birinci boyut jeli ile hizalayın ve a ve b değerlerinde yatay olarak kesin. Ardından, her numune için ayrılan 3 cm'lik boşluğu dikey olarak kesin. Görsel bir şema için Şekil 3'e bakın.
- Yeni bir döküm tepsisinde, segmentleri saat yönünde çevirin ve numune kuyucuklarının konumuna yerleştirin (Şekil 3).
- İkinci boyut agaroz jeli, 0.3 μg / mL etidyum bromürde 1x TBE'de% 1-1.3'lük bir konsantrasyonda hazırlayın.
- Yaklaşık 55 °C'ye soğuduktan sonra, ikinci boyut jelini döndürülmüş birinci boyut segmentlerinin üzerine dökün ve en az 1 saat katılaşmasına izin verin.
- İkinci boyutu 0.3 μg / mL etidyum bromürde 1x TBE içeren bir odaya aktarın ve jelin en az 30 dakika dengelenmesine izin verin.
- Ara ürünleri şekillerine göre ayırmak için jeli tekrar üstü kapalı olarak 9.23 ° C'de 4.23 V / cm'de 4-4 saat çalıştırın.
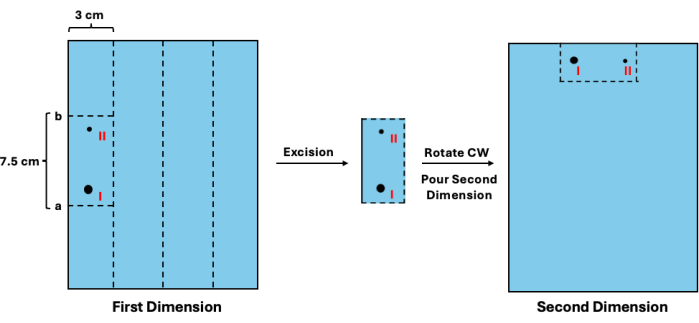
Şekil 3: İkinci boyut ayrımından önce birinci boyut ara ürünlerinin eksizyonu. Merdivenin görselleştirilmesini takiben, kopyalanmamış parçaların hareketliliği tahmin edilebilir. (I) Bu değer daha sonra onu ve çoğaltılmış muadillerini (II) çıkarmak için uygun kesim yerlerini (a ve b) belirlemek için kullanılabilir. Jelin bölümü daha sonra döndürülmeli ve ikinci boyut ayrımı için kuyucukların konumuna yerleştirilmelidir. Kısaltma: CW = saat yönünde. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
4. Radyoaktif işaretli prob ile güney lekeleme ve hibridizasyon
- İkinci boyutlu jeli hazneden çıkarın ve DNA parçalarını hafifçe sallayarak 0.24 M HCl çözeltisinde 10 dakika boyunca depurine edin. Jeli deiyonize su ile durulayın ve 10-15 dakika boyunca 0.4 M NaOH içinde bekletin.
DİKKAT: HCl ve NaOH aşındırıcıdır ve kimyasal davlumbazda uygun KKD ile kullanılmalıdır. - Ayrılan ara ürünlerin jelden zara transferini kolaylaştırmak için Güney lekesini monte etmeye başlayın. Kapsamlı bir şema için Şekil 4'e bakın.
- Yeterli büyüklükte bir kabı 1 L 0,4 M NaOH ile doldurun.
- Uzun bir cam tabakayı kabın üzerine hizalayın ve NaOH kabına uzanan cam levha boyunca dik olarak iki uzun kromatografi kağıdı yaprağını katlayın (uzunluk boyunca).
- Kağıdın üst kısmını NaOH ile ıslatın ve yüzeyinin altındaki hava kabarcıklarını dikkatlice çıkarın.
- Üç yaprak kromatografi kağıdını NaOH ile ıslatın ve katlanmış kağıdın üzerine yerleştirin, tekrar kabarcıkları giderin.
- İkinci boyutlu jeli ters çevirin ve kağıtların üzerine aktarın.
- Pozitif yüklü bir naylon membranı (0,45 μm gözenek boyutu) DI su ile ıslatın ve jelin üzerine yerleştirin.
- Son olarak, zarın üzerine DI suyla ıslatılmış üç yaprak daha kağıt ekleyin.
- Buharlaşmayı önlemek için alt kapta açıkta kalan NaOH'yi plastik sargı ile örtün. Lekenin üzerine bir yığın peçete veya kağıt havlu yerleştirin ve 0,3-0,5 m boyunda olduğundan emin olun. Sıkı kılcal hareketi kolaylaştırmak için tüm lekeyi sıkıştırarak üstüne bir ağırlık koyun. DNA'nın zara geçmesi için en az 2 gün bekleyin.
NOT: Kromatografi kağıdının ve zarının uzunluğu ve genişliği, ikinci boyut için kullanılan agaroz jelin boyutuna bağlıdır. En verimli aktarım için, jelinkiyle aynı boyutlara sahip kağıt ve membran kullanın.
- Transferin ardından, 1 dakika boyunca 120 μJ /cm2'de bir UV çapraz bağlayıcı kullanarak DNA'yı zara çapraz bağlayın.
NOT: Membran oda sıcaklığında hava geçirmez, kuru ve temiz bir tabaka koruyucuya yerleştirilerek deney burada duraklatılabilir. - Membranı 2x 5 dakika boyunca 2x salin sodyum sitrat (SSC tamponu) (0.3 M NaCl, 0.03 M sodyum sitrat) ile yıkayın.
- Membranı 0.18 mL /cm2 Church & Gilbert'in hibridizasyon tamponu [1 mM EDTA,% 1 sığır serum albümini (BSA), 0.5 M sodyum fosfat,% 7 SDS] ile 65 ° C'de önceden hibridize edin, en az 2 saat boyunca bir hibridizasyon inkübatöründe döndürün.
NOT: Membran birkaç gün önceden hibridize olabilir. - Radyoaktif işaretli probu, üreticinin protokolüne uygun olarak α-32P, dATP veya dCTP ve bir DNA etiketleme kiti kullanarak hazırlayın.
DİKKAT: Radyo etiketli dNTP'ler tehlikelidir ve bunları kullanırken uygun KKD giyilmelidir. Tüm radyoaktif çalışmalar ekranlamanın arkasında yapılmalı ve eğitimli kişiler dozimetreler kullanılarak radyasyon alımı açısından izlenmelidir.- Kısıtlama sindirilmiş diziye tamamlayıcı 400-900 bp'lik bir doğrusal DNA fragmanı tasarlayın (Şekil 2) ve polimeraz zincir reaksiyonu (PCR) kullanarak fragmanı çoğaltın.
NOT: 50-100 ng/μL'lik bir stok PCR fragmanına sahip olmanızı öneririz - 100 ng tamamlayıcı PCR fragmanını DNA Pol I, Klenow fragmanı (3' 5' ekzo-) tamponu ve rastgele dekannükleotid oligoları ile birleştirin.
- Parçayı 100 °C'de 10 dakika denatüre edin.
- Numuneye 5 ünite DNA Pol I, Klenow fragmanı (3' 5' ekzo-), 50 μCi α-32P dNTP ve radyoaktif işaretli dNTP tipinde eksik olan 30-50 μmol dNTP karışımı ekleyin.
- Polimerizasyona ve radyoaktif işaretli dNTP katılımına izin vermek için 37 ° C'de 10 dakika inkübe edin.
- Numuneye 30-50 μmol daha önce bulunmayan dNTP tipi ekleyin ve 37 ° C'de 10 dakika inkübe edin.
- Radyoaktif işaretli parçayı 2 dakika boyunca 3.000 × g'da bir döndürme sütunu kullanarak saflaştırın.
- Kısıtlama sindirilmiş diziye tamamlayıcı 400-900 bp'lik bir doğrusal DNA fragmanı tasarlayın (Şekil 2) ve polimeraz zincir reaksiyonu (PCR) kullanarak fragmanı çoğaltın.
- 50 mL hibridizasyon tamponuna radyoaktif işaretli prob ekleyin ve bir hibridizasyon inkübatöründe dönerek 65 ° C'de gece boyunca membran ile inkübe edin.
- Ertesi gün, probu çıkarın ve membranı 2x yıkama tamponu 1 (0.1x SSC, %0.1 SDS) ile 42 °C'de ve 2x yıkama tamponu 2 (2x SSC, %0.1 SDS) ile 65 °C'de yıkayın.
NOT: Tüm yıkamalar kuvözde 15 dakika boyunca hızlı dönüşlerle yapılmalıdır. - Membranı 10 dakika kurutun ve ince, şeffaf bir tabaka koruyucuya yerleştirin. Sızdırmaz membranı, fosfora duyarlı bir ekrana sahip kontrollü, radyasyona dayanıklı bir kasette saklayın. Membranın 1-10 gün ekrana maruz kalmasına izin verin.
- Fosfor görüntülemeye ayarlanmış bir biyomoleküler görüntüleyici kullanarak sonuçları görselleştirin. Yıkayın ve gerekirse tekrar pozlayın.
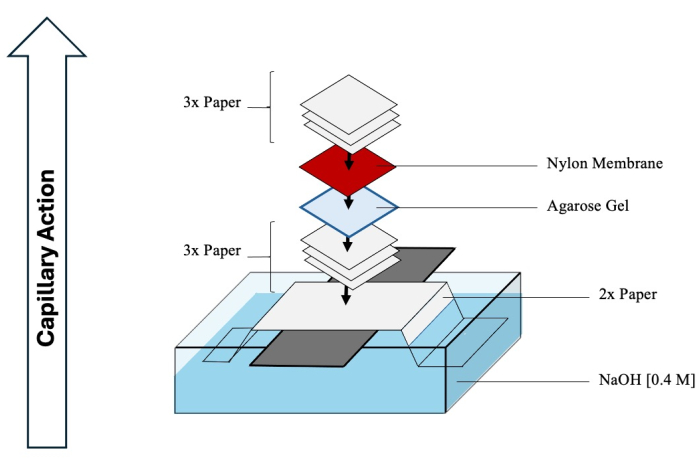
Şekil 4: Güney böceğinin montajı. Southern blot ara ürünlerinin ikinci boyuttan bir naylon membran üzerine transferi için kullanılan tipik bir aparatın kapsamlı şeması. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Sonuçlar
Başarılı olursa, görselleştirme üzerine, büyük 1n noktasından yukarı ve dışarı uzanan keskin bir replikasyon çatalı yayı gözlemlenebilir (Şekil 5A). Bir parçanın boyutu veya çoğaltılan yüzde, parçanın birinci boyuttaki hareketliliğini belirler. Ara ürünler daha eklemli bir yapı geliştirdikçe, ikinci boyutta daha yavaş hareket etmeye başlayacaklardır. Bu nedenle, bir ara ürün her iki boyutta da yavaş hareket etmişse, gel...
Tartışmalar
2DGE, belirli bir dizinin replikasyonu sırasında ortaya çıkan ara ürün nispi popülasyonlarının yarı kantitatif ve kapsamlı bir görüntüsünü sağlar. Replikasyon çatallarının kırılgan moleküler yapılarının bu prosedür boyunca korunması gerektiği göz önüne alındığında, fiziksel kesme ve kimyasal denatürasyonu önlemek için büyük özen gösterilmelidir. Bu nedenle, plazmit izolasyonu sırasında herhangi bir alkali tedaviden kaçınılması şiddetle ta...
Açıklamalar
Yazarların açıklanacak herhangi bir çıkar çatışması yoktur.
Teşekkürler
Laboratuvarımızda bu yaklaşımı geliştirmeye başlayan Jorge Cebrian ve Anastasia Rastokina'ya, bize pML113 plazmidi ve paha biçilmez tavsiyeler sağladığı için Massimo Lopes'e, anlayışlı tartışmalar için Ylli Doksani'ye ve destekleri için Mirkin laboratuvarı üyelerine teşekkür ederiz. Mirkin laboratuvarındaki çalışmalar Ulusal Genel Tıp Bilimleri Enstitüsü [R35GM130322] ve NSF-BSF [2153071] tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
Referanslar
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -. Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -. M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863 (2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23 (2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L., Adam, M. P. Emanuel Syndrome. GeneReviews®. , (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M., Kohwi, Y. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. , (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994 (2012).
- . BLAST: Basic Local Alignment Search Tool Available from: https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024)
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır