Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Sarko-Endoplazmik Retikulum Ca2+-ATPazın Tavşan Kasından Saflaştırılması
Bu Makalede
Özet
Bu protokol, son santrifüjleme adımında disakkarit trehalozu içeren geliştirilmiş bir SERCA saflaştırma yöntemini tanımlar. Bu karbonhidrat, zorlu koşullar altında proteinleri stabilize eder. Saflaştırılmış SERCA, katalitik olarak aktifti ve yüksek saflık sergiledi, bu da onu yapısal ve fonksiyonel çalışmalar için uygun hale getirdi.
Özet
Sarko/endoplazmik retikulum Ca2+-ATPaz (SERCA) gibi bazı P-tipi ATPazlar, yüksek saflık ve yapısal kalitede ve katalitik olarak aktif bir formda elde etmek için saflaştırma sırasında spesifik fizikokimyasal koşullar gerektiren, doğası gereği kararsız zar proteinleridir. Disakkarit trehaloz, zarları ve proteinleri stabilize etmek için maya sitoplazmasında yüksek konsantrasyonlarda sentezlenen ve biriktirilen uyumlu bir çözünen maddedir. Plazma membranı H + - ATPaz'ın saflaştırılması için protokolde bir katkı maddesi olarak trehalozun kullanılması, heksamerik yapısı biyokimyasal analitik yöntemlerle gösterilen yüksek kaliteli bir preparat ile sonuçlanır. Bu nedenle trehaloz, zar proteinlerinin (P-ATPazlar) saflaştırılması için stabilize edici bir katkı maddesi olarak kullanılabilir. Bu protokol, SERCA'yı bir trehaloz konsantrasyon gradyanı üzerinde santrifüjlemeye tabi tutarak SERCA saflaştırması için klasik protokolün modifikasyonunu açıklar. Bu karbonhidratın dahil edilmesi, SERCA'nın yüksek saflıkta katalitik olarak aktif bir formda ve daha da önemlisi stabil bir formda saflaştırılmasına yol açmıştır. Saflaştırılmış SERCA'nın kısmi biyokimyasal karakterizasyonu (SDS-PAGE, enzim kinetiği, FITC etiketleme, dairesel dikroizm spektroskopisi) enzimin fonksiyonel ve yapısal çalışmalar için uygun olduğunu göstermiştir. P-tipi ATPazların ve diğer kararsız membran (ve sitosolik) proteinlerin saflaştırma protokolünde trehalozun kullanılması önerilmektedir.
Giriş
Membran proteinleri/enzimleri, çeşitli süreçlerde kritik roller oynadıkları için hücrelerin temel biyolojik bileşenleridir 1,2,3. Fonksiyonlardan bazıları, iyonların ve moleküllerin hücre / iç bölmelerin içine ve dışına taşınmasını (aktif / pasif), hücre-hücre tanıma, hücreler arası bağlanma, ankraj / bağlanma ve normal ve sert fiziksel ve kimyasal koşullar altında sinyal iletim makinesi ile entegrasyon yoluyla dış ortamın algılanmasını içerebilir (yüksek tuz, düşük su, yüksek sıcaklık, ilaç direnci, vb.)3 Bu nedenle, zar proteinlerinin ve/veya enzimlerin üç boyutlu (3D) yapısının belirlenmesi hem temel hem de uygulamalı araştırmalar için büyük önem kazanmıştır 4,7,6. Daha da önemlisi, zar proteinleri/enzimleri, ilaç keşfi için (doğal veya tasarım gereği) hedef olarak yaygın bir şekilde kullanılmıştır7,8,9. Yani, zar proteinleri sağlıkta doğal bir öneme sahiptir 9,10,11.
Membran proteinlerinin/enzimlerinin hidrofobik karakteri, deney laboratuvarı12,13,14 için teknik olarak en zorlu fizikokimyasal özelliktir, oligomerik ve/veya oldukça kararsız integral membran proteinleri15,16 ile çalışırken daha da fazladır. Fonksiyonel deneysel testler ve yapısal çalışmalar için mümkün olan en yüksek kaliteye sahip uygun miktarlarda membran proteini/enziminin izolasyonu oldukça arzu edilir17. Doğası gereği hidrofobik olan membran proteinlerinin saflaştırılması çok zordur ve deterjan seçimi genellikle dikkate alınması gereken en önemli konulardan biridir 17,18,19,20. Bu bağlamda, zar proteinlerini izole etmek için kullanılan laboratuvar yöntemleri tipik olarak protein21'in 3 boyutlu yapısal düzenlemesine bir dereceye kadar hasara neden olur. Bu yöntemlerden bazıları (a) sonikasyon (yüksek frekanslı / enerjili ultrasonik dalgalar), (b) protein çözündürücü deterjanlar (sert, orta veya nazik)22, (c) kolon kromatografisinde nispeten yüksek basınçlar ve yüksek hızlı ultrasantrifüjleme23, (d) çökeltici moleküller, (e) sindirim enzimleri ve diğerleri21. Tüm bu işlemler, saflaştırma sırasında protein destabilitesine katkıda bulunabilir veya ana neden olabilir21. Bu bağlamda, bazı protokoller belirli bir zar proteini için nispeten iyi çalışıyor gibi görünmektedir. Bununla birlikte, tatmin edici sonuçlar elde etmek için daha yüksek kalitede membran proteini hazırlığı gerektiren yeni modern tahliller veya yöntemler kullanıldığında optimizasyon her zaman memnuniyetle karşılanır24. Optimizasyon adımları, bunlarla sınırlı olmamak üzere, yapı tasarımının iyileştirilmesini, membran protein ekspresyonu için en uygun koşulların bulunmasını, daha iyi kullanım koşullarının (yani pH, sıcaklık, vb.) oluşturulmasını, en iyi uyumlu deterjanın bulunmasını, sonikasyon süresi gibi saflaştırma adımlarının ayarlanmasını, santrifüjleme hızını, stabilize edici ajanlar ekleyerek tampon çözeltilerinin yeniden formüle edilmesini vb. içerebilir, ancak bunlarla sınırlı değildir.25,26,27. Bu nedenle, saflaştırılmış zar proteininin/enziminin kalitesinde (saflığında) ve aktivitesinde bir artışa yol açan saflaştırma metodolojisindeki herhangi bir değişiklik önemlidir.
P-tipi ATPaz ailesinde, maya plazma zarı H+-ATPaz en kararsız üyelerden biri gibi görünmektedir28,29. H+-ATPaz, dehidrasyon (dondurarak kurutma), ısı şoku vb. üzerine ATP hidrolize edici aktivitesini kaybeder.28,29. Trehalozun bir protein stabilizatörü olarak kullanımı, plazma zarı H+-ATPaz'ın maya K. lactis30,31'den izolasyonunda test edildi; Elde edilen H+-ATPaz preparatı yüksek saflıkta ve katalitik olarak aktifti. Daha da önemlisi, bu, enzimin oligomerik durumunun biyokimyasal yöntemlerle çözülmesine izin verdi, bunun bir heksamer olduğunu ortaya çıkardı ve daha sonra kriyo-elektron mikroskobu 31,32,33,34,35 ile doğrulandı. Bu nedenle, zar proteinlerinin hassas üç boyutlu (3D) düzenlemesinin, saflaştırma sırasında nispeten sert koşullar altında kaybolabileceği muhtemel görünmektedir36. Isı aracılı inaktivasyon29 sırasında H + -ATPaz için bifazik inaktivasyon kinetiği gözlenir. Maya hücrelerinde, diğer birçok organizmada olduğu gibi, disakkarit trehaloz, çevresel stres koşulları altında yüksek konsantrasyonlarda birikir 37,38,39. Trehaloz, proteinleri (hem zara gömülü hem de sitosolik) ve hücre zarlarını stabilize ederek zar bütünlüğünü ve taşıma işlevini korur40,41. Trehalozun stabilize edici mekanizması çeşitli gruplar tarafından ve laboratuvarımıztarafından kapsamlı bir şekilde incelenmiştir 42,43,44,45. H+-ATPaz ve diğer enzimlerle yapılan deneyler, trehalozun mono- ve disakkaritler arasında en etkili protein stabilizatörü olduğunu göstermiştir28,29. Bu, H + -ATPaz saflaştırma protokolüne30 dahil edilmesine yol açtı. Son zamanlarda, trehaloz, sarkoplazmik retikulum Ca2 + -ATPaz'ın (SERCA) tavşan hızlı kasılan kasından saflaştırılmasında da kullanılmıştır ve protein saflığı ve aktivite46'da iyi sonuçlar vermiştir. Bu nedenle, trehaloz, P-tipi ATPazların ve muhtemelen diğer membran ve sitozolik proteinlerin saflaştırılması için iyi ve uygun bir katkı maddesi gibi görünmektedir.
P-ATPazlar için, yapısal sitoplazmik alanların varlığı, özellikle substrat / ligand etkileşim çalışmaları için deneysel bir avantajdır47; ATP bağlanması, yüksek saflıkta rekombinant N-alanlarında 47,48,49 incelenmiştir, böylece diğerlerinin yanı sıra tüm zar enzimlerinin 47,48,50 saflaştırılması için teknik hususları ortadan kaldırmıştır 51. Ne yazık ki, bazı fonksiyonel (katalitik/enerji dönüşümü) ve yapısal (alt birim düzenlemesi ve diğer proteinlerle etkileşim) çalışmalar hala tüm P-ATPase52,53'ü gerektirmektedir. Bu bağlamda, SERCA'nın saflaştırılması çeşitli araştırma grupları tarafından sağlanmıştır 54,55,56,57,58. Bununla birlikte, iyileştirmeler hala uygulanabilir46, örneğin, saflaştırılmış ATPaz59'un sağlamlığının arttırılması, protein komplekslerinin protein denatürasyonunun/bozulmasının25 önlenmesi, zar proteininin denatürasyonu olmadan çözünmesinin arttırılması (yani, makromoleküler agregaların oluşumundan kaçınma)60, tahliller ve diğer aşağı akış analitik yöntemleriyle daha iyi uyumluluğa yol açar61 Ayrıca, bilimsel literatürde 61,62,63,64,65,66 yeni deneysel stratejiler, katkı maddeleri, enzim inhibitörleri vb. ortaya çıktıkça, bazen tüm P-ATPaz ile test edilmeleri gerekir. Bu çalışma, SERCA'nın saflaştırılması için protokolü ve protein yapısını ve ATPaz aktivitesini stabilize etmek için bir katkı maddesi olarak trehalozun kullanımını açıklar; Yani, enzim (yapısal) kalitesini arttırmanın yanı sıra, trehaloz, enzim izolasyonu ve depolanması sırasında enzim yapısı ve aktivitesinin kaybını önlemeye yardımcı olur, bu da biyolojik materyalin korunmasına ve böylece enzim saflaştırmalarının sayısının azaltılmasına yardımcı olur.
Protokol
Tüm hayvan prosedürleri, deney laboratuvarlarında hayvanların işlenmesine ilişkin uluslararası ve yerel (NORMA Oficial Mexicana NOM-062-ZOO-1999) yönergelere uygun olarak gerçekleştirildi 51,67,68. Kas dokusu, yerel bir hayvan işleme ünitesinden (INE/CITES/DGVS-ZOO-E0055-SLP-98)46 vahşi tip Oryctolagus cuniculus'tan elde edildi. Laboratuvar hayvanı yönetiminde uzmanlığa sahip bir veteriner, ilk kas diseksiyonunu ve işlemesini gerçekleştirdi. Bu çalışmada kullanılan reaktiflerin ve ekipmanların detayları Malzeme Tablosunda listelenmiştir.
1. Sarkoplazmik retikulumun hızlı kasılan tavşan kasından izolasyonu (Zamanlama, 7 - 9 saat)
NOT: Prosedürle ilgili ayrıntılar için Champeil ve ark.69'a bakın.
- Yabani tip Oryctolagus cuniculus'tan hızlı kasılan kas dokusu (hayvan başına her iki arka bacağından ~ 70 g) elde edin.
NOT: Diseke edilecek kasların önceden görsel olarak tanımlanması gereklidir70,71. - Hızlı kasılan kası, 1 dakika açık ve 1 dakika buz gibi soğuk dinlenme ile 100 mM KCl'lik üç hacimde süspanse ederek bir karıştırıcıda yumuşatın, üç kez tekrarlayın72.
- Doku kalıntılarını santrifüjleme (1500 × g) ile 4 ° C'de 5 dakika uzaklaştırın; Gerekirse ikinci bir santrifüjleme (4200 × g) yapılır.
- Süpernatanı toplayın ve bir doku öğütücü kullanarak homojenize edin (düşük hızda 10 vuruş, yani 500-1.500 rpm)72,73. Homojenatı (10.000 × g) 4 °C'de 15 dakika santrifüjleyin.
NOT: Nispeten yüksek bir hızın kullanılması sisteme ve operatöre zarar verebileceğinden, homojenizasyon sırasında dikkatli olunmalıdır. - Süpernatanı (sarkoplazmik retikulum içeren) (33.000 × g) 4 ° C'de 120 dakika santrifüjleyin.
- Peletleri 40 mL 0,5 M sükroz ve santrifüj (12.000 × g) içinde 4 ° C'de 15 dakika süreyle askıya alın. Süpernatanı seyreltin (0.6 M KCl ve 0.15 M sükroz) ve süspansiyonu (34.000 × g) 4 ° C'de 165 dakika santrifüjleyin.
- Sarkoplazmik retikulum vezikülleri (SRV'ler) tabakalarını içeren peleti 0.3 M sakaroz, 0.1 M KCl ve 5 mM Tris-HCl, pH 7.0 içinde askıya alın. Protein standardı olarak Lowry testi74 ve insan serum albüminini kullanarak protein konsantrasyonunu belirleyin.
- SRV'leri (30-32 mg / mL) 1 mL hacimde ayırın. SRV numunelerini daha sonraki işlemler için -72 °C'de saklayın.
NOT: Protein verimi hayvan başına 100-110 mg SRV'dir.
2. SERCA arıtma (Zamanlama, 18 - 20 saat)
NOT: Prosedürle ilgili ayrıntılar için Rivera-Morán ve ark.46'ya bakın.
- Buz gibi 75 mM Tris-HCl, pH 7.2, 0.6 M KCl, 6 mM EDTA, 1 mM EGTA ve% 0.1 (a/h) deoksikolat (DOC) kullanarak SRV süspansiyonunu 2 mg protein / mL'ye seyreltin. SRV'lerin süspansiyonunu buz üzerinde 10 dakika inkübe edin ve hafifçe çalkalayın. SRV'nin süspansiyonunu (100.000 × g) 4 ° C'de 60 dakika santrifüjleyin.
NOT: DOC damla damla eklenmelidir. - Peletleri <3 mL 25 mM Tris-HCl, pH 7.5, 0.3 M KCl, %45 gliserol (h/h) ve 2 mM EDTA içinde askıya alın. Süspansiyonu homojenize edin ve 10 mL'lik bir nihai hacme ayarlayın. Protein konsantrasyonunu belirleyin ve ardından 6.5 mg / mL'ye ayarlayın. 5 mg / mL ve 0.85 (a / a) Zwittergent 3-14'te azolektin ekleyin.
NOT: Azolektin ve Zwittergent 3-14 damla damla eklenmelidir. - Süspansiyonu ve santrifüjü (100.000 × g) 4 °C'de 60 dakika boyunca homojenize edin. Süpernatanı toplayın ve 1:2'yi 2 mM EGTA (pH 7.2) ile seyreltin.
- Süspansiyonu 10 mM Tris-HCl, pH 7.0, 1 mM EDTA, %0.1 deoksikolat ve 1 mg / mL azolektin içinde süreksiz bir trehaloz konsantrasyon gradyanına (45, 40, 35 ve %30 a/h) yavaşça dökün. Santrifüj (100.000 × g) 4 ° C'de 14 saat. Tüplerin dibinde oluşan şeffaf, hafif sarımsı peleti toplayın.
NOT: Hem trehaloz gradyanının oluşumu hem de numune süspansiyonunun eklenmesi uygun teknikler kullanılarak gerçekleştirilmelidir. Gece santrifüjleme önerilir. - Peleti 25 mM Tris-HCl, pH 7.5, 0.3 M KCl, %45 gliserol ve 2 mM EDTA'dan oluşan küçük bir hacimde (<1.5 mL) nazikçe askıya alın. Protein konsantrasyonunu belirleyin. Protein konsantrasyonunu 2 mg / mL'ye seyrelterek ayarlayın. Süspansiyonun alikotlarını (~ 50 μL) alın ve saflaştırılmış SERCA'yı kullanılana kadar -72 ° C'de saklayın.
- Saflaştırılmış SERCA72'nin ve farklı saflaştırma adımlarında alınan protein numunelerinin SDS-PAGE'sini gerçekleştirin. Jeli Coomassie Blue72 ile boyayın.
NOT: Süspansiyon tamponunda gliserol yerine 0,6 M trehaloz kullanılması, depolama ve donma-çözülme döngüleri28,29 sırasında enzimin stabilitesini arttırır.
3. ATPaz aktivitesi için test (Zamanlama, 1 - 2 saat)
- Enzim bağlı deney: ATPaz-Piruvat kinaz-laktat dehidrojenaz
- Reaksiyon karışımını hazırlayın (1 mL): 50 mM MOPS (pH 7.0), 1 mM EGTA, 80 mM KCl, 5 mM MgCl2, 3 mM CaCl2, 5 mM fosfoenolpiruvat, 250 μM NADH. Değişken ATP konsantrasyonlarını (0,001 ila 0,25 mM) dahil edin.
- Reaksiyon tahlilini girdaplama ile dikkatlice homojenize edin. Reaksiyon testini 37 ° C'de 10 dakika inkübe edin. 0.9 U L-laktat dehidrojenaz (LDH) ve 1.5 U piruvat kinaz (PK) ekleyin.
- 10 μg SERCA ilavesiyle ATPaz reaksiyonunu başlatın.
- NADH'nin oluşumunu belirleyin. Termostatlı hücre tutuculu bir spektrofotometre kullanarak zaman içinde 340 nm dalga boyunda (λ) 600 s boyunca her saniye absorbans yoğunluğundaki değişimi takip edin.
NOT: Absorbans ölçümü için zaman aralığı değişebilir, ancak ATPase reaksiyonunun ilk düz çizgisini çizmek için mümkün olduğunca çok veri noktası kaydetmeye çalışın.
- ATP hidroliz oranını belirleyin
NOT: Prosedürle ilgili ayrıntılar için Rivera-Morán ve ark.46'ya bakın.- Oluşturulan her eğrideki doğrusal kısmın eğim değerini (Δabs/dak) hesaplayın. ATPaz aktivitesini (μmol ATP hidrolize / min.mg prot) belirlemek için NADH'nin (λ = 6.220 M−1·cm−1) molar sönme katsayısını (ε) kullanın.
- Km ve Vmax kinetik parametrelerini belirleyin
- Veri analizi ve grafik yazılımı kullanarak ATP konsantrasyonuna karşı hız verilerini çizin.
- Verileri doğrusal olmayan regresyon yoluyla Michaelis-Menten Denklemine (Eşitlik 1)46 sığdırın:
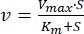
burada v hız, Vmaks maksimum hız, [S] ATP konsantrasyonu ve Km Michaelis-Menten sabitidir. - Yineleme ile K,m ve Vmax'ı belirleyin.
NOT: DOC'un dahil edilmesi ATPaz oranı yanıtını artırabilir; Optimumu bulmak için farklı DOC konsantrasyonlarının test edilmesi önerilir.
4. SERCA'nın Floresein İzotiyosiyanat (FITC) ile etiketleme deneyi (Zamanlama, 30 - 45 dakika, sadece FITC etiketlemesi için)
NOT: Prosedürle ilgili ayrıntılar için 54,75,76'ya bakın.
- 1 mM FITC içeren 50 μL (son hacim) etiketleme tamponunda (100 mM KCl, 5 mMMgCl2 ve 30 mM Tris-HCl, pH 8.9) SERCA'yı (20 μg) askıya alın. Girdap yaparak karıştırın.
- Numuneleri karanlıkta ve oda sıcaklığında farklı zamanlarda (15 dakika, 10 dakika, 7 dakika, 5 dakika ve 2 dakika) inkübe edin.
- ATP (5 mM) içeren 1 hacim buz gibi soğuk durdurma tamponu (480 mM sükroz ve 48 mM MOPS, pH 7.0) ekleyerek etiketleme reaksiyonunu durdurun. Karanlıkta 5 dakika buz üzerinde kuluçkaya yatırın.
- FITC etiketli SERCA'yı SDS-PAGE72'ye tabi tutun. Berrak jelleri UV ışığına maruz bırakın (λ = 302 nm). Sonucu fotoğrafla belgeleyin.
- Jeli Coomassie mavisi ile boyayın. Sonucu fotoğrafla belgeleyin.
NOT: FITC etiketlemesinin karanlık bir odada yapılması floresan sinyalinin kalitesini artırabilir.
5. Dairesel dikroizm (CD) spektrumları (Zamanlama, 30 dk)
NOT: Prosedürle ilgili ayrıntılar için 11,77'ye bakın.
- SERCA'yı (3 μM) 25 ° C'de 400 μL 10 mM fosfat tamponunda (pH 7.0) askıya alın.
- Numuneyi 0,1 cm yol uzunluğunda bir hücreye yükleyin. Uzak UV spektrumları λ aralığını 190-260 nm arasında ayarlayın. Dahili çözünürlüğü ve bant genişliğini 1 nm olarak ayarlayın.
- CD sinyalini 25 °C'de 50 nm/dk'da kaydedin.
Sonuçlar
Saflaştırmanın farklı aşamalarında SERCA'nın SDS-PAGE'i (Şekil 1). Coomassie mavisi lekeli jel, saflaştırma protokolü ilerledikçe SERCA protein bandının (görünür moleküler ağırlık >100 kDa) zenginleşmesini gösterir. SERCA'ya karşılık gelen protein bandı, bir trehaloz konsantrasyon gradyanı üzerinde santrifüjlemeden sonra% >90'lık bir saflık gösterir. ATP konsantrasyonuna karşı ATP hidroliz hızının grafiği (
Tartışmalar
Çoğu molekül ve iyon hücre zarlarını serbestçe geçemez, örneğin proton (H+), mitokondri83,84 gibi çeşitli organizma ve organellerin plazma zarında bir zar taşıyıcısı gerektirir. Hücre zarları seçicidir ve hücre zarlarını geçen moleküller ve iyonlar çeşitlidir, bu nedenle hücrede (a) ABC taşıyıcıları, (b) iyon kanalları, (c) zara bağlı ATPazlar, (d) SLC taşıyıcıları ve (e) su ka...
Açıklamalar
Yazar, rekabet eden hiçbir mali çıkarı olmadığını beyan eder.
Teşekkürler
Yazar, videonun düzenlenmesinde Edmundo Mata-Morales'in, VM'nin, Valentín de la Cruz-Torres'in SRV'lerin saflaştırılmasında, Miguel A. Rivera-Moran'ın SERCA saflaştırma ve analizinde ve Juan C. Gonzalez-Castro'nun yardımını kabul eder. Bu çalışmayı yürütmek için herhangi bir fon, hibe veya başka bir destek alınmamıştır.
Malzemeler
| Name | Company | Catalog Number | Comments |
| ATP | Sigma-Aldrich Corp | A2383 | |
| Azolectin from soybean | Sigma-Aldrich Corp | 44924 | |
| Benchtop UV transilluminator | Cole-Parmer | EW-97623-08 | Dual intensity High setting is ideal for analytical documentation. Low setting reduces photonicking or photobleaching of gel samples while doing preparative work. |
| CaCl2 • 2H2O | Sigma-Aldrich Corp | 223506 | |
| Coolpix B500 camera | Nikon Corp | S210 | |
| Coomassie brilliant blue G-250 | Bio-Rad | 1610406 | |
| Dodecyl maltoside | Sigma-Aldrich Corp | D4641 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid | Sigma-Aldrich Corp | E4378 | |
| Ethylenediaminetetraacetic acid tetrasodium salt dihydrate | Sigma-Aldrich Corp | E6511 | |
| Fluorescein isothiocyanate | Sigma-Aldrich Corp | F3651 | |
| KCl | JT Baker | 7447-40-7 | |
| MgCl2 • 6H2O | Sigma-Aldrich Corp | M9272 | |
| MOPS | Sigma-Aldrich Corp | M1254 | |
| NADH | Chem-Impex International Inc | 230 | |
| N-tetradecyl-N,N- dimethyl-3-ammonium-1-propanesulfonate | Sigma-Aldrich Corp | D0431 | |
| Phosphoenolpyruvate (PEP) | Chem-Impex International Inc | 9711 | |
| Rabbit muscle lactate dehydrogenase | Roche | 10003557103 | |
| Rabbit muscle pyruvate kinase | Sigma-Aldrich Corp | P1506 | |
| Sodium deoxycholate | Sigma-Aldrich Corp | D6750 | |
| Sodium dodecyl sulfate | Bio-Rad | 1610302 | |
| Spectropolarimeter | Jasco Corp. | Jasco J1500 | |
| Sucrose | Sigma-Aldrich Corp | 84100 | |
| Trehalose | Sigma-Aldrich Corp | T0167 | Dihydrate |
| Tris(hydroxymethyl)aminomethane hydrochloride | Sigma-Aldrich Corp | 857645 | |
| Ultracentrifuge | Beckman | Optima XPN | |
| UV/VIS spectrophotometer | Agilent Technologies | 8453 | The Agilent 8453 UV-Vis Spectrophotometer uses a photodiode array for simultaneous measurement of the complete ultra-violet to visible light spectrum |
| WiseStir HS- 30E | Daihan Scientific Co. | DH.WOS01010 | Ideal for all disperging and homogenizing applications, designed for tissue grinders. |
Referanslar
- Schuberth, C., Wedlich-Söldner, R. Building a patchwork - The yeast plasma membrane as model to study lateral domain formation. Biochim Biophys Acta Mol Cell Res. 1853 (4), 767-774 (2015).
- Douglas, L. M., Konopka, J. B. Fungal membrane organization: The eisosome concept. Annu Rev Microbiol. 68 (1), 377-393 (2014).
- Borrell, J. H., Domènech, &. #. 2. 1. 0. ;., Keough, K. M. W. . Membrane protein - Lipid interactions: Physics and Chemistry in the Bilayer. , (2016).
- Moraes, I., Evans, G., Sanchez-Weatherby, J., Newstead, S., Stewart, P. D. . The next generation in membrane protein structure determination. , (2016).
- White, S. H. Biophysical dissection of membrane proteins. Nature. 459 (7245), 344-346 (2009).
- Lacapère, J. -. J. Membrane protein structure determination. Methods Mol Biol. , 459 (1002).
- Palmgren, M. G. Plant plasma membrane H+-ATPases: Powerhouses for nutrient uptake. Annu Rev Plant Physiol Plant Mol Biol. 52 (1), 817-845 (2001).
- Yatime, L., et al. P-type ATPases as drug targets: Tools for medicine and science. Biochim Biophys Acta Bioenerg. 1787 (4), 207-220 (2009).
- Drews, J. Tetrahydrobiopterin biosynthesis as an off-target of sulfa drugs. Science. 287 (5460), 1960-1964 (2000).
- Xu, Z., Meshcheryakov, V. A., Poce, G., Chng, S. -. S. MmpL3 is the flippase for mycolic acids in mycobacteria. Proc Natl Acad Sci USA. 114 (30), 7993-7998 (2017).
- Siligardi, G., Hussain, R., Patching, S. G., Phillips-Jones, M. K. Ligand- and drug-binding studies of membrane proteins revealed through circular dichroism spectroscopy. Biochim Biophys Acta Biomembr. 1838 (1 PARTA), 34-42 (2014).
- Rawlings, A. E. Membrane proteins: Always an insoluble problem. Biochem Soc Trans. 44 (3), 790-795 (2016).
- Popot, J. -. L. . Membrane proteins in aqueous solutions. , 708 (2018).
- Palmgren, M. G., Sommarin, M., Ulvskov, P., Larsson, C. Effect of detergents on the H+-ATPase activity of inside-out and right-side-out plant plasma membrane vesicles. Biochim Biophys Acta Biomembr. 1021 (2), 133-140 (1990).
- McBride, Z., Chen, D., Reick, C., Xie, J., Szymanski, D. B. Global analysis of membrane-associated protein oligomerization using protein correlation profiling. Mol Cell Proteomics. 16 (11), 1972-1989 (2017).
- Yoneda, J. S., et al. Multimeric species in equilibrium in detergent-solubilized Na, K-ATPase. Int J Biol Macromol. 89, 238-245 (2016).
- Ohlendieck, K. Extraction of membrane proteins. Protein Purif Protocols. 59, 283-294 (2004).
- Champeil, P., et al. A robust method to screen detergents for membrane protein stabilization, revisited. Anal Biochem. 511, 31-35 (2016).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biol Proced Online. 18, 4 (2016).
- le Maire, M., et al. Gel chromatography and analytical ultracentrifugation to determine the extent of detergent binding and aggregation, and Stokes radius of membrane proteins using sarcoplasmic reticulum Ca2+-ATPase as an example. Nat Protoc. 3 (11), 1782-1795 (2008).
- Du, M., et al. Progress, applications, challenges and prospects of protein purification technology. Front Bioeng Biotechnol. 10, 1-26 (2022).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Harding, S. E. Analytical ultracentrifugation as a matrix-free probe for the study of kinase related cellular and bacterial membrane proteins and glycans. Molecules. 26, 6080 (2021).
- Chen, Y. -. C., et al. Thermal stability, storage and release of proteins with tailored fit in silica. Sci Rep. 7 (1), 46568 (2017).
- Montigny, C. C., Arnou, B., Marchal, E., Champeil, P. Use of glycerol-containing media to study the intrinsic fluorescence properties of detergent-solubilized native or expressed SERCA1a. Biochemistry. 47 (46), 12159-12174 (2008).
- Simongini, M., Puglisi, A., Genovese, F., Hochkoeppler, A. Trehalose counteracts the dissociation of tetrameric rabbit lactate dehydrogenase induced by acidic pH conditions. Arch Biochem Biophys. 740, 109584 (2023).
- Shivanna, B. D., Rowe, E. S. Preservation of the native structure and function of Ca2+-ATPase from sarcoplasmic reticulum: Solubilization and reconstitution by new short-chain phospholipid detergent 1,2-diheptanoyl-sn-phosphatidylcholine. Biochem J. 325 (2), 533-542 (1997).
- Sampedro, J. G., Guerra, G., Pardo, J. P., Uribe, S. Trehalose-mediated protection of the plasma membrane H+-ATPase from Kluyveromyces lactis during freeze-drying and rehydration. Cryobiology. 37 (2), 131-138 (1998).
- Sampedro, J. G., Cortés, P., Muñoz-Clares, R. A., Fernández, A., Uribe, S. Thermal inactivation of the plasma membrane H+-ATPase from Kluyveromyces lactis. Protection by Trehalose. Biochim Biophys Acta Proteins Proteomics. 1544 (1-2), 64-73 (2001).
- Sampedro, J. G., et al. Fluorescence quenching by nucleotides of the plasma membrane H+-ATPase from Kluyveromyces lactis. Biochemistry. 46 (18), 5616-5622 (2007).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Zhao, P., Zhao, C., Chen, D., Yun, C., Li, H., Bai, L. Structure and activation mechanism of the hexameric plasma membrane H+-ATPase. Nat Commun. 12 (1), 6439 (2021).
- Auer, M., Scarborough, G. A., Kühlbrandt, W. Surface crystallization of the plasma membrane H+-ATPase on a carbon support film for electron crystallography. J Mol Biol. 287 (5), 961-968 (1999).
- Hennessey, J. P., Scarborough, G. A. Secondary structure of the Neurospora CRASSA plasma membrane H+-ATPase as estimated by circular dichroism. J Biol Chem. 263 (7), 3123-3130 (1988).
- Scarborough, G. A. Crystallization, structure and dynamics of the proton-translocating P-type ATPase. J Exp Biol. 203 (Pt 1), 147-154 (2000).
- Sumida, K. H., et al. Improving protein expression, stability, and function with proteinMPNN. J Am Chem Soc. 146 (3), 2054-2061 (2024).
- Eleutherio, E., Panek, A., De Mesquita, J. F., Trevisol, E., Magalhães, R. Revisiting yeast trehalose metabolism. Curr Genet. 61 (3), 263-274 (2015).
- Attfield, P. V. Trehalose accumulates in Saccharomyces cerevisiae during exposure to agents that induce heat shock response. FEBS Lett. 225 (1-2), 259-263 (1987).
- Marunde, M. R., et al. Improved tolerance to salt and water stress in Drosophila melanogaster cells conferred by late embryogenesis abundant protein. J Insect Physiol. 59 (4), 377-386 (2013).
- Crowe, J. H. Anhydrobiosis: An unsolved problem with applications in human welfare. Subcell Biochem. 71, 263-280 (2015).
- Crowe, J. H., Crowe, L. M., Chapman, D. Preservation of membranes in anhydrobiotic organisms: The role of trehalose. Science. 223 (4637), 701-703 (1984).
- Sampedro, J. G., Rivera-Moran, M. A., Uribe-Carvajal, S. Kramers' theory and the dependence of enzyme dynamics on trehalose-mediated viscosity. Catalysts. 10 (6), 1-19 (2020).
- Sampedro, J. G., Uribe, S. Trehalose-enzyme interactions result in structure stabilization and activity inhibition. The role of viscosity. Mol Cell Biochem. 256 - 257 (1-2), 319-327 (2004).
- Crowe, J. H. Trehalose as a "chemical chaperone": Fact and fantasy. Adv Exp Med Biol. 594, 143-158 (2007).
- Crowe, J. H., Carpenter, J. F., Crowe, L. M., Anchordoguy, T. J. Are freezing and dehydration similar stress vectors? A comparison of modes of interaction of stabilizing solutes with biomolecules. Cryobiology. 27 (3), 219-231 (1990).
- Rivera-Morán, M. A., Sampedro, J. G. Isolation of the sarcoplasmic reticulum Ca2+-ATPase from rabbit fast-twitch muscle. Methods Protoc. 6 (5), 102 (2023).
- Páez-Pérez, E. D., De La Cruz-Torres, V., Sampedro, J. G. Nucleotide binding in an engineered recombinant Ca2+-ATPase N-domain. Biochemistry. 55 (49), 6751-6765 (2016).
- Ramírez-Alonso, J. I., Sampedro, J. G. Effect of cations on ATP binding to the N-domain of Na+, K+-ATPase. J Fluoresc. , (2024).
- De la Cruz-Torres, V., Cataño, V., Olivo-Rodríguez, M., Sampedro, J. G. ANS Interacts with the Ca2+-ATPase nucleotide binding site. J Fluoresc. 30 (3), 483-496 (2020).
- Sampedro, J. G., Nájera, H., Uribe-Carvajal, S., Ruiz-Granados, Y. G. Mapping the ATP binding site in the plasma membrane H+-ATPase from Kluyveromyces lactis. J Fluoresc. 24 (6), 1849-1859 (2014).
- Kiani, A. K., et al. Ethical considerations regarding animal experimentation. J Prev Med Hyg. 63 (2 Suppl 3), E255-E266 (2022).
- Ruiz-Granados, Y., De La Cruz-Torres, V., Sampedro, J. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Wang, S., et al. Structural basis for sarcolipin's regulation of muscle thermogenesis by the sarcoplasmic reticulum Ca2+-ATPase. Sci Adv. 7 (48), eabi7154 (2021).
- Champeil, P., et al. ATP regulation of sarcoplasmic reticulum Ca2+-ATPase. Metal-free ATP and 8-bromo-ATP bind with high affinity to the catalytic site of phosphorylated ATPase and accelerate dephosphorylation. J Biol Chem. 263 (25), 12288-12294 (1988).
- Møller, J. V., Olesen, C. Preparation of Ca(2+)-ATPase1a enzyme from rabbit sarcoplasmic reticulum. Methods Mol Biol. 1377, 11-17 (2016).
- Wuytack, F., De Schutter, G., Casteels, R. Partial purification of (Ca2+ + Mg2+)-dependent ATPase from pig smooth muscle and reconstitution of an ATP-dependent Ca2+-transport system. Biochem J. 198 (2), 265-271 (1981).
- Mandal, A., et al. Solubilization, purification and reconstitution of Ca2+-ATPase from bovine pulmonary artery smooth muscle microsomes by different detergents: Preservation of native structure and function of the enzyme by DHPC. Biochim Biophys Acta Gen Subj. 1760 (1), 20-31 (2006).
- Inesi, G., Ma, H., Hua, S., Toyoshima, C. Characterization of Ca2+ ATPase residues involved in substrate and cation binding. Ann NY Acad Sci. 986 (1), 63-71 (2003).
- Van Winkle, W. B., Pitts, B. J., Entman, M. L. Rapid purification of canine cardiac sarcoplasmic reticulum Ca2+-ATPase. J Biol Chem. 253 (24), 8671-8673 (1978).
- Ruiz-Granados, Y. G., De La Cruz-Torres, V., Sampedro, J. G. The oligomeric state of the plasma membrane H+-ATPase from Kluyveromyces lactis. Molecules. 24 (5), 958 (2019).
- Teramoto, N., Sachinvala, N. D., Shibata, M. Trehalose and trehalose-based polymers for environmentally benign, biocompatible, and bioactive materials. Molecules. 13 (8), 1773-1816 (2008).
- Dao, T. T., et al. Demethoxycurcumin is a potent inhibitor of P-type ATPases from diverse kingdoms of life. PLOS ONE. 11 (9), e0163260 (2016).
- Bleeker, N. P., Cornea, R. L., Thomas, D. D., Xing, C. A novel SERCA inhibitor demonstrates synergy with classic SERCA inhibitors and targets multidrug-resistant AML. Mol Pharmaceutics. 10 (11), 4358-4366 (2013).
- Michelangeli, F., East, J. M. A diversity of SERCA Ca2+ pump inhibitors. Biochem Soc Trans. 39 (3), 789-797 (2011).
- Tadini-Buoninsegni, F., Smeazzetto, S., Gualdani, R., Moncelli, M. R. Drug interactions with the Ca2+-ATPase from Sarco(Endo)Plasmic Reticulum (SERCA). Front Mol Biosci. 5, 36 (2018).
- Clausen, J. D., et al. Elucidation of antimicrobial activity and mechanism of action by N-substituted carbazole derivatives. Bioorg Med Chem Lett. 27 (19), 4564-4570 (2017).
- Leary, S., et al. . AVMA guidelines for the euthanasia of animals: 2020 Edition, Members of the Panel on Euthanasia AVMA Staff Consultants. , (2020).
- de Aluja, A. S. Laboratory animals and official Mexican norms (NOM-062-ZOO-1999). Gac Med Mex. 138 (3), 295-298 (2002).
- Champeil, P., Buschlen-Boucly, S., Bastide, F., Gary-Bobo, C. Sarcoplasmic reticulum ATPase. Spin labeling detection of ligand-induced changes in the relative reactivities of certain sulfhydryl groups. J Biol Chem. 253 (4), 1179-1186 (1978).
- Skalec, A., Janeczek, M., Czerski, A. Anatomy and histology of the rabbit common calcanean tendon. J Vet Med Series C: Anat Histol Embryol. 48 (5), 466-475 (2019).
- Janes, L. E., Mioton, L. M., Fracol, M. E., Ko, J. H. An in vivo comparison: Novel mesh suture versus traditional suture-based repair in a rabbit tendon model. J Hand Surg Glob Online. 4 (1), 32-39 (2022).
- Bollag, D. M., Rozycki, M. D., Edelstein, S. J. . Protein Methods. , (1996).
- Gagné, F. Tissue preparation and subcellular fractionation techniques. Biochem Ecotoxicol. , 21-31 (2014).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. J Biol Chem. 193 (1), 265-275 (1951).
- Autry, J. M., Rubin, J. E., Svensson, B., Li, J. L., Thomas, D. D. Nucleotide activation of the Ca-ATPase. J Biol Chem. 287 (46), 39070-39082 (2012).
- Winters, D. L., Autry, J. M., Svensson, B., Thomas, D. D. Interdomain fluorescence resonance energy transfer in SERCA probed by cyan-fluorescent protein fused to the actuator domain. Biochemistry. 47 (14), 4246-4256 (2008).
- Miles, A. J., Wallace, B. A. Circular dichroism spectroscopy of membrane proteins. Chem Soc Rev. 14 (45), 1130-1135 (2016).
- Scofano, H. M., Vieyra, A., de Meis, L. Substrate regulation of the sarcoplasmic reticulum ATPase. Transient kinetic studies. J Biol Chem. 254 (20), 10227-10231 (1979).
- Yamamoto, T., Tonomura, Y. Reaction mechanism of the Ca++-dependent ATPase of sarcoplasmic reticulum from skeletal muscle: I. Kinetic studies. J Biochem. 62 (5), 558-575 (1967).
- Murphy, A. J. Affinity labeling of the active site of the Ca2+-ATPase of sarcoplasmic reticulum. Biochim Biophys Acta - Biomembr. 946 (1), 57-65 (1988).
- Csermely, P., Katopis, C., Wallace, B. A., Martonosi, A. The E1→E2 transition of Ca2+-transporting ATPase in sarcoplasmic reticulum occurs without major changes in secondary structure. A circular-dichroism study. Biochem J. 241 (3), 663-669 (1987).
- Abràmoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Int. 11 (7), 36-42 (2004).
- Scarborough, G. A. The plasma membrane proton-translocating ATPase. Cell Mol Life Sci. 57 (6), 871-883 (2000).
- Llopis, J., McCaffery, J. M., Miyawaki, A., Farquhar, M. G., Tsien, R. Y. Measurement of cytosolic, mitochondrial, and Golgi pH in single living cells with green fluorescent proteins. Proc Natl Acad Sci. 95 (12), 6803-6808 (1998).
- Kanai, R., Vilsen, B., Cornelius, F., Toyoshima, C. Crystal structures of Na+, K+-ATPase reveal the mechanism that converts the K+-bound form to Na+-bound form and opens and closes the cytoplasmic gate. FEBS Lett. 597 (15), 1957-1976 (2023).
- Palmgren, M. P-type ATPases: Many more enigmas left to solve. J Biol Chem. 299 (11), 105352 (2023).
- Frauenfelder, H. Ask not what physics can do for biology-Ask what biology can do for physics. Phys Biol. 11 (5), 053004 (2014).
- Frauenfelder, H. The physics of proteins. Annu Rev Biophys Biomol Struct. 36, (2010).
- Narhi, L. O. . Biophysics for Therapeutic Protein Development. , (2013).
- East, J. M. Purification of a membrane protein (Ca2+/Mg2+-ATPase) and its reconstitution into lipid vesicles. Methods Mol Biol. 27, 87-94 (1994).
- Lee, E. -. H., et al. Enhancement of enzyme activity and stability by poly(γ-glutamic acid). Polymer J. 42 (10), 818-822 (2010).
- Norrild, R. K., et al. Increasing protein stability by inferring substitution effects from high-throughput experiments. Cell Rep Methods. 2 (11), 100333 (2022).
- Song, Z., Zhang, Q., Wu, W., Pu, Z., Yu, H. Rational design of enzyme activity and enantioselectivity. Front Bioeng Biotechnol. 11, 1-14 (2023).
- Jidenko, M., Lenoir, G., Fuentes, J. M., le Maire, M., Jaxel, C. Expression in yeast and purification of a membrane protein, SERCA1a, using a biotinylated acceptor domain. Protein Expr Purif. 48 (1), 32-42 (2006).
- Hua, S., Ma, H., Lewis, D., Inesi, G., Toyoshima, C. Functional role of "N" (nucleotide) and "P" (phosphorylation) domain interactions in the sarcoplasmic reticulum (SERCA) ATPase. Biochemistry. 41 (7), 2264-2272 (2002).
- Pang, Y. -. H., Chen, J. -. W. Anisodamine causes the changes of structure and function in the transmembrane domain of the Ca2+-ATPase from sarcoplasmic reticulum. Biosci Biotechnol Biochem. 68 (1), 126-131 (2004).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır