A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
عزل ناقلات العلاج الجيني من الجيل التالي من خلال الهندسة والترميز الشريطي وفحص متغيرات قفيصة الفيروس المرتبط بالغدي (AAV)
In This Article
Summary
إنشاء مكتبة عرض الببتيد AAV والتحقق اللاحق من خلال الترميز الشريطي للمرشحين ذوي الخصائص الجديدة لإنشاء الجيل التالي من AAVs.
Abstract
تعد نواقل توصيل الجينات المشتقة من الفيروس المرتبط بالغدي (AAV) واحدة من أكثر الأدوات الواعدة لعلاج الأمراض الوراثية ، ويتضح ذلك من خلال تشجيع البيانات السريرية والموافقة على العديد من العلاجات الجينية AAV. هناك سببان رئيسيان لنجاح نواقل AAV هما (i) العزل المسبق لمختلف الأنماط المصلية الفيروسية التي تحدث بشكل طبيعي مع خصائص مميزة ، و (ii) الإنشاء اللاحق لتقنيات قوية لهندستها الجزيئية وإعادة توظيفها في إنتاجية عالية. ومما يعزز إمكانات هذه التقنيات مؤخرا استراتيجيات تم تنفيذها لترميز قفيصات AAV مختارة على مستوى الحمض النووي والحمض النووي الريبي ، مما يسمح بتقسيمها الطبقي الشامل والمتوازي في الجسم الحي في جميع الأعضاء الرئيسية وأنواع الخلايا في واحد. هنا ، نقدم خط أنابيب أساسي يشمل هذه المجموعة من السبل التكميلية ، باستخدام عرض الببتيد AAV لتمثيل الترسانة المتنوعة لتقنيات هندسة القفيصة المتاحة. وفقا لذلك ، نصف أولا الخطوات المحورية لإنشاء مكتبة عرض الببتيد AAV للاختيار في الجسم الحي للمرشحين ذوي الخصائص المطلوبة ، متبوعا بعرض توضيحي لكيفية ترميز متغيرات القفيصة الأكثر إثارة للاهتمام للفحص الثانوي في الجسم الحي . بعد ذلك ، نمثل منهجية إنشاء مكتبات لتسلسل الجيل التالي (NGS) ، بما في ذلك تضخيم الباركود وربط المحول ، قبل أن نختتم بنظرة عامة على الخطوات الأكثر أهمية أثناء تحليل بيانات NGS. نظرا لأن البروتوكولات المذكورة هنا متعددة الاستخدامات وقابلة للتكيف ، يمكن للباحثين تسخيرها بسهولة لإثراء متغيرات قفيصة AAV المثلى في نموذج المرض المفضل لديهم ولتطبيقات العلاج الجيني.
Introduction
العلاج بنقل الجينات هو إدخال مادة وراثية في الخلايا لإصلاح المادة الوراثية الخلوية أو استبدالها أو تغييرها للوقاية من المرض أو علاجه أو علاجه أو تحسينه. يعتمد نقل الجينات ، سواء في الجسم الحي أو خارج الجسم الحي ، على أنظمة توصيل مختلفة ، غير فيروسية وفيروسية. تطورت الفيروسات بشكل طبيعي لتحويل خلاياها المستهدفة بكفاءة ويمكن استخدامها كناقلات توصيل. من بين الأنواع المختلفة من النواقل الفيروسية المستخدمة في العلاج الجيني ، تم استخدام الفيروسات المرتبطة بالغدي بشكل متزايد ، نظرا لافتقارها إلى الإمراضية والسلامة وانخفاض المناعة ، والأهم من ذلك قدرتها على الحفاظ على التعبير طويل الأجل وغير المتكامل1،2،3. حقق العلاج الجيني AAV إنجازات كبيرة على مدى العقد الماضي. تمت الموافقة على ثلاثة علاجات من قبل وكالة الأدوية الأوروبية وإدارة الغذاء والدواء الأمريكية للاستخدام في البشر 3,4. كما تجري العديد من التجارب السريرية لعلاج مجموعة متنوعة من الأمراض ، مثل الهيموفيليا وأمراض العضلات والقلب والأمراض العصبية ، كما تمت مراجعته في مكان آخر3. على الرغم من عقود من التقدم ، شهد مجال العلاج الجيني سلسلة من النكسات في السنوات الأخيرة4 ، وأهمها الوفيات في التجارب السريرية5 التي تم تعليقها بسبب السمية التي تحد من الجرعة ، خاصة بالنسبة للأنسجة الضخمة ، مثل العضلات ، أو التي يصعب الوصول إليها ، مثل الدماغ6.
تنتمي ناقلات AAV المستخدمة حاليا في التجارب السريرية إلى الأنماط المصلية الطبيعية مع استثناءات قليلة1. توفر هندسة AAV الفرصة لتطوير ناقلات ذات خصوصية وكفاءة فائقة للأعضاء أو الخلايا. في العقدين الماضيين ، تم تطبيق العديد من الأساليب بنجاح ، مثل عرض الببتيد ، ومبادلة الحلقة ، وخلط الحمض النووي للقفيصة ، وتفاعل البوليميراز المتسلسل المعرض للخطأ ، والتصميم المستهدف ، لإنشاء متغيرات AAV فردية أو مكتبات لها بخصائص متنوعة7. ثم تخضع هذه لجولات متعددة من التطور الموجه لتحديد المتغيرات داخلها مع الخصائص المطلوبة ، كما تمت مراجعته في مكان آخر 1,3. من بين جميع استراتيجيات تطور القفيصة ، كانت مكتبات عرض الببتيد AAV هي الأكثر استخداما ، نظرا لبعض الخصائص الفريدة: فهي سهلة التوليد نسبيا ، ويمكنها تحقيق تنوع عال وتسلسل عالي الإنتاجية ، مما يسمح بتتبع تطورها.
تم وصف أول مكتبات AAV ناجحة لإدخال الببتيد منذ ما يقرب من 20 عاما. في واحدة من الأولى ، قام Perabo et al.8 ببناء مكتبة من قفيصات AAV2 المعدلة ، حيث تم إدخال مجموعة من قليل النوكليوتيدات المولدة عشوائيا في بلازميد في موضع يتوافق مع الأحماض الأمينية 587 من بروتين قفيصة VP1 ، في المحور ثلاثي الأضواط البارز من القفيصة. باستخدام العدوى المشتركة للفيروس الغدي ، تم تطوير مكتبة AAV من خلال جولات متعددة من الاختيار ، وتبين أن المتغيرات النهائية المعاد استهدافها قادرة على تحويل خطوط الخلايا المقاومة إلى AAV28 الأبوي. بعد ذلك بوقت قصير ، قدم Müller et al.9 نظام الخطوتين لإنتاج المكتبات ، وهو تحسن كبير في البروتوكول. في البداية ، يتم استخدام مكتبة البلازميد ، جنبا إلى جنب مع البلازميد المساعد للفيروسات الغدية ، لإنتاج مكتبة AAV تحتوي على قفيصات خيمرية. تستخدم مكتبة مكوك AAV هذه لإصابة الخلايا بمضاعفات منخفضة للعدوى (MOI) ، بهدف إدخال جينوم فيروسي واحد لكل خلية. تضمن العدوى المشتركة بالفيروس الغدي إنتاج AAVs مع جينوم مطابق وقفيصة9. بعد حوالي عقد من الزمان ، استخدم Dalkara10 التطور الموجه في الجسم الحي لإنشاء متغير 7m8. يحتوي هذا المتغير على 10 إدخال حمض أميني (LALGETTRPA) ، ثلاثة منها تعمل كروابط ، وتستهدف الشبكية الخارجية بكفاءة بعد الحقن داخل الجسم الزجاجي10. هذه القفيصة الهندسية هي قصة نجاح استثنائية ، لأنها واحدة من القفيصات الهندسية القليلة التي وصلت إلى العيادة حتى الآن11.
شهد المجال دفعة ثانية مع إدخال تقنيات التسلسل من الجيل التالي (NGS). عرض منشوران من Adachi et al.12 في عام 2014 ومن Marsic et al.13 في عام 2015 ، قوة NGS لتتبع توزيع مكتبات قفيصة AAV المشفرة بدقة عالية. بعد بضع سنوات ، تم تكييف NGS لمناطق الباركود مع منطقة إدخال الببتيد لمتابعة تطور القفيصة. أجرى Körbelin et al.14 فحصا موجها ب NGS لتحديد قفيصة تعتمد على AAV2 تستهدف الرئة. ساعد تحليل NGS في حساب ثلاث درجات تصنيف: درجة الإثراء بين جولات الاختيار ، ودرجة الخصوصية العامة لتحديد خصوصية الأنسجة ، وأخيرا النتيجة المجمعة14. نشر مختبر Gradinaru15 نظام التطور المستهدف AAV القائم على Cre-reالتركيب (CREATE) في نفس العام ، مما يسهل الاختيار الخاص بنوع الخلية. في هذا النظام ، تحمل مكتبة capsid مفتاحا قابلا للعكس Cre ، حيث أن إشارة polyA محاطة بموقعين loxP. ثم يتم حقن مكتبة AAV في فئران Cre ، حيث يتم عكس إشارة polyA فقط في خلايا Cre + ، مما يوفر نموذجا لربط التمهيدي PCR العكسي مع التمهيدي الأمامي داخل جين القفيصة. مكن هذا الإنقاذ PCR المحدد للغاية من تحديد AAV-PHP. البديل B الذي يمكن أن يعبر حاجز الدم في الدماغ15. تم تطوير هذا النظام بشكل أكبر إلى M-CREATE (Multiplexed-CREATE) ، حيث تم دمج NGS وتوليد المكتبة الاصطناعية في خط الأنابيب16.
تسمح نسخة محسنة من هذا النظام القائم على الحمض النووي الريبي من مختبر ماجواير17 ، iTransduce ، بالاختيار على مستوى الحمض النووي للقفيصة التي تحول الخلايا وظيفيا وتعبر عن جينومها. يشتمل الجينوم الفيروسي لمكتبة عرض الببتيد على جين Cre تحت سيطرة مروج في كل مكان وجين قفيصة تحت سيطرة مروج p41. يتم حقن المكتبة في الفئران التي تحتوي على كاسيت loxP-STOP-loxP في المنبع من tdTomato. يمكن فرز الخلايا التي تم تحويلها باستخدام متغيرات AAV التي تعبر عن الجينوم الفيروسي وبالتالي Cre express tdTomato ، وبالاقتران مع علامات الخلايا ، واختيارها17. وبالمثل ، وضع Nonnenmacher et al.18 و Tabebordbar et al.19 مكتبة جينات القفيصة تحت سيطرة المروجين الخاصين بالأنسجة. بعد الحقن في نماذج حيوانية مختلفة ، تم استخدام الحمض النووي الريبي الفيروسي لعزل متغيرات القفيصة.
هناك طريقة بديلة تتمثل في استخدام الترميز الشريطي لوضع علامة على مكتبات capsid. استخدم مختبر Björklund20 هذا النهج لمكتبات قفيصة إدخال الببتيد الباركود وطور تطور متجه AAV العقلاني المشفر بالباركود (BRAVE). في بلازميد واحد ، يتم استنساخ كاسيت Rep2Cap بجوار التكرارات الطرفية المقلوبة (ITR) ، والبروتين الفلوري الأصفر (YFP) - الذي يعبر عن الجينات المحورة ذات العلامات الشريطية . باستخدام مواقع loxP بين نهاية الغطاء وبداية الرمز الشريطي ، تولد إعادة التركيب Cre في المختبر جزءا صغيرا بما يكفي ل NGS ، مما يسمح بربط إدخال الببتيد بالرمز الشريطي الفريد (جدول البحث ، LUT). يتم تنفيذ إنتاج AAV باستخدام مكتبة البلازميد ويتم فحص الرموز الشريطية المعبر عنها في mRNA بعد تطبيق الجسم الحي ، مرة أخرى باستخدام NGS20. عندما تشتمل مكتبات القفيصة على متغيرات من جين القفيصة بالكامل (أي المكتبات المختلطة) ، يجب استخدام تسلسل القراءة الطويلة. استخدمت العديد من المجموعات الرموز الشريطية لوضع علامة على هذه المكتبات المتنوعة ، مما يتيح NGS بعمق قراءة أعلى. قام Kay lab21 بوضع علامة على مكتبات مختلطة شديدة التنوع مع الرموز الشريطية في اتجاه مجرى إشارة cap polyA. في الخطوة الأولى ، تم إنشاء مكتبة بلازميد مشفرة بالباركود ، وتم استنساخ مكتبة جينات القفيصة المختلطة فيها. ثم تم استخدام مزيج من MiSeq (قراءة قصيرة ، عمق قراءة أعلى) و PacBio (قراءة طويلة ، عمق قراءة أقل) NGS بالإضافة إلى تسلسل Sanger لإنشاء LUT21. في عام 2019 ، حدد Ogden وزملاؤه من مختبر Churchlab 22 ملاءمة قفيصة AAV2 لوظائف متعددة باستخدام المكتبات التي تحتوي على طفرات أحادية النقطة وعمليات إدراج وحذف في كل موضع ، مما مكن في النهاية من التصميم الموجه آليا. لتوليد المكتبة ، تم تصنيع أجزاء أصغر من جين القفيصة ، ووضع علامة عليها برمز شريطي ، وتسلسل الجيل التالي ، ثم استنساخها في جين القفيصة الكامل. تم استخدام بيانات NGS لإنشاء جدول بحث. ثم تم فحص المكتبة باستخدام الرموز الشريطية فقط وتسلسل القراءة القصيرة ، والذي بدوره يسمح بعمق قراءة أعلى22.
تم استخدام المكتبات المشفرة في الغالب لفحص مجموعة من المتغيرات المعروفة والطبيعية والهندسية بعد عدة جولات من اختيار مكتبات القفيصة أو مستقلة عن دراسة تطور القفيصة. ميزة هذه المكتبات هي فرصة فحص قفيصات متعددة ، مع تقليل أعداد الحيوانات وتقليل التباين بين الحيوانات. تم نشر الدراسات الأولى التي أدخلت هذه التكنولوجيا إلى مجال AAV منذ ما يقرب من عقد من الزمان. قام مختبر ناكاي 12 بوضع علامة على 191 طفرا مزدوجا من الألانين تغطي الأحماض الأمينية من 356 إلى 736 على VP1 من AAV9 مع زوج من الرموز الشريطية المكونة من12 نيوكليوتيد. باستخدام NGS ، تم فحص المكتبة في الجسم الحي لربط الجالاكتوز وخصائص أخرى12. حدد مارسيك وزملاؤه التوزيع الحيوي لمتغيرات AAV باستخدام تحليل مزدوج الشريط 1 بعد عام13. قارنت دراسة حديثة أجريت على الرئيسيات غير البشرية التوزيع الحيوي في الجهاز العصبي المركزي ل 29 قفيصة باستخدام طرق مختلفة للتوصيل23. نشر مختبرنا مؤخرا شاشات مكتبة AAV ذات الرموز الشريطية ل 183 متغيرا تضمنت AAVs طبيعية وهندسية. أدت هذه الشاشات على مستوى الحمض النووي والحمض النووي الريبي إلى تحديد متغير AAV عالي الانتحاءالعضلي 24 في الفئران بالإضافة إلى الآخرين الذين يعرضون خصوصية عالية من نوع الخلية في دماغالفأر 25.
هنا ، نصف المنهجية المستخدمة في هذا العمل ونتوسع فيها لتشمل فحص مكتبات عرض الببتيد AAV. ويشمل ذلك إنشاء مكتبات عرض الببتيد AAV2 ، وطريقة PCR الرقمية للقطرات (dd-PCR) للقياس الكمي ، وأخيرا خط أنابيب NGS لتحليل متغيرات AAV ، استنادا جزئيا إلى عمل Weinmann وزملائه24. أخيرا ، يتم تقديم وصف لإنشاء مكتبات AAV المشفرة بالباركود وخط أنابيب NGS المستخدم في نفس المنشور.
Protocol
1. AAV2 عشوائي 7-مير عرض الببتيد إعداد مكتبة
ملاحظة: لإعداد مكتبة عرض الببتيد العشوائي AAV2 ، قم بتجميع قليل النيوكليوتيدات المتدهورة كحمض نووي أحادي الشريط ، وقم بتحويله إلى حمض نووي مزدوج تقطعت به السبل ، وهضمه ، وربطه بالبلازميد المستقبلة ، والكهرباء الكهربائية.
- تصميم قليل النيوكليوتيدات المتدهورة
- اطلب قليل النيوكليوتيدات المتحلل وتجنب تحيز الكودون. في قليل النوكليوتيد 5' CAGTCGGCCAG AG W GGC (X01) 7 GCCCAGGCGGCTGACGAG 3 '، X01 يتوافق مع 20 كودونا ، كل منها يشفر واحدا من 20 حمضا أمينيا. يمكن أن يكون W A أو T ، مما ينتج عنه الكودونات AGA أو AGT ، والتي تشفر الأحماض الأمينية أرجينين (R) أو سيرين (S).
- اطلب التمهيدي للتضخيم: 5 'CTCGTCAGCCGCCTGG 3' (انظر الشكل 1 للحصول على التفاصيل). ينتج عن هذا إدراج البروتين التالي: R / S GX 7. يتم حساب التنوع النظري على النحو التالي: 1 × 2 × 207 = 2.56 × 109 متغيرات فريدة.
ملاحظة: تجدر الإشارة إلى أن هذا التنوع قد يكون مقيدا بكفاءة التحويل.
- توليف الخيط الثاني
- إعادة تعليق كل من oligonucleotides (oligonucleotides المتدهورة والتمهيدي التضخيم) إلى تركيز نهائي 100 μM مع العازلة TE.
- بالنسبة لتفاعل تفاعل البوليميراز المتسلسل ، قم بإعداد تفاعل 50 ميكرولتر مع 1 ميكرولتر من كل برايمر ، و 10 ميكرولتر من المخزن المؤقت ، و 1.5 ميكرولتر من DMSO ، و 0.5 ميكرولتر من dNTPs (10 mM) ، و 0.5 ميكرولتر من بوليميراز البدء الساخن عالي الدقة II ، و 35.5 ميكرولتر من الماء الخالي من النيوكلياز.
- انقل التفاعل إلى جهاز تدوير حراري وقم بتشغيل خطوة ما قبل الحضانة لمدة 10 ثوان عند 98 درجة مئوية ، تليها ثلاث دورات من 10 ثوان عند 98 درجة مئوية ، و 30 ثانية عند 59 درجة مئوية ، و 10 ثوان عند 72 درجة مئوية ، ثم 5 دقائق عند 72 درجة مئوية وخطوة تبريد نهائية.
- قم بتنقية التفاعل باستخدام مجموعة إزالة النوكليوتيدات وقم بإزالته في 100 ميكرولتر من الماء الخالي من النيوكلياز.
- تأكيد كفاءة تخليق الشريط الثاني عن طريق التحليل على محلل حيوي (انظر الشكل 2). قم بتحليل حجم ونقاء الملحق المزدوج الذي تقطعت به السبل عن طريق تحميل 1 ميكرولتر من التفاعل مع شريحة الموائع الدقيقة من مجموعة كواشف DNA 1000 وفقا لتعليمات الشركة المصنعة. تم تحسين هذه المجموعة لقياس حجم وتركيز شظايا الحمض النووي المزدوجة التي تقطعت بها السبل من 25-1000 بت في الثانية.
- إعادة تعليق كل من oligonucleotides (oligonucleotides المتدهورة والتمهيدي التضخيم) إلى تركيز نهائي 100 μM مع العازلة TE.
- هضم إدراج وناقلات البلازميد
- هضم 85 ميكرولتر من الملحق المنقى مع 10 ميكرولتر من المخزن المؤقت 10x و 5 ميكرولتر من إنزيم BglI في حجم تفاعل نهائي 100 ميكرولتر (انظر الشكل 1 للحصول على التفاصيل). احتضان في 37 درجة مئوية بين عشية وضحاها. قم بالتنقية باستخدام مجموعة إزالة النوكليوتيدات ، وقم باستخلاص 50 ميكرولتر من الماء الخالي من النيوكلياز ، وقم بالقياس الكمي باستخدام النوع "Oligo DNA" في مقياس الطيف الضوئي.
- هضم 10 ميكروغرام من بلازميد AAV (pRep2Cap2_PIS) 26 (الجينوم الفيروسي المحاط ب ITR) مع 20 ميكرولتر من المخزن المؤقت 10x و 10 ميكرولتر من إنزيم SfiI في حجم تفاعل نهائي 200 ميكرولتر (انظر الشكل 1 للحصول على التفاصيل). احتضان في 50 درجة مئوية بين عشية وضحاها. قم بتنقية الناقل على جل أغاروز 1٪ باستخدام مجموعة استخراج الجل متبوعة بخطوة تنقية إضافية باستخدام مجموعة تنقية الحمض النووي. تحديد التركيز في مقياس الطيف الضوئي.
- ربط الإدراج بالمتجه
- Ligate 955 نانوغرام من ناقل البلازميد مع 45 نانوغرام من الإدراج مع 2 ميكرولتر من المخزن المؤقت و 2 ميكرولتر من ligase في تفاعل ربط 20 ميكرولتر. احتضان في 16 درجة مئوية بين عشية وضحاها، تليها 10 دقائق في 70 درجة مئوية لتسخين لتعطيل الرباط.
- التحويل وحساب التعقيد وإعداد مكتبة البلازميد
- قم بتنقية التفاعل باستخدام مجموعة تنقية الحمض النووي باتباع تعليمات الشركة المصنعة. قم بسحب التفاعل في حوالي 80٪ من حجم البدء للمياه الخالية من النيوكلياز وتخزينها على الجليد للتحول اللاحق.
- تحويل الخلايا ذات الكفاءة الكهربائية: قم بإذابة قارورة واحدة من الخلايا المختصة بالكهرباء على الجليد لمدة 10 دقائق. ثم أضف 1-2 ميكرولتر من تفاعل الربط المنقى إلى 30 ميكرولتر (قارورة واحدة) من الخلايا المختصة كهربائيا واخلطها عن طريق النقر برفق. بعد ذلك ، ماصة بعناية خليط الخلية / الحمض النووي إلى كوفيت التثقيب الكهربائي المبرد مسبقا بفجوة 1 مم دون إدخال فقاعات هواء.
- بالكهرباء باستخدام الإعدادات التالية: 1800 فولت و 600 Ω و 10 ميكروفولت. في غضون 10 ثوان من نبضة التثقيب الكهربائي ، أضف 970 ميكرولتر من وسائط الاسترداد الدافئة مسبقا (المزودة بالخلايا المختصة بالكهرباء) إلى الكوفيت واخلطها عن طريق الماصة. أخيرا ، انقل الخلايا إلى أنبوب طرد مركزي صغير واحتضانها لمدة 1 ساعة عند 37 درجة مئوية عند 250 دورة في الدقيقة. لتحقيق التنوع المطلوب ، قم بإجراء 10-100 تفاعل ، وبعد الحضانة ، قم بتجميع جميع ردود الفعل في أنبوب واحد.
- احسب التنوع عن طريق تخفيف 10 ميكرولتر من التحولات المجمعة 10 أو 100 أو 1000 ضعف في PBS ونشر 100 ميكرولتر على ألواح أجار المغذيات التي تحتوي على المضاد الحيوي المناسب (75 مجم / مل من الأمبيسلين). احتضان ألواح الآجار طوال الليل عند 37 درجة مئوية ثم عد المستعمرات على ألواح الآجار.
- احسب التنوع النظري على النحو التالي:
التنوع الأقصى النظري = 10 × عامل التخفيف × عدد المستعمرات × عدد تفاعلات التثقيب الكهربائي.
ملاحظة: لتأكيد جودة المكتبة ، قم بتسلسل 20 مستعمرة على الأقل بواسطة تسلسل سانجر. يجب أن تحتوي معظم الحيوانات المستنسخة على ملحق ، ويجب أن تكون جميعها فريدة من نوعها. - تلقيح 400-1000 مل من وسط LB الذي يحتوي على المضاد الحيوي المناسب مع بقية التحولات المجمعة واحتضانها طوال الليل عند 37 درجة مئوية ، 180 دورة في الدقيقة.
- إعداد مكتبة البلازميد
- من المزرعة الليلية ، قم بإعداد مخزون الجلسرين (امزج كميات متساوية من الثقافة البكتيرية ومحلول الجلسرين بنسبة 50٪ في ماء خال من النيوكلياز وقم بتجميده عند -80 درجة مئوية) وقم بتنقية مكتبة البلازميد باستخدام مجموعة ماكسي البلازميد.
- إنتاج مكتبة AAV الفيروسية
- قم بإعداد المكتبة الفيروسية كما هو موضح سابقا27. قم بنقل مكتبة البلازميد (pRep2Cap2_PI ، إدراج الببتيد) مع بلازميد مساعد غدي إلى خلايا HEK293T باستخدام كاشف نقل مثل polyethylenimine (PEI).
- اجمع الخلايا بعد 3 أيام وأخضعها لثلاث دورات من ذوبان الجليد. تنقية المحللة الفيروسية باستخدام الطرد المركزي الفائق التدرج كلوريد السيزيوم ، تليها تبادل العازلة إلى PBS ، وأخيرا تركيز الجسيمات الفيروسية.
- معايرة متجه AAV باستخدام dd-PCR
- قم بتخفيف 2 ميكرولتر من مخزون ناقل AAV بشكل متسلسل في 198 ميكرولتر من الماء الخالي من النيوكلياز لإنتاج تخفيف نهائي 1:106 . تخلط جيدا في كل مرة باستخدام ماصة 200 ميكرولتر. إضافة عنصر تحكم واحد بدون قالب (NTC) كعنصر تحكم سالب.
ملاحظة: يمكن فحص تخفيفات إضافية أقل أو أعلى (1:105-1:107). - تحضير 20x مزيج مسبار التمهيدي. أضف 3.6 ميكرولتر من كل من البادئات 100 ميكرومتر (الأمامي والخلفي ، Rep2 ، و ITR) ، و 1 ميكرولتر لكل من مجسات 100 ميكرومتر dd-PCR (Rep2 و ITR) ، و 3.6 ميكرولتر من الماء الخالي من النيوكلياز إلى أنبوب طرد مركزي سعة 1.5 مل.
ملاحظة: يتم قياس مكتبة AAV باستخدام مجموعة مسبار أولي مستهدف بالجينات المحورة (Rep2) تم اكتشافه باستخدام مسبار يحمل علامة FAM ، ومجموعة مسبار أولي مستهدف بواسطة ITR تم اكتشافه باستخدام مسبار يحمل علامة HEX. - قم بإعداد تفاعل تفاعل البوليميراز المتسلسل 22 ميكرولتر عن طريق إضافة 5.5 ميكرولتر من العينة ، و 1.1 ميكرولتر من مزيج مسبار التمهيدي 20x ، و 11 ميكرولتر من المزيج الفائق dd-PCR للمجسات (بدون dUTP) ، و 4.4 ميكرولتر من الماء الخالي من النيوكلياز. ينتج عن هذا تركيزات 900 نانومتر و 250 نانومتر للبادئات والمسبار ، على التوالي.
- قم بتوليد القطرات باستخدام مولد قطرات ، وانقل التفاعل إلى لوحة 96 بئرا ، وضع اللوحة في دورة حرارية ، وقم بتشغيل خطوة تمسخ لمدة 10 دقائق عند 94 درجة مئوية ، تليها 40 دورة من 30 ثانية عند 94 درجة مئوية و 1 دقيقة عند 58 درجة مئوية. بعد ذلك ، قم بتعطيل البلمرة بالحرارة لمدة 10 دقائق عند 98 درجة مئوية وإضافة خطوة تبريد نهائية. اقرأ ردود الفعل في قارئ القطيرات وانتقل إلى التحليل28.
- افتح ملف لوحة dd-PCR المحفوظ باستخدام برنامج التحليل. استخدم أداة العتبة في علامة التبويب 1D Amplitude (السعة الفلورية مقابل رقم الحدث) لفصل القطرات السالبة والموجبة لكل قناة ، باستخدام NTC كدليل ، وتصدير البيانات إلى ملف csv.
- لحساب تركيز المتجه ، احسب أولا عامل التصحيح CF باستخدام الصيغة:
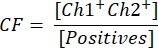
يحدد التليف الكيسي نسبة القطرات الموجبة لجين التحوير [الإيجابيات] الإيجابية لكل من جين التحوير و ITR [Ch1+ Ch2+] ، لضمان الكشف عن جسيمات النواقل الوظيفية. يمكن الآن حساب تركيز المتجه النهائي c باستخدام المعادلة التالية: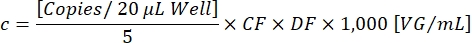
DF هو عامل التخفيف (1:105-1:107 كما هو محدد سابقا). تتوافق النسخ لكل تفاعل 20 ميكرولتر / بئر مع 5 ميكرولتر من العينة المخففة. العامل 1,000 يصحح المقياس إلى VG / mL (الجينوم الفيروسي / مل). يوضح الجدول 1 والشكل 3 نتيجة المعايرة بالتحليل الحجمي النموذجية.
- قم بتخفيف 2 ميكرولتر من مخزون ناقل AAV بشكل متسلسل في 198 ميكرولتر من الماء الخالي من النيوكلياز لإنتاج تخفيف نهائي 1:106 . تخلط جيدا في كل مرة باستخدام ماصة 200 ميكرولتر. إضافة عنصر تحكم واحد بدون قالب (NTC) كعنصر تحكم سالب.
- تحليل المكتبة الفيروسية AAV بواسطة NGS
- قم بتضخيم جزء إدخال الببتيد 96 نيوكليوتيد عن طريق إعداد تفاعل تفاعل البوليميراز المتسلسل 20 ميكرولتر باستخدام مجموعة بوليميراز قراءة التدقيق (2x ؛ انظر الشكل 4). أضف 1 ميكرولتر من مخزون AAV يحتوي على 1 × 108 vg ، 0.5 ميكرولتر من كل من 100 ميكرومتر التمهيدي (NGS_forward و NGS_reverse) ، و 10 ميكرولتر من مزيج الإنزيم إلى التفاعل. اضبط الحجم النهائي على 20 ميكرولتر بالماء الخالي من النيوكلياز.
- انقل التفاعل إلى جهاز تدوير حراري وقم بتشغيل خطوة تمسخ لمدة 3 دقائق عند 98 درجة مئوية ، تليها 30-35 دورة من 10 ثوان عند 98 درجة مئوية ، و 10 ثوان عند 59 درجة مئوية ، و 20 ثانية عند 72 درجة مئوية ، تليها 5 دقائق عند 72 درجة مئوية وخطوة تبريد نهائية.
- تنقية العينات باستخدام مجموعة تنقية PCR. حدد التركيز في مقياس الطيف الضوئي وقم بتشغيل هلام الأغاروز بنسبة 3٪ للتحقق من النقاء وحجم الشظية.
- قم بمعالجة شظايا تفاعل البوليميراز المتسلسل باستخدام نظام المكتبة لمجموعة العينات منخفضة التعقيد وفقا لتعليمات الشركة المصنعة لإعداد مكتبة NGS. قم بإجراء تفاعل الإصلاح النهائي باستخدام 30 نانوغرام من جزء تفاعل البوليميراز المتسلسل ، متبوعا بربط المحول وتضخيم تفاعل البوليميراز المتسلسل لمدة 10 دورات. استخدم مجموعة تنقية PCR لتنقية التفاعلات.
- قم بمعالجة المنتجات النهائية على محلل حيوي للتحقق من الحجم والنقاء ، باستخدام مجموعة كواشف الحمض النووي وفقا لتعليمات الشركة المصنعة.
- حدد كمية الأمبليونات باستخدام مقياس الفلور وقم بتجميعها. قم بتحديد مكتبة NGS المجمعة النهائية مرة أخرى على مقياس الفلور (وفقا لتعليمات الشركة المصنعة) وتحقق من الجودة على محلل حيوي.
- قم بتسلسل مكتبات NGS في وضع أحادي الطرف (SE) ، باستخدام مجموعة مخرجات عالية 75 دورة ، بطول قراءة 84 وفهرس 1 من 8.
ملاحظة: تم إجراء تسلسل الأمثلة في هذه المقالة في منشأة GeneCore التابعة لشركة EMBL Heidelberg (http://www.genecore.embl.de/). - تحليل بيانات تسلسل NGS باستخدام Python 3 و biopython. يمكن العثور على الملفات في https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 (بدلا من ذلك في https://doi.org/10.5281/zenodo.7032215). يتكون تحليل NGS من خطوتين.
- في الخطوة الأولى ، ابحث في ملفات التسلسل عن التسلسلات التي تفي بمعايير معينة (وجود تسلسلات التعرف المحيطة بموقع الإدراج) (انظر الشكل 4 ، الخطوة 1.9.8.5.). يتم ذلك باستخدام برنامج نصي (Script # 1) وملف تكوين يوفر المعلومات المطلوبة. بمجرد تحديد التسلسل الصحيح ، يقوم البرنامج باستخراج وتخزين التسلسل في ملف الإخراج ، وهو ملف txt يحمل نفس اسم ملف التسلسل.
- الخطوة الثانية هي تحليل ملفات الإخراج. تبدأ التسلسلات في المكتبة بأي من النيوكليوتيدات الستة (AGWggc ، W = A / T) في إدراج الأحماض الأمينية التسعة. بناء على تسلسل البداية هذا ، يتم ترجمة الببتيد. يؤدي هذا إلى إنشاء ملفات الإخراج التي تحتوي على متغيرات الببتيد (PVs).
- قم بإعداد مجلدين: البرنامج النصي والبيانات. إلى مجلد البيانات ، انسخ ملفات gzip المضغوطة الناتجة عن التسلسل. إلى مجلد البرنامج النصي ، انسخ الملفات التالية ، ملف Python: Script#1_DetectionExtraction_JoVE_Py3.py; ملف بايثون: البرنامج النصي # 2_PV_extraction_and_ranking_Py3.py ؛ ملف التكوين: Barcode_Script_JoVE.conf; وملف جدول البحث (LUT): Zuordnung.txt.
- قبل تشغيل البرامج النصية، قم بتحرير الملفات التالية في مجلد البرنامج النصي. افتح ملف "Zuordnung.txt" وأضف في عمودين مفصولين بعلامات جدولة ، أسماء ملفات gzip (العمود 1) ، والاسم النهائي المطلوب (العمود 2 ؛ قيم مفصولة بعلامات جدولة).
ملاحظة: تم العثور على نماذج ملفات txt في المجلد GitHub "PV_analysis_script". يتم إعداد الملفات المتوفرة في مجلد GitHub لتحليل ثلاثة بيانات نموذجية من المكتبة أعلاه: xaa.txt.gz و xab.txt.gz و xac.txt.gz. يتم توفير ملفات الإخراج أيضا. - تغيير المتغيرات التالية في ملف التكوين "Barcode_Script_JoVE.conf":
my_dir = "~/البيانات/"
filename_sample_file = "~/Script/Zuordnung.txt"
المتغيرات الخاصة بالتسلسل: BCV_size = 27 ، BCVاليسار = TCCAGGGCCAG ، BCVاليمين = GCCCAGG ، BCVloc = 30 ،هامش BCV = 8 ، BCVleft_revcomp = GCCGCCTGGGC ، BCVright_revcomp = CTGGCCC ، و BCVloc_revcomp = 41 (انظر الشكل 4 للحصول على التفاصيل). - استخدم الأمر التالي لاستدعاء الكشف عن تسلسل المتغير واستخراجه:
>python3 ~ / البرنامج النصي # 1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
ملاحظة: الإخراج عبارة عن ملفات txt مع تسلسلات الحمض النووي المستخرجة وأعداد قراءاتها. يحتوي رأس هذا الملف على بيانات إحصائية (أي العدد الإجمالي للقراءات والقراءات المستخرجة). يتم نقل هذه البيانات إلى الملفات التالية. بيانات txt هذه هي ملفات الإدخال الخاصة ب Script # 2 ، حيث يتم ترجمة تسلسلات الحمض النووي وترتيبها وتحليلها. - قم بإجراء استخراج وتحليل PV باستخدام الأمر التالي:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - تحليل ملفات إخراج النص من البرنامج النصي # 2. تتم تسمية ملفات إخراج Script#2 باستخدام العمود الثاني من جدول البحث في "Zuordnung.txt" مع امتدادات تستند إلى نوع التحليل.
ملاحظة: تأكد من أن ملفات الإخراج الثلاثة تحتوي على بيانات إحصائية في الصفوف الأولى ("# من قراءات PV صالحة" و "# من قراءات PV غير صالحة" و "# من قراءات PV الفريدة") ، عمود أول يحتوي على فهرس كل تسلسل DNA من ملفات txt الإدخال (إخراج البرنامج النصي # 1) ، والأعمدة التالية: (1) "... analyzed_all.csv": "عينة:" (تسلسل الحمض النووي) ، "#" (عدد القراءات) ، "Frw أو Rev" (قراءة أمامية أو عكسية) ، و "PVs" (تسلسل الببتيد المترجم). تحتوي التسلسلات غير الصالحة على "NA" و "غير صالح" في العمودين الأخيرين. (2) "... analyzed_validSeq.csv": نفس الملف السابق ، تمت تصفيته للتسلسلات الصالحة. (3) "... analyzed_PV.csv": "PVs" (تسلسل الببتيد المترجم) و "#" (عدد القراءات) و "العد" (يتم دمج عدد frw و rev في الملفات السابقة ويتم إعطاء العدد 1 أو 2). - تصور ملفات الإخراج باستخدام البرامج المتاحة بناء على احتياجات المستخدم.
2. اختيار مكتبة عرض الببتيد العشوائي AAV2 7 مير
- استخدم مكتبة AAV بعد القياس الكمي ومراقبة الجودة (القسم 1) للتطور الموجه في نموذج الاختيار للاختيار المتكرر للمرشحين ذوي الخصائص المطلوبة (انظر الشكل 5)16،18،21.
ملاحظة: يتم استخدام هؤلاء المرشحين بعد ذلك لإنشاء مكتبة مشفرة كما هو موضح أدناه في القسم 3.
3. إعداد وتحليل مكتبة قفيصة AAV المشفرة بالباركود
ملاحظة: بعد تحديد مجموعة من قفيصات AAV التي يحتمل أن تكون محددة وفعالة في شاشة عرض الببتيد ، تحقق من وظائف تسلسلات الببتيد المحددة ومقارنتها بمجموعة من متغيرات قفيصة AAV المرجعية شائعة الاستخدام أو الموصوفة جيدا. للقيام بذلك ، يتم إدراج تسلسل capsid في بناء مساعد Rep / Cap بدون لوائح الاتصالات الدولية.
- إنتاج مكتبة AAV ذات الرمز الشريطي
- قم بإجراء إنتاج AAV المؤتلف لكل متغير قفيصة باستخدام نظام ثلاثي البلازميد ، كما هو موضح سابقا24.
ملاحظة: للتمييز بين متغيرات القفيصة المختلفة ، يحتوي بلازميد التحوير المراسل المحاط ب ITR على رمز شريطي فريد من 15 نيوكليوتيد في الطول. يقع الرمز الشريطي في 3 'UTR (المنطقة غير المترجمة) بين البروتين الفلوري الأصفر المحسن (EYFP) وإشارة polyA (انظر الشكل 6A). يتم تحفيز تعبير EYFP بواسطة مروج قوي للفيروس المضخم للخلايا (CMV) في كل مكان يوفر مستويات كافية من نسخ الحمض النووي الريبي. - تصميم الرموز الشريطية من 15 نيوكليوتيدات في الطول مع البوليمرات المتجانسة من أقل من ثلاثة نيوكليوتيدات ، ومحتوى GC من <65 ٪ 29 ، ومسافة هامينغ أكبر من أربعة نيوكليوتيدات24.
- أنتج كل قفيصة على حدة مع بلازميد جين محوري يحمل رمزا شريطيا فريدا. بهذه الطريقة ، يتم تمييز كل متغير قفيصة برمز شريطي مميز يتيح تتبعه المحدد (انظر الشكل 6 ب).
- قم بإجراء إنتاج AAV المؤتلف لكل متغير قفيصة باستخدام نظام ثلاثي البلازميد ، كما هو موضح سابقا24.
- معايرة متجه AAV باستخدام dd-PCR
- قم بإجراء معايرة AAV كما هو موضح سابقا في القسم 1.8 ، عن طريق استبدال زوج التمهيدي Rep2 بزوج التمهيدي YFP.
- تحديد كمية إنتاجات AAV الفردية وتجميع كميات متساوية من كل إنتاج لإنشاء مكتبة الباركود النهائية.
- حدد المكتبة النهائية مرة أخرى للتحقق من التركيز النهائي والجودة (انظر الشكل 7).
- مكتبة AAV المشفرة بالباركود في تطبيق vivo
- قم بتطبيق مكتبة AAV المشفرة بشكل منهجي على النظام النموذجي المختار (على سبيل المثال بشكل منهجي في الفئران24).
- اجمع الأنسجة داخل وخارج الهدف (مثل الكبد والرئة والقلب والحجاب الحاجز والعضلات الملساء والاثني عشر والبنكرياس والقولون والعضلة ذات الرأسين والمبيضين والمعدة والأذن الداخلية والكلى والشريان الأورطي البطني والشريان الأورطي الصدري والدماغ والدهون البنية والبيضاء والطحال) أو أنواع الخلايا بناء على التجربة. قم بتجميدها عند -80 درجة مئوية ، واستخرج الحمض النووي / الحمض النووي الريبي ، وقم بتطبيق التحليل الكمي NGS ، كما هو موضح في القسم التالي.
- استخراج الحمض النووي / الحمض النووي الريبي
- استخرج الحمض النووي والحمض النووي الريبي من الأنسجة ذات الأهمية باستخدام مجموعة DNA / RNA Mini.
- ضع قطعة صغيرة من الأنسجة المهمة (1 مم3 ، حوالي 5 ملغ) في أنبوب تفاعل 2 مل.
- أضف 350 ميكرولتر من محلول التحلل الممزوج ب β-mercaptoethanol (1٪) وحبيبات فولاذية 5 مم إلى الأنسجة (تعامل مع العينات باستخدام β-mercaptoethanol تحت غطاء الدخان).
- تجانس الأنسجة في الأنسجةمحلل لمدة 45 ثانية عند 40 هرتز.
- أضف 10 ميكرولتر من البروتيناز K (10 مجم / مل) واحتضانه لمدة 15 دقيقة عند 55 درجة مئوية أثناء رجه عند 400 دورة في الدقيقة.
- أجهزة الطرد المركزي عند 20000 × جم لمدة 3 دقائق في درجة حرارة الغرفة ، وجمع المادة الطافية ، والمضي قدما في بروتوكول الشركة المصنعة لمجموعة DNA / RNA.
- قسم خطوة الغسيل إلى خطوتين مع 350 ميكرولتر من المخزن المؤقت للغسيل في كل خطوة. بين خطوات الغسيل هذه ، اهضم الحمض النووي المتبقي على العمود باستخدام DNase I. أضف 80 ميكرولتر من محلول DNase I ، المحضر وفقا لتعليمات الشركة المصنعة ، على العمود واحتضانه في درجة حرارة الغرفة لمدة 15 دقيقة.
- قم باستخلاص الحمض النووي الريبي / الحمض النووي من العمود بالماء الخالي من النيوكلياز. قم بتخزين الحمض النووي الريبي المعزول عند -80 درجة مئوية والحمض النووي المعزول عند -20 درجة مئوية.
- تخليق الحمض النووي cDNA
- إخضاع عينات الحمض النووي الريبي لجولة أخرى من معالجة DNase I لمدة 15-30 دقيقة (للإزالة الكاملة للحمض النووي الملوث من عينات الحمض النووي الريبي) قبل تفاعل النسخ العكسي. أضف 1 ميكرولتر من محلول DNase I ، و 4 ميكرولتر من المخزن المؤقت (مزود مع المجموعة) ، والماء الخالي من النيوكلياز إلى الحجم النهائي من 40 ميكرولتر إلى 212 نانوغرام من الحمض النووي الريبي. احتضان لمدة 30 دقيقة في درجة حرارة الغرفة والحرارة تعطيل عند 70 درجة مئوية لمدة 10 دقائق.
- توليف cDNA ، باستخدام 150 نانوغرام من الحمض النووي الريبي باستخدام مجموعة وفقا لتعليمات الشركة الصانعة. قم بتضمين عناصر التحكم بدون النسخ العكسي ، لضمان عدم وجود الحمض النووي الفيروسي الملوث من العينة. يتم تخزين cDNA عند -20 درجة مئوية.
ملاحظة: يمكن أن تختلف كمية الحمض النووي الريبي المدخل للنسخ العكسي الأمثل اعتمادا على نوع الأنسجة وكفاءة النقل المتوقعة في الأنسجة المعنية.
- تحليل المكتبة الفيروسية AAV (in-vivo) بواسطة NGS
- لتحقيق عمق تسلسل عالي بتكلفة منخفضة ، قم بإجراء NGS عبر تسلسل Illumina كما هو موضح سابقا (القسم 1.9). قم بتضخيم تسلسل الباركود ، ثم قم بربط محولات التسلسل بمكبر الصوت.
- نظرا لطول القراءة القصيرة وربط محولات التسلسل على جانبي amplicon ، عند التصميم ، تحقق من أن amplicon صغير بما يكفي لضمان وجود تسلسل الباركود داخل قراءة NGS. لتسلسل الرموز الشريطية داخل الجينومات الفيروسية والنصوص الفيروسية ، تم تصميم amplicon PCR ليكون طوله 113 نقطة أساس (انظر الشكل 8).
- قم بتضخيم المنطقة المشفرة باستخدام البادئات BC-seq للأمام و BC-seq للخلف. تحضير تفاعل تفاعل البوليميراز المتسلسل التالي: 0.5 ميكرولتر من بوليميراز الحمض النووي عالي الدقة ، 10 ميكرولتر من 5x buffer ، 0.25 ميكرولتر من كل 100 ميكرومتر التمهيدي (BC-seq fw / BC-seq rv) ، و 1 ميكرولتر من 10 mM dNTPs. استخدم 25 نانوغرام من cDNA أو DNA / التفاعل كقالب واضبط الحجم النهائي على 50 ميكرولتر بالماء الخالي من النيوكلياز.
- قم بإعداد المزيج الرئيسي PCR تحت غطاء PCR نظيف لتجنب التلوث. استخدم ظروف ركوب الدراجات التالية: 30 ثانية عند 98 درجة مئوية ، تليها 40 دورة عند 98 درجة مئوية لمدة 10 ثوان و 72 درجة مئوية لمدة 20 ثانية ، وخطوة أخيرة لمدة 5 دقائق عند 72 درجة مئوية.
- قم بتضمين عناصر تحكم تفاعل البوليميراز المتسلسل لتأكيد عدم وجود حمض نووي ملوث في المزيج الرئيسي لتفاعل البوليميراز المتسلسل. بالنسبة لعينات cDNA ، قم بتضمين عناصر التحكم بدون النسخ العكسي. أخيرا ، قم بتضمين عينة مع مكتبة إدخال AAV. سيتم استخدام هذه المعلومات لإنشاء ملف Normalization_Variant.txt المستخدم في التحليل.
- تحقق من حجم جزء PCR لكل عينة عن طريق الرحلان الكهربائي للهلام قبل تنقية PCR. يتم تحقيق هذا الأخير إما باستخدام الخرز المغناطيسي المتاح تجاريا أو أنظمة تنقية الحمض النووي القائمة على العمود (انظر جدول المواد).
- قم بإعداد مكتبة NGS باستخدام نظام المكتبة للعينات منخفضة التعقيد وفقا لتعليمات الشركة المصنعة ، كما هو موضح سابقا في القسم 1.9.
- حدد تركيز الحمض النووي عبر مجموعة dsDNA HS وقم بتحليل جودة المكتبة كما هو موضح سابقا (القسم 1.9.6) ، متبوعا بالتجميع. تحديد المكتبة المجمعة على مقياس الفلور وتقييم الجودة على محلل حيوي.
- إجراء تسلسل NGS كما تمت مناقشته في القسم 1.9.7.
- حدد بواسطة qPCR رقم نسخة جين التحوير (الجينومات الفيروسية) وجين التدبير المنزلي لتقييم توزيع المكتبة المجمعة بين الأنسجة أو الأعضاء على الحمض النووي.
- قم بإعداد تفاعل qPCR سعة 30 ميكرولتر على النحو التالي ، لتحديد عدد نسخ EYFP (جين التحوير) و GAPDH (غليسيرالدهيد 3-فوسفات ديهيدروجيناز ، جين التدبير المنزلي):
- قم بإعداد مزيج تمهيدي / مسبار 60x ل EYFP (1.5 ميكرومتر YFP_fw ، 1.5 ميكرومتر YFP_rv ، و 0.6 ميكرومتر YFP_probe ؛ انظر جدول المواد). استخدم مزيج التمهيدي / المسبار GAPDH (انظر جدول المواد) لتحديد رقم نسخة جين مدبرة المنزل. إعداد رد الفعل على الجليد.
- قم بإعداد مزيج رئيسي PCR (15 ميكرولتر ، انظر جدول المواد) وأضف 60x مزيج التمهيدي / المسبار (0.5 ميكرولتر) لجميع العينات والمعايير (لحساب أرقام النسخ للمعايير ، استخدم الرابط التالي: http://cels.uri.edu/gsc/cndna.html). إعداد رد الفعل على الجليد.
- انقل 15.5 ميكرولتر من المزيج الرئيسي إلى صفيحة 96 بئرا وأضف 14.5 ميكرولتر من العينة (75 نانوغرام من إجمالي تركيز الحمض النووي) أو قياسي إلى البئر المعني. أغلق اللوحة المكونة من 96 بئرا بورق القصدير والدوامة والدوران لفترة وجيزة.
- نقل 10 ميكرولتر من كل عينة إلى لوحة 384 بئر في نسخ. أغلق اللوحة بورق القصدير وقم بتدويرها عند 800 × جم لمدة 5 دقائق عند 4 درجات مئوية.
- احتضان مزيج التفاعل في جهاز تدوير حراري باستخدام درجة حرارة أولية تبلغ 50 درجة مئوية لمدة 2 دقيقة ، تليها خطوة تنشيط أولية مدتها 10 دقائق عند 95 درجة مئوية. قم بإجراء 40 دورة من التمسخ عند 95 درجة مئوية لمدة 15 ثانية والتلدين / التمديد عند 60 درجة مئوية لمدة 1 دقيقة24.
- للحصول على عدد الجينومات ثنائية الصيغة الصبغية (dg) ، استخدم رقم نسخة GAPDH واقسم على اثنين. بعد ذلك ، خذ قيمة رقم نسخة EYFP واقسمها على عدد dg ، مما ينتج عنه جينومات متجهة لكل جينوم ثنائي الصيغة الصبغية (vg / dg). استخدم هذه القيمة لإنشاء ملف Normalization_Organ.txt للتحليل المعلوماتي الحيوي.
- قم بإجراء تحليل بيانات تسلسل NGS مثل Weinmann et al.24، باستخدام التعليمات البرمجية المخصصة في Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). يتضمن سير العمل الكشف عن تسلسلات الباركود التي تسترشد بالتسلسلات المرافقة وطولها وموقعها (Script # 1_BarcodeDetection.py) ، بالإضافة إلى تحليل إثراء الباركود وتوزيعه على مجموعة الأنسجة (Script # 2_BarcodeAnalysis.py).
- كشف الباركود وتعيينها لمتغيرات AAV. ضع بيانات التسلسل كملفات fastq مؤرشفة في دليل واحد (على سبيل المثال ، "Data_to_analyze"). يتم تضمين ملف بيانات التسلسل لمكتبة الإدخال في هذا الدليل ويستخدم فقط لحساب نسب القفيصة في مكتبة الإدخال.
- قبل تنفيذ البرنامج النصي ، قم بإنشاء ملفين نصيين محددين بعلامات جدولة: ملف متغيرات capsid (انظر مثال الملف "Variants.txt") مع تسلسلات الباركود المخصصة لأسماء متغيرات AAV capsid ، وملف التلوث (انظر "التلوث.txt") مع تسلسلات الباركود التي تأتي من التلوث المحتمل (الرموز الشريطية الأخرى المتوفرة في المختبر ، مما يساهم في التلوث).
- أخيرا ، قم بتحرير ملف التكوين "Barcode_Script.conf" لتضمين المعلومات التالية: المسار إلى المجلد الذي يحتوي على بيانات التسلسل (على سبيل المثال ، "Data_to_analyze") ، وتسلسل المناطق الجانبية للرموز الشريطية ، وموضعها ، وحجم النافذة لاكتشاف الباركود (على غرار 1.9.8.5 ، انظر الشكل 8).
- استخدم الأمر التالي للاتصال باكتشاف الرمز الشريطي مع المسارات المتوفرة إلى Script#1_BarcodeDetection.py وملفات التكوين:
>python3 ~ / البرنامج النصي # 1_BarcodeDetection.py ~/Barcode_Script.conf
ملاحظة: إخراج تنفيذ Script # 1_BarcodeDetection.py هو ملفات نصية مع عدد القراءة لكل متغير capsid بالإضافة إلى إجمالي عدد القراءات المستردة من البيانات الأولية. - تقييم توزيع قفيصات AAV المشفرة بالباركود بين الأنسجة أو الأعضاء ، عن طريق تنفيذ Script # 2_BarcodeAnalysis.py مع ملفات txt التالية:
- في ملف "Zuordnung.txt" ، قم بتعيين الاسم لكل ملف txt تم الحصول عليه من تشغيل اكتشاف الباركود إلى اسم الأنسجة / الأعضاء: أسماء ملفات txt في العمود الأول وأسماء الأنسجة / الأعضاء المقابلة في التعيين المحدد بعلامات جدولة.
ملاحظة: على سبيل المثال، تحقق في المجلد "مثال" (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). من الجدير بالذكر أن اسم النسيج / العضو يمكن أن يتضمن أحرف تحدد قياس cDNA أو gDNA ورقم النسخ المتماثل البيولوجي (M1 ، M2 ، إلخ). - قم بإنشاء ملف نصي "أعضاء.txt" مع قائمة أسماء الأجهزة المستهدفة وخارج الهدف ، والتي تتوافق مع الأسماء الواردة في ملف "Zuordnung.txt" (انظر مجلد "مثال": https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022).
- قم بإنشاء ملفات نصية محددة بعلامات جدولة "Normalization_Organ.txt" و "Normalization_Variant.txt" بقيم طبيعية لجميع متغيرات capsid وجميع الأعضاء / الأنسجة. في العمود الأول من ملف "Normalization_Organ.txt" ، اكتب الأسماء المعطاة لكل عضو (كما في ملف المهمة "Zuordnung.txt") وفي العمود الثاني قيم التطبيع للأنسجة المقابلة ، التي تم إنشاؤها في القسم 3.6.11.
- املأ العمود الأول من ملف "Normalization_Variant.txt" بقائمة أسماء capsid والعمود الثاني بالقيم المعيارية لأعداد القراءة لكل قفيصة في المكتبة المجمعة (يمكن حساب التسوية بناء على ملف إخراج txt لمكتبة الإدخال الناتجة عن البرنامج النصي الأول).
- قم بتحرير ملف التكوين عن طريق تحديد المسارات الكاملة لجميع الملفات الإضافية المذكورة أعلاه. تنفيذ البرنامج النصي # 2_BarcodeAnalysis.py على النحو التالي:
>python3 / البرنامج النصي # 2_BarcodeAnalysis.py ~/Barcode_Script.conf
ملاحظة: يقوم البرنامج النصي لتحليل الباركود بإخراج عدة ملفات: ملفات نصية ذات قيم تركيز نسبي (RC) لتوزيع القفيصة داخل أنسجة مختلفة بناء على خطوات التطبيع المتعددة الموضحة سابقا ، وملف جدول البيانات الذي يجمع بيانات الملف النصي في بيانات المصفوفة المدمجة. يمكن استخدام هذا الأخير للتحليل العنقودي والتصور. - تصور البيانات وإجراء تحليل عنقودي لبيانات المصفوفة من أجل التمييز بين خصائص القفيصة وتقييم أوجه التشابه بينها بناء على ملفات تعريف RC عبر الأنسجة. استخدم البرنامج النصي الإضافي PCA_heatmap_plot. R وضعت في المستودع:
> Rscript - الفانيليا ~ / PCA. R ~ / التركيز النسبي.xls
ملاحظة: يأخذ البرنامج النصي ملفات التركيز النسبي.xls كمدخلات ويولد مخططين من خريطة الحرارة الهرمية للكتلة وتحليل المكون الرئيسي (PCA). - لتعديل المؤامرات (محاور خريطة الحرارة ، المكونات الرئيسية ل PCA) أو معلمات png (اللون والحجم ووضع العلامات) ، افتح البرنامج النصي R واتبع الإرشادات الواردة في الأقسام التي تم التعليق عليها.
- في ملف "Zuordnung.txt" ، قم بتعيين الاسم لكل ملف txt تم الحصول عليه من تشغيل اكتشاف الباركود إلى اسم الأنسجة / الأعضاء: أسماء ملفات txt في العمود الأول وأسماء الأنسجة / الأعضاء المقابلة في التعيين المحدد بعلامات جدولة.
النتائج
إنشاء مكتبة عرض الببتيد AAV2. كخطوة أولى نحو اختيار AAVs الهندسية ، يتم وصف إنشاء مكتبة البلازميد. يتم إنتاج إدراج الببتيد باستخدام الاشعال المتحللة. إن تقليل اتحاد الكودونات في تلك من 64 إلى 20 له مزايا القضاء على كودونات التوقف وتسهيل تحليل NGS ، عن طريق تقليل تنوع المكتبة على الحمض ال?...
Discussion
في هذا البروتوكول ، يتم تحديد الخطوات اللازمة لعرض الببتيد هندسة قفيصة AAV ولفحص مكتبة AAV المشفرة بالباركود ، وكذلك للتحليل المعلوماتي الحيوي لتكوين المكتبة وأداء القفيصة. يركز هذا البروتوكول على الخطوات التي تسهل التحليل المعلوماتي الحيوي لهذه الأنواع من المكتبات ، لأن معظم مختبرات علم ?...
Disclosures
D.G. هو أحد مؤسسي شركة AaviGen GmbH. D.G. و K.R. مخترعون في طلب براءة اختراع معلق يتعلق بتوليد متغيرات قفيصة AAV المتهربة من المناعة. بقية المؤلفين ليس لديهم ما يكشفون عنه.
Acknowledgements
تقدر D.G. تقديرا كبيرا الدعم المقدم من مؤسسة الأبحاث الألمانية (DFG) من خلال مراكز البحوث التعاونية DFG SFB1129 (Projektnummer 240245660) و TRR179 (Projektnummer 272983813) ، وكذلك من قبل المركز الألماني لأبحاث العدوى (DZIF، BMBF; TTU-فيروس نقص المناعة البشرية 04.819).
Materials
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
References
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621 (2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756 (2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075 (2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041 (2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432 (2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819 (2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899 (2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272 (2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200 (2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved