Method Article
miRNA的目标使用的任务目标ID库的全基因组屏幕
摘要
目标ID图书馆是一个基于质粒,用于识别miRNA靶基因的克隆基因的全基因组的集合。在这里,我们展示它的使用和应用。
摘要
目标ID库的设计,以协助发现和鉴定的microRNA(miRNA)的目标。目标ID图书馆是一个基于质粒的全基因组cDNA文库中克隆到双选zeocin胸苷激酶(TKzeo)融合蛋白,3'UTR下游。第一轮的选择是稳定的转化,引进miRNA的利益,最后,选择的cDNA包含的miRNA的目标。确定选定的cDNA序列(目标ID图书馆的工作流程和细节,请参阅图1-3)。
人类转录,以确保覆盖面广,目标编号图书馆的cDNA产生寡-dT通过使用准备从多种人体组织和细胞系的总RNA池启动。造成cDNA的范围从0.5到4 KB,一个1.2 kb的平均规模,并克隆到的p3TKzeo双选质粒(质粒图谱见图4)。在李代表的目标基因brary Sigma-Aldrich公司的网页上可以找到。 ( 见表3),Illumina的测序结果表明,该库包含16922 21518在UCSC的RefGene独特的基因(79%),或14,000 10或更多的内容(66%)的基因。
研究方案
1。转染与目标识别库和稳定细胞系的选择
1。 zeocin杀曲线
zeocin用于选择稳定转染细胞。然而,zeocin过剩导致不受欢迎的表型反应,在大多数细胞类型。因此,必须执行KILL曲线分析,建立最低致死剂量。
- 板1.6×10 4成井的细胞在96孔板120μL媒体。
- 第二天,添加浓度的增加,从50微克/毫升1毫克/毫升,以适当的井zeocin。
- 检查的可行性,每2天。
- 更换媒体包含zeocin每3天。选择试剂的最低浓度,导致完整的细胞死亡所需的时间后,应使用该细胞类型和实验。我们的研究结果表明,500微克/毫升的zeocin是最佳的A549,HeLa细胞,MCF7细胞。
2。图书馆通过核转染和选择
- 选择一个单元格的行要么不表达或表达水平低的你感兴趣的miRNA。稳定表达目标ID库后,将在B节中介绍的miRNA实现的目标选择。
- 文化/扩大细胞。 2×10 7%,库转染的细胞,我们已取得优异的成绩。
- Trypsinize细胞> 80%汇合,2×10 7细胞转移至15毫升无菌螺钉平顶管。
- 在200 XG颗粒胰酶消化细胞5分钟。
- 取出介质的HBSS或1×PBS洗涤细胞沉淀。
- 200 XG离心5分钟,吸洗。
- 重复洗涤细胞沉淀的一步。
- 预温6孔板2毫升完全培养基,在37°C
- 每0.5毫升管加入转染对每个目标ID图书馆2微克(不超过10μL)。
- 在15毫升管重悬细胞,每2×10 6细胞的Amaxa核转解100μL(细胞特异性)以上。例如,添加10 Nucleofections 1毫升的试剂。
- 一次在一个反应,添加100μL细胞质粒2微克。混合用吸管。
- 将混合传输以Nucleofector试管。
- 插入Nucleofector仪器试管和运行优化方案适当的细胞株(高效率,多细胞活力的首选)。
- 填写预温中移液管。占用相同的吸管和转移到6孔板中的细胞。重复为每个核转,每口井之一。
- 返回到生长室过夜培养。
- 第二天,更换培养基,使细胞恢复为3-5天。
- 更换介质含有完整的中等zeocin适当的水平,从杀曲线分析确定。
- 莫zeocin选择nitor细胞(细胞死亡)。
- zeocin每2-3天更换培养基。
- 一旦合流,在6孔板,通道,游泳池和拓展更大的烧瓶的细胞。
- 细胞体外扩增的时间长度是用户和依赖细胞株,但强烈建议扩大zeocin耐药细胞冷冻库存,为今后筛选产生(〜2-3周;细胞系而定)。
2。染图书馆细胞miRNA的表达构建,选择稳定的细胞系,并选择miRNA靶
注:Zeocin选择不再需要或想要的。 zeocin中miRNA的表达和更昔洛韦(目标)选择的细胞接触,可能会导致miRNA靶基因的损失。
3。嘌呤霉素,G418筛选,和更昔洛韦杀曲线
- 执行杀野生型细胞更昔洛韦稳定表达目标ID库和嘌呤或G418的曲线细胞。
- 板1.6×10 4成井与96孔板120μL新鲜媒体的细胞。第二天从0.1到10微克/毫升,puromycin/G418或2至32μM更昔洛韦选定井。
- 检查的可行性,每2天。含有选择试剂,每3天更换介质。所需的时间后,导致完整的细胞死亡*选择试剂的最低浓度,应使用的细胞类型和实验。我们的研究结果表明,嘌呤0.25到1微克/毫升是最佳的A549细胞,HeLa细胞,MCF7细胞,0.3μg/ ml的G418和MCF7细胞8-16μM的更昔洛韦最佳的A549细胞,HeLa细胞,MCF7细胞。
*注意:随着更昔洛韦选择,可能会不完整的细胞死亡观察细胞生长缓慢或没有。观察了几天的治疗,以验证他们不积极分裂的细胞。如果是这样的情况,然后在中酚红仍然是红色的。这也可能是必要的执行基目标ID库LL曲线分析转染细胞,如果在发生的变化是观察。然而,这将需要的细胞类型的具体基础上确定。
4。通过核转染和目标选择的miRNA转染
- 成长/扩大目标ID图书馆细胞,达到2×10 7细胞,,并trypsinize细胞> 80%汇合时。
- 转让2×10 7细胞15毫升无菌螺丝突破在5分钟的200 XG管和沉淀。
- 取出介质的HBSS或1×PBS洗涤细胞沉淀。
- 200 XG离心5分钟,吸洗。
- 重复洗涤细胞沉淀的一步。
- 预温6孔板2毫升每口井的完全培养基,在37°C
- 新增2微克的miRNA表达质粒(不超过10μL)每0.5毫升每转管。 origene microRNA表达质粒(新霉素 - G已成功用于418选择)或pBABE-PURO(质粒1764自我克隆miRNA基因; Addgene)。对于后者,一方〜200 bp的miRNA的发夹,PCR克隆人类DNA。
- 在15毫升管重悬细胞,每2×10 6细胞的Amaxa核转解100μL(细胞特异性)以上。例如:对于10 Nucleofections,加入1毫升的试剂。
- 一次在一个反应,添加100μL细胞质粒2微克。混合用吸管。
- 将混合传输以Nucleofector试管。
- 插入Nucleofector仪器试管和运行优化方案适当的细胞株(高效率的多细胞活力的首选)。
- 填写预温中移液管。占用相同的吸管和转移到6孔板中的细胞。重复为每个核转,每口井之一。
- 返回到生长室过夜培养。 <李>替换介质,使细胞恢复为3-5天。
- 取代完整的杀曲线的miRNA的构造与转染前进行的测试确定的培养基含有嘌呤或G418的适当水平中等。
- 监测细胞嘌呤/ G418筛选(细胞死亡)。
- 嘌呤/ G418的每2-3天更换培养基。
- 一旦合流,在6孔板,通道,游泳池和拓展更大的烧瓶的细胞。
- 细胞扩张的时间长度是用户的依赖,但它强烈建议扩大嘌呤/ G418抗性细胞冷冻库存,为今后筛选产生(〜2-3周;细胞系而定)。
- 适当水平的更昔洛韦(GCV),和嘌呤/ G418的决心杀曲线的miRNA的构造与转染前进行的测试,更换介质。
- 监测选择的细胞(细胞快要枯萎G,miRNA的目标和TK-的ZEO击倒)。
- 扩大细胞GCV和嘌呤/ G418的存在。
- 准备选择在GCV的细胞基因组DNA。
- PCR扩增引物试剂盒(PCR扩增)插入。
- 标准测序或提交深度测序的PCR产物克隆到TOPOTA载体(Invitrogen公司)。
3。 PCR的扩增选定库插入和序列
注:此程序进行的PCR扩增库,幸存下来的更昔洛韦选择目标。
5。收集更昔洛韦选定的单元格准备染色体DNA,用1 GenElute哺乳动物的基因组DNA小量提取试剂盒(目录号G1N10)或同等学历。我们建议准备从母细胞株不包含目标ID库,用于与DNA相比,从目标选定在聚合酶链反应的细胞作为阴性对照DNA。
- 聚合酶链反应(上午)plify基因组DNA扩增引物和扩增引物2。已获得成功扩增的JumpStart REDTaq预拌反应预混 - 聚合酶链反应(目录号P0982)。可能是必要的,如果使用另一个聚合酶条件的优化。请参考表1样本PCR PCR循环条件设置和表2。
- 解决2-5μL的PCR产物在1%琼脂糖凝胶。预期的产品应该具有一些明显的DNA带的涂抹。比较来控制细胞基因组DNA的PCR产品,没有目标的ID库。
- 进行克隆和/或深层测序的PCR产物。如果没有观察扩增产物,或者是相同的DNA来控制,优化PCR扩增条件(即,引物浓度,退火温度和循环)。
6。克隆和测序
注:我们强烈建议克隆的PCR产物s,然后使用克隆扩增试剂盒提供的引物至少96执行标准测序。执行此程序已成功使用96过夜培养和质粒纯化系统。即使所需的深度测序,克隆和标准测序的初步结果可以用来作为质量检查,以确定是否是必要的额外费用的深度测序。
- 按照TA克隆试剂盒制造的克隆PCR扩增产物(以上)的协议。
- 改造为主管细菌细胞的克隆和选择抗生素含中等过夜。
- 孤立和生长在液体培养基中的单个菌落,含有适当的抗生素。
- 净化质粒DNA。 6.5。扩增引物进行测序反应。
- 识别靶基因与人类转录组(已知的成绩单)和人类基因组序列的BLAST对齐(新型TRanscripts)。
*克隆故障排除:如果一个从序列插入质粒序列,优化等克隆/排序条件:
- PCR扩增产品(插入)的质量和数量
- 连接反应
- 质粒制备(数量和质量)
- 测序反应条件
4。代表结果
图书馆屏幕的miR-373的目标
为了评估性能的任务目标ID图书馆中,miR-373的目标选择的MCF-7表达细胞库。 MCF-7选择,因为它表达很少或根本没有检测到miR-373的(数据未显示)。 miR-373的选择,其生物的兴趣。 miR-373的表达促进MCF-7在肿瘤侵袭和转移,通常是一个非转移性细胞株[2]。此外,鼠标的miR-373同源的小鼠胚胎干细胞中的miR-290簇涉及电池保养[3]的miR-373家族成员中,miR-372,促进成纤维细胞重编程诱导多能干细胞[4]。最后,我们用锌指核酸插入PGK启动子的miR-373表达建成AAVS1网站在MCF-7细胞,并产生潜在的miR-373的RNA微阵列分析(数据未显示)的目标清单。
目标ID图书馆MCF-7细胞的转染,稳定人口的细胞被选为和zeocin含中等扩增。由此产生的细胞(MCF-7细胞库)稳定转染与miR-373的表达结构,选择和更昔洛韦含中等,以丰富的细胞表达的miR-373的目标。我们观察阴性对照图书馆的MCF-7细胞的miR-373(无)没有分离,成为圆润如预期的死细胞。然而,他们没有停止生长,这是很容易发现,因为酚红含中没有变成橙黄色作为观察FOR细胞表达的miR-373( 图5)。细胞含有更昔洛韦的培养基中,扩大和侧翼目标ID cDNA文库插入和准备从幸存的细胞DNA引物的PCR目标序列分离。 Illumina的测序PCR产物。
如表4所示,我们得到的cDNA 17740719读取,这映射到11,076独特的基因。这些独特的基因,2898人被发现超过40条,因此被认为是可靠的命中。 13106469载体读取预计因为PCR引物,用于扩增的cDNA插入40个和300个碱基的插入部位远离。正因为如此,它预计将不同于较传统的基因材料的深度测序载体,导致大部分序列。百分之十四没有映射到任何载体或基因,但没有配合NCBI的非冗余核酸数据库(即,与NR命中),只有1%的人没有cDNA片段。
初步比较发现,10个目标ID库确定了独特的基因是先前发现miR-373的在TarBase的目标( 见表5)的名单也。这些10 miR-373的目标是先前发现的一种人工合成的miR-373的模拟[5]瞬时转染的HeLa细胞与RNA芯片。此外,我们发现这些相同的10个基因下调的MCF-7细胞表达miR-373的AAVS1网站(数据未显示)。因此,这10个是可能的miR-373的有效目标。正在进行的工作是表征的潜在目标名单,并尝试实验验证定量PCR,Western印迹和荧光素酶报告基因检测的选择命中。
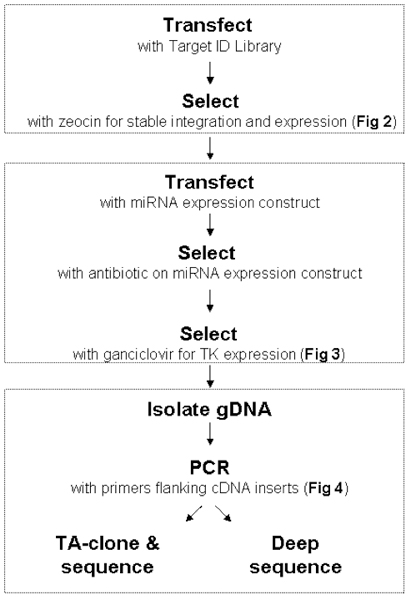
图1。目标ID库的工作流程。部分AC:参考过程中的部分。每一步都说明,并详细描述图2和3。

图2转染和Zeocin选择。 目标ID图书馆是一个质粒池(A)与人类基因插入后,将胸苷激酶zeocin融合蛋白(TKzeo图4)每到3'-UTR。细胞转染目标ID图书馆(二),并允许恢复3-5天。回收期(三)在此期间,可以整合到基因组的结构和表达编码的谈话全文(四)。恢复后,细胞暴露zeocin(五)。稳定的综合目标ID结构TKzeo融合蛋白表达细胞生存zeocin选择(F)的。转染细胞死亡(G)的。此外,任何细胞含有一个结构,是一种内源性的miRNA的目标,或任何超视距呃目标ID结构会死(高)TKzeo表达的细胞,抑制因子表达。
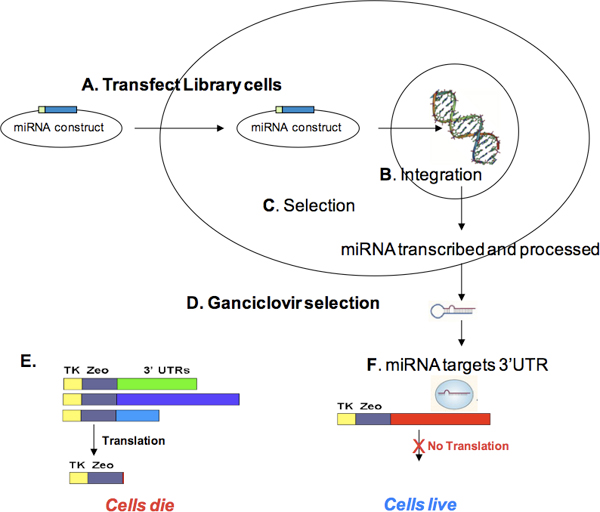
图3 miRNA的转染和更昔洛韦的选择。包含目标ID图书馆(即选定zeocin-细胞)的细胞转染与可选择的microRNA表达构造( 一 )。在恢复过程中,miRNA的表达结构可以集成(B)和表达miRNA的构造上的编码选择标记。选择稳定的集成(C)和细胞扩张后,细胞治疗用更昔洛韦(四)。 (五)另一方面生产中存在更昔洛韦(即细胞结构没有针对性的miRNA TKzeo)胸苷激酶(TK)的细胞就会死亡,细胞中含有与miRNA靶小号图书馆结构的ITES将不会产生传统知识,因此,将生存更昔洛韦选择(F)的。可以生长,存活的细胞基因组DNA隔离,含基因的miRNA靶位点PCR扩增使用的目标ID扩增引物。 PCR产物可识别miRNA靶基因与人类基因组测序和对齐。
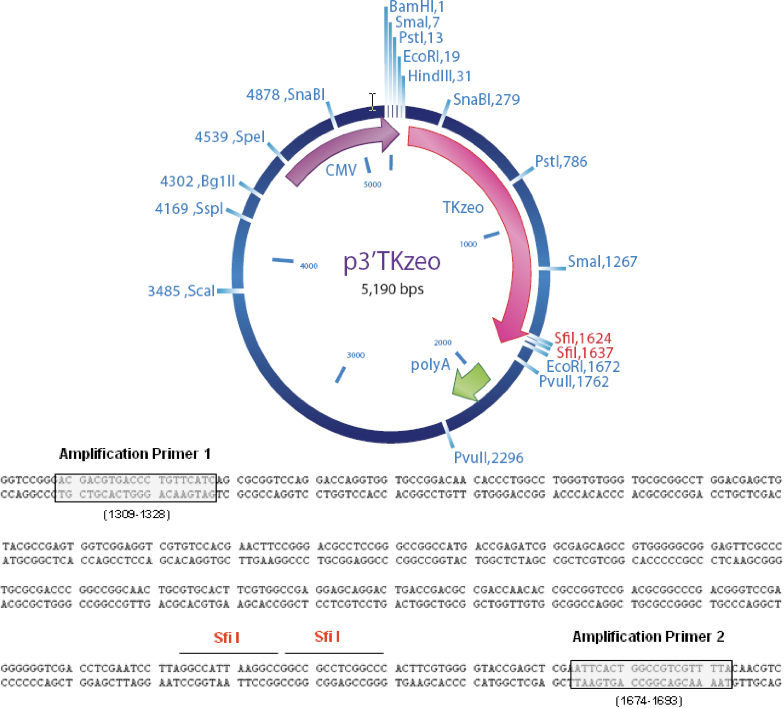
图4质粒图谱和扩增引物的位置。 SFI我cDNA的克隆网站。
引物序列
任务目标ID扩增引物1
5 ACGACGTGACCCTGTTCATC 3
任务目标ID扩增引物2
5 TAAAACGACGGCCAGTGAAT 3
ig5.jpg“ALT =”图5“/>
图5。细胞生长的影响更昔洛韦。二十四孔板接种二十万MCF-7细胞MCF-7细胞含有目标ID库。二十四小时后,培养基含有0,8,或16μM更昔洛韦的培养基取代。 15天之后,该板块被拍到(6上井),然后与HBSS中与艳蓝R染色液(B6529)(6井)染色洗井。请注意,更昔洛韦有没有图书馆MCF-7细胞没有影响,因为他们不表达胸苷激酶(TK)。另一方面,MCF-7细胞库细胞做明确的传统知识,但不完全由更昔洛韦杀害在8或16μM,采取了Brillinat蓝色的活细胞中。然而,图书馆细胞停止生长,更昔洛韦,苯酚在他们介质的红色染料的颜色证明。被捕的细胞不酸化他们的媒介,和颜色保持红色,而不是改变或昂热 - 黄色。
| 试剂 | 体积/反应 |
| Jumpstart的REDTaq预拌反应混合物 | 10μL |
| 扩增引物1(25μM的) | 0.2μL |
| 扩增引物2(25μM的) | 0.2μL |
| 基因组DNA | 50-200纳克 |
| 水,分子级 | 20μL |
表1。
| 步骤 | 温度。 | 时间 | 周期 |
| 初始变性 | 95℃ | 5分钟。 | 1 |
| 变性 | 95℃ | 30秒。 | 泛=“3”> 40次 |
| 退火* | 62℃ | 30秒。 | |
| 延期 | 68℃ | 2分钟。 | |
| 最后一次延长 | 68℃ | 5分钟。 | 1 |
| 举行 | 4℃ | 举行 |
表2。
*退火温度可能会有所不同,但我们已经观察到最好的扩增产物与退火温度在53和64之间不等°C。
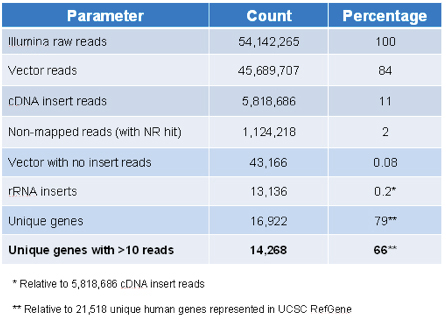
表3。目标ID Illumina的测序图书馆内容。
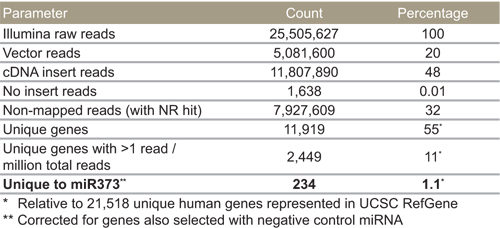
表4。miR-373的Illumina的测序选定的目标。
les/ftp_upload/3303/3303table5.jpg“ALT =”表5“/>表5。先前由RNA微阵列确定了10个目标。
讨论
microRNA是20-24核苷酸的RNA调节基因表达的转录后通过抑制mRNA的翻译,经常动摇的目标mRNA(综述[6])。单个的miRNA可以调节数百个基因来控制细胞的发展和环境信号的响应。识别和验证目标基因是决定一个miRNA在这些途径中的作用和功能是必不可少的。然而,目标识别,因为在动物中,miRNA和其目标网站不完全互补的,并不是一件简单的。 “种子”的地区,基地通过7 miRNA的5'-端,通常是互补的目标。不过,也有许多例外的种子规则,和下游的碱基配对可以弥补不完善的种子比赛。已开发的计算机算法来预测种子匹配和下游的补偿,目标结构上的miRNA靶D的位置,序列保守性,以及各种实验验证目标(综述[7])已观察到的其他参数。而在硅片的预测是方便做识别许多有效的miRNA靶基因,预测基因实验验证测试失败,许多实际的目标是不预测。此外,由于计算机算法根据先前确定的目标功能,他们根本不容许什么是已知的偏离目标的发现。
一些实验系统已成功地用于识别或发现在活细胞功能的miRNA靶基因。这些全球性的筛查方法包括微阵列和RNA测序,RNA的免疫共沉淀(RIP)和氨基酸在细胞培养稳定同位素标记(SILAC的),蛋白质的方法(综述[7])。每种方法都有优点和缺点。由于miRNA的目标往往破坏的mRNA,并导致其降解,损失ØR GAIN后引进的miRNA模仿或抑制剂,分别在mRNA水平,可以识别miRNA靶标。很容易被检测芯片或深度测序表达这方面的损失或收益。虽然mRNA的检测方法是蛋白检测方法相比更简单,更敏感,基因检测将错过任何miRNA的目标是没有退化。从巴特尔实验室SILAC的那些miRNA靶RNA检测结果比较从最近的报告[8]或核糖体分析[9]表明,哺乳动物的miRNA主要采取行动,减少目标mRNA水平。然而,许多其他实验室工作与个人的miRNA靶基因检测中蛋白质的变化,但在mRNA水平没有变化。什么似乎是没有检测到mRNA的损失与翻译调控的报告包括:miR-10B的上HOXD10 [10],miR221/222上p27kip1的[11]中,miR-21 PDCD4 [12],miR-126的p85β[13]中,miR-34对SIRT1的[14],miR-21的PTEN [15]中,miR-302D Arid4b [16],miR-200C的JAG1 [17],和miR-299,297,567,ND VEGFA 609 [18]。此外,克兰西等[19]最近报道,let-7的翻译调控只发现当包含let-7的目标网站的个人表达亚型选择性检测。后者表明,至少在某些情况下,翻译调控可能被忽略在复合型材 - 也就是说,当所有的mRNA异构体作为一个mRNA的检测 - 与许多芯片和序列分析获得。通过Argonaute蛋白(通常AGO2)或其他相关蛋白(RNA免疫沉淀,或RIP)的miRNA-mRNA的复合物的免疫共沉淀分离miRNA靶基因调控机制。此外,RIP检测内源性的,想必生物学相关,相互作用。但是,不知道是否所有的miRNA-mRNA的相互作用功能,RIP会错过任何mRNA的目标关联只是短暂的,或者是正在迅速退化。此外,特定的miRNA-mRNA的伙伴必须推断bioinformati的美云,因为所有的miRNA表达对共沉淀一起。最后,SILAC的直接识别miRNA调控,蛋白质本身的最终产品,但不敏感,因此错过难得的蛋白质和蛋白水平的小折变化。
鉴于当前miRNA的目标识别方法的局限性,我们认为需要一个附加的全局分析,识别功能miRNA靶基因的一种替代机制。为了满足这种需要,我们许可由JOOP Gaken伦敦大学国王学院和阿齐姆Mohamedali发明的技术。他们的发明是一个双选择融合蛋白,特别是一个胸苷激酶zeocin融合的,在其3'UTR潜在的miRNA靶序列的cDNA文库由监管。可以选择与表达基因TKzeo,结构稳定转染细胞与zeocin,在图2所示。 miRNA的表达了兴趣后,可以选择的miRNA的目标无线网络日更昔洛韦,在图3所示。更昔洛韦杀死细胞表达胸苷激酶,也就是说,任何细胞表达TKzeo的基因缺乏利益的miRNA的目标。有针对性的基因可以分离的DNA的PCR扩增细胞的生存更昔洛韦使用侧翼的cDNA和PCR产品可引物测序,以确定目标。
我们已经发展成为一个全球性的鉴定和功能的人类miRNA靶基因的发现,新工具的国王学院的技术 - 任务目标ID图书馆。与目标识别库,用户可以通过一系列哺乳动物细胞培养转染和药物筛选的步骤隔离miRNA靶基因。图书馆是全面的,包含66-79%的人类基因。初步结果表明,先前发现以及新的目标,可以从任务目标ID图书馆隔离。
虽然该协议是一些长度,目标ID库需要使用标准的分子实验室技术 - 哺乳动物细胞培养,转染,药物筛选,PCR扩增,测序 - 因此,应该在大多数生物学家的手段。我们建议支付足够的重视,适当的实验设计,优化培养条件,最重要的是,预先测试每一个新的细胞类型选择步骤(杀死曲线)与目标识别库,以确保成功,以确定最佳的药物浓度。
与所有的miRNA目标识别方法,它强烈建议用第二种方法,如芯片,定量RT-PCR,记者法(即荧光素酶),或西方的分析,验证,筛选目标ID图书馆确定的目标。
披露声明
作者声明在提交的工作有兴趣的商业实体的财务关系。
致谢
我们感谢她的支持和参与丰硕的故障排除讨论整个西格玛生命科学任务目标的ID库的开发团队,尤其是凯文为寻找技术和谈判授权条款Gutshall,希瑟Holemon的。我们也感谢伦敦大学国王学院的博士JOOPGäken自由分享它克隆的cDNA伟大的批次和他坚持准备在未发表的结果和建议,和RxBiosciences哈米德·卡齐。我们想南邻和斯科特·巴哈尔承认SAFC进行cDNA阵列和数据分析。
任务是Sigma-Aldrich公司的生物技术LP的注册商标。
材料
| Name | Company | Catalog Number | Comments |
| 试剂名称 | 公司 | 目录编号 | 评论 |
| 订JumpStart REDTaq预拌 | Sigma-Aldrich公司 | P0982 | |
| 更昔洛韦 | Sigma-Aldrich公司 | G2536 | |
| G418的 | Sigma-Aldrich公司 | G8168 | |
| 嘌呤 | Sigma-Aldrich公司 | P9620 | |
| TOPO TA | Invitrogen公司 | KNM4500-01 | |
| GenElute哺乳动物的基因组DNA小量提取试剂盒 | Sigma-Aldrich公司 | G1N10 | |
| 细胞特异性核转ķ它 | 龙沙 | ||
| nucleofector | 龙沙 | 反倾销协定,1001 | |
| zeocin | Invitrogen公司 | R250-01 | |
| 目标ID库 | Sigma-Aldrich公司 | MREH01 |
参考文献
- Huang, Q., Gumireddy, K., Schrier, M. The microRNAs miR-373 and miR-520c promote tumour invasion and metastasis. Nat. Cell Biol. 10 (2), 202-210 (2008).
- Lichner, Z., Pall, E., Kerekes, A. The miR-290-295 cluster promotes pluripotency maintenance by regulating cell cycle phase distribution in mouse embryonic stem cells. Differentiation. 81 (1), 11-24 (2011).
- Subramanyam, D., Lamouille, S., Judson, R. L. Multiple targets of miR-302 and miR-372 promote reprogramming of human fibroblasts to induced pluripotent stem cells. Nat. Biotechnol. 29 (5), 443-448 (2001).
- Lim, L. P., Lau, N. C., Garrett-Engele, P. Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs. Nature. 433 (7027), 769-773 (2005).
- Huntzinger, E., Izaurralde, E. Gene silencing by microRNAs: contributions of translational repression and mRNA decay. Nat. Rev. Genet. 12 (2), 99-110 (2011).
- Thomas, M. Desperately seeking microRNA targets. Nat. Struc. Mol. Biol. 17 (10), 1169-1174 (2010).
- Baek, D. The impact of microRNAs on protein output. Nature. 455 (7209), 71-71 (2008).
- Guo, H. Mammalian microRNAs predominantly act to decrease target mRNA levels. Nature. 466 (3708), 835-840 (2010).
- Ma, L. Tumour invasion and metastasis initiated my microRNA-10b in breast cancer. Nature. 449 (7163), 682-688 (2007).
- Visone, R., et al. MicroRNAs miR-221 and miR-222, both overexpressed in human thyroid papillary carcinomas, regulate p27Kip1 protein levels and cell cycle. Endocrine-Related Cancer. 14 (3), 791-798 (2007).
- Lu, Z., et al. MicroRNA-21 promotes cell transformation by targeting the programmed cell death 4 gene. Oncogene. 27 (31), 4373-4379 (2008).
- Guo, C., et al. The noncoding RNA, miR-126, suppresses the growth of neoplastic cells by targeting phosphatidylinositol 3-kinase signaling and is frequently lost in colon cancers. Genes Chrom. Cancer. 47 (11), 939-946 (2008).
- Yamakuchi, M., et al. miR-34a repression of SIRT1 regulates apoptosis. Proc. Natl. Acad. Sci. U.S.A. 105 (36), 13421-13426 (2008).
- Wickramasinghe, N. S., et al. Estradiol downregulates miR-21 expression and increases miR-21 target gene expression in MCF-7 breast cancer cells. Nucleic Acid Res. 37 (8), 2584-2595 .
- Ciaudo, C., et al. Highly dynamic and sex-specific expression of microRNAs during early ES cell differentiation. PLoS Genetics. 5, e1000620 (2009).
- Vallejo, D. M., et al. Targeting Notch signaling by the conserved miR-8/200 microRNA family in development and cancer cells. EMBO J. 30 (4), 756-769 (2011).
- Jafarifar, F., et al. Repression of VEGFA by CA-rich element-binding microRNAs is modulated by hnRNAP. L. EMBO J. 30 (7), 1324-1334 (2011).
- Clancy, J. L., et al. mRNA isoform diversity can obscure detection of miRNA-mediated control of translation. RNA. 17 (6), 1025-1033 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。