Method Article
发现蛋白质相互作用和检定蛋白质功能使用的HaloTag技术
摘要
的HaloTag技术是显示在小和大的蛋白质复合物从哺乳动物细胞中分离显著成功的多功能技术。在这里,我们重点介绍了这项技术的优势比现有的替代品,并展示其效用研究蛋白质功能的许多方面里面的真核细胞。
摘要
研究蛋白质组学已经爆炸,近年来随着质谱能力的进步,导致了众多的蛋白质组,包括那些来自病毒,细菌和酵母的特性。相比之下,人类蛋白质组的分析滞后,部分原因是由于绝对数量必须研究的蛋白质,而且网络和这些存在相互作用的复杂性。专门解决理解人类蛋白质组的挑战,我们已经开发的HaloTag技术,蛋白分离,多蛋白复合物分离尤为强劲,并允许在低丰度弱或短暂的相互作用和/或蛋白质的更有效的捕获。的HaloTag是一种基因编码的蛋白融合标签,专为共价键,具体,并迅速固定或与各种配体蛋白的标记。凭借这些特性,开发,以人物众多应用的哺乳动物细胞IZE蛋白质的功能,在这里,我们提出的方法,包括:用于发现新的相互作用和功能分析蛋白质的下拉功能,以及细胞定位。我们发现显著优势,在速度,特异性,和融合蛋白对表面进行蛋白质组分析,相对于其他传统的非共价方法的共价捕获。我们证明这些和使用两个重要的后生蛋白质作为例子,人的溴区蛋白BRD4和组蛋白脱乙酰基酶HDAC1该技术的广泛的实用性。这些例子说明了该技术在实现新颖的交互的发现和真核生物中表征细胞定位的权力,这将进一步共同了解人性化的功能蛋白质组学。
引言
了解细胞功能,对刺激的反应,并转变发展方式和/或疾病状态是密切关联的去卷积的蛋白质在整个这些不同的动态状态1,2。已作出很大努力来理解低等生物的蛋白质组,但是,给定的蛋白质的数量与所有可能的相互作用,这是非常具有挑战性的寻址人类蛋白质组1,3-7。在质谱的进展,极大地激活可进行这些研究的能力,在这里,我们提出了一个额外的和显著的进步与的HaloTag技术的发展为蛋白质复合物和哺乳动物细胞8-10人体蛋白质功能特性的既有效捕获。此技术最近已显示在几项研究是为发现的重要的相互作用的一个关键因素,允许洞察新的蛋白质功能和understandi吴疾病11-13。
该蛋白质的HaloTag融合最初开发是为了解决传统的亲和标签和抗体可作为相关蛋白捕获,纯化和标记8-10几个挑战。内几乎所有方法的蛋白质捕获和纯化,有其用于特定蛋白14的浓缩的非共价相互作用的步骤。在此步骤中,蛋白和/或络合物可以解离,由于扩散,特别是如果该过程需要时间,而不是分钟来完成。为了解决这一问题,该融合蛋白是专门设计,以快速和可逆地与其配体的相互作用,这可以是颗粒,表面,或荧光团9。因此,一旦它被绑定到其配体,它仍然绑定,这是最差分系数,相对于其他的亲和标签中的一个。这里还展示,是能够以解决不同的生物学问题用单个构建体,其可以通过交替配体9( 图1)。例如,询问蛋白质相互作用或功能,表面配体可用于捕获的蛋白质或复合物( 图1)。在另一项实验中,同样融合蛋白可被荧光标记,研究细胞定位,贩运,或蛋白质周转( 图1)。要记住然而,一旦配体结合,无论是表面或荧光团,它是不可逆的,因此,其它的配体不能再被随后捆绑在相同的实验是非常重要的。
现有的技术来映射蛋白质相互作用的分析揭示了难度,有效地隔离特定的相互作用,包括那些更短暂的或现在的低丰度1,6,7,14,15。此外,最近的工作由无数组s显示了在传统的分离方法,特别是在人类蛋白质组学领域,也有显著数量的蛋白质污染物可能掩盖真正来自于交互件质谱分析16个信号的关注。为的HaloTag蛋白及其共同开发的树脂地址以及这些关注开发的物业。在可以在15分钟内实现的协议复杂的下拉( 图1),复合物的结合,既促进复合物捕获和减少非特异性结合8水平。另外,不可逆地结合允许对低丰度蛋白的高效捕获并允许使用少得多的细胞,再一次减少背景8。一旦复合物的捕获建立后,协议包括温和的洗涤条件,以维护复杂的完整性。捕获复合物的洗脱可以通过两种方法和所选择哪种方法由下游肛门的目标决定进行ysis。如果愿望是分析或通过蛋白质印迹或质谱发现相互作用的蛋白质,那么建议以洗脱物与变性剂如SDS或尿素( 图1)。这是用于质谱测试的特别有利的,因为最初稠合的HaloTag蛋白,称为诱饵蛋白,将留在后面结合到树脂中,因此不会主导的质谱检测的肽的人口。这也意味着然而,该诱饵蛋白很可能是不可见的由免疫印迹,一个显著差异,相对于其他的非共价亲和纯化或共免疫沉淀分析。如果目标是达到净化复杂原封不动地用于功能研究,洗脱可以使用TEV(烟草蚀纹病毒)蛋白酶,其识别的融合蛋白和诱饵蛋白( 图1)之间的编码其同源序列裂解来进行。 Ŧ淡褐色样品也可以通过质谱法进行分析,但如后所示,它们将包含一个显著量诱饵蛋白,可能会阻止检测低丰度的,特定交互件。
在这里,我们提出了适用于两个重要的治疗蛋白,该蛋白溴区BRD4和组蛋白去乙酰化酶,HDAC1 17下拉和成像协议。我们已经证明具有这些例子中,与预期伙伴相互作用如通过质谱法,经过复杂的隔离HDAC1酶活性,并为适当的细胞定位来确定。总之,这些结果证明了该技术的真核细胞蛋白质的相互作用和功能特性的多功能性质和强度。
研究方案
注意:下面的协议可以与任何的HaloTag融合和选择稳定或瞬时转染的任何细胞系中使用。对于这些例子,我们会给协议进行的HaloTag融合在HEK293T或HeLa细胞的瞬时转染。对于所有实验,我们建议使用蛋白质只控制。
1,融合蛋白表达测试
- 获得融合蛋白的结构:
- 获得预先制作的HaloTag融合载体或构建使用现有的载体。有关可用的结构的更多信息,请参阅表中的特定试剂。
- 如果进行任何克隆,建议进行测序验证该DNA。
- 检测融合蛋白与控制的表达式:
- 对于每个样品,准备加入250μl的1×SDS上样染料缓冲液(60毫摩尔Tris HCL PH6.8,0.75 mM的溴酚蓝,12.5%甘油,100mM的二硫苏糖醇,0.5%SDS)。
- 对于每一个融合蛋白和控制,板0.5毫升HEK293T或HeLa细胞在其相应的媒体在2-4×10 5细胞/在一个标准的24孔板ml的密度。
- 培养18〜24小时,在37℃和5%CO 2中 ,然后转染的建议。
- 24小时转染后,加入0.5微升的5mM四甲基若丹明(TMR)的配位体的每一个的媒体,并轻轻混合板。
- 孵育含有配位体为15分钟,在37℃和5%CO 2的转染细胞。
- 含配体吸去培养基,轻轻地用1 ml RT PBS以及细胞每洗。
- 吸过PBS和执行第二次洗1毫升RT PBS。
- 除去PBS中,加入200μl1X的SDS直接上样染料缓冲区细胞。
- 吸取裂解液从平板到1.5ml离心管中。
- 在95℃下煮沸裂解物5分钟
- 除去5-10微升的反应和负荷到SDS-PAGE凝胶中。
- 使用荧光检测仪(TMR激励:555 nm,发射:585纳米)来检测条带。使用荧光蛋白标记物为标准。
- 如果荧光检测仪是不可用时,使用抗两种诱饵蛋白或蛋白融合标记,以检测该融合蛋白的表达进行蛋白质印迹。
2,蛋白质下拉
- 制备瞬时转染细胞的:
- 对于每一个融合或控制准备一张15cm培养皿用30毫升的细胞在3-4×10 5细胞/ ml或1〜1.2×10 7细胞的总每个构建。
- 培养18〜24小时,在37℃和5%CO 2中 ,然后转染构建的建议。
- 经过24-48小时转染后,取出介质,并轻轻地用20-25毫升冰冷的PBS洗细胞层。
- 除去PBS液冲洗,再加入25-30毫升4℃冷藏的PBS,轻轻刮去细胞脱盘。
- 收集细胞成锥形管和离心福哥5-10分钟在2000 XG和4°C。
- 丢弃上清液,并将细胞沉淀在-80℃下至少30分钟,或最多6个月。
- 平衡的HaloLink树脂:
- 对于每种融合或对照样品,制备将12ml树脂平衡/洗涤缓冲液(100mM的Tris-HCl pH值7.5,150mM的NaCl和0.005%IGEPAL CA-630) 注意:这是非常重要的,以及使用该树脂平衡缓冲液中12小时的稀释IGEPAL CA-630是不稳定的或有效的超出了这个时间框架。
- 轻轻摇动或混合树脂以获得均匀的悬浮液。
- 对于每一个下拉实验,取200微升树脂至1.5 ml离心管。
- 离心机在800×g离心1分钟( 例如,每分钟3000转的离心),然后小心地取下并丢弃上清液(乙醇),而不必在管的底部扰乱树脂。
- 加入800μL树脂平衡/华的SH缓冲区和颠倒离心管数次调匀。
- 离心800×g离心2分钟,然后小心地取出并弃去上清液。
- 总共3次洗涤,重复步骤2.2.5和2.2.6 2次以上。
- 请勿移除最后一次洗涤液或上清液,直到准备加入下述细胞裂解液(步骤2.3.8),以防止树脂干燥。
- 结合与融合复合物的洗涤:
- 对于每个样品,制备500μl的哺乳动物细胞裂解缓冲液(50mM的Tris-HCl pH为7.5,150 mM氯化钠,1%的Triton X-100,0.1%脱氧胆酸钠)和1的1X TBS缓冲液(100mM的Tris-盐酸pH值7.5和150mM的NaCl)中。
- 移液器向上和向下或短暂涡旋解冻的细胞沉淀重悬在300微升哺乳动物裂解液。
- 加入6微升的50X蛋白酶抑制剂(800微克/毫升苯甲脒盐酸,500微克/毫升邻二氮菲,500微克/毫升抑肽酶,500微克/毫升亮肽素,500&#181克/毫升胃蛋白酶抑制剂A,50mM的PMSF) 注:蛋白酶鸡尾酒,其中包括AEBSF不能使用,因为这些干扰蛋白质融合标签绑定。
- 加入3微升RQ1 DNA酶和转化在室温10分钟。
- DOUNCE用玻璃匀浆器2.0毫升大小; 25-30招冰,或者通过25或27号针头通过细胞的5-10倍,完成裂解注:超声是不建议作为复合物可能散架和过热可能会影响蛋白质的融合标签的活动。
- 离心机以14,000×g离心5分钟,在4℃下以清除裂解物。
- 传输清晰的裂解液,约300μl总体积,以新管置于冰上。
- 加入澄清的裂解增加700微升的1X TBS缓冲液,并通过上下吹打拌匀。
- 取步骤2.2.8制备平衡树脂管和从树脂除去最后的洗涤/上清液没有在管的底部扰乱树脂。
- 添加到每个TU是树脂中,加入1ml稀释裂解物。
- 孵育15分钟,在22°C。 注混合在一个管旋转(或温柔的混频器):树脂在这段时间建立具有约束力降低效率。如果在4℃下结合需要,通过混合1小时,这样做。
- 树脂离心管中在800×g离心2分钟,弃上清。
- 加入1 ml的树脂平衡/洗涤缓冲液的步骤2.2.1提出和通过反相树脂管的手几次调匀。
- 树脂离心管中在800×g离心2分钟,弃去洗。
- 重复步骤2.3.12其次是2.3.14三个额外的时间。
- 加入1 ml的树脂平衡/洗涤缓冲液和孵化在22℃下5分钟,不断旋转。
- 树脂离心管中在800×g离心2分钟,弃去洗。
- 这取决于最终的应用(参见导言作进一步的解释),进行任第2.4或2.5。
- SDS洗脱变性凝胶,印迹或质谱分析:
- 从每个样品重悬在树脂中加入50μlSDS洗脱缓冲液(1%SDS和50mM的Tris-HCl pH7.5的)
- 摇动试管在室温下30分钟。
- 离心机在800 XG和洗脱液转移到新管中进行分析2分钟。
- 免疫印迹或银染色凝胶,负载5-10微升在SDS变性凝胶。
- 对于质谱,保存将40μl各样品于-20℃。
- TEV蛋白酶洗脱进行功能分析,蛋白质印迹,或质谱:
- 去掉最后一次洗涤后,悬浮树脂在50微升ProTEV裂解缓冲液和30个单位的TEV酶。
- 孵育在25℃下振荡1小时。
- 离心2分钟,在800×g的。
- 洗脱液转移至新管。
- 在4℃下,在功能测定立即使用存储样本。
3。将荧光标记的融合蛋白的细胞成像用胞质和核配体透水
- 转染,标签和图像细胞:
- 在以1-2×10 5细胞/ ml的密度为8以及腔玻璃罩每个融合蛋白或控制,板400μl的HeLa细胞在每孔中的适当的媒介。
- 培养18〜24小时,在37℃和5%CO 2中 ,然后转染的建议。
- 18-24小时转染后,稀TMR配体在适当的细胞介质1:200,然后加入100μl的该溶液,以每孔中,并轻轻混合。
- 孵育含有配位体为15分钟,在37℃和5%CO 2的转染细胞。
- 含有配位体和替换用500μl的适当的介质缺乏蛋白融合标记的配体,其中已经预先加热至37℃,吸去培养基
- 总共3次洗涤重复两次。
- 把细胞放回培养箱中(376,C和5%CO 2)30分钟。
- 吸断纸张,换用500微升适当的媒体,已预热至37°C。
- 图像上使用适当的采集参数(TMR激励:555 nm,发射:585 nm)的显微镜。
结果
当任何新的融合蛋白的工作,重要的是要对转染后的该蛋白质的表达第一测试也验证了正确分子量的蛋白质被生产。如的HaloTag融合蛋白可以是荧光和共价标记的可渗透的或取决于定位,不透水的配体,它可以通过施加蜂窝裂解物以变性凝胶电泳随后扫描的fluorimager快速确定表达。使用在第1.2节,最后一战- BRD4(189 KD)的表达和单独的HaloTag控制(CTRL)观察(34 kD的, 图2A)中描述的协议。中所提到的协议,也可以使用传统的Western印迹用抗的HaloTag抗体来检测融合蛋白的表达,或者如果它们是可用的,抗体与诱饵蛋白。如果可能的话,建议使用荧光配体,而不是因为它是更具体,更快速,更容易吨汉抗体检测,同时还定量10。
适当的全长融合蛋白的表达被证实后,蛋白质下拉可以被执行。 如图2B所示的是用SDS(协议第2.4节),用卤代BRD4和Ctrl下拉而展示出高再现性生物学重复的银染色凝胶。在银染凝胶上显示出显著数目的蛋白质被发现与BRD4蛋白和对照组( 图2B)非常低的背景相互作用。如引言中所述,在洗脱的过程中,卤代 - BRD4不会从树脂上洗脱下来,因为它仍然是共价结合。因此,不有显著带在银染色( 图2B),或蛋白印迹分析(数据未示出)被检测到这样的分子量。以确定是否这些蛋白质所特有的BRD4,液相色谱质谱法(LC-MS/MS)关于c进行SDS洗脱后获得omplex的混合物。 如图2C所示的光谱数和归一化光谱丰度因子为18-20 BRD4的已知相互作用物的卤代BRD4质谱分析测定值(NSAF)值。高丰组件从pTEFb 18,20,也是BRD9 19蛋白的确认BRD4复合物的特异性捕获。作为预测的银染色凝胶( 图2B),许多其他的蛋白质也被确定为BRD4的那些没有在对照(数据未示出)观察到潜在干扰作用。因为这些是先前未知的,它们需要被独立地通过其他方法验证,以确认该蛋白是否是一个真实的交互件,并且如果是这样,如果它是直接或间接与BRD4相关联。
分离的复合物也可以用于研究活性;它是推荐使用TEV蛋白酶(协议第2.5节),使他们保持functionali洗脱物TY。在图3A中 ,从使用TEV蛋白酶的树脂释放晕-HDAC1下拉复合银染色凝胶结果。如TEV蛋白酶将裂解的蛋白融合标记和其融合伴侣,显著量的诱饵蛋白,在这种情况下,HDAC1,观察到( 图3A)之间的连接区域。如果要判断这部分包含HDAC1活性,TEV洗脱样品中的荧光检测HDAC,HDAC-21格洛进行了测试。 如图3B所示 ,HDAC1下拉样品显示出高水平的HDAC1活性的(第1栏),它是专门通过已知的HDAC抑制剂,SAHA 22(第2栏)抑制。作为对照,以进一步证明特异性,无HDAC抑制观察到与一个相关的沉默信息调节因子家族的抑制剂,EX-527 22(第3栏),用缓冲液单独无HDAC1下拉样品中加入(第4列)中没有检测到信号。
的功能的显著组成部分人蛋白质组学和理解配合,也理解蛋白质的定位和/或贩卖。由于这些相同的融合构建可荧光标记的细胞内,我们使用共焦成像仪来监控他们的定位。以下第3节中的协议,HeLa细胞瞬时转染晕-BRD4( 图4A)和Halo-HDAC1( 图4B)进行荧光标记的配体的TMR和成像。 如图4A和4B所示 ,这两个定位于预期17细胞核。这些数据表明,标签的存在并没有改变它的融合配偶体的生理细胞定位。

图1。原理蛋白质下拉和共聚焦成像应用中,使用的英格尔构建多个应用程序对于了解蛋白质的功能在哺乳动物细胞中是可能的。对于所有,卤代融合构建体可以是稳定或瞬时表达于贴壁或悬浮的哺乳动物细胞。对于复杂的蛋白质下拉,将细胞裂解,然后,复合物被共价捕获于树脂,并通过任一SDS洗脱(左通道)或TEV切割位(右通道)洗脱。 SDS洗脱被推荐用于在进行质谱分析,而TEV切割位是最优的,用于执行功能的分析。表征的表达,细胞定位,贩卖事件,或蛋白质周转,活细胞表达融合蛋白的荧光标记和SDS-PAGE凝胶或使用共焦成像进一步分析。两种细胞可渗透的或不可渗透的荧光配体,可根据该融合蛋白的细胞内定位或文稿。
IGURE 2“FO:内容宽度=”6英寸“SRC =”/ files/ftp_upload/51553/51553fig2highres.jpg“宽度=”600“/>
图2:最后一战- BRD4蛋白的表达,下拉,和质谱分析。 (A)SDS-PAGE凝胶显示晕- BRD4融合蛋白,189 kD的,独自晕蛋白融合标签,34 kD的控制(CTRL)的标有TMR配体(协议第2.1节)一个HEK293T细胞裂解液中的表达。凝胶进行扫描的fluorimager检测和荧光分子量标记,使用(B)生物学重复进行纯化,用SDS卤代BRD4和Ctrl样品的下拉的银染色凝胶。分子量大小是表示用于凝胶(C)光谱计数(左图)和归一化光谱丰度(NSAF)值(右图)占蛋白在卤代的生物学重复的质谱分析鉴定的蛋白质的分子量BRD4和Ctrl键。显示的是众所周知的互动瓦特蛋白质第i个BRD4,包括pTEFb(CDK9和Cyclin T)的组成部分18,20,以及BRD9 19。从这些蛋白肽没有被确定在Ctrl键。
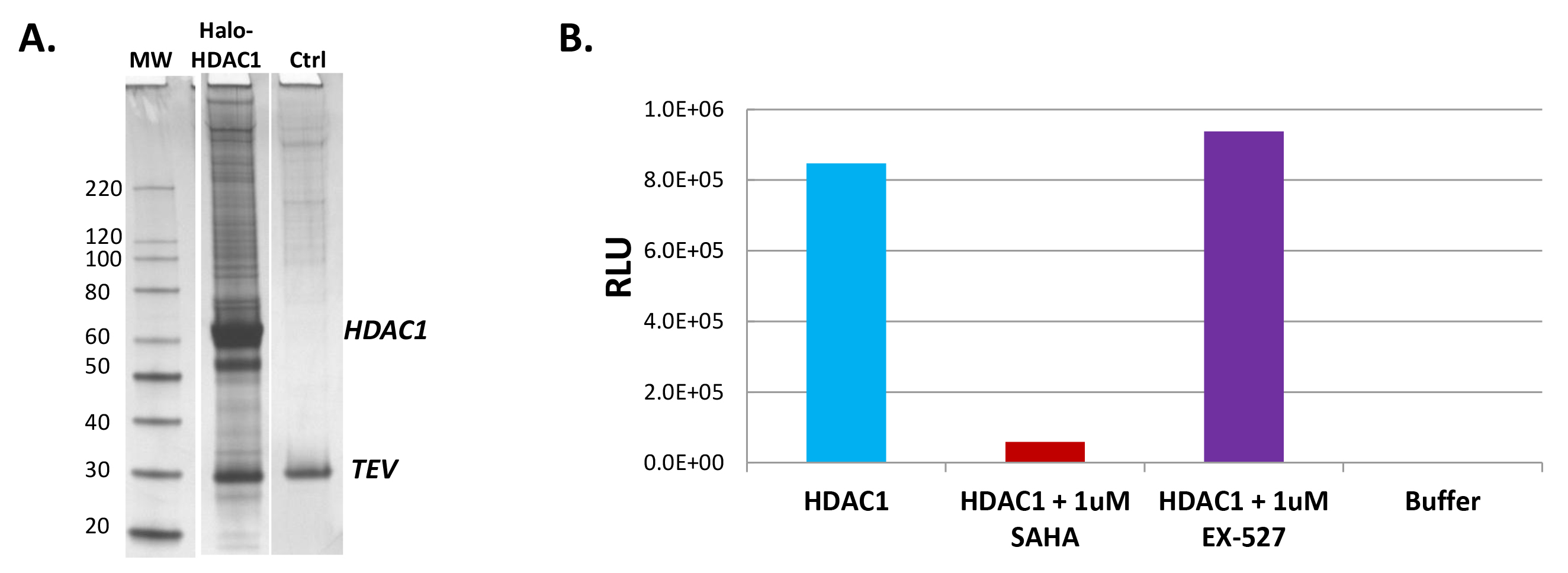
图3:最后一战,HDAC1复合物分离和活性分析。 (一)银染凝胶呈现出晕-HDAC1复合物和背景,从TEV酶切后,按Ctrl(协议第2.5节)的隔离。突出HDAC1带(55 kDa的)和TEV蛋白酶带标示。自由HDAC1通过TEV切割捕获在树脂上( 图1)(B)示出图形HDAC1复合隔离样品中的荧光组蛋白脱乙酰酶测定中,HDAC-活性后的HaloTag和HDAC1融合序列之间的最佳连接物内产生的GLO 21。图中第1栏高升HDAC活性的包含与光晕-HDAC1下拉样品(HDAC1)evels。第2栏显示这个活动可以通过另外的HDAC抑制剂,萨哈22的具体下降,到HDAC1下拉样品。作为对照,第3栏显示特定沉默信息调节因子家人去乙酰化酶抑制剂,但不是乙酰化酶,EX-527 22,不抑制HDAC1活性和第4列显示没有活动是单独使用的缓冲观察。
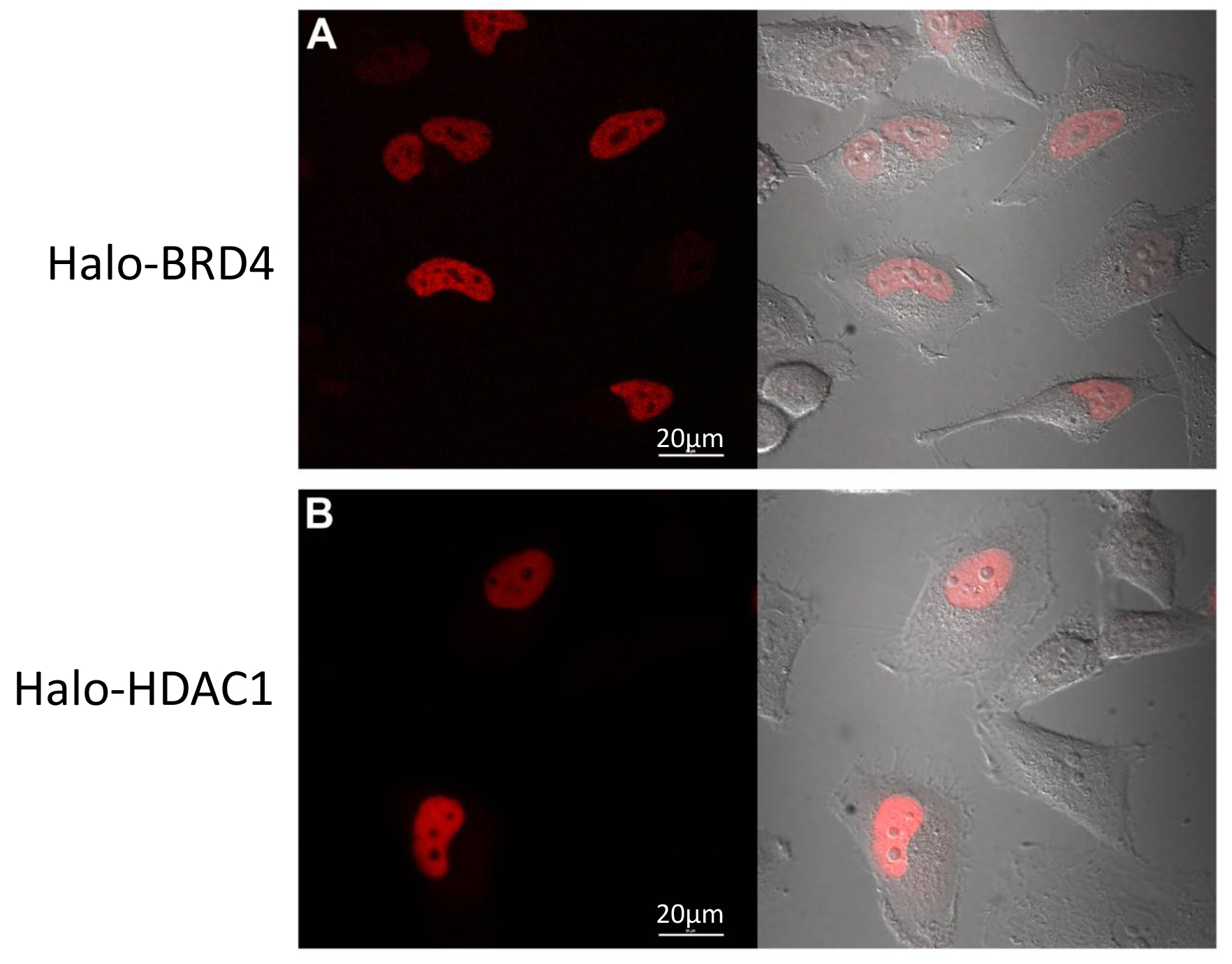
图4:最后一战- BRD4和Halo-HDAC1共聚焦成像。活HeLa细胞转染晕- BRD4(A)或晕-HDAC1(B)荧光标记的TMR配体细胞共聚焦成像。(a)卤素- BRD4的表达被限制到细胞核和(B)晕-HDAC1表达主要是NUC利尔。面板的左侧是荧光通道和右侧是荧光通道与DIC通道的每一个的叠加。被收购的共聚焦显微镜配备使用适当的过滤器套37°C + CO 2环境室的图像。比例尺=20μm左右。
讨论
这里介绍两种融合蛋白,晕 - BRD4和Halo-HDAC1,其特征在于真核哺乳动物细胞中进行表达,蛋白复合物的分离和活性,细胞定位。通过这些不同的协议工作,有针对每个实验的成功几个重要的步骤。由于是与它本身可以保持生理的关键变量的任何融合蛋白的表达水平和位置的情况下。因此,重要的是要考虑到的N-和/或C-末端融合将需要进行设计,如果先验知识或与其他标记工作尚未得到证实供选择的特定的蛋白质。对于表达,如果电平太高,可以转染的载体或使用过程中执行较弱的启动子的DNA稀释的都可以实现,是适当的水平。前期工作已经完成,显示在勒内源性大分子复合物的高效分离表达式8的VELS,使工作在表达水平非常低。如果可能的话,细胞定位研究也将能够提供答案为需要进行适当的生理最优标签放置位置以及相对表达水平。
一旦融合蛋白是准备下拉实验,以获得最大的成功与该协议是非常重要的,以跟随在裂解建议的时间帧,绑定和洗涤部分,作为最大的优点之一是复杂的隔离的速度过程。如果上述任何步骤被延长时间,例如,需要对基于抗体的捕获方法,存在复杂的解离的危险或增加非特异性结合8。同样,如果在次期间,细胞裂解或有约束力的缩短,电池可能无法完全溶解或分别有效地捕获。如果洗涤次数是DECR缓解或在结合或洗涤液,不会发生树脂的良好混合,然后非特异性蛋白的背景水平会有所提高。此外,裂解的手段是作为相关的结合效率与树脂非常重要的。企图超声处理的样品中,去掉了推荐的洗涤剂,添加SDS或其它强的洗涤剂和/或包含所述蛋白酶抑制剂AEBSF将导致减少或融合蛋白和它们的复合物结合到树脂上的损失。
从哺乳动物细胞和人类蛋白质组分析的奋斗与减少的背景中分析通过质谱16的挑战,复杂的隔离现有的方法。这一直显著,杂蛋白的存储库已经建立了众多的质谱组16。背景质谱下拉样品中可以被定义为任何能够防止识别诱饵蛋白的真实干扰作用。因此,背景可以产生污染的非特异性蛋白质或也较大浓度诱饵蛋白或用来沉淀诱饵蛋白的抗体。显著工作是在优化二者这里提出的下拉协议以及该树脂,以减少在下拉过程中的非特异性的杂蛋白的水平进行。这是显而易见的,在银染的凝胶和控制的质谱分析( 图2)。解决高层次诱饵蛋白,或可以同样有害的污染物,并与其他方法斗争的抗体,用SDS洗脱下拉建议(协议第2.4节)。由于共价连接到树脂上,这个过程会留下大部分的起始融合蛋白的共价连接到树脂上。诱饵的一小部分在质谱分析,这被认为是观察发生通过水解,但它不用于检测其它较弱或瞬时相互作用8提出一个问题。
正如引言中所述,在蛋白质组学显著的进步已经由质谱1,7显著的进步启用。因此,突出质谱分析的选择的重要参数,以解卷积的下拉得到的蛋白质的混合物是重要的。仪器必须可靠并且能够定期地和高效地分析含有少量的蛋白质,通常比<1微克更少的样品。涉及50-75微米内径HPLC柱用的流速在100〜300升/分钟范围内的纳米级色谱通常采用用于与小样本的相容性和最大化质谱仪的灵敏度。为了最大限度地在一个单一的分析状态Ø获得的信息f能够在时间尺度与前述的纳米级分离兼容,≥10赫兹获取高分辨质谱的技术质谱仪,通常采用。这些仪器具有灵敏度attomolar水平,并能经常获得与子ppm的前体和产物离子质量误差容限的数据。这些性能特征,以提高识别的蛋白的数量的产率和与这些标识关联的置信度。
有了这些数据,我们证明生理细胞定位,适当的蛋白质:蛋白质相互作用以及潜在的发现新的相互作用,以及所有使用单个构造活跃的复合物隔离。确的替代技术可用于所有这些不同的方面,但可能不适合所有23,24。用的HaloTag技术,多官能的方法都可以使用,前进蛋白质组学ICS的研究并获得在哺乳动物细胞中的蛋白质功能的更完整的理解。
披露声明
本文刊登费用由Promega公司赞助。达内特L.丹尼尔斯,杰奎·门德斯,埃莱娜Benink,安德鲁·奈尔斯,南希·墨菲和玛列塔乌尔赫是Promega公司,由的HaloTag技术及其应用的专利转让的商业所有者的雇员。迈克尔·福特,理查德·琼斯,拉维阿穆努加马,和大卫艾伦是MSBioworks,它提供了在这个手稿中描述的质谱分析服务的员工。
致谢
我们感谢马丁·罗森伯格博士,加里·塔普利博士和Keith木博士支持这项工作,和詹姆斯卡利博士的手稿的批判性阅读。 DLD,JM,HB,NM,AN,JC,和MU是Promega公司的雇员。 MF,RJ,RA和DA是MS Bioworks,有限责任公司的员工。
材料
| Name | Company | Catalog Number | Comments |
| HeLa Cells | ATCC | CCL-2 | Adherent |
| HEK293T Cells | ATCC | CRL-11268 | Adherent |
| Cellular growth media | Invitrogen/Gibco | ||
| PBS - tissue culture certified | Invitrogen/Gibco | ||
| FuGENE HD Transfection Reagent | Promega | E2311 | |
| HaloTag Control Vector | Promega | G6591 | |
| HaloTag-BRD4 | Kazusa DNA Research Institute | FHC11882 | N-terminal HaloTag fusion |
| HaloTag-HDAC1 | Kazusa DNA Research Institute | FHC02563 | N-terminal HaloTag fusion |
| HaloTag Clones containing content | Promega | Various | http://www.promega.com/FindMyGene/ |
| HaloTag Flexi Vectors for cloning (N- or C-terminal) | Promega | Various | Choose based upon expression level and HaloTag orientation |
| HaloTag TMR Ligand | Promega | G8251 | |
| IGEPAL CA-630 | Sigma | 18896 | |
| HaloTag Mammalian Pull-Down and Labelling System | Promega | G6500 | Contains HaloTag TMR ligand |
| HaloTag Mammalian Pull-Down System | Promega | G6504 | |
| Protease Inhibitor Cocktail, 50X | Promega | G6521 | |
| Mammalian Lysis Buffer | Promega | G9381 | |
| HaloTag Monoclonal Antibody | Promega | G9211 | |
| ProTEV Plus | Promega | V6101 | |
| RQ1 Dnase | Promega | M6101 | |
| HaloLink Resin | Promega | G1912 | |
| HDAC-Glo | Promega | G6420 | |
| PBS + 0.1% Triton X-100 | For optional imaging | ||
| 4% paraformaldehyde/0.2 M sucrose/PBS | For optional imaging | ||
| Ethanol | |||
| Tissue culture vessels and dishes | |||
| Cell culture incubator | |||
| Disposable cell lifter | Thermo Fisher Scientific | 08-773-1 | |
| Glass homogenizer such as Kontes Dounce Tissue Grinder | Thermo Fisher Scientific | K885300-0002 | 25-27 G needle could also be used |
| Microcentrifuge | |||
| Tube rotator such as Labquake | Thermo Fisher Scientific | 4002110Q | Ideal for binding and washing steps |
| Shaking platform such as Eppendorf Thermomixer | Thermo Fisher Scientific | 05-400-200 | Ideal for SDS elution and TEV cleavage steps |
| Chambered cover glass | Thermo Fisher Scientific | 155409 | For optional imaging |
| Fluorescent Microscrope (555 nm Ex/585 nm Em) | For optional imaging |
参考文献
- Altelaar, A. F., Munoz, J., Heck, A. J. Next-generation proteomics: towards an integrative view of proteome dynamics. Nat Rev Genet. 14, 35-48 (2013).
- Tyers, M., Mann, M. From genomics to proteomics. Nature. 422, 193-197 (2003).
- Coulombe, B., et al. Interaction networks of the molecular machines that decode, replicate, and maintain the integrity of the human genome. Mol Cell Proteomics. 3, 851-856 (2004).
- Gavin, A. C., et al. Proteome survey reveals modularity of the yeast cell machinery. Nature. 440, 631-636 (2006).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Sardiu, M. E., et al. Probabilistic assembly of human protein interaction networks from label-free quantitative proteomics. Proc Natl Acad Sci U S A. 105, 1454-1459 (2008).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312, 212-217 (2006).
- Daniels, D. L., et al. Examining the complexity of human RNA polymerase complexes using HaloTag technology coupled to label free quantitative proteomics. J Proteome Res. 11, 564-575 (2012).
- Encell, L. P., et al. Development of a dehalogenase-based protein fusion tag capable of rapid, selective and covalent attachment to customizable ligands. Curr Chem Genomics. 6, 55-71 (2012).
- Ohana, R. F., et al. HaloTag-based purification of functional human kinases from mammalian cells. Protein Expr Purif. 76, 154-164 (2011).
- Black, J. C., et al. KDM4A Lysine Demethylase Induces Site-Specific Copy Gain and Rereplication of Regions Amplified in Tumors. Cell. 154, 541-555 (2013).
- Deplus, R., et al. TET2 and TET3 regulate GlcNAcylation and H3K4 methylation through OGT and SET1/COMPASS. EMBO J. 32, 645-655 (2013).
- Galbraith, M. D., et al. HIF1A Employs CDK8-Mediator to Stimulate RNAPII Elongation in Response to Hypoxia. Cell. 153, 1327-1339 (2013).
- Collins, M. O., Choudhary, J. S. Mapping multiprotein complexes by affinity purification and mass spectrometry. Curr Opin Biotechno. 19, 324-330 (2008).
- Collins, S. R., et al. Toward a comprehensive atlas of the physical interactome of Saccharomyces cerevisiae. Mol Cell Proteomics. 6, 439-450 (2007).
- Mellacheruvu, D., et al. The CRAPome: a contaminant repository for affinity purification-mass spectrometry data. Nat Methods. 10, 730-736 (2013).
- Dawson, M. A., Kouzarides, T. Cancer epigenetics: from mechanism to therapy. Cell. 150, 12-27 (2012).
- Jang, M. K., et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription. Mol Cell. 19, 523-534 (2005).
- Rahman, S., et al. The Brd4 extraterminal domain confers transcription activation independent of pTEFb by recruiting multiple proteins, including NSD3. Mol Cell Biol. 31, 2641-2652 (2011).
- Yang, Z., et al. Recruitment of P-TEFb for stimulation of transcriptional elongation by the bromodomain protein Brd4. Mol Cell. 19, 535-545 (2005).
- Halley, F., et al. A bioluminogenic HDAC activity assay: validation and screening. J Biomol Screen. 16, 1227-1235 (2011).
- Mack, G. S. To selectivity and beyond. Nat Biotechnol. 28, 1259-1266 (2010).
- Daniels, D. L., Urh, M. Isolation of intracellular protein--DNA complexes using HaloCHIP, an antibody-free alternative to chromatin immunoprecipitation. Methods Mol Biol. 977, 111-124 (2013).
- Urh, M., Rosenberg, M. HaloTag a Platform Technology for Protein Analysis. Curr Chem Genomics. 6, 72-78 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。