Method Article
通过捕获Hi-C 破 译高分辨率3D染色质组织
摘要
该协议描述了用于以高分辨率表征超大型靶基因组区域的3D组织的Capture Hi-C方法,包括拓扑关联结构域(TAD)的边界以及调控和其他DNA序列元件之间的长程染色质相互作用。
摘要
基因组的空间组织有助于其在许多情况下的功能和调节,包括转录、复制、重组和修复。因此,了解基因组拓扑和功能之间的确切因果关系至关重要,并且越来越成为深入研究的主题。染色体构象捕获技术(3C)允许通过测量基因组任何区域之间的相互作用频率来推断染色质的3D结构。在这里,我们描述了一种快速简单的协议来执行Capture Hi-C,这是一种基于3C的靶标富集方法,可在高分辨率下表征超大型基因组靶标的等位基因特异性3D组织。在捕获Hi-C中,在下游高通量测序之前,由一系列生物素化探针捕获目标区域。因此,实现了更高的分辨率和等位基因特异性,同时提高了该技术的时间效率和可负担性。为了证明其优势,将Capture Hi-C协议应用于小鼠X灭活中心(Xic),这是X染色体失活 (XCI)的主要调控位点。
引言
线性基因组保存了生物体经历胚胎发育并在整个成年期生存所需的所有信息。然而,指导基因相同的细胞执行不同的功能对于准确控制在特定情况下使用哪些信息至关重要,包括不同的组织和/或发育阶段。基因组的三维组织被认为通过促进或阻止线性基因组中可以相隔几百千碱基的调控元件之间的物理相互作用来参与基因活动的这种准确的时空调控(评论1,2,3).在过去的20年中,我们对基因组折叠和活性之间相互作用的理解迅速增加,这主要是由于染色体构象捕获技术(3C)的发展(综述4,5,6,7)。这些方法测量基因组任何区域之间相互作用的频率,并依赖于细胞核内3D接近的DNA序列的连接。最常见的3C方案从用交联剂(如甲醛)固定细胞群开始。然后用限制性内切酶消化交联染色质,尽管MNase消化也被使用8,9。消化后,空间接近的游离DNA末端被重新连接,交联被逆转。这一步产生了3C"文库"或"模板",这是一个混合的杂交片段池,其中3D接近细胞核的序列有更高的机会连接在同一DNA片段中。这些杂交片段的下游定量能够推断基因组区域的3D构象,这些基因组区域在线性基因组中相距数千个碱基对,但可能在3D空间中相互作用。
已经开发了许多不同的方法来表征3C文库,在分析连接片段的哪些子集以及使用哪种技术进行下游定量方面都有所不同。最初的3C方案依赖于选择两个感兴趣的区域,并通过PCR10,11量化它们的"一对一"相互作用频率。4C方法(环状染色体构象捕获)测量单个兴趣位点(即"视点")与基因组其余部分("一个与所有")之间的相互作用12,13,14。在4C中,3C文库经历第二轮消化和重新连接,以产生小的环状DNA分子,这些分子通过视点特异性引物15进行PCR扩增。5C(染色体构象捕获碳拷贝)能够表征更大感兴趣区域的3D相互作用,从而深入了解该区域内高阶染色质折叠("多与多")16。在5C中,3C文库杂交到重叠限制性内切位点的寡核苷酸池中,随后可以通过具有通用引物15的多重PCR扩增。在4C和5C中,信息丰富的DNA片段最初通过微阵列定量,后来通过下一代测序(NGS)定量17,18,19。这些策略表征了感兴趣的目标区域,但不能应用于绘制全基因组相互作用。后一个目标是通过Hi-C实现的,Hi-C是一种基于3C的高通量策略,其中3C模板的大规模并行测序允许在全基因组水平("全部与全部")对染色质折叠进行无偏表征20。Hi-C方案包括在消化片段的末端掺入生物素化残基,然后用链霉亲和素珠下拉连接片段,以提高连接片段的回收率20。
Hi-C揭示了哺乳动物基因组在3D细胞核中的多个尺度上结构组织。在巨碱基尺度上,基因组分为活性和非活性染色质区域,A和B区室分别为20,21。随后还显示了由不同染色质和活性状态表示的进一步亚区室的存在22。在更高的分辨率下,基因组进一步划分为称为拓扑关联域(TADs)的亚兆碱基自相互作用域,首先通过人类和小鼠基因组的Hi-C和5C分析23,24揭示。与以组织特异性方式变化的隔室不同,TAD往往是恒定的(尽管有许多例外)。重要的是,TAD边界在物种中是保守的25。在哺乳动物细胞中,TADs通常包含具有相同调控景观的基因,并且已被证明代表一种结构框架,该结构框架促进基因共调控,同时限制与相邻调控域的相互作用(综述3,26,27,28)。此外,在TADs中,由于凝聚素挤出环底部的CTCF位点引起的相互作用可能会增加启动子-增强子或增强子-增强子相互作用的可能性(综述29)。
在Hi-C中,可以在1 Mb至40 kb的分辨率下检测隔室和TAD,但可以实现更高的分辨率来表征较小尺度的接触,例如远端元件之间的循环相互作用,分辨率为5-10 kb。然而,提高分辨率以便能够通过HiC有效地检测此类环路需要显着增加测序深度,从而增加测序成本。如果分析需要具有等位基因特异性,则情况会加剧。事实上,分辨率的X倍提高需要测序深度增加X2 ,这意味着高分辨率和等位基因特异性全基因组方法可能非常昂贵30。
为了在保持高分辨率的同时提高成本效益和可负担性,可以在下游测序之前与互补的生物素标记寡核苷酸探针杂交后,从全基因组3C或Hi-C文库中物理提取感兴趣的靶区。这些靶标富集策略被称为Capture-C方法,允许询问分散在基因组中的数百个靶位点的相互作用(即启动子捕获(PC)Hi-C;下一代 (NG) 捕获-C;低输入 (LI) 捕获-C;核滴定 (NuTi) 捕获-C;Tri-C)31,32,33,34,35,36,37,38,39,40,或跨越多个巨型碱基的区域(即捕获HiC;HYbrid Capture Hi-C (Hi-C 2);平铺-C)41,42,43。在基于捕获的方法中,两个方面可能有所不同:(1)生物素化寡核苷酸的性质和设计(即RNA或DNA,捕获分散基因组靶标的单个寡核苷酸或将感兴趣区域平铺的多个寡核苷酸);(2)用于下拉靶标的模板可以是3C或Hi-C文库,后者由从3C文库拉下的生物素化限制性内切片段组成。
本文描述了一种基于3C库中目标触点富集的Capture Hi-C协议。该方案依赖于定制生物素化RNA探针平铺阵列的设计,并且可以在从3C文库制备到NGS测序的1周内完成。该协议快速、简单,允许以 5 kb 分辨率表征兆基大小感兴趣区域的高阶 3D 组织,同时与其他 3C 方法相比提高了时间效率和可负担性。Capture Hi-C方案被应用于X染色体失活(XCI)的主调控位点,即X失活中心(Xic),其宿主Xist非编码RNA。Xic以前一直是广泛的结构和功能分析的主题(审查44,45)。在哺乳动物中,XCI补偿雌性(XX)和雄性(XY)之间X连锁基因的剂量,并涉及雌性细胞中两条X染色体中几乎全部的转录沉默。Xic代表了3D基因组拓扑学研究以及与基因调控44相互作用的强大黄金标准位点。对小鼠胚胎干细胞(mESCs)中Xic的5C分析导致了TAD的发现和命名,为拓扑分区和基因共调控的功能相关性提供了初步的见解24。Xic的拓扑组织随后被证明与Xist上调和XCI46的适当发育时间密切相关,并且最近在Xic 47,48,49中也发现了可以影响TAD内部和之间基因活性的未被怀疑的顺式调节元件。将捕获Hi-C应用于跨越Xic的小鼠X染色体的3 Mb,证明了这种方法在高分辨率下解剖大规模染色质折叠方面的力量。从设计感兴趣区域内每个DpnII限制性位点的生物素化探针阵列开始,到全基因组3C文库的生成、靶标接触的杂交和捕获以及下游数据分析,提供了详细且易于遵循的方案。还概述了适当的质量控制和预期成果,并根据类似的现有方法讨论了该方法的长处和局限性。
研究方案
本研究中使用的小鼠胚胎干细胞(mESCs)来源于TX/TX R26rtTA/rtTA 雌性50 与雄性 Mus musculus castaneus 的杂交,根据居里研究所(巴黎)的动物护理指南51。
1. 探头设计
- 设计一系列覆盖目标区域的生物素化探针(120-mer RNA 寡核苷酸)。
- 用重叠的寡核苷酸平铺感兴趣的区域,以便平均目标中的每个序列都被两个独特的探针覆盖(2倍覆盖率)(图1)。
- 从探针覆盖中排除重复序列,以避免富集非特异性相互作用。
注意:为了最大限度地提高信息连接片段的富集率,定义了靶标上每个DpnII限制性位点上游和下游300 bp的区域(ChrX:102,475,000-105,475,000),并通过Sure Design平台根据SureSelect DNA靶标富集技术设计了28,913个生物素化探针52.根据该策略,每个寡核苷酸中最多允许40个碱基的重复序列,以最大程度地减少非特异性相互作用的富集。探针阵列由安捷伦合成。在这里,DpnII用作限制性内切酶有两个原因:(1)它是一种四刀,常规用于几种基于3C的方法53;(2)与本研究中使用的F1杂交 系( C57BL / 6J x CASTEi / J)中计算机测试的其他限制性内切酶相比,它最大限度地提高了在切割位点附近捕获信息性单核苷酸多态性(SNP)的机会。
2. 实验程序
- 细胞制备
- 在一个或多个细胞培养板上接种适当数量的细胞,以在固定当天获得≥ 5 x 107 个细胞的总细胞数。
注意:本研究使用了小鼠胚胎干细胞(mESC)。将mESCs接种在含有2i + LIF和批量测试胎牛血清(DMEM,15%FBS,0.1mM β-巯基乙醇,1,000U / mL−1白血病抑制因子(LIF),CHIR99021(3μM)和PD0325901(1μM)的mESC培养基中的糊化(0.1%明胶在1x PBS - o / n中),CHIR99021(3μM)和PD0325901(1μM))。对于这种细胞类型,一个80%汇合的10cm板包含大约2 x 107 个细胞。 - 准备一个额外的细胞培养板用于细胞计数。
注意:可以使用较小的细胞培养板来减少培养基的使用。在这种情况下,需要相应地调整较小平板上接种的细胞数量(例如,与15 cm平板相比,10 cm平板上的细胞数量减少了3倍)。
- 在一个或多个细胞培养板上接种适当数量的细胞,以在固定当天获得≥ 5 x 107 个细胞的总细胞数。
- 甲醛固定
- 估计要交联的细胞总数。
- 在开始交联反应之前,根据制造商的说明,使用自动细胞计数器对专门准备用于细胞计数的对照板中的细胞进行胰蛋白酶消化和计数。
- 包括活力染色(例如台盼蓝)以确定活细胞54的百分比。根据该细胞计数,估计准备用于交联的平板中的细胞总数。
- 从准备用于交联的平板中取出培养基,并用适量的固定溶液(细胞培养基中的2%甲醛)代替。在 10 cm 平板上使用 10 mL(例如,15 cm 平板使用 ~20 mL)。
注意:添加精确体积的固定溶液。如果无法固定贴壁细胞,则可以将此步骤适用于胰蛋白酶消化的细胞,并在50 mL锥形离心管中的30 mL固定溶液中进行。甲醛不得超过1年。最好使用一次性小瓶。固定溶液在使用前必须达到室温(RT)。
注意:甲醛是危险的,需要根据适当的健康和安全法规进行处理。 - 在室温下在摇床上轻轻混合下固定 10 分钟。
- 通过加入 2.5 M 甘氨酸-1x PBS 至终浓度为 0.125 M 来淬灭固定反应。 在 10 cm 板上将 530 μL 2.5 M 甘氨酸-1x PBS 加入 10 mL 中(例如,在 15 cm 板上加入 1060 μL 至 20 mL)。
注意:如果将细胞固定在溶液中,则用1590μL的2.5M甘氨酸-1x PBS淬灭固定反应。 - 在室温下孵育5分钟,在摇床上轻轻混合。
- 将板转移到冰上,在冰上再孵育15分钟,同时在摇床上轻轻混合。
注意:从现在开始,细胞必须保存在冰上,缓冲液必须预冷以避免进一步交联。如果需要处理许多板,请移至冷藏室。 - 通过将固定溶液倒入烧杯中,从细胞中取出固定溶液,以确保快速处理。
注意:确保根据适当的健康和安全法规处理含甲醛的液体废物。 - 用 5 mL 冷的 0.125 M 甘氨酸-1x PBS(15 cm 板为 8 mL)快速冲洗 10 cm 板两次,以洗去碎片和死细胞。通过将液体倒入烧杯中来去除板中的液体,以确保快速处理。
- 将 5 mL 冷的 0.125 M 甘氨酸-1x PBS 加入 10 cm 平板(15 cm 平板为 10 mL),并使用塑料细胞刮刀快速从平板上刮下细胞。
- 使用血清移液器将细胞悬液转移到预冷的 50 mL 锥形离心管中。
- 用5 mL冷的0.125 M甘氨酸-1x PBS冲洗板两次,并将细胞悬液加入锥形离心管中。
- 在4°C下以480× g 旋转10分钟。
注意:如果将细胞固定在溶液中,则将细胞转移到预冷的锥形离心管中,并在4°C下以480× g 旋转10分钟。 通过将固定溶液倒入烧杯中除去固定溶液,并在10mL冷的0.125M甘氨酸-1x PBS中洗涤三次。确保在每个洗涤步骤中重悬细胞。 - 用台式抽吸系统吸出去除上清液。用 P1000 移液器小心地上下移液,将细胞重悬于每 1 x 107 个细胞的 500 μL 1x PBS 中。要以准确的体积重悬细胞,请参阅2.2.1中获得的总细胞数估计值。
- 将 500 μL 细胞悬液等分到计算数量的 1.5 mL 微量离心管(1 x 107 个细胞/管)中。
- 在4°C下以480× g 旋转10分钟。
- 用台式抽吸系统除去上清液,并在液氮中快速冷冻细胞沉淀。将干细胞颗粒储存在-80°C。
注意:样品可以保存至少 1 年。
- 估计要交联的细胞总数。
- 细胞裂解
- 将冷冻颗粒在冰上解冻。
- 每个样品在 H20 中制备 1.5 mL 裂解缓冲液:加入 10 mM Tris-HCl、pH 8.0、10 mM NaCl 和 0.2% NP40。
- 加入 600 μL 冷裂解缓冲液,并在冰上重悬。
- 在冰上孵育15分钟,让细胞膨胀。
- 在4°C下以2655× g 旋转5分钟,并使用台式抽吸系统除去上清液。
- 为了去除碎片,将沉淀重悬于1mL冷裂解缓冲液中,在4°C下以2655× g 旋转5分钟,然后除去上清液。
- 在2655× g 和4°C下再次旋转,并使用装有P200吸头的台式抽吸系统尽可能多地去除剩余的上清液。
- 重悬于 100 μL 0.5%(体积/体积)SDS 中。
- 在62°C的热混合器中孵育,以1400rpm旋转10分钟。
- 加入 290 μL H2O + 50 μL 10% TritonX-100 并充分混合,避免气泡。
- 在37°C的热混合器中孵育,以1400rpm旋转15分钟。
- 加入 50 μL 10x Dpnll 缓冲液并倒置试管进行混合。
- 取 50 μL 未消化的 DNA 进行质量控制,放入单独的试管中。不要忘记取未消化的对照样品。
- DpnII消化
- 加入 10 μL Dpnll 高浓度(总共 500 U)并通过倒置混合。
- 将样品和未消化的对照在37°C的热混合器中孵育,以1400rpm旋转>4小时。
- 在一天结束时加入 10 μL Dpnll 高浓度(总共 500 U)。
- 将样品和未消化的对照在37°C孵育,以1400rpm旋转过夜。
- 第二天开始时向样品中加入 10 μL Dpnll 高浓度(总共 500 U)。
- 将样品和未消化的对照在37°C的热混合器中孵育,以1400rpm旋转4小时。
- 交联的连接和逆转
- 将试管在65°C以1400rpm孵育20分钟。
注意:此时不要添加 SDS。这个想法是为了保持核完整性,因此连接是在细胞核内进行的,避免了极端稀释的需要。 - 将样品在冰上冷却最多5-10分钟。为避免SDS沉淀,请勿将样品留在冰上超过此时间。
- 取 50 μL 未连接的消化 DNA 在单独的管中进行质量控制。将未消化和未连接的对照储存在-20°C。
注意:不要忘记采集未连接的对照样品。 - 加入 800 μL 连接混合物:122 μL 10x 连接酶缓冲液、8 μL T4 连接酶 (30 U/μL) 和 670 μL H20。
- 在16°C孵育,以1000rpm旋转过夜。
- 向样品中加入 7.5 μL 蛋白酶 K (20 mg/mL),向对照中加入 2 μL。
- 在65°C下以1000rpm孵育4小时。
- 将试管在65°C以1400rpm孵育20分钟。
- 脱氧核糖核酸纯化
- 将冰上的样品转移到预冷的 15 mL 锥形离心管中,加入 2 mL 水、10.5 mL 冰冷的 EtOH 和 583 μL 3 M NaAC。
注意:额外的水旨在防止DTT带入颗粒中。 - 向未消化和未连接的质控品中加入 200 μL 冰冷的 EtOH、10.8 μL NaAC 和 1 μL 共沉淀剂。
- 在-80°C孵育至少4小时至过夜。
- 在4°C下以2200× g 旋转15mL管45分钟。
- 将1.5 mL对照管在4°C下以20,500 x g 旋转30分钟。
- 用 3 mL(样品)和 1 mL(对照)冰冷的 70% EtOH 洗涤一次。
- 在4°C下以2200×g(样品)或20,500 ×g(对照)旋转10分钟。
- 小心地去除EtOH并在室温下风干10-15分钟;不要过度干燥。
- 将样品和对照分别重悬于 100 μL 和 20 μL H20 中。
- 加入 1 μL RNAseA 并在 37 °C 下孵育,以 1400 rpm 旋转 30 分钟。
- 将冰上的样品转移到预冷的 15 mL 锥形离心管中,加入 2 mL 水、10.5 mL 冰冷的 EtOH 和 583 μL 3 M NaAC。
- 3C模板制备的质量控制
- 使用荧光计试剂盒定量每个样品和对照,以进行高灵敏度 DNA 浓度测量。
- 在 1% 琼脂糖/1x TBE 凝胶上加载 100-200 ng 的每个样品和每个对照。
- 通过比较对照和3C模板的DNA片段大小差异,验证凝胶图像是否显示了预期结果,如图 2A所示。
- 将样品和对照储存在-20°C。
- 用于多重测序的杂交、捕获和样品处理
- 要将生物素化RNA探针阵列与3C模板杂交,捕获靶向连接片段,并根据本研究中使用的靶标富集系统制备样品以进行多重测序( 目录).根据制造商的说明遵循协议,同时进行以下小修改:
- 制造商协议第2部分:样品制备
- 按照说明从 3 μg gDNA 起始进行靶标富集。
- 使用以下规格在超声仪中剪切DNA:10%占空比,4强度,200 cyc/突发和130 s。首先将 4 μg 3C 模板重悬于 130 μL 水中进行每次捕获反应,以确保有足够的材料继续使用 3 μg 剪切的 DNA 进行样品制备。
- 评估剪切DNA的质量。根据高灵敏度方案在 DNA 生物分析仪上运行 1 μL 剪切的 DNA。预计片段大小分布在150-700 bp之间(图2)。
- 使用固相可逆固定化(SPRI)珠纯化样品。根据制造商的说明,将 124 μL SPRI 磁珠加入 124 μL DNA 样品中,进行 1:1 左侧尺寸选择,并在 25 μL 无核酸酶水中洗脱。该纯化步骤将去除较短的片段,以富集约300 bp的片段(图2)。
注意:此步骤中使用的样品和SPRI微球的数量考虑了将样品转移到新管并在生物分析仪上运行质量控制时发生的体积损失。所有后续尺寸选择步骤均根据制造商协议推荐的比例执行。在整个实验方案中,SPRI磁珠的DNA洗脱在室温下进行。 - 评估大小选择的剪切DNA的质量。根据高灵敏度 (HS) 方案在 DNA 生物分析仪上运行 1 μL 剪切的 DNA。预期片段大小分布在300 bp时富集度最高(图2)。如果剪切成功,请继续对剪切的DNA进行定量。
- 使用荧光计试剂盒定量剪切的 DNA,用于 HS DNA 浓度测量。
注意:如果DNA剪切导致DNA产量为<3μg,请用另外4μg的DNA进行第二轮DNA剪切,并在第一个SPRI磁珠纯化步骤后合并剪切的DNA样品,以获得总共3μg的剪切DNA。 - 将无核酸酶的水加入到尺寸选择的清洁DNA样品(总共3 μg)中,最终体积为48 μL,并根据制造商的方案进行最终修复反应。
- 连接配对端接头后,根据制造商的说明(试剂盒中提供了PCR的条件)进行五次预捕获PCR循环,从而扩增文库。
- 制造商协议第4部分:杂交和捕获
- 要将制备的 DNA 样品与靶标特异性 RNA 探针杂交,以 3.4 μL 的最终体积稀释 750 ng 的 DNA 样品,初始浓度为 221 ng/μL。对于以较大体积稀释的 DNA 样品,请使用速度真空浓缩器减少到最终体积。速度真空浓度(250 x g ≤45 °C)持续15-20分钟通常足以使重悬于10 μL中的样品。 在启动速度真空浓缩器之前,请确保每个样品的输入体积相同。
- 根据制造商的说明,将杂交混合物在65°C下用105°C的加热盖孵育16-18小时。
- 制造商协议第 5 部分:多重测序的索引和样品处理
- 要使用索引引物扩增捕获的文库,请根据制造商的说明执行12个捕获后PCR循环(试剂盒中提供了PCR和引物的条件)。
- 制造商协议第2部分:样品制备
- 要将生物素化RNA探针阵列与3C模板杂交,捕获靶向连接片段,并根据本研究中使用的靶标富集系统制备样品以进行多重测序( 目录).根据制造商的说明遵循协议,同时进行以下小修改:
- 二代测序
- 要在同一流通池上运行多个捕获 Hi-C 文库,请准备捕获文库的等摩尔混合物,并对每个谱库 100-120 M 读数进行测序。
- 如果需要等位基因特异性分析,则对 150 bp 配对末端进行测序,以确保足够的 SNP 覆盖率。
3. 数据分析
- 应用 HiC-Pro 管道执行捕获 Hi-C 数据分析55.HiC-Pro在处理的每个步骤中提供质量控制,包括(图3):
(一)参考基因组上的比对率,指定跨越连接位点的读段比例,以及配对和单例的数量。
(二)有效连接产物和非信息读对(悬垂端、自连接等)的比例。
(三)短距离/长距离和染色体内/染色体间接触的比例。
(四)捕获 Hi-C 的目标联系人的比例。
(五)等位基因特异性读取的比例(如果指定)。
注意:HiC-Pro 支持多种协议,包括原位 Hi-C 和 Capture Hi-C。在后一种情况下,用户只需在配置文件中指定目标区域(BED 格式)。处理完数据后,HiC-Pro输出可以很容易地转换为较冷的对象,用于下游分析56。在此步骤中,使用Imakaev及其同事先前描述的ICE方法对各种分辨率的接触图进行标准化57。然后可以运行几种分析来调用染色体区室,TAD或染色质环(用于综述58)。该协议的工作流程如图 4 所示。在这里,"cooltools"套件用于计算绝缘分数和TADs边界,如图5和图6所示 59。
结果
所描述的捕获Hi-C方案基于使用四碱基切割器(DpnII)制备全基因组3C模板。根据本研究中使用的靶标富集系统,通过杂交一系列平铺RNA探针及其基于链霉亲和素的捕获,获得了跨感兴趣基因组区域的连接片段的后续富集(图1)。之所以选择生物素化RNA探针,是因为与DNA探针52,60相比,它们与靶标的结合亲和力更强。然后对捕获的文库进行索引和汇集,以进行多路复用的高通量测序。捕获Hi-C数据可以可视化为高分辨率Hi-C相互作用图,也可以可视化为类似4C的单视点接触图,以专门可视化整个捕获区域内较小序列(如启动子或增强子)的相互作用。该协议的工作流程如图 4 所示。测序前质控品如图 2 所示,包括评估3C模板的正确消化和重新连接及其在方案不同步骤中的高效剪切和纯化。剪切后的3C模板DNA预计在150至700 bp之间运行,并且不应检测到富集的片段>2 kb。在以下步骤中,首先在剪切后,然后在捕获前和捕获后PCR之后执行几个基于磁珠的DNA纯化和大小选择步骤。 清洁后的文库显示出在高灵敏度DNA生物分析仪上可视化的独特片段富集谱(图2)。由于衔接子的连接、测序和索引引物,平均片段大小在文库制备过程中增加。测序后质量控制通过Hi-C Pro 获得 ,如图 3所示。已经提出了许多不同的生物信息学软件应用程序,用于类似3C的数据处理和分析。其中,HiC-Pro流水线是最受欢迎的解决方案之一,允许以各种分辨率55将原始测序数据处理为最终接触图。HiC-Pro使用两步定位策略来对齐参考基因组上的测序读数。然后重建并过滤掉3C产品,以去除无信息的接触对并生成接触图。此外,它能够使用已知多态性列表来执行等位基因特异性分析,并在不同的接触图中分离来自两个亲本等位基因的接触。最近,HiC-Pro已被纳入并扩展到nf-core框架(nf-core-hic)中,提供了一个高度可扩展和可重现的社区驱动管道61,62。
为了捕获小鼠Xic,设计了一个由28,913个RNA探针组成的阵列,平铺3 Mb的X染色体。该区域包括XCI中的关键参与者,长非编码基因Xist及其已知的~800 kb调控环境(图5)。这个~800 kb区域被划分为两个TAD:一个包括Xist启动子及其已知的阳性调节因子(即,非编码转录本Ftx,Jpx和Xert以及蛋白质编码基因Rnf12),以及包含Xist负顺式调节因子(即其反义转录本Tsix,增强子元素Xite和非编码转录本Linx)的相邻TAD(用于审查44, (第45页)。
通过将所描述的捕获Hi-C协议应用于 Xic, 以前所未有的分辨率获得了该位点的拓扑组织(图6 和 图7)。将采集 Hi-C 配置文件与之前发布的 5C47 进行比较时,这一点尤其明显(图 6 和 图 7; 补充表1)和 Hi-C61 (图 6 和 图 7; 补充表1)配置 文件。例如,亚TAD结构更为明显 - 包含Xist启动子( Xist-TAD )的TAD明显细分为两个较小的结构域(图6A,蓝色箭头)。以前,这只能从5C剖面(图6B)中目视"猜测",尽管使用绝缘评分算法检测该区域的边界。同样,捕获Hi-C谱的分辨率允许识别相邻TAD中的两个较小的结构域(图6A, B),其中包含Tsix位点( Tsix-TAD )的启动子;这在以前是用5C实现的(图6B)。值得注意的是,由Capture Hi-C和5C数据的绝缘分数确定的拓扑边界通常在略有不同的位置和不同的相对强度处检测到。
此外,从Capture Hi-C数据中可以清楚地看到其他子TAD结构,例如接触环路,例如Xist和Ftx之间的环路(图7A),之前用Capture-C63标识,以及Xist和Xert之间的环路(图7B),最近使用类似的协议识别了捕获Hi-C 48。.由于捕获Hi-C配置文件的分辨率提高,其他接触也可以更精确地映射,例如在Linx,Chic1和Xite位点之间的Tsix-TAD中形成已知接触热点的触点(图7A)。
与 图7所示的Hi-C数据相比,捕获Hi-C使分辨率提高了四倍,但它只需要四分之一的测序深度(即126 M读数对571 M读数)(补充表1)。分辨率的提高允许检测Hi-C在图 6 和 图7所示的测序深度无法检测到的亚TAD和循环相互作用。因此,与以前的方法相比,所描述的Capture Hi-C协议允许对感兴趣的大基因组区域进行更详细,高分辨率的表征。
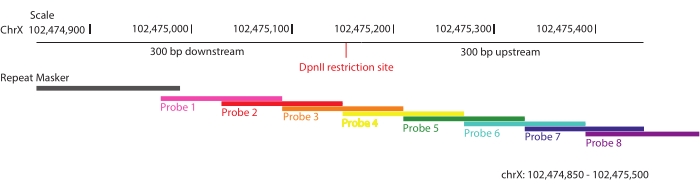
图 1:探头设计。用于探头设计的策略的示意图。 选择跨3 Mb靶区的每个DpnII限制性位点上游和下游300 bp的区域,并用重叠的生物素化RNA探针平铺。显示其中一个选定区域,chrX:102,474,805-102,475,500。每个探针中允许的重复序列碱基不超过40个。 请点击此处查看此图的大图。
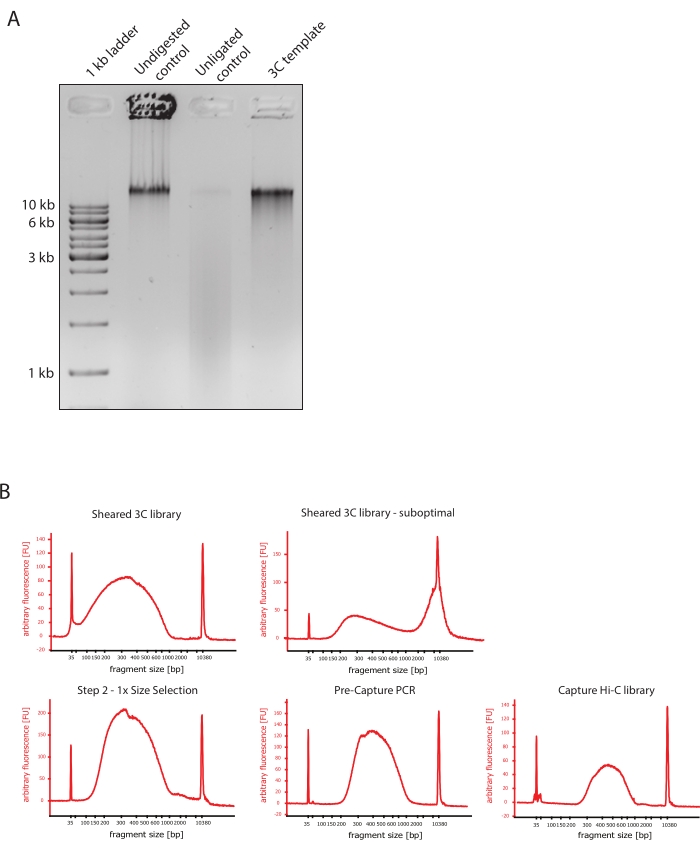
图 2:捕获 Hi-C 预测序质量控制 。 (A)3C模板质量控制的代表性实例。将200 ng DNA上样于1%琼脂糖凝胶上。车道 1:1 kb 梯子。泳道 2:未消化、交联和完整的染色质在 >10 kb 处以尖锐的条带形式运行。泳道 3:DpnII 消化的交联染色质以 1 kb 至 3 kb 大小的涂片形式运行。通道4:最终3C库或模板;重新连接消化的交联DNA片段的自由末端。较低分子大小的DNA涂片几乎检测不到,连接产物检测为>10 kb的条带。(B)高灵敏度生物分析仪DNA图谱的代表性例子。左上:成功剪切的3C文库显示片段大小分布在150 bp和700 bp之间。右上:不满意的剪切3C库。未剪切的DNA被检测为片段的广泛富集>2 kb。(C) 左下角:使用 SPRI 磁珠进行 1:1 左侧尺寸选择后的剪切 DNA 样品。~300 bp的片段被富集。底部中间:根据制造商的方案连接配对端接头后预捕获PCR谱。右下:最终的 Capture Hi-C 文库,包括用于多重测序的衔接子、测序和索引引物。缩写:bp = 碱基对,FU = 任意荧光单位。 请点击此处查看此图的大图。
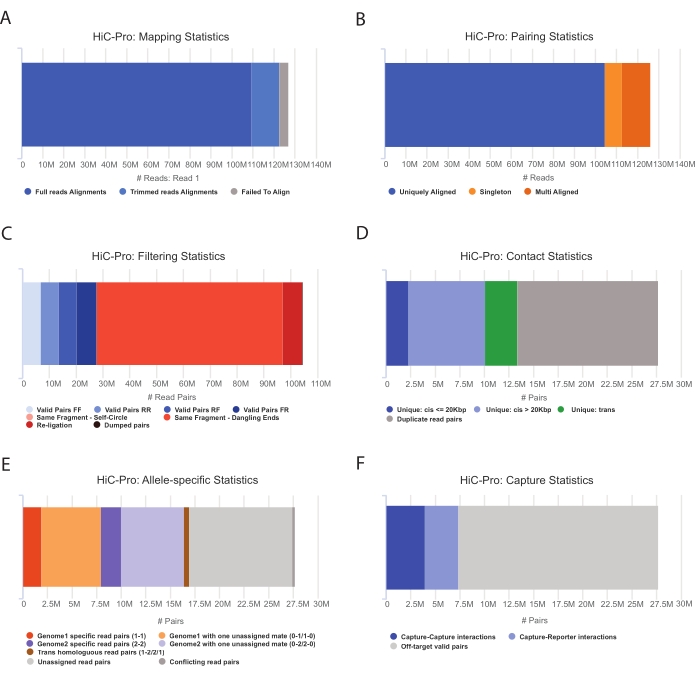
图 3:使用 HiC-Pro 捕获 Hi-C 测序后质控品 。 (A) 测序对第一配偶的参考基因组映射率示例。浅蓝色部分表示由HiC-Pro对齐并跨越连接结的读数。因此,该指标可用于验证实验连接步骤。(B)一旦测序伙伴在基因组上比对,只保留唯一比对的读对进行分析。(C) 从分析中丢弃无效对(红色),例如悬端、自圆或重新连接。有效对的分数是连接和下拉效率的良好指标。(D)有效对可进一步分为染色体内/染色体间和短距离/长距离接触。可能代表PCR伪影的重复读对将从分析中丢弃。(E)对于等位基因特异性分析,HiC-Pro报告每个亲本基因组(即C57BL / 6J x CASTEi / J)的一个或两个配偶支持的等位基因读取数。预计分配给母系和父系等位基因的读段比例相同。(F)最后,仅选择与捕获区域重叠的有效对来构建联系人映射。捕获-捕获对表示目标区域内的接触,而捕获-报告器对涉及目标区域与脱靶区域之间的交互。 请点击此处查看此图的大图。
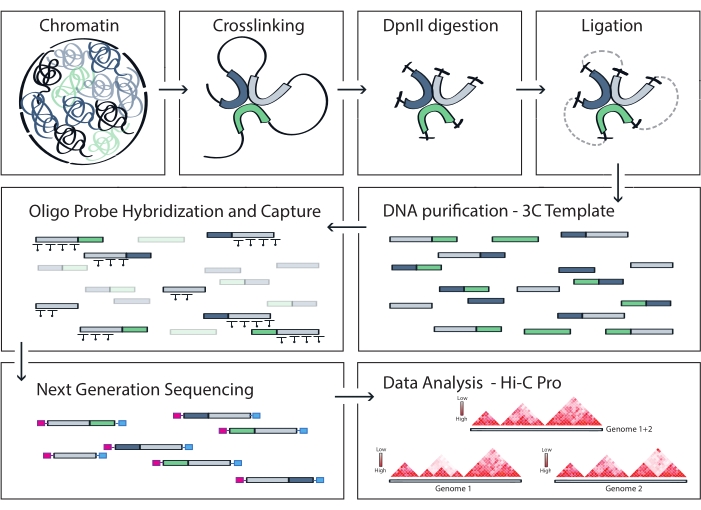
图 4:采集 Hi-C 协议的工作流程。 不同协议步骤的示意图。为了生成全基因组3C模板,染色质首先与甲醛交联,然后用DpnII限制性内切酶消化。然后游离的DNA末端被重新连接,交联被逆转,DNA被纯化。为了富集包含靶区域的片段,将一系列生物素化RNA探针杂交到3C模板中,并通过链霉亲和素介导的下拉捕获。处理捕获文库以进行多重测序,并对有效的连接片段进行定量,以推断靶标上染色质接触的频率,这些频率可视化为高分辨率相互作用图。 请点击此处查看此图的大图。
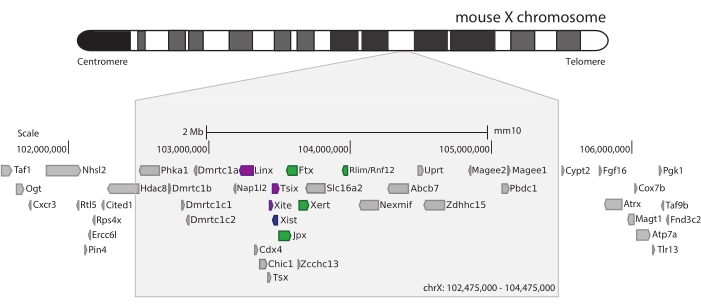
图 5:包含小鼠 X 染色体上的 Xic 的区域概述。 鼠标X染色体的示意图和3 Mb捕获区域的放大(ChrX:102,475,000-105,475,000)。靶区包括~800 kb对应于Xic的DNA, Xic是XCI的主要调控位点。Xic包括长非编码基因Xist, XCI 的关键参与者,及其监管环境。Xist的正调节剂以绿色显示,负调节剂以紫色显示。 请点击此处查看此图的大图。
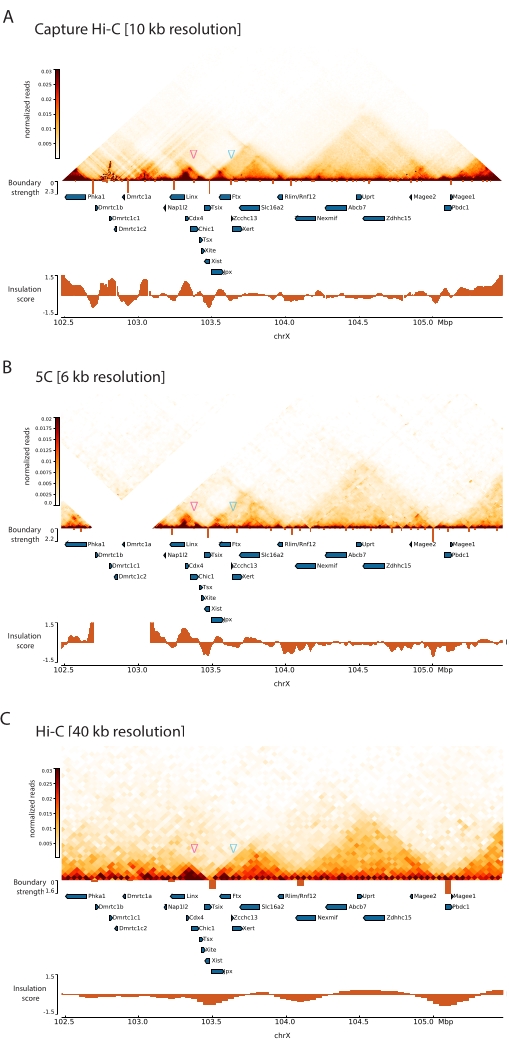
图 6:捕获 3 Mb 捕获区域的 Hi-C、5C 和 Hi-C 交互图。 (A) 以 10 kb 分辨率捕获包含鼠标 Xic 的 3 Mb 目标的 Hi-C 交互图(本研究)。(B)6 kb分辨率下与A中相同目标区域的5C交互图(从47重新处理的数据)。未包含在分析中的重复区域用白色掩盖。5C数据需要自己的生物信息学处理(见47)。清洁和对齐后,使用运行中位数(窗口= 30 kb,步骤= 5)对引物分辨率的5C图进行分箱,以达到6 kb的最终分辨率。(C)在40 kb分辨率下与A和B相同的基因组区域的Hi-C相互作用图(数据从64重新处理)。所有交互映射都是从鼠标ESC生成的。绝缘分数是使用冷却工具计算的,并以直方图表示,在TAD边界处具有绝缘最小值。TAD 边界在地图下方显示为垂直线。每条线的高度表示边界强度。基因显示为指向转录方向的箭头。在捕获 Hi-C 地图中专门或更精确地检测到的子 TAD 边界分别由 Tsix 和 Xist TAD 中的子 TAD 的洋红色和蓝色箭头表示。请点击此处查看此图的大图。
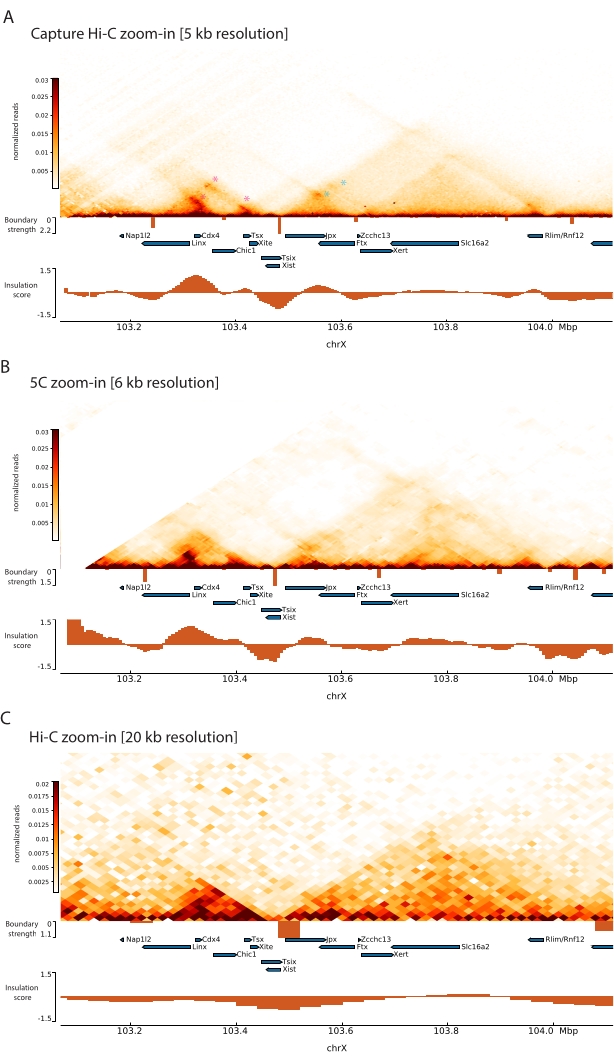
图 7:捕获区域内 1 Mb 的 Hi-C、5C 和 Hi-C 交互图。 (A)以5 kb分辨率捕获包含小鼠Xic的1 Mb基因组区域的Hi-C相互作用图(本研究)。(B)与A相同的基因组区域的5C相互作用图。分辨率为 6 KB(数据从47 重新处理)。未包含在分析中的重复区域用白色掩盖。值得注意的是,5C数据需要自己的生物信息学处理(见47)。清洁和对齐后,使用运行中位数(窗口= 30 kb,步骤= 5)对引物分辨率的5C图进行分箱,以达到6 kb的最终分辨率。(C)在20 kb分辨率下与Hi-C的A和B相同的基因组区域的Hi-C相互作用图(数据从64重新处理)。所有交互映射都是从mESC生成的。绝缘分数是使用冷却工具计算的,并以直方图表示,在TAD边界处具有绝缘最小值。TAD 边界在地图下方显示为垂直线。每条线的高度表示边界强度。基因显示为指向转录方向的箭头。在 Capture Hi-C 中专门或更精确地检测到的接触环分别由洋红色和蓝色星号表示,表示 Tsix 和 Xist TAD 中的环路。请点击此处查看此图的大图。
补充表1:本手稿中使用的数据集的测序后统计数据:捕获Hi-C(本研究),Hi-C64和5C47。请点击此处下载此文件。
讨论
在这里,我们描述了一种相对快速和简单的Capture Hi-C协议,以5-10 kb分辨率表征兆碱基大小基因组区域的高阶组织。Capture Hi-C属于Capture-C技术家族,旨在富集全基因组3C或Hi-C模板的靶向染色质相互作用。迄今为止,绝大多数Capture-C应用已被用于绘制分散在整个基因组中的相对较小的调控元件的染色质接触。在第一个Capture-C方案中,使用多个重叠的RNA生物素化探针在由红系细胞制备的3C文库中捕获>400个预选启动子31。随后在下一代 (NG) 和核滴定 (NuTi) 捕获-C 中改进了相同的策略,通过使用跨越单个限制性位点的单个 120 bp DNA 诱饵和两轮连续捕获,实现 >8,000 个启动子的高分辨率相互作用谱,以最大限度地提高信息连接片段的富集32,40.这些策略导致在许多不同的背景下对顺式作用元件进行功能解剖,包括小鼠胚胎发育,细胞分化,X染色体失活和病理条件下的基因错误调节46,63,65,66,67,68,69,70,71。
在启动子捕获Hi-C(PCHi-C)中,通过在限制性内切片段34,72的两端杂交单个RNA 120-mer生物素化探针,从Hi-C文库中拉下>22,000个含有限制性内切酶片段的注释启动子。该方法允许解剖快速增长的细胞类型中数千个启动子的相互作用组,包括小鼠胚胎干细胞,胎儿肝细胞和脂肪细胞34,35,72,73,以及人淋巴母细胞系,造血祖细胞,表皮角质形成细胞和多能细胞37,74,75,76,77.
与这些靶标富集技术相比,Capture Hi-C靶向连续的基因组区域,最高可达兆碱基规模,从而跨越一个或多个TAD,并涵盖基因的调控环境。整个感兴趣区域必须用一系列生物素化探针平铺,这些探针包含靶标内的每个DpnII限制性位点。生物素化阵列与3C模板的杂交,随后基于链霉亲和素的捕获以及多重测序的处理使用Illumina配对端多重测序的靶标富集系统进行。整个实验方案速度很快,从3C文库制备到NGS测序只需1周即可完成,并且只需要稍作调整和/或定制特定的故障排除。
与其他基于3C的方法相比,该协议还具有优势。为了获得分辨率为5-10 kb的交互图,我们对100-120 M配对端读取进行了测序。作为比较,我们在这里使用了 571 M 读取的 Hi-C 数据集来达到 20 kb 分辨率64 (GSM2053973),并且至少需要 10 亿次读取才能达到染色体宽Hi-C 22 的 5 kb 分辨率。
本研究中使用的捕获Hi-C比先前发表的基于6 bp切割限制性内切酶47 的5C分辨率高得多(补充表1)。重要的是,旨在丰富和扩增5C中靶向相互作用的策略不允许对染色质相互作用进行等位基因特异性分析。相反,Capture Hi-C数据可以特异性地映射等位基因,从而允许解剖成对同源染色体的3D结构景观,例如在人类细胞或通过杂交遗传不同的小鼠品系衍生的F1杂交细胞系中78。为了生成5 kb分辨率的等位基因特异性Capture Hi-C相互作用图,我们对150 bp配对末端读取进行了测序,以增加SNP覆盖率。类似的等位基因特异性方法可以应用于人类细胞系,SNP的注释是可用的22。
重要的是,尽管Capture Hi-C通常可确保高分辨率,同时提高测序成本的可承受性,但定制生物素化寡核苷酸的生产确实会影响该方法的总体成本。因此,对于不同的应用,最合适的3C方法的选择会有所不同,并且取决于正在解决的生物学问题和所需的分辨率,以及感兴趣区域的大小。开发的其他 Capture Hi-C 协议与此处描述的协议具有相同的关键功能。例如,应用Capture Hi-C策略来表征~50 kb至1 Mb基因组区域,这些基因组区域跨越与乳腺癌和结直肠癌风险相关的非编码变异;在该协议中,通过杂交 120 聚体 RNA 诱饵以 3 倍覆盖率33,38,79 平铺目标区域,从 Hi-C 文库中拉下目标区域。同样,HYbrid Capture Hi-C(Hi-C 2)用于定位感兴趣区域内高达2 Mb80的相互作用。在这两种方案中,与我们的方案相比,使用富含生物素下拉连接片段的Hi-C模板增加了总信息读段的百分比。例如,在我们用于比较64 (GSM2053973) 的 Hi-C 数据集中,删除重复项后的有效对百分比比在 Capture Hi-C 中获得的有效对高 4.8 倍,如图 3 和补充表 1 中所述。然而,生物素化连接片段和杂交探针的连续下拉使该方案更加复杂和耗时,同时可能降低捕获区域的复杂性。
另一种用平铺探针富集3C模板的可用方法是Tiled-C,它被用于在小鼠红系分化过程中以高空间和时间分辨率研究染色质结构43。在Tiled-C中,一组70 bp生物素化探针用于在连续两轮捕获中富集大规模区域内的接触,以生成靶向相互作用的高分辨率图谱43,81。与Capture Hi-C相比,双重捕获富集也使协议更长,更复杂。然而,与针对单个限制性位点的Capture-C策略不同,在Tiled-C中,第二轮捕获似乎并没有显着提高捕获效率,因此可能可以省略43。最后,基于本研究中使用的相同靶标富集策略的类似平铺方法应用于解剖调控景观,包括先天性畸形患者中描述的结构变异,并在转基因小鼠中重新设计41,42。在这种情况下,探针的平铺阵列是在整个目标上设计的,而不是在DpnII限制性位点41附近设计的。尽管如此,这项工作在强调该策略在不同背景下实现大型基因组区域的高分辨率表征的敏感性和能力方面具有开创性意义41,42,48。
总之,这里描述的协议代表了一种简单、稳健且强大的策略,用于对任何感兴趣的基因组区域进行高分辨率 3D 表征。将这种方法应用于不同的模型系统,细胞类型,发育调节的染色质景观以及健康和病理条件下的基因调控,可能有助于我们理解基因组拓扑和基因调控之间的相互作用和因果关系,这是表观遗传学领域的基本开放问题之一。此外,应用Capture Hi-C来绘制GWAS研究确定的风险变异的远程相互作用和高阶染色质折叠,有可能揭示在不同背景下与人类疾病相关的非编码基因组位点的功能相关性,从而为潜在的潜在发病机制过程提供新的见解。
披露声明
Kai Hauschulz 是安捷伦科技公司诊断和基因组学事业部的现场应用科学家。所有其他作者声明没有竞争利益。
致谢
赫德实验室的工作得到了欧洲研究委员会高级研究员奖(XPRESS - AdG671027)的支持。A.L.得到了欧盟玛丽·斯克沃多夫斯卡-居里行动个人奖学金(IF-838408)的支持。A.H.得到了ITN创新和跨学科网络ChromDesign的支持,根据Marie Skłodowska-Curie赠款协议813327。作者感谢Daniel Ibrahim(MPI分子遗传学,柏林)提供的有用的技术建议,感谢居里研究所(巴黎)的NGS平台,感谢弗拉基米尔·贝内斯(Vladimir Benes)和EMBL的基因组学核心设施(海德堡)的支持和帮助。
材料
| Name | Company | Catalog Number | Comments |
| 10x PBS pH 7.4 | Gibco | 10010-023 | |
| 37% (vol/vol) paraformaldehyde solution | Electron Microscopy Sciences | 15686 | single use glass-vials; do not reuse |
| 50 mL PP conical tube | Falcon | 352070 | |
| Agarose | Sigma | A9539-500g | |
| Bioanalyzer | Agilent | G2939BA | |
| Cell Scrapers - 25 cm Handle and 3.0 cm Blade | Falcon | 353089 | |
| CHIR99021 | Axon Medchem BV | Axon 1386 | |
| cOmplete Mini, Protease inhibitor cocktail (EDTA-free) | Merck | 11836170001 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL | Invitrogen | ZGEXSCCOUNTESS2FL | Automated cell counter |
| Covaris S2 | Covaris | 500217 | Sonicator |
| DNA LoBind tube, 1.5 mL | Eppendorf | 30108051 | |
| DpnII (50000 units/mL) | New England Biolabs | R0543M | |
| Dulbecco's Modified Eagle Medium (DMEM) | Merck | D6429 | |
| Ethanol (100%) | Merck | 1.00983.2500 | |
| Fetal Bovine Serum (FBS) | Thermo Scientific | 10270106 | |
| gelatine from porcine skin | Sigma | G1890 | |
| GeneRuler 1 kb Plus DNA Ladder | Thermo Scientific | SM0313 | |
| GlycoBlue | Thermo Scientific | AM9516 | Coprecipitant |
| High-Sensitivity Bioanlayzer chips | Agilent | 5067-4626 | |
| Large Cooling Centrifuge 5920 R | Eppendorf | 5948000018 | |
| leukaemia inhibitory factor (LIF) | Merck | ESG1107 | |
| Liquiport | KNF | NF300 | Benchtop aspiration system |
| Low-binding filter tips | Biozym | VT0260U, VT0240, VT0220, VT0200U | |
| Molecular biology grade water | Merck | W3500-6x500ML | |
| Next Seq 500 | Illumina | SY-415-1001 | |
| Next Seq 500 High Output v2 Kit (300 cycles) | Illumina | FC-404-2004 | |
| Nonidet P40 Substitute (NP40) | Merck | 11332473001 | |
| PD0325901 | Axon Medchem BV | Axon 1408 | |
| Protease inhibitor cocktail (EDTA-free) | Merck | 11873580001 | |
| Proteinase K - recombinant, PCR-grade (20 mg/mL) | Thermo Scientific | EO0491 | |
| Qubit 2.0 | Thermo Scientific | Q32871 | |
| Qubit assay tubes | Thermo Scientific | Q32856 | |
| Qubit dsDNA High Sensitivity kit | Thermo Scientific | Q32851 | |
| RNase A (10 mg/mL) | Thermo Scientific | EN0531 | |
| Sodium acetate pH 5.2 (3M) | Merck | S7899 | |
| speed vacuum concentrator | Eppendorf | EP5305000100-1EA | |
| Agencourt AMPureXP | Beckman Coulter | A63881 | SPRI beads |
| SureSelect Target Enrichment Box 1 | Agilent | 5190-8645 | |
| SureSelect Target Enrichment Kit ILM Indexing Hyb Module Box 2 | Agilent | 5190-4455 | |
| SureSelect XT Library Prep Kit ILM | Agilent | 5500-0132 | |
| T4 ligase (30 units/µL) | Thermo Scientific | EL0013 | |
| table-top Centrifuge 5427 R | Eppendorf | 5409000012 | |
| Triton-X-100 (500 mL) | Merck | X100-500ML | |
| Trypan Blue | Invitrogen | T10282 | |
| Trypsine | Thermo Scientific | 25300054 | |
| UltraPure Glycine | Thermo Scientific | 15527013 | |
| β-mercaptoethanol | Thermo Scientific | 31350010 |
参考文献
- Ibrahim, D. M., Mundlos, S. The role of 3D chromatin domains in gene regulation: a multi-facetted view on genome organization. Current Opinion in Genetics & Development. 61, 1-8 (2020).
- Bolt, C. C., Duboule, D. The regulatory landscapes of developmental genes. Development. 147 (3), (2020).
- Glaser, J., Mundlos, S. 3D or not 3D: Shaping the genome during development. Cold Spring Harbor Perspectives in Biology. 14 (5), 040188 (2021).
- Denker, A., De Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Kempfer, R., Pombo, A. Methods for mapping 3D chromosome architecture. Nature Reviews Genetics. 21 (4), 207-226 (2020).
- McCord, R. P., Kaplan, N., Giorgetti, L. Chromosome conformation capture and beyond: Toward an integrative view of chromosome structure and function. Molecular Cell. 77 (4), 688-708 (2020).
- Jerkovic, I., Cavalli, G. Understanding 3D genome organization by multidisciplinary methods. Nature ReviewsMolecular Cell Biology. 22 (8), 511-528 (2021).
- Hsieh, T. -. H. S., et al. Mapping nucleosome resolution chromosome folding in yeast by Micro-C. Cell. 162 (1), 108-119 (2015).
- Krietenstein, N., et al. Ultrastructural details of mammalian chromosome architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Naumova, N., Smith, E. M., Zhan, Y., Dekker, J. Analysis of long-range chromatin interactions using Chromosome Conformation Capture. Methods. 58 (3), 192-203 (2012).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Zhao, Z., et al. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra-and interchromosomal interactions. Nature Genetics. 38 (11), 1341-1347 (2006).
- Würtele, H., Chartrand, P. Genome-wide scanning of HoxB1-associated loci in mouse ES cells using an open-ended Chromosome Conformation Capture methodology. Chromosome Research. 14 (5), 477-495 (2006).
- De Wit, E., De Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes & Development. 26 (1), 11-24 (2012).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Splinter, E., et al. The inactive X chromosome adopts a unique three-dimensional conformation that is dependent on Xist RNA. Genes & Development. 25 (13), 1371-1383 (2011).
- Ferraiuolo, M. A., Sanyal, A., Naumova, N., Dekker, J., Dostie, J. From cells to chromatin: capturing snapshots of genome organization with 5C technology. Methods. 58 (3), 255-267 (2012).
- Kim, J. H., et al. 5C-ID: Increased resolution Chromosome-Conformation-Capture-Carbon-Copy with in situ 3C and double alternating primer design. Methods. 142, 39-46 (2018).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Zhang, Y., et al. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations. Cell. 148 (5), 908-921 (2012).
- Rao, S. S. P., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Dixon, J. R., et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485 (7398), 376-380 (2012).
- Nora, E. P., et al. Spatial partitioning of the regulatory landscape of the X-inactivation centre. Nature. 485 (7398), 381-385 (2012).
- Krefting, J., Andrade-Navarro, M. A., Ibn-Salem, J. Evolutionary stability of topologically associating domains is associated with conserved gene regulation. BMC Biology. 16 (1), 87 (2018).
- Galupa, R., Heard, E. Topologically associating domains in chromosome architecture and gene regulatory landscapes during development, disease, and evolution. Cold Spring Harbor Symposia on Quantitative Biology. 82, 267-278 (2017).
- Tena, J. J., Santos-Pereira, J. M. Topologically associating domains and regulatory landscapes in development, evolution and disease. Frontiers in Cell and Developmental Biology. 9, 702787 (2021).
- Lupiáñez, D. G., Spielmann, M., Mundlos, S. Breaking TADs: How alterations of chromatin domains result in disease. Trends in Genetics. 32 (4), 225-237 (2016).
- Davidson, I. F., Peters, J. -. M. Genome folding through loop extrusion by SMC complexes. Nature Reviews Molecular Cell Biology. 22 (7), 445-464 (2021).
- Schmitt, A. D., Hu, M., Ren, B. Genome-wide mapping and analysis of chromosome architecture. Nature Reviews Molecular Cell Biology. 17 (12), 743-755 (2016).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Davies, J. O. J., et al. Multiplexed analysis of chromosome conformation at vastly improved sensitivity. Nature Methods. 13 (1), 74-80 (2016).
- Jäger, R., et al. Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci. Nature Communications. 6, 6178 (2015).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Research. 25 (4), 582-597 (2015).
- Sahlén, P., et al. Genome-wide mapping of promoter-anchored interactions with close to single-enhancer resolution. Genome Biology. 16, 156 (2015).
- Joshi, O., et al. Dynamic reorganization of extremely long-range promoter-promoter interactions between two states of pluripotency. Cell Stem Cell. 17 (6), 748-757 (2015).
- Mifsud, B., et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nature Genetics. 47 (6), 598-606 (2015).
- Dryden, N. H., et al. Unbiased analysis of potential targets of breast cancer susceptibility loci by Capture Hi-C. Genome Research. 24 (11), 1854-1868 (2014).
- Oudelaar, A. M., Davies, J. O. J., Downes, D. J., Higgs, D. R., Hughes, J. R. Robust detection of chromosomal interactions from small numbers of cells using low-input Capture-C. Nucleic Acids Research. 45 (22), 184 (2017).
- Oudelaar, A. M., et al. Single-allele chromatin interactions identify regulatory hubs in dynamic compartmentalized domains. Nature Genetics. 50 (12), 1744-1751 (2018).
- Franke, M., et al. Formation of new chromatin domains determines pathogenicity of genomic duplications. Nature. 538 (7624), 265-269 (2016).
- Despang, A., et al. Functional dissection of the Sox9-Kcnj2 locus identifies nonessential and instructive roles of TAD architecture. Nature Genetics. 51 (8), 1263-1271 (2019).
- Oudelaar, A. M., et al. Dynamics of the 4D genome during in vivo lineage specification and differentiation. Nature Communications. 11 (1), 1-12 (2020).
- Galupa, R., Heard, E. X-chromosome inactivation: A crossroads between chromosome architecture and gene regulation. Annual Review of Genetics. 52, 535-566 (2018).
- Loda, A., Collombet, S., Heard, E. Gene regulation in time and space during X-chromosome inactivation. Nature Reviews. Molecular Cell Biology. 23 (4), 231-249 (2022).
- van Bemmel, J. G., et al. The bipartite TAD organization of the X-inactivation center ensures opposing developmental regulation of Tsix and Xist. Nature Genetics. 51 (6), 1024-1034 (2019).
- Galupa, R., et al. A conserved noncoding locus regulates random monoallelic Xist expression across a topological boundary. Molecular Cell. 77 (2), 352-367 (2020).
- Gjaltema, R. A. F., et al. Distal and proximal cis-regulatory elements sense X chromosome dosage and developmental state at the Xist locus. Molecular Cell. 82 (1), 190-208 (2022).
- Galupa, R., et al. Inversion of a topological domain leads to restricted changes in its gene expression and affects inter-domain communication. Development. 149 (9), (2022).
- Savarese, F., Flahndorfer, K., Jaenisch, R., Busslinger, M., Wutz, A. Hematopoietic precursor cells transiently reestablish permissiveness for X inactivation. Molecular and Cellular Biology. 26 (19), 7167-7177 (2006).
- Schulz, E. G., et al. The two active X chromosomes in female ESCs block exit from the pluripotent state by modulating the ESC signaling network. Cell Stem Cell. 14 (2), 203-216 (2014).
- Gnirke, A., et al. Solution hybrid selection with ultra-long oligonucleotides for massively parallel targeted sequencing. Nature Biotechnology. 27 (2), 182-189 (2009).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Piccinini, F., Tesei, A., Arienti, C., Bevilacqua, A. Cell counting and viability assessment of 2D and 3D Cell cultures: Expected reliability of the trypan blue assay. Biological Procedures Online. 19 (1), 8 (2017).
- Servant, N., et al. HiC-Pro: an optimized and flexible pipeline for Hi-C data processing. Genome Biology. 16, 259 (2015).
- Abdennur, N., Mirny, L. A. Cooler: scalable storage for Hi-C data and other genomically labeled arrays. Bioinformatics. 36 (1), 311-316 (2020).
- Imakaev, M., et al. Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nature Methods. 9 (10), 999-1003 (2012).
- Forcato, M., et al. Comparison of computational methods for Hi-C data analysis. Nature Methods. 14 (7), 679-685 (2017).
- Venev, S., et al. . open2c/cooltools: v0.4.1. , (2021).
- Wages, J. M. NUCLEIC ACIDS | Immunoassays. Encyclopedia of Analytical Science. , 408-417 (2005).
- Ewels, P. A., et al. The nf-core framework for community-curated bioinformatics pipelines. Nature Biotechnology. 38 (3), 276-278 (2020).
- Servant, N., Peltzer, A. nf-core/hic: Initial release of nf-core/hic. Zenodo. , (2019).
- Furlan, G., et al. The Ftx noncoding locus controls X chromosome inactivation independently of its RNA products. Molecular Cell. 70 (3), 462-472 (2018).
- Giorgetti, L., et al. Structural organization of the inactive X chromosome in the mouse. Nature. 535 (7613), 575-579 (2016).
- Simon, C. S., et al. Functional characterisation of cis-regulatory elements governing dynamic Eomes expression in the early mouse embryo. Development. 144 (7), 1249-1260 (2017).
- Williams, R. M., et al. Reconstruction of the global neural crest gene regulatory network in vivo. Developmental Cell. 51 (2), 255-276 (2019).
- Godfrey, L., et al. DOT1L inhibition reveals a distinct subset of enhancers dependent on H3K79 methylation. Nature Communications. 10 (1), 2803 (2019).
- Hanssen, L. L. P., et al. Tissue-specific CTCF-cohesin-mediated chromatin architecture delimits enhancer interactions and function in vivo. Nature Cell Biology. 19 (8), 952-961 (2017).
- Larke, M. S. C., et al. Enhancers predominantly regulate gene expression during differentiation via transcription initiation. Molecular Cell. 81 (5), 983-997 (2021).
- Oudelaar, A. M., et al. A revised model for promoter competition based on multi-way chromatin interactions at the α-globin locus. Nature Communications. 10 (1), 5412 (2019).
- Long, H. K., et al. Loss of extreme long-range enhancers in human neural crest drives a craniofacial disorder. Cell Stem Cell. 27 (5), 765-783 (2020).
- Schoenfelder, S., Javierre, B. -. M., Furlan-Magaril, M., Wingett, S. W., Fraser, P. Promoter Capture Hi-C: High-resolution, genome-wide profiling of promoter interactions. Journal of Visualized Experiments. (136), e57320 (2018).
- Siersbæk, R., et al. Dynamic rewiring of promoter-anchored chromatin loops during adipocyte differentiation. Molecular Cell. 66 (3), 420-435 (2017).
- Rubin, A. J., et al. Lineage-specific dynamic and pre-established enhancer-promoter contacts cooperate in terminal differentiation. Nature Genetics. 49 (10), 1522-1528 (2017).
- Freire-Pritchett, P., et al. Global reorganisation of cis-regulatory units upon lineage commitment of human embryonic stem cells. eLife. 6, 21926 (2017).
- Javierre, B. M., et al. Lineage-specific genome architecture links enhancers and non-coding disease variants to target gene promoters. Cell. 167 (5), 1369-1384 (2016).
- Miguel-Escalada, I., et al. Human pancreatic islet three-dimensional chromatin architecture provides insights into the genetics of type 2 diabetes. Nature Genetics. 51 (7), 1137-1148 (2019).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Baxter, J. S., et al. Capture Hi-C identifies putative target genes at 33 breast cancer risk loci. Nature Communications. 9 (1), 1028 (2018).
- Sanborn, A. L., et al. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes. Proceedings of the National Academy of Sciences. 112 (47), 6456-6465 (2015).
- Owens, D. D. G., et al. Dynamic Runx1 chromatin boundaries affect gene expression in hematopoietic development. Nature Communications. 13 (1), 773 (2022).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。