需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
生成转录组学特征的转录调控视图,用于小型数据集上的预测任务和暗生物标志物检测
摘要
在这里,我们介绍了一种将转录组数据转换为mqTrans视图的协议,从而能够识别暗生物标志物。虽然在常规转录组学分析中没有差异表达,但这些生物标志物在 mqTrans 视图中表现出差异表达。该方法是对传统方法的补充技术,揭示了以前被忽视的生物标志物。
摘要
转录组代表样品中许多基因的表达水平,已广泛应用于生物学研究和临床实践中。研究人员通常专注于在表型组和对照组样本之间具有差异表示的转录组生物标志物。本研究提出了一个多任务图-注意力网络(GAT)学习框架来学习参考样本的复杂基因间相互作用。在健康样本 (HealthModel) 上预训练了一个示范参考模型,该模型可直接用于生成独立测试转录组的基于模型的定量转录调控 (mqTrans) 视图。通过预测任务和暗生物标志物检测证明了生成的转录组 mqTrans 视图。创造的术语“暗生物标志物”源于其定义,即暗生物标志物在 mqTrans 视图中显示差异表示,但在其原始表达水平上没有差异表达。由于缺乏差异表达,在传统的生物标志物检测研究中,深色生物标志物总是被忽视。管道 HealthModelPipe 的源代码和手册可以从 http://www.healthinformaticslab.org/supp/resources.php 下载。
引言
转录组由样品中所有基因的表达组成,可通过微阵列和 RNA-seq1 等高通量技术进行分析。数据集中一个基因的表达水平称为转录组学特征,表型组和对照组之间转录组特征的差异表示将该基因定义为该表型的生物标志物 2,3。转录组生物标志物在疾病诊断4、生物学机制5和生存分析6、7等研究中得到广泛应用。
健康组织中的基因活性模式携带有关生命的重要信息8,9。这些模式提供了宝贵的见解,并作为理解良性疾病10,11和致命疾病12的复杂发展轨迹的理想参考。基因相互作用,转录组代表其复杂相互作用后的最终表达水平。这种模式被表述为转录调控网络13和代谢网络14等。信使RNA(mRNA)的表达可以通过转录因子(TFs)和长基因间非编码RNA(lincRNA)进行转录调控15,16,17。传统的差异表达分析忽略了这种复杂的基因相互作用,并假设特征间独立性18,19。
图神经网络 (GNN) 的最新进展表明,在从基于 OMIC 的数据中提取重要信息用于癌症研究20 方面具有非凡的潜力,例如,识别共表达模块21。GNNs的先天能力使它们成为模拟基因之间错综复杂的关系和依赖关系的理想选择22,23。
生物医学研究通常侧重于准确预测对照组的表型。这些任务通常表述为二元分类24,25,26。在这里,两个类标签通常编码为 1 和 0、true 和 false,甚至正负27。
本研究旨在提供一种易于使用的协议,用于基于预训练的图注意力网络 (GAT) 参考模型生成转录组数据集的转录调控 (mqTrans) 视图。先前发表的工作26 中的多任务 GAT 框架用于将转录组学特征转换为 mqTrans 特征。使用来自加州大学圣克鲁斯分校 (UCSC) Xena 平台28 的大型健康转录组数据集对参考模型 (HealthModel) 进行预训练,该模型定量测量了从调节因子(TF 和 lincRNA)到靶 mRNA 的转录调控。生成的 mqTrans 视图可用于构建预测模型和检测暗生物标志物。该协议利用来自癌症基因组图谱(TCGA)数据库29 的结肠腺癌(COAD)患者数据集作为说明性示例。在这种情况下,I 期或 II 期的患者被归类为阴性样本,而 III 期或 IV 期的患者被视为阳性样本。还比较了 26 种 TCGA 癌症类型中深色和传统生物标志物的分布。
HealthModel 管道的说明
该协议中采用的方法基于先前发布的框架26,如 图 1 所示。首先,用户需要准备输入数据集,将其输入到建议的 HealthModel 管道中,并获取 mqTrans 特征。详细的数据制备说明在协议部分的第 2 节中提供。随后,用户可以选择将 mqTrans 特征与原始转录组特征相结合,或者仅继续使用生成的 mqTrans 特征。然后,生成的数据集将进行特征选择过程,用户可以灵活地在 k 倍交叉验证中选择他们的首选 k 值进行分类。该协议中使用的主要评估指标是准确性。
HealthModel26 将转录组学特征分为三个不同的组:TF(转录因子)、lincRNA(长基因间非编码 RNA)和 mRNA(信使 RNA)。TF 特征是根据人类蛋白质图谱30,31 中可用的注释定义的。这项工作利用了 GTEx 数据集32 中 lincRNA 的注释。属于 KEGG 数据库33 中第三级通路的基因被认为是 mRNA 特征。值得注意的是,如果 mRNA 特征表现出 TRRUST 数据库34 中记录的靶基因的调控作用,则将其重新分类为 TF 类。
该协议还手动生成调节因子 (regulatory_geneIDs.csv) 和靶标 mRNA (target_geneIDs.csv) 的基因 ID 的两个示例文件。调控特征(TFs和lincRNAs)之间的成对距离矩阵由Pearson相关系数计算,并通过流行的工具加权基因共表达网络分析(WGCNA)36 (adjacent_matrix.csv)进行聚类。用户可以直接将 HealthModel 管道与这些示例配置文件一起使用,以生成转录组数据集的 mqTrans 视图。
HealthModel的技术细节
HealthModel 将 TF 和 lincRNA 之间的复杂关系表示为图形,输入特征用作 V 表示的顶点,顶点间边矩阵指定为 E。每个样品都具有 K 调控特征,符号为 VK×1。具体来说,数据集包含 425 个 TF 和 375 个 lincRNA,样本维数为 K = 425 + 375 = 800。为了建立边矩阵 E,这项工作使用了流行的工具 WGCNA35。连接两个顶 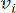 点的成对权重表示为 和
点的成对权重表示为 和 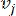 ,由 Pearson 相关系数确定。基因调控网络表现出无标度拓扑结构36,其特征在于存在具有关键功能作用的枢纽基因。我们使用拓扑重叠度量 (TOM) 计算两个要素或顶点
,由 Pearson 相关系数确定。基因调控网络表现出无标度拓扑结构36,其特征在于存在具有关键功能作用的枢纽基因。我们使用拓扑重叠度量 (TOM) 计算两个要素或顶点 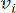 之间的相关性,如下
之间的相关性,如下 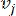 所示:
所示:
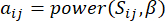 (1)
(1)
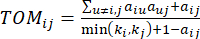 (2)
(2)
软阈值β是使用 WGCNA 软件包中的“pickSoft Threshold”函数计算的。应用幂指数函数 a ij,其中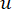 表示不包括 i 和 j 的基因,并表示
表示不包括 i 和 j 的基因,并表示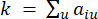 顶点连通性。WGCNA 使用常用的差异度量将转录组学特征的表达谱聚类到多个模块中 (
顶点连通性。WGCNA 使用常用的差异度量将转录组学特征的表达谱聚类到多个模块中 (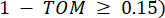 37.
37.
HealthModel 框架最初被设计为多任务学习架构26.该协议仅利用模型预训练任务来构建转录组mqTrans视图。用户可以选择使用额外的特定任务转录组样本在多任务图注意力网络下进一步完善预训练的 HealthModel。
特征选择和分类的技术细节
特征选择池实现了 11 种特征选择 (FS) 算法。其中,基于滤波器的FS算法有三种:使用最大信息系数(SK_mic)选择K个最佳特征,根据MIC的FPR(SK_fpr)选择K个特征,选择MIC错误发现率最高的K个特征(SK_fdr)。此外,三种基于树的 FS 算法使用具有 Gini 指数 (DT_gini)、自适应提升决策树 (AdaBoost) 和随机森林 (RF_fs) 的决策树来评估单个特征。该池还包含两种包装方法:使用线性支持向量分类器 (RFE_SVC) 的递归特征消除和使用逻辑回归分类器 (RFE_LR) 的递归特征消除。最后,包括两种嵌入算法:具有排名靠前的 L1 特征重要性值 (lSVC_L1) 的线性 SVC 分类器和具有排名靠前的 L1 特征重要性值 (LR_L1) 的逻辑回归分类器。
分类器池使用七种不同的分类器来构建分类模型。这些分类器包括线性支持向量机 (SVC)、高斯朴素贝叶斯 (GNB)、逻辑回归分类器 (LR)、k 最近邻(k 默认设置为 5 (KNN)、XGBoost、随机森林 (RF) 和决策树 (DT)。
可以在命令行中设置数据集的随机拆分为训练:测试子集。演示的示例使用火车的比率:test = 8:2。
Access restricted. Please log in or start a trial to view this content.
研究方案
注意:以下协议描述了主要模块的信息学分析过程和 Python 命令的详细信息。图 2 说明了该协议中使用的示例命令的三个主要步骤,有关更多技术细节,请参阅以前发表的作品26,38。在计算机系统中的普通用户帐户下执行以下协议,并避免使用管理员或 root 帐户。这是一个计算协议,没有生物医学危害因素。
1. 准备 Python 环境
- 创建虚拟环境。
- 本研究使用了 Python 编程语言和 Python 3.7 的 Python 虚拟环境 (VE)。请按照下列步骤操作(图 3A):
conda create -n healthmodel python=3.7
conda create 是用于创建新 VE 的命令。参数 -n 指定新环境的名称,在本例中为 healthmodel。 python=3.7 指定要安装的 Python 版本。选择支持上述命令的任何首选名称和 Python 版本。 - 运行命令后,输出类似于 图 3B。输入 y 并等待该过程完成。
- 本研究使用了 Python 编程语言和 Python 3.7 的 Python 虚拟环境 (VE)。请按照下列步骤操作(图 3A):
- 激活虚拟环境
- 在大多数情况下,使用以下命令激活创建的 VE(图 3C):
conda 激活 healthmodel - 如果某些平台要求用户上传特定于平台的配置文件进行激活,请按照特定于平台的说明进行 VE 激活。
- 在大多数情况下,使用以下命令激活创建的 VE(图 3C):
- 安装 PyTorch 1.13.1
- PyTorch 是用于人工智能 (AI) 算法的流行 Python 包。以基于 CUDA 11.7 GPU 编程平台的 PyTorch 1.13.1 为例。在 https://pytorch.org/get-started/previous-versions/ 查找其他版本。使用以下命令(图 3D):
pip3 安装 torch torchvision torchaudio
注意:强烈建议使用 PyTorch 版本 1.12 或更高版本。否则,安装所需的软件包 torch_geometric 可能具有挑战性,如torch_geometric官方网站上所述:https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html。
- PyTorch 是用于人工智能 (AI) 算法的流行 Python 包。以基于 CUDA 11.7 GPU 编程平台的 PyTorch 1.13.1 为例。在 https://pytorch.org/get-started/previous-versions/ 查找其他版本。使用以下命令(图 3D):
- 安装火炬几何的其他软件包
- 按照 https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html 中的准则,使用命令安装以下软件包:torch_scatter、torch_sparse、torch_cluster 和 torch_spline_conv(图 3E):
pip install pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html
- 按照 https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html 中的准则,使用命令安装以下软件包:torch_scatter、torch_sparse、torch_cluster 和 torch_spline_conv(图 3E):
- 安装 torch-geometric 软件包。
- 本研究需要 火炬几何 包的特定版本 2.2.0。运行命令(图 3F):
pip 安装 torch_geometric==2.2.0
- 本研究需要 火炬几何 包的特定版本 2.2.0。运行命令(图 3F):
- 安装其他软件包。
- 像 pandas 这样的软件包通常默认可用。如果没有,请使用 pip 命令安装它们。例如,要安装 pandas 和 xgboost,请运行:
pip 安装 pandas
pip 安装 xgboost
- 像 pandas 这样的软件包通常默认可用。如果没有,请使用 pip 命令安装它们。例如,要安装 pandas 和 xgboost,请运行:
2. 使用预训练的 HealthModel 生成 mqTrans 特征
- 下载代码和预训练模型。
- 从网站下载代码和预训练的 HealthModel:http://www.healthinformaticslab.org/supp/resources.php,该网站名为 HealthModel-mqTrans-v1-00.tar.gz (图 4A)。下载的文件可以解压缩为用户指定的路径。已实施协议的详细表述和支持数据可在26 中找到。
- 引入参数以运行 HealthModel。
- 首先,在命令行中将工作目录更改为 HealthModel-mqTrans 文件夹。使用以下语法运行代码:
python main.py <数据文件夹> <模型文件夹> <输出文件夹>
有关每个参数以及数据、模型和输出文件夹的详细信息如下:
data 文件夹:这是源数据文件夹,每个数据文件都是 csv 格式。此数据文件夹包含两个文件(请参阅步骤 2.3 和 2.4 中的详细说明)。这些文件需要替换为个人数据。
data.csv:转录组基质文件。第一行列出了特征(或基因)ID,第一列给出了样本ID。基因列表包括调控因子(TF 和 lincRNA)和调控的 mRNA 基因。
label.csv:示例标签文件。第一列列出示例 ID,名称为“label”的列提供示例标签。
model 文件夹:用于保存模型信息的文件夹:
HealthModel.pth:预先训练的 HealthModel。
regulatory_geneIDs.csv:本研究中使用的调控基因 ID。
target_geneIDs.csv:本研究中使用的靶基因。
adjacent_matrix.csv:调控基因的相邻基质。
输出文件夹:输出文件写入此文件夹,由代码创建。
test_target.csv:Z-归一化和插补后靶基因的基因表达值。
pred_target.csv:靶基因的预测基因表达值。
mq_target.csv:靶基因的预测基因表达值。
- 首先,在命令行中将工作目录更改为 HealthModel-mqTrans 文件夹。使用以下语法运行代码:
- 准备csv格式的转录组学矩阵文件。
- 每行代表一个样本,每列代表一个基因(图4B)。将转录组数据矩阵文件命名为 data.csv 在 data 文件夹中。
注意:此文件可以通过从 Microsoft Excel 等软件中以 .csv 格式手动保存数据矩阵来生成。转录组基质也可以通过计算机编程生成。
- 每行代表一个样本,每列代表一个基因(图4B)。将转录组数据矩阵文件命名为 data.csv 在 data 文件夹中。
- 准备 csv 格式的标签文件。
- 与转录组基质文件类似,将标签文件命名为数据文件夹中的label.csv(图4C)。
注意:第一列给出了示例名称,每个示例的类标签在标题 为标签的列中给出。标签列中的 0 值表示此样本为负数,1 表示为阳性样本。
- 与转录组基质文件类似,将标签文件命名为数据文件夹中的label.csv(图4C)。
- 生成 mqTrans 特征。
- 运行以下命令以生成 mqTrans 特征并获取 图 4D 所示的输出。mqTrans 特征将生成为文件 ./output/mq_targets.csv,标签文件将重新保存为文件 ./output/label.csv。为了便于进一步分析,mRNA基因的原始表达值也被提取为文件 ./output/test_target.csv。
python ./Get_mqTrans/code/main.py ./data ./Get_mqTrans/model ./输出
- 运行以下命令以生成 mqTrans 特征并获取 图 4D 所示的输出。mqTrans 特征将生成为文件 ./output/mq_targets.csv,标签文件将重新保存为文件 ./output/label.csv。为了便于进一步分析,mRNA基因的原始表达值也被提取为文件 ./output/test_target.csv。
3. 选择 mqTrans 功能
- 功能选择代码的语法
- 首先,将工作目录更改为 HealthModel-mqTrans 文件夹。使用以下语法:
python ./FS_classification/testMain.py
各参数详情如下:
in-data-file:输入数据文件
in-label-file:输入数据文件的标签
输出文件夹:此文件夹中保存两个输出文件,包括 Output-score.xlsx (特征选择方法和对应分类器的精度)和 Output-SelectedFeatures.xlsx (每个特征选择算法的所选特征名称)。- select_feature_number:选择要素数,范围从 1 到数据文件的特征数。
- test_size:设置要拆分的测试样品的比例。例如,0.2 表示输入数据集按 0.8:0.2 的比例随机拆分为 train: 测试子集。
- combine:如果为 true,则将两个数据文件组合在一起以进行特征选择,即原始表达式值和 mqTrans 特征。如果为 false,则仅使用一个数据文件进行特征选择,即原始表达式值或 mqTrans 特征。
- 合并文件:如果合并为 true,请提供此文件名以保存合并的数据矩阵。
注: 此管道旨在演示生成的 mqTrans 特征在分类任务中如何执行,并直接使用第 2 节生成的文件进行以下操作。
- 首先,将工作目录更改为 HealthModel-mqTrans 文件夹。使用以下语法:
- 运行特征选择算法以进行 mqTrans 特征选择。
- 如果用户选择 mqTrans 特征或原始特征,则将 combine = False 。
- 首先,选择 800 个原始特征,并将数据集拆分为训练:test=0.8:0.2:
python ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 False - 如果用户希望将 mqTrans 特征与原始表达式值组合以选择特征,则将 combine = True 转为。在这里,示范示例是选择 800 个特征并将数据集拆分为训练:test=0.8:0.2:
python ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 True ./output/test_target.csv
注意: 图 5 显示了输出信息。此协议所需的补充文件位于HealthModel-mqTrans-v1-00.tar文件夹(补充编码文件 1)中。
Access restricted. Please log in or start a trial to view this content.
结果
评估转录组数据集的 mqTrans 视图
测试代码使用 11 种特征选择 (FS) 算法和 7 种分类器来评估转录组数据集生成的 mqTrans 视图对分类任务的贡献(图 6)。测试数据集由来自癌症基因组图谱 (TCGA) 数据库29 的 317 个结肠腺癌 (COAD) 组成。I期或II期的COAD患者被视为阴性样本,而III期或IV期的COAD患者为阳性样本。
测试代?...
Access restricted. Please log in or start a trial to view this content.
讨论
该协议的第 2 部分(使用预先训练的 HealthModel 生成 mqTrans 特征)是该协议中最关键的步骤。在第 1 节中准备计算工作环境后,第 2 节基于预训练的大型参考模型生成转录组数据集的 mqTrans 视图。第 3 节是为生物标志物检测和预测任务选择生成的 mqTrans 特征的示范性示例。用户可以使用自己的工具或代码在此 mqTrans 数据集上进行其他转录组学分析。
原始 HealthModel 框架可以使用...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
本研究得到了贵州省科技项目(ZK2023-297)、贵州省卫健委科技基金(gzwkj2023-565)、吉林省教育厅科技项目(JJKH20220245KJ JJKH20220226SK)、国家自然科学基金(U19A2061)、吉林省大数据智能计算重点实验室等单位的支持(20180622002JC)和中央高校基础科研基金(JLU.我们衷心感谢审稿编辑和三位匿名审稿人的建设性批评,这些批评在大大提高本协议的严谨性和清晰度方面发挥了重要作用。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
参考文献
- Mutz, K. -O., Heilkenbrinker, A., Lönne, M., Walter, J. -G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516(2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684(2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352(2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528(2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. Analysis of Survival Data. , Chapman and Hall/CRC. London. (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866(2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307(2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119(2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239(2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334(2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338(2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , Springer. Cham. (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738(2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5(2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883(2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582(2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801(2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138(2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308(2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。