Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Osmotische Vermeidung in Caenorhabditis elegans: Synaptic Function von zwei Genen, Orthologe der Menschenrechte NRXN1 Und NLGN1, Die als Kandidaten für Autismus
In diesem Artikel
Zusammenfassung
Neurexine und Neuroligine sind Membran-Neuron Haftung Proteine, die wichtige Rollen in synaptic Differenzierung und die Übertragung durchzuführen. Neuroligin defizienten Mutanten von C. elegans Defekt sind bei der Aufdeckung von osmotischen Kraft, aber wenn sie enthalten auch eine Mutation im Gen Neurexin, erholen sie den Wildtyp-Phänotyp.
Zusammenfassung
Neurexine und Neuroligine sind Zelladhäsionsmoleküle in erregenden und hemmenden Synapsen, und sie sind für die korrekte neuronales Netzwerk-Funktion 1 erforderlich. Diese Proteine sind an der präsynaptischen und postsynaptischen Membranen 2 gefunden. Studien an Mäusen zeigen, dass Neurexine und neurologins eine wesentliche Rolle in der synaptischen Übertragung 1 haben. Jüngste Berichte haben gezeigt, dass einer veränderten neuronalen Verbindungen während der Entwicklung des menschlichen Nervensystems könnten die Basis für die Ätiologie der zahlreichen Fälle von Autismus-Spektrum-Störungen 3 bilden.
Caenorhabditis elegans konnte als experimentelles Werkzeug, um die Untersuchung der Funktionsweise der synaptischen Komponenten zu erleichtern verwendet werden, wegen seiner Einfachheit für Labor-Experimente, und da sein Nervensystem und synaptische Verkabelung wurde vollständig charakterisiert. In C. Elegans NRX-1 und NLG-1-Gene sind orthologen die menschliche NRXN1 und NLGN1 Gene, die alpha-Neurexin-1 und Neuroligin-1-Proteine, bzw. zu codieren. Bei Menschen und Nematoden, ist die Organisation von Neurexine und Neuroligine ähnlich in Bezug auf die funktionellen Domänen.
Der Kopf des Nematoden enthält die amphid, ein Sinnesorgan des Nematoden, die Antworten vermittelt auf verschiedene Reize, einschließlich osmotische Stärke. Die amphid ist von 12 sensorischen Neuronen bipolar mit Flimmerepithel Dendriten und ein Axon präsynaptischen Terminal 4. Zwei von diesen Neuronen, genannt ASHR und ASHL sind besonders wichtig in osmotischen sensorische Funktion, Aufdeckung wasserlösliche Repellents mit hohen osmotischen Stärke 5. Die Dendriten der beiden Neuronen an der Spitze des Mundes zu verlängern und die Axone zu verlängern, um den Nerv Ring, wo sie synaptische Verbindungen mit anderen Neuronen Bestimmung der Verhaltensreaktion 6 machen.
Zur Beurteilung der Auswirkungen der Neurexin und Neuroligin in hohen osmotischen Kräfte Vermeidung, zeigen wir die verschiedenen Reaktion von C. elegans-Mutanten in NRX-1 und NLG-1-Gene, mit einer Methode auf einem 4M Fructose Ring 7 basiert. Die Verhaltens-Phänotypen wurden bestätigt mit spezifischen RNAi-Klone 8. In C. elegans, kann die dsRNA erforderlich, um RNAi auslösen durch die Fütterung 9 verabreicht werden. Die Lieferung der dsRNA durch Nahrung induziert die RNAi Interferenz des Gens von Interesse so dass die Identifizierung von genetischen Komponenten und Netzwerk-Pfade.
Protokoll
1: Osmotische Vermeidung Assay.
- Über 16-24 Stunden vor dem Test, Pick L4 Larvenstadium Tiere jeder Genotyp eine frische NGM-Platte mit OP50 E. coli andincubate es bei 20 ° C. Am nächsten Tag starten das Experiment mit jungen Erwachsenen.
- Es wird empfohlen, die Durchführung des Tests "blind". Die Platten mit jeder Stamm zu untersuchenden sollte durch einen zweiten Experimentator neu etikettiert werden, der Durchführung des Tests mit dem neu etikettiert Platten, entlarvt als das Experiment beendet werden würde.
- Der Tag des Tests, bereiten Sie einen 4M Stammlösung von Fructose mit 1% Kongorot Lösung auflösen und bei Raumtemperatur. Wir empfehlen, die Lösung vor jedem Test, da der Farbstoff mit der Zeit ausfallen könnte.
- Kreisring (1 cm Durchmesser) auf NGM befindet sich auf der Mitte der festen Medium beschrieben, mit 15 ul der roten 4M Fructose-Lösung. Lassen Sie die Fructose-Lösung in den Agar einweichen, dies dauert in der Regel zwischen 2 und 5 Minuten.
- Legen Sie einzelne junge erwachsene Tiere von jedem Stamm in den Ring und folgen in den nächsten 10 Minuten, um die Antwort auf die osmotische Barriere zu bestimmen. Tiere Vermeidung der Ring mehr als sechs Mal in Folge als normal eingestuft werden; Ausscheidenden am Ring in weniger als sechs Versuche sind als fehlerhaft in osmotische Empfindlichkeit.
Hinweis: Die Steuerung Stammes muss in jedem Assay verwendet werden. N2 junge erwachsene Tiere werden als positive Kontrollen verwendet, da sie zu vermeiden entscheidend die Ringwallanlage. Tiere, die zuvor verhungert, ging durch Dauer-Larven oder aus Platten, die zu trocken kommen sollten nicht verwendet werden. Zu Beginn sollte der Kontrolltiere in zweifacher Ausfertigung ausgewertet werden, um zu bestätigen, dass die Assay-Platten und die Lösung korrekt sind.
2: Generierung von Knockdown Würmer durch RNAi Fütterung.
- RNAi Platten: NGM RNAi Fütterung Platten enthält pro Liter: 17 g Agar, 2,5 g Pepton, 3,0 g NaCl, 1 ml 5 mg ml -1 Cholesterin; füllen die Flasche zu 1 Liter mit H 2 O und Autoklaven. Nach Agar kühlt auf etwa 65 ° C, 25 ml 1 M KPO 4, pH 6,0, 1 ml 1 M CaCl 2, 1 ml 1M MgSO 4, 0,5 ml Carbenicillin (50 mg ml -1) und 1 ml 1M IPTG. Wenn Sie mit vorbereiteten soliden NGM beginnen, schmelzen festes Medium durch die Mikrowelle und anschließend mit Flüssigkeiten NGM auf der Bank zu kühlen und fügen Sie dann KPO 4, pH 6,0, CaCl 2, MgSO 4, Carbenicillin und IPTG wie zuvor angegeben.
10 ml NGM in jede 60 mm Petrischale. Abkühlen lassen und umkehren Platten Beibehaltung über Nacht bei RT vor dem Gebrauch. Die Platten können in einer Plastiktüte bei 4 ° C für ca. 4-5 Tage gelagert. - Bakterielle und Einleitung: Isolieren Sie Kolonien von E.coli HT115 (DE3)-Stamm, mit L4440 Vektor, der ein Fragment, das dem Ziel-Gen transformiert, auf Luria-Bertani (LB)-Agarplatten (17 g Agar, 10 g Trypton, 5 g Hefeextrakt und 10 g NaCl pro Liter) mit Ampicillin (50 ug / ml) und Tetracyclin (15 ug / ml).
Wählen Sie eine Kolonie von Bakterien und impfen in LB mit 50 pg / ml Ampicillin, gewachsen und für 6 8 h unter Schütteln bei 37 ° C; Samen ein Tropfen dieser Kultur auf die vorbereiteten Platten NGM und trocknen die Platten gründlich, bevor sie Inkubation über Nacht ( 12 24 h) bei Raumtemperatur, damit die Bakterien wachsen und der Induktion zu beginnen. Die Inkubation wurde im Dunkeln, weil Tetracyclin ist lichtempfindlich und kann die Variabilität der RNAi-Assays unter Platten beeinflussen.
Es ist notwendig, um eine positive und eine negative Kontrolle für RNAi Fütterungsversuche verwenden. Die positive Kontrolle ist E.coli HT115 Zelle mit der L4440 Vektor, der die unc-22-Gen-Sequenz umgewandelt. Der Knockdown von unc-22-Gen produziert ein "Zucken Phänotyp". Die negative Kontrolle ist E.coli HT115 Zelle mit leeren L4440 transformiert. - Worm Handhabung und Scoring: Der erste Tag, Ort L4 Stadium Würmer aus einer NGM-Platte mit OP50 auf NGM-Platten ohne Bakterien und inkubieren ausgesät bei 20 ° C für 12 Stunden (Fasten).
Am nächsten Tag, Transfer jungen Erwachsenen fasteten Zwitter auf einen Teller mit den Bakterien Ausdruck der spezifischen Zielgens RNAi seedded. Lassen Sie 40-48 Stunden bei 20 ° C bis F1-Nachkommen zu erzeugen.
Dann legen Sie ein paar F1 erwachsenen Würmer auf eine andere Platte mit der gleichen Bakterien geimpft. Nach 24-48 Stunden, auszuwählen und zu isolieren, aus der F2-Nachkommen, junge Erwachsene (voll ausgebildete überstehende Vulva mit wenigen Eier) und Note für Phänotypen.
Hinweis: der RNAi-Induktion in Neuronen haben einige Einschränkungen, weil der feuerfesten Eigenschaften der C elegans Nervensystem RNAi.. Zur Überwindung der Probleme dieser Ineffizienz in Neuron ist es empfehlenswert, die GebO-3-Stamm, eine hypersensible Hintergrund der Auswirkungen der RNAi in Neuronen 10 zu verwenden. In unseren Experimenten keine differences wurden zwischen Bristol N2 und RRF-3-Stämme für die Gene gefunden und den Phänotyp analysiert.
3: Die Stämme verwendet.
Die C. elegans Stämme in dieser Arbeit sind in Tabelle 1 dargestellt. Mutanten-Stämme wurden gekreuzt gegen N2 Wildtyp - mindestens vier Mal, um unerwünschte zufällige Mutationen, die während der Mutagenese Protokoll erzeugt worden sein könnten, zu entfernen.
.. OP50 E-coli-Stamm wurde von Caenorhabditis Genetic Center, University of Minnesota, USA E.coli HT115 (DE3) mit dem Plasmid pL4440 Durchführung NRX-1 (JA: C29A12.5) suppliied und NLG-1 (JA: C40C9.5 Gen)-Fragmente wurden durch Dr. Peter Askjaer, Centro Andaluz de Biología del Desarrollo (CABD), CSIC Universidad Pablo Olavide, Sevilla, Spanien zur Verfügung gestellt.
4: Repräsentative Ergebnisse.
Illustrative Ergebnisse sind in den Abbildungen 1 und 2 dargestellt. Zehn Tiere von jedem Stamm und ein Minimum von drei Replika-Experimente wurden durchgeführt.
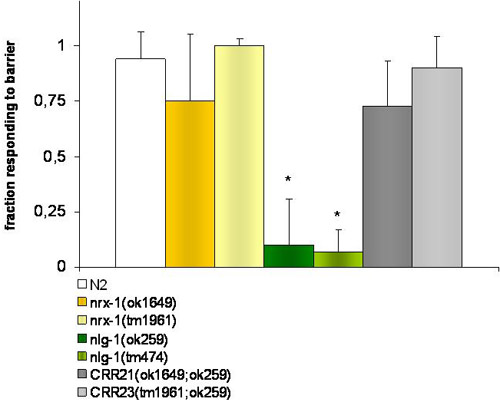
Abbildung 1. Experimente des osmotischen Vermeidungsverhalten.
Control-Stämme und NRX-1 (ok1649 und tm1961) V mangelhaft mutantworms durch Umkehren rückwärts, wenn sie einen osmotischen Barriere (4M Fructose) Begegnung reagieren. NLG-1 (ok259 und tm474) X-Mutanten nicht, diese Barriere zu erkennen. Doppel-Mutanten in NLG-1 und NRX-1, Stämme CRR21 (ok1649; ok259) VX und CRR23 (tm1961; ok259) VX erholte sich der Wildtyp-Phänotyp. Der * zeigt signifikante Unterschiede (P ≤ 0,001) von t-Student-Test, in der Antwort von jedem Stamm der Bristol N2 Wildtyp von t-Student-Test
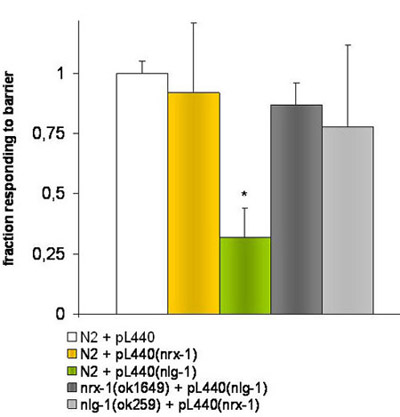
Abbildung 2. Experimente mit Knockdown Würmer durch RNAi Fütterung.
E.coli HT115 (DE3) mit leeren pL4440 Vektor transformierte oder mit einem Fragment, das dem Zielgene NRX-1 oder NLG-1 wurden gefüttert anderer Wurm Stämme verwendet. Der * zeigt signifikante Unterschiede (P ≤ 0,001) von t-Student-Test, in der Reaktion des N2-Stamm mit Bakterien, die die pL4440 vector mit einem Fragment Targeting der NLG-1-Gens im Vergleich mit dem N2-Stamm mit leerem Vektor pL4440 eingespeist .
Access restricted. Please log in or start a trial to view this content.
Diskussion
Neurexine und Neuroligine übernehmen wichtige Rollen in der synaptischen Übertragung 11 und Differenzierung von synaptischen Verbindungen 12. Beide Moleküle haben als Kandidatengene für Autismus 13,14 identifiziert worden.
In diesem Video zeigen wir eine einfache Methode, die uns um die Wirkung der Gene, die den osmotischen Vermeidung Antwort in C. Studie erlaubt elegans. Neuroligin Mutanten defekt sind bei der Aufdeckung von osmotischen...
Access restricted. Please log in or start a trial to view this content.
Danksagungen
Wir möchten dankt Herrn Dr. Antonio Miranda-Vizuete für seine wertvolle Hilfe wollen. Wir möchten auch unseren Dank an Salma Boulayoune und Isabel Caballero Ausdruck für wertvolle technische Hilfe. Diese Arbeit wurde durch einen Zuschuss von der Junta de Andalucía (BIO-272) finanziert. Diese Forschung wurde in Absprache mit den aktuellen Gesetzen genetische Experimente in Europa durchgeführt.
Access restricted. Please log in or start a trial to view this content.
Materialien
Tabelle 1. C. elegans Stämme.
| Name | Company | Catalog Number | Comments |
| Belastung | Gen | Allele | Quelle |
| Bristol N2 | - | - | ÖCGK |
| VC228 | NLG-1 | ok259 | CGC |
| FX00474 | NLG-1 | tm474 | bNBP-JAPAN |
| VC1416 | NRX-1 | ok1649 | CGC |
| FX1961 | NRX-1 | tm1961 | NBP-JAPAN |
| NL2099 | GebO-3 | pk1436 | CGC |
| CRR21 | NRX-1; NLG-1 | ok1649; ok259 | Diese Arbeit |
| CRR22 | NRX-1; NLG-1 | tm1961; ok259 | Diese Arbeit |
a. Caenorhabditis Genetic Center, University of Minnesota, USA.
b. Nationale Bioresource Projekt für das Experimental Animal "Nematoden C. elegans". Tokyo Womens Medical University, Japan.
Referenzen
- Sudhof, T. C. Nature. 455 (7215), 903-903 (2008).
- Fabrichny, I. P., Leone, P., Sulzenbacher, G. Neuron. 56 (6), 979-979 (2007).
- Garber, K. Science. 317 (5835), 190-190 (2007).
- Wang, K., Zhang, H., Ma, D. Nature. 459 (7246), 528-528 (2009).
- Ward, S., Thomson, N., White, J. G. The Journal of comparative neurology. 160 (3), 313-313 (1975).
- Bargmann, C. I., Thomas, J. H., Horvitz, H. R. Cold Spring Harbor symposia on quantitative biology. 55, 529-529 (1990).
- White, J. G., Southgate, E., Thomsom, J. N., Brenner, S. Philos. Trans. R. Soc. Lond. B Biol. Sci. 314, 1-1 (1986).
- Culotti, J. G., Russell, R. L. Genetics. 90 (2), 243-243 (1978).
- Fire, A., Xu, S., Montgomery, M. K. Nature. 391 (6669), 806-806 (1998).
- Timmons, L., Fire, A. Nature. 395 (6705), 854-854 (1998).
- Simmer, F., Tijsterman, M., Parrish, S., Koushika, S. P., Nonet, M. L., Fire, A., Ahringer, J., Plasterk, R. H. Curr Biol. 12, 1317-1317 (2002).
- Missler, M., Zhang, W., Rohlmann, A. Nature. 423 (6943), 939-939 (2003).
- Varoqueaux, F., Aramuni, G., Rawson, R. L. Neuron. 51 (6), 741-741 (2006).
- Graf, E. R., Zhang, X., Jin, S. X., Scheiffele, P., Fan, J., Choih, J. Cell. 119 (7), 1013-1013 (2004).
- Scheiffele, P., Fan, J., Choih, J. Cell. 101 (6), 657-657 (2000).
- Jamain, S., Quach, H., Betancur, C. Nature genetics. 34 (1), 27-27 (2003).
- Szatmari, P., Paterson, A. D., Zwaigenbaum , L. Nature genetics. 39 (3), 319-319 (2007).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten