Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Laser Capture Mikrodissektion von Drosophila Peripheren Neuronen
In diesem Artikel
Zusammenfassung
In diesem Video-Artikel präsentieren wir eine Methode zur Isolierung von einzelnen oder mehreren Drosophila Da Neuronen aus dritten Larvenstadium mit der Infrarot-Aufnahme (IR)-Klasse von Laser Capture Mikrodissektion (LCM). RNA aus den isolierten Neuronen erhalten können leicht für nachgelagerte Anwendungen wie qRT-PCR oder Microarray-Analysen verwendet werden.
Zusammenfassung
Die dendritischen Verzweigung (da) Neuronen des Drosophila peripheren Nervensystem (PNS) liefern ein hervorragendes Modell, in dem die zugrunde liegenden molekularen Mechanismen klassenspezifischen Dendriten Morphogenese 1,2 untersuchen. Zur Erleichterung der molekularen Analyse von klassenspezifischen da Neuron Entwicklung ist es wichtig, diese Zellen in eine reine Population zu erhalten. Obwohl eine Reihe von verschiedenen Zell-und Gewebe-spezifische RNA-Isolierung Techniken existieren für Drosophila-Zellen, einschließlich Magnetic-Bead-basierten Zellreinigung 3,4, Fluorescent Activated Cell Sorting (FACS) 5-8, und RNA bindenden Proteins Strategien 9, keine Diese Methoden können leicht zur Isolierung von einzelnen oder mehreren Klassen-spezifischen Drosophila da Neuronen mit einem hohen Grad an räumlicher Präzision eingesetzt werden. Laser Capture Mikrodissektion (LCM) wurde als ein äußerst leistungsfähiges Werkzeug, das benutzt, um bestimmte Zelltypen aus Gewebeschnitten isoliert mit einem hohen Grad an räumlicher Auflösung und Genauigkeit kann entstanden. RNA aus isolierten Zellen erhalten kann dann für Analysen einschließlich qRT-PCR und Microarray-Expression Profiling innerhalb einer bestimmten Zelltyp 10-16 verwendet werden. Bis heute hat LCM nicht umfassend in die Analyse von Drosophila Gewebe und Zellen 17,18, einschließlich da Neuronen an der dritten Larvenstadium Larvenstadium der Entwicklung eingesetzt.
Hier präsentieren wir unsere optimiertes Protokoll für die Isolierung von Drosophila da Neuronen mit der Infrarot-(IR)-Klasse von LCM. Diese Methode ermöglicht die Erfassung von Einzel-, Klassen-spezifischen oder mehrere da Neuronen mit hoher Spezifität und räumlicher Auflösung. Alter abgestimmten dritten Larvenstadium Ausdruck eines UAS-mCD8:: GFP 19 Transgen unter der Kontrolle von entweder der Klasse IV da Neuron spezifischen ppk-GAL4 20 Treiber oder in den gesamteuropäischen da Neuron spezifischen 21-7-GAL4 21 Treiber wurden für diese verwendet Experimente. RNA aus den isolierten da Neuronen erhalten ist von sehr hoher Qualität und kann direkt für nachgelagerte Anwendungen, einschließlich qRT-PCR oder Microarray-Analysen verwendet werden. Darüber hinaus kann diese LCM-Protokoll leicht angepasst werden, zu erfassen anderen Drosophila Zelltypen eine verschiedene Stadien der Entwicklung abhängig vom Zelltyp spezifisch, GAL4-driven Expressionsmuster von GFP.
Protokoll
Allgemeine Bemerkungen zum LCM von Drosophila peripheren Neuronen
Allow from 6 Stunden, bis zu einer Woche oder länger für LCM je nach Gewebetyp und die Anzahl der Zellen erforderlich.
Alle Verfahren sind in streng RNase-freien Bedingungen gemäß Standardverfahren durchgeführt. Larven, die entweder 21-7-GAL4, UAS-mCD8:: GFP oder ppk-GAL4, UAS-mCD8:: GFP transgenen Reporter Linien wurden für diese Experimente verwendet.
1. Vorbereitung der Larven
- Sammeln 30-40 gleichaltrigen dritten Larvenstadium und waschen Sie sie in ddH 2 O, gefolgt von kurzen Spülen in RNAse Away (Sigma-Aldrich) und einem letzten Waschen in ddH 2 O auf RNase AWAY entfernen. Wick das überschüssige ddH 2 O vollständig mit einem sauberen Kimwipe vor dem Einbetten die Larven in Oktober
- Nehmen Sie ein sauberes Gewebe Einbettform und Schicht mit einer dünnen (1,5-2mm) Schicht von OCT, gerade genug, um eine einzige Schicht von Larven zu decken.
- Vor Einbettung der Larven, wenn nötig, kühle der OCT auf 0 ° C in das Gewebe Einbettformen. Tun Sie dies, indem Sie die Form mit ÜLG auf einem Eisblock. Dies wird dazu beitragen, Larven Bewegung während der Einbettung.
- Legen Sie die sauberen Larven auf die vorgekühlte OCT und ordnen sie in parallel zueinander. Sobald die Larven haben angeordnet worden, langsam füllt die Form mit OCT, ohne die Larven Anordnung. Snap Einfrieren der Form, indem sie auf einem Trockeneis-Block. [Critical Schritt:. Snap-freeze die Larven sofort an Bewegung zu reduzieren] Diese Methode erlaubt die Larven in einer Ebene sein, die Maximierung der Anzahl von Zellen zur Verfügung pro Abschnitt für die Erfassung.
Wenn es notwendig ist, um das Gewebe Morphologie für die Identifizierung der Zellen von Interesse zu wahren, oder wenn die erwartete Anzahl von Zellen pro Abschnitt ist als zufriedenstellend befunden dann gehen Sie direkt zu Schritt 11 fort.
Alternativ, wenn die Zellen, die spezifisch und gekennzeichnet sind, können mit einer leicht erkennbaren Marker, wie GFP oder RFP identifiziert werden, aber die Anzahl der Zellen pro Abschnitt gefunden wird gering sein, dann die Schritte 5-10 kann hilfreich sein für die Erhöhung der Anzahl von Zellen zur Verfügung für die Erfassung pro Abschnitt. Dies sind optionale Schritte und sollte, falls erforderlich, als eine Methode zur Erhöhung der Zahl der Neuronen da für LCM pro Abschnitt, wenn die andere Methode versagt, um günstige Ergebnisse zu liefern. [Achtung: Diese Methode kann stören die gesamte Gewebemorphologie und machen Gewebe Identifikation auf bestimmte räumliche Lage der Grundlage sehr schwierig.]
- Wash 50-70 dritten Larvenstadium wie in Schritt 1 beschrieben. Legen Sie die Larven in einem 1,5 ml Mikrozentrifugenröhrchen mit 500 ul RNAse freien 1X PBS.
- Dounce die Larven mit einem Polypropylen-Stößel (USA Scientific), mit 6-7 langsamen kräftigen Griffen.
- Zentrifugieren Sie die Lösung bei 16.000 (x) g für 5-10 Sekunden (bis die Larven Nagelhaut nieder auf den Boden der Mikrozentrifugenröhrchen]. Überstand verwerfen. Das Pellet sollte in erster Linie von Larvencuticula bestehen, auf die die PNS, einschließlich da Neuronen ist dicht verklebt.
- Waschen Sie die Nagelhaut Pellet 2-3 mal in 1X PBS und das Pellet in 500 ul 1X PBS. Drehen Sie die Lösung bei 16.000 (x) g für 1 Minute zu einer kompakten Form von Pellets. Saugen Sie den Überstand vollständig mit einer feinen Pasteurpipette.
- Entfernen Sie vorsichtig das Pellet aus der Mikrozentrifugenröhrchen mit einem sauberen Spatel, und Docht das überschüssige PBS mit einem sauberen Kimwipe Gewebe. [Anmerkung: Es ist wichtig, um das Pellet so kompakt wie möglich zu halten, um die Zelle Ertragssteigerung]. Platzieren Sie den kompakten Nagelhaut Pellets auf einer Kunststoff-Form mit einer dünnen Schicht von OCT (1mm ca.).
- Vorsichtig aus dem Pellet in einer dünnen, kreisförmigen Fläche verteilt. Füllen Sie die Form mit OCT, und frieren das Gewebe wie in Schritt 4 beschrieben.
- Mit Hilfe eines Kryostaten geschnitten Gefrierschnitten bei 5-8 mu m Dicke auf Normalpapier, etikettiert, ungeladen, RNAse freie Objektträger aus Glas. [Critical Schritt: Positionieren Sie den Gewebeschnitten in der Nähe der Mitte der Folie.]
- Bewahren Sie die Folien entweder direkt auf Trockeneis oder bei -80 ° C in einen sauberen Objektträger-Box, bis sie zur Mikrodissektion. Führen LCM vorzugsweise innerhalb einer Woche nach Schneiden des Gewebes.
2. Dehydration und Nagelhaut entfernen von Frozen Larven Sections
[Critical Schritt: Alle Lösungen zur Entwässerung müssen bereit sein, vor jeder Sitzung LCM frisch. Vollständige Dehydratisierung von gefrorenen Larven Gewebeschnitten für die Erreichung optimaler Mikrodissektion Effizienz Essenziell]
- Entfernen Sie die Folien mit den gefrorenen Larven Gewebeschnitten aus der -80 ° C Gefrierschrank und legen Sie sie auf Trockeneis.
- Entfernen Sie eine einzelne Folie aus Trockeneis und sofort legen Sie es direkt in eine 50 ml konische tumit 70% Ethanol Fixativ, durch ein kurzes Spülen in RNAse freien ddH 2 O entsprechend der empfohlenen Zeiten in Tabelle 1 dargestellt, gefolgt gefüllt werden.
- Die Anwesenheit von Larvencuticula in den Gefrierschnitten kann die Effizienz des Fanges durch die Verhinderung effizienter LCM cap-Zell-Kontakt, die unspezifische Aufnahme führen kann beeinträchtigt werden. Wenn nötig, führen Sie die folgenden Schritte (4-5), um die Schuppenschicht aus Gewebeschnitten klar.
- Gently Pipette 50 ul 2,5% Trypsin direkt auf den Gewebeschnitten und inkubieren für 5-30 Sekunden bei Raumtemperatur. [Critical Schritt: Dieser Schritt kann optimiert werden müssen. Die Inkubationszeit hängt von der Gewebe mikrodissezierten und Schnittdicke. Längere Inkubation kann klar alles, einschließlich der Zellen von Interesse, während einer kurzen Inkubationszeit kann nicht ausreichen, um die Nagelhaut entfernen]
- Spülen Sie die Teile kurz in einer 50 ml konische Röhrchen mit RNAse-freie ddH 2 O, um Trypsin zu entfernen, und locker eingehalten Nagelhaut Fragmente.
- Tauchen Sie die Folien der Reihe nach in jedem der verbleibenden Ethanol und Xylol-Lösungen für die empfohlenen Zeiten (Tabelle 1), um die Entwässerung zu vervollständigen.
- Nach der Entwässerung Gradienten werden die Objektträger kurz unter einer milden Luftstrom für 60-120 Sekunden bei Raumtemperatur getrocknet vor der Durchführung LCM. [Achtung: Dry das Xylol vollständig von der Gewebeschnitte vor der Durchführung LCM. Xylol ist bekannt, die LCM Kappe resultierende Polymer in Mikrodissektion Fehler] auflösen.
| 70% Ethanol (Fixativ) | 3-10 Sekunden |
| ddH 2 O | 5-10 Sekunden |
| 2,5% Trypsin Incubation | 5-30 Sekunden |
| ddH 2 O | 5-10 Sekunden |
| 70% Ethanol | 60 Seconds |
| 95% Ethanol | 60 Seconds |
| 100% Ethanol | 120 Sekunden |
| 100% Ethanol | 120 Sekunden |
| 100% Xylol | 120 Sekunden |
| 100% Xylol | 120 Sekunden |
| Der Luft trocknen in einem milden Luftstrom | 60-120 Sekunden |
Tabelle 1: Empfohlene Verfahren für die LCM Dehydrierung von gefrorenen Larven Gewebeschnitten.
3. Laser Capture Mikrodissektion
PixCell IIe LCM Instrument mit Fluor 300 Epifluoreszenz-Optik für EGFP optimiert ausgestattet war für die Durchführung der LCM verwendet.
[Critical Schritt: Wenn möglich, führen Sie die Mikrodissektion in einer Feuchte-kontrollierten Raum zu einer Minderung Mikrodissektion Effizienz aufgrund der erhöhten Feuchtigkeit zu verhindern. Raumfeuchte ist ein kritischer Faktor für die Mikrodissektion Effizienz. Niedrige Raumfeuchte kann eine erhöhte statische klammern sich in unspezifischen erfassen, während hohe Raumfeuchte in niedrigen Mikrodissektion Effizienz führen kann. Wir erzielten eine optimale LCM Effizienz zwischen 25-50% relative Luftfeuchtigkeit.]
- Schalten Sie die Stromversorgung für das Mikroskop und Laser-Schaltkasten.
- Laden Sie die CapSure Kappe Halter Montage mit HS LCM caps (Molecular Devices): Zwei Patronen von LCM Kappen können auf einmal geladen werden.
- Öffnen Sie das Molecular Devices Software und geben Sie das Experiment Details einschließlich Foliennummer und Cap Losnummer.
- Legen Sie eine neue HS LCM Kappe von der Patrone auf die PixCell IIe LCM Instrument und positionieren Sie es korrekt in Bezug auf den Joystick, um die richtige Positionierung der Kappe in Bezug auf die Capture-Bereich zu gewährleisten.
- Legen Sie den Objektträger mit frisch entwässertem Larvengewebe Abschnitte auf dem Mikroskoptisch für die Mikrodissektion.
- Suchen Sie die fluoreszenzmarkierten Zellen mit den Okularen oder dem Computer-Bildschirm. Aufgrund der hohen Spezifität der PPK-GAL4, UAS-mCD8:: GFP transgenen Reporter Linie, kann die Klasse IV da Neuronen leicht von GFP-Fluoreszenz identifiziert werden.
- Betreff des Gewebes zu LCM mit CapSure HS LCM Kapseln [für eine detaillierte Anweisungen für die Einstellung des Gerätes, Fokussierung des Laserstrahls und Durchführung LCM siehe Referenz 22].
- Stellen Sie die "Power" und "Dauer"-Laserpuls Parameter, um eine präzise geschmolzene Polymer vor Ort, dessen Größe entspricht dem gewählten Messfleck zu erreichen. Passen Sie die Einstellungen für die Größe der geschmolzene Polymer-Bereich anpassen. Die folgenden Parameter wurden für microdissecting einzigen Klasse IV da Neuronen verwendet: Spot Durchmesser 7,5 um, Laser-Stärke 30-50 mW, Laser-Zeit und 2-4 Ms und 1-2 Schüsse pro Zelle verwendet wurden. [Critical Schritt: die Laser-Parameter für einen richtigen Stelle erforderlich kann mit Gewebedicke und Raumfeuchte Bedingungen variieren. Stellen Sie die "Power" und "Dauer"-Einstellungen, um die erforderliche geschmolzene Polymer Spotgröße für microdissecting einzelne oder mehrere Zellen zu erreichen.]
- Zur Maximierung der Spezifität, microdissect Zellen mit minimaler Überlappung und eine starke Fluoreszenz. Jede Kappe ist für den Fang zahlreicher Zellkörper. [Critical Schritt: Zur Vermeidung von RNA-Abbau, schränken die gesamte Messzeit einschließlich Entwässerung und Mikrodissektion zu weniger als 45 Minuten]
- Sobald alle Zellen von Interesse aufgenommen wurden, heben Sie den HS LCM Kappe mit dem mikrodissezierten Zellen und legte es auf einen sauberen Objektträger um die Präsenz der eingefangenen Zellen zu bestätigen und eine Sichtprüfung der Kappe auf das Vorhandensein von unerwünschten Zellen oder Schmutz.
4. RNA-Isolierung aus LCM abgeleiteten Zellen
- Bringen Sie die Kappe mit dem mikrodissezierten Zellen zu einem ExtracSure (Molecular Devices) Gerät. Fügen Sie 12μl der RNA-Extraktionspuffer (PicoPure, Molecular Devices) auf die Kappe Oberfläche mit den Zellen und verbinden ein umgekehrtes dünnwandigen Reaktionsrohr (GeneAmp, Applied Biosystems), um die ExtracSure Gerät. Legen Sie die Montage auf eine Ausrichtung Fach (Molecular Devices) und decken Sie es mit einer Inkubationszeit Block (Molecular Devices) vorgewärmt auf 42 ° C innerhalb eines Inkubators.
- Nach einer Inkubationszeit von 30 Minuten, Zentrifuge die Röhrchen mit den Extrakten bei 800 (x) g für 2 Minuten. Schließen Sie die Rohre und speichern Sie die Zellextrakte bei -80 ° C bis sie für RNA Aufreinigung.
- Extrahieren und Spalte reinigen die RNA nach dem PicoPure (Molecular Devices) RNA-Extraktion Kit Anweisungen. DNAse Behandlung ist optional und kann auf der Säule während der RNA-Reinigung nach Bedarf durchgeführt werden. Falls erforderlich, können mehrere LCM-Proben zusammen während der Reinigung in einer einzigen Spalte zur endgültigen RNA-Ausbeute zu erhöhen gebündelt werden und kann in einem kleinen Volumen (11-30 ul) Elutionspuffer (PicoPure, Molecular Devices) eluiert und bei -80 ° C bis zur Verwendung. Auf Wunsch eine 1 ul-Aliquot verwendet werden, um Gesamt-RNA Qualität auf einem Bioanalyzer 2100 (Agilent Technologies, Inc.) zu beurteilen.
Repräsentative Ergebnisse
Laser Mikrodissektion (LCM) wurde verwendet, um einzelne, klassenspezifische oder mehrere Drosophila da Neuronen aus dritten Larvenstadium (Abb. 1) zu isolieren. LCM ermöglicht eine hohe Präzision und Spezifität bei der Isolierung von Drosophila da Neuron Zellkörper (Abbildung 2a-c). Darüber hinaus wurde die Gesamt-RNA aus diesen isoliert da Neuronen gereinigt gefunden von ausgezeichneter Qualität sein, wie durch das Vorhandensein von scharfen 5.8S, 18S und 28S ribosomalen RNA Spitzen angezeigt, wenn auf einem Agilent 2100 Bioanalyzer (Agilent Technologies, Inc.) (Abbildung analysiert 2d).
Zur Beurteilung der neuronal-spezifische Anreicherung unserer isolierten Zellen führten wir real-time quantitative reverse Transkription PCR (qRT-PCR) mit Hilfe der neuronalen Gen-spezifischer Marker, Elav. Für die qRT-PCR-Analysen berechneten wir die relativen Pegel der Elav Ausdruck in der LCM mikrodissezierten da Neuronen und normalisiert diesen Ausdruck, dass der endogene Kontrolle (rp49) und bezogen auf Gesamt-RNA aus ganzen Larven mit dem ΔΔCt Methode 23 isoliert. Diese Analysen zeigten, über 2,5 fache Anreicherung in den relativen Pegel der Elav in LCM Mikro-seziert da Neuron Proben, ganze Tiere (Abbildung 3) verglichen.
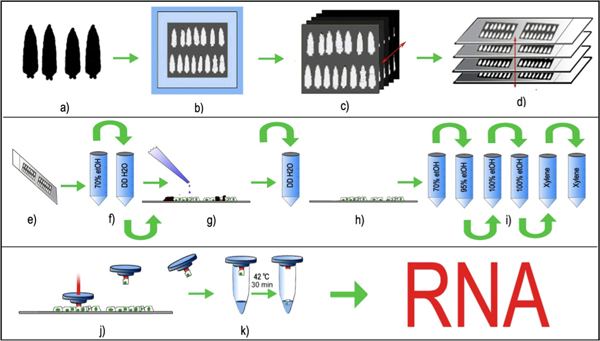
Abbildung 1: LCM von Drosophila peripheren Neuronen. (A) Alter abgestimmt dritten Larvenstadium ausgewählt, gewaschen und (b) in einer cryomold mit OCT eingebettet und bei -80 ° C, (c) 8μm serielle Gewebeschnitte werden mit einer Standard-Kryostaten und (d) platziert gleichmäßig auf sauberen Glasobjektträger. Die Glasträger mit den angebrachten Gewebeschnitte werden bei -80 ° C vor dem LCM Verarbeitung. (E, f) Unmittelbar vor LCM, die Gewebeschnitte werden aufgetaut und kurz fixiert in 70% igem Ethanol durch kurze in ddH 2 O. spülen gefolgt (g) Die Folien werden im Folgenden kurz mit Trypsin inkubiert und gespült mit ddH 2 O auf Nagelhaut (schwarz) aus dem larvalen Abschnitte. (h, i) Gewebeschnitte ohne Larvencuticula entfernen werden dann weiter dehydriert in ein Ethanol-Gradienten und schließlich gelöscht in Xylol . (j) Die LCM Kappe mit einem thermolabile Polymer ist auf dem Gewebeschnitt platziert und der Laser wird auf den ausgewählten fluoreszierende Zellkörper gepulst. Der Laserpuls schmilzt das Polymer und verschlingt die markierte Zelle Körper. Das Polymer Kappe, zusammen mit den erfassten Zellkörper angehoben wird, und (k) RNA-Extraktionspuffer wird in die Zelle aufgenommen. Die aufgenommenen Zelle, zusammen mit der RNA-Extraktionspuffer ist bei 42 ° C für 30 Minuten inkubiert und kann entweder bei -80 ° C gelagert werden, oder direkt für die RNA-Reinigung weiterverarbeitet.
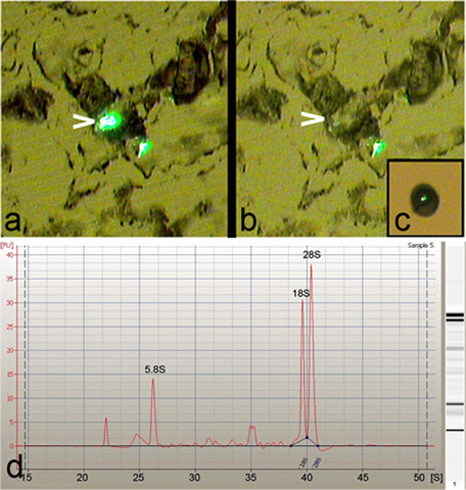
Abbildung 2: LCM ermöglicht eine hohe Präzision einfangen von da Neuronen. (A) Repräsentative Bild eines dehydriert und Trypsin behandelt 8 Mikron Gewebeschnitt vor der Durchführung LCM, mit zwei Klasse-IV da Neuronen mit GFP durch die PPK-GAL4 gekennzeichnet, UASmCD8:: GFP-Reporter-Stamm. Beachten Sie, dass eine der Neuronen markiert (Pfeilspitze) für die Erfassung. (B) Zellkörper der Neuronen markiert (Pfeilspitze) sauber ist mit hoher Spezifität aus dem Gewebeschnitt Mikro-seziert. (C) End-on Ansicht einer einzelnen Klasse -IV da Neuron auf der LCM cap erfasst. (d) Agilent 2100 Bioanalyzer (Agilent Technologies, Inc.) Elektropherogramm Gesamt-RNA aus LCM-derived da Neuronen isoliert, die eine hervorragende RNA-Qualität als durch das Vorhandensein von scharfen 5.8S, 18S angegeben und 28S rRNA Peaks.
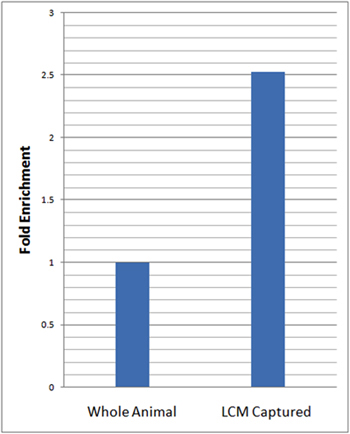
Abbildung 3:. QRT-PCR zeigt wesentliche Bereicherung der neuronalen Marker Genexpression in LCM eingefangen da Neuronen relativ zu ganzen Tier qRT-PCR-Analysen von Neuronen-spezifische Marker Genexpression (Elav) in LCM eingefangen da Neuronen (21-7-GAL4, UAS-mCD8:: GFP) und ganze Tiere im Alter abgestimmt dritten Larvenstadium wurde in dreifacher Ausführung durchgeführt. Relative Ebenen Elav Ausdruck in LCM eingefangen da Neuronen wurden die endogenen Kontrolle (rp49) und relativ zum gesamten Larven mit dem ΔΔCt Methode 23 normalisiert. Ein 2,5-fache Anreicherung in den relativen Pegel der Elav in LCM eingefangen da Neuron Proben relativ zu ganzen Tieren beobachtet wurde.
Fehlerbehebung
Problem: RNA abgebaut, oder von geringer Qualität.
Stellen Sie sicher, RNAse freien Bedingungen im gesamten Experiment. Verringern Sie viel Zeit auf LCM pro Objektträger verbracht bis 30 Minuten. Halten Sie die Folien und die Proben auf Trockeneis, außer bei der Durchführung von LCM. Vermeiden Sie Auftauen der Gewebeschnitte, wenn sie geschnitten werden.
Problem: Die meisten Zellen sind aus der Folie während Trypsin-Behandlung (Schritt 17-18) verloren.
Reduzieren Sie die Zeit der Trypsin-Behandlung. Reduzieren Sie die Trypsin-Konzentration.
Problem: Präsenz der Kutikula und anderen unspezifischen Gewebereste auf der Kappe Polymer.
Versuchen Sie, die Gewebereste, indem man die Polymer-Oberfläche mit der klebrigen Seite eines Klebers beachten 22.
Problem: Geringe LCM Effizienz.
Erhöhen Sie die Inkubationszeit in 100% Ethanol und Xylol. Reduzieren Raumfeuchte mit Entfeuchter.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Das Protokoll hier vorgestellte beschreibt unsere optimierte Methode zur Isolierung von Drosophila peripheren Neuronen über LCM. Während dieser LCM-Protokoll für die spezifische Isolierung von Einzel-, Klassen-spezifischen oder mehrere Drosophila da Neuronen ab dem dritten Larvenstadium Larvenstadium der Entwicklung konzipiert wurde, konnte geringfügige Änderungen des Protokolls ohne weiteres für den Fang von anderen Drosophila Zelltypen aus allen Entwicklungs angepasst werden Phasen mit...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Danksagungen
Wir danken Drs. Yuh-Nung Jan und Wes Grueber für die Bereitstellung von fly Aktien in dieser Studie verwendet wird, und Virginia Espina, Dr. Emanuel Petricoin und Dr. Lance Liotta für die Unterstützung bei LCM. Die Autoren danken Thomas F. und Kate Miller Jeffress Memorial Trust für die Unterstützung dieser Forschung (DNC) und der George Mason University Provost s Office (EPRI).
Access restricted. Please log in or start a trial to view this content.
Materialien
Ausstattung:
- Kryostat
- 50 ml konischen Rohr für Dia-Fixierung, Spülen, Trypsin-Behandlung und Ethanol / Xylol Dehydration
- -80 ° C Gefrierschrank
- Inkubator
- PixCell IIe LCM Instrument mit Fluor 300 Epifluoreszenz-Optik für EGFP (Molecular Devices-Molecular Devices) optimiert
Referenzen
- Corty, M. M., Matthews, B. J., Grueber, W. B. Molecules and mechanisms of dendrite development in Drosophila. Development. 136, 1049-1061 (2009).
- Parrish, J. Z., Emoto, K., Kim,, Jan, Y. N. Mechanisms that regulate establishment, maintenance, and remodeling of dendritic fields. Annu. Rev. Neurosci. 30, 399-423 (2007).
- Wang, X., Starz-Gaiano, M., Bridges, T., Montell, D. Purification of specific cell populations from Drosophila tissues by magnetic bead sorting, for use in gene expression profiling. Nature Protocols. , (2008).
- Iyer, E. P. R., Iyer, S. C., Sulkowski, M. J., Cox, D. N. Isolation and purification of Drosophila peripheral neurons by magnetic bead sorting. J Vis Exp. 34, 2912-2917 (2004).
- Tirouvanziam, R., Davidson, C. J., Lipsick, J. S., Herzenberg, L. A., Tirouvanziam, R., Tirouvanziam, R. Fluorescence-activated cell sorting (FACS) of Drosophila hemocytes reveals important functional similarities to mammalian leukocytes. Proc. Natl. Acad. Sci. 101, 2912-2917 (2004).
- Shigenobu, S., Arita, K., Kitadate, Y., Noda, C., Kobayashi, S. Isolation of germline cells from Drosophila embryos by flow cytometry. Dev. Growth Differ. 48, 49-57 (2006).
- Reeves, N., Posakony, J. W. Genetic programs activated by proneural proteins in the developing Drosophila PNS. Dev. Cell. 8, 413-425 (2005).
- Jinushi-Nakao, S. Knot/Collier and Cut control different aspects of dendrite cytoskeleton and synergize to define final arbor shape. Neuron. 56, 963-978 (2007).
- Yang, Z., Edenberg, H. J., Davis, R. L. Isolation of mRNA from specific tissues of Drosophila by mRNA tagging. Nucl. Acids Res. 33, (2005).
- Appay, V. Sensitive Gene Expression Profiling of Human T Cell Subsets Reveals Parallel Post-Thymic Differentiation for CD4+ and CD8+ Lineages. J. Immunol. 179, 7406-7414 (2007).
- Ginzinger, D. G. Gene quantification using real-time quantitative PCR: An emerging technology hits the mainstream. Exp. Hematol. 30, 503-512 (2002).
- Keays, K. M., Owens, G. P., Ritchie, A. M., Gilden, D. H., Burgoon, M. P. Laser capture microdissection and single-cell RT-PCR without RNA purification. J. Immunol. Methods. 302, 90-98 (2005).
- Volgin, D. V., Swan, J., Kubin, L. Single-cell RT-PCR gene expression profiling of acutely dissociated and immunocytochemically identified central neurons. J. Neurosci. Methods. 136, 229-236 (2004).
- Emmert-Buck, M. R. Laser capture microdissection. Science. 274, 998-1001 (1996).
- Xiao, W. Gene expression profiling in embryonic mouse lenses. Mol. Vis. 12, 1692-1698 (2006).
- Scharschmidt, T. Analysis of human osteoarthritic connective tissue by laser capture microdissection and QRT-PCR. Connect. Tissue Res. 48, 316-323 (2007).
- Spletter, M. L. regulates Drosophila olfactory projection neuron identity and targeting specificity. Neural Dev. 2, 14-14 (2007).
- Hoopfer, E. D., Penton, A., Watts, R. J., Luo, L. Genomic analysis of Drosophila neuronal remodeling: a role for the RNA-binding protein Boule as a negative regulator of axon pruning. J. Neurosci. 28, 6092-6103 (2008).
- Lee, T., Luo, L. Mosaic analysis with a repressible cell marker for studies of gene function in neuronal morphogenesis. Neuron. 22, 451-461 (1999).
- Grueber, W. B. Projections of Drosophila multidendritic neurons in the central nervous system: links with peripheral dendrite morphology. Development. 134, 55-64 (2007).
- Song, W., Onishi, M., Jan, L. Y., Jan, Y. N. Peripheral multidendritic sensory neurons are necessary for rhythmic locomotion behavior in Drosophila larvae. Proc. Natl. Acad. Sci. USA. 104, 5199-5204 (2007).
- Espina, V. Laser capture microdissection. Nat. Prot. 1, 586-603 (2006).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method. Methods. 25, 402-408 (2001).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten