Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
DNA Stable-Isotope Probing (DNA-SIP)
In diesem Artikel
Zusammenfassung
DNA stabilen Isotopen ist ein Anbau-unabhängige Methode zur Identifizierung und Charakterisierung aktiver Gemeinschaften von Mikroorganismen, die fähig werden bestimmte Substrate sind. Assimilation von Substrat in schweren Isotop angereichert führt zu Einbau von markierten Atome in mikrobielle Biomasse. Dichtegradienten-Ultrazentrifugation ruft markierter DNA für nachgeschaltete molekulare Analysen.
Zusammenfassung
DNA stabilen Isotopen (DNA-SIP) ist ein leistungsfähiges Verfahren zur Identifizierung von aktiven Mikroorganismen, insbesondere Kohlenstoff-Substraten und Nährstoffe in zelluläre Biomasse zu assimilieren. Als solcher hat dieser Anbau-unabhängige Technik eine wichtige Methodik für die Vergabe metabolische Funktion, um die verschiedenen Gemeinschaften bewohnen eine Vielzahl von terrestrischen und aquatischen Umgebungen. Nach der Inkubation eines Umwelt-Probe mit stabilen Isotopen markierten Verbindungen, extrahiert Nukleinsäure Dichtegradienten-Ultrazentrifugation und die anschließende Steigung Fraktionierung unterzogen, um Nukleinsäuren unterschiedlicher Dichte zu trennen. Reinigung von DNA aus Cäsiumchlorid ruft beschriftet und unbeschriftet DNA für die anschließende molekulare Charakterisierung (z. B. Fingerabdrücke, Microarrays, Klon-Bibliotheken, Metagenomik). Diese JoVE Video-Protokoll bietet visuelle Schritt-für-Schritt-Erklärungen für das Protokoll für die Dichtegradienten-Ultrazentrifugation, Gradienten Fraktionierung und Rückgewinnung von markierten DNA. Das Protokoll enthält außerdem Beispielcode SIP Daten und hebt wichtige Tipps und Hinweise, die zu berücksichtigen, um eine erfolgreiche DNA-SIP-Analyse zu gewährleisten.
Protokoll
1. Vorbereitung der Reagenzien
DNA-SIP erfordert die Verwendung von Reagenzien, die im Vorfeld der eigentlichen Verfahren hergestellt werden sollte. Die Vorschrift zur Herstellung jedes Reagenz werden in diesem Abschnitt aufgeführt sind und aus einer früheren SIP-Protokoll 1 geändert.
- Cäsiumchlorid (CsCl)-Lösung für die Vorbereitung SIP Gradienten - Bereiten Sie eine 7,163 M CsCl-Lösung durch die schrittweise Auflösung 603,0 g CsCl in destilliertem und entionisiertem Wasser (ddH 2 O) auf ein Endvolumen von 500 mL. Achten Sie darauf, nicht zu 500 mL überschreiten! Erwärmen der Lösung leicht unter Rühren auflösen helfen alle CsCl. Aliquot der endgültigen Lösung in verschlossenen Aliquots. In unserem Labor ist eine gemeinsame Lagerung der Praxis bis 100-ml-Aliquoten in 125-mL Serumfläschchen, die dann Crimp-versiegelt mit Butylgummistopfen vorzubereiten. Die versiegelten Aliquots unbegrenzt bei Raumtemperatur (20 ° C) gelagert werden. Die Dichtungen verhindern Verdunstung und CsCl "Kruste" Bildung. Bestimmen Sie die Dichte der Lösung durch Wiegen dreifach 100-ul Aliquots, oder mit einem digitalen Refraktometer (z. B. Reichert AR200), die sorgfältig für CsCl-Lösungen kalibriert. Nach der Kalibrierung erfolgreich, wird die Reichert AR200 konsistent und liefert präzise Messwerte für mehrere Jahre. Bei Raumtemperatur (20 ° C), die endgültige Dichte dieser Lösung liegt typischerweise im Bereich von 1,88 bis 1,89 g ml -1. Die Dichte variiert leicht jedes Mal ein neues Lager vorbereitet.
- Cäsiumchloridlösung Vorbereitung Gradienten mit Ethidiumbromid (EtBr) - Kombinieren Sie 250 g CsCl mit 250 ml sterile ddH 2 O Wasser. Aliquot dieser Lösung in separate Serumfläschchen, die Crimp-versiegelt wurden mit Butyl-Kautschuk-Dichtungen, wie in 1.1 beschrieben.
- Gradient Buffer - Kombinieren Sie 50 ml von 1 M Tris-HCl, 3,75 g KCl und 1 ml 0,5 M EDTA auf 400 ml Wasser. Lösen Sie die KCl, dann fügen Sie ddH 2 O auf 500 ml. Filter-sterilisiert und Autoklaven. Die endgültige Lösung ist 0,1 M Tris, 0,1 M KCl und 1 mM EDTA.
- Polyethylenglykol (PEG)-Lösung - Bereiten Sie die PEG-Lösung durch Auflösen von 150 g Polyethylenglykol 6000 und 46,8 g NaCl in sterilem ddH 2 O Wasser auf ein Gesamtvolumen von 500 mL (30% PEG, 1,6 M NaCl). Autoclave.
Hinweis: Diese Lösung trennt sich in zwei Phasen mit Autoklavieren. Fügen Sie einen Rührstab in der Flasche autoklaviert, so dass die Lösung richtig gemischt werden, wenn dies der Fall ist. - TE-Puffer - Bereiten Sie eine Lösung aus 10 mM Tris-HCl (pH 8,0) und 1 mM EDTA (pH 8,0) in sterilen ddH 2 O Wasser, mit autoklaviert Stammlösungen von 1 M Tris-HCl (pH 8,0) und 0,5 M EDTA ( pH 8,0). Filter sterilisieren und Autoklaven.
- 70% Ethanol - Kombinieren Sie 350 ml von hochreinem Ethanol mit 150 ml sterilem ddH 2 O Wasser.
2. Probeninkubation und DNA-Extraktion
Für die DNA-SIP Inkubationen werden die Proben in der Regel mit schweren Isotop Kohlenstoff (13 C) inkubiert. Inkubationszeiten und Bedingungen (zB Nährstoff-Supplementierung, Feuchtigkeit, Licht) ist abhängig von der Art der Probe, inkubiert wird und die Beschaffenheit des Untergrundes. DNA-SIP Experimente erfolgreich durchgeführt wurden mit einer Vielzahl von einzelnen Kohlenstoff-Verbindungen 2,3, multi-Kohlenstoff-Verbindungen 4,5,6 und mit Hilfe markierter Stickstoff oder Sauerstoff 7,8 9. Allerdings ist ein Nachteil bei der Verwendung von 15 N-oder 18 O-markierten Verbindungen der verminderten physischen Trennung der markierten Nukleinsäure, in erster Linie auf das Vorhandensein von weniger Stickstoff-und Sauerstoffatomen in DNA und RNA relativ zur C-Atomen.
Ein kritischer Kontrollpunkt für die DNA-SIP Experimente ist eine identische Inkubation mit nativen (zB 12 C)-Substrat hergestellt. Diese Inkubation bietet einen anschließenden Vergleich zu gewährleisten, dass offensichtliche Kennzeichnung von Nukleinsäure nicht ein Artefakt der Ultrazentrifugation oder G + C-Gehalt Dichteunterschiede in DNA Beitrag zur Trennung 10. Es ist auch wichtig, um gefrorene Probenmaterial für den Vergleich mit "light" und "schweren" DNA zu halten, und den Wert mit einem nicht-Substrat-Kontrolle im Hintergrund zu Änderungen in der gesamten SIP Inkubation zu beurteilen.
- Inkubieren Umweltproben in Mikrokosmen mit markiertem Substrat (Abbildung 1). Nach unserer Erfahrung haben wir festgestellt, dass ein Minimum Einbeziehung zwischen 5-500 pmol 13 C Kohlenstoff pro Gramm der Probe werden für Proben mit hoher Biomasse wie Bodenproben 1. Für im Wasser lebende Proben mit weniger Biomasse als Böden, kann 100 bis 100 pmol integriert 13 C Kohlenstoff pro Liter ergeben einen nachweisbaren schweren Isotopensignatur 1. Die Menge an Kohlenstoff Änderung wird Anteil von Kohlenstoff in Biomasse und Voraussetzung für die zusätzliche Nährstoff-Ergänzung für die Assimilation integriert alle auf die Eigenschaften der Proben analysiert hängen und die gezielte organisms von Interesse. Ein einziger Satz von Probeninkubation Richtlinien sind nicht anwendbar für alle Proben. Wichtig ist, dass die Substratkonzentration für SIP Inkubation verwendet idealerweise so nahe wie möglich, um die Konzentration in der Regel in situ angetroffen; experimentellen Bias kann eine Folge der Anreicherung Kulturbedingungen 10 sein.
- Nach der Inkubation der Probe mit stabilen Isotopen markiertes Substrat, extrahieren DNA aus Mikrokosmen mit einem rigorosen Extraktion Protokoll (PCR oder kleine Einlage Klonen) oder einem vertrauenswürdigen enzymatische Lyse für hochmolekulare Klonen (z. B. große einfügen Metagenomik). RNA co-Extraktion in der Regel nicht beeinflussen Analyse, so Protokolle, die Rendite RNA sowie DNA verwendet werden können. Die Ultrazentrifugation der extrahierten DNA wird nicht Scher-Fragmente kürzer als ~ 50 kb Paare 1.
- Quantify extrahierte DNA vor der CsCl Gradienten-Ultrazentrifugation Röhren-Setup. Quantifizierung der DNA mit einem Spektralphotometer (zB Nanodrop 2000), wenn die Extraktion-Protokoll liefert nur DNA (zB spaltenbasierte Kits). Alternativ zu quantifizieren mittels Agarose-Gelelektrophorese.
3. Vorbereitung Gradient Lösungen für Ultrazentrifugation
Dieses Verfahren beinhaltet das Hinzufügen von DNA zu Rohren Ultrazentrifuge. Es gibt mehr als eine Art von Schlauch und Rotor so das genaue Protokoll wird variieren und sich auf die Anweisungen des Herstellers abhängig. Das heißt, empfehlen wir die Verwendung eines Vertikal-Rotor auch um den maximal möglichen Trennung von leichten und schweren DNA zu gewährleisten. Wir verwenden einen Beckman-Coulter Vti 65,2 Rotor mit 16 Vertiefungen zur Aufnahme von 5,1 ml QuickSeal Polyallomer Rohre und das Protokoll werden die Schritte und Überlegungen für diese Bedingungen zu gewährleisten.
- Mit dem DNA-Konzentrationen in Schritt 2.3 ermittelt, berechnen Sie die benötigte Menge der extrahierten DNA, die erforderlich sind, um 0,5 ug geben wird - 5 ug DNA in der Ultrazentrifuge Röhren.
- Kombinieren Sie extrahierten DNA (0,5 bis 5 ug) mit Gradient Buffer (siehe Schritt 1.3) und 4,8 ml 7,163 M CsCl auf ein Gesamtvolumen von ca. 6 ml in einer sterilen Einweg-15-ml-Tube. Beachten Sie, dass die Dichte der CsCl-Lösung kann auch variieren bei gleicher Molarität (siehe Schritt 1.1). Die folgende Gleichung kann verwendet werden, um das Volumen der Gradient Buffer / DNA-Mischung, die erforderlich sind, um eine angemessene Mischungsverhältnis zu erzeugen, ist zu bestimmen:
Gradient Puffer und DNA-Lösung (ml) = (CsCl Stammlösung Dichte - gewünschte Enddichte) x Volumen von CsCl Stammlösung x 1,52
Geben Sie die Lautstärke des CsCl Stammlösung bei 4,80 ml. Die gewünschte Enddichte sollte 1,725 g ml -1. Die Stammlösung Dichte wurde in Schritt 1.1 bestimmt.
Beachten Sie auch, dass die relative Volumen von CsCl und Gradient Buffer / DNA wird in einem Gesamtvolumen von mehr als 5,1 ml führen. Vorbereitung Volumes größer als die maximale Lautstärke Kapazität der Ultrazentrifuge Röhren (größer als 5,1 ml) wird dafür sorgen, dass genügend Lösung zum vollständigen Füllen des Rohres. - Mischen durch Invertieren 10-mal. DNA wird bei Raumtemperatur in CsCl stabil.
4. Erstellen einer EtBr Kontrolle Gradient (optional)
Da EtBr ist ein interkalierende Farbstoff-Komplexe mit DNA sichtbar unter UV-Licht machen, sind Kontrolle Gradienten mit EtBr hilfreich, weil sie unmittelbare visuelle Bestätigung Gradientenbildung vor der Fraktionierung von Probenröhrchen (zB siehe Abbildung 1). Die Aufnahme eines Steuerrohr mit EtBr und eine Mischung aus beiden 12 C-DNA und 13 C-DNA (oder 14 N-DNA und 15 N-DNA) ermöglicht die sofortige Visualisierung der Band Formation innerhalb der Rohre nach Abschluss der Ultrazentrifugation. Dies ist wichtig, weil eine gebrochene Rohr während Ultrazentrifugation oder falsch programmiert Laufbedingungen in scheiterte Gradientenbildung führen kann. Gebunden an DNA, senkt EtBr die Dichte der DNA und als Ergebnis ein anderes Protokoll ist, gefolgt von Gradienten vorzubereiten. Beachten Sie, dass andere Nukleinsäure Flecken anstelle von EtBr 11 kann verwendet werden, aber das Protokoll-Optimierung mit anderen Fluorophoren erfordern.
- Die Steuerung Gradienten erfordert zwei Bänden von genomischer DNA: ein vollständig mit stabilen Isotopen markiert und einmal ohne Label. Wir verwenden in der Regel entweder Sinorhizobium meliloti in Medien mit 13 C-bzw. 12 C-Glukose als einzige Kohlenstoffquelle oder Methylococcus capsulatus Stamm Bath in Gegenwart von 13 C-bzw. 12 C-Methan als unsere Kontrollen gezüchtet, kultiviert.
- Kombinieren Sie einen 5 -10 pg Menge sowohl der 12 C-DNA und 13 C-DNA mit Gradient Puffer auf ein Endvolumen von 1,00 ml in eine Einweg-15-ml mit Schraubverschluss Röhre.
- Add 1,00 g festem CsCl, um das gleiche Rohr. Durch Umdrehen mischen.
- Add 110 uleiner 10 mg ml -1 EtBr-Lösung und 4,3 ml einer 1 g ml -1 CsCl Stammlösung zu den gleichen Schraubverschluss Rohr in Schritt 4.2 verwendet. Die endgültige Dichte der Lösung wird etwa, dass der ursprüngliche CsCl Stammlösung.
- Eine zusätzliche "leere" Control-Lösung mit EtBr wird auch erforderlich, um ein Gegengewicht die Lösung in Schritt 4.4 erstellt werden. Kombinieren Sie 1,00 mL der Gradient Buffer, 1,00 g CsCl, 110 ul einer 10 mg ml -1 EtBr-Lösung und 4,3 ml einer 1 g ml -1 CsCl-Stammlösung in einem separaten 15 ml mit Schraubverschluss Röhrchen und mischen durch Umdrehen.
5. Ultrazentrifugation
- Mit einer Glühbirne und Pasteurpipette sorgfältig ausfüllen Ultrazentrifuge Rohre mit Gefälle Lösungen in Schritt 3.2 (oder Schritte 4.4 Falls Vorbereitung einer EtBr Kontrolle Gradient) vorbereitet. Vorsichtig die Lösungen für die Rohre mit einer Pasteurpipette. Beschriften Sie die Röhrchen auf der Tubenschulter mit einem feinen Filzstift. VORSICHT: Sicherstellen, dass die Rohre exakt auf den Boden des Rohres Hals gefüllt. Unzureichend gefüllte Röhrchen sind wahrscheinlich während Ultrazentrifugation platzen.
- Wenn alle erforderlichen Rohre mit Musterlösungen gefüllt sind, notieren Sie die genauen Masse jedes Rohr. Paar Rohre und Ausgeglichenheit sie auf, binnen 0-10 mg. Zum Auswuchten, finden fast paarweise und hinzuzufügen oder zu entfernen winzige Mengen der Lösung, bis sie ausgeglichen sind, wobei die Lösung auf möglichst nahe an den Boden des Rohres Hälse wie möglich. Beachten Sie, dass für Wägegefäße wir ein umgekehrtes 15-ml mit Schraubverschluss Röhre, in der Hälfte wurde als Rohr Halterung für das Gleichgewicht geschnitten verwenden.
- Verschließen Sie die Röhrchen mit einer "Röhre Topper" nach den Anweisungen des Herstellers.
- Prüfen Sie, ob die Rohre richtig durch Invertieren sie versiegelt und mit mäßigem Druck. Wiegen Sie die Rohre wieder zu überprüfen, ob sie noch nach der Versiegelung innerhalb 0-10 mg ausgewogen.
- Prüfen Sie die einzelnen Rotor auch sorgfältig, um sicherzustellen, dass der Brunnen sauber und frei von Schmutz oder Staub, die vielleicht Punktion der Rohre während der Ultrazentrifugation.
- Legen Sie die Rohre in den Rotor mit der ausgewogenen Paare einander gegenüber. Notieren Sie den Rotor Lage von jeder Probe, weil die Ultrazentrifugation Prozess kann dazu führen, Marker Etiketten beschädigt oder gelöscht werden. Sorgfältig verschließen Rotor Brunnen, wie vom Hersteller angegeben.
- Legen Sie den Rotor in der Ultrazentrifuge. Schließen Sie die Ultrazentrifuge Tür und ein Vakuum. Bei Verwendung eines Vti 65,2 Rotor, stellen Sie die Drehzahl auf 44.100 Umdrehungen pro Minute (~ 177.000 xg av), die Temperatur bei 20 ° C, und Ultrazentrifugation Zeit für 36-40 Stunden. Wählen Sie Vakuum, maximale Beschleunigung, und schalten Sie die Bremse (sorgt Gradienten nicht durch Verzögerung unterbrochen). Beachten Sie, dass das Ausschalten der Bremse wird eine zusätzliche 1-2 Stunden, um die Laufzeit hinzufügen. Beachten Sie auch, dass kürzere Laufzeiten kann keine ausreichende Band-Auflösung. Lange Ultrazentrifugation läuft empfohlen, da sie eine höhere Auflösung von unterschiedlichen Nukleinsäure-Bands führen.
- Unmittelbar nach Abschluss der Ultrazentrifugation Verfahren, entfernen Sie den Rotor vorsichtig. Vermeidung jeglicher Kippen oder Anstoßen des Rotors, entfernen Rohre aus dem Rotor zu verhindern, daß das Gefälle innerhalb der Rohre. In seltenen Fällen wird eine Röhre während des Laufs platzen. Wenn ja, gibt es eine Chance, dass die Gradienten in den anderen Röhren nicht richtig bilden. Wenn ein Steuerelement Gradienten aufgenommen wurde, überprüfen Sie dieses Rohr sorgfältig unter UV-Licht zu Gradientenbildung bestätigen. Wenn das Gefälle nicht richtig in das Steuerrohr gebildet wird, ist es am besten, alle Schritt 5 wiederholen. Beachten Sie, dass die EtBr Steuerrohr und seine Blindkontrolle in der Dunkelheit können gespeichert und wiederverwendet werden bis zu sechs Monaten. Achten Sie darauf, den Rotor sauber nach den Anweisungen des Herstellers nach dem Platzen Rohr entfernt worden ist. Verwenden Sie keine Metall-Bürsten oder Scheuermittel zum Rotor Brunnen zu reinigen, um Kratzer auf den Rotor Brunnen zu vermeiden! Rotor-spezifischen Bürsten und Reinigungsmittel können von Beckman erworben werden.
6. Gradienten Fraktionierung
Es gibt zwei Methoden, die derzeit verwendet werden, um DNA aus der Ultrazentrifuge Röhren wieder her: Fraktionierung und Nadel-Extraktion. Dieses Protokoll wird nur beschreiben den Prozess der Gewinnung von DNA mit Hilfe der Fraktionierung Technik. Dies liegt daran, für die meisten SIP Experimenten markierter DNA nicht sichtbar gemacht werden können mit EtBr und muss stattdessen durch einen Vergleich entspricht leichte und schwere Fraktionen aus mehreren Probenröhrchen erkannt werden. Eine Spritzenpumpe wird dringend empfohlen, gleich Dichtegradienten-Fraktionen aus Ultrazentrifuge Rohre abzurufen. Wir arbeiten mit einem BSP-Modell Infusionspumpe (Braintree Scientific Inc.). Ein Low-Flow-Schlauchpumpe oder einer HPLC-Pumpe kann ebenfalls verwendet werden.
- Füllen Sie eine sterile 60-ml-Spritze mit sterilem ddH 2 O enthält, ausreichend Bromphenolblau Farbstoff eine dunkelblaue Farbe geben. Legen Sie die Spritze auf dem Ladearm der Spritzenpumpe.Attach Pumpe-Schlauch mit einem 23-Spur 1 "die Nadel an und schalten Sie die Pumpe, bis einige ddH 2 O bis zum Ende der Nadel. Beachten Sie, dass eventuell vorhandene Luftblasen in diesem ddH 2 O Versorgung wird sich negativ auf die Zerlegung gekommen ist eingebaut.
- Fix eines der Ultrazentrifugation Rohre eine Klammer stehen. Stellen Sie sicher, dass die Klemme fest genug ist, um den Schlauch vom verdrängt zu verhindern, aber nicht so, dass Druck auf die Tube wäre eine Freigabe der CsCl-Lösung, wenn das Rohr durchbohrt verursachen. Pierce ganz unten das Rohr entlang der Röhre Naht mit einem frischen 23 Gauge 1 "Nadel. Für die besten Ergebnisse, durchbohren die Röhre, in einer kontrollierten, schnellen und sicheren Auftreten. Diese sehr schwierig, gut zu machen ist, der Praxis mehrere Male, bevor Diese wird zunächst mit Probenröhrchen versucht.
- Für jede Probe vorbereiten 12 sterile 1,5 ml Reaktionsgefäße, deren Kennzeichnung der Probe Anzahl und Anteil (1-12; schwer zu leicht). Mit der Nadel, um die Pumpe-Schlauch (Schritt 6,1), durchbohren die Spitze der Tube auf das obere Rohr Schulter, entlang der Naht. Sammeln Sie die Gradienten-Lösung mit dem Mikrozentrifugenröhrchen. Wie für den unteren Teil der Röhre, durchbohren die Röhre in eine schnelle und kontrollierte Art und Weise durchgeführt. Üben vorher und sehr vorsichtig sein, um eine kontrollierte Zugbewegung verwenden, um die erzwungene Nadel aus, die durch die Röhre und in einen Finger zu verhindern! Verwenden eines zuvor kalibrierten Pumprate, dass 12 x 425 ul Fraktionen in 12 Minuten (425 ul min -1) erbringt.
- Verwenden Sie ein digitales Refraktometer (z. B. Reichert AR200; empfohlen) oder eine analytische Waage, um die Dichte der Fraktionen von einem Gefälle zu überprüfen, um eine ordnungsgemäße Gradientenbildung bestätigen. Sie müssen ~ 50 ul Probe für den Test verwenden. Wir haben oft gehört Reinkultur DNA in einem Röhrchen (wie für die Vorbereitung der EtBr Kontrolle Gradienten beschrieben), um als Kontrolle für die Fraktionierung dienen und nutzen diese zur Dichtebestimmung. Erwarten Sie das Dichten von ~ 1,690-1,760 g ml -1 Bereich, mit einer mittleren Dichte von ~ 1,725 g ml -1.
7. DNA Niederschlag
- Niederschlag DNA aus allen Fraktionen, indem zuerst 20 ug lineares Polyacrylamid als Träger für die Fällung. Durch Umdrehen mischen. Add 2 Volumen PEG-Lösung (siehe Schritt 1) und mischen durch Umdrehen. Beachten Sie, dass ein Träger für die Fällung (z. B. Glykogen oder linearem Polyacrylamid) von entscheidender Bedeutung für quantitative Rückgewinnung von DNA aus Gradienten Fraktionen, aber Vorsicht ist angebracht, wenn Glykogen als Träger für die Fällung für dieses Protokoll verwendet wird. Glykogen Vorbereitungen haben gezeigt, dass mit bakteriellen Nukleinsäuren kontaminiert sein und Verunreinigungen können leicht verwirren die Interpretation der SIP Gradientenfraktionen 12.
- Lassen Sie die Röhrchen bei Raumtemperatur für 2 Stunden, um die DNA zu fällen können. Falls gewünscht, können Rohre über Nacht bei Raumtemperatur stehen gelassen werden.
- Zentrifuge bei 13.000 g für 30 Minuten mit der Rückseite der Röhren nach außen für eine konsequente Orientierung Rohr in den Rotor. Vorsichtig absaugen und den Überstand verwerfen. Ein Pellet sichtbar sein soll, kann aber sehr schwierig, in diesem Stadium zu sehen. Arbeiten unter eine helle Lichtquelle (zB Schreibtischlampe) bis in die Visualisierung der Pellets zu unterstützen.
- Waschen des Pellets mit 500 ul 70% Ethanol. Zentrifuge bei 13.000 g für 10 Minuten. Vorsichtig absaugen und den Überstand verwerfen. Das Pellet wird in der Regel besser sichtbar für diesen Schritt, wird aber von der Rohrwand leichter distanzieren.
- Lassen Sie das Pellet bei Raumtemperatur für 15 Minuten trocknen lassen.
- Suspend jedes Pellet in 50 ul TE Puffer (siehe Schritt 1.5). Run 5 ul jeder Fraktion auf einem Agarosegel nach Standard Labor-Protokolle.
8. Fraction Charakterisierung
Die Methode zur Gradientenfraktionen charakterisieren den Erfolg eines SIP Inkubation Beurteilung wird je nach Labor und Verfügbarkeit der Ausrüstung. Mit einem Fingerprint-Methode für die Ausrichtung der 16S rRNA-Gens ist ein gemeinsames Vorgehen und Methoden wie Terminal Restriktionsfragmentlängenpolymorphismus (T-RFLP) oder denaturierende Gradienten-Gelelektrophorese (DGGE) angemessen sind (Abbildung 1). Nach dem oben beschriebenen Protokoll, erwarten, dass die Licht-DNA mit den Fraktionen 9-11 (~ 1,705-1,720 g ml -1) und die schweren DNA-Fingerprints in Verbindung gebracht werden, um innerhalb von Bruchteilen 5-8 (~ 1,720-1,735 g ml -1 in Verbindung gebracht werden ). Einzigartige Fingerabdrücke mit den Fraktionen 5-8 von stabilen Isotopen inkubierten Proben assoziiert, aber nicht mit native-Substrat inkubiert steuert bietet starke Beweise, die bestimmte Organismen mit dem Stoffwechsel von besonderer markiertem Substrat. Wenn nicht genügend markierter DNA bleibt für einige Anwendungen (Hybridisierung, Metagenomik), kann Multiple Displacement Amplification verwendet werden, um zu produzieren größere Mengen von 13 bis 15 werden, aber dies kann Chimären in die amplifizierte DNA 14,16 einzuführen.
9. Ergebnisse
Typische DNA-SIP Ergebnisse werden zeigen, eine Trennung von markierten und unmarkierten DNA in den Gradienten durch Ultrazentrifugation gebildet. Im Idealfall wird eine vollständige Rückbildung der hochmolekularen genetisches Material (z. B. 13 C, 15 N) von unmarkierten Materialien erreicht werden. Auflösung kann visuell durch Beobachtung Band Formation in EtBr Röhren beobachtet werden kann. Die Konzentrationen der abgerufenen genomischer DNA in den einzelnen Gradientenfraktionen enthalten sind, können auch verwendet werden, um eine ordnungsgemäße Gradientenbildung bestätigen.
Aus diesem Protokoll sind wir repräsentative Ergebnisse der Ultrazentrifugation erfolgt mit Nukleinsäure aus zwei reinen Kulturen (Abbildung 2). Die fraktionierten Gradienten, die hier wurde unter Verwendung von genomischer DNA aus S. extrahiert meliloti (ATCC 1021) und 13 C-markierten M. capsulatus str. Bath. Nach Ultrazentrifugation, Fraktionierung und DNA-Gewinnung, markierten und unmarkierten genomischen DNA getrennt in die jeweiligen Gradienten-Fraktionen mit unterschiedlicher Dichte (Abbildung 2A). Heavy-Isotop markierter DNA kann in Fraktionen 4-5 beobachtet werden, während unmarkierte DNA in hohen Konzentrationen in den Fraktionen 9-10 zu finden ist. Die DNA aus jeder Fraktion wurde mit denaturierende Gradienten-Gelelektrophorese 17 und der PCR-amplifizierten Produkte erzeugt diskrete Bandenmuster entsprechend den beiden Organismen in der Steigung (Abbildung 2B) enthalten charakterisiert. Die Dichte der Fraktionen reichten von ~ 1,580 bis 1,759 g ml -1, und sie sind in der Reihenfolge abnehmender Dichte von links nach rechts dargestellt.
Obwohl die Trennung von reinem 13 C-und 12 C-DNA kann gesprochen (Abbildung 2), kann die Umwelt Probe Inkubationen werden schwieriger zu interpretieren. Zum Beispiel, inkubierten wir Tundra Böden von Resolute Bay (Nunavut, Kanada) mit 12 C-bzw. 13 C-markierter Glukose für eine 14-Tage-Zeitraum bei 15 ° C. Die Agarosegele von gereinigtem Gradienten Bruchteil DNA nachgewiesen, dass genomische DNA wurde "verschmiert" über Fraktionen 7-10 für beide 12 C-und 13 C-Inkubationen (Abbildung 3A und 3C, jeweils). In diesem Fall können 13 C-Anreicherung von Biomasse aus bestimmten mikrobiellen Taxa nur mit einem Ansatz, wie DGGE von 16S rRNA-Gene ermittelt werden. Die 12 C-Glucose inkubiert Boden-DNA erzeugt ähnliche Muster in allen Gradientenfraktionen (3B), aber die 13 C-Glucose inkubiert Probe erzeugten DGGE Fingerabdrücke eindeutig mit den Fraktionen 5-8 (Abbildung 3D) assoziiert sind. Von besonderem Interesse sind die konservierten Bands durch die Pfeile angedeutet. Das dominante "phylotype" ist in allen Gradientenfraktionen aber verschiebt sich schwereren Fraktionen für die DNA von 13 C-Glucose inkubiert Boden gewonnen. Nachfolgende DNA-Sequenzierung dieser Band und / oder Klon-Bibliothek Analyse würde bestätigen die Identität von diesem besonderen 16S rRNA-Gens und anschließende Führung metagenomische oder Anbau-basierten Ansätzen.
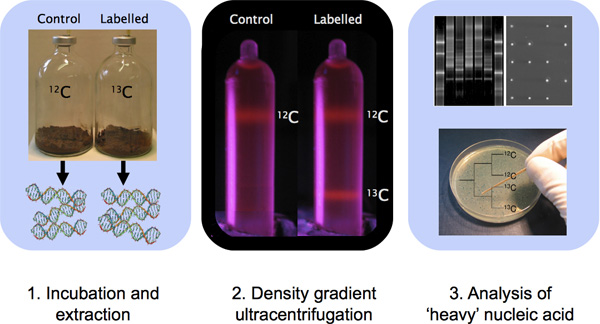
Abbildung 1. Eines DNA-SIP Experiment mit Probeninkubation, DNA-Extraktion, CsCl Dichtegradienten-Ultrazentrifugation und DNA Charakterisierung mit molekularen Techniken Gliederung.
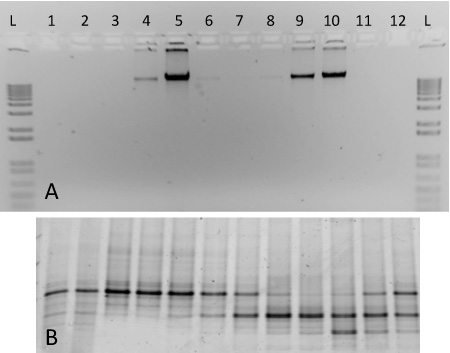
Abbildung 2. Erwartete Ergebnisse für eine SIP-Gradienten Fraktionierung einschließlich DNA von zwei reinen Kulturen. (A) Aliquots von DNA aus Gradientenfraktionen 1-12 wurden auf einem 1% Agarosegel aus einem Verlauf mit 13 C-markierten M. laufen capsulatus Stamm Bath (Fraktionen 4-6) und 12 C-markierten S. meliloti (Fraktionen 8-10). A 1-kb Leiter ist für den Vergleich einbezogen (B) PCR-amplifizierte DNA aus dem gleichen Fraktionen wurden auf einem 10% DGGE-Gel laufen gelassen. Fingerprint-Muster zeigen deutliche Unterschiede zwischen den Fraktionen 5 und 9, zum Beispiel.
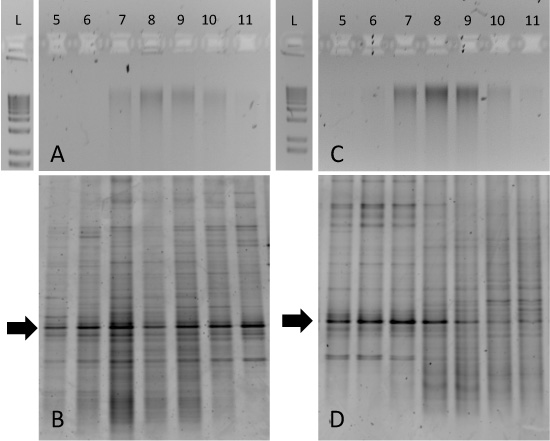
Abbildung 3. Erwartete Ergebnisse für die SIP-Gradienten Fraktionierungen aus Bodenprobe Inkubationen. Aliquots von Gradienten-Fraktionen aus beiden 12 C-Glucose geändert Boden (A) und 13 C-Glucose geändert Boden (C) wurden auf 1% Agarosegelen laufen und eine 1-kb Leiter ist für den Vergleich einbezogen. Entsprechende DGGE Fingerabdrücke für jede dieser Proben sind in (B) und (D). Fingerprinting von Fraktionen enthüllt Anreicherung bestimmter bakterieller Taxa in den 13 C-Glucose geändert Probe in Fraktionen 5-8 (D).
Diskussion
Eine sachgerechte Konstruktion der stabilen Isotopen Experimente ist von entscheidender Bedeutung für den Erhalt von markierter DNA vor dem Hintergrund unbeschrifteten Gemeinschaft. Überlegungen im Zusammenhang mit Inkubationszeiten Substratkonzentrationen, Inkubationsbedingungen (z. B. Nährstoffe, Feuchtigkeitsgehalt des Bodens), Cross-Fütterung und Replikation Probe wurden an anderer Stelle 10,18 und diskutiert, empfehlen wir dem Leser wenden diese Veröffentlichungen bei der Gestaltung eines SIP Inkuba...
Offenlegungen
Danksagungen
Diese Arbeit wurde von Strategic Project and Discovery Zuschüsse an JDN aus dem Natural Sciences and Engineering Research Council of Canada (NSERC) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments | |
| Bromophenol Blue | Reagent | Fisher Scientific | BP115-25 | |
| Cesium chloride | Reagent | Fisher Scientific | BP210-500 | |
| Ethanol, reagent grade | Reagent | Sigma-Aldrich | 652261 | |
| Ethidium bromide | Reagent | Sigma-Aldrich | E1510 | |
| Hydrochloric acid | Reagent | Fisher Scientific | 351285212 | |
| Linear polyacrylamide | Reagent | Applichem | A6587 | |
| Polyethylene Glycol 6000 | Reagent | VWR international | CAPX1286L-4 | |
| Potassium Chloride | Reagent | Fisher Scientific | AC42409-0010 | |
| Sodium Chloride | Reagent | Fisher Scientific | S2711 | |
| Sodium Hydroxide pellets | Reagent | Fisher Scientific | S3181 | |
| Tris base | Reagent | Fisher Scientific | BP1521 | |
| Dark Reader | Equipment | Clare Chemical | DR46B | |
| Microcentrifuge | Equipment | Eppendorf | 5424 000.410 | |
| Nanodrop 2000 | Equipment | Fisher Scientific | 361013650 | |
| Infusion pump | Equipment | Braintree Scientific, Inc. | N/A | Model Number: BSP See www.braintreesci.com for ordering details. |
| Tube sealer | Equipment | Beckman Coulter Inc. | 358312 | |
| Ultracentrifuge | Equipment | Beckman Coulter Inc. | ||
| Ultracentrifuge rotor | Equipment | Beckman Coulter Inc. | 362754 | |
| Ultraviolet light source | Equipment | UVP Inc. | 95-0017-09 | Any UV source will suffice |
| Ultraviolet light face shield | Equipment | Fisher Scientific | 114051C | |
| Butyl rubber stoppers, gray | Material | Sigma-Aldrich | 27232 | |
| Centrifuge tubes | Material | Beckman Coulter Inc. | 342412 | |
| Hypodermic needle, 23 gauge, 2” length | Material | BD Biosciences | 305145 | |
| Microfuge tubes, 1.5 mL | Material | DiaMed | AD151-N500 | |
| Open center seals, 20 mm diameter | Material | Sigma-Aldrich | 27230-U | |
| Pasteur pipettes, glass | Material | Fisher Scientific | 13-678-6C | |
| Pipet tips | Material | DiaMed | BPS340-1000 | Catalogue number is for 200 μl tips. 10 or 20 μl tips may be purchased from the same source |
| Pump tubing 1.5 mm bore x 1.5 mm wall | Material | Appleton Woods | ||
| Screw-cap tubes, 15 mL | Material | DiaMed | AD15MLP-S | |
| Serum vials, 125 mL volume | Material | Sigma-Aldrich | Z114014 | |
| Syringe, 60 mL | Material | BD Biosciences | 309653 |
Referenzen
- Neufeld, J. D. DNA stable-isotope probing. Nat. Protocols. 2, 860-866 (2007).
- Neufeld, J. D., Boden, R., Moussard, H., Schäfer, H., Murrell, J. C. Substrate-specific clades of active marine methylotrophs associated with a phytoplankton bloom in a temperate coastal environment. Appl. Environ. Microbiol. 74, 7321-7328 (2009).
- Nercessian, O., Noyes, E., Kalyuzhnaya, M. G., Lidstrom, M. E., Chistoserdova, L. Bacterial populations active in metabolism of C1 compounds in the sediment of Lake Washington, a freshwater lake. Appl. Environ. Microbiol. 71, 6885-6899 (2005).
- Padmanabhan, P. Respiration of 13C-labelled substrates added to soil in the field and subsequent 16S rRNA gene analysis of 13C-labelled soil DNA. Appl. Environ. Microbiol. 69, 1614-1622 (2003).
- Bernard, L. Dynamics and identification of soil microbial populations actively assimilating carbon from 13C-labelled wheat residue as estimated by DNA- and RNA-SIP techniques. Environ. Microbiol. 9, 752-764 (2007).
- Haichar, e. l. Z. a. h. a. r., F, . Identification of cellulolytic bacteria in soil by stable isotope probing. Environ. Microbiol. 9, 625-634 (2007).
- Addison, S., McDonald, I., Lloyd-Jones, G. Stable isotope probing: Technical considerations when resolving 15N-labelled RNA in gradients. J. Microbiol. Meth. 80, 70-75 (2009).
- Buckley, D. H., Huangyutitham, V., Hsu, S. -. F., Nelson, T. A. Stable isotope probing with 15N achieved by disentangling the effects of genome G + C content and isotope enrichment on DNA density. Appl. Environ. Microbiol. 73, 3189-3195 (2007).
- Schwartz, E. Characterization of growing microorganisms in soil by stable isotope probing with H218O. Appl. Environ. Microbiol. 73, 2541-2546 (2007).
- Neufeld, J. D., Dumont, M. G., Vohra, J., Murrell, J. C. Methodological considerations for the use of stable isotope probing in microbial ecology. Microb. Ecol. 53, 435-442 (2007).
- Martineau, C., Whyte, L., Greer, C. Development of a SYBR safe technique for the sensitive detection of DNA in cesium chloride density gradients for stable isotope probing assays. J. Microbiol. Meth. 73, 199-202 (2008).
- Bartram, A. K., Poon, C., Neufeld, J. D. Nucleic acid contamination of glycogen used in nucleic acid precipitation and assessment of linear polyacrylamide as an alternative co-precipitant. Biotechniques. 47, 1019-1022 (2009).
- Chen, Y. Revealing the uncultivated majority: combining DNA stable-isotope probing, multiple displacement amplification and metagenomic analyses of uncultivated Methylocystis in acidic peatlands. Environ. Microbiol. 10, 2609-2622 (2008).
- Neufeld, J. D., Chen, Y., Dumont, M. G., Murrell, J. C. Marine methylotrophs revealed by stable-isotope probing, multiple displacement amplification and metagenomics. Environ. Microbiol. 10, 1526-1535 (2008).
- Kalyuzhnaya, M. High-resolution metagenomics targets specific functional types in complex microbial communities. Nat. Biotechnol. 26, 1029-1034 (2008).
- Binga, E. K., Lasken, R. S., Neufeld, J. D. Something from (almost) nothing: the impact of multiple displacement amplification on microbial ecology. ISME J. 2, 233-241 (2008).
- Green, S. J., Leigh, M. B., Neufeld, J. D., Timmis, K. N. . Microbiology of Hydrocarbon and Lipid Microbiology. , 4137-4158 (2010).
- Neufeld, J. D., Wagner, M., Murrell, J. C. Who eats what, where and when? Isotope-labelling experiments are coming of age. ISME J. 1, 103-110 (2007).
- Gallagher, E., McGuinness, L., Phelps, C., Young, L. Y., Kerkhof, L. J. DNA shortens the incubation time needed to detect benzoate-utilizing denitrifying bacteria by stable-isotope probing. Appl. Environ. Microbiol. 71, 5192-5196 .
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten