Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Doppel-Fluoreszenz- In situ Hybridisierung in Fresh Hirnschnitten
In diesem Artikel
Zusammenfassung
Dieses Protokoll umfasst eine nicht-radioaktive In-situ Hybridisierungsverfahren, dass die gleichzeitige Identifizierung von zwei Transkript Spezies ermöglicht, bei einer einzigen Zelle Auflösung, in dünne Schnitte von Gehirn der Wirbeltiere.
Zusammenfassung
Hier beschreiben wir eine modifizierte Version des Doppel-Fluoreszenz-
Protokoll
Dieses Protokoll wurde entwickelt und basiert auf Standard-radioaktive und nicht-radioaktiven in-situ-Hybridisierung Methoden, die zuvor von uns und anderen entwickelt wurde, um ein oder zwei Transkript Arten in Hirngewebe 1-7 erkennen verfeinert. Die unten beschriebenen Protokoll hat eine Gesamtlänge von 2 oder 3 Tage, je nach der Anzahl der prozeduralen Unterbrechungen durch den Endbenutzer gewählt. Alle Schritte nachfolgend beschrieben werden, um bei Raumtemperatur durchgeführt werden, mit Ausnahme der Ribosonde Hybridisierung und post-Hybridisierung wäscht. Die Lösungen und Puffer für alle Schritte in diesem Verfahren Beteiligten erforderlich finden Sie am Ende dieses Protokoll gefunden werden.
1. Tissue Vorbereitung und Schnitte
- Enthaupten und extrahieren Thema Gehirn schnell ab, und legen Sie sie in eine Kunststoff-Formenbau von ausreichender Größe.
- Cover Gehirn mit Tissue-Tek Einbettmedium und schnell statt der Kunststoff-Form in einem dry-ice/alcohol Bad für schnelles Einfrieren. Gefrorenes Gewebe kann bei -80 ° C bis zur Verwendung gelagert werden.
- Mit einem Kryostaten, sammeln 2 oder 3 Hirnschnitten pro Folie auf Rechnung Superfrost Plus Objektträger. Die Dicke der Abschnitte sollten 10-12 um betragen. Dias können bei -80 ° C bis zur Verwendung gelagert werden.
2. Vorbereitung der Sephadex G50 Säulen für Probe Purification
"Sephadex-Säulen aus kommerziellen Quellen erworben werden kann, jedoch bieten wir Ihnen nachfolgend eine kostengünstige Alternative für die Erzeugung von Spalten, die für die Sonde Reinigung benötigt werden.
- Hydrate ausreichende Menge an Sephadex G50-Pulver mit RNase-freiem, DEPC-behandeltem Wasser (z. B. 2 g Pulver in 100 ml DEPC-behandeltem Wasser), Mix-Lösung kurz und bei Raumtemperatur lagern, um Niederschlag über Sephadex G50.
- Überstand entfernen (obere Schicht des Wassers) nach Sephadex G50 Niederschlag.
- Wiederholen Sie den Vorgang über 3 bis 5 mal.
- Nach dem letzten Waschen, wieder aussetzen Sephadex G50-Lösung in TE-Puffer (1:1), und lagern bei 4 ° C bis zur Verwendung.
- Legen Sie autoklaviert Glaswolle in eine sterile 1 ml-Spritze und komprimiert sie mit dem Stößel zu einer kompakten Schicht an der Unterseite machen, und dann legen Sie die Spritze in eine 15 ml Falcon-Röhrchen.
- Gut mischen die Lösung von Sephadex G50 in TE. Füllen Sie die Spritze / Spalte mit dieser Lösung.
- Zentrifugieren Sie die Spalte für 30 sec bei 1000 Umdrehungen pro Minute.
- Wiederholen Sie diesen Vorgang, bis die Spalte ist fast vollständig mit Sephadex G50 Perlen gefüllt.
- Apply 200 pl Säule Waschpuffer auf die Säule und zentrifugieren Sie für 2 min bei 1000 Umdrehungen pro Minute. Durchfluss verwerfen.
- Apply 200 pl Säule Blocking-Puffer in die Säule und spinnen sie für 2 min bei 1000 Umdrehungen pro Minute. Wiederholen Sie diesen Schritt 4-5 mal in die Spalte Gleichgewicht zu bringen. Spalten können bei 4 ° C gelagert werden, bis zu verwenden, wenn mit Parafilm abgedichtet.
3. Beschriftung und Reinigung von Ribosonden
Im Folgenden ausführlich die Erzeugung und Reinigung eines einzelnen Ribosonde. Für dFISH, wird die Vorbereitung der einzelnen Sonde beinhalten die gleiche Methode, außer dass einer der Sonden mit Digoxigenin (DIG)-markierten UTP, während die anderen werden mit Biotin-markierten UTP markiert.
- Bereiten Sie eine konzentrierte (> 150 ng / ul) und gereinigtes linearisierten cDNA-Lösung von Interesse, um entweder (Kontrolle) oder antisense Ribosonden generieren.
- In einem 1,5 ml Mikrozentrifugenröhrchen, fügen 0,5-1 pg gereinigte cDNA-Template, 2 ul 5X Sondenmarkierung Puffer, 1 ul 10x DIG (oder Biotin)-Labeling Mix, 0,5 ul RNasin und 1 ul der entsprechenden RNA-Polymerase und bringt die endgültige Volumen der Lösung auf 10 ul mit RNase-freiem (DEPC-behandelt) Wasser.
- Inkubieren Sie die Lösung in einem Wasserbad bei 37 ° C für 2 Stunden.
- Add 1 ul tRNA (Lager 20 ug / ul) und 39 ul der Spalte Blocking-Puffer in der Lösung.
- Bereiten Sie die Sephadex G50-Säule für die Sonde Reinigung. Zu diesem Zweck werden 50 ul Blocking-Puffer in die Säule und drehen Sie es für 10 sec bei 1000 Umdrehungen pro Minute.
Wiederholen Sie diesen Schritt 2-3 mal auf die Säule (also die vollen 50 ul auf die Säule sind nach Zyklus der Zentrifugation gewonnen) ausgleichen. - Übernehmen Sie die Sonde Lösung der Sephadex G50-Säule, Position ein neues Mikrozentrifugenröhrchen am unteren Rand der Säule, und drehen Sie es für 3 min bei 1000 rpm, um das gereinigte 50 ul Ribosonde Lösung zu erhalten.
- Beurteilen Sie die Qualität und Ausbeute der markierten Ribosonde entweder mit einem Standard-Formaldehyd-Agarose-RNA-Gel oder ein Spektralphotometer.
4. Post-Fixierung, Acetylierung und Hybridisierung
- Entfernen Sie Ausschnitte aus dem -80 ° C Gefrierschrank und ermöglichen eine Raumtemperatur ins Gleichgewicht. Anschließend inkubieren Abschnitte in einem kalten, frisch zubereitete 3% Paraformaldehyd-Lösung für 5 min.
- Kurz spülen Abschnitte in phosphate Kochsalzlösung (PBS) zweimal.
- Entwässern Schnitte durch ein Standard-Alkohol-Reihe (70, 95 und 100%; je 2 min), und lassen Sie sie an der Luft trocknen.
- Inkubieren Abschnitte in einer Acetylierung Lösung für 10 min.
- Spülen Sie den Abschnitten 3 mal in 2X SSPE.
- Entwässern Abschnitte noch einmal durch die Standard-Alkohol-Reihe oben beschrieben, und es ihnen an der Luft trocknen.
- Bereiten Sie ein angemessenes Volumen der Hybridisierungslösung und fügen Sie beide Ribosonden die Lösung (Sonde Konzentration: 1 ng / ul für jede Sonde). Das Gesamtvolumen der Hybridisierungslösung wird auf der Grundlage der Anzahl der Abschnitte, um hybridisiert (16 ul Hybridisierungslösung pro Abschnitt) bestimmt.
- Bewerben ausreichendes Volumen der Hybridisierungslösung, um Gewebe und Deckglas gleitet sicher, dass keine Luftblasen Overlay das Gewebe.
- Die Objektträger in einem Metall Diahalter. Tauchen Halter in ein Mineral Ölbad bei 65 ° C über Nacht eingestellt. Stellen Sie sicher, dass Deckgläser sind nach oben gerichtet (dh der lateralen Seite des Diahalter sollten in Kontakt mit dem Boden des Mineralöl-Container werden).
5. Post-Hybridisierung Wäscht
- Am folgenden Tag, entfernen Sie vorsichtig den Diahalter aus der Mineralöl-Bad und kurz ausspülen in Chloroform, um überschüssiges Öl von den Folien zu entfernen.
- Die Objektträger in einem 2X SSPE-Lösung für 5-10 min. Deckgläser sollten von Dias, während in Lösung zu lösen.
- Die Objektträger in eine neue 2X SSPE-Lösung, und halten Sie sie für 1 Stunde bei Raumtemperatur.
- Transfer-Abschnitte in einer Lösung mit 2X SSPE plus 50% Formamid. Die Temperatur dieser Lösung sollte mit der Temperatur in der Nacht Hybridisierung Verfahren verwendet. Halten Sie Folien in dieser Lösung für 1,5 Stunden.
- Transfer-Abschnitte, um eine 0,1 × SSPE-Lösung auf die gleiche Temperatur der Hybridisierung vorgewärmt. Inkubieren Abschnitte in dieser Lösung für 30 min. Wiederholen Sie diesen Schritt für weitere 30 min.
6. Erkennung und Visualisierung von Ribosonden
- Transfer-Folien TNT-Puffer, dem 0,3% Wasserstoffperoxid wurde hinzugefügt, für 10 min.
- Wash Abschnitte in TNT-Puffer, 3-mal (10 Min.).
- Mit einem DAKO Pen zeichnen ein gut in die Umgebung mit den Hirnschnitten. Wichtig ist, ist Vorsicht geboten, um sicherzustellen, dass Teile nicht trocken werden.
- Apply 150 ul TNB-Puffer zu jedem Dia, und inkubieren Abschnitte in dieser Lösung für 30 min in einer feuchten Kammer.
- Entfernen Sie überschüssiges TNB Lösung durch Kippen Dias.
- Apply 150 ul TNB Lösung mit Peroxidase-konjugierten anti-DIG-Antikörper, und speichern Sie gleitet in einer feuchten Kammer für 2 Stunden. Antikörper-Konzentration sollte individuell für jedes Transkript von Interesse vor der Durchführung dFISH bestimmt werden.
- Wash Abschnitte in TNT-Puffer, 3-mal, jeweils 10 Minuten.
- Apply 150 ul Alexa 594-konjugierten Tyramid funktionierende Lösung zu jeder Folie, und speichern sie in einer feuchten Kammer für 1 Stunde. Diese Lösung muss nach den Anweisungen des Herstellers zubereitet werden.
- Wash Abschnitte in TNT-Puffer, 3-mal, jeweils 10 Minuten.
- 30 min - inkubieren Abschnitte in TNT-Puffer, dem 0,3% Wasserstoffperoxid für 10 aufgenommen worden.
- Wash Abschnitte in TNT-Puffer, 3-mal, jeweils 10 Minuten.
- Apply 150 ul TNB-Puffer pro Objektträger, und bewahren Sie sie in einer feuchten Kammer für 30 min.
- Entfernen Sie überschüssiges TNB Lösung durch Kippen Dias.
- Add 150 ul TNB Lösung mit Peroxidase-konjugierten anti-Biotin-Antikörper, und speichern Sie gleitet in einer feuchten Kammer für 2 Stunden.
- Wash Abschnitte in TNT-Puffer, 3-mal, jeweils 10 Minuten.
- Apply 150 pl einer Alexa 488-konjugierten Tyramid funktionierende Lösung, um die Folien und inkubieren Abschnitte für 1 Stunde. Diese Lösung sollte nach den Empfehlungen des Herstellers vorbereitet werden.
- Wash Abschnitte in TNT-Puffer, 3-mal, jeweils 10 Minuten.
- Add 150 ul Hoechst-Lösung (1:1000 in TNT-Puffer) zu den Abschnitten und halten sie in der feuchten Kammer für 2 min.
- Waschen Sie die Abschnitte in TNT-Puffer, 3-mal, jeweils 5 min.
- Deckglas Abschnitte mit einem Fluoreszenz-kompatible Mounting Medium (zB Vectashield oder verlängern Antifade).
7. Repräsentative Ergebnisse
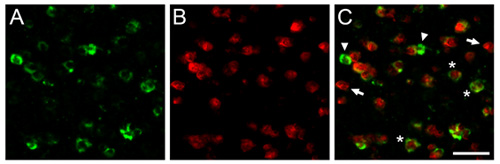
Abbildung 1. Wir zeigen hier Vertreter dFISH Ergebnisse in der Zebrafinken Gehirn erhalten. Gezeigt werden Aufnahmen aus dem caudomedial nidopallium (NCM), der Singvogel Analogon des auditorischen Kortex von Säugetieren erhalten. Gehirn-Schnitte wurden mit einem biotinylierten Ribosonde gegen Parvalbumin (A), ein Marker für eine Subpopulation der hemmenden Neuronen gerichtet hybridisiert und einer DIG-markierten RiboSonde gegen die Aktivitäts-abhängigen Gen Zenk (B), ein zuverlässiger Marker für Song-driven Neuronen gerichtet. C) Überlagerung von (A) und (B) zeigt eine Bevölkerung von hemmenden Neuronen, die durch Hörerlebnis aktiviert werden. Pfeile und Pfeilspitzen zeigen Zellen ausschließlich mit jeder der beiden Ribosonden beschriftet und Sternchen zeigen repräsentative Neuronen co-exprimierenden beide Transkripte von Interesse. Balken = 25 um.
Diskussion
Wir haben dieses Protokoll verwendet, um zu untersuchen, wie Gehirn der Wirbeltiere neurochemisch und funktional organisiert ist, und um festzustellen, wie verhaltensgestörte relevanten sensorischen Reizen Auswirkungen der genomischen Maschinerie der Nervenzellen im erwachsenen Gehirn 8-10. Wir haben erfolgreich diese Methode in Hirngewebe von Mäusen, Ratten und Singvögel, aber erwarten, dass dieses Protokoll wird einfach an Hirnschnitten aus einem Array von Wirbeltierarten und, vielleicht, nicht-neuralen ...
Offenlegungen
Danksagungen
Arbeit unterstützt durch Zuschüsse der NIH / NIDCD und der Schmitt-Stiftung zur RP.
Materialien
Lösungen
- Column Waschpuffer: 10 mM Tris-HCl, 0,15 M NaCl, 0,05 mM EDTA, 50 ug / ul tRNA, 0,1% SDS in 50 ml DEPC-behandeltem Wasser.
- Column Blockierungspuffer: 10 mM Tris-HCl, 50 mM NaCl, 0,1 mM EDTA in 50 ml DEPC-behandeltem Wasser.
- TNT-Puffer: 60 ml von 1 M Tris-HCl, 18 ml 5 M NaCl und 1,8 ml Triton X-100 in 600 ml DEPC-behandeltem Wasser.
- TNB Puffer: 100 mM Tris-HCl, 8,3 ug / ul BSA, 0,15 M NaCl und 3% Triton X-100 in DEPC-behandeltem Wasser.
- TE-Puffer: 10 mM Tris-HCl, pH 7,5 plus 1 mM EDTA, pH 7,5.
- Acetylierung Lösung: 2,7 ml Triethanolamin plus 0,5 ml Acetanhydrid in 200 ml DEPC-behandeltem Wasser.
- Hybridisierungslösung: 50% Formamid, 2X SSPE, 2 ug / ul tRNA, 1 ug / ul BSA und 1 ug / ul poly A in DEPC-behandeltem Wasser. Es braucht 16 l pro Abschnitt.
Referenzen
- Jin, L., Lloyd, R. V. In situ hybridization: methods and applications. J Clin Lab Anal. 11, 2-9 (1997).
- Stoler, M. H. In situ hybridization. Clin Lab Med. 10, 215-236 (1990).
- Komminoth, P., Werner, M. Target and signal amplification: approaches to increase the sensitivity of in situ hybridization. Histochem Cell Biol. 108, 325-3233 (1997).
- Kessler, C. The digoxigenin:anti-digoxigenin (DIG) technology--a survey on the concept and realization of a novel bioanalytical indicator system. Mol Cell Probes. 5, 161-205 (1991).
- Panoskaltsis-Mortari, A., Bucy, R. P. In situ hybridization with digoxigenin-labeled RNA probes: facts and artifacts. Biotechniques. 18, 300-307 (1995).
- Qian, X., Lloyd, R. V. Recent developments in signal amplification methods for in situ hybridization. Diagn Mol Pathol. 12, 1-13 (2003).
- Mello, C. V., Jarvis, E. D., Denisenko, N., Rivas, M. Isolation of song-regulated genes in the brain of songbirds. Methods Mol Biol. 85, 205-217 (1997).
- Pinaud, R. GABAergic neurons participate in the brain's response to birdsong auditory stimulation. Eur J Neurosci. 20, 1318-1330 (2004).
- Velho, T. A., Pinaud, R., Rodrigues, P. V., Mello, C. V. Co-induction of activity-dependent genes in songbirds. Eur J Neurosci. 22, 1667-1678 (2005).
- Tremere, L. A., Jeong, J. K., Pinaud, R. Estradiol shapes auditory processing in the adult brain by regulating inhibitory transmission and plasticity-associated gene expression. J Neurosci. 29, 5949-5963 (2009).
- Pinaud, R., Mello, C. V., Velho, T. A., Wynne, R. D., Tremere, L. A. Detection of two mRNA species at single-cell resolution by double-fluorescence in situ hybridization. Nat Protoc. 3, 1370-1379 (2008).
- Pinaud, R., Jeong, J. K. Duplex fluorescence in situ hybridization in the study of gene co-regulation in the vertebrate brain. Methods Mol Biol. 611, 115-129 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten