Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Die Produktion von C. elegans Transgene via Recombineering mit dem GalK Selektionsmarker
In diesem Artikel
Zusammenfassung
Die Fähigkeit, Transgene für produzieren Caenorhabditis elegans Verwendung von genomischer DNA von Fosmide durchgeführt Besonders attraktiv ist wie alle nativen regulatorischen Elemente beibehalten werden. Beschrieben wird eine einfache und robuste Verfahren zur Herstellung von Transgenen über Rekombination mit dem GalK Selektionsmarker.
Zusammenfassung
Die Erzeugung transgener Tiere ist weit verbreitet in C genutzt elegans Forschung einschließlich der Verwendung von GFP Fusionsproteine auf die Regulation und Expression von Genen von Interesse oder die Erzeugung von Tandem-Affinitätsreinigung (TAP) markierten Versionen bestimmter Gene, um ihre Reinigung zu erleichtern studieren. Typischerweise Transgene werden durch einen Promotor stromaufwärts von einem GFP-Reportergen oder cDNA von Interesse erzeugt, und dies führt oft zu einer repräsentativen Expressionsmuster. Allerdings kritischen Elemente der Genregulation, wie Steuerelemente in der 3 'untranslatierten Region oder alternative Promotoren, konnte durch diesen Ansatz fehlen. Weitere nur eine einzige Spleißvariante können in der Regel auf diese Weise untersucht werden. Im Gegensatz dazu die Verwendung von Wurm genomischer DNA von Fosmid DNA-Klone durchgeführt beinhaltet wahrscheinlich die meisten, wenn nicht alle Elemente in der Genregulation in vivo, die die größere Fähigkeit, die echten Ausdruck Muster und Timing zu erfassen erlaubt beteiligt. Zur Erleichterung der Erzeugung von Transgenen mit Fosmid DNA beschreiben wir ein E. coli basierten Recombineering Verfahren zur GFP, einem TAP-tag oder andere Sequenzen von Interesse in jeder beliebigen Stelle im Gen einzufügen. Das Verfahren nutzt die galK-Gen als Selektionsmarker für die positiven und negativen Selektion Schritte in die Rekombination in den Erhalt der gewünschten Modifikation mit hohem Wirkungsgrad zur Folge. Außerdem sind Plasmide, die die galK Gens durch Homologie Arme auf häufig verwendete GFP flankiert und TAP Fusionsgene zur Verfügung, die Reduzierung der Kosten für Oligos um 50% bei der Erzeugung eines GFP-oder TAP-Fusionsprotein. Diese Plasmide verwenden R6K Replikationsursprung, der die Notwendigkeit einer umfassenden PCR-Produkt Reinigung ausschließt. Schließlich haben wir auch zeigen, eine Technik, um die unc-119-Marker auf die Fosmid Rückgrat der die Fosmid direkt injiziert werden oder bombardiert zu Würmern, um transgene Tiere zu erzeugen erlaubt integrieren. Dieses Video zeigt die Verfahren bei der Erzeugung eines Transgens über Rekombination mit dieser Methode beteiligt.
Protokoll
Überblick
Viele Transgene in der Erzeugung transgener C eingesetzt elegans besteht aus Promotorsequenzen und vielleicht auch ein Gen-cDNA in einer der Vektoren, die durch das Labor von Dr. Andy Fire 1 erzeugt geklont. Während diese Transgene oft erfolgreich mit Bezug auf die Herstellung eines GFP Reportergen oder Ausdruck einer cDNA in einem gewünschten Muster können diese Transgene Mangel der alternativen Promotoren, Enhancer-Elemente, und 3 'untranslatierten Region (UTR) Elemente, die eine wichtige Rolle spielen bei der Kontrolle der Genexpression in vivo 2. Zum Beispiel haben sowohl die daf-12 und fah-1-Gene wichtig Enhancer-Elemente, die außerhalb liegen, der proximalen Promotor, der in Promoter verpasste nur Konstrukte 3,4,5. Weitere zahlreiche transgene Konstrukte mit dem unc-54 3'UTR die Regelung verhindert, dass durch die entsprechende microRNA Gene 6,7,8. Folglich würde Erzeugung Transgene mit großen Teilen der Wurm genomischer DNA als ideal für die Erfassung aller Promotoren, Splice-Varianten und 3 'UTR Bedienelemente. Vor kurzem wurde ein C. elegans Fosmid Bibliothek, die von ~ 40 kb Regionen von genomischer DNA aus und deckt nahezu alle im Genom konstruiert worden. Die Verwendung von Wurm genomischer DNA durchgeführt, indem diese Fosmid DNA-Klone Ergebnisse in der größeren Fähigkeit zum echten Ausdruck Muster und Timing von spezifischen Genen 2,8,9,10,11 erfassen.
Doch die Arbeit mit großen Regionen der genomischen DNA stellt praktische Herausforderungen wie die großen Schwierigkeiten im Umgang mit Standard-Techniken der Molekularbiologie 12. Um diese Einschränkungen zu überwinden, um Techniken Fosmide oder bakterielle künstliche Chromosomen durch homologe Rekombination in E. ändern coli wurden entwickelt und werden als Recombineering 12,13. Recombineering ermöglicht die nahtlose Einfügung von GFP, einem Tandem-Affinitätsreinigung (TAP)-tag oder andere Sequenzen von Interesse in jeder beliebigen Stelle im Gen durch die C elegans Fosmid Klon 2,10,14. Die homologe Rekombination erfolgt zwischen einem PCR-Produkt von 50 bp Homologiebereiche an den Zielort und die Ziel-DNA flankiert in speziell modifizierten E. coli-Stämmen.
Vor kurzem haben wir ein zweistufiges Verfahren zur Modifikation von C beschrieben elegans Fosmide durch Rekombination, die das Einfügen der galK Gen an der gewünschten Stelle und dann wieder an das Gen mit der gewünschten Sequenz 2 beinhaltet. Die galK Gen dient als wirksames Selektionsmarker für beide Schritte in dem Prozess, wie es für und gegen über den Einsatz von selektiven Nährmedium 15 kann ausgewählt werden. In der ersten Stufe der Fosmid Modifikation wird die galK Gen durch homologe Rekombination an der gewünschten Stelle eingefügt und die korrekt geändert Fosmide durch positive Selektion für die Fähigkeit, Galactose als Kohlenstoffquelle nutzen 2,15 identifiziert. In der zweiten Stufe wird das galK Gens durch die gewünschte Sequenz ersetzt, und die korrekt geändert Fosmide werden durch negative Selektion gegen die galK Gen durch die Verwendung des giftigen Galactosederivat Desoxygalactose die galK + Bakterien 2,15 tötet identifiziert. Ein Vorteil der galK ist die Fähigkeit eines einzelnen Gens für die positive und negative Selektion Schritte verwendet werden, anstatt andere Marker, die getrennte Gene für jeden Schritt, und die Ergebnisse sind in den Erhalt der gewünschten Modifikation mit hohem Wirkungsgrad 2,15.
Um die Anwendung dieser Technik zu erleichtern C. elegans Forschung, haben wir einige Änderungen an den verfügbaren Ressourcen. Erstens, die GFP und TAP-Tags werden üblicherweise verwendet, um Wurm Transgene zu generieren, so bauten wir in 50 bp Regionen Homologie zu jedem dieser Tags in den pMOD4 galK-G und pMOD4 galK-GT-Plasmide, die als Quelle der galK Gens dienen 2. Diese Regionen ermöglichen einen einzigen Satz von Oligos für beide Stufen der Fosmid Modifikation, die die Notwendigkeit, einen zweiten Satz etwas teuer Oligos bestellen spart verwendet werden. Zweitens verwenden diese Plasmide der R6K Replikationsursprung, der die Notwendigkeit für die Verdauung der Muttergesellschaft Plasmid oder umfangreiche PCR-Produkt Reinigung als Muttergesellschaft Plasmid ist nicht in der Lage, in die Bakterien für Rekombination verwendet replizieren entgegensteht, und kann nur in besonderen Belastungen wie EC100 2 nur , 16 (Tabelle 1 und Tabelle 2). Schließlich, eine gemeinsame Methode zur Generierung transgener C. elegans ist durch den Einsatz von biolistischen Bombardement durch Selektion auf transgene Würmer über die Rettung der unc-119-Mutation 17 an. Um die Fosmide mit Bombardements, entwickelten wir die pLoxP unc-119-Plasmid, das benutzt, um die unc-119-Marker auf die Fosmid Rückgrat 2 zu integrieren lassen.
I. Oligo Design
Mit Recombineering die gewünschten Sequenzen können an einer beliebigen Stelle innerhalb des Gens eingefügt werden. Gemeinsame Seiten sind am 5'-Ende oder 3'-Ende je nach funktionellen Domänen, Splice-Varianten oder post-translationale Modifikationen wie die Spaltung durch Proteasen. Die pMOD4 GFP-Plasmid von unserem Labor erstellt wurden, können verwendet werden, um eine FLAG-markierte GFP an beliebiger Stelle einzufügen, da das Plasmid enthält ein Startcodon und keine 3 'Stop-Codon (Abbildung 1). Im Gegensatz dazu hat die TAP-Tag Versionen für 5 'und 3' Fusionen durch TEV-Spaltung bei der Aufreinigung 18,19 verwendet.
- Planen Sie die Website von Tag-Insertion innerhalb des Gens. Betrachten Sie alternative Promotoren, funktionellen Domänen, alternative Slicing und post-translationale Modifikationen, wenn man die Einstichstelle. Verschiedene Insertionsstellen könnte verwendet werden, um alle, einen oder einige Isoformen von einem spezifischen Gen-Tag werden. Identifizieren 50 bp Regionen stromaufwärts und stromabwärts von der Einfügemarke.
- Entwerfen Sie die Oligos (Tabelle 1). Entweder 100 nM Skala - Gel gereinigt Oligos oder Ultramer Oligos von Integrated DNA Technologies kann für das Verfahren verwendet werden.
Um galK Recombineering, müssen Sie galK Primer mit 50 bp Homologie Design zu einem Bereich flankieren die gewünschte Stelle geändert werden und das 3'-Ende dieser Primer binden an die galK Kassette, die in beiden pMOD4 galK-G und pMOD4 ist galK-GT (Abbildung 1). Die zukunftsgerichteten Oligo werden 5'------- 50 bp Homologie ------- CCTGTTGACAATTAATCATCGGCA-3 'und umgekehrt eine als 5'------- 50 bp Homologie auf dem komplementären Strang - ------ TCAGCACTGTCCTGCTCCT-3 '.
Um galK Rekombination mit GFP, müssen Sie pMOD4 galK-G oder pMOD4 galK-GT-Primer mit 50 bp Homologie Design zu einem Bereich flankieren die gewünschte Stelle zu modifizieren (Abbildung 1). Achten Sie darauf, das Fusionsprotein in Rahmen zu halten. Die ATG kann verworfen werden, falls gewünscht. Das 3'-Ende dieser Primer binden an Regionen der GFP Homologie flankierenden galK Kassette. Hinweis: Die ersten und letzten Codons GFP sind unterstrichen, um das Leseraster zu demonstrieren. Die zukunftsgerichteten Oligo werden 5'------- 50 bp Homologie ------- ATG GATTACAAGGACGATGACGATAAGATGAG -3 '3' und den Rückwärtsgang ein 5'------- 50 bp Homologie auf den komplementären Strang ------- CAA AGCTTGTGGGCTTTTGTATAG-3 '
Um galK C-term TAP-Rekombination, müssen Sie pMOD4 galK-GT-Primer mit 50 bp Homologie Design zu einem Bereich flankieren die gewünschte Stelle zu modifizieren (Abbildung 1). Achten Sie darauf, das Fusionsprotein in Rahmen zu halten. Das 3'-Ende dieser Primer binden an Regionen der TAP Homologie flankierenden galK Kassette. Hinweis: Die ersten und letzten Codons TAP sind unterstrichen, um das Leseraster zu demonstrieren. Die zukunftsgerichteten Oligo werden 5'------- 50 bp Homologie ------ ATG GAAAAGAGAAGATGGAAAAAG - -3 'und umgekehrt eine 5'------- 50 bp Homologie auf dem komplementären Strang - ------ GGT TGACTTCCCCGC -3 '
Um galK N-term TAP-Rekombination, müssen Sie pMOD4 galK-GT-Primer mit 50 bp Homologie Design zu einem Bereich flankieren die gewünschte Stelle zu modifizieren (Abbildung 1). Achten Sie darauf, das Fusionsprotein in Rahmen zu halten. Das 3'-Ende dieser Primer binden an Regionen der TAP Homologie flankierenden galK Kassette. Hinweis: Die ersten und letzten Codons TAP sind unterstrichen, um das Leseraster zu demonstrieren. Die zukunftsgerichteten Oligo werden 5'------- 50 bp Homologie ------ ATG GCAGGCCTTGCGC - -3 'und umgekehrt eine 5'------- 50 bp Homologie auf dem komplementären Strang - ------ AAG TGCCCCGGAGGATGAGATTTTCT -3 ' - Erzeugen Sie eine Reihe von flankierenden Oligos für die PCR in späteren Schritten sowie für die Sequenzierung des Fosmid. Dies sind Standard-PCR-Oligos, die ~ 100 bp binden sollte vor und hinter der Insertionsstelle.
II. Transfer zum SW016 Bakterien Fosmid
Die Fosmide aus dem C. elegans Fosmid Bibliothek sind in der EPI300 Bakterienstamm (F-mcrA Δ (MRR-hsdRMS-McrBC) φ80dlacZΔM15 ΔlacX74 recA1 endA1 araD139 Δ (ara, leu) 7697 galu galK λ-rpsL nupG TRFA tonA) (Epicentre Biotechnologies, Madison, WI vorgesehen ), mit dem Fosmid Ausdruck über eine einzelne Kopie pro Zelle, um DNA-Ausbeuten bei der Aufreinigung (Tabelle 2) zu verbessern erhöht werden. Für Rekombination wird die Fosmid müssen auf die SW106 Bakterienstamm (mcrA Δ (MRR-hsdRMS-McrBC) ΔlacX74 DeOr endA1 araD139 Δ (ara, leu) 7697 rpsL recA1 nupG φ80dlacZΔM15 [λc1857 (cro-BIOA) <> Tet übertragen werden ] (cro-BIOA) <> araC-PBAD Cre ΔgalK) (NCI-Friedrich) Stamm, der das λred homologe Rekombination Gene unter der Kontrolle eines temperatursensitiven λ-Repressor und Arabinose induzierbare Cre-Rekombinase (Tabelle 2) 15 trägt.
- Bestellen Sie die Fosmid Klon von Gene von Interesse (GOI) von Geneservice (Cambridge, UK) mit Wormbase als Leitfaden. Bei der Auswahl der Klone, wählen wir diejenigen, die die indische Regierung habe in der Mitte der Sequenz. Ones, dass die benachbarten Gene auszuschließen vorzuziehen wäre, könnte aber schwer zu finden.
- Kultur der Fosmid Klon von der indischen Regierung in LB mit 12,5 pg / mL Chloramphenicol bei 37 ° C.
- Wachsen einer 1,5 ml Übernachtkultur der Fosmid und mini-prep die Fosmid DNA unter Verwendung des Epicentre Fosmid Prep Kit (Epicentre Biotechnologies, Madison, WI). Wir folgen dem alternativen Protokoll in den Anweisungen, die Addition der Riboshredder Mix zu einem früheren Schritt wird beschrieben.
- Bestimmen Sie die Fosmid DNA-Konzentration mit einem Spektralphotometer.
- Bereiten Sie die elektrokompetente SW106 Zellen durch Aufwachsen einer 5 ml Übernachtkultur von SW106 in einem 14 mL-Snap-Cap-Rohr mit LB-Medium mit 12,5 pg / mL Chloramphenicol bei 32 ° C.
- Inoculate 1 mL in 100 ml LB mit Chloramphenicol in einem 2 L Flasche. Wachsen SW106 Bakterien zu einer OD 600 von 0,6 bis 0,8. NICHT Hitze-Schock.
- Pellet durch Zentrifugation bei 5000xg für 5 Minuten, das Pellet durch vorsichtiges Vortexen, und mit 50 mL eiskaltem 10% Glycerin. Wiederholen Sie diesen Waschschritt einmal.
- Pellet der SW106 durch Zentrifugation und saugen alle, aber ~ 500 ul von jedem Überstand
- Resuspendieren der Pellets durch sanftes Vortexen. Freeze-100 ul Aliquots in flüssigem Stickstoff oder mit Trockeneis und bei -80 ° C für eine spätere Verwendung.
- Transformieren Sie die Fosmid DNA in elektrokompetente SW106 Zellen durch Elektroporation der Bakterien mit ~ 50 ng Fosmid DNA mit einer Eppendorf 2510 Elektroporator bei 1350 Volt in 0,1 cm Abstand Küvetten.
- Recover Bakterien in 1 ml LB für 1 Stunde bei 32 ° C.
- Platte Aliquots auf LB-Platten mit Chloramphenicol (12,5 pg / mL) und inkubieren bei 32 ° C über Nacht.
- Überprüfen Sie, ob die Anwesenheit der indischen Regierung durch Kolonie-PCR. Wachsen einer 5-ml Übernachtkultur in LB mit 12,5 pg / mL Chloramphenicol bei 32 ° C. Add 0,5 ul der Kultur zu einer Standard-PCR-Reaktion mit den flankierenden Oligos und erhöhen die ersten 95 ° C Inkubation für 5 Minuten, um die Bakterien zu lysieren vor der PCR.
- Bereiten Sie eine Glycerin Lager für die langfristige Lagerung.
III. Insertion von galK Gene durch Recombineering
In der ersten Stufe der Fosmid Modifikation wird die galK Gen in die Fosmid durch homologe Rekombination eingefügt, und die korrekt geändert Fosmide sind durch Wachstum auf Minimalmedium mit Galactose als einzige Kohlenstoffquelle (Abbildung 2A) ausgewählt. Der SW106 Bakterien wachsen langsam über die minimal Medien und 3-5 Tage benötigt, um Kolonien zu sehen.
- Bereiten Sie MOPS Minimalmedium-Platten mit 0,2% Galaktose. MOPS minimal Medien sind von Teknova Inc. (Hollister, CA) (Katalog # M2106), aber nicht mit dem mitgelieferten Glukose.
MOPS Minimalmedium mit 0,2% Galactose (1 L)
Autoclave 15 Gramm Agar in 870 ml Wasser
Abkühlen auf 55 ° C und fügen:100 mL 10x MOPS minimal Medien 5 mL 0,2 mg / ml d-Biotin (sterilfiltriert) 4,5 ml 10 mg / ml L-Leucin (1%, erhitzt, dann abgekühlt und sterilfiltriert) 10 mL 20% Galaktose (autoklaviert) 1 mL 12,5 mg / ml Chloramphenicol in EtOH 2,55 ml 20% NH 4 Cl 10 mL 0,132 M zweibasische Kaliumphosphat - PCR Verstärkung des pMOD4 galK-G oder pMOD4 galK-GT-Kassetten unter Verwendung der Primer entworfen oben. Wir haben Phusion (New England Biolabs, Ipswich, MA) oder GoTaq (Promega, Madison, WI) verwendet.
- Gel reinigt die daraus resultierende Band. Quantifizierung der Ausbeute durch Gel oder Nanodrop Spektralphotometer. Dieses PCR-Produkt ist bereit für Schritt 3,14.
- Impfen einer Übernachtkultur von SW106 Zellen mit den Fosmid DNA in 5 ml LB mit Chloramphenicol (12,5 pg / mL). Wachsen bei 32 ° C.
- Set Schüttelwasserbad bis 42 ° C zu warm-up mit einem sterilen 250 ml Flasche in die Halterung ein. Die Verwendung eines Schüttelwasserbad ist entscheidend für immer hohen Wirkungsgrad.
- 1 ml der Übernacht-Kultur in 100 ml LB und Chloramphenicol in einem 2 L Flasche. Wachsen bis zu einer OD 0,6-0,8. Dies dauert in der Regel 3-4 Stunden.
- Transfer 50 ml SW106 Zellen an die 250 ml Flasche und Hitze-Schock bei 42 ° C für genau 20 min. in einem Schüttel-Wasserbad bei 100 rpm Lassen Sie die übrigen Bakterien bei 32 ° C als nicht induzierte Kontrolle.
- Kühlen Sie die induzierten und induzierten Bakterien auf Eis für 10 Minuten.
- Übertragen Sie die Proben auf zwei sterile Zentrifugenröhrchen und Pellet bei ~ 5000xg für 5 Minuten.
- Abgießen alle Überstand und das Pellet in 1 ml eiskaltem 10% Glycerin durch sanftes Vortexen (dh Einstellung 3-4).
- Wenn resuspendiert, fügen Sie eine weitere 49 ml eiskaltem 10% Glycerin und Pellet die Proben bei ~ 5000xg für 5 Minuten.
- Wiederholen Sie Schritt 3.9, 3.10 und 3.11 wieder.
- Entfernen Sie alle Überstand durch Umdrehen der Röhrchen, und das Pellet in der verbleibenden Flüssigkeit (ca. 500 ul pro Stück). Aliquot in 100 ul Proben auf Trockeneis eingefroren und bei -80 ° C. Diese sind gut für Wochen bis Monate. (Wir normalerweise hier aufhören und führen Sie die Elektroporation des folgenden Tages).
- Elektroporieren der induzierten und nicht-induzierten SW106-Zellen mit 150 ng des PCR-Produktes mit 0,1 cm Abstand Küvetten in einer Eppendorf 2510 Elektroporator Satz bei 1350 Volt.
- Recover die Bakterien in 1 ml LB in einem 14 ml Falcon-Röhrchen. Inkubation bei 32 ° C für 4,5 Stunden.
- Pellet die Bakterien in einer Mikrozentrifuge bei 13.200 rpm für 15 Sekunden. Die Bakterien sind in M9 resuspendiert und anschließend zweimal gewaschen entfernen Vollmedium (siehe unten für Rezept).
- M9-Medium (1 L)
- 6g Na 2 HPO 4
- 3g KH 2 PO 4
- 1g NH 4 Cl
- 0,5 g NaCl
- AUTOCLAVE
- Nach dem zweiten Waschen wird der Überstand entfernt und das Pellet in 1 ml M9 vor dem Ausplattieren serielle Verdünnungen in M9 (100 ul, 100 ul einer Verdünnung von 1:10 und 100 ul 1:100) auf MOPS Minimalmedium resuspendiert.
- Inkubieren 3-5 Tage bei 32 ° C im Brutschrank. Hinweis: Seien Sie geduldig, wie das wahre Positive langsam wachsen.
- Streak ein paar Kolonien auf MacConkey Agar-Indikator-Platten (BD # 281810) mit 1% Galaktose und Chloramphenicol 12,5 ug / mL ergänzt. Alle Kolonien erscheinen nach dem letzten Schritt sollten galK + sein, aber um keine loszuwerden galK - Verunreinigungen, ist es wichtig, einzelne, leuchtend rosa Kolonien, bevor Sie den zweiten Schritt zu erhalten. Die galK - Kolonien werden weiß / farblos und die galK + Bakterien werden leuchtend rot / pink durch eine pH-Änderung, die aus vergorenen Galaktose nach einer Inkubation über Nacht bei 32 ° C.
- Wählen Sie eine einzelne Kolonie und impfen eine 5 ml LB + Chloramphenicol-Nacht-Kultur für das Wachstum bei 32 ° C.
- Bestätigen Einsetzen der galK Gen an der richtigen Stelle über PCR unter Verwendung der flankierenden Oligos. Add 0,5 ul der Kultur zu einer Standard-PCR-Reaktion und erhöhen die ersten 95 ° C Inkubation für 5 Minuten, um die Bakterien zu lysieren. Das PCR-Produkt sollte in der Größe durch die Anwesenheit des galK Gen hochgeschaltet werden.
- Bereiten Sie eine Glycerin Lager für die Lagerung.
IV. Ersatz von galK mit Tag-Sequenzen von Recombineering
In diesem Stadium der galK Gen wird durch den gewünschten Tag-Sequenzen und die korrekt geändert Fosmide werden durch Selektion gegen die galK Gens ausgewählt von den toxischen Galaktose analog Desoxygalactose (DOG) (Abbildung 2B) ersetzt.
- Bereiten Sie MOPS Minimalmedium-Platten mit 0,2% Desoxygalactose (DOG) und 0,2% Glycerin. MOPS minimal Medien sind von Teknova Inc. (Hollister, CA) (Katalog # M2106), aber nicht mit dem mitgelieferten Glukose.
MOPS Minimalmedium mit 0,2% DOG und Glycerin (1 L)
Autoclave 15 Gramm Agar in 860 ml Wasser
Abkühlen auf 55 ° C und fügen:100 mL 10x MOPS minimal Medien 5 mL 0,2 mg / ml d-Biotin (sterilfiltriert) 4,5 ml 10 mg / ml L-Leucin (1%, erhitzt, dann abgekühlt und sterilfiltriert) 10 mL 20% Desoxygalactose (sterilfiltriert) 10 mL 20% Glycerin (autoklaviert) 1 mL 12,5 mg / ml Chloramphenicol in EtOH 2,55 ml 20% NH 4 Cl 10 mL 0,132 M zweibasische Kaliumphosphat - PCR zu amplifizieren die Tag-Fragmente aus pMOD4 GFP, pBS1761 (N-term TAP) oder pBS1479 (C-term TAP) unter Verwendung der gleichen Oligos in der ersten Runde eingesetzt oder bei kürzeren GFP oder TAP-spezifischen Oligos (die innere Sequenzen in Schritt 1.2 ). Wenn Sie mehrere Konstrukte sind, ist es besonders nützlich, um die kürzere Oligos als die gleiche PCR-Produkt für alle Konstrukte benutzen kann.
- Gel reinigt die PCR-Produkt und messen die Konzentration über Gel oder Spektrophotometrie.
- Generieren induzierten und nicht-induzierten zuständige SW106 Durchführung der Fosmid mit dem galK Gen eingefügt folgenden Schritte 3,4-3,13 oben.
- Elektroporieren der induzierten und nicht-induzierten SW106 Zellen mit ~ 100 ng des PCR-Produktes mit 0,1 cm Abstand Küvetten in einer Eppendorf 2510 Elektroporator Satz bei 1350 Volt.
- Erholen Sie sich in 1 ml LB in einer 14 ml-Snap-Cap-Röhrchen und inkubieren in einem 32 ° C Schüttler für 4,5 Stunden.
- Wash und verdünnten Bakterien wie in den Schritten 3,16 und 3,17. Platte Bakterien auf MOPS Minimalmedium-Platten mit 0,2% 2-Desoxy-Galactose (DOG) und 0,2% Glycerin.
- Inkubation bei 32 ° C für 3 Tage.
- Vier Kolonien werden verwendet, um 5 ml machenÜbernacht-Kulturen in LB mit 12,5 pg / mL Chloramphenicol. Diese sind für die Kolonie-PCR verwendet, wie oben zu bestätigen, dass die Kassette eingelegt wurde. Wir verwenden sowohl die kürzere GFP / TAP spezifischen Oligos und die flankierenden Oligos auf der rechten Seite einfügen und rechten Seite zu zeigen. GFP ist ~ 800 bp und TAP ist ~ 550 bp, während galK beträgt 1,4 kb.
- Bereiten Sie eine Glycerin Lager.
V. Die Zugabe von unc-119 Gene von cre-loxP Rekombination
Eine gemeinsame Einrichtung zur Erzeugung transgener Tiere mit dem modifizierten Fosmide wird durch den Einsatz von biolistischen Bombardement. Diese Technik verwendet DNA-beschichtete Goldpartikel auf Fosmid DNA in C. Einführung elegans. Transgene Tiere sind in der Regel über Rettung der unc-119-Mutante mit einem UNC-119 Transgen identifiziert. In diesem Schritt wird das unc-119-Gen auf die Fosmid Rückgrat in cis von cre-loxP Rekombination mit dem pLoxP hinzu unc-119 Plasmid (Abbildung 2C).
- Bereiten Sie zuständige SW106 Bakterien, die die modifizierte Fosmid Schritt 4,9 mit 2,5-2,9 Schritten. NICHT bei 42 ° C auslösen
- Elektroporieren mit 50 ng. pLoxP unc-119 von einem Mini-Prep mit 0,1 cm Abstand Küvetten in einer Eppendorf 2510 Elektroporator bei 1350 Volt eingestellt.
- Recover Bakterien in LB mit 0,1% Arabinose für 1 Stunde bei 32 ° C.
- Platte Aliquots auf LB-Platten mit Ampicillin 50μg/mL und 12,5 pg / mL Chloramphenicol. Inkubation bei 32 ° C über Nacht. die Auswahl für die Integration von pLoxP unc-119 in die Fosmid.
- Wachsen einer Übernachtkultur in LB mit Ampicillin 50μg/mL und 12,5 pg / mL Chloramphenicol. Verwenden Sie 0,5 ul für PCR überprüft die Anwesenheit der unc-119-Gen mit dem unc-119 F (5'-CAAATCCGTGACCTCGACAC-3 ') und unc-119 R (5'-CACAGTTGTTTCTCGAATTTGG-3') Oligos (Tabelle 1).
- Machen Sie eine Glycerin Bilanz der letzten Fosmid.
VI. Large Scale Fosmid Vorbereitung
Zur Erleichterung der Einholung der größere Mengen an DNA für Fosmid Bombardement nötig, in diesem Schritt die Fosmid ist es, die EPI300 Bakterien übertragen. Dieser Stamm hat die Fähigkeit, die Fosmid Kopienzahl zu erhöhen, um die Erträge während der DNA-Präparation zu erhöhen.
- Wachsen einer 5-ml Übernachtkultur der Bakterien aus Schritt 5.5 in LB mit Ampicillin und Chloramphenicol bei 32 ° C. Verwenden Sie Epicentre Fosmid prep Kit zur Fosmid von 1,5 mL der Kultur zu isolieren.
- Elektroporieren ~ 50 ng in die EPI300 Bakterien mit 0,1 cm Abstand Küvetten in einer Eppendorf 2510 Elektroporator Satz bei 1350 Volt. Die EPI300 Bakterien können von Epicentre Biotechnologies (Madison, WI) erworben werden.
- Recover Bakterien in LB für 1 Stunde bei 37 ° C. Platte Aliquots auf LB Agar mit 50 ug / mL und 12,5 pg / mL Chloramphenicol.
- Wachsen und induzieren die EPI300 Bakterien mit den geänderten Fosmid mit der mitgelieferten Anleitung. Eine 50 ml induzierten Kultur geben> 10 ug von gereinigtem Fosmid DNA. Purify die Fosmid mit dem Epizentrum Fosmid prep kit.
VII. Beschuss
- Verwenden Sie 10 ug. von Fosmid DNA zu bombardieren die DP38 Wurmstamm wie beschrieben (D. Hochbaum, A. Ferguson, und A. Fisher, Jupiter, in press).
VIII. Repräsentative Ergebnisse
Die Änderung der Fosmide über Recombineering robust und Erfolgsquoten von> 90% in die negative Selektion Schritt werden routinemäßig 2 beobachtet. Dieses Protokoll nimmt auch ~ 2 Wochen in Anspruch was die Herstellung von Transgenen ziemlich schnell. Das Protokoll wurde auch von anderen Laboren mit Erfolg 20 versucht worden.
| Oligo | Reihenfolge |
| C-term TAP F | ATGGAAAAGAGAAGATGGAAAAAG |
| C-term TAP R | GGTTGACTTCCCCGC |
| FLAG-GFP F | ATGGATTACAAGGACGATGACGATAAGATGAG |
| FLAG-GFP R | CAAAGCTTGTGGGCTTTTGTATAG |
| N-term TAP F | ATGGCAGGCCTTGCGC |
| N-term TAP R | AAGTGCCCCGGAGGATGAGATTTTCT |
| galK F | CCTGTTGACAATTAATCATCGGCA |
| galK R | TCAGCACTGTCCTGCTCCT |
| unc-119 F | CAAATCCGTGACCTCGACAC |
| unc-119 R | CACAGTTGTTTCTCGAATTTGG |
Tabelle 1. Oligonukleotide für die PCR verwendet.
| Plasmide | Quelle | Verfügbar unter |
| Fosmid Klon | Geneservice Ltd | Geneservice |
| pGalK | 15 | NCI-Friedrich |
| pMOD4-RT-G | 2 | Addgene |
| pMOD4-galK-G | ||
| pMOD4-galK-GT | ||
| pLoxP-unc-119 | ||
| pMOD4-GFP | ||
| Bakterien | ||
| SW106 | 15 | NCI-Friedrich |
| EPI300 | Epicentre Biotechnologies | Epicentre |
| EC100D pir-116 | ||
Tabelle 2. Dehnungs-und Vektor-Verfügbarkeit.
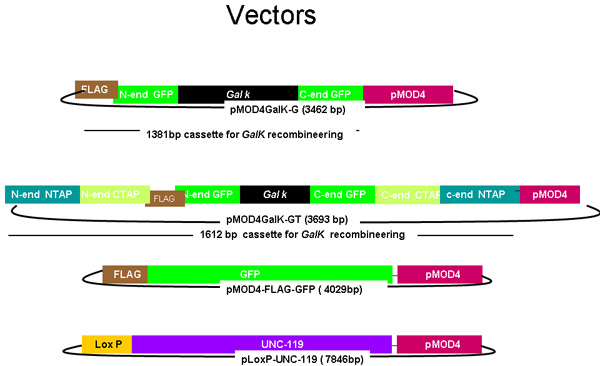
Abbildung 1.
Schematische Darstellung der pMOD4-galK-G, und pMOD4-galK-GT, pMOD4 GFP, pLoxP-unc-119
Die pMOD4-galK-G-Plasmid besteht aus dem galK Kassette (schwarz) um 50 Nukleotid-Regionen identisch mit der 5 'und 3' Enden der FLAG (Brown)-GFP (grün) flankiert, während PMOD 4-galK-GT besteht aus den galK Kassette sowohl von der FLAG-GFP Homologieregionen und 50 Nukleotid-Regionen identisch mit der 5 'und 3' Enden der N-terminalen und C-terminalen TAP (blau und orange, jeweils) flankiert. pMOD4-FLAG-GFP besteht aus dem vollen GFP Kassette mit einem 5 'FLAG-Tag und pLoxP unc-119 besteht aus dem unc-119 genomischen Sequenz (lila) in ein Plasmid mit einer loxP site. Alle Plasmide nutzen die R6K-basierte pMOD4 (rot)-Backbone, die nicht in SW106 replizieren.
Abbildung 2.
Übersicht der galK Recombineering Prozess
Abbildung 2A-2C Separate Zahlen, die die Schritte und die Zeit in Recombineering mit dem galK Kassette beteiligt. Dies sind die gleichen Figuren, die in Abbildung 2d zusammengeführt werden, sondern getrennt für Klarheit und gute Lesbarkeit. Ein Fosmid von Interesse ist zunächst in einem zweistufigen Verfahren mit dem Einsetzen der galK Kassette 50 bp Regionen Homologie zu FLAG-GFP oder TAP (Abbildung 2A) durch Austausch von dieser Kassette von FLAG-GFP oder TAP gefolgt flankiert geändert ( Abbildung 2B). Später wurde das unc-119-Marker für den Einsatz bei der Erzeugung transgener Tiere wird in den loxP-Ort auf der Fosmid Rückgrat (Abbildung 2C) eingesetzt.
2D zeigt eine zusammengeführte Figur des galK Recombineering Verfahren.
Outline of galK Recombineering Verfahren wie in der obigen einschließlich der Zeit für jeden Schritt aus der Verschmelzung der 2A-2C erforderlich beschrieben.
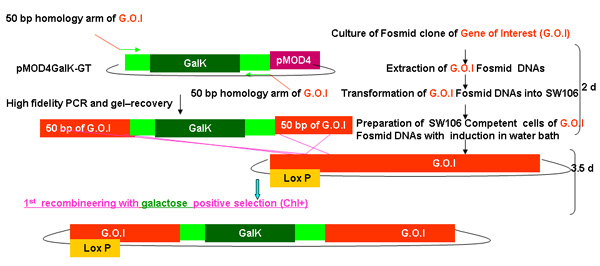
Abbildung 2a. Die galK Einfügung in galK Rekombination.
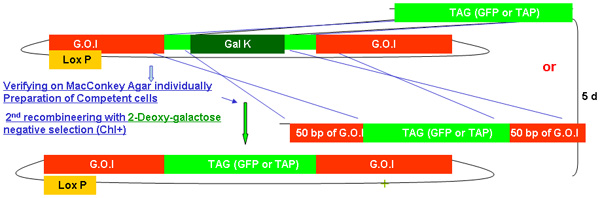
Abbildung 2b. Die TAG (GFP / TAP)-Insertion in galK Rekombination.
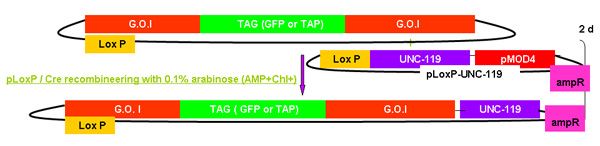
Abbildung 2c. Die Zugabe von unc-119.
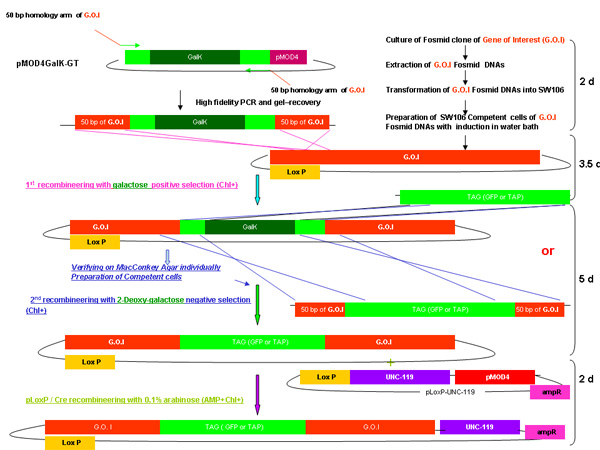
Abbildung 2d. Das fusionierte Überblick über galK Rekombination.
Diskussion
Die Erzeugung von Transgenen aus Fosmide bietet den Vorteil der Beibehaltung aller von den nativen Promotor-Elemente, Splice-Varianten und 3 'UTR regulatorische Elemente. Dies kann mit dem Bau eines Transgens, das eher auf den einheimischen Ausdruck Muster oder den Bau eines funktionalen Transgen, wenn andere Ansätze 5 alle fehlschlagen wird führen können. Die daraus resultierende Transgene tragen kann eine Vielzahl von Epitop-Tags einschließlich GFP oder ein TAP-Tag.
Der ...
Offenlegungen
Danksagungen
Die Autoren bedanken sich bei Lindsey Nash für die Hilfe bei der Entwicklung der Technik zu danken. Diese Arbeit wurde vom NIH AG028977 zu ALF, ein Pilotprojekt Zuschuss von der University of Pittsburgh OAIC (AG024827), und Anschubfinanzierung von der University of Pittsburgh finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| FosmidMAX kit | Epicentre Biotechnologies | FMAX046 | |
| GoTaq | Promega Corp. | M7122 | |
| MOPS Media | TEKnova, Inc. | M2120 | |
| 0.132 M Potassium phosphate solution | TEKnova, Inc. | M2102 | |
| D-galactose | Sigma-Aldrich | G0750 | |
| 2-deoxygalactose | Sigma-Aldrich | D4407 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Leucine | Sigma-Aldrich | L8000 | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Phusion DNA polymerase | New England Biolabs | F-530S | |
| MacConkey agar base | BD Biosciences | 281810 | |
| Arabinose | Sigma-Aldrich | A3131 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S5136 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Sodium chloride | Sigma-Aldrich | S5886 | |
| Glycerol | Sigma-Aldrich | G2025 | |
| Bacto Agar | BD Biosciences | 214010 |
Referenzen
- Mello, C., Fire, A. DNA transformation. Methods Cell Biol. 48, 451-482 (1995).
- Zhang, Y., Nash, L., Fisher, A. L. A simplified, robust, and streamlined procedure for the production of C. elegans transgenes via recombineering. BMC Dev Biol. 8, 119-119 (2008).
- Antebi, A., Yeh, W. H., Tait, D., Hedgecock, E. M., Riddle, D. L. daf-12 encodes a nuclear receptor that regulates the dauer diapause and developmental age in C. elegans. Genes and Development. 14, 1512-1527 (2000).
- Snow, M. I., Larsen, P. L. Structure and expression of daf-12: a nuclear hormone receptor with three isoforms that are involved in development and aging in Caenorhabditis elegans. Biochim. Biophys. Acta. 1494, 104-116 (2000).
- Fisher, A. L., Page, K. E., Lithgow, G. J., Nash, L. The Caenorhabditis elegans K10C2.4 Gene Encodes a Member of the Fumarylacetoacetate Hydrolase Family. A CAENORHABDITIS ELEGANS MODEL OF TYPE I TYROSINEMIA. J Biol.Chem. 283, 9127-9135 (2008).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75, 855-862 (1993).
- Lehrbach, N. J. LIN-28 and the poly(U) polymerase PUP-2 regulate let-7 microRNA processing in Caenorhabditis elegans. Nat Struct Mol Biol. 16, 1016-1020 (2009).
- Tursun, B., Cochella, L., Carrera, I., Hobert, O. A toolkit and robust pipeline for the generation of fosmid-based reporter genes in C. elegans. PLoS One. 4, e4625-e4625 (2009).
- Bamps, S., Hope, I. A. Large-scale gene expression pattern analysis, in situ, in Caenorhabditis elegans. Brief. Funct. Genomic. Proteomic. , (2008).
- Dolphin, C. T., Hope, I. A. Caenorhabditis elegans reporter fusion genes generated by seamless modification of large genomic DNA clones. Nucleic Acids Res. 34, e72-e72 (2006).
- Sarov, M. A recombineering pipeline for functional genomics applied to Caenorhabditis elegans. Nat. Methods. 3, 839-844 (2006).
- Yang, X. W., Model, P., Heintz, N. Homologous recombination based modification in Escherichia coli and germline transmission in transgenic mice of a bacterial artificial chromosome. Nat Biotechnol. 15, 859-865 (1997).
- Court, D. L., Sawitzke, J. A., Thomason, L. C. Genetic engineering using homologous recombination. Annu.Rev.Genet. 36, 361-388 (2002).
- Westenberg, M., Bamps, S., Soedling, H., Hope, I. A., Dolphin, C. T. Escherichia coli MW005: lambda Red-mediated recombineering and copy-number induction of oriV-equipped constructs in a single host. BMC Biotechnol. 10, 27-27 (2010).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36-e36 (2005).
- Penfold, R. J., Pemberton, J. M. An improved suicide vector for construction of chromosomal insertion mutations in bacteria. Gene. 118, 145-146 (1992).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Puig, O. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat.Biotechnol. 17, 1030-1032 (1999).
- Achilleos, A., Wehman, A. M., Nance, J. PAR-3 mediates the initial clustering and apical localization of junction and polarity proteins during C. elegans intestinal epithelial cell polarization. Development. 137, 1833-1842 (2010).
- Maduro, M., Pilgrim, D. Identification and cloning of unc-119, a gene expressed in the Caenorhabditis elegans nervous system. Genetics. 141, 977-988 (1995).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten