Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Sammeln Variable-Konzentration Isotherme Titrationskalorimetrie Datasets in Auftrag, um bindende Mechanismen bestimmen
In diesem Artikel
Zusammenfassung
ITC ist ein leistungsfähiges Werkzeug für die Untersuchung der Bindung eines Liganden an seinen Wirt. In komplexen Systemen kann jedoch mehrere Modelle die Daten gleich gut passen. Die hier beschriebene Methode bietet eine Möglichkeit, die entsprechende Bindung Modell für komplexe Systeme aufzuklären und extrahieren Sie die entsprechende thermodynamische Parameter.
Zusammenfassung
Isotherme Titrationskalorimetrie (ITC) wird häufig verwendet, um die thermodynamischen Parameter der Bindung eines Liganden an einen Host-Makromolekül verbunden zu bestimmen. ITC hat einige Vorteile gegenüber gängigen spektroskopischen Methoden zur Untersuchung von Host / Ligand-Wechselwirkungen. Zum Beispiel wird die freiwerdende Wärme oder absorbiert wird, wenn die beiden Komponenten interagieren direkt gemessen und erfordert keine exogenen Reporter. So ist die Bindungsenthalpie und der Assoziationskonstante (Ka) werden direkt von ITC-Daten erhalten, und kann verwendet werden, um die entropische Beitrag berechnen. Darüber hinaus ist die Form der Isotherme abhängig von der c-Wert und die mechanistischen Modell beteiligt. Die c-Wert ist definiert als c = n [P] TKA, wobei [P] t ist die Protein-Konzentration und n die Anzahl der Liganden-Bindungsstellen innerhalb der Host. In vielen Fällen sind mehrere Bindungsstellen für einen bestimmten Liganden nicht gleichwertig und ITC ermöglicht die Charakterisierung des thermodynamischen Bindungsparameter für jedes einzelne Bindungsstelle. Dies erfordert jedoch, dass die korrekte Bindung Modell verwendet werden. Diese Wahl kann problematisch sein, wenn verschiedene Modelle der gleichen experimentellen Daten passen. Wir haben bereits gezeigt, dass dieses Problem durch die Durchführung von Experimenten an mehreren c-Werte können nicht umgangen werden. Die multiple Isothermen bei verschiedenen c-Werte erhalten werden gleichzeitig verschiedene Modelle passen. Das richtige Modell ist neben basierend auf die Güte der Anpassung über die gesamte variable-c Datensatz identifiziert. Dieser Prozess ist hier, um die Aminoglykosid-Resistenz verursachenden Enzym Aminoglycosid N-6'-Acetyltransferase-II (AAC (6 ')-II) beantragt. Obwohl unsere Methode ist anwendbar auf jedem System, ist die Notwendigkeit, diese Strategie besser mit einem Makromolekül-Ligand-System zeigt Allosterie oder Kooperativität gezeigt, und wenn verschiedene verbindliche Modelle bieten im Wesentlichen identisch passt auf die gleichen Daten. Nach unserer Kenntnis gibt es keine derartigen Systeme im Handel erhältlich. AAC (6 ')-II ist ein Homo-Dimer mit zwei aktiven Zentren, welche Kooperativität zwischen den beiden Untereinheiten. Allerdings ITC-Daten an einem einzigen c-Wert erhalten kann ebenso gut fit sein, um mindestens zwei verschiedene Modelle einer Zwei-sets-of-Sites eigenständiges Modell und eine Zwei-Seiten sequentielle (Genossenschaft) Modell. Durch Variation der c-Wert wie oben erläutert, wurde festgestellt, dass die korrekte Bindung Modell für AAC (6 ')-II a two-site sequenzielle Bindung Modell ist. Hier beschreiben wir die Schritte, die bei der Durchführung von ITC Experimenten werden, um Datensätze für variable-c-Analysen erhalten müssen.
Protokoll
1. Vorbereitung Stammlösungen
- Purify das Makromolekül von Interesse. (In diesem Fall ist Aminoglykosid N-6'-Acetyltransferase-II (AAC6'-II), isoliert wie an anderer Stelle berichtet. 13)
- Bereiten Sie 4 Liter Dialyse-Puffer. (In diesem Fall haben wir 25 mM 4 - (2-hydroxyethyl)-1-piperazineethanesulfonic Säure (HEPES, MW 238,3 g / mol), enthaltend 2 mM Ethylendiamintetraessigsäure (EDTA, MW 292,2), bei pH 7,5.)
- Dialysieren das Protein Das AAC (6 ')-II Probe (5 mL bei 400 uM) verwendet in der Dialyse-Puffer (3 x 1,3 l) dialysiert werden müssen. Die endgültige Dialyselösung gehalten wird, um die Maschine spülen und die Proben zu verdünnen.
- Filtern Sie die letzte Dialyse-Lösung durch einen 0,45 um Filter aus Zellulose, und bei 4 ° C. Will als "Laufpuffer" genannt werden
- Filter Protein-Lösung durch einen 0,2 um Spritzenfilter, die gespült worden ist gründlich mit Laufpuffer. Messen Sie die Endkonzentration des Proteins mit einem Standard-Test (Bradford, Lowry, etc.) oder UV-Absorption. Shop des Proteins an langfristige Stabilität zu maximieren. (Im Falle von AAC (6 ')-II bedeutet dies, Lagerung bei 4 ° C. AAC (6')-II nicht behalten Aktivität nach dem Einfrieren und Auftauen).
- Bereiten Sie 200 ul 25 mM Stammlösung von Acetyl-Coenzym A (AcCoA, den Liganden, MW 809,57) durch Lösen von 4,0 mg in Laufpuffer. Einfrieren bei -78 ° C bis zur Verwendung.
- Alle Protein und Ligand Proben müssen aus dem gleichen Stammlösungen stammen, zu Zufallsstichprobe-to-sample Schwankungen in der Konzentration zu minimieren. Eine einzige Korrekturfaktor für die Protein-Konzentration über die gesamte variable-c Datensatz wird in der Analyse angepasst. 10
2. Vorbereitung ITC Proben
- Verdünnen Sie die Enzymlösung zu einer c-Wert von 64 mit einem Gesamtvolumen von 2 mL. (Im Falle von AAC (6 ')-II, das entspricht zu 192 pM.)
- Schnell Tauwetter der Ligand (AcCoA) Stammlösung in Eiswasser.
- Bereiten Sie 0,5 ml einer Lösung AcCoA 10-mal stärker konzentriert sich die Anzahl der Bindungsstellen des Proteins, in diesem Fall 4 mM, die durch Verdünnen 80 ul der AcCoA Stammlösung in 420 ul der Laufpuffer. Transfer in eine Pipette Füllrohr. Schnell wieder die AcCoA Stammlösung zu -78 ° C.
- Degas das Protein und Lösungen unter Vakuum für 5 min bei einer Temperatur 1 ° C unterhalb der gewünschten Betriebstemperatur (19 ° C).
3. Einrichten der Spritze 14
- Legen Sie die Injektionsspritze durch Spritzenhalter bis die vormontierten Spritzenklemme ist auf der gleichen Höhe wie der Inhaber.
- Vorschub der zweiten Spritzenklemme über die Injektionsspritze, bis es fest gegen den Boden der Spritze Halterung gedrückt. Leicht anziehen Klemme mit dem mitgelieferten 0,050 "Ball Point Hex Drive.
- Legen Spritze Halter in den Pipettenhalter.
- Schieben Sie die Pipette Injektor in die Injektionsspritze. Achten Sie darauf, den Kolben Spitze direkt in das Loch der Spritze gefüttert. Einmal vollständig eingeführt ist, schrauben Sie den Spannring der Spritze Halter in die Pipette Injektor.
4. Lädt die Probe Zelle 14
- Wash Probenzelle mit einem Minimum von 50 ml Laufpuffer und entfernen Sie alle verbleibenden Flüssigkeit unter Verwendung eines langnadligen 2,5 ml Glasspritze.
- Langsam ziehen ein Minimum von 1,8 ml Protein Probenlösung in den sauberen und trockenen langnadligen 2,5 ml Spritze. Achten Sie darauf, keine Blasen einzuführen.
- Vorsichtig Nadel in Probenzelle und sanft berühren den Boden der Zelle. Raise Spitze leicht (~ 1 mm) und leicht zu injizieren AAC (6 ')-II-Lösung in die Zelle, bis die überschüssige Flüssigkeit sichtbar ist über dem oberen Ende der Probe Zelle.
- Langsam heben Sie die Nadel etwa 1 cm und gleichzeitig Flüssigkeit bleibt in den Überlauf. Schnell zurückziehen und injizieren eine kleine Menge der Lösung (~ 0,25 ml) um eingeschlossene Luftblasen in der Probe Zelle zu entfernen.
- Entfernen Sie alle Lösungen Überlauf. Dies wird durch leichtes Verschieben der Nadel an der Seite der Überlauf in die Probenzelle erreicht. Die Spitze der Spritze wird eine Leiste getroffen, das ist der Wunsch Höhe für das laufende Lösung. Entfernen Sie alle Flüssigkeit, die oberhalb dieser Linie sitzt.
5. Loading Injektionsspritze und Einleitung laufen 14
- Befestigen Sie den Kunststoffschlauch loading Spritze in Einfüllöffnung.
- Lower Kolbenspitze an die Spitze der Einfüllöffnung.
- Legen Sie die AcCoA Lösung in die Pipette Füllrohr an der Unterseite des Pipettenhalter. Die Spitze der Spritze nicht berühren den Boden des Füllrohr.
- Langsam ziehen Sie die Lösung in die Spritze, bis eine kleine Menge in die Röhre des Be-Spritze.
- Schließen Sie die Einfüllöffnung durch eine Senkung der Kolbenspitze und klicken Sie auf die Schließen-Schaltfläche Port Fill. Purge und füllen, indem Sie auf die Purge-> Refill-Taste, 3-mal um die Luftblasen zu entfernen, dass kann eingeschlossen werden während des Ladens haben.
- Entfernen Sie den Füllschlauch vom Pipettenhalter und wischen Sie die Spitze der Spritze.
- Nehmen Pipette Montage und senken Spritze in die Messküvette. Gehen Sie langsam, wie die Spritze leicht verbiegen kann, so große Sorgfalt erforderlich ist. Sicherstellen, dass die Spritze vollständig durch Drücken auf Basis der Spannring eingesetzt.
- Stellen Sie die gewünschte Betriebstemperatur (in diesem Fall 20 ° C), und wählen Sie einen Hinweis Kraft, die etwas größer ist als der maximale Einspritzdruck Wärmestrom erwartet (in diesem Fall 20μcal/sec) ist.
- Programmieren Sie die gewünschte Injektionsvolumina und Verzögerungen. (In diesem Fall wurden 28 Injektionen eingesetzt. Die erste Injektion hatte ein Volumen von 2 ul mit 60 s Verzögerung. Alle nachfolgenden Injektionen hatten Volumina von 10 ul mit 330 s verzögert.)
6. Nachfolgende Durchläufe
- In allen nachfolgenden Läufen wiederholen Sie die Schritte 2-5, während abnehmende AAC (6 ')-II und AcCoA Konzentrationen mit einem Faktor von 2,4,8,16 und 32.
7. Data Analysis
- Verwenden Sie ein passendes Verfahren, das weltweit für alle der Isothermen zu einer einzigen Reihe von verbindlichen Parameter, wie zuvor beschrieben 10.
8. Repräsentative Ergebnisse
Repräsentative Daten sind in Abbildung 1 dargestellt. Die Formen der Isothermen sollte mit der Konzentration variieren. Schärfere Übergänge sind für höhere erwartete c-Werte (dh höhere Protein und Ligand-Konzentrationen) (Abbildung 2).
Im Falle von AAC (6 ')-Ii, gibt den beiden vor Ort sequenziellen Modell eine bessere Passform als eine Beschreibung zwei Sätze identisch, unabhängig Sites mit verstellbaren Stöchiometrien.
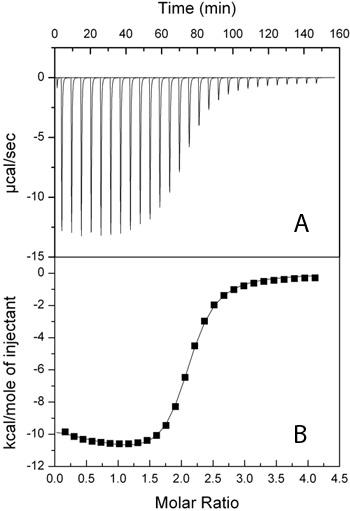
Abbildung 1. Isothermen durch Titration AcCoA (3,86 mM) in AAC (6 ')-II (192 nM) hergestellt. A) Raw ITC Spur. B) Integrierte Werte für die Ermittlung Bindungsparameter (Quadrate) mit einem 2-site sequentielle fit (verwendet -).

Abbildung 2. ITC Isothermen für AcCoA in AAC (6 ')-II in unterschiedlichen Konzentrationen titriert. Die experimentellen Daten (offene Kreise) wurden auf ein 2-sets-of-Sites unabhängigen Modell (gestrichelte magenta) und ein 2-site sequentielle Modell (durchgezogene blaue) passen. Die 2-site sequentional Modell eindeutig eine bessere allgemeine Einigung. Die eingesetzten Konzentrationen waren A) 6 uM, 0,25 mm, B) 12 uM, 0,25 mm, C) 24 uM, 0,5 mm, d) 48 uM, 1,0 mm, E) 96 uM, 1,9 mm, und F) 196 uM, 3,86 mm, für AAC (6 ')-II und AcCoA jeweils.
Diskussion
Diese analytische Teil der variablen c Montage wurde bereits im Detail 10 beschrieben. Hier berichten wir über die praktischen Aspekte des Sammelns variable-c-Datensätze für diesen Ansatz. Es ist wichtig, dass alle Protein und Ligand Proben aus dem gleichen Stammlösungen gezogen werden. Deshalb ist es wichtig, dass genügend Stammlösung wird zunächst die gesamte Reihe von Experimenten vollständig vorbereitet. Dadurch wird sichergestellt, das Verhältnis von AAC (6 ')-II und AcCoA ist bei allen Expe...
Offenlegungen
Danksagungen
Diese Arbeit wurde von den Canadian Institutes of Health Research (CIHR), National Science and Engineering Research Council (NSERC) und eine CIHR Ausbildungsförderung Stipendium (LF) unterstützt. Wir danken Prof. Gerard D. Wright (McMaster University, Kanada) für den AAC (6)-Ii Expressionsplasmid.
Materialien
| Name | Company | Catalog Number | Comments |
| Acetyl c–nzyme A (AcCoA) | Sigma-Aldrich | A2056 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Fisher Scientific | 7365-45-9 | |
| ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 431788 | |
| Spectra/Por 2 Dialysis Tubing | Spectrum Labs | 132678 | |
| Sterile Syringe Filter (0.2 μm) | VWR international | 281445-477 | |
| Cellulos Nitrate Membrane Filters (0.45 μm) | Whatman, GE Healthcare | 7184-004 | |
| VP-ITC | MicroCal | VP-ITC | Microcalorimeter used for measurements |
| ThermoVac | MicroCal | USB Thermo Vac | Temperature Controlled Degassing Station |
Referenzen
- Cliff, M. J., Ladbury, J. E. A survey of the year 2002 literature on applications of isothermal titration calorimetry. Journal of Molecular Recognition. 16, 383-391 (2003).
- Cliff, M. J., Gutierrez, A., Ladbury, J. E. A survey of the year 2003 literature on applications of isothermal titration calorimetry. Journal of Molecular Recognition. 17, 513-523 (2004).
- Ababou, A., Ladbury, J. E. Survey of the year 2004: literature on applications of isothermal titration calorimetry. Journal of Molecular Recognition. 19, 79-89 (2006).
- Ababou, A., Ladbury, J. E. Survey of the year 2005: literature on applications of isothermal titration calorimetry. Journal of Molecular Recognition. 20, 4-14 (2007).
- Okhrimenko, O. k. s. a. n. a., J, I. A survey of the year 2006 literature on applications of isothermal titration calorimetry. Journal of Molecular Recognition. 21, 1-19 (2008).
- Bjelic, S., Jelesarov, I. A survey of the year 2007 literature on applications of isothermal titration calorimetry. Journal of Molecular Recognition. 21, 289-312 (2008).
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Current Opinion in Structural Biology. 11, 560-566 (2001).
- Wiseman, T., Williston, S., Brandts, J. F., Lin, L. -. N. Rapid measurement of binding constants and heats of binding using a new titration calorimeter. Analytical Biochemistry. 179, 131-137 (1989).
- Capaldi, S. The X-Ray Structure of Zebrafish (Danio rerio) Ileal Bile Acid-Binding Protein Reveals the Presence of Binding Sites on the Surface of the Protein Molecule. Journal of Molecular Biology. 385, 99-116 (2009).
- Freiburger, L. A., Auclair, K., Mittermaier, A. K. Elucidating Protein Binding Mechanisms by Variable-c ITC. ChemBioChem. 10, 2871-2873 (2009).
- Wybenga-Groot, L. E., Draker, K. -. a., Wright, G. D., Berghuis, A. M. Crystal structure of an aminoglycoside 6'-N-acetyltransferase: defining the GCN5-related N-acetyltransferase superfamily fold. Structure. 7, 497-507 (1999).
- Draker, K., Northrop, D. B., Wright, G. D. Kinetic Mechanism of the GCN5-Related Chromosomal Aminoglycoside Acetyltransferase AAC(6')-Ii from Enterococcus faecium: Evidence of Dimer Subunit Cooperativity. Biochemistry. 42, 6565-6574 (2003).
- Wright, G. D., Ladak, P. Overexpression and characterization of the chromosomal aminoglycoside 6'-N-acetyltransferase from Enterococcus faecium. Antimicrob. Agents Chemother. 41, 956-960 (1997).
- MicroCal. . ITC Data Analysis in Origin. , (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten