Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Single Particle Electron Microscopy Rekonstruktion der Exosom Complex Mit der Random-konische Tilt-Methode
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt ein standardisiertes Verfahren, um eine dreidimensionale (3D) Rekonstruktion biologischer Makromoleküle mit Hilfe Negativfärbung Elektronenmikroskopie (EM) zu bekommen. In diesem Protokoll erklären wir, wie die 3D-Struktur des Saccharomyces cerevisiae Exosom Komplex in mittlerer Auflösung unter Verwendung des Random konische Neigung Rekonstruktionsverfahren (RCT) zu bekommen.
Zusammenfassung
Einzelne Partikel-Elektronenmikroskopie (EM) Rekonstruktion hat vor kurzem ein beliebtes Mittel, um die dreidimensionale (3D) Struktur von großen makromolekularen Komplexen erhalten werden. Im Vergleich zu Röntgenkristallographie, hat es einige einzigartige Vorteile. Erstens, einzelne Partikel EM Rekonstruktion nicht erforderlich, die Proteinprobe, die den Engpass in der Röntgenkristallographie ist kristallisieren, insbesondere bei großen makromolekularen Komplexen. Zweitens braucht es nicht große Mengen von Protein-Proben. Verglichen mit Milligramm Proteine, die für Kristallisation, einzelne Partikel EM Wiederaufbau braucht nur einige Mikroliter Proteinlösung bei nano-molaren Konzentrationen, wobei die negative Färbung EM-Methode. Doch trotz ein paar makromolekularen Anordnungen mit hoher Symmetrie, einzelne Partikel EM ist bei relativ niedrigen Auflösung begrenzt (weniger als 1 nm Auflösung) für viele Exemplare vor allem ohne Symmetrie. Diese Technik wird auch von der Größe der Moleküle unter-Studie beschränkt, dh 100 kDa für negativ gefärbten Proben und 300 kDa für frozen-hydratisierten Proben im Allgemeinen.
Für eine neue Probe mit unbekannter Struktur, verwenden wir grundsätzlich eine Heavy Metal-Lösung, um die Moleküle durch negative Färbung einbinden. Die Probe wird dann in einem Transmissionselektronenmikroskop untersucht, um zweidimensionale (2D) Aufnahmen der Moleküle statt. Idealerweise haben die Proteinmoleküle einer homogenen 3D-Struktur, sondern weisen unterschiedliche Orientierungen in den Aufnahmen. Diese Aufnahmen werden digitalisiert und verarbeitet in Computern als "single-Teilchen". Mit Hilfe von zweidimensionalen Ausrichtung und Klassifizierung Techniken, homogen sind Moleküle in die gleichen Ansichten in Klassen gruppiert. Ihre Mittel erhöhen das Signal des Moleküls 2D-Formen. Nachdem wir die Teilchen mit der richtigen relativen Orientierung (Euler-Winkel) zuordnen, können wir die 2D-Partikel-Bilder in einer virtuellen 3D-Volumen zu rekonstruieren.
In einzelnen Partikels 3D-Rekonstruktion, ist ein wesentlicher Schritt, um richtig zuordnen die richtige Ausrichtung der einzelnen Teilchen. Es gibt mehrere Methoden, um die Ansicht für jedes Teilchen zuordnen, einschließlich der eckigen Rekonstitution 1 und zufälligen konische Neigung (RCT) Methode 2. In diesem Protokoll, beschreiben wir unsere Praxis in immer der 3D-Rekonstruktion von Hefe Exosom Komplex mit negativen Färbung EM und RCT. Anzumerken ist, dass unser Protokoll der Elektronenmikroskopie und Bildverarbeitung das Grundprinzip der RCT folgt, ist aber nicht der einzige Weg, die Methode auszuführen. Wir beschreiben zunächst, wie die Protein-Probe in einer Schicht von Uranyl-Formate mit einer Dicke vergleichbar mit der Größe des Proteins eingebettet, mit einem löchrigen Kohlenstoff-Gitter mit einer Schicht durchgehende dünne Kohlenstoff-Folie abgedeckt. Dann wird die Probe in einem Transmissions-Elektronenmikroskop zu sammeln eingefügt ungekippten (0-Grad) und neigbar (55-Grad-) Paare von Aufnahmen, die später für die Verarbeitung und den Erhalt einer ersten 3D-Modell der Hefe Exosom verwendet werden. Zu diesem Zweck führen wir RCT und dann zu verfeinern den ersten 3D-Modell mit Hilfe der Projektion passenden Verfeinerung der Methode 3.
Protokoll
1. Prinzip des Zufalls Konische Tilt-Methode
- Das Prinzip des Zufalls konische Neigung Methode erfordert unter ein Paar Aufnahmen der gleichen Region der Probe im Elektronenmikroskop. Ein Bild der Probe in einem ungekippten Position (Abbildung 1A) genommen und das andere Bild ist von der Probe in einem Winkel zwischen 50 bis 70 Grad (in unserem Fall verwenden wir 55 Grad) geneigt gemacht. (Abbildung 1B)
- Mit dem Computer die digitalisierten Aufnahme Paar nebeneinander und Bilder aus dem gleichen Teilchen setzen gewählt werden. (Abbildung 1C)
- In dreidimensionalen Koordinaten werden die Bilder von ungekippten Partikel und ihre gekippt Partnern, die untereinander durch die Richtung der Kippachse und der Kippwinkel korreliert. (1D)
- Die Ausrichtung der ungekippten Teilchen Bilder bringt die Bilder der geneigte Partikel zu den entsprechenden azimutalen Standorten. (Abbildung 1E)
- Die Verwendung mehrerer Bilder gekippt Partikel Füllung der azimutalen Raum, kann die dreidimensionale Struktur des Moleküls rekonstruiert mit Hilfe eines Rückprojektion Algorithmus. (Abbildung 1F)
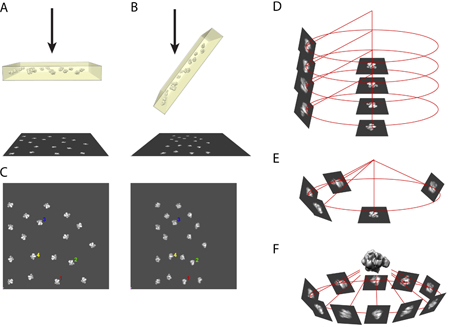
Abbildung 1. Ein Beispiel für das Prinzip für RCT Rekonstruktion.
2. Bereiten Sie die Grids von Holey Carbon mit dünnen Kohlenstoff-Covered
Begründung: Wir verwenden Negativfärbung Methode, um die Proteinprobe für zufällige konische Neigung Wiederaufbau zu beheben. Um die Makromoleküle, ohne zu viel Abflachung während der Trocknung zu erhalten, versuchen wir, die Proteinmoleküle in eine tiefe Fleck mit einer Dicke über die Dimension der Proteine 4 einbinden. In der Regel ist eine kontinuierliche Kohlenstoff bei der Herstellung negativ gefärbten Proben verwendet. Solche Art von Kohlenstoff, ist jedoch schwierig, den Fleck Dicke um das Protein zu steuern. Wir benutzen also hausgemacht löchrigen Netze Kohlenstoff mit einer dünnen Schicht von Kohlenstoff-Film (~ 5 nm Dicke) abgedeckt, um negativ gefärbten Proben zu machen. Kleine Vertiefungen durch die Löcher gebildet ermöglichen Beibehaltung der Protein-Lösung und die Färbelösung auf dem Gitter so dass es viel leichter ist, Protein in einem optimalen Fleck Dicke einbinden. Darüber hinaus reduziert die dünne Schicht von Kohlenstoff über dem Loch das Hintergrundrauschen stark.
- Bereiten Sie 0,5% Formvar Lösung. In der Abzugshaube, fügen 0,45 g Polyvinylformalharz und 90 ml Chloroform in einem 100 mL Becherglas. Verwenden Sie Alufolie in das Becherglas bedecken und mit einem kleinen Rührfisch zu helfen, löst formal Harz auf einem Magnetrührer. Es dauert etwa 15 Minuten, um das Harz auflösen.
- Während die Auflösung der Formvar, Dias sauber Mikroskopie Glas in Methanol und trocken wischen mit Kimwipes.
- Nach dem Formvar Harz ist voll in Chloroform gelöst, 1 mL 50% Glycerin an die Oberfläche der Lösung. Einstellen der Lautstärke des zugesetzten Glycerin wirkt sich auf die Dichte der Bohrungen auf der löchrigen Kohlenstoff. Tauchen Sie die Spitze eines Ultraschallgerät in der Lösung bei ungefähr 1 Zoll Tiefe und Nutzung maximaler Leistung bis 1 min beschallen zu Emulsion Glycerintröpfchen in Formvar Lösung zu machen. Die Lösung wird milchig, nachdem Sie diesen Schritt. Längere Beschallung Ursachen geringere Größe der Löcher auf dem löchrigen Kohlenstoff.
- Unmittelbar nach der Beschallung, tauchen die gereinigte Glas gleitet vertikal in die Emulsion für 1 Sekunde, nehmen Sie sie heraus, und tupfen Sie die Folien "Boden mit Filterpapier auf eine dünne Kunststofffolie über die Oberfläche der Folien zu bilden. Nach dem choloroform verdunstet, überprüfen Sie die Dichte und Größe der Löcher in der Folie unter einem Lichtmikroskop. Passen Sie die Vorbereitung Zustand entsprechend den Bedürfnissen. Mit der Bedingung hier beschriebene wir in der Regel erhalten Löcher mit einem Durchmesser von 3 bis 4 Mikrometer und 10 ~ 20 Löcher in jedes Quadrat von einem 400 Mesh Grid.
- Nach dem Glasplättchen trocken sind, schneiden Sie die Kante der Folie auf der Gleitfläche. Float die Folie auf der Oberfläche von destilliertem Wasser. Der dünne Film auf der Wasseroberfläche kann bei einem Glanzwinkel gegen die Lichtreflexion beobachtet werden. Ort 400-Mesh-Kupfer-Gitter auf dem Film eine nach der anderen, mit den Gittern "glatte Oberfläche nach unten zeigt.
- Nehmen Sie den Kunststoff-Folie mit Gitter auf ihn mit einem Stück Papier. Flip das Papier und lassen Sie ihn in eine Petrischale trocken. Weichen Sie das Papier in Methanol, um das restliche Glycerin in die Löcher zu entfernen und lassen Sie das Papier an der Luft trocknen.
- Coat die Netze mit einer Schicht aus Kohlenstoff mit einer Dicke von ca. 20 Nanometer im Carbon-Verdampfer. Die Dicke kann durch die graue Farbe des Kohlenstoffs bestimmt werden.
- Weichen Sie das Gitter mit Kohlenstoff in Chloroform eine halbe Stunde, um die Formvar entfernen. Nach den Gittern getrocknet sind, haben wir gewonnen hausgemachte löchrigen Kohlenstoff Gittern.
- Dampft dünne Schicht von Kohlenstoff mit etwa 5 Nanometer dick auf frisch gespaltenem Glimmer-Oberfläche.
- Vorsichtig in den löchrigen Kohlenstoff Gitter unter destilliertem Wasser. Float der dünnen Kohlenstoff aus der Glimmer-Oberfläche auf die Wasseroberfläche und Hinterlegung es auf den löchrigen KohlenstoffGitter langsam. Trocknen Sie die Netze in einem Abzug.
3. Negative Färbung der Exosom Complex
Begründung: Es gibt durchaus ein paar Heavy-Metal-Fleck-Lösungen, die für negative Färbung EM verwendet werden können, einschließlich Uranylacetat, Uranyl Formiat, Phosphorwolframsäure, Ammoniummolybdat, und andere. Verschiedene Färbelösung hat verschiedene einzigartige Eigenschaften. Zum Beispiel bietet Uranylacetat hohen Kontrast des Teilchens kann aber Protein-Komplexe, die nicht wie saures Milieu zum Absturz bringen. Für diejenigen Proben kann phosphotungestic Säure bei neutralen pH-Wert eine gute Färbelösung werden. Wir wählen gesättigten Uranyl Formate (UF)-Lösung durch ihre feine Granularität und hoher Durchdringungsfähigkeit in Molekülen.
- Boil destilliertem Wasser für 1 min. Cool it langsam auf Raumtemperatur. Dieser Schritt ist für gelösten Sauerstoff aus dem Wasser zu entfernen.
- Machen Sie frische 2% Uranyl Formate (UF)-Lösung. Mix 1 ml Wasser und 20 mg UF in ein 1,5 ml-Röhrchen. Vortex für 10 min.
- Der pH-Wert auf 5,0 durch Zugabe von 2 Mikroliter 10 M Kaliumhydroxid. Mix sofort. Die Lösung Farbe sollte mehr gelb. Der pH-Wert der Lösung sollte nicht zu hoch sein, da sonst die Färbung ausfällt.
- Setzen Sie das Rohr auf Vortexer für weitere 10 min.
- Drehen Sie die Lösung auf einem Desktop-Zentrifuge bei maximaler Geschwindigkeit für 10 min.
- Filter die Lösung durch ein 0,2 Mikrometer PVDF-Membran. Dies ist die frische UF Lösung. Decken Sie die Lösung Rohr in einem Stück Alufolie, um Licht zu verhindern. Die Lösung muss in der gleichen Tag verwendet werden.
- Glimmentladung eine dünne-Kohlenstoff-over-löchrigen Kohlenstoff Gitter mit einem Glimmentladung Gerät für 30 Sekunden bei 25 mA.
- Legen Sie ein Stück sauberer Parafilm auf der Bank. Put 3 Tröpfchen von 50 Mikroliter UF Färbelösung auf dem Parafilm.
- Verdünnen Sie die Exosom komplex, um eine Konzentration von 50 ~ 100 nm mit dem Verdünnungspuffer (25 mM Tris-HCl pH 7,5, 100 mM NaCl, 2 mM DTT). Setzen Sie 4 Mikroliter der verdünnten Protein auf der glow-entladen Netz. Lassen Sie die Probe auf das Netz für eine Minute zu bleiben. (Hinweis:. Solche eine endgültige Konzentration von Molekülen in der Regel ergibt eine optimale Dichte von gut dispergierten Teilchen auf der negativ gefärbten Gitter für Uranyl Formiat oder Uranyl acecate Färbelösung ist die Phosphat-oder hoher Salzkonzentration (mehr als 0,5 M) im Allgemeinen nicht gut für die immer gute Ergebnisse Färbung. Unsere Erfahrung zeigt, dass Hepes oder Pipes gut mit dem Uranyl-Fleck-Lösungen.)
- Verwenden Sie ein Stück Filterpapier, um die restliche Lösung von der Kante des Gitters Blot und Flip das Netz sofort auf den Fleck Tropfen und spülen Sie das Netz für etwa 10 Sekunden auf jedem Tröpfchen.
- Nach dem letzten Spülgang, lassen Sie den Fleck zu bleiben in der Startaufstellung noch 1 min und dann blot die entfernt Flecken von einem Stück Filterpapier. Halten Sie eine dünne Schicht von Flecken Lösung auf dem Grid Oberfläche, um einen guten tiefen Fleck Ergebnisse zu erhalten. Lassen Sie das Gitter trocknen schnell in einem Abzug.
4. Elektronenmikroskopie der Exosom Complex
Begründung: Jede Transmissionselektronenmikroskop mit einem Kippen der Bühne lässt sich kippen Paare der Probe für RCT Rekonstruktion zu sammeln. In der Theorie können die höheren Winkel der Probe gekippt werden, um Daten, desto besser einzuziehen. In der Praxis aufgrund der Konstruktion des Probenhalters und der Geometrie des Rasters ist die maximale bedienbar Winkel von 50 bis 70 Grad beschränkt. In diesem Protokoll, wir beschreiben nur unser Verfahren mit einem FEI Tecnai-12-Elektronenmikroskop. Für die anderen Modelle von Mikroskopen, müssen die Operationen entsprechend der Anforderung des Projekts und Eigentum des Gerätes eingestellt werden.
- Legen Sie die Probe Gitter in den Probenhalter und setzen Sie dann den Halter in ein FEI Tecnai -12 Elektronenmikroskop. Das Mikroskop ist bei 120 kV betrieben. Wir verwenden Gatan Ultrascan4000 CCD-Kamera, um Fotos zu machen. Stellen Sie sicher, dass die "Flip um die vertikale Achse 'im' config-Kamera"-Dialog von Digital Micrograph Schnittstelle deaktiviert, um die richtige Händigkeit Bestimmung zu gewährleisten. (Hinweis:. Das ist wichtig vor allem, wenn der Leser setzt auf SPIDER RCT Rekonstruktion Verfahren zu einem 3D-Modell zu erhalten)
- Überprüfen Sie die Probe Raster bei geringer Vergrößerung zu den besten Plätzen gefärbt finden. Solche Art von Plätzen sollten ein Dutzend Löcher mit Abmessung von etwa 1 bis 2 Mikrometer und dunklen Fleck Bereiche in ihnen. (Abbildung 2)

Abbildung 2. Ein geringer Vergrößerung Aufnahme eines Gitters zeigt Quadrate mit guten und schlechten Flecken. - Schalten Sie den niedrigen Dosis-Modus des FEI User Interface und richten Sie die Suche, Fokus und setzen Position in niedriger Dosis-Modus. Wir verwenden Vergrößerungen von 150.000 für Fokus, 52.000 für die Belichtung und 1,5 Meter Länge Kamera in Beugung zu suchen. Die Belichtungszeit wird auf 1 Sekunde eingestellt. Stellen Sie den Fokus Position 2 Mikrometer weg von der Exposition position entlang der Kippachse.
- Finden Sie die Löcher mit guter Färbung in den Suchmodus, und speichern Sie die Standorte. Die gute Löcher haben in der Regel dunkler Fleck Gradienten in ihnen beobachteten unter den Suchmodus. Werfen Sie einen CCD Bild des Platzes. Kippen Sie die Probe auf 55 Grad, nehmen Sie ein anderes Bild. Vergleichen Sie die beiden Bilder, identifizieren die gepaart Löcher in den beiden Aufnahmen. (Abbildung 3)
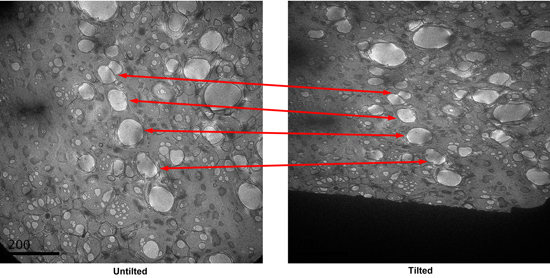
Abbildung 3. Eine Neigung Paar Aufnahmen von einem Platz in den Suchmodus. Entsprechende gepaart Löcher sind angegeben. - Tilt die Bühne zurück, um 0 Grad. Mit der low-dose-Kit, um Bilder von jedem Loch in den Suchmodus bei hoher Vergrößerung identifiziert nehmen. Die Unschärfe verwendet wird ca. -0,7 Mikrometer.
- Nachdem alle Löcher Bilder gemacht haben, neigen Sie die Bühne, um 55 Grad. Nehmen Sie Aufnahmen von der gekippten Probe bei gleicher Vergrößerung wie ungekippten diejenigen mit einer Unschärfe von ca. -1,2 Mikrometer. Identifizieren Sie die entsprechenden gekippt Paar Aufnahmen auf die Muster in der niedrigen Vergrößerung Aufnahmen basiert. Die unregelmäßige Muster der hausgemachten löchrigen Kohlenstoff Gitter hilft die Korrelation. Untersuchen Sie neigen Paare von Aufnahmen und entfernen Sie die Aufnahmen mit schlechten Flecken wie seicht gefärbten Bereichen (Partikel scheinen einen Heiligenschein um sich herum unter gekippten Zustand haben). (Abbildung 4)
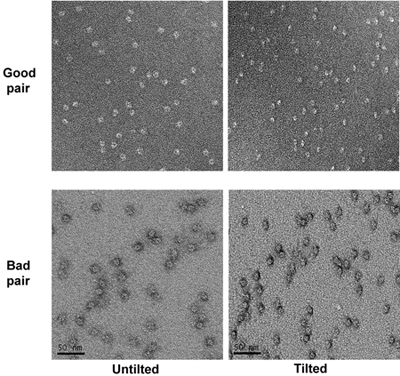
Abbildung 4. Zwei Tilt Paar Aufnahmen der Probe bei hoher Vergrößerung. Die guten und schlechten Aufnahmen sind markiert.
5. Bildverarbeitung der Daten
Begründung: Es gibt verschiedene Optionen und Softwarepakete für die RCT Rekonstruktion im Computer durchzuführen. Die am häufigsten verwendet wird SPIDER 5. Eine grundlegende Protokoll zur RCT in SPIDER ausführen können in der Web-Seite gefunden werden http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Ein detailliertes Protokoll zur RCT in SPIDER führen in dem Artikel wird von Shaikh et al. 6 In unserem Protokoll, verwenden wir eine Kombination aus IMAGIC-5 7 und SPIDER in der Video-Version des Protokolls. Wir bieten auch ein alternatives Verfahren zur Verwendung ausschließlich SPIDER in die Text-Version des Protokolls.
- Setup-Programme. Wir nutzen die proc2d in der EMAN 8-Gehäuse, um das Bild-Format von Gatan Digitale Bild SPIDER-Bildformat zu ändern. IMAGIC-5 wird verwendet, um die 2D-Ausrichtung zu tun. SPIDER wird verwendet, um die 3D-Rekonstruktion und Verfeinerung zu tun.
Abs. 1: Picking neigen Paare der Partikel.
- Konvertieren Sie die *. dm3 Gatan Digitale Bild SPIDER-Bildformat mit dem proc2d Befehl in EMAN. Die gekippt und ungekippten Paare sind in Muster wie *** t.spi und *** u.spi benannt, bzw..
- Wählen Sie die Teilchenpaaren mit "WEB"-Programm in SPIDER Programm-Paket verteilt. Folgen Sie den Anweisungen in der Die Koordinaten sind automatisch in dcu ***. spi und dct ***. Spi für ungekippten geneigt und Partikel bzw. gespeichert. Ein weiteres Dokument dcb ***. spi enthält die Neigungswinkel von Informationen zwischen gekippt und ungekippten Partikel (. Hinweis: Die drei Winkel bei der Anprobe in WEB zwischen der Neigung Paaren keine Garantie für die korrekte Händigkeit der endgültigen Rekonstruktion, weil theta nicht die Zeichen (positiver Wert) und phi-und Gamma verlassen sich auf die ursprünglichen Werte gesetzt zu ihnen. Untersuchen der Richtung der Unschärfe-Gradienten auf die gekippte Aufnahme würde helfen, die richtige Startwerte der phi-und Gamma vor der Montage, um eine korrekte bekommen Händigkeit des Wiederaufbaus. Abbildung 5 zeigt die richtige Konvention.)
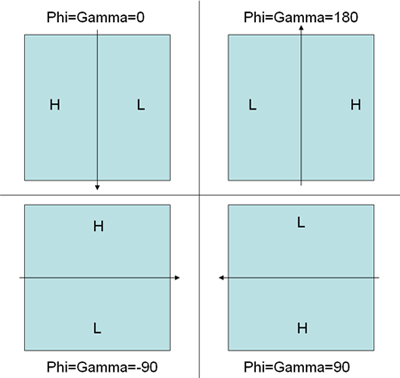
Abbildung 5. Eine Illustration zu dem Übereinkommen vom Winkel Bestimmung in WEB Kipp-pair Partikel Kommissionierung Montage-Modus durch die Unschärfe-Gradienten bestimmt erklären. H steht für High-Unschärfe-Bereich, während L steht für Low-Unschärfe-Bereich. Der Pfeil stellt Kippachse. Die entsprechenden korrekten anfänglichen Winkel für PHI und GAMMA sind für jedes System angegeben. - Box aus allen abgeholt Partikel mit einer modifizierten Version des SPIDER-Skript als in der Webseite gezeigt http://www.wadsworth.org/spider_doc/spider/docs/partpick.html . Script speichert die ungekippten geneigt und Partikel-Stacks als u.spi und t.spi. Die Zahl der Aufnahme, wo die Partikel aus sollte in particle_list.spi gespeichert werden. (Hinweis: Dies ist sehr wichtig für die Erzeugung des korrekten Eulerwinkel-Dateien fürRCT).
Abs. 2: Zweidimensionale Ausrichtung und Klassifizierung der ungekippten Teilchen Bilder.
- Konvertieren Sie die ungekippten Partikel in IMAGIC-5-Format mit em2em Programm in Imagic-5-Gehäuse. Richten und Klassifizierung der Teilchen in homogenen Klassen iterativ mit dem IMAGIC-5-Programme (Anhang A). Mit der msa-Namen-in-class-Befehl in IMAGIC-5 zu generieren Klassen Lookup-Tabelle der Teilchen, die wir als imagic_classes.lis speichern. (Hinweis:. Die Klassifizierung und Ausrichtung ist es, die Zahl der Teilchen mit der gleichen Form zu erhöhen sowie eine Reduzierung der Variation in einer Klasse Variance Karte jede Klasse kann Informationen über die Qualität der Klasse.)
- Generieren Sie einen Plot-Datei (ali_50.plt in der Video-Demonstration) für Translation und Rotation Werte der Ausrichtung der einzelnen Teilchen mit header-Befehl in IMAGIC-5.
- Konvertieren Sie die Klassen Look-Up-Tabelle in SPIDER Dokumentdateien base_file ***. spi mit einem Perl-Skript lis2spi.pl in verteilten http://cryoem.berkeley.edu .
- Konvertieren Sie die Translation und Rotation Werte der Ausrichtung der einzelnen Teilchen aus dem Plot-Datei für Translation und Rotation Werte in Schritt 5,6 in SPIDER Dokumentdatei ali_50.spi mithilfe eines Skripts plt2spi.pl in verteilten generiert http://cryoem.berkeley.edu .
Wir verwenden IMAGIC-5 für die 2D-Alignment und Klassifizierung, weil es die beste Leistung bei diesem Job gibt in unseren Händen. Alternative Strategie in SPIDER für die zweidimensionale Ausrichtung und Klassifizierung finden Sie unter http://www.wadsworth.org/spider_doc/spider/docs/align.html . Wir haben auch SPIDER verwendet werden, um die 2D-Analyse auf der ungekippten Teilchen des Exosom durchzuführen. Es folgt ein einfaches Verfahren.
Alternative 5,5) Verwenden Sie die Referenz-free Ausrichtung wie in der beschriebenen Richten Sie die Bilder. Zwei einfache Skripte gefunden werden können Speichern Sie die Rotation und Verschiebung aller Teilchen in einem Dokument Datei angular_file.spi.
Alternative 5,6) Klassifizieren Sie die ausgerichteten Partikel in Gruppen mit der gleichen Ansicht wie in Wir haben K-means-Methode für die Klassifizierung verwendet. Generieren base_file ***. spi über die Einstufung basiert.
Abs. 3: Dreidimensionale Rekonstruktion mit dem gekippten Teilchen Bilder.
- Band-Pass-Filter, Masken-und Mittelpunkt der gekippten Partikel ebenso wie für die ungekippten Partikel in IMAGIC-5. Generieren Sie einen neuen Datensatz für den Titel Partikel. (Hinweis: Dieser Schritt ist optional in SPIDER getan werden kann..)
- Erstellen anglular Dokument-Dateien aus den Klassen look-up table Dokumente ali_50.spi, die DCB ***. spi-Dateien von Web in Schritt 5.3 und die Partikel-List-Datei partile_list.spi aus Schritt 5.4 mit der SPIDER-Skript, wie in Anhang B. generiert
- Verwenden Sie den Wiederaufbau Skripte in SPIDER für den Wiederaufbau der einzelnen Klassen zu tun wie in http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Jede Klasse von Teilchen trägt zu einer 3D-Rekonstruktion Volumen. Die 3D-Modelle können in UCSF-Chimera 9 untersucht werden. Eine zweidimensionale Projektion des 3D-Modells bei Euler-Winkel (0,0,0) zu den entsprechenden 2D-Klasse Durchschnitt aus den ungekippten Partikel verglichen werden, um die Qualität der Rekonstruktion zu überprüfen. Finden Sie ähnliche Volumina, ausrichten und verschmelzen sie zu ersten Bänden nach dem Verfahren erzeugen http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html .
Abs. 4: Verfeinerung der 3D-Rekonstruktion mit ungekippten Teilchen Bilder.
- Do Projektion passenden Verfeinerung der fusionierten ursprünglichen Volumens gegen alle ungekippten Partikel auf eine höhere Auflösung zu bekommen volume ohne fehlende Kegel und Abflachung Artefakt mit SPIDER-Skripte wie in http://www.wadsworth.org/spider_doc/spider/docs/techs/recon/refine.html .
6. Repräsentative Ergebnisse:
Mit dem RCT-Methode haben wir etwa 50 Rekonstruktionen der Exosom von insgesamt 5.000 kippen Paaren (Abbildung 6) erhalten. Von den 50 3D-Modelle können wir verschiedene Orientierungen des Komplexes sitzt auf dem Gitter mit hauptsächlich zwei orthogonalen Ansichten zu sehen. Eine Abflachung Artefakt ist auch in vielen der Bände in der Richtung senkrecht zu den Carbon-Oberfläche nachweisbar. Wir führten Ausrichtung und Zusammenführung der 3D-Volumen mit zwei ersten Bände in orthogonalen Ansichten zu generieren. Mit den 5.000 ungekippten Teilchen Bilder, wir haben die gleichen endgültigen 3D-Rekonstruktion der Exosom bei etwa 18 Angström-Auflösung erhalten von beiden ersten Modelle (Abbildung 7). Die Struktur offenbart die Architektur der Hefe Exosom und Einblicke auf die RNA-Substrat Rekrutierung Weg 10.

Abbildung 6. 50 3D-Modelle der Exosom Komplex durch RCT Rekonstruktion.
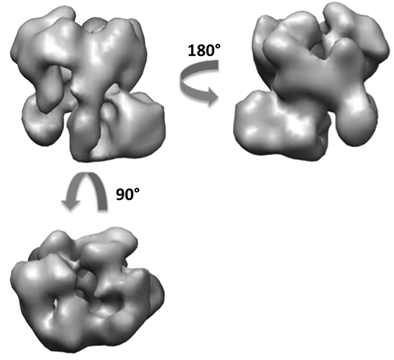
Abbildung 7. 3D-Rekonstruktion der Exosom Komplex nach Verfeinerung.
Anhang:
Anhang A. Die Script-Datei für 2D-Alignment und Klassifizierung in IMAGIC-5.
Datei: auto_align_i.sh
Klicken Sie hier für Datei
Anhang B. Die Script-Datei zur Erzeugung eckig-Datei für 3D-Rekonstruktion in SPIDER.
Datei: generate_angular_file.spi
Klicken Sie hier für Datei
Diskussion
In diesem Artikel präsentieren wir ein detailliertes Protokoll der Probenvorbereitung und dreidimensionale Rekonstruktion des Exosom Komplex mit negativen Färbung Elektronenmikroskopie. Mit dieser Methode erhalten wir die 3D-Rekonstruktion mit zufälligen konische Neigung Methode ohne vorherige Kenntnis der Struktur. Zufällige konische Neigung Methode erfordert nicht unbedingt eine homogene Probe, aber die folgenden Projektion passenden Verfeinerung Schritt wäre eine homogene Probe braucht, um eine hohe Auflösung z...
Offenlegungen
Danksagungen
Die Autoren bedanken sich bei den Mitgliedern des Nogales Labor an der UC-Berkeley danken an der Erstellung der ersten Protokolle und die Mitglieder des Wang-Labor an der Yale University in ihre Hilfe, um die volle Protokolle zu etablieren. Wir erkennen auch die Mitarbeiter in Kryo-EM-Anlage und High-Performance Computation-Center an der Yale School of Medicine für ihre Unterstützung. HW ist ein Smith Family Preisträger.
Materialien
| Name | Company | Catalog Number | Comments |
| Polyvinyl Formal Resin | Electron Microscopy Sciences | 63450-15-7 | |
| Uranyl Formate | Electron Microscopy Sciences | 22451 | |
| Superfrost Microscope Slides | Thermo Fisher Scientific, Inc. | 4951F-001 | |
| 400 mesh grid regular | SPI Supplies | 3040C | |
| Carbon coater Auto 306 | Edwards Lifesciences | ||
| Tecnai-12 Electron Microscope | FEI | ||
| Glow Discharger | BAL-TEC | Sputter Coater SCD 005 |
Referenzen
- van Heel, M. Angular reconstitution: a posteriori assignment of projection directions for 3D reconstruction. Ultramicroscopy. 21, 111-123 (1987).
- Radermacher, M. Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J Electron Microsc Tech. 9, 359-394 (1988).
- Penczek, P. A., Grassucci, R. A., Frank, J. The ribosome at improved resolution: new techniques for merging and orientation refinement in 3D cryo-electron microscopy of biological particles. Ultramicroscopy. 53, 251-270 (1994).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative staining and image classification - powerful tools in modern electron microscopy. Biol Proced Online. 6, 23-34 (2004).
- Frank, J., Radermacher, M., Penczek, P., Zhu, J., Li, Y., Ladjadj, M., Leith, A. SPIDER and WEB: processing and visualization of images in 3D electron microscopy and related fields. J Struct Biol. 116, 190-199 (1996).
- Shaikh, T. R., Gao, H., Baxter, W. T., Asturias, F. J., Boisset, N., Leith, A., Frank, J. SPIDER image processing for single particle reconstruction of biological macromolecules from electron micrographs. Nat Protoc. 3, 1941-1974 (2008).
- Heel, M. v. a. n., Harauz, G., Orlova, E. V., Schmidt, R., Schatz, M. A new generation of the IMAGIC image processing system. J Struct Biol. 116, 17-24 (1996).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: semiautomated software for high-resolution single-particle reconstructions. J Struct Biol. 128, 82-97 (1999).
- Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C., Ferrin, T. E. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25, 1605-1612 (2004).
- Wang, H. W., Wang, J., Ding, F., Callahan, K., Bratkowski, M. A., Buttler, J. S., Nogales, E., Ke, A. Architecture of the yeast Rrp44 exosome complex suggests routes of RNA recruitment for 3' end processing. Proc Natl Acad Sci USA. 104, 16844-16849 (2007).
- Scheres, S. H., Nunez-Ramirez, R., Sorzano, C. O., Carazo, J. M., Marabini, R. Image processing for electron microscopy single-particle analysis using Xmipp. Nat Protoc. 3, 977-990 (2008).
- Yoshioka, C., Pulokas, J., Fellmann, D., Potter, C. S., Milligan, R. A., Carragher, B. Automation of random conical tilt and orthogonal tilt data collection using feature-based correlation. J Struct Biol. 159, 335-346 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten