Method Article
Assoziierte Chromosome-Trap zur Identifizierung weiträumige DNA-Wechselwirkungen
In diesem Artikel
Zusammenfassung
Die damit verbundenen Chromosom Trap (ACT)-Assay ist ein neuartiges unvoreingenommene Verfahren zur Identifizierung von Langstrecken-DNA-Wechselwirkungen. Die Charakterisierung der Long Range DNA-Wechselwirkungen wird es uns ermöglichen, die Beziehung der nuklearen Architektur, um die Genexpression zu bestimmen, sowohl die normale Physiologie und in krankhaften Zuständen.
Zusammenfassung
Genetische Informationen, die von DNA codiert wird, in einer komplexen und stark regulierten Chromatin-Struktur organisiert. Jedes Chromosom befindet sich in einem bestimmten Gebiet, die nach Stadium der Entwicklung oder Zellzyklus ändern kann. Genexpression kann in spezialisierten transkriptionelle Fabriken dort auftreten, wo Chromatin Segmente Schleife aus verschiedenen Chromosomen Territorien, die zu Co-Lokalisation von DNA-Segmenten, die auf unterschiedlichen Chromosomen oder weit voneinander entfernt auf demselben Chromosom vorhanden sein können. The Associated Chromosome Trap (ACT)-Assay bietet eine effektive Methode, um diese Langstrecken-DNA Verbände in einem unvoreingenommenen Weise durch die Erweiterung und Änderung des Chromosoms Konformation Capture-Technik zu identifizieren. Der ACT-Test macht es uns möglich, Mechanismen der Regulation der Transkription in trans zu untersuchen, und können bei der Erläuterung der Beziehung der nuklearen Architektur, die Genexpression in der normalen Physiologie und während Krankheitszuständen.
Protokoll
1. Formaldehydfixierung von Langstrecken-Chromatin-Wechselwirkungen
- Kultur humanen Zelllinie HL-60 in RPMI1640 Medium mit 15% FBS und 1 x Penicillin / Streptomycin auf 80-90% Konfluenz in einem Inkubator mit 5% CO 2 bei 37 ° C geliefert
- Sammeln Sie die Zellen in einem 50-ml Nunc Röhrchen, Zentrifuge bei 1.200 Umdrehungen pro Minute für 15 Minuten, und entfernen Sie dann das Medium durch Absaugen
- 5 ml Kulturmedium auf die Zellpellets resuspendiert, und zählen Sie die Zellen mit Hilfe einer Zählkammer. Nehmen Sie ca. 1 x 10 7 Zellen auf ein Volumen von 40 ml mit RPMI1640 / 10% FBS, dann fügen Sie 1,7 ml 37% Formaldehyd zu der verdünnten Chromatin zu beheben.
- Inkubieren bei Raumtemperatur für 10 Minuten unter leichtem Schütteln und dann löschen mit 2,4 ml 2 M Glycin. Zentrifuge für 15 Minuten bei 1.200 rpm bei 4 ° C, und entfernen Sie den Überstand. Waschen des Pellets einmal mit 40 ml eiskaltem PBS, dann drehen Sie den Pellets und entfernen Sie die PBS.
2. Zelllyse zu Kernen isolieren
- Die Zellen in 40 ml eiskaltem Lysepuffer (10 mM Tris-HCl, pH 8,0, 10 mM NaCl, 0,2% NP-40) mit frisch hinzu Protease-Inhibitoren (1:500 Verdünnung) und 0,1 mM PMSF. Inkubieren in einem kalten Raum mit Rotation für 90 Minuten, Zentrifuge bei 2.500 rpm für 15 Minuten, und entfernen Sie den Überstand.
3. Restriktionsenzymverdau mit Bgl II
- Resuspendieren der Kerne in 0,5 ml 1 x NEB Puffer 3 und fügen Sie 15 μlof 10% SDS. Bei 37 ° C für 1 Stunde unter Schütteln, dann fügen Sie 45 ul von 20% Triton X-100 auf die SDS zu binden. Bei 37 ° C für 1 Stunde unter Schütteln.
- Verwenden Sie ein Aliquot von 1 x 10 6 Kerne (ca. 15 pg, ein Zehntel der ursprünglichen Zellen) für Restriktionsenzymverdau. Nehmen Sie 55 ul der Kerne Lösung aus Schritt 3.1 und machen bis zu 500 ul mit 433 ul 1 x NEB Puffer 3 und 12 ul der Bgl II (50U/ul). Bei 37 ° C über Nacht.
4. Ligation von interagierenden DNA-Segmenten
- Inaktivierung des Restriktionsenzyms, indem 95 ul 10% SDS und denaturieren durch Erhitzen bei 65 ° C für 20 Minuten in einem Wasserbad.
- 7 ml 1 x Ligation Puffer (30 mM Tris-HCl, pH 8,0, 10 mM MgCl 2, 10 mM DTT, 1 mM ATP) und 360 ul 20% Triton X-100 und bei 37 ° C für 1 Stunde .
- Senken Sie die Temperatur auf 16 ° C und 50 ul von 400 U / ul T4 DNA-Ligase. Inkubieren Sie die Probe bei 16 ° C für 4 Stunden und dann bei Raumtemperatur für 30 Minuten.
5. DNA Purification
- Add 300 ug Proteinase K, und inkubieren Sie bei 65 ° C über Nacht.
- Add 5 ug RNase A und Inkubation bei 37 ° C für 30 Minuten
- Purify DNA durch Phenol / Chloroform-Extraktion und Fällung DNA in Isopropanol. Lösen DNA in 150 ul sterilem destilliertem Wasser.
6. Aufschluss mit Msp I und Ligation mit Oligonukleotid-Linker
- Inkubieren 2μg der gereinigten DNA mit 5 Einheiten Msp I bei 37 ° C für 4-6 Stunden. Inactivate Msp I bei 65 ° C für 10 Minuten, dann fallen die DNA in Ethanol mit 1 ul von 5 mg / ml Glykogen. Lösen Sie die DNA-Pellet in 50 ul sterilem destilliertem Wasser.
- Mix 50 ul Msp I-behandelter DNA mit 2 ul einer 20 uM Linker-Oligonukleotid L (5'-gctgaccctgaattcgcacgtgcctgtcgttagcggacacagggcgattcac-3 '), 1 ul einer 20 uM Oligonukleotid S (5'-cggtgaatc-3'), 1 ul sterilem destilliertem Wasser und 6 ul 10 x T4 DNA-Ligase-Puffer. Decken Sie die Mischung mit flüssigem Wachs.
- Denaturieren Oligonukleotide bei 50 ° C für 1 Minute und abkühlen lassen schrittweise auf 10 ° C in einer 0,5 ° C / Minute Gradienten in einem Thermocycler.
- Add 1 ul 400 U / ul T4 DNA-Ligase und Inkubieren bei 15 ° C über Nacht. Purify der Linker-DNA ligiert mit einem QIAquick PCR Purification Kit und eluieren in 50 ul sterilem destilliertem Wasser.
7. PCR-Amplifikation und Sequenzanalyse
- Wählen Sie ein Primer für die spezifische DNA-Region, die Sie für langreichweitige Wechselwirkungen (dh der "Köder") untersuchen möchten. In diesem Beispiel verwenden wir ABL-1.
- Nehmen Sie 1 ul der gereinigten DNA in die erste Runde PCR mit 1 ul 20 uM ABL -1 spezifischen Primer # 4656 (5'-gttcaagcgattctcctgcctcga-3 '), 1 ul 20 uM Linker spezifischen Primer # 2963 (durchführen 5'- gctgaccctgaattcgcacgtgcct-3 '), 3μl von 3 x Klen Taq DNA Polymerase I Cocktail und 3 ul sterilem destilliertem Wasser. Zwei ul von 32 P-dCTP ist zu 100 ul 3 x Klen Taq DNA Polymerase I Cocktail vor dem PCR-Amplifikation aufgenommen. Die thermischen Zyklus für einen heißen Start-PCR liegt bei 72 ° C für 2 min, 95 ° Cfor 2 Minuten, 18 Zyklen von 95 ° C für 20 Sekunden, 65 ° C für 40 Sekunden und 72 ° C für 1 Minute, die letzte Verlängerung ist bei 72 ° Cfür 5 Minuten.
- Purify der ersten Runde PCR-Produkten mit einem QIAquick PCR Purification Kit, und eluieren DNA in 30 ul sterilem destilliertem Wasser.
- Nehmen Sie 1 ul 100 x verdünnt erste PCR-Produkt zu einem zweiten PCR-Runde mit verschachtelten Primer durch Zugabe von 1 ul 20 uM ABL-1-spezifischen Primer # 4626 (5'-ggagaatttttatctgcctctgtga-3 ') (Abbildung 1a), 1 ul durchführen von 20 pM des Linkers spezifischen Primer # 2961 (5'-gtcgttagcggacacagggcgattc-3 '), 3 ul 3 x Klen Taq DNA Polymerase I Cocktail und 3 ul sterilem destilliertem Wasser. Der Temperaturwechsel Zeitplan ist 25 Zyklen von 95 ° C für 20 Sekunden, 67 ° C für 40 Sekunden und 72 ° C für 1 Minute, nach Verlängerung bei 72 ° C, gefolgt von 5 Minuten.
- Visualisieren Sie die PCR-Produkte, indem Sie einen 5% Urea-PAGE-Gel und das Scannen der exponierten Bildschirm in einem Phosphoimager (Abb. 1b). Jeder PCR-Bande aus dem Gel durch Auflösen des Gels Streifen in ein Eppendorf-Röhrchen mit 60 ul sterilem destilliertem Wasser und Inkubation bei 95 ° C für 5 Minuten recycelt werden. Kurz zentrifugieren bei 10.000 rpm für 10 sec, um alle Proben zu sammeln. Nehmen Sie 1 ul als die DNA-Vorlage verwenden, um PCR mit dem Primerpaar 2961/4626 unter den gleichen Bedingungen wie oben beschrieben durchführen. Die Sequenzanalyse können nach der Reinigung mit einem QIAquick PCR Purification Kit durchgeführt werden.
- DNA-Sequenzen werden mit Hilfe eines Online-Tools, um ihre chromosomale Lage auf der Website zu ermitteln: http://genome.ucsc.edu (Abbildung 1c). Klicken Sie auf "BLAT 'ein neues Fenster, das eine DNA-Sequenz einfügen können eingegeben haben, erscheint ein neues Fenster nach dem Klicken auf' Senden 'und zeigt die" BLAT Search Results'. Klicken Sie auf "Browser" der Hit mit 100% Identität zu einer nächsten Fenster geben Sie den Speicherort der DNA-Sequenz zu zeigen.
8. Repräsentative Ergebnisse
1. ACT-Assay mit ABL-1 Region als Köder, um seiner großen Reichweite DNA Wechselwirkungen bestimmen
Wie in Abbildung 1a, zwei Bgl II-und einer BamHI site dargestellt wurden für die ACT-Test ausgewählt. In der zweiten Runde der PCR, Primer-Set 4626/2961 verwendet wurde, um ABL-M1 zu verstärken, wurde 4630/2961 für ABL-M2 verwendet, und 4636/2961 war für die ABL-M3 verwendet. Ein typisches Gel Muster zeigt eine bis mehrere Banden (Abb. 1b). Jedes Fragment aus einem ACT-Test besteht aus zwei kombinierten DNA-Abschnitte: ein Segment aus dem Köder DNA-Region stammen, während das zweite Segment kommt aus den assoziierten Partner, der den Köder Region Segment durch die erste Restriktionsenzym-Erkennungssequenz verbunden war. Das zweite Enzym Erkennungssequenz wird am Ende der zugehörigen Partner-Sequenz (Abbildung 1c) erscheinen. Das geklonte ABL-M1-Fragment enthält DNA aus der Region auf Chromosom 9q32.4 ABL1 aus +133,592,306-133,592,399 befindet, und die damit verbundenen Partner ist auf Chromosom 3p13 aus +71,869,882-71,870,107 entfernt. Die Identitäten der beteiligten Partner sind durch Strahlen ihre Sequenzen mit der UCSC Genom-Browser (GRCh7/hg19 im Februar veröffentlicht, 2009) entdeckt. PROK2 als assoziierter Partner an der chr3p13 Locus identifiziert wurde.
Ebenso wurde das ABL-M2 assoziierter Partner zu chr5q21.1 lokalisiert, während Klon ABL-M3 als intra-chromosomale Verein in der Nähe des ABL-1-Locus identifiziert wurde.
2. Bestimmen weniger verbreitet langreichweitige Wechselwirkungen mit ACT-Test
Andere Tests auf Basis 3C haben viele weitere Interaktionspartner als dass wir in Abbildung 1 und in der Igf2 / H19-Locus 12 gezeigt gemeldet. Die Methode, die wir skizziert haben wählt die am weitesten verbreitete langreichweitige Wechselwirkungen. Doch durch die Erhöhung der Anzahl der PCR-Zyklen, ist es möglich, zusätzliche, weniger häufige Assoziationen zu identifizieren sowie (Abbildung 2).
3. Unterschiede in langreichweitige Wechselwirkungen in Krebszellen zu normalen Geweben verglichen.
Der ACT-Test kann auch verwendet werden, um Unterschiede in der nuklearen Architektur und langreichweitige Wechselwirkungen zwischen normalen und Krebszellen (Abbildung 3) zu identifizieren. Diese Unterschiede können reflektieren nuklearen Architektur Änderungen, die während Zelltransformation auftreten, und somit diesen Test kann letztlich gelten für diagnostische Zwecke. Die ähnliche Gel Muster, die in normalen und Krebsgewebe auftreten angedeutet, dass die ACT-Test zuverlässig und wiederholbar ist. Während die ACT-Assay identifizieren können Long Range DNA-Partner, weitere Analysen, wie zB Fisch-und Chip-Assays sind erforderlich, um die Anwesenheit der identifizierten Verbindungen zwischen entfernten Loci zu überprüfen. Genetische, physiologische und biochemische Untersuchungen können dann durchgeführt werden, um die biologischen Auswirkungen dieser langen Strecke DNA Verbänden aufzuklären.
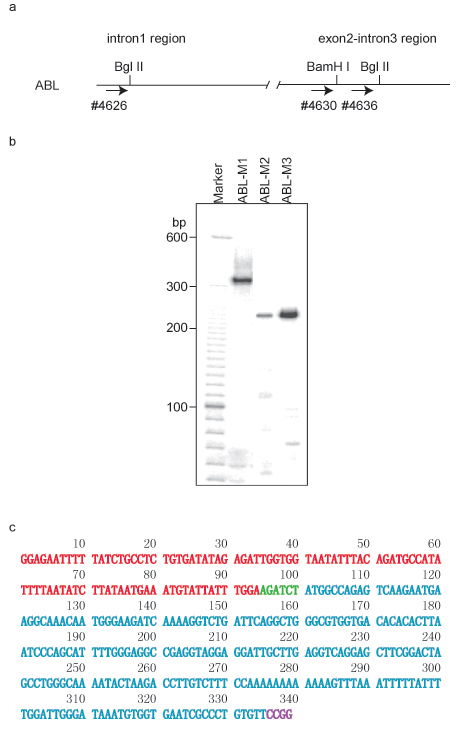
Abbildung 1 ACT-Assay mit ABL-1 Region als Köder DNA in HL-60 Zellen. a. DNA-Strukturure in ABL-1 Region. Die erste Restriktionsenzym in ACT-Assay verwendet wurde Bgl II. Die Primer für die zweite Runde der PCR werden auch durch die Pfeile und die entsprechenden Primer-Nummern gekennzeichnet. b. Gel Muster der ACT-Test in 5% Harnstoff-PAGE. Primerpaar 4626/2961 zur Verstärkung Klon ABL-M1 in der nested PCR verwendet wurde, wurde 4630/2961 für Klon ABL-M2 verwendet, und 4636/2961 war für Klon ABL-M3 verwendet. c. DNA-Sequenz von Klon ABL-M1. DNA-Fragment aus ABL-1 Köder DNA ist rot markiert, und die damit verbundene DNA-Partner ist in den Farben Cyan bezeichnet. Bgl II-Stelle (AGACTC) wurde in grün markiert, und Msp I-Stelle (CCGG) ist in lila markiert.

Abbildung 2 PCR-Zyklen beeinflussen ACT Testergebnisse. Mit dem Imprinting Kontrollregion (ICR) in der Igf2/H19 locus als Köder DNA in Maus-Fibroblasten-Zellen wurden verschiedene Rad-Programme in der ersten und zweiten Runde der PCR in der ACT-Test angewendet. 18-20 Zyklen in der ersten Runde der PCR nicht verstärken genug Signal visualisiert werden. Fünfundzwanzig Zyklen in der zweiten PCR-Ergebnisse in deutliche Banden, bei gleichzeitiger Erhöhung der Anzahl der Zyklen für die zweite Runde der PCR induzierte ein Abstrich Muster neben mehr Bands.

Abbildung 3: Nachweis von Unterschieden in der nuklearen Architektur zwischen normalen Kolon-und Darmkrebs Gewebe in ACT-Test. A. DNA-Struktur des ICR Region IGF2/H19 Locus und IGF2 Gen. Dpn II Standorte für ACT-Assay sind beschriftet. Primer in der zweiten Runde der PCR verwendet werden durch Pfeile und Zahlen in verschiedenen Farben, die auf jeder Bahn in Panel b der Figur entsprechen gekennzeichnet. b. Gel Muster der ACT-Test in 5% Harnstoff-PAGE. Normale Dickdarmgewebe MAD03-1423-und Darmkrebs Gewebe MAD04-149 wurde von Cooperative Human Tissue Network (CHTN) Western Division erhalten. Nach jedem Doppelpunkt Gewebe wurde homogenisiert, war ACT-Test nach dem hier beschriebenen Verfahren durchgeführt. Spur 1 stellt die PCR-Ergebnisse mit Primerpaar # 2961and # 4161 (5'-tctgcgccatcagggcagtgagac-3 ') in pink in Panel ein markiertes, Spur 2 stellt die PCR-Ergebnisse mit Primerpaar # 2961 und # 4163 (5'-gccgcgcggccacttccgattcc-3 ') in orange in Panel ein markiertes, Spur 3 zeigt die PCR-Ergebnisse mit Primerpaar # 2961 und # 5145 (5'-gccatgcaggtaggatttgagctg-3') in blau in Panel ein markiertes, Spur 4 ist die PCR-Ergebnisse mit Primerpaar # 2961 und # 5151 (5'-gtctcaaataggggccagctagcttgg-3 ') in grün in Panel a. beschriftet Einzigartige Bands, die nur in normalen Kolon sind durch gelbe Pfeile gekennzeichnet sind, und jenen Bands, die nur in Darmkrebsgewebe erschienen sind in roten Pfeilen gekennzeichnet. ICR, Imprinting Kontrollregion; DMR, verschiedene methylierte Region.
Diskussion
Dekker et al. Entwickelte das Chromosom Konformation erfassen Ansatzes (3C), um die Häufigkeit der Interaktion zwischen zwei genomischen Loci 1 zu erfassen, und 3C wurde ausgiebig auf intra-chromosomale und inter-chromosomal Assoziationen zwischen zwei bekannten DNA-Regionen in Säugetier-Zellen 2 zu untersuchen verwendet -9. Obwohl neu entwickelten Hallo-C Methodik zur Untersuchung der genomweiten DNA Verein aufgebracht werden können, ist ACT-Test noch eine effektive Technik zur Untersuchung von Locus-spezifischen DNA-Interaktion 10-11. Wir haben diesen Ansatz modifiziert werden, um unbekannte DNA Regionen, die mit einer bekannten DNA-Region in kultivierten Maus und humanen Zellen (Abbildung 1) verbunden sind, zu identifizieren. Wir nannten diese Methode der damit verbundenen Chromosom Trap (ACT)-Assay, da es uns eine verlässliche und reproduzierbare Methode, um neue unbekannte DNA-Partnern, die mit einem bekannten Ziel-DNA Bereich 12 Mitarbeiter zu identifizieren. Eine erfolgreiche 3C-Assay mit geeigneten Kontrollen wird vor der Ausführung der neuartigen Aspekte der ACT-Test 13 durchgeführt. Um möglichst viele zugehörige DNA-Regionen wie möglich tofind, ist es notwendig, verschiedene Kombinationen der ersten und zweiten Restriktionsenzyme verwenden. Es ist besonders wichtig, Restriktionsenzyme, die unempfindlich gegen CpG-Methylierung auf den ersten 3C Ligationsschritt durchführen verwenden. Protein-Bindung und DNA-Methylierung können auch Einfluss Restriktionsenzymverdau Effizienz und kann zum Ausfall der Ligation der damit verbundenen DNA-Bereiche für bestimmte Restriktionsenzym Aufschlüsse führen. Das Auftreten von intra-oder inter-chromosomal Ligation ist abhängig von Protein-DNA-Vernetzung und entsprechende physische Karten von beiden der zugehörigen DNA-Regionen. So sind einige Vorversuche notwendig, um eine praktikable wirksame Formaldehyd-Konzentration und Behandlungsdauer in der ACT-Tests erstellen. Eine Reihe von Endkonzentrationen von Formaldehyd (from1.5% bis 2%) wurden in mehreren Labors wurde während der 3C Teil des Tests 7,9 verwendet. Alternativ können die Oligonukleotide als Linker verwendet entworfen, um den Zusammenhalt Ende durch den zweiten Restriktionsenzym geschnitten angepasst werden. Obwohl wir festgestellt, dass 18-20 Zyklen in der ersten Runde der PCR und 20-25 Zyklen in der zweiten Runde der PCR konnten deutliche Banden liefern, ist es notwendig, die beste PCR-Bedingungen für jedes Experiment (Abbildung 2) zu etablieren. Intra-Molekül Glühen zwischen 5'-Ende und 3'-Ende komplementär Adapter Sequenz eines DNA-Stranges kann in PCR auftreten, hemmt es Adapter-spezifischen Primer mit den DNA-Molekül, und führte zu viel niedrigeren Amplifikationseffizienz in der ersten mehreren Zyklen. Nachdem die Ziel-DNA für die Zyklen amplifiziert wurde, kann ihre Höhe sehr viel größer sein als diese unspezifischen Reaktionen und kann der Konkurrenz der Primer an die Target-DNA-Moleküle zu erleichtern. Dies gilt auch, warum wir Hintergrundamplifikation sehen kann, und warum die erste Runde PCR-Produkt muss verdünnt Hintergrund amplification.It zu verringern ist wichtig, um überschüssige Primer aus der PCR-Reaktion zu entfernen, um den Hintergrund in die zweite Runde der PCR verringern. Wie in allen PCR-Experimenten, ist es wichtig, Primer, die nicht in Repeat-Sequenz Regionen, die die Mehrheit der Menschen und der Maus DNA bilden befinden Design. Obwohl neu entwickelten Hallo-C Methodik zur Untersuchung der genomweiten DNA Verein aufgebracht werden können, ist ACT-Test noch eine effektive Technik zur Untersuchung von Locus-spezifischen DNA-Interaktion.
Offenlegungen
Danksagungen
Wir bedanken uns Adelle Murell und Wolf Reik sehr viel für die gemeinsame Nutzung ihrer 3C-Protokoll. Diese Arbeit wurde vom Department of Defense und dem Research Service des Department of Veterans Affairs unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| RPMI1640 medium | Invitrogen | 22400-105 | |
| acrylamide | Invitrogen | 15512-023 | |
| ATP solution, 10mM | Invitrogen | AM8110G | |
| fetal bovine serum | Invitrogen | 16000-044 | |
| penicillin-streptomycin | Invitrogen | 15140-122 | |
| 1M Tris pH8.0 | Invitrogen | AM9856 | |
| RNase A | Invitrogen | 12091-039 | |
| SDS | Invitrogen | 15525-017 | |
| urea | Invitrogen | 15505-035 | |
| BamH I | New England Biolabs | R0136T | |
| Bgl II | New England Biolabs | R0144M | |
| Dpn II | New England Biolabs | R0543T | |
| Msp I | New England Biolabs | R0106S | |
| dNTPs | New England Biolabs | N0447L | |
| proteinase K | New England Biolabs | P8102S | |
| T4 DNA ligase | New England Biolabs | M0202T | |
| 37% formaldehyde | Sigma-Aldrich | F8775 | |
| Bis-acrylamide | Sigma-Aldrich | 146072 | |
| dithiothreitol | Sigma-Aldrich | 43815 | |
| glycine | Sigma-Aldrich | 50046 | |
| PMSF | Sigma-Aldrich | 93482 | |
| proteinase inhibitor | Sigma-Aldrich | S8830 | |
| Nonidet P-40 | Roche Group | 11754599001 | |
| KlenTaq1 | Ab peptides | 1001 | |
| dCTP alpha P32 | PerkinElmer, Inc. | BLU513H250UC | |
| PTC-100 Thermal Cycler | MJ Research | mjptc100 | |
| Power Supply | Bio-Rad | 164-5056 | |
| OmniPAGE Maxi | Aurogene Life Science | VS20D | |
| Typhoon 9400 | GE Healthcare | 63-0055-78 |
Referenzen
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295, 1306-1311 (2002).
- Apostolou, E., Thanos, D. Virus Infection Induces NF-kappaB-dependent interchromosomal associations mediating monoallelic IFN-beta gene expression. Cell. 134, 85-96 (2008).
- Duan, Z. A three-dimensional model of the yeast genome. Nature. 465, 363-367 (2010).
- Lomvardas, S. Interchromosomal interactions and olfactory receptor choice. Cell. 126, 403-413 (2006).
- Murrell, A., Heeson, S., Reik, W. Interaction between differentially methylated regions partitions the imprinted genes Igf2 and H19 into parent-specific chromatin loops. Nat Genet. 36, 889-893 (2004).
- Schoenfelder, S. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. Nat Genet. 42, 53-61 (2010).
- Spilianakis, C. G., Lalioti, M. D., Town, T., Lee, G. R., Flavell, R. A. Interchromosomal associations between alternatively expressed loci. Nature. 435, 637-645 (2005).
- Vakoc, C. R. Proximity among distant regulatory elements at the beta-globin locus requires GATA-1 and FOG-1. Mol Cell. 17, 453-462 (2005).
- Xu, N., Tsai, C. L., Lee, J. T. Transient homologous chromosome pairing marks the onset of X inactivation. Science. 311, 1149-1152 (2006).
- Lieberman-Aiden, E. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Berkum, N. L. v. a. n. Hi-C: a method to study the three-dimensional architecture of genomes. J Vis Exp. , (2010).
- Ling, J. Q. CTCF mediates interchromosomal colocalization between Igf2/H19 and Wsb1/Nf1. Science. 312, 269-272 (2006).
- Dekker, J. The three 'C's of chromosome conformation capture: controls, controls, controls. Nat Methods. 3, 17-21 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten