Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
High Throughput Screening von Fungal Endoglucanaseaktivität in Escherichia coli
In diesem Artikel
Zusammenfassung
Wir beschreiben eine niedrige Kosten, hohe Durchsatz-Verfahren zum Screening von Pilzen Endoglucanaseaktivität in E. coli. Die Methode beruht auf einer einfachen visuellen Ablesung der Substratabbau, erfordert keine Enzymreinigung und ist hoch skalierbar. Dies ermöglicht das schnelle Screening von großen Bibliotheken der Enzym-Varianten.
Zusammenfassung
Cellulase-Enzyme (Endoglucanasen Cellobiohydrolasen und β-Glucosidasen) hydrolysieren Zellulose in Zucker-Komponente, die wiederum in Kraftstoff Alkohole 1 umgewandelt werden kann. Das Potenzial für die enzymatische Hydrolyse von Zellulose-Biomasse zu erneuerbarer Energie zu versorgen hat sich bemüht, Cellulasen für sparsamen Produktion 2 Ingenieur intensiviert. Von besonderem Interesse sind Pilzcellulasen 3-8, wobei die bereits industriell für Lebensmittel und Textilien verarbeitet wird.
Kennzeichnung der aktiven Varianten unter einer Bibliothek von mutierten Cellulasen ist entscheidend für den Engineering-Prozess, aktive Mutanten können weitere für die verbesserten Eigenschaften und / getestet werden oder eine zusätzliche Belastung von Mutagenese. Effizientes Engineering von Pilzcellulasen hat durch einen Mangel an genetischer Werkzeuge für die einheimische Organismen und durch die Schwierigkeiten bei der Expression der Enzyme in heterologen Wirten behindert. Kürzlich entwickelte Morikawa und Mitarbeiter ein Verfahren zur Expression in E. coli der katalytischen Domäne von Endoglucanasen von H. jecorina 3,9, ein wichtiges Industriezentrum Pilz mit der Fähigkeit, Cellulasen in großen Mengen ausscheiden. Functional E. coli Expression wurde auch für Cellulasen aus anderen Pilzen, einschließlich Macrophomina phaseolina 10 und Phanerochaete chrysosporium 11-12 berichtet worden.
Wir stellen eine Methode zur Hochdurchsatz-Screening von Pilzen Endoglucanaseaktivität in E. coli. (Abb. 1) Diese Methode verwendet das gemeinsame mikrobielle Farbstoff Kongorot (CR) auf den enzymatischen Abbau der Carboxymethylcellulose (CMC) von wachsenden Zellen auf festem Medium zu visualisieren. Der Aktivitätstest erfordert kostengünstige Reagenzien, minimale Manipulation, und gibt eindeutige Ergebnisse als Zonen des Abbaus ("Halos") in der Kolonie vor Ort. Obwohl ein quantitatives Maß für enzymatische Aktivität kann mit dieser Methode nicht ermittelt werden, haben wir festgestellt, dass Halo Größe mit insgesamt enzymatische Aktivität in der Zelle korreliert. Die weitere Charakterisierung der einzelnen positiven Klone bestimmt, relative Protein Fitness.
Traditionelle bakterielle ganze Zelle CMC / CR Aktivitätstests 13 beinhalten Gießen Agar mit CMC auf Kolonien, die Gegenstand einer Kreuzkontamination oder Inkubation Kulturen in CMC-Agar Brunnen, die weniger für großflächige Experimente ist. Hier berichten wir über ein verbessertes Protokoll, dass die bestehenden Waschverfahren 14 für Cellulase-Aktivität verändert: Zellen, die auf CMC-Agarplatten gezüchtet werden vor CR Färbung entfernt. Unser Protokoll reduziert Kreuzkontamination und ist hoch skalierbar, so dass das schnelle Screening von tausenden von Klonen. Neben H. jecorina Enzyme, haben wir zum Ausdruck gebracht und gesiebt Endoglucanase Varianten aus dem Thermoascus aurantiacus und Penicillium decumbens (siehe Abbildung 2), was darauf hindeutet, dass dieses Protokoll für Enzyme, die aus einer Reihe von Organismen.
Protokoll
1. Abschirmblech Vorbereitung
- Add 25g LB Nährmedium, 15g Agar und 1,5 g Carboxymethylcellulose (CMC) Substrat 1L destilliertes Wasser, und Autoklaven zu sterilisieren.
- Nach dem Autoklavieren können mittel-bis kühl, und fügen Sie geeignete Antibiotika und IPTG zu einer Endkonzentration von 100 &mgr;.
- Gießen Sie 200 ml Medium in je 5 große quadratische Petrischalen / Bioassay Tabletts (240 x 240 x 20mm oder größer). Lassen Platten vollständig trocknen.
2. Endoglucanase Bibliothek Generation und Bildschirm
- Generieren Sie eine Bibliothek von Endoglucanase katalytische Domäne (n). Dies kann in vielerlei Hinsicht erreicht werden (z. B., site-directed oder zufällige Mutagenese, Rekombination, etc.) und ist durch den Forscher bestimmt.
- Klon der Bibliothek in einen Vektor, so dass sie unter der Kontrolle des T7-Promotors mit dem lac-Operator sind, und enthalten eine pelB Signalsequenz für die Ausrichtung in das Periplasma. Ein Beispiel für einen geeigneten Vektor ist pET22b (+) von Novagen.
- Transformieren Sie die Endoglucanase Bibliothek in E. coli-Stamm BL21 (DE3) und Platte für einzelne Kolonien.
- Inkubieren Transformation über Nacht bei 37 ° C.
- Mit sterilen Zahnstochern, impfen Klone in 96-Well Deep-Well-Platten mit 400μL LB-Medium mit entsprechenden Antibiotika ergänzt. Bei 37 ° C über Nacht unter Schütteln bei 250 Umdrehungen pro Minute.
- Add steriles Glycerin auf 96 gut Nacht-Kulturen bis zu einer Endkonzentration von 10% für die langfristige Lagerung bei -80 ° C. Dies wird die Master-Platte sein.
- Mit einem 96-poligen Stempel, replizieren Platte über Nacht Kulturen auf die Screening-Platten mit IPTG und CMC. Bis zu 5 Sets von Übernacht-Kulturen können auf jeder Vorführung Typenschild geprägt werden.
- Beschriften Sie die Screening-Platten, so dass die Identität und Orientierung der einzelnen 96-Well-Platte bekannt ist.
- Inkubieren Sie die Stempel-Bibliotheken bei Raumtemperatur (17-25 ° C) über Nacht oder bis sichtbare Kolonien gebildet haben.
- Spülen Platte mit destilliertem Wasser, bis alle Spuren von den Kolonien entfernt werden.
- Gießen Kongorot (CR; 0,5% in Wasser) auf gewaschene Platte. Achten Sie darauf, gerade genug, CR hinzufügen, um die Platte zu decken.
- Inkubieren 15 Minuten bei Raumtemperatur. Nicht mehr als 15 Minuten für CR Inkubation. Längere Inkubationszeiten wird in weniger deutliche Halos führen.
- Abgießen CR und fügen Sie eine großzügige Menge (mehr als genug, um die Platte cover) von 1M NaCl.
- Inkubieren 15 Minuten bei Raumtemperatur.
- Abgießen NaCl zu CMC Abbau Halos zu offenbaren. Für eine bessere Auflösung des Halos kann längere Inkubationszeiten mit NaCl oder mehrere NaCl wäscht erforderlich sein.
- Mit einem sterilen Zahnstocher holen aktive Enzyme Klone aus der Master-Platte für die weitere Charakterisierung.
3. Repräsentative Ergebnisse:
Ein Beispiel für die Anzeige für diesen hohen Durchsatz-Bildschirm ist in Abbildung 3 dargestellt. Klone können als inaktiv, schwach aktiv, der hochwirksam und von der Größe des Abbaus Zonen identifiziert werden. Dies ist besonders nützlich, wenn ein Enzym mit bekannter Aktivität in der Bibliothek als Referenz enthalten ist. Wie hier gezeigt, ist die Identifizierung von aktiven Enzymen robust und reproduzierbar. Aber bitte beachten Sie, dass eine angemessene Kennzeichnung der Screening-Platten extrem wichtig ist. Der Forscher muss sich aktiv Varianten nach dem Waschen den Kolonien weg zu identifizieren, um sie von der Master-Platte bei -80 ° C für die weitere Charakterisierung gespeichert holen. Schließlich ist es schwierig zu beurteilen, a priori die Beziehung zwischen Aktivitäten auf künstliche vs natürliche Substrate. Deshalb müssen die Forscher testen alle positiven Klone auf industriell relevante Material Protein Fitness zu bestimmen.
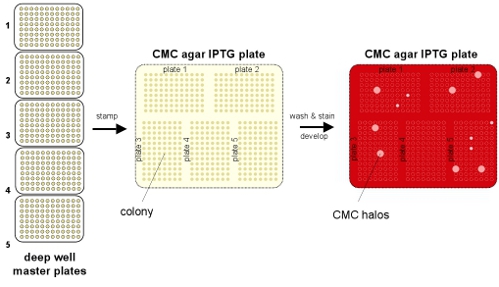
Abbildung 1. Schematische Darstellung der Screening-Verfahren. Die Zellen sind mit einer Endoglucanase-Bibliothek transformiert und selektiv für einzelne Kolonien überzogen. Klone gepickt und über Nacht gezüchtet in 96-Well Deep Well Platten für (1) Lagerung bei -80 ° C und (2) Replika-Plattierung auf Platten mit CMC und IPTG zu Endoglucanase Expression zu induzieren. Aktive Klone werden von der Master-Platte bei -80 ° C gelagert abgeholt

Abbildung 2. Klonierung und Expression von Pilz-katalytischen Domänen in E. coli. (A) Endoglucanase Expressionsvektor. Gene wurden in pET22b (+) (Novagen) unter der Kontrolle des T7-Promotor kloniert. (B) Rechts: Endoglucanase-exprimierenden BL21 (DE3) Kolonien. Linkes Bild: CMC Abbau Halos nach dem Waschen und Entwicklung mit Kongorot.
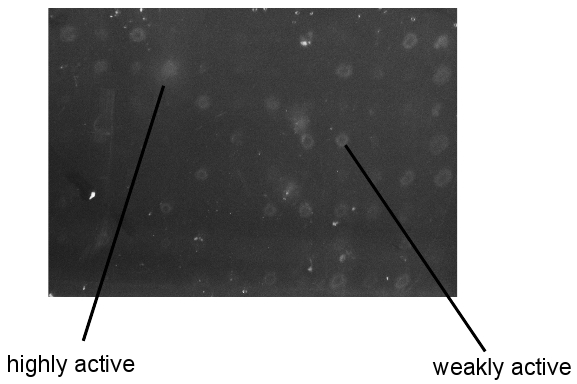
Abbildung 3. Die Ergebnisse der Proben aus CMC / Kongorot Bildschirm. Bilder von Screening-Platten nach dem Waschen und Kongorot Entwicklung genommen. Alle Kolonien, die auf screening Platten wurden von ähnlicher Größe.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die hier beschriebene Protokoll ermöglicht eine schnelle und hohen Durchsatz Identifizierung von aktiven Enzymen, mit minimaler Manipulation. Bewegungsmelder ist ziemlich empfindlich und qualitativ spiegelt das Ausmaß der Aktivität innerhalb der Zelle. Seine einfache Bedienung macht diese Methode eignet sich für eine breite Palette von Enzym-Bibliotheken, die nur durch funktionelle Expression in E. begrenzt coli. Darüber hinaus ist Aktivitätsscreening dieser Art nicht zu Pilzcellulasen beschränk...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde von der Gordon and Betty Moore Foundation, und durch die UNCF / Merck Science Initiative finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagens | Firma | Produkt-Nummer | |
|---|---|---|---|
| IPTG | Sigma | I1284 | |
| Kongorot | Sigma | C6277 | |
| Carboxymethylcellulose | Sigma | 360384 | |
| NaCl | Sigma | S1679 | |
| Bacto-Agar | BD Biosciences | 214030 | |
| Bioassay-Platten | Thermo Scientific | 240845 |
Referenzen
- Atsumi, S., Hanai, T., Liao, J. C. Non-fermentative pathways for synthesis of branched-chain higher alcohols as biofuels. Nature. 451, 86-89 (2008).
- Wilson, D. B. Cellulases and biofuels. Curr Opin Biotechnol. 20, 295-299 (2009).
- Qin, Y. Engineering endoglucanase II from Trichoderma reesei to improve the catalytic efficiency at a higher pH optimum. J Biotechnol. 135, 190-195 (2008).
- Nakazawa, H. Directed evolution of endoglucanase III (Cel12A) from Trichoderma reesei. Appl Microbiol Biotechnol. 83, 649-657 (2009).
- Heinzelman, P. A family of thermostable fungal cellulases created by structure-guided recombination. Proc Natl Acad Sci. 106, 5610-5615 (2009).
- Heinzelman, P. SCHEMA recombination of a fungal cellulase uncovers a single mutation that contributes markedly to stability. J Biol Chem. 284, 26229-26233 (2009).
- Lantz, S. E. Hypocrea jecorina CEL6A protein engineering. Biotechnol Biofuels. 8, 3-20 (2010).
- Mahadevan, S. A. Site-directed mutagenesis and CBM engineering of Cel5A (Thermotoga maritima). FEMS Microbiol Lett. 287, 205-211 (2008).
- Nakazawa, H. Characterization of the catalytic domains of Trichoderma reesei endoglucanase I, II, and III, expressed in Escherichia coli. Appl Microbiol Biotechnol. 81, 681-689 (2008).
- Wang, H., Jones, R. W. Properties of the Macrophomina phaseolina endoglucanase (EGL 1) gene product in bacterial and yeast expression systems. Appl Biochem Biotechnol. 81, 153-160 (1999).
- Howard, R. L. Enzyme activity of a Phanerochaete chrysosporium cellobiohydrolase (CBHI.1) expressed as a heterologous protein from Escherichia coli. Afr J Biotechnol. 2, 296-300 (2003).
- Howard, Characterisation of a chimeric Phanerochaete chrysosporium cellobiohydrolase expressed from Escherichia coli. Afr J Biotechnol. 3, 349-352 (2004).
- Teather, R. M., Wood, P. J. Use of Congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen. Appl Env Microbiol. 43, 777-780 (1982).
- Gilkes, N. R. Mode of action and substrate specificities of cellulases from cloned bacterial genes. J Bio Chem. 259, 10455-10459 (1984).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten