Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verstärkung und Quantifizierung von HIV-1 RNA in HIV-infizierten Personen mit Viruslast unter der Nachweisgrenze von Standard Clinical Assays
In diesem Artikel
Zusammenfassung
Die Quantifizierung der HIV-1 RNA im Plasma und Sequenzierung einzelner HIV-1 Genome von Individuen mit einer Viruslast unterhalb der Nachweisgrenze (50-75 Kopien / ml) ist schwierig. Hier beschreiben wir, wie zu extrahieren und zu quantifizieren, Plasma-Virus-RNA mit Hilfe eines Echtzeit-PCR-Assay, der zuverlässig misst HIV-1 RNA bis zu 0,3 Kopien / ml und wie man virale Genome von einzelnen Genomsequenzierung verstärken, von Proben mit sehr niedrigen Viruslast.
Zusammenfassung
Amplifikation viraler Gene und Quantifizierung von HIV-1 RNA in HIV-1 infizierten Patienten mit einer Viruslast unterhalb der Nachweisgrenze von Standard-Tests (unter 50-75 Kopien / ml) ist notwendig, um einen Einblick zu viralen Dynamik und Virus Wirt-Interaktionen zu gewinnen bei Patienten, die natürlich Kontrolle der Infektion und diejenigen, die auf einer antiretroviralen Kombinationstherapie (cART) sind.
Hier beschreiben wir, wie man virale Genome von einzelnen Genoms (die SGS-Protokoll) 13, 19 und wie genau quantifizieren HIV-1 RNA bei Patienten mit niedrigen Viruslast (die Single-Copy-Test (SCA)-Protokoll) 12, 20 zu verstärken.
Die Single-copy-Assay ist ein real-time PCR-Assay mit Empfindlichkeit in Abhängigkeit vom Volumen des Plasmas untersucht. Wenn ein einzelner Virus-Genom in 7 ml Plasma erkannt wird, dann ist die RNA-Kopienzahl gemeldet zu 0,3 Kopien / ml liegen. Der Test hat ein internes Kontrollsystem Tests für die Effizienz der RNA-Extraktion und Steuerungen für möglich Amplifikation aus DNA oder Verunreinigung. Patientenproben sind in dreifacher Ausfertigung gemessen.
Die Single-Genom-Sequenzierung Assay (SGS), mittlerweile weit verbreitet und als nicht arbeitsintensiv 3, 7, 12, 14, 15, ist ein limitierender Verdünnung Assay, in dem Endpunkt verdünnten cDNA Produkt über einen 96-Loch verteilt Platte. Laut einer Poisson-Verteilung, wenn weniger als 1 / 3 der Brunnen Produkt geben, gibt es eine 80% ige Chance, das PCR-Produkt wird resultierenden Verstärkung von einer einzigen cDNA-Molekül. SGS hat den Vorteil, über das Klonen von nicht zu Resampling unterzogen und nicht durch PCR eingeführt Rekombination 19 vorgespannt. Allerdings hängen die Verstärkung Erfolg von SCA und SGS auf Primer-Design. Beide Tests wurden für HIV-1 Subtyp B entwickelt, kann aber für andere Subtypen und anderen Regionen des Genoms, indem Primer, Sonden und Standards angepasst werden.
Protokoll
1. Extraktion von RNA aus große Mengen an Plasma-
Eine Übersicht über die einzelne Kopie Assay (SCA) und die einzigen Genoms (SGS)-Protokoll sind in den Abbildungen 1 und 2 vorgesehen.
- Um 7 ml Plasma, Spin ca. 14 ml Blut in 15 ml EDTA (nicht Heparin) Sammelröhrchen bei 2.600 xg für 15 Minuten ohne zu bremsen. Pipette Plasma (achten Sie darauf, die Buffy-Coat-Schicht zu vermeiden) in 15 ml Tuben.
- Wenn RNA wird für die einzelne Kopie Assay (SCA), spike das Plasma mit 30.000 Kopien des Rous Sarcoma Virus internen Virion-Steuerung (RCAS) extrahiert. Die RCAS Virus ist vor der Durchführung SCA wie zuvor beschrieben (20) vorbereitet. Kurz gesagt, ist RCAS Virus aus Zellkulturüberstand durch RT-Aktivität quantifiziert, verdünnt, um eine endgültige Konzentration von 30.000 Virionen pro 200 ul, in RPMI Gewebekultur-Medien mit 5% FBS verdünnt und bei -80 ° C in einzelnen Aliquots. Die RCAS Virus sollte nicht gefroren / werden vor in der Assay Gebrauch aufgetaut. RCAS in den Patienten-Plasma für die Effizienz der RNA-Extraktion und für die Richtigkeit der PCR Quantifizierung Kontrolle versetzt. : Weitere Informationen über RCAS kann unter folgendem Link gefunden werden http://home.ncifcrf.gov/hivdrp/RCAS/ .
- "Pre-spin"-Plasma, in 15 ml konische Röhrchen, bei 2.600 xg für 15 Minuten zu trennen keine Lipide und Zelltrümmer, die genaue RNA-Quantifizierung stören können.
- Label Seton Easy-Seal Röhrchen mit einem Permanent-Marker zur Probe Nummer und der Ort, wo die Pellets im Rohr Formular zu identifizieren. Nach der Vor-Spin, Transfer-Plasma zu einem Seton Easy-Seal Röhrchen durch Pipettieren des Plasmas und zur Vermeidung jeglicher Zelltrümmer und / oder Lipide an der Oberfläche. Notieren Sie sich den Eingang Volumen von Plasma.
- Add Tris-gepufferter Kochsalzlösung (TBS), um verbleibende Volumen der Seton-Easy Seal Rohr an der Unterseite der Gewindezapfen zu füllen. Achten Sie darauf, dass sich keine Luftblasen im Schlauch oder das Rohr wird in der Ultrazentrifuge Zusammenbruch. Stellen Sie sicher, dass die Marke auf den Röhren ist nach außen bei der Platzierung der Röhrchen in den Rotor.
- Seal mit wiederverwendbaren Kappen und Ort Proben in einer vorgekühlten Sorval T1270 Rotor und Zentrifuge in der Sorval 90SE Ultrazentrifuge auf 170.000 xg (50.000 rpm) bei 4 ° C für 30 min.
- Nach der Zentrifugation zu entfernen Überstand und fügen 90 ul molekularen grade Wasser und 10 ul Proteinase-K (20 mg / ml) an die virale Pellet (dies wird nicht angezeigt).
- Die Röhrchen in einem 55 ° C Wasserbad inkubieren für 30 Minuten. Achten Sie darauf, die Rohre in einem Winkel, so dass die Seite mit dem Pellet in die Proteinase-K-Wasser-Gemisch getaucht wird eingestellt sind.
- Nach der Inkubation, kurz drehen Sie das Rohr in einer Tischzentrifuge für alle Flüssigkeit am Boden des Röhrchens zu sammeln (etwa 5 Sekunden) und fügen 315 UL von 6M Guanidiniumthiocyanat-Lösung und 10 ul von 20 mg / ml Glykogen (Hinweis: Befolgen Sie entsprechende Entsorgung Richtlinien für dieses gefährlichen Stoffes; nicht mit Bleichmittel oder Säuren mischen und entsorgen in separaten Behältern).
- Vortex leicht und Spin kurz (für ca. 5 Sekunden). Inkubieren für 10 Minuten bei Raumtemperatur und anschließend Transfer Inhalt jedes Röhrchens in eine neu beschriftet 1,5 ml RNAse freie Zentrifugenröhrchen.
- Add 495 UL der molekularen grade Isopropylalkohol in jedes Röhrchen, Wirbel für 10 Sekunden und Zentrifuge mit 21.000 xg für 30 Minuten bei Raumtemperatur.
- Überstand verwerfen und fügen Sie 500 ul 70% Ethanol.
- Vortex für 10 Sekunden und Zentrifuge mit 21.000 xg für 15 Minuten bei Raumtemperatur. Entfernen Ethanol mit einer Transferpipette. Spin wieder und entfernen Sie alle verbleibenden Ethanol mit einer Pipette. Luft für 10 Minuten trocknen lassen. (Für die SGS-Protokoll lösen RNA in 40 ul 5 mM Tris-HCl pH 8,0, und fahren Sie mit dem SGS-Protokoll).
- Lösen Sie Pellet in 55 ul der RNA-Puffer. RNA-Puffer wird durch Zugabe von 10 ul 100 mM DTT und 25 ul von 40 U / ul RNasin zu erhalten 965 ul Tris-HCl (pH 8,0, 5 mM). Auf Eis legen.
2. Die Single Copy-Test
Ein 96-Well-Platte besitzt 8 Patientenproben einschließlich HIV und RCAS Standards und Kontrollen. Dieses Protokoll beschreibt die Einrichtung einer einzigen Platte.
- Beginnen Sie mit der Herstellung von 2 Aliquots von 1 ml RNA-Puffer, in dem die HIV-1 RNA und RNA-Transkripte RCAS für die RNA-Standard-Kurven verdünnt werden. RNA-Puffer ist unter 1.13 beschrieben.
- Verdünnungen auf Eis in 2 ml Zentrifugenröhrchen. Machen halblogarithmische Verdünnungen von HIV-1 RNA-Transkripte in der RNA-Puffer durch Zugabe von 25 ul des HIV-1 RNA Lager (1x10 6 Kopien / ul) auf 54 ul RNA-Puffer geben 3x10 5 Kopien / ul.
- Weiter, indem aufeinanderfolgende halbe log Verdünnungen (25 ul + 54 ul) bis zu 0,3 Kopien pro 10 ul (Note: 0,3, 1 und 3 Verdünnungen werden nicht Teil des Standard-Kurve während der Analyse für diese werden als Extrapolation verwendeten Werte und sollte Unbekannten dur eingestellt werdenIng.-Analyse). RCAS Transkripte sind bei 1x10 6 Kopien / ul bestückt. Ebenso bereiten halblogarithmische Verdünnungen RCAS Transkripte in RNA-Puffer, aber bis zu 100 Kopien pro 10 ul.
- Bereiten Sie die RT-Cocktail für die cDNA-Synthese, wie in Tabelle 1 aufgeführt. Bereiten RT und NRT Reaktionen wie in Tabelle 1 aufgeführt unter Berücksichtigung zu machen, ein wenig mehr zu berücksichtigen, für Verluste, die während der Übertragung auftreten können. Hinweis: für die NRT-Cocktail, der RT-Enzym wird durch Wasser ersetzt, um für die Amplifikation von DNA zu kontrollieren.
Hinweis: Dieser Schritt hat vor kurzem zusätzlich zu Platte auf einem Qiagen Robot (ursprünglich Corbett) gesetzt wurden automatisiert. - Richten Sie das optische 96-Well-Platte (wie in Abbildung 3 dargestellt), durch Zugabe von 20 ul RT Reaktionsgemisch in jede Vertiefung. In den 8 Kavitäten mit der Aufschrift "NRT", fügen Sie die Cocktail ohne die reverse Transkriptase (NRT Reaktionsgemisch). Nach der Zugabe des Cocktails an der Platte, mit 10 ul Wasser in die Vertiefungen der Aufschrift "No-Template-Kontrollen (NTC)". Add 10 ul der HIV-Transkripte in die Vertiefungen der Aufschrift "HIV" in Konzentrationen in Abbildung 3 dargestellt. Add 10 ul RCAS Transkripte in die Vertiefungen der Aufschrift "RCAS" in nach-Konzentrationen. Add 10 ul der Proben in den 3 benachbarten Vertiefungen mit der Aufschrift "Muster" auf der Platte Diagramm. Add 10 ul der eluierten Probe in die Vertiefungen der Aufschrift "NRT". Add 5 ul der gleichen Probe und 5 ul Wasser in die Vertiefungen der Aufschrift "internen Kontrolle."
- Verschließen Sie die Platte und laufen auf Thermocycler bei 25 ° C für 15 Minuten, 42 ° C für 40 Minuten, 85 ° C für 10 Minuten, 25 ° C für 30 Minuten und 5 ° C zu halten.
- Bereiten PCR Master Mix gemäß Tabelle 2.
- Wenn die cDNA-Synthese abgeschlossen ist, um einen Bereich für die Verwendung von cDNA bezeichnet bewegen. In diesem bestimmten Bereich, mit 20 ul des PCR-Mastermix in jede Vertiefung der cDNA Platte für beide HIV und RCAS. Es ist wichtig, diesen Schritt in einem bestimmten Bereich durchzuführen, um mögliche Verunreinigungen zu vermeiden.
- Spin Platte, Dichtung mit ABI oder Roche Optical und Deckel mit optischer Decke (Decke nur für ABI 7700). Run auf Roche 480 oder ABI 7700. PCR Bedingungen: 95 ° C für 10 Minuten, dann 45 Zyklen von 95 ° C für 15 Sekunden, 60 ° C für 1 min. Eine getrennte Analyse für RCAS und HIV erforderlich. Beispiele für Ergebnisse sind in Abbildung 4 dargestellt. Die Steigung der Standardkurve wird durch einen normalen Poisson-Verteilung mit geringer Kopienzahl Transkripte betroffen sein. Um die genaue Steigung der Standardkurve, diese niedrigen Kopienzahl Standards (0,3, 1 und 3 Kopien) sicherzustellen, werden als set "Unbekannten." 17
3. Einzel-Genome Sequencing
- cDNA-Synthese. Add 5 ul 10 mM dNTPs und 5 ul der 2 mM Gen-spezifischen Primer (pol oder env) zu einem gut in einer 96-well PCR-Platte (Biorad). Add 40 ul der extrahierten RNA. Seal Platte.
- Denaturieren RNA-Mischung in Thermocycler bei 65 ° C für 10 min.
- Mischen Sie die Reagenzien für die cDNA-Synthese, in Tabelle 3 aufgeführt. Nach dem Denaturierungsschritt, Ort PCR-Platte auf Eis, fügen Sie die 50 ul der cDNA-Gemisch zu jeder Probe.
- Run PCR-Platte auf Thermocycler: 45 ° C für 50 min, 85 ° C für 10 min und 4 ° C zu halten.
- Erste PCR, 10 ul Reaktionen. Die PCR-Reaktionsgemisch in einem Reagenzträgers entweder mit dem Primer für die erste Runde Amplifikation von p6-rt oder env (Reagenzien in Tabelle 3 aufgeführten Primer in Tabelle 5 aufgeführt).
- Mit einer Mehrkanal-Pipette verzichtet 8,0 ul der PCR-Ansatz auf 73 Bohrungen auf dem 96-Well-PCR-Platte (70 Proben und 3 negative Kontrollen).
- Nach cDNA-Synthese beendet ist ziehen die cDNA und PCR-Platte mit PCR-Reaktionsmischung in einen Bereich für die Verwendung von cDNA bezeichnet. In dem bezeichneten Gebiet, dann werden 40 ul Tris-HCl pH 8,0 bis jeder cDNA-Probe bringt das Endvolumen auf 140 ul. Fügen Sie 2,0 ul der cDNA zu jedem der 70 Brunnen und 2,0 ul Wasser zu jedem der negativen Kontrollen. Seal PCR-Platte. Run auf Thermocycler-Programm in Tabelle 4.
- Nested PCR - 20ul Reaktionen. Mischen Sie die Reagenzien für die nested PCR in dem Reagenzträger (Reagenzien in Tabelle 3 aufgeführten Primer in Tabelle 5 aufgeführt).
- Add 18 &mgr; nested PCR-Gemisch auf 73 Bohrlöchern auf einer 96-well PCR-Platte, die von Mehrkanalpipette
- Verdünnen ersten PCR 1:5 und fügen Sie 2 ul der ersten Runde PCR, um die verschachtelte PCR. Run PCR-Reaktion auf den Thermocycler unter den Bedingungen in Tabelle 4 aufgeführt.
- Run Proben auf einem 1% Agarosegel. Um sicherzustellen, dass die Mehrheit der PCR-Produkte das Ergebnis von einem einzigen Molekül der cDNA sind, sollten nicht mehr als 30% der Brunnen positiv sein. Der Prozess wurde automatisiert mit dem Beckman Coulter Biomek FM-Roboter, um den Inhalt von PCR-Platten mit 1% Last, 96 Wells E-Gel (Invitrogen). E-Gele sind für 7 Minuten auf dem E-base laufen. Sequence-Produkte unter Verwendung der Primer in Tabelle 5 aufgelistet.
4. Repräsentative Ergebnisse:
SCA:
Alle Bedienelemente sollte passieren, um zu prüfen, die HIV-1 RNA-Messung zu korrigieren. Wenn eine negative Kontrolle ist Positive, sollte der Lauf wegen möglicher Verunreinigungen außer Acht gelassen werden. Um Verluste von RNA während der Extraktion Kontrolle, sollte mindestens 50% der RCAS im Plasma versetzt wiederhergestellt werden. Wenn die durchschnittliche RCAS ist <15.000 Exemplare, schlägt der Probe und sollte ignoriert werden, weil eine erhebliche Menge an RNA konnte während der Extraktion oder andere Schritte des Protokolls verloren gegangen sein. Dies geschieht in etwa 10% aller Patientenproben untersucht wahrscheinlich aufgrund der hohen Lipide in Plasma 6. Die Erholung der RCAS Virus soll für die Qualität der Extraktion und die Effizienz der cDNA-Synthese und PCR-Amplifikation zu kontrollieren. Es ist nicht verwendet werden, um für die HIV Erholung korrekt, da HIV wahrscheinlich Immunglobulinen und anderen menschlichen Proteinen macht es etwas anders aus Virus aus Gewebekultur isoliert gebunden. Die Erholung der RCAS in unserem Labor wurde im Durchschnitt 25.716 + / - 4.057 Kopien / Reaktionsgemisches, oder etwa 95% + / -15% des RCAS RNA 17 hinzugefügt. Die NRT (kein Reverse-Transkriptase-) Kontrolle wird parallel laufen, um für die Verstärkung von DNA-Test. Das NRT-Wert von jedem der drei HIV-1 RNA-Werte subtrahiert, dann der Durchschnitt errechnet und wenn diese Zahl kleiner als Null ist (die Amplifikation von DNA übersteigt die Amplifikation von RNA), das Ergebnis wäre fehl und sollte ignoriert werden, weil der Gefahr der Verstärkung von DNA als RNA. Wenn HIV-1 RNA ist jedoch erst ab 1 / 3 Brunnen verstärkt, eine erneute Prüfung der Probe wird empfohlen, um sicherzustellen, dass die Verstärkung wahr ist für die eigentliche Probe untersucht und nicht wegen einer möglichen Verunreinigung.
Wenn alle Kontrollen passieren, kann die durchschnittliche HIV-1 RNA-Kopienzahl im Plasma berechnet werden.
Beispiel 1: Wenn der durchschnittliche HIV-1-Kopienzahl Null ist, wird die Nachweisgrenze bezogen auf das Volumen des Plasmas untersucht berechnet. Als ein Beispiel, sagen wir mal 7 ml Plasma untersucht wurde. Die kleinste Menge an RNA, die mit diesem Test wurde nachgewiesen hätte 1 Exemplar in einem der Brunnen und 0 Kopien in den beiden anderen Brunnen, die eine durchschnittliche von 0,33 Exemplaren pro gut gewesen. Die durchschnittliche Kopienzahl muss dann mit einem Faktor von 5,5 für die gesamte Kopienzahl in der RNA Elution (es gibt 10 ul in jede Vertiefung, sondern die gesamte Elutionsvolumen betrug 55 ul) bekommen multipliziert werden. Die untere Nachweisgrenze ist dann: 5,5 x 0,33 Kopien von 7ml = 1,83 / 7 = 0,3 Kopien / ml aufgeteilt. Die Konzentration von HIV-1 RNA im Plasma in diesem Beispiel wurde <0,3 Kopien / ml.
Beispiel 2: HIV-1 RNA nachweisbar ist. RNA wurde aus 7 ml Plasma extrahiert. Die durchschnittliche Kopienzahl von HIV-1 RNA pro Well wird berechnet auf 2,0 Kopien pro Bohrung. Dann wird der durchschnittliche Kopienzahl pro ml Plasma beträgt 5,5 x 2,0 Kopien / 7 ml = 1,6 HIV-1 RNA / ml Plasma.
Eine Vorlage Excel-Sheet zum Kopieren Zahlen berechnen kann sich von der Website geladen werden.
SGS:
Wenn einer der negativen Kontrollen ist positiv, die ausgeführt werden soll wegen der möglichen Verunreinigung außer Acht gelassen werden. Die Anzahl der Produkte aus jedem Lauf wird auf die Viruslast in jeder Probe und der Zustand der Probe ab. In unserer Erfahrung eine Menge von Lipiden oder Zellen im Plasma reduziert die Chancen auf eine Produkt. Lagerbedingungen und früheren Einfrieren und Auftauen der Probe wird auch Einfluss auf die Erholung der RNA stark. In der Regel bei der Arbeit mit Proben mit einer Viruslast unter 50 Kopien / ml, Produkt (Bands auf Gel) ist in etwa ein Drittel -1 / 2 der Proben verarbeitet zu erwarten. Abhängig von der oben genannten Faktoren, sollte 1-5 Bands pro Platte ein gutes Ergebnis aufgrund der geringen Viruslast bei diesen Patienten in Betracht gezogen werden.
| cDNA-Synthese (RT Cocktail / cDNA-Reaktion) | 1-Reaktion | Kein Reverse-Transkriptase (NRT) Cocktail / cDNA-Reaktion (1-Reaktion) |
| Molecular Grade H 2 O | 8,1 ul | 8,2 ul |
| 25 mM MgCl 2 | 6 &mgr; | 6 &mgr; |
| 25 mM dNTPs | 0,6 ul | 0,6 ul |
| 100 mM DTT | 0,2 ul | 0,2 ul |
| Zufällige Hexamere (0,1 ug / ul) | 1,5 ul | 1,5 ul |
| 10X TaqMan Buffer A | 3,0 ul | 3,0 ul |
| RNasin (40 U / ul) | 0,5 ul | 0,5 ul |
| Strategene RT (200 U / ul) | 0,1 ul | Fügen Sie nicht |
| Gesamt | 20 ul | 20 ul |
| Proben-RNA | 10 ul | 10 ul |
| Gesamtvolumen | 30 ul | 30 ul |
Tabelle 1. Reaction mixtnahmen zur cDNA-Synthese in Single Copy-Test.
| PCR-Mastermix | 1-Reaktion | Grundierungen |
| H 2 O | 15,1 ul | HIV Vorwärtsprimer 5'-CATGTTTTCAGCATTATCAGAAGGA-3 ' |
| 10X PCR Gold-Buffer | 2,0 ul | HIV Reverse-Primer 5'-TGCTTGATGTCCCCCCACT-3 ' |
| 25 mM MgCl 2 | 2,0 ul | HIV Probe5'FAM-CCACCCCACAAGATTTAAACACCATGCTAA-TAMRA 3 ' |
| * Forward-Primer (100 um) | 0,3 ul | |
| * Reverse Primer (100 um) | 0,3 ul | RCAS Vorwärts Primer5'-GTCAATAGAGAGAGGGATGGACAAA-3 ' |
| * Probe (100 um) | 0,05 ul | RCAS umgekehrt Primer5'-TCCACAAGTGTAGCAGAGCCC-3 ' |
| TaqGold (5 U / ul) | 0,25 ul | RCAS Probe 5'FAM-TGGGTCGGGTGGTCGTGCC-TAMRA 3 ' |
| Gesamt | 20,0 ul |
Tabelle 2. PCR Master Mix und Primer für Single Copy-Test.
| cDNA-Cocktail / cDNA-Reaktionen | 1-RNA-Probe | Erste PCR-Cocktail | 1 Platte (75 Reaktionen) | Nested PCR-Cocktail | 1 Platte (75 Reaktionen) |
| 10x RT-Puffer (Invitrogen) | 10 ul | 10x PCR-Puffer (Invitrogen) | 75 ul | 10x PCR-Puffer | 150 ul |
| 25 mM MgCl 2 | 20 ul | 50 mM MgSO 4 | 30 ul | 50 mM MgSO 4 | 60 ul |
| 0,1 M DTT | 1 ul | 10 mM dNTPs | 15 ul | 10 mM dNTPs | 30 ul |
| RNase-freies Wasser | 17,5 ul | 50 uM Primer (ea) | 3 ul. | 50 uM Primer (ea) | 6 &mgr; |
| RNase-Out (Invitrogen) | 1 ul | Platinum Taq Hallo Fi Enzyme (Invitrogen) | 6 &mgr; | Platinum Taq Hallo Fi Enzyme (Invitrogen) | 12 ul |
| Superscript III (200 U / ul) (Invitrogen) | 0,5 ul | Molecular-grade Wasser | 468 ul | Molecular-grade Wasser | 1086 ul |
Tabelle 1. CDNA und PCR Mischungen für Single Genome Sequencing.
| P6-RT 1 PCR-Programm | Env 1 PCR-Programm |
| 1. 94 ° C für 2 Minuten | 1. 94 ° C für 2 Minuten |
| 2. 94 ° C für 30 Sekunden | 2 0,94 ° C für 30 Sekunden |
| 2 0,94 ° C für 30 Sekunden | 3. 52 ° C für 30 Sekunden |
| 4. 72 ° C für 1 Minute und 30 Sekunden | 4. 72 ° C für 1 Minute |
| 5. Zur Nr. 2, 44 Zyklen | 5. Zur Nr. 2, 44 Zyklen |
| 6. 72 ° C für 3 Minuten | 6. 72 ° C für 3 Minuten |
| 7. 4 ° C zu halten | 7. 4 ° C zu halten |
| P6-RT 2 PCR-Programm | Env 2 PCR-Programm |
| 1. 94 ° C für 2 Minuten | 1. 94 ° C für 2 Minuten |
| 2. 94 ° C für 30 Sekunden | 2. 94 ° C für 30 Sekunden |
| 3. 55 ° C für 30 Sekunden | 3. 56 ° C für 30 Sekunden |
| 4. 72 ° C für 1 Minute | 4. 72 ° C für 45 Sekunden |
| 5. Zum Nr. 1, 40 (41 Zyklen insgesamt) | 5. Zum Nr. 1, 40 (41 Zyklen insgesamt) |
| 6. 72 ° C für 3 Minuten | 6. 72 ° C für 3 Minuten |
| 7. 4 ° C zu halten | 7. 4 ° C zu halten |
Tabelle 4. Thermocycler Bedingungen für die Einzel-Genome Sequencing Assay.
| Reaktion | Grundierung | Reihenfolge |
| P6-RT cDNA | 3500 - | 5 'CTATTAAGTATTTTGATGGGTCATAA 3' |
| env cDNA | E115- | 5'AGAAAAATTCCCCTCCACAATTAA 3 ' |
| P6-RT 1. PCR | 3500 - | 5 'CTATTAAGTATTTTGATGGGTCATAA 3' |
| P6-RT 1. PCR | 1849 + | 5 'GATGACAGCATGTCAGGGAG 3' |
| P6-RT2.PCR | 1870 + | 5 'GAGTTTTGGCTGAGGCAATGAG 3' |
| P6-RT 2.PCR | 3410 - | 5 'CAGTTAGTGGTATTACTTCTGTTAGTGCTT 3' |
| env 1. PCR | E115- | 5'AGAAAAATTCCCCTCCACAATTAA 3 ' |
| env 1. PCR | E20 + | 5'GGGCCACACATGCCTGTGTACCCACAG 3 ' |
| env 2. PCR | E30 + | 5'GTGTACCCACAGACCCCAGCCCACAAG3 " |
| env 2. PCR | E125- | 5'CAATTTCTGGGTCCCCTCCTGAGG 3 ' |
| P6-RT-Sequenzierung | 2030 + | 5'TGTTGGAAATGTGGAAAGGAAGGAC 3 ' |
| P6-RT-Sequenzierung | 2600 + | 5'ATGGCCCAAAAGTTAAACAATGGC3 " |
| P6-RT-Sequenzierung | 2610 - | 5'TTCTTCTGTCAATGGCCATTGTTTAAC3 " |
| P6-RT-Sequenzierung | 3330 - | 5'TTGCCCAATTCAATTTTCCCACTAA 3 ' |
| env-Sequenzierung | E30 + | 5'GTGTACCCACAGACCCCAGCCCACAAG3 " |
| env-Sequenzierung | E125- | 5'CAATTTCTGGGTCCCCTCCTGAGG 3 ' |
Tabelle 5. Primer für die Einzel-Genome Sequencing Assay.
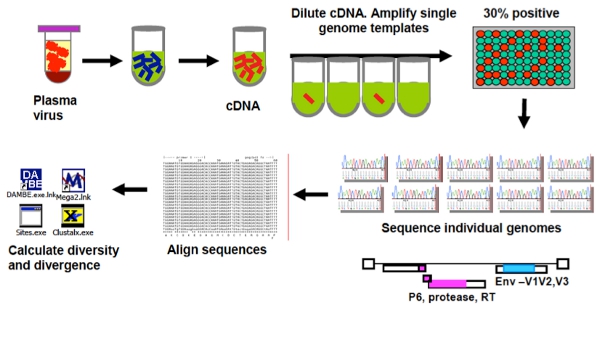
Abbildung 1. Übersicht über die Einzel-Genome Sequencing Assay (SGS).
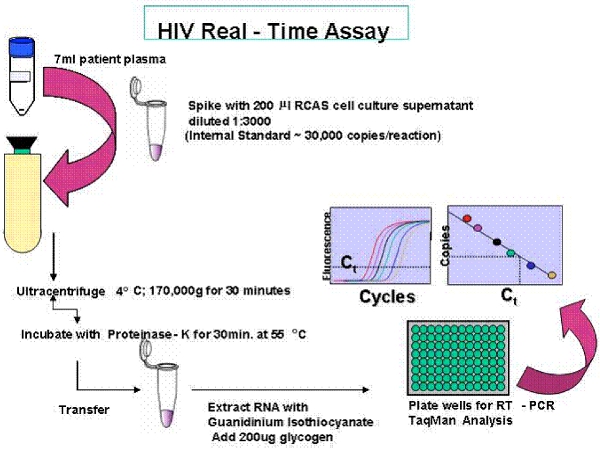
Abbildung 2. Überblick über die Single Copy-Test (SCA).
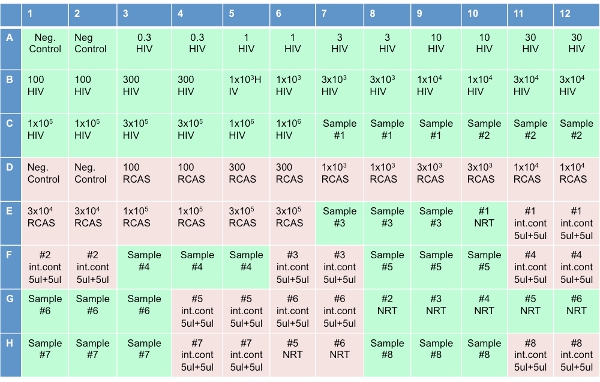
Abbildung 3. Plate Set-up für die Single Copy-Test.
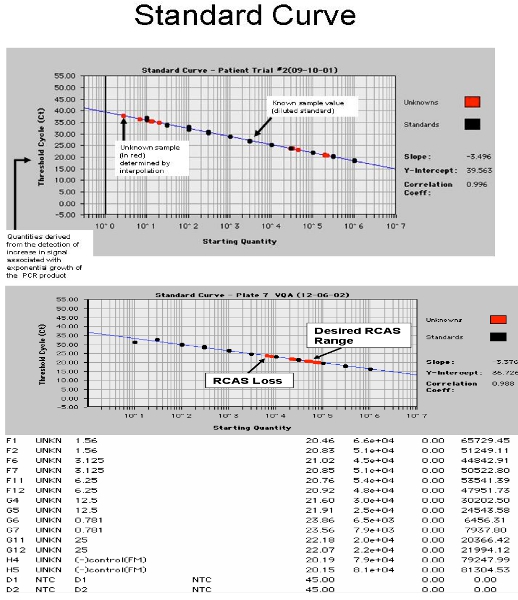
Abbildung 4. Bildschirm von der ABI 7700 erschossen. A. Darstellung der HIV-1 RNA-Standard-Kurve mit Patientenproben und B. zeigt RCAS Standardkurve mit Spikes Patientenproben.
Diskussion
HIV-1 infizierten Personen auf einer antiretroviralen Kombinationstherapie behandelt (cART) oder die natürlich kontrollieren die Infektion haben eine sehr niedrige Viruslast, typischerweise etwa 1 Exemplar pro ml Plasma 4, 11, 12, 17, 18. Viruslast bei Patienten mit natürlichen Kontrolle, oft um einen individuellen Sollwert 1, 2, 17 schwanken. Die Empfindlichkeit des hier beschriebenen Tests ist stark abhängig von der Eingabe Volumen von Plasma. Generell haben wir mit 7 ml Plasma gearbeitet, abe...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren möchten die Patienten, die in den zahlreichen Studien von HIV-1 beteiligt zu bestätigen.
JMC war ein Research Professor der American Cancer Society mit Unterstützung der FM Kirby Foundation.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz | Firma | Katalog-Nummer | Kommentare |
| Zufällige Hexameren (500 ug / ml) | Promega | C118A | |
| Taqman Puffer A | Applied Biosystems | 4304441 | |
| RNAsin (40 U / ul) | Promega | N211B | |
| RT-Enzym (200 U / ul) | Strategene | 600107-51 | |
| AmpliTaq Gold DNA-Polymerase | Applied Biosystems | 4311814 | 6-pack |
| Amplitaq 10XGold PCR-Puffer II | Applied Biosystems | 4311814 | kommt mit dem Enzym |
| Magnesiumcloride 25 mM | Applied Biosystems | 4311814 | kommt mit dem Enzym |
| 1M Tris-HCl-Puffer, pH 8,0, 5 mM | Invitrogen | AM9855G | |
| Superscript III Enzym reverse Transkriptase, 200 U / ul | Invitrogen | 18080-044 | |
| Superscript III 10X-Puffer | Invitrogen | kommt mit dem Enzym | |
| 100 mM DTT | Invitrogen | kommt mit dem Enzym | |
| Platinum Taq DNA Polymerase High Fidelity 5 U / ul | Invitrogen | 11304-102 | |
| 10x High Fidelity PCR Buffer | Invitrogen | 11304-102 | kommt mit dem Enzym |
| Proteinase-K 20 mg / ml | Ambion | 2546 | |
| Guanidiniumthiocyanat Lösung 6M | FlukaBiochemica | 50983 | |
| Glykogen 20 mg / ml | Roche | 10901393001 | |
| Isopropanol | Sigma-Aldrich | 19516 | |
| Ethanol 70% | Sigma-Aldrich | E702-3 | |
| Molecular-Wasser | Gibco / Invitrogen | 10977-015 | |
| dNTPs (25 mmol pro Stück) | Bioline | BIO-39025 |
| Name des Euipment | Firma | Katalog-Nummer | Kommentare |
| Einfache Seal Zentrifugenröhrchen (16x73mm) | Seaton Scientific | 6041 | |
| Weiß Delrin Kronen | Seaton Scientific | 4020 | Caps für den Easy Seal Tubes |
| Tube Rack für Optiseal Bell-Top Tubes, 8,9 ml | Beckman | 361642 | |
| Hex-Treiber ½ inc Eröffnung | Seaton Scientific | 4013 | |
| Kappe Entfernung tupe | Seaton Scientific | 4014 | |
| 5 ml serologischen Pipetten | Costar | 4051 | |
| Pipetboy | alle | ||
| 15 ml Transferpipetten | ISC Bioexpress | P-5005-7 | |
| 5,8 ml Dispossable Transferpipetten, feine Spitze | VWR | 14670-329 | |
| 15 ml Zentrifugenröhrchen | Falke | ||
| 1 ml Zentrifugenröhrchen | alle | ||
| Tris-Puffer Saline Tabletten | Sigma-Aldrich | T5030-100TAB | |
| Ultrazentrifuge und Rotor | Sorval | 90SE und T-1270 | |
| 96-well PCR-Platten | Biorad | HSS-9601 | |
| 50 ml Reagenz-Reservoir | Costar | 4870 | |
| Microamp optischen 96-Well-Reaktionsplatte | Applied Biosystems | N801-0560 | |
| Optische Platte deckt | Applied Biosystems | 4333183 | |
| MicroAmp Optical Compression Pad | Applied Biosystems | 4312639 | |
| MicrosealEin Film | Biorad | MSA-5001 | |
| 2 ml Zentrifugenröhrchen | Alle | ||
| 1% igen Agarosegelen (E-Gel, Invitrogen) | Invitrogen | G-7008 bis 01 | |
| E-base (Invitrogen) | Invitrogen | EB-M03 |
EDTA Sammelröhrchen jede Marke
Referenzen
- Amara, R. R., Villinger, F., Altman, J. D., Lydy, S. L., O'Neil, S. P., Staprans, S. I., Montefiori, D. C., Xu, Y., Herndon, J. G., Wyatt, L. S. Control of a mucosal challenge and prevention of AIDS by a multiprotein DNA/MVA vaccine. Vaccine. 20, 1949-1955 (2002).
- Dinoso, J., Kim, S., Blankson, J., Siliciano, R. F. Viral Dynamics of Elite Suppressors in HIV-1 Infection. Conference on Retroviruses and Opportunistic Infections. , (2008).
- Dinoso, J. B., Kim, S. Y., Wiegand, A. M., Palmer, S. E., Gange, S. J., Cranmer, L., O'Shea, A., Callender, M., Spivak, A., Brennan, T., Kearney, . Treatment intensification does not reduce residual HIV-1 viremia in patients on highly active antiretroviral therapy. Proc. Natl. Acad. Sci. U.S.A. 106, 9403-9408 (2009).
- Doria-Rose, N. A., Klein, R. M., Manion, M. M., O'Dell, S., Phogat, A., Chakrabarti, B., Hallahan, C. W., Migueles, S. A., Wrammert, J., Ahmed, R., Nason, M., Wyatt, R. T., Mascola, J. R., Connors, M. Frequency and phenotype of human immunodeficiency virus envelope-specific B cells from patients with broadly cross-neutralizing antibodies. J. Virol. 83, 188-199 (2009).
- Dornadula, G., Zhang, H., Uitert, B. V. a. n., Stern, J., Livornese, L., Ingerman, M. J., Witek, J., Kedanis, R. J., Natkin, J., DeSimone, J., Pomerantz, R. J. Residual HIV-1 RNA in blood plasma of patients taking suppressive highly active antiretroviral therapy. JAMA. 282, 1627-1632 (1999).
- Gandhi, R. T., Zheng, L., Bosch, R. J., Chan, E. S., Margolis, D. M., Read, S., Kallungal, B., Palmer, S., Medvik, K., Lederman, M. M., Alatrakchi, N., Jacobson, J. M., Wiegand, A., Kearney, M., Coffin, J. M., Mellors, J. W., Eron, J. J. The effect of raltegravir intensification on low-level residual viremia in HIV-infected patients on antiretroviral therapy: a randomized controlled trial. PLoS medicine. 7, (2010).
- Gay, C., Dibben, O., Anderson, J. A., Stacey, A., Mayo, A. J., Norris, P. J., Kuruc, J. D., Salazar-Gonzalez, J. F., Li, H., Keele, B. F., Hicks, C., Margolis, D., Ferrari, G., Haynes, B., Swanstrom, R. Cross-sectional detection of acute HIV infection: timing of transmission, inflammation and antiretroviral therapy. PLoS One. 6, 19617-19617 (2011).
- Hatano, H., Delwart, E. L., Norris, P. J., Lee, T. H., Dunn-Williams, J., Hunt, P. W., Hoh, R., Stramer, S. L., Linnen, J. M., McCune, J. M., Martin, J. N., Busch, M. P., Deeks, S. G. Evidence for persistent low-level viremia in individuals who control human immunodeficiency virus in the absence of antiretroviral therapy. J. Virol. 83, 329-335 (2009).
- Havlir, D. V., Bassett, R., Levitan, D., Gilbert, P., Tebas, P., Collier, A. C., Hirsch, M. S., Ignacio, C., Condra, J., Gunthard, H. F., Richman, D. D., Wong, J. K. Prevalence and predictive value of intermittent viremia with combination hiv therapy. JAMA. 286, 171-179 (2001).
- Jordan, M. R., Kearney, M., Palmer, S., Shao, W., Maldarelli, F., Coakley, E. P., Chappey, C., Wanke, C., Coffin, J. M. Comparison of standard PCR/cloning to single genome sequencing for analysis of HIV-1 populations. J Virol Methods. 168, 114-120 (2010).
- Kaufmann, D. E., Kavanagh, D. G., Pereyra, F., Zaunders, J. J., Mackey, E. W., Miura, T., Palmer, S., Brockman, M., Rathod, A., Piechocka-Trocha, A., Baker, B., Zhu, B., Gall, S. L. e., Waring, M. T., Ahern, R., Moss, K., Kelleher, A. D. Upregulation of CTLA-4 by HIV-specific CD4+ T cells correlates with disease progression and defines a reversible immune dysfunction. Nat Immunol. 8, 1246-1254 (2007).
- Kearney, M., Maldarelli, F., Shao, W., Margolick, J. B., Daar, E. S., Mellors, J. W., Rao, V., Coffin, J. M., Palmer, S. HIV-1 Population Genetics and Adaptation in Newly Infected Individuals. J. Virol. 83, 2715-2727 (2008).
- Kearney, M., Palmer, S., Maldarelli, F., Shao, W., Polis, M. A., Mican, J., Rock-Kress, D., Margolick, J. B., Coffin, J. M., Mellors, J. W. Frequent polymorphism at drug resistance sites in HIV-1 protease and reverse transcriptase. AIDS. 22, 497-501 (2008).
- Kearney, M., Spindler, J., Shao, W., Maldarelli, F., Palmer, S., Hu, S. L., Lifson, J. D., Kewal Ramani, V. N., Mellors, J. W., Coffin, J. M., Ambrose, Z. Genetic diversity of simian immunodeficiency virus encoding HIV-1 reverse transcriptase persists in macaques despite antiretroviral therapy. Journal of Virology. 85, 1067-1076 (2011).
- Keele, B. F., Giorgi, E. E., Salazar-Gonzalez, J. F., Decker, J. M., Pham, K. T., Salazar, M. G., Sun, C., Grayson, T., Wang, S., Li, H., Wei, X., Jiang, C., Kirchherr, J. L., Gao, F., Anderson, J. A., Ping, L. H., Swanstrom, R. Identification and characterization of transmitted and early founder virus envelopes in primary HIV-1 infection. Proc. Natl. Acad. Sci. U.S.A. , 105-7552 (2008).
- Liu, S. L., Rodrigo, A. G., Shankarappa, R. G., Learn, H., Hsu, L., Davidov, O., Zhao, L. P., Mullins, J. I. HIV quasispecies and resampling. Science. 273, 415-416 (1996).
- Mahalanabis, M., Jayaraman, P., Miura, T., Pereyra, F., Chester, E. M., Richardson, B., Walker, B., Haigwood, N. L. Continuous viral escape and selection by autologous neutralizing antibodies in drug-naive human immunodeficiency virus controllers. J. Virol. 83, 662-672 (2009).
- Migueles, S. A., Connors, M. The Role of CD4(+) and CD8(+) T Cells in Controlling HIV Infection. Curr. Infect. Dis. Rep. 4, 461-467 (2002).
- Palmer, S., Kearney, M., Maldarelli, F., Halvas, E. K., Bixby, C. J., Bazmi, H., Rock, D., Falloon, J., Davey, R. T., Dewar, R. L., Metcalf, J. A., Hammer, S., Mellors, J. W., Coffin, J. M. Multiple, linked human immunodeficiency virus type 1 drug resistance mutations in treatment-experienced patients are missed by standard genotype analysis. J. Clin. Microbiol. 43, 406-413 (2005).
- Palmer, S., Wiegand, A. P., Maldarelli, F., Bazmi, H., Mican, J. M., Polis, M., Dewar, R. L., Planta, A., Liu, S., Metcalf, J. A., Mellors, J. W., Coffin, J. M. New real-time reverse transcriptase-initiated PCR assay with single-copy sensitivity for human immunodeficiency virus type 1 RNA in plasma. J. Clin. Microbiol. 41, 4531-4536 (2003).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten