Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Biophysikalische Assays auf die mechanischen Eigenschaften der Interphase Zellkern Probe: Substrat Dehnungs-Anwendung und Mikronadel Manipulation
In diesem Artikel
Zusammenfassung
Wir stellen zwei unabhängige, Mikroskop-basierten Tools zur induzierten Nuklear-und Zytoskelett-Verformungen in Einzel-, Wohn adhärente Zellen in Reaktion auf die globale oder lokale Dehnung Anwendung zu messen. Diese Techniken werden verwendet, um nukleare Steifigkeit (dh, Verformbarkeit) zu ermitteln und intrazellulären Kraftübertragung zwischen dem Kern und dem Zytoskelett-Sonde.
Zusammenfassung
In den meisten eukaryotischen Zellen ist der Kern der größten Organelle und ist in der Regel 2 bis 10 mal steifer als die umgebenden Zytoskeletts, folglich tragen die physikalischen Eigenschaften des Kerns wesentlich zur umfassenden biomechanischen Verhalten von Zellen unter physiologischen und pathologischen Bedingungen. Zum Beispiel bei der Migration von Neutrophilen und eindringende Krebszellen, nukleare Steifigkeit kann ein großes Hindernis bei der Extravasation oder Durchfahrt durch Engstellen im Gewebe darstellen. 1 Auf der anderen Seite erfordert der Kern der Zellen in mechanisch aktive Gewebe wie Muskeln ausreichende strukturelle Unterstützung standhalten repetitive mechanische Beanspruchung. Wichtig ist, dass der Kern fest in der zellulären Architektur integriert, es ist physikalisch mit dem umgebenden Zytoskelett, das eine entscheidende Voraussetzung für die intrazelluläre Bewegung und Positionierung des Kerns, zum Beispiel in polarisierten Zellen, synaptische Kerne an neuromuskulären Synapsen verbunden ist, oder bei der Migration von Zellen. 2 Nicht überraschend, dass Mutationen in Kernhüllenproteine wie Lamine und nesprins, die eine entscheidende Rolle spielen bei der Bestimmung nuklearen Steifigkeit und nucleo-Zytoskelett-Kopplung, wurden kürzlich in einer Reihe von menschlichen Krankheiten, darunter ausgewiesen Emery- Dreifuss Muskeldystrophie, Körper-Muskeldystrophie und Kardiomyopathie. 3 Um die biophysikalischen Funktion der verschiedenen Kernhüllenproteine und die Wirkung der spezifischen Mutationen zu untersuchen, haben wir experimentelle Methoden, um die physikalischen Eigenschaften des Kerns in einzelnen lebenden Zellen Studie entwickelt unterzogen, um globale oder lokale mechanische Störung. Mess-induzierte atomare Verformungen in Reaktion auf genau angewendet Substrat Belastung Anwendung liefert wichtige Informationen über die Verformbarkeit des Kerns und ermöglicht quantitative Vergleich zwischen verschiedenen Mutationen oder Zelllinien Mangel für bestimmte Kernhüllenproteine. Lokalisierte Zytoskelett-Dehnungs-Anwendung mit einer Mikronadel wird verwendet, um diesen Test zu ergänzen und zusätzliche Informationen über die intrazelluläre Kraftübertragung zwischen dem Kern und dem Zytoskelett Ertrag. Studieren nuklearen Mechanik in intakten lebenden Zellen erhält die normale intrazelluläre Architektur und vermeidet mögliche Artefakte, die entstehen, wenn die Arbeit mit isolierten Kernen kann. Darüber hinaus präsentiert Substrat Belastung Anwendung ein gutes Modell für die physiologischen Stress, der durch Zellen in Muskel oder anderen Geweben (zB vaskuläre glatte Muskelzellen Schiff Belastungen ausgesetzt) erlebt. Schließlich, während diese Werkzeuge wurden in erster Linie die nukleare Mechanik Studie entwickelt, können sie auch angewendet, um die Funktion des Zytoskelett-Proteine und Mechanotransduktion Signalisierung zu untersuchen.
Protokoll
1. Substrate Belastung Anwendung
Die Messung der normalisierten nukleare Belastung umfasst die Vorbereitung der Belastung Gerichte mit transparenten, elastischen Silikon-Membranen als Zellkultur-Oberfläche, Plattieren Zellen auf den Teller, und den Erwerb von Bildern der Zellen vor, während und nach (einachsige oder zweiachsige) Stamm-Anwendung.
Herstellung von Silicon-Membran Gerichte und die Einhaltung von Zellen
- Jeder Stamm Gericht besteht aus einem maßgeschneiderten bodenlose Plastikschale mit einem Durchmesser von 3 "und einem Kunststoff-O-Ring, um eine Silikon-Membran, die als Zellsubstrates dient zu halten. Für die Herstellung des Stammes Gerichte, Klemme 4" x 4 "Stück Silikonmembran zwischen den O-Ring und das Gericht. vorsichtig weggeschnitten das überschüssige Membran mit deionisiertem Wasser abspülen und Autoklaven die Belastung Gerichte.
- Mark einen Bezugspunkt in der unteren Mitte der Membran (außen) vor der Beschichtung der Silikon-Membranen mit extrazellulären Matrix (zB Fibronectin). Dieses Wahrzeichen wird dazu beitragen, die gleichen Zellen während der Belastung Experimente. (Optional: Für einachsige Beanspruchung Anwendung sind zwei parallel verlaufenden Streifen Tesafilm um den Bezugspunkt angewendet, um eine Verformung der Membran in einer Dimension zu beschränken.)
- Um optimale Zelladhäsion, Mantel der Silikon-Membranen mit 3 ug / ml Fibronektin in 10 ml PBS oder geeignetem Proteine der extrazellulären Matrix verdünnt sind. Decken Sie die Belastung Schüssel mit einem umgekehrten 10cm Polystyrol Schüssel und inkubieren Sie die Speisen über Nacht bei 4 ° C.
- Am nächsten Tag, spülen Sie die Membranen einmal mit Phosphat-gepufferter Saline (PBS), um überschüssiges Protein zu entfernen. Füllen Schüssel mit 10 ml Wachstumsmedium (Dulbecco Modified Eagles Medium (hohe Glukose) mit 10% fötalem Rinderserum und 1% Penicillin / Streptomycin) und beiseite stellen.
- 48 Stunden unter normalen Kulturbedingungen - Maus embryonale Fibroblasten werden mit 0,05% Trypsin und ausgesät in Wachstumsmedium bei ca. 30% Konfluenz auf die beschichtete Silikon-Membran Teller und inkubieren für 24 trypsiniert.
Substrate Belastung Experimente
- Set-up des Mikroskops für Experimente. Die Versuche werden auf einem inversen Mikroskop mit einer Digitalkamera für Fluoreszenz-Mikroskopie, Phasenkontrast oder DIC geeignet durchgeführt, unter Verwendung eines 60x objektive und angemessene Bildaufnahme-Software (zB IPLab oder Metamorph). Eine aufrechte Mikroskop eignet sich nicht für diese Anwendung. Der Stamm besteht aus einer Grundplatte, die auf dem Mikroskoptisch passt und hält eine zentrale zylindrische Walze, die sich anstrengen, um den zentralen Abschnitt der Silikonmembran, einer beweglichen Platte, dass der Stamm Gericht hält gelten dient, und das kann nach oben und unten auf vier integrierten Führungsstiften, sowie eine 5-lb Gewicht Platte, um eine Last sind.
- Um Kerne sichtbar zu machen, Inkubieren Sie die Zellen in dem Stamm Schüssel mit 1 pg / ml Hoechst 33342 für 15 min bei 37 ° C. Absaugen des Mediums und ersetzen mit 15 ml Phenolrot-freien Nährmedium (Phenol-rot freien Dulbeccos Modified Eagles Medium (hohe Glukose) mit 25 mM Hepes, mit 10% fötalem Rinderserum und 1% Penicillin / Streptomycin). Schrauben Sie die Belastung Schüssel in der Schüssel Halteplatte. Sorgfältig Fett (Braycote 804 Vacuum Grease), um den Umfang der Unterseite der Silikonmembran, um sicherzustellen, Gleiten der Membran entlang der zentralen Platte. Achten Sie darauf, halten die zentralen Abschnitt der Membran klar.
- Platzieren Sie die Grundplatte auf dem Mikroskoptisch. Berg Servierhalter Platte vorsichtig auf die Bodenplatte. Sicherstellen, dass in der ersten Ruheposition, die Silikonmembran des Stammes Schüssel locker beruht auf der zentralen Platte.
- Erstens, auf der Unterseite der Silikonmembran konzentrieren und finden Sie die zentralen Schwarzen Referenzpunkt. Der Punkt wird als Ausgangspunkt für alle Bildaufnahmen und Hilfsmittel bei der Suche nach den gleichen Zellen während und nach der Strecke dienen. Wir verwenden eine speziell geschriebenen automatisierte Imaging-Programm, um die Positionen von Zellen zu speichern und diese Zellen während des Experiments zu verlegen, dies kann aber auch manuell erfolgen.
- Ab dem Punkt, stellen Sie den Fokus auf die Zellen und die Spitze der Silikonmembran zu visualisieren. Suchen Sie auch verbreitet Zellen mit zentral gelegenen Kernen und erwerben ein Phasenkontrast-und Fluoreszenz-Bild der nuklearen Hoechst-Färbung. Die Phasenkontrast-Bild sollte auf der Zelle Gliederung und die Silikon-Membran zu konzentrieren, während die Fluoreszenz-Aufnahmen auf der Mittelebene des Kerns konzentrieren sollten.
- Nach der Übernahme von Bildern von 5 bis 15 Zellen, an die zentrale Punkt zu bewegen. Langsam gilt das Gewicht, um die Belastung Gericht, was in Uniform Belastung Anwendung in der Mitte der Schale. Die maximale angewendet Substrat Spannung wird durch die Nylon-Abstandshalter auf die vertikale Ausrichtung Pins (Leitung Pins) platziert begrenzt.
- Konzentrieren Sie sich auf der Unterseite der Silikonmembran und suchen Sie den Referenzpunkt wieder. Ausgehend von derdot, verlagern die gleichen Zellen wieder erwerben Phasenkontrast und Fluoreszenz-Bild der Zellen und Kerne unter voller Belastung, versuchen, entsprechen fast genau den Brennebenen der ersten Bilder. Dieser Vorgang sollte nicht länger als 10 min zur aktiven Umgestaltung und Anpassung der Zelle an der angespannten Substrat zu vermeiden.
- Nachdem alle entsprechenden Bilder nach dem Erwerb, bewegen Sie den Mikroskoptisch zurück zum Ausgangspunkt. Entfernen Sie vorsichtig das Gewicht von der Schale Halteplatte und ermöglichen die Silikonmembran zu entspannen. Falls erforderlich, schieben Sie die Belastung Schüssel, bis sie in die Ausgangsposition ist. Dann erwerben Phasenkontrast-und Fluoreszenz-Aufnahmen der post-Dehnungs-Zellen, wie oben für die Belastung Bildern beschrieben.
Analyse
- Bilder von Zellen und fluoreszenzmarkierten Kernen vor, während und nach der Belastung Anwendung werden analysiert, um die normierte nukleare Belastung berechnen. In unserem Labor verwenden wir eine speziell geschriebenen Matlab-Skript für die Analyse, sondern mehrere alternative Optionen zur Verfügung. Die Analyse erfolgt in drei Schritten durchgeführt.
- Erstens, um die aufgebrachte Substrat Belastung zu berechnen, werden die Positionen von 3 bis 6 Kontrollpunkte auf der Membran befindet manuell zwischen den entsprechenden Vor-, Voll-und post-Dehnungs-Bilder abgestimmt. Die MATLAB-Programm berechnet dann die aufgebrachte Membran Belastung durch den Vergleich der Positionen der passenden Kontrollpunkte zwischen den pre-Dehnungs-und Full-Dehnung Bilder und auch die Restspannung zwischen den pre-Dehnungs-und post-Dehnungs-Bilder. Zur gleichen Zeit werden die Kontrollpunkte, an denen die Bildpaare, die dazu beitragen, beschädigt oder Abnehmen Zelle (siehe Abbildung 1) erkennen wird zu registrieren.
- In einem zweiten Schritt werden Kerne manuell ausgewählt mit einem separaten MATLAB-Programm, dass die nukleare Belastung für jeden einzelnen Kern mit passenden entweder Kerngröße oder intranukleären Marker zwischen den entsprechenden Vor-, Voll-und post-Dehnungs-fluoreszierende Bilder berechnet. Zur Berücksichtigung kleinen Variationen in der angewandten Membran Spannung zwischen verschiedenen Experimenten, drücken wir Ergebnisse als Normalized Nuclear Strain, als das Verhältnis der induzierten nukleare Belastung angewendet Membran Stamm, der für jeden Kern berechnet wird definiert. Die MATLAB-Skripte werden von der Lammerding Labor auf Anfrage zur Verfügung.
- Schließlich ist jeder Kern bestätigt, ohne Messungen von Zellen, oder trennen beschädigt werden während der Belastung Anwendung (Abbildung 1).
2. Mikronadel Manipulation Test
Zubereitung der Speisen, adhärente Zellen und Mikronadeln
- Inkubieren 35 mm Glasboden Zellkulturschalen mit einer niedrigen Konzentration von Fibronektin (0.5ug/ml) in Buffered Salz Hanks Salzlösung (HBSS) oder jede geeignete Proteine der extrazellulären Matrix für 2 Stunden bei 37 ° C. Wash Gerichte mit HBSS zwei Mal und 2 ml Wachstumsmedium in die Schale, bevor Sie den nächsten Schritt.
- Maus embryonale Fibroblasten werden mit 0,05% Trypsin und ausgesät in 2 ml Wachstumsmedium bei 7,5 x 10 4 Zellen / ml auf die Fibronektin-beschichteten Glasboden ausgesät trypsiniert. Legen Zellen zurück in Inkubator über Nacht. Legen Zellen zurück in Inkubator über Nacht. Eines sollten die Optimierung der Anzahl der Zellen, um Einzel-, Haft-, nicht-konfluente Zellen für andere Zelltypen zu erhalten.
- Ziehen Sie den Mikronadeln, aus Borosilikatglas Kapillaren aus, zu einem Durchmesser von ca. 1 bis 3 mu m mit einer handelsüblichen Pipette Abzieher (z. B. Sutter Instrument Company) Spitze.
Mikronadel Manipulation Experiment
- Am nächsten Tag, inkubieren Sie die Zellen mit MitoTracker mitochondrialen Fleck (600 uM; Invitrogen) und Hoechst 33342 Kernfärbung (1 pg / mL) zugegeben, um das Wachstum Medium für 30 Minuten bei 37 ° C Inkubator.
- Waschen Sie die Zellen einmal in HBSS für 5 Minuten bei Raumtemperatur und fügen Sie dann Phenolrot-freien Nährmedium der Zellen für die Bildgebung.
- Erwerben Sie ein Bild von der einzelnen Zelle, ohne die Mikronadel in das Zytoskelett im Phasenkontrast eingesetzt, Fleck ein fluoreszierendes Abbild der Hoechst 33342-Färbung und einem fluoreszierenden Bild der Mitochondrien, mit einem 60fach-Objektiv (0,70 NA, Plan-Achromat) auf eine umgekehrte Mikroskop mit einer digitalen charge-coupled device Kamera.
- Mit einem Mikromanipulator (zB InjectMan NI 2, Eppendorf), vorsichtig die Mikronadel in das Zytoplasma einer Zelle in einem festen Abstand (typischerweise 5 um) weg von der nuklearen Peripherie und nehmen eine Phase kontrastreiches Bild, ein fluoreszierendes Abbild der Hoechst 33342 Fleck und ein fluoreszierendes Abbild der mitochondrialen Fleck. Für diese Anwendung ist es hilfreich, den Mikromanipulator über einen Computer zu steuern, z. B. Windows Hyperterminal, um konsistente Mikromanipulation Verfahren zu erreichen.
- Bewegen Sie den Mikronadel, einen bestimmten Abstand (in der Regel 10 oder 20 um) in Richtung der Zellperipherie bei 1 um / sec, während gleichzeitig das Sammeln Fluoreszenz-und Phasenkontrast-Bilder alle 10Sekunden. Als Mikronadel ist es, eine bestimmte Strecke in Richtung der Zellperipherie bewegen. Mit den gewählten Parametern, wie der Mikronadel zu einem bestimmten Abstand in Richtung der Zellperipherie in Bewegung ist, wird diese auf 2-3 Frames während der Manipulation Prozess entsprechen.
- Schließlich erwerben weitere Bilder nach dem Mikronadel aus dem Zytoskelett entfernt wird.
Analyse
- Displacement Maps berechnet mit Hilfe eines speziell geschriebenen Matlab-Skript auf Tracking fluoreszenzmarkierten Features der Zellkern und Cytoplasma auf. (Die MATLAB-Skript ist von der Lammerding Labor auf Anfrage). Das Programm verwendet eine normalisierte Kreuzkorrelation Algorithmus zwischen kleinen Bildbereichen (etwa 10 um x 10 um in Größe und Abstand von 5 mu m Abstand) in den nachfolgenden Bildrahmen. Für jede Region Mitte, die Verschiebung in x-und y-Richtung werden als die Verschiebung zwischen den ursprünglichen Speicherort und die neu identifizierten Position berechnet und angezeigt als Verschiebungsvektor und auch als Zahlenwerte gespeichert. Aus der Verschiebung Karten können durchschnittlich Verschiebungen innerhalb vordefinierter Regionen berechnet werden. Beachten Sie, dass Zytoskelett-Verschiebungen auf der Fluoreszenz-Kanal für Zytoskelett-Markern (zB MitoTracker mitochondriale Färbung) beruhen, während die Kern-Verschiebungen von der Fluoreszenz-Kanal entsprechend der Hoechst 33342-Signal berechnet werden. Für unsere Anwendung haben wir routinemäßig untersuchen die folgenden Regionen: (i) des Zytoskeletts Dehnung bei der Belastung der Applikationsstelle, dh die Mikronadel Insertionsstelle, (ii) nukleare Belastung in einer Region im Kern auf die Belastung der Applikationsstelle; (iii) nukleare Stamm in einem Kernbereich von der Anwendung vor Ort, und (iv) des Zytoskeletts Stamm in einem cytoplasmatischen Region in den Zellkern. Darüber hinaus kann man auch direkt zu messen nuklearen Dehnung von Phasenkontrast oder Hoechst 33342 Fluoreszenz Bildsequenzen. In diesem Fall müssen die nukleare Belastung durch Division des nuklearen Dehnung (AL = L - L 0) berechnet wird durch die ursprüngliche Länge L 0, wobei L die endgültige Länge des Kerns am Ende des Stammes Anwendung und L 0 ist die ursprüngliche Länge des Kerns. Für Zellen mit intaktem nucleo-Zytoskelett-Kopplung wird der Kern in Richtung der Belastung der Applikationsstelle zu verlängern. Im Gegensatz dazu in Zellen, in denen nucleo-Zytoskelett-Kopplung unterbrochen, dh Kräfte weniger effizient sind zwischen dem Zytoskelett und den Kern übertragen werden, ist der Kern erwartet, dass deutlich weniger länglich in Richtung des Stammes der Applikationsstelle. So verringerte sich nuklearen Verformungen in Reaktion auf Zytoskelett-Dehnungs-Anwendung bedeutet eine (teilweise) Entkopplung zwischen dem Kern und Zytoskelett.
3. Repräsentative Ergebnisse:
Substrate Belastung Anwendung
Wir aufgenommenen Bilder vor, während und nach der Belastung Anwendung embryonalen Maus-Fibroblasten, die aus heterozygoten und homozygoten Lamin A / C-Mangel (LMNA + / - und LMNA - / -) und Wildtyp (LMNA + / +) Mäuse und anschließend berechnet die normalisierte nukleare Belastung für jede Zelle. Nach der Analyse werden die Kerne validiert und Zellen, die beschädigt werden oder widerrufen während Belastung Anwendung werden aus der Analyse ausgeschlossen. Abbildung 1A zeigt Kerne aus drei Zellen, die gültig sind, während Abbildung 1B zeigt Zellen, die aus der Analyse ausgeschlossen werden sollten. Normalized nukleare Belastung Daten aus mindestens drei unabhängigen Experimenten (jeweils Messungen von ~ 5-10 Kerne) zusammengefasst und verglichen mit anderen Zelle oder Behandlungsgruppen durch eine statistische Analyse. Erhöhte normalisiert nukleare Belastung zeigt reduziert nuklearen Steifigkeit, als in Zellen mit verminderter Expression der Kernhüllenproteine Lamin A / C (Abb. 2) zu sehen.
Mikronadel Manipulation Test
Für die Mikronadel Manipulation Assay abgebildet wir Kern-und Zytoskelett-Verschiebungen während lokalisiert Zytoskelett-Dehnungs-Anwendung. Zellen, die beschädigt oder abgelöst werden aus der Analyse ausgeschlossen. Für die Analyse, messen wir die Größe des Kern-und Zytoskelett-Bewegungen in Richtung der Krafteinwirkung Ort in Einzel-, adhärente Zellen. Zum Beispiel in Abbildung 3, verfolgen wir mitochondriale (Marker für das Zytoskelett) Verschiebungen vor und nach dem Zytoskelett-Belastung und dann zeichnen Sie die Verschiebungen als Vektoren. Jeder Vektor repräsentiert die Verschiebung als die Verschiebung zwischen den ursprünglichen Speicherort und die neu identifizierten Position berechnet. Regionen mit niedrigen Bildintensität oder unzureichende Struktur (zB Regionen außerhalb der Zelle) werden aus der Analyse ausgeschlossen. Das Zytoskelett und nukleare Verschiebungen werden dann in ausgewählten Bereichen mit zunehmender Entfernung von den Strapazen der Applikationsstelle (Abbildung 4, Bereiche entsprechend der farbigen bo quantifiziertxes in Kasten). In embryonalen Maus-Fibroblasten mit intakten nucleo-Zytoskelett-Kopplung werden die Kräfte durch den gesamten Zellen übertragen, was zu einer induzierten Nuklear-und Zytoskelett-Deformationen, die sich langsam weg von den Strapazen der Applikationsstelle (Abbildung 4) abzuführen. Im Gegensatz dazu anzuzeigen Fibroblasten mit gestörter nucleo-Zytoskelett-Kupplung (oder verändert Zytoskelett-Organisation) lokalisiert Verschiebungen in der Nähe der Applikationsstelle, wie in Abbildung 4 dargestellt und nur wenig induzierte Verformungen weiter weg. Vergleichbare Zytoskelett-Dehnungs-Anwendung auf der Mikronadel Insertionsstelle (orange box) ist sowohl für Kontrolle Fibroblasten (mCherry allein) und Fibroblasten, die mit einer gestörten nucleo-Zytoskelett-Kupplung (DN KASH) beobachtet. Allerdings induziert wurden Kern-und Zytoskelett-Verschiebungen (blau, gelb und rot ankreuzen) am anderen Regionen deutlich kleiner in den Fibroblasten mit einer gestörten nucleo-Zytoskelett-Kupplung (DN KASH) als in Kontrollzellen (mCherry allein) (Abbildung 4). So Rückgang des Zytoskeletts und nukleare Verschiebungen weg von den Strapazen der Applikationsstelle, zeigt an, dass die Kraftübertragung zwischen dem Zytoskelett und Kern gestört wurde.
Wichtig ist, haben wir auch validiert, dass Mitochondrien geeignet Zytoskelett-Marker sind, durch die Durchführung Mikronadel Manipulation an embryonalen Maus-Fibroblasten, die mit GFP-oder mCherry Aktin und GFP-Vimentin transfiziert und fluoreszierend mit Mitotracker Grün oder Rot gekennzeichnet. Zytoskelett-Displacement-Maps wurden unabhängig von der Fluoreszenz-Signal der Mitochondrien und der Aktin-Zytoskeletts oder Vimentin berechnet. Die durchschnittliche absolute Verschiebung wurde für vier verschiedene Zytoskelett-Regionen mit zunehmender Entfernung vom Stamm Einsatzort berechnet. Die Steigung und der R-squared-Werte wurden aus der linearen Regression zwischen den Messungen von Mitochondrien und von Aktin oder Vimentin, jeweils erhaltenen berechnet. Für Aktin, war die Neigung 0,99 und der R 2-Wert war 0,986; für Vimentin, war die Steigung 1,04 und der R2-Wert betrug 0,971, die bestätigt, dass mitochondriale Verschiebungen als zuverlässige Indikatoren für Zytoskelett Verformungen dienen.
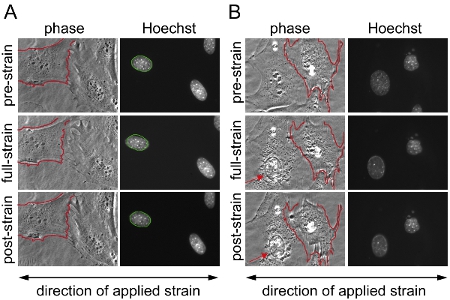
Abbildung 1. Substrat-Stamm-Anwendung auf embryonalen Maus-Fibroblasten (MEFs). Embryonalen Maus-Fibroblasten über zwei unterschiedliche Bereiche auf dem Silizium-Membran verteilt wurden mit Phasenkontrast-und Fluoreszenz-Mikroskopie, bevor abgebildet, während und nach der Anwendung der 20% einachsigen Belastung. (A) Beispiel für ein gelungenes Experiment mit gültigen Kerne von Zellen, die die Belastung Anwendung ohne Beschädigung oder Ablösung und (B) beispielsweise von Zellen, die / Einfahren teilweise während Belastung Anwendung abgelöst überlebt; Ergebnisse aus den Zellen dargestellt in (B) sind ausgeschlossen aus der Analyse. In (B), zeigt die Zelle auf der linken Seite Anzeichen von Zytoskelett-Schäden und nuklearen Zerfall (Pfeil), während die Zelle auf der rechten Seite löst teilweise und fährt während der Belastung Anwendung. Dies kann ein Indiz für übermäßige Belastung Anwendung. Zum besseren Vergleich in (A) und (B) die Grenze von einer der ungedehnten Zellmembranen ist in rot dargestellt und überlagert auf der gleichen Zelle während und nach der Belastung Anwendung. In (A) die Grenze des ungedehnten Kern ist in grün dargestellt und überlagert auf dem gleichen Kern während und nach der Belastung Anwendung.
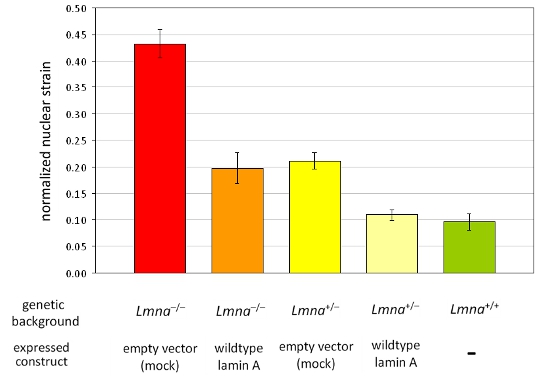
Abbildung 2. Die Analyse der normalisierten nukleare Belastung in einem Panel von verschiedenen MEF-Zelllinien MEFs der LMNA -. / - Und LMNA + / - genetischen Hintergrund ektopisch, die entweder einen leeren Vektor-oder Wildtyp-Lamin A analysiert wurden. Im Vergleich zu MEFs von Wildtyp-Geschwister (LMNA + / +), Verlust von Lamin A / C-Ausdruck führt zu verminderten nuklearen Steifigkeit, die vollständig durch die Wiedereinführung von Wildtyp-Lamin A. wiederhergestellt werden können Bemerkenswert ist, reduziert nuklearen Steifigkeit ist durch reflektiertes erhöhte Werte der normalisierten nukleare Belastung. Die Fehlerbalken stellen Standardfehler.

Abbildung 3. Mikronadel Manipulation Assay intrazellulären Kraftübertragung zu messen. Phasenkontrast (A, B) und Fluoreszenz (C, D) Bilder einer Fibroblasten mit Kernfärbung (blau) gekennzeichnet und MitoTracker mitochondriale Färbung (grün). Ein Mikronadel wurde in das Zytoskelett in einem definierten Abstand vom Kern (A und C) eingeführt und anschließend in Richtung der Zellperipherie (B, D) verschoben. Zytoskelett und nukleare Verschiebungen wurden von Tracking fluoreszenzmarkierten Kern und Mitochondrien mit Hilfe eines speziell geschriebenen Kreuzkorrelation Algorithmus quantifiziert. (E) Displacement-Map des letzten Zytoskeletts (grün) Verformungen von Fluoreszenz Bildserien berechnet; Pfeillänge ist 2x besser visibi vergrößertkeit. Scale-Bars, 10 pm.
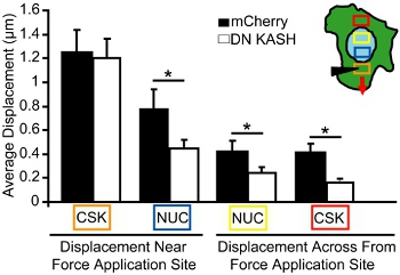
Abbildung 4. Die Analyse der intrazellulären Kraftübertragung während Mikronadel Manipulation. Induced Zytoskeletts und nukleare Verschiebungen während Mikronadel Manipulation, gemessen in den Bereichen entsprechend der farbigen Kästchen (siehe Einschub in A). Die Orange Box ist die Belastung der Applikationsstelle. Trotz ähnlicher Stamm Anwendung in das Zytoskelett (orange box), induziert Nuklear-und Zytoskelett-Verschiebungen (blau, gelb und rot ankreuzen) waren signifikant kleiner in der embryonalen Maus-Fibroblasten, die mit einer gestörten nucleo-Zytoskelett-Kupplung (DN KASH) im Vergleich zur Kontrollgruppe ( mCherry alone)-Zellen.
Diskussion
Substrat-Stamm-Test
Dehnungs-Anwendung wurde erfolgreich von uns und anderen Gruppen verwendet werden, um induzierte atomare Verformungen in den Zellen unterzogen, um mechanische Belastungen zu untersuchen und den Beitrag der spezifischen Kernhüllenproteine der nuklearen Steifigkeit zu untersuchen. 4-8 Der Vorteil dieser Technik ist, dass es Sonden mechanischen Eigenschaften Lebensstandard Kerne in ihrer normalen zellulären und Zytoskelett-Umgebung und das Substrat Belastung ...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde von den National Institutes of Health (R01 HL082792 und R01 NS059348) und dem Brigham and Women Hospital Cardiovascular Leadership Group-Award unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz | Firma | Katalog-Nummer | |
| Fibronectin | Millipore | FC010 | |
| MitoTracker Red FM und Green FM | Invitrogen | M22425 und M-7514 | |
| Hoechst 33342 | Invitrogen | H3570 | |
| Hanks Buffered Saline Salz | Invitrogen | 14185 | |
| Phenol frei, DMEM | Invitrogen | 21063 | |
| Rinderfötenserum | Aleken Biologicals | FBSS500 | |
| Penicillin / Streptomycin | Sigma | P0781-100ML | |
| Borosilikatglas mit Filament | Sutter Instrument | BF100-78 bis 10 | |
| Gloss / Gloss unverstärkten Silikon-Folien, 0,005 " | Sonstige Manufacturing Inc. | ||
| Dulbeccos Phosphate Buffered Saline | Invitrogen | 14200 | |
| 35 mm Glasboden Kulturschalen (FluoroDish) | World Precision Instruments, Inc | FD35-100 | |
| Braycote 804 Vakuumfett | Spi liefert | 05133A-AB |
Referenzen
- Friedl, P., Wolf, K., Lammerding, J. Nuclear mechanics during cell migration. Curr Opin Cell Biol. 23, 55-64 (2011).
- Mejat, A., Misteli, T. LINC complexes in health and disease. Nucleus. 1, 40-52 (2010).
- Worman, H. J., Fong, L. G., Muchir, A., Young, S. G. Laminopathies and the long strange trip from basic cell biology to therapy. J Clin Invest. 119, 1825-1836 (2009).
- Caille, N., Tardy, Y., Meister, J. J. Assessment of strain field in endothelial cells subjected to uniaxial deformation of their substrate. Ann Biomed Eng. 26, 409-416 (1998).
- Lammerding, J. Lamins A and C but not lamin B1 regulate nuclear mechanics. J Biol Chem. 281, 25768-25780 (2006).
- Lammerding, J. Abnormal nuclear shape and impaired mechanotransduction in emerin-deficient cells. J Cell Biol. 170, 781-791 (2005).
- Lammerding, J. Lamin A/C deficiency causes defective nuclear mechanics and mechanotransduction. J Clin Invest. 113, 370-378 (2004).
- Verstraeten, V. L., Ji, J. Y., Cummings, K. S., Lee, R. T., Lammerding, J. Increased mechanosensitivity and nuclear stiffness in Hutchinson-Gilford progeria cells: effects of farnesyltransferase inhibitors. Aging Cell. 7, 383-393 (2008).
- Brooks, S. V., Zerba, E., Faulkner, J. A. Injury to muscle fibres after single stretches of passive and maximally stimulated muscles in mice. J Physiol. 488, 459-469 (1995).
- Maniotis, A. J., Chen, C. S., Ingber, D. E. Demonstration of mechanical connections between integrins, cytoskeletal filaments, and nucleoplasm that stabilize nuclear structure. Proc Natl Acad Sci. 94, 849-854 (1997).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten