Method Article
Live Cell Imaging von Bacillus subtilis Und Streptococcus pneumoniae Verwendung von Automated Zeitraffer-Mikroskopie
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet eine Schritt-für-Schritt-Verfahren, um einzelne Zelle Verhalten verschiedener Bakterien in der Zeit mit Hilfe automatisierter Fluoreszenz-Zeitraffer-Mikroskopie zu überwachen. Darüber hinaus bieten wir Richtlinien, wie die Mikroskopie-Aufnahmen zu analysieren.
Zusammenfassung
In den letzten Jahren beschäftigen sich Wissenschaftler zunehmend bewusst, dass die durchschnittlichen Daten von mikrobiellen Population basierten Experimenten nicht repräsentativ für das Verhalten, Status oder Phänotyp der einzelnen Zellen. Aufgrund dieser neuen Erkenntnisse die Anzahl der einzelnen Zelle Studien steigt kontinuierlich (neuere Übersichten siehe 1,2,3). Allerdings wissen viele der einzelnen Zelle angewandten Techniken nicht zulassen, dass die Überwachung der Entwicklung und das Verhalten einer bestimmten Zelle in der Zeit (zB Durchflusszytometrie oder Standard-Mikroskopie).
Hier bieten wir Ihnen eine detaillierte Beschreibung eines Mikroskopie-Methode in mehreren neueren Studien 4, 5, 6, 7, die nach und Aufzeichnung (Fluoreszenz) einzelnen bakteriellen Zellen von Bacillus subtilis und Streptococcus pneumoniae durch Wachstum und Teilung für viele Generationen ermöglicht werden. Die resultierenden Filme können verwendet werden, um phylogenetische Stammbäume durch Rückverfolgung der Geschichte einer einzelnen Zelle innerhalb einer Population, die von einem gemeinsamen Vorfahren entstanden zu konstruieren. Das Zeitraffer-Fluoreszenzmikroskopie Methode kann nicht nur verwendet werden, um Wachstum, Teilung und Differenzierung der einzelnen Zellen zu untersuchen, sondern auch um die Wirkung von Zelle Geschichte und Abstammung auf spezifische zelluläre Verhalten zu analysieren. Darüber hinaus ist Zeitraffer-Mikroskopie ideal geeignet, um die Genexpression Dynamik und Proteinlokalisierung während der bakteriellen Zellzyklus zu untersuchen. Die Methode wird erläutert, wie die Bakterienzellen vorbereiten und bauen den Objektträger zu den Auswuchs von einzelnen Zellen in eine Mikrokolonie ermöglichen. Kurz gesagt, sind einzelne Zellen auf einem semi-feste Oberfläche aus Wachstumsmedium mit Agarose, auf dem sie wachsen und sich teilen unter dem Fluoreszenzmikroskop in einem temperierten Klimakammer ergänzt gesichtet. Die Bilder werden in bestimmten Abständen erfasst und später analysiert mit dem Open-Source-Software ImageJ.
Protokoll
1. Vorbereitung von B. subtilis Kulturen
- Impfen Zellen von -80 ° C Bestände in 10 ml Zeitraffer-Mikroskopie (TLM)-Medium (62 mM K 2 HPO 4, 44mm KH 2 PO 4, 15 mM (NH 4) 2 SO 4, 6,5 mM Natriumcitrat, 0,8 mM MgSO 4, 0,02% Casaminosäuren, 27,8 mM Glukose, 0,1 mM L-Tryptophan, wurde der pH auf 7 eingestellt mit einer KOH-Lösung), ergänzt mit Antibiotika, falls erforderlich.
- Wachsen die Zellen über Nacht in einem Schüttelkolben (30 ° C, 225 rpm).
- Am nächsten Morgen, verdünnte die Zellen 1:10 in vorgewärmten chemisch definiertem Medium (CDM) (62 mM K 2 HPO 4, 44mm KH 2 PO 4, 15 mM (NH 4) 2 SO 4, 6,5 mM Natriumcitrat, 0,8 mM MgSO 4, 2,2 Glukose, 2,1 mM L-Glutaminsäure, 6 uM L-Tryptophan, 7,5 uM MnCl 2, 0,15 x Metall-Mix (50x vorzubereiten MT-Mix Lager (ref 8), enthält: 0,2 M MgCl 2, 70 mM CaCl 2, 5 mM MnCI 2, 0,1 mM ZnCl 2, 0,2 mM Thiamin-Hydrochlorid, 2 mM HCl, 0,5 mM FeCl 3 (add letzten)) ohne Antibiotika.
- Wachsen die B. subtilis-Zellen bis Mitte exponentiellen Phase (30 ° C, 225 rpm). Normalerweise dauert dies etwa vier Stunden. Wichtig ist, bereiten die Agarose slide 1 Stunde, bevor die Zellen Mitte exponentiellen Phase zu erreichen (siehe Abschnitt 2).
- Messen Sie die Absorption der Kultur bei 600 nm (A 600) und verdünnen Sie die Zellen, um eine ungefähre A 600 von 0,035 mit CDM. Das OD ist sichergestellt, dass einzelne Zellen mit den entsprechenden Abstand auf dem Objektträger für Zeitraffer-Mikroskopie werden gesichtet.
2. Vorbereitung des Mikroskops Probe (siehe auch Abbildung 2)
Eine Stunde vor-Zellen Mitte exponentielles Wachstum zu erreichen, bereiten Sie die Objektträger wie folgt:
- Saubere Zwei mikroskopische Objektträger (z. B. Knittel Glas, 7,6 x 2,6 cm,) mit 70% Ethanol und Wasser.
- Werfen Sie einen Gen-Rahmen (ABgene; 1,7 x 2,8 cm) und entfernen Sie vorsichtig eine der Kunststoff-Folien aus dem Gen-Rahmen ohne Demontage der Kunststoffabdeckung auf der anderen Seite der Gen-Rahmen.
- Befestigen Sie den Gen-Rahmen in der Mitte einer der Glasträger, indem Sie zunächst die Erleichterung von Kontakten auf nur einer Seite, durch geführte Anlage der übrigen Gen-Rahmen mit dem Fingernagel an. Verhindern Luftblasen beim Anbringen des Gens Rahmen auf die Glasplatte.
- Verwenden Sie eine Mikrowelle auf 150 mg (1,5%) mit hoher Auflösung niedrig schmelzende Agarose (Sigma) in 10 ml CDM aufzulösen. Die Agarose muss vollständig aufgelöst werden, um minimal Hintergrund für die Zeitraffer-Mikroskopie Experimente einzuholen. Falls erforderlich, ergänzen die Agarose-CDM mit Induktor oder andere Verbindungen zu diesem Zeitpunkt
- Transfer 500 ul des warmen Agarose-CDM in der Mitte des Gens Rahmen. Achten Sie darauf, das gesamte Gebiet, einschließlich (die Grenzen) vollständig abgedeckt ist.
Die folgenden Schritte (2,6-2,10) müssen durchgeführt werden, schnell auf übermäßige Trocknung der Agarose-CDM zu verhindern. - Legen Sie die zweite Glasplatte auf dem Agarose-CDM gefüllt Gen Rahmen. Versuchen Sie, um Luftblasen zu vermeiden. Legen Sie die Sandwich-Dias in eine horizontale Position für 45 min bei 4 ° C im Kühlschrank, damit die Agarose-CDM ausreichend zu verfestigen.
- Schieben Sie den oberen Glasplatte. Verwenden einer Rasierklinge herausgeschnitten Agarstreifen von ~ 5 mm Breite innerhalb des Gens Rahmen, auf dem die Zellen gezüchtet werden. Es können maximal drei Streifen können pro Folie verwendet werden, getrennt durch ~ 4 mm Platz auf beiden Seiten. Diese Räume liefert Luft, die wesentlich ist für B. subtilis Wachstum. Wenn vier verschiedene Stämme in der Zeit verfolgt werden müssen, können zwei Streifen gemacht werden und in zwei Hälften geschnitten werden, um in vier kleine Quadrate ergeben. Entfernen Sie alle verbleibenden festen Medium.
- Entfernen Sie vorsichtig die zweite und letzte Plastikabdeckung von der Gen-Rahmen auf die klebrige Seite des Gens belichten
- Laden einzelner Zellen (aus Schritt 1,5) auf dem festen Medium ohne es zu berühren mit der Pipettenspitze. Verwenden Sie 2,5 ul für einen ganzen Streifen oder 1 ul für einen kleinen Platz. Always on top der Agarose-Pad starten und damit die Flüssigkeit gleichmäßig verteilen auf dem zugewiesenen Wachstumsbereich, indem die Folie auf und ab. Der Schlitten ist bereit, sobald die Ränder des flüssigen Wellpappe und Bewegung der Flüssigkeit werden nicht mehr sichtbar ist beim Drehen der Folie.
- Legen Sie einen sauberen Objektträger Deckgläschen (24 x 50 mm) auf dem Gen-Rahmen von einer Seite zur anderen (keine Luftblasen). Assure komplette Anlage durch Druck auf das Deckglas auf dem Gen-Rahmen mit dem Fingernagel. Wenn das Deckglas auf die Zellen, ohne dass diese lange genug trocken gelegt, neigen Zellen auf der jeweils anderen während des Experiments zu wachsen. Auch darauf achten, nicht zu lange warten, bevor das Deckglas, da die Agarose dann wird zu trocken.
- Pre-warm die Folie für 1 h bei 30 ° C. Wenn die Folie would direkt in den vorgewärmten Klimakammer gestellt werden (siehe Schritt 3.1) des Mikroskops, könnte die Temperaturschwankungen Autofokus Probleme in den ersten Stunden des Experiments führen.
3. Zeitraffer-Fluoreszenzmikroskopie (siehe auch Abbildung 3 und Movie 1)
- Pre-warmen der Klimakammer auf Zeit (in unseren Händen mindestens 2 Stunden vor Beginn des Experiments), um Autofokus Probleme nach dem Start des Experiments zu verhindern. Die benötigte Zeit ist abhängig von der Klimakammer sowie die Heizung und das Mikroskop verwendet.
- Wählen Sie das entsprechende Ziel, Filter und dichroitische Spiegel nach Ihren Versuchsaufbau. Für lange Experimente sicher, dass ein UV-Filter zwischen Lichtquelle und die Probe gestellt zu machen. Auch, wenn möglich, Block einige der Anregungslicht mit Neutralfilter, um die Exposition zu minimieren.
Die folgende Ausrüstung (bereitgestellt von DeltaVision, UK) wurde für die Zeitraffer-Mikroskopie Experimente in de Jong et al verwendet 2010 5:. IX71 Mikroskop (Olympus), CoolSNAP HQ2 Kamera (Princeton Instruments), 300W Xenon-Lichtquelle, 60x heller Feld Ziel (1,25 NA), GFP Filtersatz (Chroma, Anregung bei 470/40 nm, Emission 525/50 nm), mCherry Filtersatz (Chroma, Anregung bei 572/35 nm, Emission 632/60 nm). Autofokus wurde mit Durchlichtstativ Licht und mit der Autofokus-Routine in Deltavision ist Softworx Software. Es wird darauf hingewiesen, dass es inzwischen eine Reihe von anderen Autofokus-Systeme, die auch geeignet sind, wie die Zeiss Definite Focus, die Nikon Perfect Focus System und die Leica Adaptive Focus Control werden.
- Programmieren Sie Ihr Experiment nach Ihren Versuchsaufbau. Es ist ratsam, die Menge an Licht für spezifische Konstrukte erforderlich, ebenso wie der Autofokus-Einstellungen für andere Zeitraffer-Mikroskop oder Bakterien vor dem eigentlichen Experiment zu bestimmen. Kürzere Belichtungszeiten und weniger Menge an Anregungslicht minimiert Bleichen und Phototoxizität. Verwenden Sie Durchlichtstativ Licht für den Autofokus-Routine.
Die folgenden Einstellungen wurden für die Zeitraffer-Mikroskopie Experimente in de Jong et al verwendet 2010 5:. Snapshots für Filme wurden in Intervallen von 8 oder 12 Minuten mit 10% APLLC Weiß übernommen LED-Licht und 0,05 s Belichtungszeit für Hellfeld Bilder, 10% Xenon-Licht und 0,5 s Belichtung für GFP-Erkennung, und 32% Xenon-Licht und 0,8 s Belichtung für mCherry Erfassen von. Raw-Daten gespeichert wurden mit Softworx 3.6.0 (Applied presicion). Der Autofokus wurde für 0,06 um Schritte und eine Gesamtreichweite von 1,2 um programmiert.
- Legen Sie die vorbereiteten Objektträger (Abschnitt 2) in den vorgewärmten Klimakammer des Mikroskops und überwachen das Auswachsen einzelner Zellen in eine Mikrokolonie Monoschicht bei 30 ° C.
Spezielle Tipps:
- Wählen Sie einzelne Zellen, die in der Mitte des Agar-Pad befinden. Die Kanten der Agar-Pad austrocknen leichter. Bewahren Sie die X, Y, Z-Position mit dem Mikroskop-Software.
- Große Bewegungen der Bühne in der X-, Y-und Z-Richtung könnte stören die Agarose und damit behindern Identifikation von Zellen durch die Autofokus-Routine. Im Allgemeinen zu minimieren X, Y, Z-Bewegung, wissen wir nicht wählen mehr als 10 Positionen pro Experiment, auch wenn ein Bild enthält mehrere Stämme.
- Nachdem Sie die erste Zelle, nur passen Sie die Z-Fokus mit der Software. Von diesem Punkt nicht verändern den Fokus manuell am Mikroskop Körper mit dem "Z-Knopf", es sei denn diese digital codiert ist. Stellen Sie sicher, dass nach jeder Autofokus Routine der neuen X, Y, Z-Position von der Software gespeichert ist.
- Prüfen Sie, ob der Autofokus-Einstellungen geeignet für das Experiment sind, bevor der Flucht. Die Verwendung von Phasenkontrast-Mikroskopie können zur Verbesserung der Autofokus-Routine im Vergleich zur Verwendung Hellfeld oder DIC-Mikroskopie, dank verbesserter Kontrast. Allerdings macht die Phase Ring in Phasen-Kontrast-Ziele sie weniger empfindlich (ca. 10%) bei der Erhebung Fluoreszenzlicht. Also für schwache fluoreszierende Proben, ist ein Ziel, ohne Phase Ring besser geeignet.
- Prüfen Sie, ob die ausgewählten Zellen noch im Fokus jede halbe Stunde, bis das Experiment stabil läuft. Wenn die Zellen zu diesem Zeitpunkt nicht scharf eingestellt sind, manuell einstellen. Durch Temperaturschwankungen, sowie schlecht getrockneten Proben, kann dies notwendig sein, während der ersten Stunden. Darüber hinaus dank verbesserter Kontrast, arbeitet der Autofokus besser, wenn mehr Zellen in das Blickfeld sind.
- Nach dem Experiment beendet ist, trennen Sie die verschiedenen Kanäle des Films und sichere sie als separate Dateien (zB Phasenkontrast, GFP, mCherry) bei Bedarf (bestimmte Erwerb Pakete aller Kanäle in einem einzigen gestapelt Datei findet). Für die Veröffentlichung kann Bilder von 2D Dekonvolution, die vor allem ist uns verstärkt werdeneful für Proteinlokalisierung Studien. Deconvolve die Bilder mit Ihrem Mikroskop-Software oder mit einem kommerziellen Paket wie Huygens ( www.svi.nl ).
- Analysieren Sie die Daten mit ImageJ ( http://rsbweb.nih.gov/ij/ ) (Verwendung der rohen, unbearbeiteten Bilddateien für diese) und Microsoft Excel oder Sigma Plot. Stacks können zum Beispiel als ". Avi" Filmdatei in ImageJ gespeichert werden. Eine detaillierte Beschreibung, wie Fluoreszenz einzelner Zellen können in Zeit gemessen werden wird unten gegeben.
4. Die Datenanalyse der Promotoraktivität Dynamik mit ImageJ
Wir nehmen zur Kenntnis, dass andere gute Software-Pakete verfügbar sind, die bei der Analyse Zeitraffer-Mikroskopie-Aufnahmen wie BHV Software 9, 4, Schnitzcell 10, PSICIC 11 und Microbe-Tracker 12, spezialisiert, aber hier sind wir auf der frei verfügbaren ImageJ Paket konzentrieren.
- Herunterladen ImageJ ( http://rsbweb.nih.gov/ij/ ) und (falls nötig) das richtige Plugin, um Ihre (gestapelt) Datei zu öffnen. Zum Beispiel, Filme aufgenommen mit einem deltavision Mikroskop kann nur in ImageJ mit dem deltavision Opener Plugin geöffnet werden. Kopieren Sie die DV-Plugin in der ImageJ Plugin-Ordner und starten Sie das Programm. Ändern Sie die Speicherkapazität in edit / option / Speicher & Themen bis 1250. Dies ermöglicht das Arbeiten mit größeren Dateien wie die von Zeitraffer-Filme erhalten.
- Zur Beurteilung der Zell-Geschichte aus einer einzigen Zelle, öffnen Sie die ursprüngliche Phasenkontrast Film von einem Mikrokolonie und blättern Sie zum letzten Frame von Interesse in den Film. Wählen Sie den "segmentierte Linien" Auswahl-Taste in das Menü.
- Zeichnen Sie eine Linie in den Hintergrund, und drücken Sie "Strg" + "T". Dies öffnet den regions of interest (ROI)-Manager. (Die entsprechende Hintergrundfluoreszenz Wert kann verwendet werden, um manuell subtrahieren Hintergrund werden -.. Siehe unten Alternativ können Sie die Hintergrund-Subtraktion Routine derzeit in ImageJ) Auch ziehen Sie eine Linie in der Zelle von Interesse und fügen Sie den ROI auf die ROI-Manager. Da wir untersuchen Promotor-GFP-Fusionen sind in dieser Fallstudie und der GFP ist im gesamten Zytoplasma diffus, sollte die gesamte Zelle ähnliche Fluoreszenz-Werte für jedes einzelne Pixel über die Länge der Zelle haben. Scrollen von einem Bild in der Zeit zurück und wählen einen neuen ROI in der gleichen Zelle von Interesse. Speichern dieser dritten ROI und das Verfahren fortsetzen, bis die entsprechenden ROI in den ersten Frame des Films gespeichert wurde.
Seien Sie sich bewusst, dass die Fluoreszenz der Tochterzellen kann stark variieren nach der Zellteilung. Verwenden Sie entweder eine Membran Farbstoff, der gemeinsam mit der Fluoreszenz-Proteine von den Zellen (wie z. B. die Kombination der roten Membran Farbstoff FM 5-95 ® (Invitrogen) und GFP) produzierten Septenbildung visualisieren angewendet werden. In diesem Fall wird die entsprechende Fluoreszenz-Kanal und nicht der Phasenkontrast-Film sollte verwendet werden, um Zellen mit der Zeit zu folgen. Alternativ kann man auf der sicheren Seite, indem Sie ROI in nur eine Hälfte einer Zelle zu bleiben. - Innerhalb der ROI-Manager, klicken Sie auf "Speichern". Wenn das Ende der Datei ist ". Roi", dann roi ist in der Liste ausgewählt, und nur diese wird gespeichert. Wenn das Ende der Datei ist ". Zip", dann das ganze Set wird gerettet (erforderlich).
- Schließen Sie die Phasenkontrast-Film und öffnen Sie die ursprüngliche Fluoreszenz Film (zB GFP). Klicken Sie auf "Alle zeigen" und "Maßnahme" in die ROI-Manager. Ein neues Fenster wird geöffnet (Ergebnisse). Kopieren Sie die Ergebnisse in einem Excel-Sheet und subtrahieren die mittlere Fluoreszenz für jede einzelne Zelle aus der Hintergrund-Fluoreszenz des Mediums. Der daraus resultierende Fluoreszenz gegen die Zeit, um die Promotor-Aktivität der Zelle von Interesse in der Zeit offenbaren geplottet werden.
- Alternativ kann die Fluoreszenz aller Zellen eines Mikrokolonie zu einer bestimmten Zeit-Punkt analysiert werden. Um dies zu tun wählen und speichern ROI ist für den Hintergrund und jede einzelne Zelle in einem Rahmen, wie oben beschrieben, kopieren Sie die Fluoreszenz-Werte nach Excel und produzieren Histogramme mit dem "Histogramm"-Funktion im Menü "Tool".
- Um Bilder von Einzelbildern für die Veröffentlichung, wählen Sie den Rahmen des Interesses, wählen Sie "Bild" - "Kopie des Bildes" und ändern Sie die Art des Bildes auf "RGB" oder "8-Bit" durch Auswahl von "Bild" - "Typ" - "RGB-Farbe" oder "8-Bit". Speichern Sie die duplizierten Rahmen als ". Tiff". Die RGB/8-bit Bilder können durch herkömmliche Zeichnung Programmen wie CorelDraw oder Adobe Illustrator geöffnet werden. Falls erforderlich, kann die Bilder angepasst mit "Bild" werden - "anpassen" - "Helligkeit / Kontrast" oder "Bild" - "anpassen" - "Fenster / Ebene" in ImageJ.
5. Producing Filme für die Veröffentlichung mit ImageJ
- Öffnen Sie die ursprüngliche Phasenkontrast und die entsprechende Fluoreszenz Film (e) in ImageJ wie oben beschrieben. Wählen Sie die rechteckige Auswahl-Taste (1 st auf der linken Seite) und zeichnen Sie ein Rechteck ROI in den ersten Frame in einer Weise, dass die Entwicklungsländer Mikrokolonie durch die ROI throu ist beigefügtghout den ganzen Film. Wählen Sie "Bild" - "Ernte" und sichere diese kleinere Version des Films unter einem neuen Namen. Wählen Sie den gleichen ROI über die ROI-Manager in der Fluoreszenz-Film und gehen wie zuvor.
- So kombinieren Sie die Filme entweder horizontal oder vertikal, wählen Sie "plugin" - "Stack Kombinierer". Wenn gewünscht, kann eine Zeitstempler via "plugin" hinzugefügt - "Zeitstempler". Speichern Sie die kombinierte Stapel einst als ". Dv" oder ". Tiff" und einmal ". Avi" oder QuickTime-Film.
Alternative Protocol Anpassungen für Streptococcus pneumoniae (Abb. 4 und Movie 2):
6. Vorbereitung von S. pneumoniae Kulturen
- Wachsen S. pneumoniae-Zellen (gekapselt Stammes D39 13 oder unverkapseltem Stamm R6 14 als stehende Kulturen in C + Y Medium 15 bei 37 ° C bis zu einer OD bei 600nm A von ungefähr 0,4 erreicht ist. Zentrifugieren Sie die Zellen für 2 min bei 14000 rpm und resuspendieren Zellpellet in einem Volumen von frischem C + Y-Medium mit 14,5% Glycerin (v / v), die in einem A 600nm von genau 0,4 ergeben würde. Aliquot der Zellen und lagern Sie sie bei -80 ° C für eine spätere Verwendung.
- Für Zeitraffer-Mikroskopie wird ein aliquoter von zuvor kultivierten S. pneumoniae-Zellen. Impfen 4 ml frisches C + Y Medium 1:100 mit Zellen aus dem -80 ° C Aliquot. Wachsen die Zellen bis Mitte exponentiellen Phase zu einer OD von A 600nm von 0,1 bis 0,2. Normalerweise dauert dies etwa 2 Stunden bei der Verwendung von Zellen von -80 ° C Aliquots.
7. Vorbereitung des Mikroskops Probe
- Bereiten Sie einen Objektträger wie oben beschrieben für B. subtilis, aber stellen Sie sicher, dass die Agarose-Komplex C + Y Medium enthält. Da S. pneumoniae ist ein mikroaerophilen, sollte die Lufteinschlüsse zwischen Agarose Streifen kleiner sein als für B. subtilis (~ 1 mm Platz auf beiden Seiten).
- Messen Sie die Absorption der Kultur bei 600 nm (A 600), verdünnte der exponentiell wachsenden S. pneumoniae-Zellen, um eine ungefähre A 600 von 0,05 unter Verwendung von C + Y Medium und nutzen diese Verdünnung auf die Agarose Rutsche laden.
8. Zeitraffer-Phasenkontrast-Mikroskopie
Passen Sie die Einstellungen des Mikroskops für S. pneumoniae: Verwendung der Phasenkontrast-Mikroskopie seit S. pneumoniae ist schwierig zu identifizieren mit Hellfeld-Mikroskopie. Fahren Sie mit der Protokoll wie für B beschrieben subtilis (folgen Sie den Schritten von 2,9 bis 3,7). S. pneumoniae-Zellen können entweder 30 angewachsen ° C oder 37 ° C (sie wachsen schneller bei 37 ° C).
9. Repräsentative Ergebnisse:
Die Zeitraffer-Fluoreszenz-Experiment wurde erfolgreich durchgeführt, wenn die Bakterien wuchsen in eine Mikrokolonie Monoschicht, die vollständig innerhalb des Sichtfeldes befindet sich am Ende des Experiments befindet (siehe Abbildung 5A-C). Wenn Zellen wuchsen auf der jeweils anderen, ist es nicht nur unmöglich zurückzuverfolgen ihre Geschichte genau, sondern auch die Fluoreszenz-Ebenen überlappenden Zellen nicht korrekt gemessen werden. Zellen neigen dazu, auf der jeweils anderen zu wachsen, wenn die gefleckte Zellen wurden nicht ausreichend (Stufe 2,9) getrocknet oder, wenn das Medium Zusammensetzung angepasst werden muss, zu einem langsameren Wachstum zu erzielen. Wenn ein Mikrokolonie entstand aus der Sicht, dann ist die Verteilung der Fluoreszenz-Signale innerhalb einer Kolonie kann nicht bestimmt werden. Ursachen für "Mikrokolonie Bewegung" können unzureichende Trocknung gesichtet Zellen (Schritt 2,9), oder wenn die Software nicht so programmiert, dass die Mikrokolonie während der Entwicklung zu verfolgen. Darüber hinaus ist es wichtig, dass lokale Flecken erhöhte Fluoreszenz nicht in das Medium nachweisbar, da dies verschleiert die Fluoreszenz-Signale aus den Zellen (siehe Abbildung 5D-F). Background Probleme können von Medien-Verbindungen, Luftblasen oder ungelöste Agarose Klumpen entstehen. Zur Visualisierung dieser zeigen wir in Abb.. 5F Hintergrund Signale dieser bestimmten Folie, wenn das Bild aufgenommen wurde mittels Anregung / Emission Filter für rot fluoreszierende Farbstoffe. Wie man sieht, sind hell autofluoreszierenden Flecken vorhanden, könnten bildgebende behindern. Zur Vermeidung solcher Stellen sicher, dass die Agarose vollständig gelöst ist und es gibt keine Luftblasen bei der Platzierung das Deckglas auf dem Objektträger.
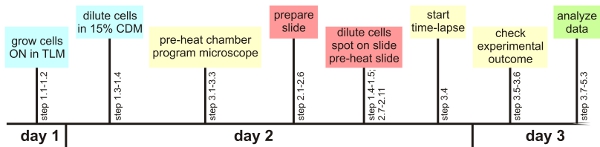
Abbildung 1: Experimentelle Überblick

Abbildung 2: Vorbereitung des Mikroskops Probe
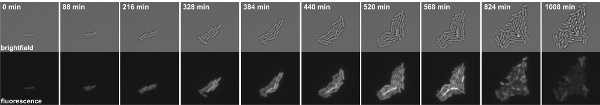
Abbildung 3: Zeitraffer-Fluoreszenzmikroskopie von B. subtilis-Zellen, die ein P kinB-gfp Fusion. Snapshots werden von Movie 1 übernommen. Top-Panels: Hellfeld, Bodenplatten: GFP-Kanal.
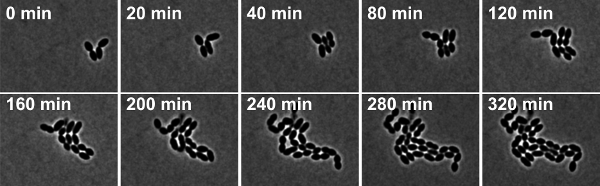
FeigeAbbildung 4: Zeitraffer-Phasenkontrast-Mikroskopie von S. pneumoniae Wildtyp-Stamm R6. Snapshots werden von Movie 2 übernommen.
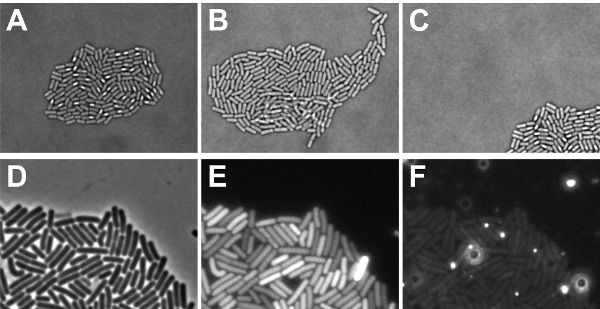
Abbildung 5: Illustration der möglichen (Time-Lapse) Mikroskopie Ergebnisse. AC zeigt Faktoren, die für Daten mit Durchlichtstativ Licht-Einstellungen erhalten berücksichtigt werden müssen. (A) Hellfeld-Aufnahme eines Mikrokolonie Monoschicht (positives Ergebnis) von sporulierenden B. subtilis-Zellen (B) Hellfeldaufnahme eines B. subtilis Mikrokolonie, in dem einige Zellen auf der jeweils anderen wuchs (negatives Ergebnis) (C) Hellfeldaufnahme einer sporulierenden B. subtilis Mikrokolonie, dass aus dem Bereich der Scharfeinstellung (negatives Ergebnis) wuchs. DF zeigen Faktoren, die für Daten mit episkopisch Licht-Einstellungen erhalten berücksichtigt werden müssen (D) Phasenkontrast-Bild von B. subtilis-Zellen in der exponentiellen Phase dargestellt zu visualisieren, wo die Fluoreszenz-Signale in E und F von (E) GFP-Signale der Zellen in D. Beachten Sie, dass der Hintergrund-Signale ähnlich in jedem Pixel (positives Ergebnis) angezeigt stammen. Beachten Sie auch, dass die Belichtungszeit kann zu viel sein, da eine Zelle zeigt einen gesättigten Signal (negatives Ergebnis) (F)-Signale über den roten Kanal der Zellen in D. Hinweis angezeigt, dass der Hintergrund Bereichen mit erhöhter roter Fluoreszenz Ebenen (enthält erhaltenen negativen Outcome).
Movie 1. Zeitraffer-Fluoreszenzmikroskopie von B. subtilis-Zellen, die ein P kinB-gfp Fusion. Snapshots sind in 8 Minuten-Intervallen entnommen. Links: Hellfeld, rechts: GFP-Kanal. Klicken Sie hier, um den Film zu sehen.
Movie 2. Zeitraffer-Phasenkontrast-Mikroskopie von S. pneumoniae Wildtyp-Stamm R6. Snapshots sind in 10 min Intervallen genommen. Klicken Sie hier, um den Film zu sehen.
Diskussion
Im Gegensatz zu vielen anderen einzelnen Zelle Techniken kann der Zeitraffer-Fluoreszenzmikroskopie hier beschriebene Methode verwendet, um die Geschichte einer bestimmten Zelle mit Hinweis auf seine Vorfahren, sein Verhalten und Division Veranstaltungen folgen werden. In Kombination mit fluoreszenzmarkierten Ziel Promotoren oder Proteine, können umschriebene Entwicklungsstörung Stoffwechselwegaktivierung in Zeit-und Protein-Lokalisierung sowie Protein Dynamik kann während bakterielle Entwicklung beobachtet werden folgen.
Wie bereits erwähnt, können Studien konzentriert sich auf verschiedene Bakterienarten durch die Anpassung der Wachstumsbedingungen nach den Anforderungen für ein bestimmtes Bakterium durchgeführt werden. Die einzigen Einschränkungen stießen wir an die Wachstumsbedingungen und Stichprobengröße zusammen. Aufgrund einer geschlossenen Umgebung können mittlere Bedingungen nicht während des Experiments geändert werden. Darüber hinaus kann ein Maximum von vier Stämme pro Experiment effizient überwacht werden.
Angesichts einige kritische Schritte beschrieben, die einzelne Zelle Analyseverfahren kann hier leicht unter Verwendung eines automatisierten Mikroskop werden. Im Folgenden wird ein Überblick über diese wichtigen Schritte gegeben werden. Detaillierte Informationen finden Sie im Haupttext zu finden Allgemeine Vorbereitung:. Es ist ratsam, den Autofokus-Einstellungen für ein bestimmtes Bakterium vor dem Experiment erforderlich zu überprüfen. Ebenso sollte in etwa die optimalen Einstellungen für die Visualisierung von Fluoreszenz im Voraus bestimmt werden, wenn möglich. Darüber hinaus folgt eine vorbereitete time-line hilft, alle wesentlichen bereit, in der Zeit verwenden (Vorwärmen des Mikroskops Kammer, die Programmierung der Einstellungen des Mikroskops, die Vorbereitung der Folie 1 Stunde, bevor die Zellen in der gewünschten Wachstumsphase befinden, siehe Abbildung 1) haben . Das Wachstum der B. subtilis in TLM und CDM: TLM und CDM sind chemisch Hunger Medien definiert, in dem B. subtilis wächst nur langsam. Der Zeitraum, in dem die Zellen in den Medien angebaut werden müssen möglicherweise verlängert abhängig von der spezifischen Belastung werden. Das langsame Wachstum verhindert, dass Zellen häufen sich gegenseitig Vorbereitung des Mikroskops Probe:. Luftblasen zwischen den Gen-Rahmen, den Objektträger und dem Deckglas müssen verhindert werden, um umfangreiche Trocknen der Agarose-basierte Medium zu verhindern. Das gleiche gilt für das Medium / Deckglas-Schnittstelle. Es ist unverzichtbar, dass die Zellen genügend trocken, zum Schwimmen und / oder mehrschichtigen Wachstum zu verhindern Zeitraffer-Fluoreszenzmikroskopie:. Vorwärmen der Folie sowie die Klimakammer ist entscheidend für die großen Autofokus Probleme zu vermeiden. Die Zellen sind in der Mitte einer Agar-Pad gewählt werden, da diese die höchste Chance, in Feld und Fokus während des Experiments (vorausgesetzt, die Probe wurde gut genug getrocknet) zu bleiben. Es können maximal 10 Stellen pro Experimente noch einwandfrei funktioniert. Nachdem Sie die erste Zelle des Interesses nur die Software, um den Fokus einzustellen (siehe Text für Details). Prüfen Sie, ob die Zellen noch in Fokus in den ersten Stunden des Versuches in 30 min Abständen Analyse:. Es ist wichtig, vor der erweiterten Analyse-Verfahren zu überprüfen, ob der Hintergrund des Mediums ähnliche Werte hat in der Fluoreszenz-Kanäle. Kleine Staubpartikel, mittel-Komponenten, verschmutzte Linsen oder winzige Agarose Klumpen können lokal erhöhte Fluoreszenz beitragen, dass der Film schwer oder unmöglich zu Trouble Shooting zu analysieren:. Wenn die Zellen auf der jeweils anderen zu wachsen, könnte dies entweder darauf hin, dass das Deckglas wurde angebracht zu früh oder dass das Medium nicht für das Wachstum von Mikrokolonien Monoschichten geeignet. Wenn Zellen von Interesse kontinuierlich vorzeitig sterben, während andere Zellen auf dem Objektträger glücklich zu teilen, möchten Sie vielleicht zu prüfen, ob Sie die UV-Filter setzen in Position. Es könnte auch dazu beitragen, die Belichtungszeit oder die Lichtintensität während der langen Experimenten zu verringern.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Arbeit in der Gruppe der JWV wird von einem EU Marie-Curie Reintegration Fellowship, ein Sysmo2 Grant (NWO-ALW/ERASysBio), eine Horizon-Stipendium (ZonMw) und durch eine VENI Gemeinschaft (NWO-ALW) unterstützt. Die Gruppe der OPK wird von mehreren Standard-Zuschüsse (NWO), ein SYSMO1 (IGdeJ) und SYSMO2 gewähren, eine ESF Eurocores SynBio gewähren (SynMod) und durch die Kluyver Center for Genomics of Industrial Fermentation und die Top-Instituts für Lebensmittel und Ernährung unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare (optional) |
|---|---|---|---|
| Gene Rahmen | ABgene | AB-0578 | 1,7 x 2,8 cm |
| hochauflösende niedrig schmelzenden Agarose | Sigma | A4718 | |
| großen Deckglas | mehrere | 24 x 50 mm | |
| wenn gewünscht, Membran-Farbstoff, z. B. FM 5-95 | Invitrogen | T23360 | andere Membran Farbstoffe sind ebenfalls erhältlich: http://probes.invitrogen.com/media/pis/mp34653.pdf |
| Zeitraffer-Mikroskop mit Klimakammer | mehrere | Details für unser Gerät in den entsprechenden Abschnitten |
Referenzen
- Veening, J. W., Smits, W. K., Kuipers, O. P. Bistability, epigenetics, and bet-hedging in bacteria. Annu. Rev Microbiol. 62, 193-193 (2008).
- Dubnau, D., Losick, R. Bistability in bacteria. Mol Microbiol. 61, 564-564 (2006).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nat. Rev. Microbiol. 7 (5), 383-383 (2009).
- Veening, J. W. Bet-hedging and epigenetic inheritance in bacterial cell development. Proc. Natl. Acad. Sci. U. S. A. 105 (11), 4393-4393 (2008).
- de Jong, I. G., Veening, J. W., Kuipers, O. P. Heterochronic phosphorelay gene expression as a source of heterogeneity in Bacillus subtilis spore formation. J. Bacteriol. 192 (8), 2053-2053 (2010).
- Veening, J. W., Murray, H., Errington, J. A mechanism for cell cycle regulation of sporulation initiation in Bacillus subtilis. Genes Dev. 23 (16), 1959-1959 (2009).
- Eberhardt, A. Cellular localization of choline-utilization proteins in Streptococcus pneumoniae using novel fluorescent reporter systems. Mol Microbiol. 74 (2), 395-395 (2009).
- Vasantha, N., Freese, E. Enzyme changes during Bacillus subtilis sporulation caused by deprivation of guanine nucleotides. J Bacteriol. 144 (3), 1119-1119 (1980).
- Stewart, E. J. Aging and death in an organism that reproduces by morphologically symmetric division. PLoS. Biol. 3, 45-45 (2005).
- Rosenfeld, N. Gene regulation at the single-cell level. Science. 307 (5717), 1962-1962 (2005).
- Guberman, J. M. PSICIC: noise and asymmetry in bacterial division revealed by computational image analysis at sub-pixel resolution. PLoS. Comput. Biol. 4 (11), 1000233-1000233 (2008).
- Montero, L. lopis P. Spatial organization of the flow of genetic information in bacteria. Nature. 466, 77-77 (2010).
- Avery, O. T., Macleod, C. M., McCarty, M. Studies on the chemical nature of the substance inducing transformation of pneumococcal types: Induction of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III. J Exp. Med. 79, 137-137 (1944).
- Hoskins, Genome of the bacterium Streptococcus pneumoniae strain R6. J Bacteriol. 183, 5709-5709 (2001).
- Martin, B. The recA gene of Streptococcus pneumoniae is part of a competence-induced operon and controls lysogenic induction. Mol Microbiol. 15, 367-367 (1995).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten